Method Article
Screening dei frammenti basato su NMR in un campione minimo ma in modalità di automazione massima
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Lo screening basato su frammenti mediante NMR è un metodo robusto per identificare rapidamente i leganti di piccole molecole alle biomacromolecole (DNA, RNA o proteine). Vengono presentati protocolli che descrivono la preparazione dei campioni basata sull'automazione, gli esperimenti NMR e le condizioni di acquisizione e i flussi di lavoro di analisi. La tecnica consente lo sfruttamento ottimale di entrambi i nuclei NMR-attivi 1H e 19F per il rilevamento.
Abstract
Lo screening basato su frammenti (FBS) è un concetto ben convalidato e accettato all'interno del processo di scoperta di farmaci sia nel mondo accademico che industriale. Il più grande vantaggio dello screening dei frammenti basato su NMR è la sua capacità non solo di rilevare leganti oltre 7-8 ordini di grandezza di affinità, ma anche di monitorare la purezza e la qualità chimica dei frammenti e quindi di produrre risultati di alta qualità e falsi positivi o falsi negativi minimi. Un prerequisito all'interno dell'FBS è quello di eseguire il controllo di qualità iniziale e periodico della libreria di frammenti, determinando la solubilità e l'integrità chimica dei frammenti nei buffer pertinenti e stabilendo più librerie per coprire diversi scaffold per adattarsi a varie classi target di macromolecole (proteine / RNA / DNA). Inoltre, è richiesta un'ampia ottimizzazione del protocollo di screening basato su NMR rispetto alle quantità del campione, alla velocità di acquisizione e all'analisi a livello di costrutto biologico / spazio dei frammenti, nello spazio condizione (buffer, additivi, ioni, pH e temperatura) e nello spazio dei ligando (analoghi del ligando, concentrazione del ligando). Almeno nel mondo accademico, questi sforzi di screening sono stati finora intrapresi manualmente in modo molto limitato, portando a una disponibilità limitata di infrastrutture di screening non solo nel processo di sviluppo dei farmaci, ma anche nel contesto dello sviluppo della sonda chimica. Al fine di soddisfare i requisiti in modo economico, vengono presentati flussi di lavoro avanzati. Sfruttano l'hardware avanzato più recente, con il quale la raccolta di campioni liquidi può essere riempita in modo controllato a temperatura nelle provette NMR in modo automatizzato. 1Gli spettri basati sul ligando NMR H/19F vengono quindi raccolti a una data temperatura. Lo scambiatore di campioni ad alta produttività (scambiatore di campioni HT) può gestire più di 500 campioni in blocchi a temperatura controllata. Questo, insieme a strumenti software avanzati, accelera l'acquisizione e l'analisi dei dati. Inoltre, l'applicazione di routine di screening su campioni di proteine e RNA sono descritti per rendere consapevoli dei protocolli stabiliti per un'ampia base di utenti nella ricerca biomacromolecolare.
Introduzione
Lo screening basato su frammenti è ora un metodo comunemente usato per identificare molecole piuttosto semplici e a basso peso molecolare (MW <250 Da) che mostrano un debole legame con bersagli macromolecolari tra cui proteine, DNA e RNA. I colpi iniziali dagli schermi primari servono come base per condurre uno screening secondario di analoghi più grandi disponibili in commercio degli hit e quindi per utilizzare strategie di crescita o collegamento dei frammenti basate sulla chimica. Per una piattaforma di scoperta di farmaci basata su frammenti (FBDD) di successo, in generale, è necessario un solido metodo biofisico per rilevare e caratterizzare i colpi deboli, una libreria di frammenti, un bersaglio biomolecolare e una strategia per la chimica di follow-up. Quattro metodi biofisici comunemente applicati all'interno delle campagne di scoperta di farmaci sono saggi di spostamento termico, risonanza plasmonica di superficie (SPR), cristallografia e spettroscopia di risonanza magnetica nucleare (NMR).
La spettroscopia NMR ha mostrato vari ruoli all'interno delle diverse fasi dell'FBDD. Oltre a garantire la purezza chimica e la solubilità dei frammenti in una libreria di frammenti disciolti in un sistema tampone ottimizzato, gli esperimenti NMR osservati con ligando possono rilevare il legame del frammento a un bersaglio con bassa affinità e gli esperimenti NMR osservati possono delineare l'epitopo di legame del frammento, consentendo così studi dettagliati sulla relazione struttura-attività. All'interno della mappatura degli epitopi, i cambiamenti di spostamento chimico basati sulla NMR non solo possono identificare i siti di legame ortosterici, ma anche i siti allosterici che potrebbero essere criptici e accessibili solo nei cosiddetti stati conformazionali eccitati del bersaglio biomolecolare. Se il bersaglio biomolecolare lega già un ligando endogeno, i colpi di frammento identificati possono essere facilmente classificati come allosterici o ortosterici eseguendo esperimenti di competizione basati su NMR. Determinare la costante di dissociazione (KD) dell'interazione ligando-bersaglio è un aspetto importante nel processo FBDD. Le titolazioni di spostamento chimico basate su NMR, sia ligando che bersaglio osservate, possono essere facilmente eseguite per determinare il KD. Uno dei principali vantaggi della NMR è che gli studi di interazione vengono eseguiti in soluzione e in condizioni fisiologiche. Pertanto, tutti gli stati conformazionali per l'analisi dell'interazione ligando/frammento con il suo bersaglio possono essere sondati. Inoltre, gli approcci basati sulla NMR non sono solo limitati allo screening di proteine solubili ben ripiegate, ma vengono anche applicati per adattarsi a uno spazio target più ampio tra cui DNA, RNA, proteine legate alla membrana e intrinsecamente disordinate1.
Le librerie di frammenti sono una parte indispensabile del processo FBDD. In generale, i frammenti agiscono come precursori iniziali che alla fine diventano parte (sottostruttura) del nuovo inibitore sviluppato per un bersaglio biologico. Diversi farmaci (Venetoclax2, Vemurafenib3, Erdafitinib4, Pexidartnib5) sono stati segnalati come frammenti e sono ora utilizzati con successo nelle cliniche. Tipicamente, i frammenti sono molecole organiche a basso peso molecolare (<250 Da) con un'elevata solubilità e stabilità acquosa. Una libreria di frammenti accuratamente realizzata contenente in genere poche centinaia di frammenti, può già promettere un'esplorazione efficiente dello spazio chimico. La composizione generale delle librerie di frammenti si è evoluta nel tempo e il più delle volte sono state derivate sezionando farmaci noti in frammenti più piccoli o progettati computazionalmente. Queste diverse librerie di frammenti contengono principalmente aromatici piatti o eteroatomi e aderiscono alla regola di Lipinski di 5 6, o all'attuale regola di tendenza commerciale di 3 7, ma evitano i gruppi reattivi. Alcune librerie di frammenti sono state anche derivate o composte da metaboliti altamente solubili, prodotti naturali e/o loro derivati8. Una sfida generale posta dalla maggior parte delle librerie di frammenti è la facilità della chimica a valle.
Il Center for Biomolecular Magnetic Resonance (BMRZ) della Goethe-University di Francoforte, è partner di iNEXT-Discovery (Infrastructure for NMR, EM and X-rays for Translational research-Discovery), un consorzio per le infrastrutture di ricerca strutturale per tutti i ricercatori europei di tutti i campi della ricerca biochimica e biomedica. Nell'ambito della precedente iniziativa di iNEXT che si è conclusa nel 2019, è stata realizzata una libreria di frammenti composta da 768 frammenti con l'obiettivo di "minimi frammenti e massima diversità" che copre un ampio spazio chimico. Inoltre, a differenza di qualsiasi altra libreria di frammenti, anche la libreria di frammenti iNEXT è stata progettata sulla base del concetto di "frammenti in bilico" con l'obiettivo di facilitare la sintesi a valle di ligandi complessi e ad alta affinità e d'ora in poi nota come libreria interna (Diamond, Structural Genomic Consortium e iNEXT).
La creazione di FBDD tramite NMR richiede manodopera, conoscenza e strumentazione. Al BMRZ sono stati sviluppati flussi di lavoro ottimizzati per supportare l'assistenza tecnica allo screening dei frammenti mediante NMR. Questi includono il controllo di qualità e la valutazione della solubilità della libreria di frammenti 9, l'ottimizzazione del buffer per i bersagli scelti, lo screening basato su 1H o 19F- osservato 1D-ligando, esperimenti di competizione per differenziare tra legame ortosterico e allosterico, esperimenti NMR osservati su target 2D per la mappatura degli epitopi e per caratterizzare l'interazione con il set secondario di derivati dei colpi iniziali del frammento. BMRZ ha stabilito routine automatizzate per l'analisi, come discusso anche in precedenza nella letteratura 10,11, delle interazioni piccole molecole-proteine e dispone di tutte le infrastrutture automatizzate necessarie per lo screening dei frammenti basato su NMR. Ha implementato la differenza di trasferimento di saturazione NMR (STD-NMR), water-ligando osservato tramite spettroscopia a gradiente (waterLOGSY) e esperimenti di rilassamento basati su Carr-Purcell-Meiboom-Gill (basati su CPMG) per identificare frammenti all'interno di una vasta gamma di regimi di affinità, nonché strumentazione NMR automatizzata all'avanguardia e software per la scoperta di farmaci. Mentre lo screening dei frammenti basato su NMR è ben consolidato per le proteine, questo approccio è meno comunemente usato per trovare nuovi ligandi che interagiscono con RNA e DNA. BMRZ ha stabilito prove di concetto per nuovi protocolli che consentono l'identificazione di piccole interazioni molecola-RNA/DNA. Nelle sezioni seguenti di questo contributo, l'applicazione di routine di screening su campioni di proteine e RNA è riportata per rendere consapevoli dei protocolli stabiliti per un'ampia base di utenti nella ricerca biomacromolecolare.
Protocollo
1. Libreria di frammenti
- Libreria di frammenti interna
NOTA: Nell'ambito di una delle attività di ricerca congiunte di iNEXT, è stata sviluppata una libreria di frammenti di prima generazione robusta e favorevole alla chimica a valle12 e successivamente una seconda generazione della biblioteca è stata messa insieme in collaborazione con Enamine ed è nota come DSI (Diamond-SGC-iNEXT)-poised fragment library (d'ora in poi denominata "In-house library"). Questa biblioteca può essere resa disponibile presso il BMRZ a scopo di screening.- Valutare la libreria di frammenti per la sua integrità e solubilità utilizzando un protocollo basato su NMR precedentemente riportato9.
NOTA: La biblioteca interna è composta da 768 frammenti con una diversità chimica molto elevata (>200 Singletons). L'esecuzione dello screening in miscele di frammenti può accelerare significativamente la campagna di screening; tuttavia, il numero di frammenti in una miscela è limitato a causa della sovrapposizione del segnale nello spettro 1H-NMR. La maggiore diversità chimica offerta dalla libreria interna consente la preparazione di miscele contenenti 12 frammenti diversi senza alcuna significativa sovrapposizione di spostamento chimico negli spettri NMR osservati 1H. - 103 frammenti all'interno dei 768 frammenti possiedono un atomo di fluoro. Per scopi di screening 19 F, dividere tutti i 103 frammenti che possiedono un gruppo fluoro in 5 miscele basate sulla sovrapposizione minima di spostamento chimico di 19F. Per ridurre al minimo la sovrapposizione del segnale nello screening 19F, utilizzare le informazioni sullo spostamento chimico dalle misurazioni di singoli composti alla progettazione di miscele con numero massimo di frammenti e minima sovrapposizione del segnale. Ogni miscela ha 20-21 frammenti con distinti spostamenti chimici di 19F che consentono un'assegnazione univoca dei frammenti.
- Valutare la libreria di frammenti per la sua integrità e solubilità utilizzando un protocollo basato su NMR precedentemente riportato9.
- Libreria di frammenti definita/fornita dall'utente
- Eseguire campagne di screening con la libreria di frammenti definita o fornita dall'utente; Tuttavia, i seguenti passaggi devono precedere la campagna di screening.
- Se non specificato dall'utente in anticipo, eseguire il controllo di qualità basato su NMR dei frammenti (presso il BMRZ, vengono utilizzati strumenti software avanzati per questo; 9, capitolo 6.1.1).
- Controllare la solubilità dei frammenti nel buffer-of-choice per il bersaglio biomolecolare, l'integrità della struttura e la concentrazione dei frammenti prima dell'uso.
- Progettare la miscela in modo da ridurre sia la sovrapposizione del segnale negli spettri NMR che il tempo di misurazione.
- Progettare le miscele secondo il punto 4.2.
- Scherma singoli frammenti o un sottoinsieme di miscele invece dell'intera libreria.
2. Preparazione del campione
NOTA: lo screening ad alta produttività mediante NMR utilizza un robot di pipettaggio per la preparazione dei campioni. Gli spettri NMR, ma anche le stabilità per diversi giorni di acquisizione del segnale di proteine, RNA e DNA sono estremamente sensibili alle fluttuazioni di temperatura e quindi i sistemi automatizzati a temperatura controllata faciliteranno notevolmente la stabilità dei campioni sottoposti a pipetta. A tale scopo, un dispositivo aggiuntivo aggiuntivo, che funziona da 4 a 40 °C, è accoppiato al robot di pipettaggio per la gestione dei liquidi dei campioni NMR in un ambiente a temperatura controllata.
- Preparazione della miscela di leganti
- Preparare campioni di screening per le misurazioni NMR utilizzando un robot di preparazione dei campioni. La configurazione flessibile del robot consente un'ampia gamma di applicazioni (ad esempio, recupero dei campioni da tubi NMR in contenitori di stoccaggio o attività generali di movimentazione dei liquidi). Possono essere utilizzati tubi NMR con diversi diametri (1,7, 2,0, 2,5, 3,0 e 5,0 mm). Il sistema robotizzato campione insieme al software di controllo avanzato legge il codice a barre assegnato per ogni tipo di contenitore ed esegue il protocollo di riempimento del liquido in modo ottimale.
- Per la preparazione delle miscele di leganti della libreria interna, utilizzare fiale con codice a barre. Le fiale con codice a barre garantiscono il massimo livello di affidabilità e una tracciabilità ottimale dei campioni.
- Distribuire 768 composti in 8 piastre di formato a 96 pozzetti. La concentrazione stock di ogni singolo frammento è di 50 mM in d6-DMSO/D2O (9:1). In totale, preparare 64 miscele contenenti ciascuna 12 frammenti. La concentrazione finale di ciascun frammento in una miscela è di 4,2 mM.
NOTA: Il robot di pipettaggio può ospitare una varietà di tipi di contenitori con geometrie diverse (fiale di criocampionatore o autocampionatore, piastre a 96 pozzetti rotonde o quadrate di profondità, fiale standard con codice a barre, tubi per microcentrifuga) e assiste l'esecuzione efficiente del trasferimento del liquido a una varietà di tubi e rack NMR.

Figura 1: (A) Preparazione del campione NMR ad alta produttività e robot di riempimento dei tubi NMR installato presso BMRZ. (B) Scambiatore di campioni ad alta produttività con rack individuali a temperatura controllata installati su uno spettrometro a 600 MHz presso l'impianto BMRZ. Fare clic qui per visualizzare una versione ingrandita di questa figura.
- Preparazione del campione di screening in bianco (spettro del ligando di riferimento) e con target (ligando in presenza del bersaglio)
- Per la preparazione dei campioni di screening NMR, in presenza della biomolecola bersaglio (proteina/RNA/DNA) e della miscela di ligando, utilizzare provette per scambiatori di campioni NMR HT da 3 mm selezionate dal portafoglio Bruker NMR di tubi NMR standard.
- Trasferire il bersaglio biomolecolare (ad esempio, 1H Screening: 10 μM RNA o Proteina) in un tampone di screening definito nel tubo NMR da 3 mm (volume finale di 200 μL) manualmente o utilizzando il robot di pipettaggio.
- Trasferire 10 μL (ad esempio, 1H Screening) della miscela di leganti nella fase successiva utilizzando il sistema robotico nei tubi NMR da 3 mm con codice a barre contenenti la biomolecola bersaglio e miscelare utilizzando il protocollo integrato del software di controllo.
NOTA: Il numero di codice a barre del tubo NMR è comodamente e automaticamente incorporato nel set di dati NMR acquisito, garantendo così un flusso di lavoro orientato all'ID senza alcuna confusione. L'accessorio di controllo della temperatura del robot di pipettaggio consente di mantenere i campioni preparati nelle provette NMR a temperatura costante.
- Condizioni e parametri definiti internamente
- Stabilire condizioni tampone ottimali per eseguire lo screening di RNA e proteine rispetto alla libreria di frammenti interna. I seguenti campioni condizionati sono utilizzati per l'RNA al BMRZ: 25 mM KPi, 50 mM KCl, pH 6,2. Mg2+ è facoltativo.
- Le proteine sono estremamente sensibili alle condizioni di soluzione; Utilizzare buffer ottimali per la destinazione scelta. Per ciascuno di questi buffer, acquisire spettri di riferimento aggiuntivi dei ligandi da utilizzare come vuoto per l'analisi.
- Condizioni specificate dall'utente
NOTA: Nei casi in cui le condizioni stabilite internamente non sono adatte per i target da sottoporre a screening da un potenziale utente, è necessario implementare i seguenti passaggi.- Eseguire 1H-NMR del tampone da solo per garantire interferenze minime dai componenti del tampone nell'esecuzione e nell'analisi degli esperimenti di screening osservati con ligando. I componenti interferenti potrebbero essere opportunamente sostituiti con equivalenti deuterati.
- Limitazioni nella produzione di campioni (quantità target)/condizioni e disponibilità
NOTA: L'isolamento o la produzione ricombinante di alcune biomacromolecole può in alcuni casi rivelarsi impegnativo e comportare una disponibilità limitata del target per perseguire una campagna di screening farmacologico di successo. In caso di disponibilità limitata o illimitata dei bersagli, le seguenti alternative potrebbero essere utilizzate per condurre con successo uno screening dei frammenti basato su NMR.- Se limitato, utilizzare 19screening basati su F-NMR. I tipici ligandi fluorurati hanno un singolo segnale 19F; Pertanto, utilizzare cocktail con 25-30 frammenti senza alcuna sovrapposizione di segnale. Ci sono meno segnali da analizzare, nessuna interferenza di segnale dai componenti del buffer e meno segnali su cui fare affidamento per l'identificazione dei colpi.
- Se illimitato, utilizzare schermi più grandi come 1H-NMR. La libreria di frammenti più grande può essere sottoposta a screening. Tipicamente, i frammenti sono composti da più di un protone, il che significa più segnali su cui fare affidamento per l'analisi.
3. Condizioni di acquisizione NMR
- Condizioni interne generalmente definite
- Spettrometro dotato di scambiatore di campioni HT (Automazione)
- Per lo screening ad alta produttività, utilizzare piastre a 96 pozzetti che possono essere misurate solo utilizzando uno scambiatore di campioni HT. Lo scambiatore di campioni HT offre anche la possibilità di temprare singolarmente ogni rack.
- Per un rapporto segnale-rumore ottimale, utilizzare uno spettrometro con una sonda criogenica raffreddata ad elio o azoto. Per l'automazione è necessario un modulo di sintonizzazione e corrispondenza automatizzato (ATM).
- Set di parametri e sequenze di impulsi
NOTA: Molti esperimenti NMR possono caratterizzare eventi di legame. L'identificazione dei riscontri varia a seconda della configurazione sperimentale. I seguenti esperimenti sono utilizzati di routine nelle campagne di screening BMRZ. Tuttavia, è possibile apportare modifiche per le campagne di screening definite dall'utente e in base alle specifiche dell'utente.- Se si utilizza il software TopSpin, includere il set di parametri per gli esperimenti basati su ligando: SCREEN_STD, SCREEN_T1R, SCREEN_T2 SCREEN_WLOGSY. Il set di parametri include tutti i parametri necessari e le sequenze di impulsi: STD: stddiffesgp.3; T1ρ: t1rho_esgp2d; T2: cpmg_esgp2d; e waterLOGSY: ephogsygpno.2.
- Per tutti gli esperimenti elencati, utilizzare l'eccitazione scultorea13 come soppressione dell'acqua. Per un riferimento, utilizzare la scultura dell'eccitazione 1D (zgesgp). Il numero di scansioni dipende dalla sensibilità del sistema (intensità del campo magnetico e testa della sonda), dalla concentrazione del campione e dalla scelta dell'esperimento. Una raccomandazione è: 1D con NS=64, T1ρ & T2 con NS=128, STD con NS=256 e waterLOGSY con NS= 384 o 512.
- Per lo screening 19 F, utilizzare entrambi gli esperimenti 1D e T2: 1D: F19CPD (pp=zgig) per 19 teste di sonda F{1 H} e F19(pp=zg) per 19teste di sonda F/1H; SCREEN_19F_T2 (pp = cpmgigsp).
- Utilizzare una larghezza spettrale di 220 ppm e una frequenza di eccitazione a -140 ppm. Il tempo di esperimento è compreso tra 1 e 5 ore (garantire la stabilità a lungo termine della biomacromolecola) a seconda dell'hardware e della concentrazione del campione. Per T2, il tempo CPMG dovrebbe alternarsi tra 0 ms e 200 ms.
- Elaborazione
- Registra gli esperimenti STD, T1ρ e T2 come pseudo 2D. Per elaborare i due singoli spettri 1D, IconNMR utilizza il programma au-proc_std con o senza l'opzione relax. La prima opzione fornisce il riferimento 1D e la differenza di due spettri. La seconda opzione produce due spettri separati con tempi di rilassamento brevi e lunghi. Il waterLOGSY è un singolo 1D che dovrebbe essere sfasato con un negativo per il segnale del solvente.
- Spettrometro dotato di scambiatore di campioni HT (Automazione)
- Condizioni specifiche per l'utente
- Adattare uno qualsiasi dei parametri menzionati in precedenza alle condizioni definite dall'utente. Ad esempio, se una proteina fornita dall'utente non è stabile alla temperatura generalmente utilizzata, è possibile condurre esperimenti di ottimizzazione variando temperatura, concentrazione, condizioni tampone, ecc.
4. Analisi dei dati
- QC della libreria di frammenti (d6-DMSO/buffer specifico) e quantificazione
- CMC-q
NOTA: Il controllo di qualità delle librerie di frammenti è essenziale prima dell'avvio delle campagne di screening. Inoltre, è necessario garantire la stabilità a lungo termine della biblioteca di frammenti per l'applicazione di diverse campagne di screening, motivo per cui deve essere condotta una valutazione periodica della qualità della biblioteca. A tale scopo, i software integrati CMC-q e CMC-a di TopSpin vengono utilizzati per la valutazione della qualità e della quantità. CMC-q e CMC-a sono moduli software all'interno di Topspin che consentono un'acquisizione fluida, l'analisi e la verifica della struttura utilizzando 1spettro H-NMR ottenuto da piccole molecole organiche 9.- Per l'integrità, preparare campioni di valutazione con una concentrazione di frammenti di 1 mM in d6-DMSO. Preparare i campioni in modo automatizzato con un robot di pipettaggio riempiendo la raccolta di campioni liquidi in una provetta NMR da 3 mm.
- Per la valutazione della solubilità, utilizzare un campione costituito da 1 mM di composto in tampone fosfato di sodio da 50 mM a pH 7,4, 150 mM di cloruro di sodio, 90% H 2 O/ 10% D 2 O e 1 mM di sale sodico acido 3-(trimetilsilil)propionico-2,2,3,3-d4 (TMSP-Na).
- Raccogliere spettri NMR a 298 K o 293 K utilizzando uno spettrometro NMR a 600 MHz dotato di sonda criogenica TCI a tripla risonanza da 5 mm e uno scambiatore di campioni HT, in grado di gestire 579 campioni contemporaneamente.
- Per configurare il software CMC-q seguire le istruzioni del manuale utente, che implementa la creazione di un utente IconNMR, l'attivazione di FastLaneNMR e la modifica dello scambiatore di campioni HT.
- Calibrare l'impulso di 90° e salvarlo nella tabella TopSpin prosol.
- Posizionare la piastra del pozzetto di campionamento 96 in una delle 5 posizioni rack nello scambiatore di campioni HT.
- Per caricare un file SDF (file di dati della struttura) che deve contenere la struttura chimica proposta, un identificatore univoco e la posizione nello scambiatore di campioni HT di ciascun campione in un batch, andare su Sfoglia nella finestra di configurazione CMC-q e fare clic su Apri dopo aver selezionato un file che termina con .sdf.
- Nelle impostazioni di CMC.q Batch Automation, impostare il tipo di verifica che definisce l'esperimento che verrà misurato, l'utente IconNMR e definire il solvente.
- Definire i file SDF per il percorso per il file SDF, l'ID della molecola e la posizione del campione.
- Avviare l'acquisizione facendo clic su Start. Fare nuovamente clic su Avvia acquisizione . Il setup CMC-q può anche essere salvato cliccando su Salva.
- Per una descrizione dettagliata dei passaggi di configurazione di CMC-q, seguire le istruzioni del manuale utente di Bruker.
- CMC-a
- Per CMC-a, utilizzare il modulo software all'interno di Topspin che consente l'analisi, inclusa la verifica della struttura utilizzando lo spettro 1H-NMR ottenuto da piccole molecole organiche9.
- CMC-q
- Design della miscela
NOTA: Una corretta progettazione della miscela svolge un ruolo importante per lo screening utilizzando NMR come piattaforma. Un numero elevato di frammenti per miscela consente uno screening più rapido, ma aumenta il rischio di falsi positivi e negativi. Un numero inferiore riduce tale rischio, ma aumenta il tempo necessario per condurre lo screening. In generale, una sovrapposizione di segnali deve essere evitata durante la creazione di miscele. Utilizzando la libreria interna, questo può essere trascurato per lo screening 1H in quanto la libreria è stata specificamente progettata per essere diversificata e mostrare poca sovrapposizione di segnali pur mantenendo un'elevata diversità chimica. Ciò a sua volta significa che non è necessario sottoporsi a una procedura di progettazione speciale per la creazione delle 64 miscele.- Poiché lo screening 19F si basa sui frammenti della libreria interna che contengono fluoro e la libreria non è stata creata per ridurre la sovrapposizione del segnale per questi frammenti specifici, progettare una miscela adeguata.
- Misurare spettri composti singoli per tutti i frammenti contenenti 19F.
- Notare le informazioni sullo spostamento chimico di ciascun segnale.
- In base a queste informazioni, scegliere 20-21 frammenti per miscela. Questo a sua volta fornisce 5 miscele contenenti ciascuna 20-21 frammenti senza sovrapposizione di segnale e consente un'analisi semi-automatica dei dati.
- Eseguire l'identificazione dei colpi all'interno di un'interazione biomacromolecola-ligando osservata da un ligando
NOTA: Esistono diverse definizioni di hit tra la procedura di screening 19F e 1H. Le seguenti identificazioni dei riscontri sono state create da noi e seguono regole specifiche. L'oggetto della determinazione del colpo è molto soggettivo e può differire da utente a utente. Tuttavia, è della massima importanza che le regole per l'identificazione dei riscontri positivi non cambino una volta concordate per mantenere la convalida e la credibilità.- 1Schermo H
- Per determinare con sicurezza i colpi, acquisire spettri 1D 1H, waterLOGSY e esperimenti di rilassamento T2 sia in presenza che in assenza di bersaglio per identificare i leganti. Tutti e tre gli esperimenti hanno il potenziale per mostrare un evento vincolante. Se un CSP superiore a 6 Hz è visibile negli spettri del campione rispetto agli spettri vuoti, questo è considerato come un'indicazione per un hit. Lo stesso vale se è visibile un forte segnale positivo nel waterLOGSY e una riduzione di oltre il 30% di T2 negli spettri del campione. Gli eventi di legame possono essere mostrati in tutti e tre gli esperimenti, quando si confronta il campione contenente spettri con i rispettivi spettri vuoti. Tuttavia, gli eventi di legame potrebbero non essere visibili in tutti e tre gli esperimenti. Per questo motivo è stato concordato che almeno due degli eventi sopra descritti devono verificarsi per classificare un frammento come un colpo vincolante.
- Utilizzare lo strumento FBS in TopSpin per definire lo stato dei frammenti in binding, ambiguo, sconosciuto, aggregato e non vincolante.
- Quando hai finito con un mix, approvalo all'interno dello strumento FBS.
- Nella scheda di riepilogo all'interno del progetto FBS, fai clic su Crea un rapporto di screening. Si aprirà una finestra che crea un file .xlsx. L'utente può quindi scegliere tra tutti i leganti, solo ligando vincolante, solo ligando non vincolante e leganti ambigui da riportare nel foglio di calcolo.
- 19Schermo F
- Per distinguere tra non legante, raccoglitore settimanale e raccoglitore forte, dividere il quoziente di integrazione tra la misurazione target di 200 ms e la misurazione di 200 ms in bianco per il quoziente della misurazione target di 0 ms e viene utilizzata la misurazione in bianco di 0 ms:
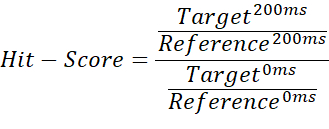
NOTA: fornisce valori compresi tra 0 e ~ 1 (l'hit-score), rendendo possibile l'assegnazione di soglie per ogni stato di associazione. - Utilizzare la media della misurazione di riferimento di 200 ms come soglia di base, per contrassegnare i casi in cui l'hit-score supera 1. Ciò può verificarsi se gli integrali importati contengono valori negativi o la misura di riferimento è superiore alla misura di destinazione. Un hit-score di ≤ 0,67 è considerato un colpo debole, < 0,33 un colpo forte, e qualsiasi cosa > 0,67 come no-hit. Un esempio è illustrato nella Figura 2.
- Per distinguere tra non legante, raccoglitore settimanale e raccoglitore forte, dividere il quoziente di integrazione tra la misurazione target di 200 ms e la misurazione di 200 ms in bianco per il quoziente della misurazione target di 0 ms e viene utilizzata la misurazione in bianco di 0 ms:
- 1Schermo H
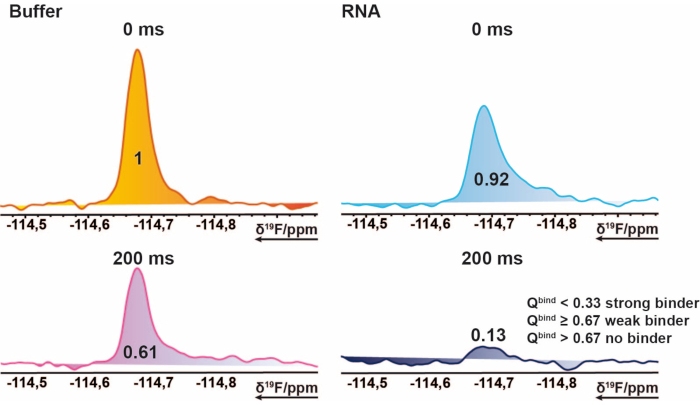
Figura 2: Identificazione dei colpi per lo screening 19F. Sezione di 19spettri F CPMG NMR di un composto esemplare. Questa rappresentazione pittorica spiega le proprietà di un legante. 19Spettri F-CPMG di un composto acquisito da campioni di miscela in presenza e assenza di RNA. I valori rappresentano i valori integrali normati del picco corrispondente. Fare clic qui per visualizzare una versione ingrandita di questa figura.
- Analisi dei dati
- Preparare i dati per l'analisi
NOTA: È importante che i dati acquisiti non presentino difetti visibili. Ciò significa che i dati in cui lo shimming era problematico o la soppressione dell'acqua era insufficiente non dovrebbero essere considerati per l'analisi. Piuttosto, si consiglia di registrare nuovamente i dati e assicurarsi che tutto vada bene con il campione (ad esempio, senza bolle d'aria), con la temperatura, lo shimming e la soppressione dell'acqua. La correttezza dei dati può sempre essere valutata quando si confrontano i segnali DMSO. - 1Screening H
- Per analizzare i dati di screening 1H, utilizzare lo strumento FBS (richiede una licenza aggiuntiva) in TopSpin 4.0.9.
- Seguire le istruzioni nel manuale dello strumento FBS per iniziare con l'analisi dei dati. I passaggi seguenti riassumono la procedura riportata nel manuale.
- Memorizzare i dati NMR BMRZ dalle campagne di screening in modo tale che ogni diversa miscela di screening abbia la propria directory in cui una sottodirectory contiene i diversi esperimenti misurati sul campione.
- Per utilizzare lo strumento FBS, memorizzare gli spettri di riferimento che hanno tutti i dati salvati dai campioni senza il bersaglio biomolecolare ma con le miscele e il singolo composto misurato in diverse directory /nmr. Questo è importante in quanto lo strumento FBS chiederà il percorso della directory di ciascuno individualmente.
NOTA: lo strumento FBS riconoscerà una directory come progetto di screening se i seguenti set di dati sono stati memorizzati nella stessa directory in cui sono memorizzate le combinazioni di un campione di screening (csv, documenti XML FragmentScreen e file BAK). - Quando si utilizza TopSpin 4.0.9, creare un percorso diretto alla directory contenente i dati acquisiti, un cosiddetto DIR. Scegliere la directory /nmr in cui tutte le miscele devono avere una directory distinta.
- Per avviare lo strumento FBS di un esempio schermato, trascinare il progetto FBS simbolo al centro della finestra TopSpin. Nella directory scelta dovrebbe apparire il simbolo del progetto FBS se in precedenza tali set di dati sono stati copiati in esso.
- La finestra Opzioni di screening basate su frammenti dovrebbe aprirsi automaticamente al primo caricamento di un nuovo progetto FBS. In questa finestra scegli un file cocktail. Il file cocktail è un file csv contenente l'assegnazione del nome delle miscele, il nome di ogni frammento e la loro divisione nelle miscele. Definite anche una cartella degli spettri del ligando di riferimento che contiene tutti gli spettri misurati dei singoli frammenti. Infine, definire una cartella di esperimenti vuota di riferimento, che di solito è la cartella contenente i set di dati dei mix senza la destinazione indagata.
- Le opzioni di screening basate su frammenti hanno una scheda chiamata Tipi di spettri che consente di definire gli spettri studiati e il colore per la visualizzazione degli spettri . Impostare lo Spectype in base ai dati elaborati in precedenza. Nella scheda Layout di visualizzazione , definire gli spettri che verranno confrontati tra loro in base alle relative specifiche.
- Premere OK per avviare il progetto FBS.
- Mentre si guardano i dati, si aprirà una finestra separata, che riassume tutte le miscele di cocktail e tutti i ligandi di ciascuna miscela in una tabella. Facendo doppio clic su una cella, si apriranno i rispettivi set di dati, confrontando ad esempio gli spettri vuoti 1H 1D con il set di dati contenente il target.
- Prima di assegnare i leganti, assicurarsi che i picchi di riferimento (DMSO di tutte le misurazioni e dei singoli composti) corrispondano tra loro e abbiano lo stesso spostamento chimico. Se si osservano differenze, correggerle utilizzando l'opzione di elaborazione seriale di TopSpin.
- L'opzione di elaborazione seriale si trova nella scheda Processo in Avanzate. Applica le modifiche a tutti gli spettri selezionati da un set di dati. In questo modo, gli Spectype possono essere facilmente assegnati ai numeri degli esperimenti e tutti gli spettri possono essere spostati contemporaneamente per allinearsi con il riferimento.
- 19F Screening
- Per la prima analisi delle 19miscele F, creare un file di integrazione per ogni miscela. Per definire la regione di integrazione, fare clic sulla funzione Integra nella scheda Analizza . Assicurarsi che per ogni frammento della miscela sia definita una chiara regione di integrazione per i corrispondenti 19F-singal.
- Utilizzare il pulsante Salva/Esporta regioni di integrazione per esportare il file di integrazione per un utilizzo futuro. Salvare tutti i file di integrazione utilizzati in C:\Bruker\TopSpin4.0.9\exp\stan\nmr\lists\intrng, o il percorso corrispondente della directory di installazione di TopSpin.
- Per i dati 19F, aprire un set di dati con o senza la destinazione analizzata.
- Per caricare il file di integrazione nello spettro corrente, aprire nuovamente la scheda Analizza , andare su Integra e utilizzando il pulsante Leggi/Importa regioni di integrazione, caricare il file di integrazione corrispondente. In questo modo verranno caricate tutte le regioni definite del file nello spettro corrente.
- Salvare e tornare per trovare un elenco di tutte le regioni integrate nella scheda Integrali . Copialo in un foglio di calcolo o in qualsiasi altro strumento utilizzato per l'ulteriore analisi dei dati.
- Ripetere questa procedura per ogni mix, con e senza target.
- Gestione dei dati
- Per facilità d'uso e produttività, disporre di un flusso di lavoro uniforme per l'ulteriore analisi e archiviazione dei dati acquisiti. Per lo screening 1 H e 19F, utilizzare un foglio di calcolo appositamente progettato per ciascuno.
NOTA: Per lo screening 1H questo è stato utilizzato esclusivamente per la gestione dei dati e per riassumere ciascun bersaglio, mentre per lo screening 19F è stato utilizzato il quoziente spiegato nel capitolo 4.3 per etichettare automaticamente ogni frammento come hit/no hit dopo che i dati integrali sono stati copiati in esso. Ciò riduce il rischio di errore umano durante l'analisi, supponendo che il file sia stato impostato correttamente, e rende più facile la condivisione delle informazioni, poiché tutte le informazioni importanti vengono raccolte in un unico posto in un file che può essere aperto praticamente da chiunque senza la necessità di ulteriori programmi per dare un'occhiata iniziale ai dati.
- Per facilità d'uso e produttività, disporre di un flusso di lavoro uniforme per l'ulteriore analisi e archiviazione dei dati acquisiti. Per lo screening 1 H e 19F, utilizzare un foglio di calcolo appositamente progettato per ciascuno.
- Preparare i dati per l'analisi
Risultati
Controllo di qualità della libreria di frammenti
I frammenti della libreria interna sono stati consegnati come soluzioni madre da 50 mM in 90% d6-DMSO e 10% D 2 O (il10% di D2O garantisce la minimizzazione della degradazione del composto dovuta ai ripetuti cicli di congelamento-disgelo14). I campioni composti singoli consistevano in 1 mM di ligando in tampone fosfato da 50 mM (25 mM KPi pH 6,2 + 50 mM KCl + 5 mM MgCl 2), pH 6,0 in 90% H 2 O/9% D2O/1% d6-DMSO. 1Gli esperimenti H-NMR di frammenti della libreria iNEXT sono stati misurati su uno spettrometro NMR a 500/600 MHz. Questi dati sono stati ulteriormente utilizzati per identificare i singoli composti nelle campagne di screening 1H utilizzando il software CMC-q che consente all'utente di acquisire completamente spettri in modo automatizzato e l'aggiunta di analisi CMC-a la qualità (solubilità e integrità) dei frammenti è stata valutata. I risultati dell'analisi automatizzata di CMC-a sono mostrati come output grafico simile a quello rappresentato nella Figura 3. L'output grafico mostra una rappresentazione di una lastra da 96 pozzetti. Un cerchio di colore rosso significa che questo frammento mostra incoerenza nella struttura o nella concentrazione. I pozzetti di colore verde indicano che il frammento è coerente.
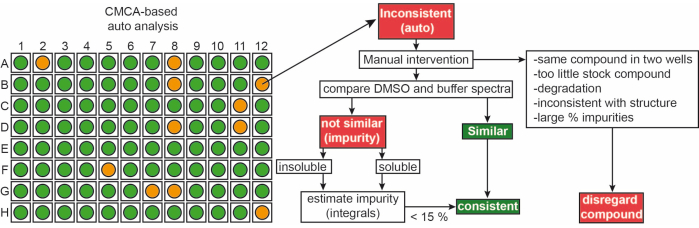
Figura 3: Controllo di qualità della libreria di frammenti. Rappresentazione schematica dell'output automatizzato basato su CMC-a. Vengono valutate le proprietà dei frammenti come la concentrazione e l'integrità strutturale. Il verde sta per coerente, l'arancione in questo caso sta per incoerente. I frammenti incoerenti vengono rivisti manualmente seguendo il flusso di lavoro mostrato. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Approssimativamente, il 65% e il 35% dei frammenti sono stati classificati come coerenti e incoerenti, rispettivamente, sia nel DMSO che nel buffer. Inoltre, il 30% dei ligandi classificati incoerenti è diventato coerente dopo un'attenta ispezione manuale degli spettri9.
19F Design della miscela
103 frammenti contenenti uno o più gruppi di fluoro dalla libreria interna sono stati suddivisi in 5 miscele (A, B, C, D, E). Ogni miscela ha da 20 a 21 frammenti. In questo caso le miscele dovevano essere attentamente progettate per evitare sovrapposizioni di segnale. 19Sono stati misurati esperimenti di rilassamento trasversale F per ogni miscela che applica treni di impulsi CPMG. Questi esperimenti possono essere modificati variando i ritardi di rilassamento. Lo spostamento chimico 19F delle miscele A-E può essere visto nella Figura 4.
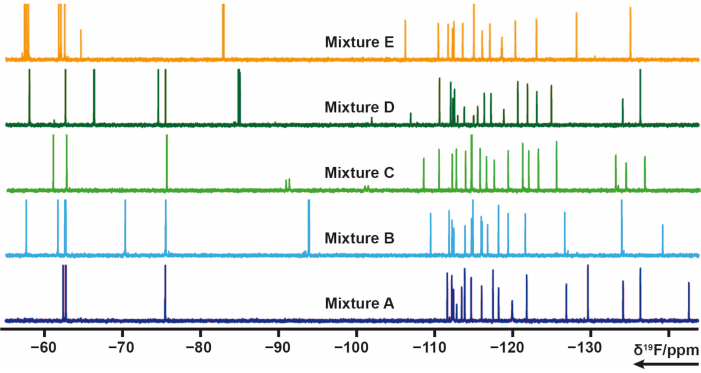
Figura 4: 19spettri F 1D-NMR di campioni di miscela dalla libreria interna. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Preparazione del campione
La preparazione del campione nella procedura di screening 19F è stata eseguita manualmente o con pipettaggio automatizzato utilizzando un robot di pipettaggio. I frammenti in ciascuna miscela avevano una concentrazione di 2,5 mM nel 90% d6-DMSO e nel 10% in D2O. Il volume finale di un campione di screening era di 170 μL con il 5% di D2O come agente bloccante. Ogni miscela è stata pipettata due volte, una in una soluzione contenente tampone (senza bersaglio) e una in una soluzione tampone contenente bersaglio. Il rapporto tra bersaglio e frammento è stato impostato a 1:1, risultando in una concentrazione finale target/ligando di 50 μM. Inoltre, i campioni di controllo sono la biomolecola bersaglio nel tampone di screening senza miscela per garantire l'integrità del bersaglio, nonché un campione di controllo con solo tampone e D2O per garantire la qualità del tampone.
I dati di screening NMR di 19 F-1D e 19F-CPMG-T2 sono stati misurati come descritto nella sezione 3.1. Ad esempio, nel caso dell'RNA è stata acquisita una sequenza di eco jump-return (pp = zggpjrse,15) per il singolo campione bersaglio in buffer.
Analisi dei dati
La procedura di screening 19F è stata applicata al TPP riboswitch thiM da E. coli e alla proteina tirosina chinasi (PtkA) da M. tuberculosis tra molti altri bersagli16. La libreria di screening 19F ha 103 frammenti che sono divisi in 5 miscele etichettate dalla miscela A alla E. La preparazione dei campioni di screening può essere eseguita manualmente senza l'uso di un robot di pipettaggio dei campioni. La soluzione contenente thiM RNA da 40 μM (condizioni tampone) è stata miscelata con 3,2 μL delle miscele. Sono stati preparati ulteriori campioni di controllo costituiti da tampone solo, tampone con il 5% di DMSO (precedentemente garantire la stabilità della biomacromolecola in presenza della concentrazione di DMSO desiderata) e tampone con RNA. Questi 13 campioni di screening sono stati preparati e trasferiti in tubi NMR da 3 mm. I codici a barre delle provette NMR vengono scansionati e ogni miscela in presenza e assenza di RNA, così come i campioni di controllo sono stati misurati secondo i suddetti esperimenti NMR 19F eseguiti a 298 K. Lo screening del thiM RNA rispetto alla libreria interna è stato eseguito conducendo misurazioni T2 con CPMG di 0 ms e 200 ms per ogni diverso campione. Il corretto shimming e la soppressione dell'acqua sono stati monitorati dopo aver terminato le misurazioni confrontando tutti i picchi di DMSO in termini di ampliamento della linea e perdita di intensità degli esperimenti 1 H1D misurati in aggiunta per tutti i campioni. L'elaborazione degli spettri di rilassamento CPMG T2 19F ottenuti è stata eseguita utilizzando rispettivamente una macro precedentemente preparata e automatizzata in TopSpin. L'analisi dei dati è stata eseguita seguendo le istruzioni nella sezione protocollo. I dati integrali ottenuti da TopSpin (seguendo le istruzioni nel protocollo) possono essere valutati rapidamente e facilmente utilizzando un foglio di calcolo pre-creato o qualsiasi programma simile, impostando le condizioni e le soglie corrette. Come descritto in precedenza, le soglie sono utili per definire raccoglitore, raccoglitore debole o non raccoglitore. La Figura 5 mostra i risultati tipici degli spettri CPMG dell'RNA thiM e del PtkA, rispettivamente. In alcuni casi, è stata necessaria un'ulteriore revisione da parte di esperti.
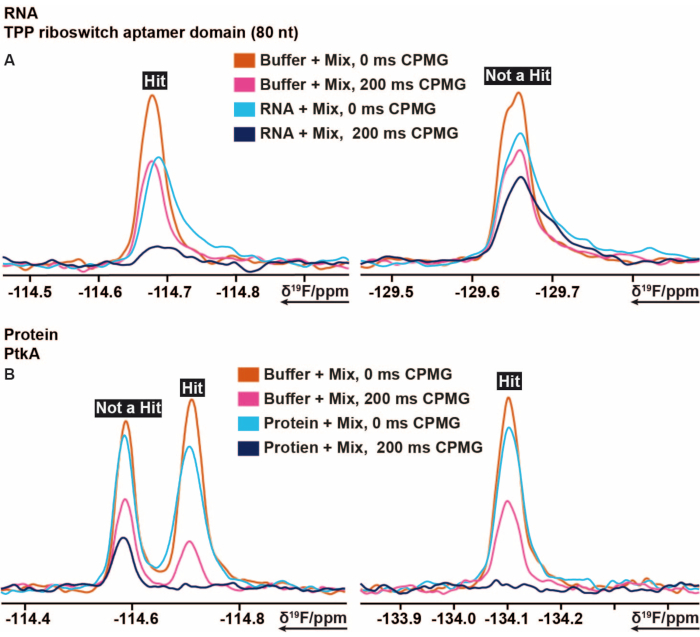
Figura 5: Taglio di 19 spettri F CPMG NMR che mostrano le variazioni di intensità ottenute da diversi tempi di ritardo degli esperimenti basati su CPMG . (A) Rappresentazione di un legante (hit) e di un non-legante nello screening basato su frammenti 19F eseguito su TPP riboswitch thiM RNA da E. coli. (B) Rappresentazione di un legante e di un non-legante nello screening basato su frammenti 19F eseguito su PtkA da M. tuberculosis. Fare clic qui per visualizzare una versione ingrandita di questa figura.
1ora di proiezione
Design della miscela
La libreria interna utilizzata è così diversificata che per scopi di screening 1H non è stato eseguito alcun design di miscela. Ciò significa che 64 miscele sono state preparate scegliendo casualmente 12 da mescolare in una miscela.
Preparazione del campione
Per lo screening 1H di un RNA SARS-CoV-2 esemplare, è stato eseguito il pipettaggio automatico utilizzando un robot di pipettaggio per preparare i campioni. I frammenti in ciascuna miscela avevano una concentrazione di 4,2 mM nel 90% d6-DMSO e nel 10% in D2O. Il volume finale di un campione di screening era di 200 μL con il 5% di D2O come agente bloccante. 64 campioni contenenti ciascuno una miscela diversa in 25 mM KPi, 50 mM KCl a pH 6,2 sono stati pipettati senza RNA bersaglio. Rispettivamente, 64 campioni sono stati pipettati con RNA bersaglio, ciascuno contenente una miscela diversa. Il rapporto RNA:Ligando è stato impostato a 1:20, risultando in una concentrazione di RNA di 10 μM e una concentrazione di ligando di 200 μM.
Analisi dei dati
Per l'analisi 1H, è stato utilizzato lo strumento FBS in TopSpin. Per determinare se un frammento è un colpo, sono stati condotti esperimenti di rilassamento 1D chemical shift, waterLOGSY e T2 . Per il rilassamento di T2 , una diminuzione dell'intensità superiore al 30% è stata contata come un colpo, mentre per lo spostamento chimico uno spostamento superiore a 6 Hz è stato il cut-off. Il waterLOGSY doveva mostrare un cambiamento significativo del segnale (da negativo a positivo in questo caso). Se due di questi tre criteri erano positivi, un frammento veniva conteggiato come un successo. Due esempi di questo possono essere visti nella Figura 6.
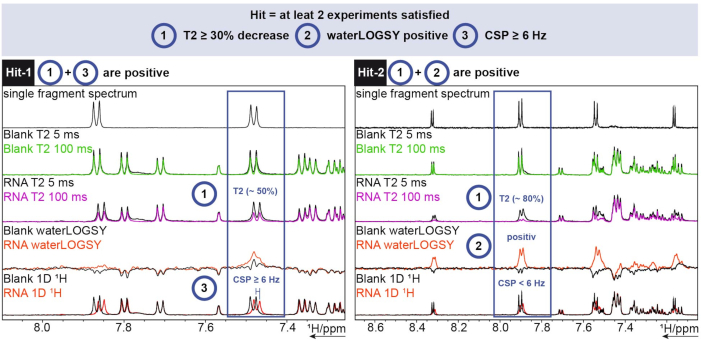
Figura 6: 1H screening eseguito su un RNA SARS-CoV-2 esemplare che mostra criteri di determinazione del riscontro cardiaco. Acquisizione di tre diversi esperimenti (1 H T2 CPMG (5/100 ms), waterLOGSY e 1D 1H). Fare clic qui per visualizzare una versione ingrandita di questa figura.
Hit-1 mostra una diminuzione di T2 di ~ 50% e un CSP ≥ 6 Hz. Il waterLOGSY non mostra un cambiamento abbastanza significativo nel segnale da essere conteggiato come positivo. Poiché due esperimenti su tre sono positivi, questo frammento viene conteggiato come un successo. Per Hit-2, il T2 mostra una diminuzione dell'intensità del segnale ~ 80% e un chiaro cambiamento del segnale può essere visto per il waterLOGSY. Il CSP non è sufficiente in questo caso, ma poiché i due criteri precedenti sono positivi, viene comunque conteggiato come un successo.
Discussione
Versatilità dello screening di frammenti/farmaci basato su NMR. BMRZ ha implementato con successo strumentazione NMR automatizzata all'avanguardia, nonché STD-NMR, waterLOGSY ed esperimenti di rilassamento per identificare frammenti all'interno di una vasta gamma di regimi di affinità per la scoperta di farmaci. L'hardware installato include un robot per la preparazione dei campioni ad alta produttività e un'unità di archiviazione, sostituzione e acquisizione dati ad alta produttività associata a uno spettrometro a 600 MHz. Una sonda criogenica acquistata di recente per 1 H, 19 F, 13C e 15N garantisce la sensibilità richiesta per le misurazioni proposte e consente il disaccoppiamento di 1 H (1) durante il rilevamento di 19F. Questa sonda è collegata alla console NMR di ultima generazione che offre la possibilità di utilizzare gli strumenti software avanzati di Bruker, tra cui CMC-q, CMC-assist, CMC-se e FBS (inclusi in TopSpin). Lo strumento di screening basato su frammenti (FBS) è incluso nell'ultima versione di TopSpin e aiuta ad analizzare i dati ad alto rendimento che comprendono esperimenti di rilassamento STD, waterLOGSY, T2 / T1r. La raccolta di campioni liquidi 1D 1H può essere riempita nelle provette NMR in modo automatizzato utilizzando il robot di riempimento del campione. In genere, un blocco di 96 tubi (3 mm) viene riempito in circa due ore. Le scaffalature a 96 pozzetti sono posizionate direttamente nello scambiatore di campioni HT, che legge il codice a barre del blocco e assegna i tubi NMR agli esperimenti controllati dal software di automazione (IconNMR). Cinque rack a piastre da 96 pozzetti possono essere immagazzinati e programmati contemporaneamente nello scambiatore di campioni HT. La temperatura di ciascuno dei singoli rack può essere controllata e regolata separatamente. Inoltre, ogni singolo campione può essere precondizionato (preriscaldamento e asciugatura del tubo per la rimozione dell'umidità condensata) alla temperatura desiderata prima della misurazione.
Idoneità per un'ampia gamma di applicazioni. Una delle ampie applicazioni di questo screening automatizzato basato su NMR è l'identificazione e lo sviluppo di nuovi ligandi che si legano a un bersaglio biomacromolecolare (DNA / RNA / proteine). Questi ligandi possono includere inibitori ortosterici e allosterici che tipicamente si legano in modo non covalente. Inoltre, FBDD da NMR è tipicamente utilizzato come primo passo per selezionare composti promettenti, i requisiti da soddisfare sono la disponibilità del bersaglio biomolecolare in quantità sufficienti. Questo obiettivo è diviso in due compiti principali.
Il primo compito è quello di sviluppare e caratterizzare una libreria di frammenti interna per i seguenti motivi: controllo di qualità iniziale e periodico, caratterizzazione e quantificazione di oltre 1000 frammenti; determinazione della solubilità dei frammenti in tamponi ottimizzati per ciascun bersaglio, in particolare per bersagli proteici; e la creazione di diverse librerie per ospitare diversi scaffold e l'estensione verso altre classi di macromolecole. Il secondo compito è quello di integrare i flussi di lavoro per la progettazione di farmaci basati su frammenti (FBDD) mediante NMR utilizzando: screening automatizzato osservato con ligando 1D (1H e 19F osservati); saggi di sostituzione automatizzati (esperimenti di competizione con ligando (naturale)) per differenziare il legame ortosterico e allosterico; screening secondari automatizzati con più frammenti; screening automatizzato delle proteine 2D e screening secondario di una serie di derivati attorno a un riscontro positivo iniziale utilizzando la libreria EU-OPENSCREEN o qualsiasi altra libreria; e riprofilare lo screening della biblioteca FDA rispetto agli obiettivi scelti.
Inoltre, la metabotipizzazione di varie linee cellulari (rilevante per la malattia) può essere condotta al fine di svelare i meccanismi regolatori che collegano il controllo del ciclo cellulare e il metabolismo. Inoltre, vi è la caratterizzazione funzionale di elementi di regolazione RNA/DNA/proteine in vivo e in vitro per l'ottimizzazione dell'ottimizzazione del costrutto/dominio (ottimizzazione della stabilità per indagini strutturali (Buffer, pH, temperatura e screening salino), e un'estensione dello screening dei frammenti basato su NMR alle proteine di membrana e alle proteine intrinsecamente disordinate, che sono generalmente inaccessibili ad altre tecniche.
Limitazioni. L'uso di librerie di frammenti 19F e 1H ha i suoi pro e contro, alcuni dei quali saranno menzionati di seguito. Il più grande vantaggio delle misurazioni 19F rispetto a 1H è la velocità sia del tempo di misurazione effettivo che dell'analisi successiva, poiché le miscele contengono quasi il doppio del numero di frammenti e devono essere condotti meno esperimenti. L'analisi di follow-up è anche più semplice per lo screening 19F, in quanto non vi è alcuna interferenza da buffer e offre inoltre una gamma di spostamento chimico più ampia con quasi nessuna sovrapposizione di segnali per una miscela di frammenti progettata in modo ottimale. Gli spettri stessi sono notevolmente semplificati, di solito hanno solo uno o due segnali per frammento, a seconda del numero di atomi di fluoro. L'analisi di questi spettri può quindi essere automatizzata, riducendo ancora una volta i tempi. Ciò avviene a scapito della diversità chimica, almeno per la biblioteca utilizzata in questo studio. Poiché solo ~ 13% della libreria contiene 19 F, ma naturalmente tutti sono utilizzabili nello screening 1H, la diversità dei frammenti di screening 19F sarà inferiore. Questo potrebbe essere aggirato utilizzando librerie 19F appositamente progettate con più frammenti e una maggiore diversità chimica. Un altro svantaggio per lo screening 19F è il basso numero di segnali per frammento. I frammenti sono generalmente composti da più di un atomo di idrogeno. Pertanto, gli esperimenti di screening osservati 1H possono fare affidamento su segnali diversi per lo stesso frammento per rilevare il legame. Ciò conferisce un grado più elevato di sicurezza quando si identificano i risultati per lo screening 1H, mentre lo screening 19F deve basarsi su uno o due segnali forniti per frammento.
È stato presentato un resoconto dettagliato della moderna strumentazione automatizzata di screening dei frammenti basata su NMR, del software e dei relativi metodi e protocolli di analisi. L'hardware installato include un robot per la preparazione dei campioni ad alta produttività e un'unità di archiviazione, cambio e acquisizione dati ad alta produttività associata a uno spettrometro a 600 MHz. Una testa di sonda criogenica installata di recente per 1 H, 19 F, 13C e 15N garantisce la sensibilità richiesta per le misurazioni proposte e consente il disaccoppiamento di 1H durante il rilevamento di 19F. Inoltre, l'ultima generazione di console NMR offre la possibilità di utilizzare software analitici avanzati per facilitare l'acquisizione e l'analisi al volo. La tecnologia sopra discussa, i flussi di lavoro e i protocolli descritti dovrebbero favorire un notevole successo per gli utenti che perseguono FBS tramite NMR.
Divulgazioni
Nessuno.
Riconoscimenti
Questo lavoro è stato supportato da iNEXT-Discovery, progetto numero 871037, finanziato dal programma Horizon 2020 della Commissione Europea.
Materiali
| Name | Company | Catalog Number | Comments |
| Bruker Avance III HD | Bruker | 600 MHz NMR Spectrometer | |
| Matrix Clear Polypropylene 2D Barcoded Open-Top Storage Tubes | 3731-11 0.75ML V-BOTTOM TUBE/LATCH RACK | ThermoFisher Scientific | Barcoded Tubes |
| Matrix SepraSeal und DuraSeal& | 4463 Cap Mat, SeptraSeal 10/CS | ThermoFisher Scientific | |
| SampleJet | Bruker | HT Sample Changer | |
| SamplePro Tube | Bruker | Pipetting Robot |
Riferimenti
- Yanamala, N., et al. NMR-Based Screening of Membrane Protein Ligands. Chemical Biology & Drug Design. 75, 237-256 (2010).
- Souers, A. J., et al. ABT-199, a potent and selective BCL-2 inhibitor, achieves antitumor activity while sparing platelets. Nature Medicine. 19, 202-208 (2013).
- Su, M. C., Te Chang, C., Chu, C. H., Tsai, C. H., Chang, K. Y. An atypical RNA pseudoknot stimulator and an upstream attenuation signal for -1 ribosomal frameshifting of SARS coronavirus. Nucleic Acids Research. 33, 4265-4275 (2005).
- Perera, T. P. S., et al. Discovery & pharmacological characterization of JNJ-42756493 (Erdafitinib), a functionally selective small-molecule FGFR family inhibitor. Molecular Cancer Therapeutics. 16, 1010-1020 (2017).
- Zhang, C., et al. Design and pharmacology of a highly specific dual FMS and KIT kinase inhibitor. Proceedings of the National Academy of Sciences of the United States of America. 110, 5689-5694 (2013).
- Lipinski, C. A., Lombardo, F., Dominy, B. W., Feeney, P. J. Experimental and computational approaches to estimate solubility and permeability in drug discovery and development settings. Advanced Drug Delivery Reviews. 23, 3-25 (1997).
- Congreve, M., Carr, R., Murray, C., Jhoti, H. A 'Rule of Three' for fragment-based lead discovery. Drug Discovery Today. 8, 876-877 (2003).
- Chávez-Hernández, A. L., Sánchez-Cruz, N., Medina-Franco, J. L. A Fragment Library of Natural Products and its Comparative Chemoinformatic Characterization. Molecular Informatics. 39, 2000050(2020).
- Sreeramulu, S., et al. NMR quality control of fragment libraries for screening. Journal of Biomolecular NMR. , 00327-00329 (2020).
- Gao, J., et al. Automated NMR Fragment Based Screening Identified a Novel Interface Blocker to the LARG/RhoA Complex. PLoS One. 9, 88098(2014).
- Peng, C., et al. Fast and Efficient Fragment-Based Lead Generation by Fully Automated Processing and Analysis of Ligand-Observed NMR Binding Data. Journal of Medicinal Chemistry. 59, 3303-3310 (2016).
- Cox, O. B., et al. A poised fragment library enables rapid synthetic expansion yielding the first reported inhibitors of PHIP(2), an atypical bromodomain. Chemical Science. 7, 2322-2330 (2016).
- Hwang, T. L., Shaka, A. J. Water Suppression That Works. Excitation Sculpting Using Arbitrary Wave-Forms and Pulsed-Field Gradients. Journal of Magnetic Resonance, Series A. 112, 275-279 (1995).
- Gossert, A. D., Jahnke, W. NMR in drug discovery: A practical guide to identification and validation of ligands interacting with biological macromolecules. Progress in Nuclear Magnetic Resonance Spectroscopy. 97, 82-125 (2016).
- Sklenar, V., Bax, A. A new water suppression technique for generating pure-phase spectra with equal excitation over a wide bandwidth. Journal of Magnetic Resonance. 75, 378-383 (1987).
- Binas, O., et al. 19F NMR-Based Fragment Screening for 14 Different Biologically Active RNAs and 10 DNA and Protein Counter-Screens. ChemBioChem. , (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon