Method Article
Triagem de fragmentos baseada em RMN em uma amostra mínima, mas no modo de automação máxima
Neste Artigo
Resumo
A triagem baseada em fragmentos por RMN é um método robusto para identificar rapidamente ligantes de pequenas moléculas a biomacromoléculas (DNA, RNA ou proteínas). Protocolos descrevendo a preparação de amostras baseada em automação, experimentos de RMN e condições de aquisição e fluxos de trabalho de análise são apresentados. A técnica permite a exploração ideal de núcleos ativos de RMN de 1H e 19F para detecção.
Resumo
A triagem baseada em fragmentos (FBS) é um conceito bem validado e aceito dentro do processo de descoberta de medicamentos, tanto na academia quanto na indústria. A maior vantagem da triagem de fragmentos baseada em RMN é sua capacidade não apenas de detectar ligantes acima de 7-8 ordens de magnitude de afinidade, mas também de monitorar a pureza e a qualidade química dos fragmentos e, assim, produzir acertos de alta qualidade e mínimos falsos positivos ou falsos negativos. Um pré-requisito dentro do FBS é realizar o controle de qualidade inicial e periódico da biblioteca de fragmentos, determinando a solubilidade e a integridade química dos fragmentos em buffers relevantes e estabelecendo várias bibliotecas para cobrir diversos arcabouços para acomodar várias classes alvo de macromoléculas (proteínas/RNA/DNA). Além disso, uma extensa otimização do protocolo de triagem baseada em RMN com relação às quantidades de amostra, velocidade de aquisição e análise no nível de construto biológico/espaço de fragmento, no espaço de condição (tampão, aditivos, íons, pH e temperatura) e no espaço ligante (análogos do ligante, concentração do ligante) é necessária. Pelo menos no meio acadêmico, esses esforços de triagem até agora foram realizados manualmente de forma muito limitada, levando a uma disponibilidade limitada de infraestrutura de triagem não apenas no processo de desenvolvimento de medicamentos, mas também no contexto do desenvolvimento de sondas químicas. A fim de atender aos requisitos economicamente, fluxos de trabalho avançados são apresentados. Eles aproveitam o mais recente hardware avançado de última geração, com o qual a coleta de amostras líquidas pode ser preenchida de forma controlada por temperatura nos tubos de RMN de maneira automatizada. 1ºOs espectros baseados em ligantes de RMN H/19F são então coletados a uma dada temperatura. O trocador de amostras de alto rendimento (trocador de amostras HT) pode lidar com mais de 500 amostras em blocos com temperatura controlada. Isso, juntamente com ferramentas avançadas de software, acelera a aquisição e análise de dados. Além disso, a aplicação de rotinas de triagem em amostras de proteína e RNA são descritas para tornar ciente dos protocolos estabelecidos para uma ampla base de usuários em pesquisa biomacromolecular.
Introdução
A triagem baseada em fragmentos é atualmente um método comumente usado para identificar moléculas bastante simples e de baixo peso molecular (MW <250 Da) que mostram ligação fraca a alvos macromoleculares, incluindo proteínas, DNA e RNA. Os acertos iniciais das telas primárias servem como base para conduzir uma tela secundária de análogos maiores disponíveis comercialmente dos hits e, em seguida, utilizar estratégias de crescimento ou ligação de fragmentos baseadas em química. Para uma plataforma de descoberta de fármacos baseada em fragmentos (FBDD) bem-sucedida, em geral, um método biofísico robusto é necessário para detectar e caracterizar acertos fracos, uma biblioteca de fragmentos, um alvo biomolecular e uma estratégia para acompanhamento químico. Quatro métodos biofísicos comumente aplicados dentro das campanhas de descoberta de fármacos são ensaios de deslocamento térmico, ressonância plasmônica de superfície (SPR), cristalografia e espectroscopia de ressonância magnética nuclear (RMN).
A espectroscopia de RMN tem mostrado papéis variados dentro dos diferentes estágios do FBDD. Além de garantir a pureza química e a solubilidade dos fragmentos em uma biblioteca de fragmentos dissolvidos em um sistema tampão otimizado, os experimentos de RMN observados por ligantes podem detectar a ligação de fragmentos a um alvo com baixa afinidade e os experimentos de RMN observados no alvo podem delinear o epítopo de ligação do fragmento, permitindo estudos detalhados da relação estrutura-atividade. Dentro do mapeamento de epítopos, as mudanças de deslocamento químico baseadas em RMN não identificam apenas os sítios de ligação ortostérica, mas também os sítios alostéricos que podem ser crípticos e acessíveis apenas nos chamados estados conformacionais excitados do alvo biomolecular. Se o alvo biomolecular já se liga a um ligante endógeno, os acertos do fragmento identificado podem ser facilmente classificados como alostéricos ou ortostéricos através da realização de experimentos de competição baseados em RMN. A determinação da constante de dissociação (KD) da interação ligante-alvo é um aspecto importante no processo de FBDD. Titulações de deslocamento químico baseadas em RMN, tanto ligante quanto alvo observado, podem ser prontamente realizadas para determinar a KD. Uma grande vantagem da RMN é que os estudos de interação são realizados em solução e próximos a condições fisiológicas. Assim, todos os estados conformacionais para a análise da interação ligante/fragmento com seu alvo podem ser sondados. Além disso, as abordagens baseadas em RMN não são apenas restritas à triagem de proteínas solúveis bem dobradas, mas também estão sendo aplicadas para acomodar um espaço alvo maior, incluindo DNA, RNA, proteínas ligadas à membrana e intrinsecamente desordenadas1.
As bibliotecas de fragmentos são uma parte indispensável do processo FBDD. Em geral, os fragmentos atuam como precursores iniciais que eventualmente se tornam parte (subestrutura) do novo inibidor desenvolvido para um alvo biológico. Vários medicamentos (Venetoclax2, Vemurafenib3, Erdafitinib4, Pexidartnib5) foram relatados como tendo começado como fragmentos e agora são usados com sucesso nas clínicas. Tipicamente, os fragmentos são moléculas orgânicas de baixo peso molecular (<250 Da) com alta solubilidade aquosa e estabilidade. Uma biblioteca de fragmentos cuidadosamente elaborada, contendo tipicamente algumas centenas de fragmentos, já pode prometer uma exploração eficiente do espaço químico. A composição geral das bibliotecas de fragmentos evoluiu ao longo do tempo e, na maioria das vezes, foi derivada dissecando drogas conhecidas em fragmentos menores ou projetados computacionalmente. Essas diversas bibliotecas de fragmentos contêm principalmente aromáticos planos ou heteroátomos e aderem à Regra de Lipinski de 5 6, ou à Regra de tendência comercial atual de 3 7, mas evitam grupos reativos. Algumas bibliotecas de fragmentos também foram derivadas ou compostas por metabólitos altamente solúveis, produtos naturais e/ou seus derivados8. Um desafio geral colocado pela maioria das bibliotecas de fragmentos é a facilidade da química a jusante.
O Centro de Ressonância Magnética Biomolecular (BMRZ) da Goethe-University Frankfurt, é parceiro do iNEXT-Discovery (Infrastructure for NMR, EM and X-rays for Translational research-Discovery), um consórcio de infraestruturas de investigação estrutural para todos os investigadores europeus de todas as áreas da investigação bioquímica e biomédica. Dentro da iniciativa anterior do iNEXT, que terminou em 2019, uma biblioteca de fragmentos composta por 768 fragmentos foi criada com o objetivo de "fragmentos mínimos e diversidade máxima" cobrindo um grande espaço químico. Além disso, ao contrário de qualquer outra biblioteca de fragmentos, a biblioteca de fragmentos iNEXT também foi projetada com base no conceito de "fragmentos equilibrados" com o objetivo de facilitar a síntese a jusante de ligantes complexos e de alta afinidade e, doravante, conhecida como biblioteca interna (Diamond, Structural Genomic Consortium e iNEXT).
Estabelecer FBDD por RMN requer mão de obra, conhecimento e instrumentação. Na BMRZ, foram desenvolvidos fluxos de trabalho otimizados para dar suporte à assistência técnica à triagem de fragmentos por RMN. Estes incluem controle de qualidade e avaliação da solubilidade da biblioteca de fragmentos 9, otimização de buffer para os alvos escolhidos, triagem baseada em ligantes 1D observados em 1H ou 19F, experimentos de competição para diferenciar entre ligação ortostérica e alostérica, experimentos de RMN observados em alvos baseados em 2D para mapeamento de epítopos e para caracterizar a interação com um conjunto secundário de derivados dos acertos iniciais dos fragmentos. A BMRZ estabeleceu rotinas automatizadas para a análise, como também discutido anteriormente na literatura 10,11, de interações molécula-proteína de pequeno porte e possui toda a infraestrutura automatizada necessária para a triagem de fragmentos baseada em RMN. Ele implementou a NMR POR DIFERENÇA DE TRANSFERÊNCIA DE SATURAÇÃO (STD-NMR), ligante de água observado via espectroscopia de gradiente (waterLOGSY) e experimentos de relaxamento baseados em Carr-Purcell-Meiboom-Gill (baseados em CPMG) para identificar fragmentos dentro de uma ampla gama de regimes de afinidade, bem como instrumentação de RMN automatizada de última geração e software para descoberta de drogas. Enquanto a triagem de fragmentos baseada em RMN está bem estabelecida para proteínas, essa abordagem é menos comumente usada para encontrar novos ligantes interagindo com RNA e DNA. A BMRZ estabeleceu uma prova de conceito para novos protocolos que permitem a identificação de interações RNA/DNA de pequenas moléculas. Nas seções seguintes desta contribuição, a aplicação de rotinas de triagem em amostras de proteína e RNA é relatada para dar conhecimento aos protocolos estabelecidos para uma ampla base de usuários em pesquisa biomacromolecular.
Protocolo
1. Biblioteca de fragmentos
- Biblioteca interna de fragmentos
NOTA: No âmbito de uma das actividades de investigação conjuntas do iNEXT, foi desenvolvida uma biblioteca de fragmentos de primeira geração robusta e a jusante12 e, subsequentemente, uma segunda geração da biblioteca foi criada em colaboração com a Enamine e é conhecida como a biblioteca de fragmentos DSI (Diamond-SGC-iNEXT) (doravante denominada "biblioteca interna"). Esta biblioteca pode ser disponibilizada na BMRZ para fins de triagem.- Avaliar a biblioteca de fragmentos quanto à sua integridade e solubilidade usando um protocolo baseado em RMN previamente relatado9.
NOTA: A biblioteca interna é composta por 768 fragmentos com uma diversidade química muito alta (>200 Singletons). A realização da triagem em misturas fragmentadas pode acelerar significativamente a campanha de triagem; no entanto, o número de fragmentos em uma mistura é limitado devido à sobreposição de sinal no espectro de 1H-RMN. A maior diversidade química oferecida pela biblioteca interna permite a preparação de misturas contendo 12 fragmentos diferentes sem qualquer sobreposição significativa de deslocamento químico nos espectros de RMN de 1H observados. - 103 fragmentos dentro dos 768 fragmentos possuem um átomo de flúor. Para fins de triagem de 19 F, divida todos os 103 fragmentos que possuem um grupo de flúor em 5 misturas com base na sobreposição mínima de deslocamento químico de 19F. Para minimizar a sobreposição de sinal na triagem de 19F, use as informações de deslocamento químico de medições de composto único para projetar misturas com número máximo de fragmentos e mínima sobreposição de sinal. Cada mistura tem 20-21 fragmentos com deslocamentos químicos distintos de 19F, permitindo a atribuição inequívoca de fragmentos.
- Avaliar a biblioteca de fragmentos quanto à sua integridade e solubilidade usando um protocolo baseado em RMN previamente relatado9.
- Biblioteca de fragmentos definida/fornecida pelo usuário
- Realizar campanhas de triagem com a biblioteca de fragmentos definida ou fornecida pelo usuário; no entanto, as etapas a seguir precisam preceder a campanha de triagem.
- Se não for especificado pelo usuário previamente, execute o controle de qualidade baseado em RMN dos fragmentos (na BMRZ, ferramentas avançadas de software são usadas para isso; 9, capítulo 6.1.1).
- Verificar a solubilidade dos fragmentos em tampão de escolha para o alvo biomolecular, integridade da estrutura e concentração dos fragmentos antes do uso.
- Projetar a mistura para diminuir a sobreposição de sinal nos espectros de RMN e no tempo de medição.
- Projetar misturas de acordo com a etapa 4.2.
- Filtre fragmentos únicos ou um subconjunto de misturas em vez de toda a biblioteca.
2. Preparo da amostra
NOTA: A triagem de alto rendimento por RMN utiliza um robô de pipetagem para a preparação da amostra. Os espectros de RMN, mas também as estabilidades ao longo de vários dias de aquisição de sinal de proteínas, RNAs e DNA são extremamente sensíveis às flutuações de temperatura e, portanto, sistemas automatizados com temperatura controlada facilitarão muito a estabilidade das amostras que estão sendo pipetadas. Para este fim, um dispositivo adicional adicional, que funciona entre 4 a 40 °C, é acoplado ao robô de pipetagem para manuseio de líquidos das amostras de RMN em um ambiente com temperatura controlada.
- Preparação da mistura de ligantes
- Prepare amostras de triagem para medições de RMN usando um robô de preparação de amostras. A configuração flexível do robô permite uma ampla gama de aplicações (por exemplo, recuperação das amostras de tubos de RMN de volta para recipientes de armazenamento ou tarefas gerais de manuseio de líquidos). Tubos de RMN com diferentes diâmetros (1,7; 2,0; 2,5; 3,0 e 5,0 mm) podem ser utilizados. O sistema de robô de amostra, juntamente com o software de controle avançado, lê o código de barras atribuído para cada tipo de recipiente e executa o protocolo de enchimento de líquidos de forma otimizada.
- Para a preparação das misturas de ligantes da biblioteca interna, use frascos com código de barras. Os frascos com código de barras garantem o mais alto nível de confiabilidade e rastreabilidade ideal das amostras.
- Distribua 768 compostos em 8 placas de formato de 96 poços. A concentração de estoque de cada fragmento individual é de 50 mM em d6-DMSO/D2O (9:1). No total, prepare 64 misturas contendo 12 fragmentos cada. A concentração final de cada fragmento em uma mistura é de 4,2 mM.
NOTA: O robô de pipetagem pode acomodar uma variedade de tipos de recipientes com geometrias variadas (frascos de crio-amostrador ou automático, placas de 96 poços de profundidade redonda ou quadrada, frascos padrão com código de barras, tubos de microcentrífuga) e auxilia a execução eficiente da transferência de líquido para uma variedade de tubos e racks de RMN.

Figura 1: (A) Preparação de amostras de RMN de alto rendimento e robô de enchimento de tubo de RMN instalado na BMRZ. (B) Trocador de amostras de alto rendimento com racks individuais com temperatura controlada instalados em um espectrômetro de 600 MHz nas instalações da BMRZ. Clique aqui para ver uma versão maior desta figura.
- Preparação da amostra de triagem em branco (espectro do ligante de referência) e com alvo (ligante na presença do alvo)
- Para a preparação das amostras de triagem de RMN, na presença da biomolécula alvo (proteína/RNA/DNA) e da mistura de ligantes, use tubos trocadores de amostra HT de RMN de 3 mm selecionados do portfólio de tubos de RMN padrão da Bruker.
- Transfira o alvo biomolecular (por exemplo, triagem de 1H: RNA ou proteína de 10 μM) em um tampão de triagem definido para o tubo de RMN de 3 mm (volume final de 200 μL) manualmente ou usando o robô pipetador.
- Transfira 10 μL (por exemplo, triagem de 1H) da mistura de ligantes na próxima etapa usando o sistema robótico para os tubos de RMN de 3 mm com código de barras contendo a biomolécula alvo e misture usando o protocolo embutido do software de controle.
NOTA: O número do código de barras do tubo de RMN é incorporado de forma conveniente e automática ao conjunto de dados de RMN adquirido, garantindo assim um fluxo de trabalho orientado para ID sem qualquer mistura. O acessório de controle de temperatura do robô pipetador permite manter as amostras preparadas nos tubos de RMN sob temperatura constante.
- Condições e parâmetros definidos internamente
- Estabelecer condições ideais de tampão para realizar a triagem de RNA e proteína contra a biblioteca interna de fragmentos. As seguintes amostras condicionadas são usadas para o RNA na BMRZ: 25 mM KPi, 50 mM KCl, pH 6,2. Mg2+ é opcional.
- As proteínas são extremamente sensíveis para condições de solução; Use buffers ideais para o destino escolhido. Para cada um desses tampões, adquira espectros de referência adicionais dos ligantes para servir como branco para a análise.
- Condições especificadas pelo usuário
NOTA: Nos casos em que as condições internas estabelecidas não são adequadas para que os alvos sejam rastreados de um usuário em potencial, as seguintes etapas devem ser implementadas.- Realizar 1H-RMN do tampão sozinho para garantir a interferência mínima dos componentes do tampão na realização e análise dos experimentos de triagem observados pelo ligante. Os componentes interferentes poderiam ser adequadamente substituídos por equivalentes deuterados.
- Limitações na produção da amostra (quantidades-alvo)/condições e disponibilidade
NOTA: O isolamento ou a produção recombinante de certas biomacromoléculas pode, em certos casos, revelar-se desafiante e resultar numa disponibilidade limitada do alvo para prosseguir uma campanha de rastreio de medicamentos bem-sucedida. Em casos de disponibilidade limitada ou ilimitada dos alvos, as seguintes alternativas podem ser utilizadas para a realização de uma triagem de fragmentos baseada em RMN bem-sucedida.- Se limitado, use 19triagem baseada em F-NMR. Os ligantes fluorados típicos têm um único sinal de 19F; Portanto, use coquetéis com 25-30 fragmentos sem qualquer sobreposição de sinal. Há menos sinais para analisar, nenhuma interferência de sinal de componentes de buffer e menos sinais para confiar para a identificação de acertos.
- Se ilimitado, use telas maiores, como 1H-NMR. A biblioteca de fragmentos maior pode ser examinada. Normalmente, os fragmentos são compostos por mais de um próton, o que significa mais sinais nos quais confiar para a análise.
3. Condições de aquisição de RMN
- Condições internas geralmente definidas
- Espectrômetro equipado com trocador de amostras HT (Automação)
- Para triagem de alto rendimento, use placas de 96 poços que só podem ser medidas usando um trocador de amostras HT. O trocador de amostras HT também oferece a possibilidade de temperar cada rack individualmente.
- Para obter um sinal para ruído ideal, use um espectrômetro com uma sonda criogênica que seja resfriada por hélio ou nitrogênio. Um módulo automatizado de sintonia e correspondência (ATM) é necessário para a automação.
- Conjuntos de parâmetros & sequências de pulso
NOTA: Muitos experimentos de RMN podem caracterizar eventos de ligação. A identificação do acerto varia de acordo com a configuração experimental. Os experimentos a seguir são rotineiramente usados em campanhas de triagem da BMRZ. No entanto, podem ser feitas alterações para campanhas de triagem definidas pelo usuário e de acordo com as especificações do usuário.- Se estiver usando o software TopSpin, inclua o conjunto de parâmetros para experimentos baseados em ligantes: SCREEN_STD, SCREEN_T1R, SCREEN_T2 SCREEN_WLOGSY. O conjunto de parâmetros inclui todos os parâmetros necessários e as sequências de pulso: STD: stddiffesgp.3; T1ρ: t1rho_esgp2d; T2: cpmg_esgp2d; e waterLOGSY: ephogsygpno.2.
- Para todos os experimentos listados, use a escultura de excitação13 como supressão de água. Para uma referência, use 1D excitation sculpting (zgesgp). O número de varreduras depende da sensibilidade do sistema (intensidade do campo magnético e cabeça da sonda), da concentração da amostra e da escolha do experimento. A recomendação é: 1D com NS=64, T1ρ & T2 com NS=128, DST com NS=256 e waterLOGSY com NS= 384 ou 512.
- Para a triagem de 19 F, use os experimentos 1D e T2: 1D: F19CPD (pp=zgig) para 19 cabeças de sonda F{1 H} e F19(pp=zg) para 19cabeças de sonda F/1H; SCREEN_19F_T2 (pp = cpmgigsp).
- Use uma largura espectral de 220 ppm e uma frequência de excitação a -140 ppm. O tempo de experimento é entre 1 e 5 horas (garantir a estabilidade a longo prazo da biomacromolécula), dependendo do hardware e da concentração da amostra. Para T2, o tempo de CPMG deve alternar entre 0 ms e 200 ms.
- Processamento
- Registre os experimentos de DST, T1ρ e T2 como pseudo 2D. Para processar os dois espectros 1D únicos, o IconNMR usa o programa au proc_std com ou sem a opção relax. A primeira opção fornece a referência 1D e a diferença de dois espectros. A segunda opção produz dois espectros separados com tempo de relaxamento curto e longo. O waterLOGSY é um único 1D que deve ser faseado com um negativo para o sinal do solvente.
- Espectrômetro equipado com trocador de amostras HT (Automação)
- Condições específicas do usuário
- Adapte qualquer um dos parâmetros mencionados anteriormente às condições definidas pelo usuário. Por exemplo, se uma proteína fornecida pelo usuário não for estável na temperatura geralmente usada, experimentos de otimização podem ser conduzidos variando temperatura, concentração, condições de tampão, etc.
4. Análise dos dados
- Biblioteca de fragmentos QC (d6-DMSO/buffer específico) e quantificação
- CMC-q
NOTA: O controle de qualidade das bibliotecas de fragmentos é essencial antes do início das campanhas de triagem. Além disso, a estabilidade a longo prazo da biblioteca fragmentada precisa ser garantida para a aplicação de várias campanhas de triagem, razão pela qual a avaliação periódica da qualidade da biblioteca deve ser realizada. Para isso, os softwares integrados CMC-q e CMC-a da TopSpin são utilizados para avaliação de qualidade e quantidade. O CMC-q e o CMC-a são módulos de software dentro do Topspin que permitem uma aquisição suave, análise incluindo verificação de estrutura usando 1espectro de RMN-H obtido de pequenas moléculas orgânicas 9.- Para integridade, preparar amostras de avaliação com uma concentração de fragmento de 1 mM em d6-DMSO. Prepare amostras de forma automatizada com um robô de pipetagem preenchendo a coleta de amostras líquidas em um tubo de RMN de 3 mm.
- Para avaliação da solubilidade, utilizar amostra constituída por composto de 1 mM em tampão fosfato de sódio 50 mM pH 7,4, cloreto de sódio 150 mM, 90% H 2 O/ 10% D 2 O e 1 mMde sal ácido de sódio 3-(trimetilsilil)propiônico-2,2,3,3-d4 (TMSP-Na).
- Colete espectros de RMN a 298 K ou 293 K usando um espectrômetro de RMN de 600 MHz equipado com sonda criogênica de TCI de 5 mm de ressonância tripla e um trocador de amostras HT, que pode lidar com 579 amostras de uma só vez.
- Para configurar o software CMC-q, siga as instruções do manual do usuário, que implementa a criação de um usuário IconNMR, a ativação do FastLaneNMR e a alteração do trocador de amostra HT.
- Calibre o pulso de 90° e guarde-o na mesa de prosol TopSpin.
- Coloque a placa do poço de amostra 96 em uma das 5 posições do rack no trocador de amostras HT.
- Para carregar um arquivo SDF (arquivo de dados de estrutura) que deve conter sua estrutura química proposta, um identificador exclusivo e a posição no trocador de amostra HT de cada amostra em um lote, vá para Procurar na janela Configuração do CMC-q e clique em Abrir depois de selecionar um arquivo que termine em .sdf.
- Nas configurações de Automação de Lote CMC.q, defina o tipo de verificação que define o experimento que será medido, o usuário IconNMR e defina o Solvente.
- Defina arquivos SDF para o arquivo Path for SDF, o ID da molécula e a posição da amostra.
- Inicie a aquisição clicando em Iniciar. Clique em Iniciar aquisição novamente. A configuração do CMC-q também pode ser salva clicando em Salvar.
- Para obter uma descrição detalhada das etapas de configuração do CMC-q, siga as instruções do manual do usuário da Bruker.
- CMC-a
- Para CMC-a, use o módulo de software dentro do Topspin que permite a análise, incluindo a verificação da estrutura usando o espectro de RMN-1H obtido de pequenas moléculas orgânicas9.
- CMC-q
- Delineamento de misturas
NOTA: Um planejamento de mistura adequado desempenha um papel importante para a triagem usando RMN como plataforma. Um alto número de fragmentos por mistura permite uma triagem mais rápida, mas aumenta o risco de falsos positivos e negativos. Um número menor diminui esse risco, mas aumenta o tempo necessário para realizar a triagem. Em geral, uma sobreposição de sinal deve ser evitada ao criar misturas. Usando a biblioteca interna, isso pode ser negligenciado para a triagem de 1H, pois a biblioteca foi projetada especificamente para ser diversificada e mostrar pouca sobreposição de sinal, mantendo uma alta diversidade química. Isso, por sua vez, significa que nenhum procedimento especial de design precisa ser submetido para criar as 64 misturas.- Como a triagem 19F depende dos fragmentos da biblioteca interna que contêm flúor e a biblioteca não foi criada para reduzir a sobreposição de sinal para esses fragmentos específicos, projete uma mistura adequada.
- Medir espectros compostos únicos para todos os fragmentos contendo 19F.
- Observe as informações de deslocamento químico de cada sinal.
- De acordo com essas informações, escolha de 20 a 21 fragmentos por mistura. Isso, por sua vez, dá 5 misturas cada uma contendo 20-21 fragmentos sem sobreposição de sinal e permite uma análise semi-automatizada dos dados.
- Realizar a identificação de acertos dentro de uma interação biomacromolécula-ligante observada
NOTA: Existem diferentes definições de acerto entre o procedimento de triagem 19F e 1H. As seguintes identificações de acerto foram configuradas por nós e seguem regras específicas. O assunto da determinação de acertos é uma maneira muito subjetiva e pode diferir de usuário para usuário. No entanto, é de extrema importância que as regras para identificação de acertos não mudem uma vez acordadas para manter a validação e a credibilidade.- 1ºTela H
- Para determinar com confiança os acertos, adquira espectros 1D 1H, waterLOGSY e experimentos de relaxamento T2 tanto na presença quanto na ausência de alvo para identificar ligantes. Todos os três experimentos têm o potencial de mostrar um evento de ligação. Se um PSC maior que 6 Hz for visível nos espectros da amostra em comparação com os espectros em branco, isso é considerado como uma indicação de um acerto. O mesmo acontece se um forte sinal positivo no waterLOGSY, bem como uma redução de mais de 30% T2 nos espectros da amostra for visível. Eventos de ligação podem ser mostrados em todos os três experimentos, quando se compara a amostra contendo espectros com seus respectivos espectros em branco. No entanto, os eventos de ligação podem não ser visíveis nos três experimentos. Por causa disso, foi acordado que pelo menos dois dos eventos descritos anteriormente devem ocorrer para classificar um fragmento como um acerto de ligação.
- Use a ferramenta FBS no TopSpin para definir o estado dos fragmentos em vinculação, ambígua, desconhecida, agregada e não vinculação.
- Quando terminar com uma mistura, aprove-a dentro da ferramenta FBS.
- Na guia resumo dentro do projeto FBS, clique em Criar um relatório de triagem. Isso abrirá uma janela que cria um arquivo .xlsx. O usuário pode então optar por escolher entre todos os ligantes, ligantes vinculantes apenas, ligantes não ligantes apenas e ligantes ambíguos a serem relatados na planilha.
- 19ºTela F
- Para diferenciar entre não-ligante, ligante semanal e ligante forte, divida o quociente de integração entre a medida alvo de 200 ms e a medida em branco de 200 ms pelo quociente da medida alvo de 0 ms e a medida em branco de 0 ms é usada:
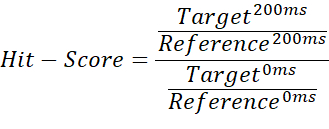
NOTA: Isso fornece valores que variam de 0 a ~1 (a pontuação de acertos), tornando possível atribuir limites para cada estado de vinculação. - Use a média da medida de referência de 200 ms como limite de linha de base, para marcar os casos em que a pontuação de acerto excede 1. Isso pode ocorrer se as integrais importadas contiverem valores negativos ou se a medida de referência for maior do que a medida de destino. Um hit-score de ≤ 0,67 é considerado um golpe fraco, < 0,33 um hit forte, e qualquer coisa > 0,67 como no-hit. Um exemplo é mostrado na Figura 2.
- Para diferenciar entre não-ligante, ligante semanal e ligante forte, divida o quociente de integração entre a medida alvo de 200 ms e a medida em branco de 200 ms pelo quociente da medida alvo de 0 ms e a medida em branco de 0 ms é usada:
- 1ºTela H
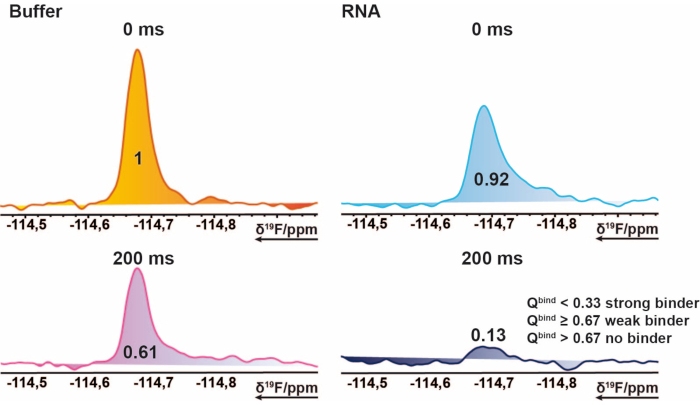
Figura 2: Identificação de acertos para a triagem de 19F. Seção de 19F CPMG NMR espectros de um composto exemplar. Essa representação pictórica explica as propriedades de um ligante. 19ºEspectros de F-CPMG de um composto adquirido de amostras de mistura na presença e ausência de RNA. Os valores representam os valores integrais normados do pico correspondente. Clique aqui para ver uma versão maior desta figura.
- Análise de dados
- Preparar dados para análise
NOTA: É importante que os dados adquiridos não apresentem falhas visíveis. Isso significa que os dados em que o shimming foi problemático ou a supressão de água foi insuficiente não devem ser considerados para análise. Em vez disso, recomenda-se registrar os dados novamente e certificar-se de que tudo está bem com a amostra (por exemplo, sem bolhas de ar), com a temperatura, o shimming e a supressão de água. A exatidão dos dados sempre pode ser avaliada ao comparar sinais DMSO. - 1ºTriagem H
- Para analisar dados de triagem de 1 H, use a ferramenta FBS (precisa delicença adicional) no TopSpin 4.0.9.
- Siga as instruções no manual da ferramenta FBS para começar com a análise de dados. As etapas a seguir resumem o procedimento relatado no manual.
- Armazene os dados de RMN da BMRZ de campanhas de triagem de modo que cada mistura de triagem diferente tenha seu próprio diretório no qual um subdiretório contém os diferentes experimentos medidos na amostra.
- Para usar a ferramenta FBS, armazene os espectros de referência que têm todos os dados salvos de amostras sem o alvo biomolecular, mas com as misturas, bem como o único composto medido em diferentes diretórios /nmr. Isso é importante, pois a ferramenta FBS solicitará o caminho do diretório de cada um individualmente.
NOTA: A ferramenta FBS reconhecerá um diretório como um projeto de triagem se os seguintes conjuntos de dados foram armazenados no mesmo diretório onde as misturas de uma amostra de triagem são armazenadas (csv, documentos XML FragmentScreen e arquivo BAK). - Ao usar o TopSpin 4.0.9, crie um caminho direto para o diretório que contém os dados adquiridos, o chamado DIR. Escolha o diretório /nmr no qual todas as misturas devem ter um diretório distinto.
- Para iniciar a ferramenta FBS de uma amostra filtrada, arraste o projeto FBS símbolo para o meio da janela TopSpin. No diretório escolhido, o símbolo do projeto FBS deve aparecer se os conjuntos de dados mencionados anteriormente foram copiados para ele.
- A janela Opções de triagem baseada em fragmento deve ser aberta automaticamente ao carregar um novo projeto FBS pela primeira vez. Nesta janela, escolha um arquivo de coquetel. O arquivo de coquetel é um arquivo csv contendo a atribuição do nome das misturas, o nome de cada fragmento e sua divisão nas misturas. Defina também uma pasta de espectros de ligantes de referência que tenha todos os espectros medidos dos fragmentos individuais. Por fim, defina uma pasta de experimento em branco de referência, que geralmente é a pasta que contém os conjuntos de dados das misturas sem o destino investigado.
- As Opções de Triagem Baseadas em Fragmentos têm uma aba chamada Tipos de espectros que permite definir os espectros investigados, bem como a cor para exibir os espectros . Defina o Spectype de acordo com os dados processados previamente. Na guia Exibir layout , defina os espectros que serão comparados entre si de acordo com suas especificações.
- Pressione Ok para iniciar o projeto FBS.
- Ao olhar para os dados, uma janela separada será aberta, resumindo todas as misturas de coquetéis e todos os ligantes de cada mistura em uma mesa. Ao clicar duas vezes em uma célula, os respectivos conjuntos de dados serão abertos, comparando, por exemplo, os espectros 1H 1D em branco com o conjunto de dados que contém o destino.
- Antes de atribuir ligantes, certifique-se de que os picos de referência (DMSO de todas as medições, bem como os compostos únicos) coincidam entre si e tenham o mesmo deslocamento químico. Se forem observadas diferenças, corrija-as usando a opção de processamento serial do TopSpin.
- A opção de processamento serial está na guia Processo em Avançado. Ele aplica alterações a todos os espectros selecionados de um conjunto de dados. Dessa forma, os Spectypes podem ser facilmente atribuídos a números de experimentos e todos os espectros podem ser deslocados de uma só vez para se alinharem com a referência.
- 19ºTriagem F
- Para a primeira análise das 19misturas F, crie um arquivo de integração para cada mistura. Para definir a região de integração, clique na função Integrar na guia Analisar . Certifique-se de que, para cada fragmento na mistura, uma região de integração clara para os 19F-singal correspondentes seja definida.
- Use o botão Salvar/Exportar regiões de integração para exportar o arquivo de integração para uso futuro. Salve todos os arquivos de integração usados em C:\Bruker\TopSpin4.0.9\exp\stan\nmr\lists\intrng ou o caminho correspondente do diretório de instalação do TopSpin.
- Para dados 19F, abra um conjunto de dados com ou sem o alvo investigado.
- Para carregar o arquivo de integração no espectro atual, abra a guia Analisar novamente, vá para Integrar e, usando o botão Ler/Importar regiões de integração, carregue o arquivo de integração correspondente. Isso carregará todas as regiões definidas desse arquivo no espectro atual.
- Salve e retorne para encontrar uma lista de todas as regiões integradas na guia Integrais . Copie isso em uma planilha ou qualquer outra ferramenta usada para a análise posterior dos dados.
- Repita este procedimento para cada mistura, com e sem alvo.
- Gerenciamento de Dados
- Para facilitar o uso e produtividade, tenha um fluxo de trabalho uniforme configurado para a análise e armazenamento posterior dos dados adquiridos. Para a triagem de 1H e 19F, use uma planilha projetada especificamente para cada um.
NOTA: Para a triagem de 1 H, isso foi usado puramente para o gerenciamento dedados e para resumir cada alvo, enquanto para a triagem de 19F foi usado o quociente explicado no capítulo 4.3 para rotular automaticamente cada fragmento como hit/no hit depois que os dados integrais foram copiados para ele. Isso reduz o risco de erro humano durante a análise, supondo que o arquivo foi configurado corretamente, e facilita o compartilhamento de informações, pois todas as informações importantes são reunidas em um só lugar em um arquivo que pode ser aberto por praticamente qualquer pessoa sem a necessidade de programas adicionais para dar uma olhada inicial nos dados.
- Para facilitar o uso e produtividade, tenha um fluxo de trabalho uniforme configurado para a análise e armazenamento posterior dos dados adquiridos. Para a triagem de 1H e 19F, use uma planilha projetada especificamente para cada um.
- Preparar dados para análise
Resultados
Controle de qualidade da biblioteca de fragmentos
Os fragmentos da biblioteca interna foram entregues como soluções estoque de 50 mM em 90% d6-DMSO e 10% D 2 O (10% de D2O garante a minimização da degradação do composto devido a repetidos ciclos de congelamento-descongelamento14). Amostras de composto único consistiram de ligante 1 mM em tampão fosfato 50 mM (25 mM KPi pH 6,2 + 50 mM KCl + 5 mM MgCl 2), pH 6,0 em 90% H 2 O/9% D2O/1% d6-DMSO. 1ºExperimentos de RMN-H de fragmentos da biblioteca iNEXT foram medidos em um espectrômetro de RMN de 500/600 MHz. Estes dados foram ainda utilizados para identificar os compostos isolados em campanhas de triagem de 1 H utilizando o software CMC-q que permite ao usuário adquirir espectros deforma automatizada e a análise addon CMC-a a qualidade (solubilidade e integridade) dos fragmentos foi avaliada. Os resultados da análise automatizada do CMC-a são apresentados como uma saída gráfica semelhante à representada na Figura 3. A saída gráfica mostra uma representação de uma placa de 96 poços. Um círculo de cor vermelha significa que este fragmento mostra inconsistência na estrutura ou concentração. Poços de cor verde indicam que o fragmento é consistente.
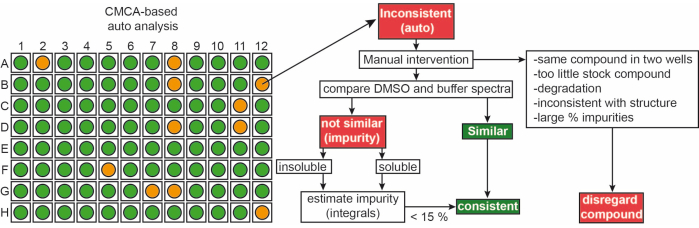
Figura 3: Controle de qualidade da biblioteca de fragmentos. Representação esquemática da saída automatizada baseada em CMC-a. Propriedades do fragmento, como concentração e integridade estrutural, são avaliadas. Verde significa consistente, laranja, neste caso, significa inconsistente. Fragmentos inconsistentes são revisados manualmente seguindo o fluxo de trabalho mostrado. Clique aqui para ver uma versão maior desta figura.
Aproximadamente, 65% e 35% dos fragmentos foram classificados como consistentes e inconsistentes, respectivamente, tanto no DMSO quanto no tampão. Além disso, 30% dos ligantes classificados inconsistentes tornaram-se consistentes após uma cuidadosa inspeção manual dos espectros9.
19F Dimensionamento de mistura
103 fragmentos contendo um ou vários grupos de flúor da biblioteca interna foram divididos em 5 misturas (A, B, C, D, E). Cada mistura tem de 20 a 21 fragmentos. Neste caso, as misturas tiveram de ser cuidadosamente concebidas para evitar a sobreposição de sinais. 19ºExperimentos de relaxação transversal F foram medidos para cada mistura que aplica trens de pulso CPMG. Esses experimentos podem ser modificados variando-se os atrasos de relaxamento. O deslocamento químico de 19F das misturas A-E pode ser visto na Figura 4.
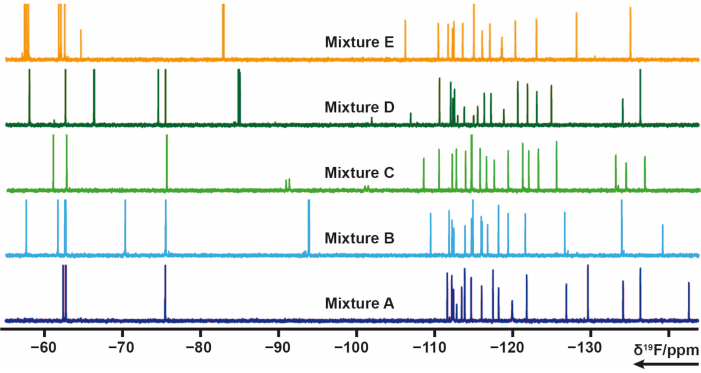
Figura 4: Espectrosde RMN 1D F 19 de amostras de mistura da biblioteca interna. Clique aqui para ver uma versão maior desta figura.
Preparo da amostra
O preparo da amostra no procedimento de triagem de 19F foi feito manualmente ou com pipetagem automatizada usando um robô pipetador. Os fragmentos de cada mistura apresentaram concentração de 2,5 mM em 90% d6-DMSO e 10% D2O. O volume final de uma amostra de triagem foi de 170 μL com 5% D2O como agente bloqueador. Cada mistura foi pipetada duas vezes, uma em um tampão contendo solução (sem alvo) e outra em um alvo contendo solução tampão. A relação entre alvo e fragmento foi ajustada para 1:1, resultando em uma concentração final/ligante de 50 μM. Além disso, as amostras de controle são a biomolécula alvo no tampão de triagem sem uma mistura para garantir a integridade do alvo, bem como uma amostra de controle com apenas tampão e D2O para garantir a qualidade do tampão.
Os dados de rastreamento de RMN de 19 F-1D e 19F-CPMG-T2 foram medidas conforme descrito na seção 3.1. Por exemplo, no caso do RNA, uma sequência de eco de retorno-salto (pp = zggpjrse,15) foi adquirida para a única amostra alvo em tampão.
Análise de dados
O procedimento de triagem de 19 F foi aplicado ao tiM riboswitch TPP de E. coli e à proteína tirosina quinase (PtkA) de M. tuberculosis, entre vários outros alvos 16. A biblioteca de triagem 19F tem 103 fragmentos que são divididos em 5 misturas rotuladas de Mix A a E. A preparação de amostras de triagem pode ser realizada manualmente sem o uso de um robô de pipetagem de amostras. 40 μM de solução contendo RNA thiM (condições tampão) foi misturada com 3,2 μL das misturas. Outras amostras-controle foram preparadas consistindo de tampão apenas, tampão com 5% de DMSO (previamente garantindo a estabilidade da biomacromolécula na presença da concentração desejada de DMSO) e tampão com RNA. Essas 13 amostras de triagem foram preparadas e transferidas para tubos de RMN de 3 mm. Os códigos de barras dos tubos de RMN são escaneados e cada mistura na presença e ausência de RNA, bem como amostras controle foram medidas de acordo com os experimentos de RMN 19F acima mencionados realizados em 298 K. A triagem do RNA thiM contra a biblioteca interna foi realizada realizando medições de T2 com CPMGs de 0 ms e 200 ms para cada amostra diferente. O shimming adequado e a supressão de água foram monitorados após o término das medições, comparando todos os picos de DMSO em termos de alargamento de linha e perda de intensidade de experimentos 1H 1D medidos adicionalmente para todas as amostras. O processamento dos espectros de relaxação obtidos de CPMG T219F foi realizado usando uma macro previamente preparada e automatizada no TopSpin, respectivamente. A análise dos dados foi realizada seguindo as instruções do protocolo. Os dados integrais obtidos do TopSpin (seguindo as instruções do protocolo) podem ser avaliados de forma rápida e fácil usando uma planilha pré-fabricada ou qualquer programa similar, definindo as condições e limites corretos. Conforme descrito anteriormente, os limites são úteis na definição de fichário, ligante fraco ou não-fichário. A Figura 5 mostra os resultados típicos dos espectros de CPMG de thiM RNA e PtkA, respectivamente. Em alguns casos, foi necessária uma nova revisão por especialistas.
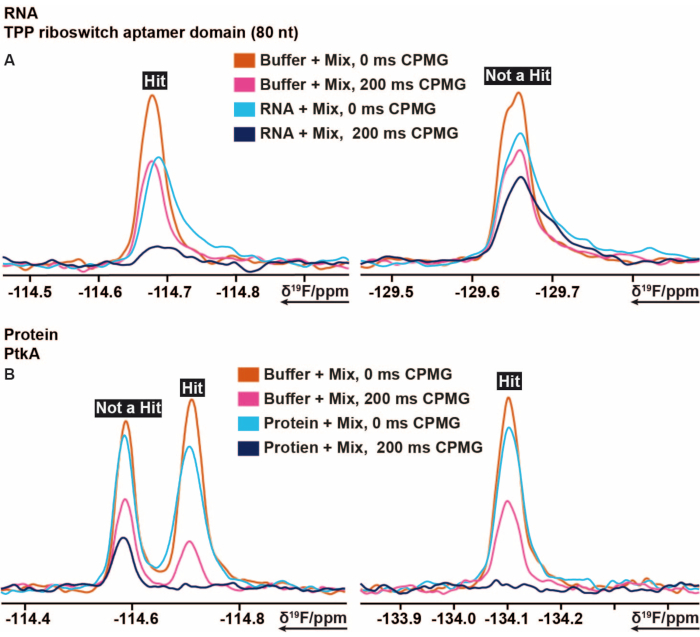
Figura 5: Recorte dos espectros de RMN de 19F CPMG mostrando as mudanças de intensidade obtidas a partir de diferentes tempos de atraso de experimentos baseados em CPMG . (A) Representação de um ligante (hit) e um não-ligante em triagem baseada em fragmentos 19F realizada em RNA thiM riboswitch TPP de E. coli. (B) Representação de um ligante e um não-ligante na triagem baseada em fragmentos de 19F realizada em PtkA de M. tuberculosis. Clique aqui para ver uma versão maior desta figura.
1H de Triagem
Delineamento de misturas
A biblioteca interna utilizada é tão diversificada que, para fins de triagem de 1 H, nenhum projeto demistura foi realizado. Isso significa que 64 misturas foram preparadas escolhendo aleatoriamente 12 para serem misturadas em uma mistura.
Preparo da Amostra
Para a triagem de 1 H de um RNA SARS-CoV-2 exemplar, a pipetagem automatizada usando um robô depipetagem foi realizada para preparar as amostras. Os fragmentos de cada mistura apresentaram concentração de 4,2 mM em 90% d6-DMSO e 10% D2O. O volume final de uma amostra de triagem foi de 200 μL com 5% D2O como agente bloqueador. 64 amostras contendo uma mistura diferente em KPi 25 mM, KCl 50 mM em pH 6,2 foram pipetadas sem RNA alvo. Respectivamente, 64 amostras foram pipetadas com RNA-alvo, cada uma contendo uma mistura diferente. A relação RNA:Ligante foi ajustada para 1:20, resultando em uma concentração de RNA de 10 μM e uma concentração de ligante de 200 μM.
Análise de dados
Para a análise de 1H, foi utilizada a ferramenta FBS no TopSpin. Para determinar se um fragmento é um sucesso, experimentos de relaxamento 1D chemical shift, waterLOGSY e T2 foram realizados. Para o relaxamento em T2 , uma diminuição na intensidade maior que 30% foi contada como um acerto, enquanto para o desvio químico um desvio maior que 6 Hz foi o ponto de corte. O waterLOGSY teve que mostrar uma mudança de sinal significativa (de negativo para positivo neste caso). Se qualquer um desses três critérios fosse positivo, um fragmento era contado como um acerto. Dois exemplos disso podem ser vistos na Figura 6.
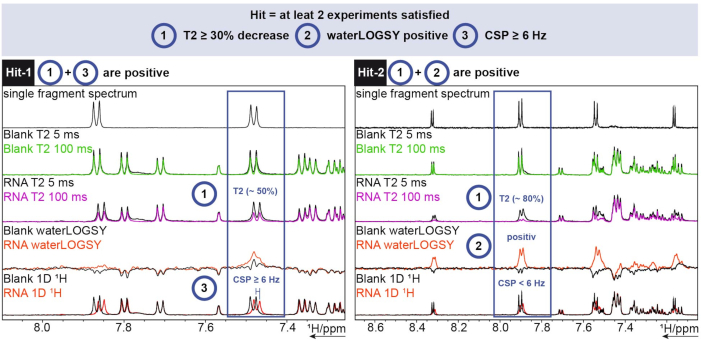
Figura 6: 1Triagem H realizada em um RNA SARS-CoV-2 exemplar mostrando critérios de determinação de acertos. Aquisição de três experimentos diferentes (1 H T2 CPMG (5/100 ms), waterLOGSY e 1D 1H). Clique aqui para ver uma versão maior desta figura.
Hit-1 mostra uma diminuição de T2 de ~50% e um CSP ≥ 6 Hz. O waterLOGSY não mostra uma mudança significativa o suficiente no sinal para também ser considerado positivo. Como dois em cada três experimentos são positivos, esse fragmento é contado como um acerto. Para Hit-2, o T2 mostra uma diminuição de ~80% da intensidade do sinal e uma clara mudança de sinal pode ser vista para o waterLOGSY. O CSP não é suficiente neste caso, mas como os dois critérios anteriores são positivos, ele ainda é contabilizado como um acerto.
Discussão
Versatilidade da triagem de fragmentos/fármacos baseada em RMN. A BMRZ implementou com sucesso instrumentação de RMN automatizada de última geração, bem como experimentos de RMN STD, waterLOGSY e relaxamento para identificar fragmentos dentro de uma ampla gama de regimes de afinidade para a descoberta de drogas. O hardware instalado inclui um robô de preparação de amostras de alto rendimento e armazenamento de amostras de alto rendimento, trocador e unidade de aquisição de dados associados a um espectrômetro de 600 MHz. Uma sonda criogênica recém-adquirida para 1 H, 19 F, 13C e 15 N garante a sensibilidade necessária para as medições propostas e permite o desacoplamento de1 H (1) durante a detecção de 19F. Esta sonda está conectada à última geração de console NMR que oferece a possibilidade de usar as ferramentas avançadas de software da Bruker, incluindo CMC-q, CMC-assist, CMC-se e FBS (incluído no TopSpin). A ferramenta de triagem baseada em fragmentos (FBS) está incluída na versão mais recente do TopSpin e ajuda a analisar os dados de alto rendimento que compreendem experimentos de relaxamento r STD, waterLOGSY, T2/T1. A coleta de amostras líquidas 1D 1H pode ser preenchida nos tubos de RMN de forma automatizada usando o robô de enchimento de amostras. Normalmente, um bloco de 96 tubos (3 mm) é preenchido em aproximadamente duas horas. Os racks de placas de 96 poços são posicionados diretamente no trocador de amostras HT, que lê o código de barras do bloco e atribui os tubos de RMN aos experimentos controlados pelo software de automação (IconNMR). Cinco racks de placas de 96 poços podem ser armazenados e programados no trocador de amostras HT ao mesmo tempo. A temperatura de cada um dos racks individuais pode ser controlada e regulada separadamente. Além disso, cada amostra individual pode ser pré-condicionada (pré-aquecimento e secagem do tubo para remoção da umidade condensada) à temperatura desejada antes da medição.
Adequação para uma ampla gama de aplicações. Uma das amplas aplicações dessa triagem automatizada baseada em RMN é identificar e desenvolver novos ligantes que se ligam a um alvo biomacromolecular (DNA/RNA/proteínas). Esses ligantes podem incluir inibidores ortostéricos e alostéricos que tipicamente se ligam de forma não covalente. Além disso, FBDD por RMN é tipicamente usado como um primeiro passo para selecionar compostos promissores, os requisitos a serem atendidos são a disponibilidade do alvo biomolecular em quantidades suficientes. Este objectivo divide-se em duas grandes tarefas.
A primeira tarefa é desenvolver e caracterizar uma biblioteca interna de fragmentos pelos seguintes motivos: controle de qualidade inicial e periódico, caracterização e quantificação de mais de 1000 fragmentos; determinação da solubilidade dos fragmentos em tampões otimizados para cada alvo, em particular para alvos proteicos; e o estabelecimento de várias bibliotecas para acomodar diversos arcabouços e estendendo-se para outras classes de macromoléculas. A segunda tarefa é integrar fluxos de trabalho para planejamento de fármacos baseado em fragmentos (FBDD) por RMN usando: triagem observada automatizada de ligantes 1D (1H e 19F observados); ensaios automatizados de substituição (experimentos de competição com ligante (natural) para diferenciar ligação ortostérica e alostérica; rastreios secundários automatizados com múltiplos fragmentos; rastreio automatizado de proteínas 2D e rastreio secundário de um conjunto de derivados em torno de um acerto inicial, utilizando a biblioteca EU-OPENSCREEN ou qualquer outra biblioteca; e reperfilamento da biblioteca da FDA em relação aos alvos escolhidos.
Adicionalmente, a metabotipagem de várias linhagens celulares (doença relevante) pode ser conduzida a fim de desvendar os mecanismos regulatórios que ligam o controle do ciclo celular e o metabolismo. Além disso, há caracterização funcional de elementos de regulação de RNA/DNA/proteínas in vivo e in vitro para otimização de construto/domínio (otimização de estabilidade para investigações estruturais (tampão, pH, temperatura e triagem salina), e uma extensão da triagem de fragmentos baseada em RMN para proteínas de membrana e proteínas intrinsecamente desordenadas, que geralmente são inacessíveis a outras técnicas.
Limitações. O uso de bibliotecas de fragmentos 19F e 1H tem seus prós e contras, poucos dos quais serão mencionados a seguir. O maior benefício das medições de 19F versus 1H é a velocidade do tempo de medição real e da análise subsequente, já que as misturas contêm quase o dobro do número de fragmentos e menos experimentos devem ser realizados. A análise de acompanhamento também é mais fácil para a triagem de 19F, pois não há interferência de buffers e, adicionalmente, oferece uma faixa de deslocamento químico mais ampla com quase nenhuma sobreposição de sinal para uma mistura de fragmentos idealmente projetada. Os espectros em si são muito simplificados, geralmente tendo apenas um ou dois sinais por fragmento, dependendo do número de átomos de flúor. A análise desses espectros pode, portanto, ser automatizada, novamente reduzindo o tempo. Isso tem o custo da diversidade química, pelo menos para a biblioteca usada neste estudo. Como apenas ~13% da biblioteca contém 19 F, mas naturalmente todos eles são utilizáveis na triagem de 1H, a diversidade dos fragmentos de triagem de 19F será menor. Isso poderia ser contornado usando bibliotecas 19F especificamente projetadas com mais fragmentos e maior diversidade química. Outra desvantagem para a triagem de 19F é o baixo número de sinais por fragmento. Os fragmentos geralmente são compostos por mais de um átomo de hidrogênio. Portanto, experimentosde triagem observados em 1 H podem contar com sinais diferentes para o mesmo fragmento para detectar ligação. Isso dá um maior grau de confiança ao identificar acertos para a triagem de 1H, enquanto a triagem de 19F deve se basear em um ou dois sinais dados por fragmento.
Um relato detalhado sobre a moderna instrumentação automatizada de triagem de fragmentos baseada em RMN, software e métodos de análise e protocolos dos mesmos foi apresentado. O hardware instalado inclui um robô de preparação de amostras de alto rendimento e uma unidade de armazenamento, trocador e aquisição de dados de amostras de alto rendimento associada a um espectrômetro de 600 MHz. Uma cabeça de sonda criogênica recentemente instalada para 1 H, 19 F, 13 C e 15 N garante a sensibilidade necessária para as medições propostas e permite o desacoplamento de 1H durante a detecção de 19F. Além disso, a última geração de console de RMN oferece a possibilidade de usar software analítico avançado para auxiliar a aquisição e análise em tempo real. A tecnologia discutida acima, os fluxos de trabalho e os protocolos descritos devem promover um sucesso notável para os usuários que buscam FBS por RMN.
Divulgações
Nenhum.
Agradecimentos
Este trabalho foi apoiado pelo iNEXT-Discovery, projeto número 871037, financiado pelo programa Horizonte 2020 da Comissão Europeia.
Materiais
| Name | Company | Catalog Number | Comments |
| Bruker Avance III HD | Bruker | 600 MHz NMR Spectrometer | |
| Matrix Clear Polypropylene 2D Barcoded Open-Top Storage Tubes | 3731-11 0.75ML V-BOTTOM TUBE/LATCH RACK | ThermoFisher Scientific | Barcoded Tubes |
| Matrix SepraSeal und DuraSeal& | 4463 Cap Mat, SeptraSeal 10/CS | ThermoFisher Scientific | |
| SampleJet | Bruker | HT Sample Changer | |
| SamplePro Tube | Bruker | Pipetting Robot |
Referências
- Yanamala, N., et al. NMR-Based Screening of Membrane Protein Ligands. Chemical Biology & Drug Design. 75, 237-256 (2010).
- Souers, A. J., et al. ABT-199, a potent and selective BCL-2 inhibitor, achieves antitumor activity while sparing platelets. Nature Medicine. 19, 202-208 (2013).
- Su, M. C., Te Chang, C., Chu, C. H., Tsai, C. H., Chang, K. Y. An atypical RNA pseudoknot stimulator and an upstream attenuation signal for -1 ribosomal frameshifting of SARS coronavirus. Nucleic Acids Research. 33, 4265-4275 (2005).
- Perera, T. P. S., et al. Discovery & pharmacological characterization of JNJ-42756493 (Erdafitinib), a functionally selective small-molecule FGFR family inhibitor. Molecular Cancer Therapeutics. 16, 1010-1020 (2017).
- Zhang, C., et al. Design and pharmacology of a highly specific dual FMS and KIT kinase inhibitor. Proceedings of the National Academy of Sciences of the United States of America. 110, 5689-5694 (2013).
- Lipinski, C. A., Lombardo, F., Dominy, B. W., Feeney, P. J. Experimental and computational approaches to estimate solubility and permeability in drug discovery and development settings. Advanced Drug Delivery Reviews. 23, 3-25 (1997).
- Congreve, M., Carr, R., Murray, C., Jhoti, H. A 'Rule of Three' for fragment-based lead discovery. Drug Discovery Today. 8, 876-877 (2003).
- Chávez-Hernández, A. L., Sánchez-Cruz, N., Medina-Franco, J. L. A Fragment Library of Natural Products and its Comparative Chemoinformatic Characterization. Molecular Informatics. 39, 2000050 (2020).
- Sreeramulu, S., et al. NMR quality control of fragment libraries for screening. Journal of Biomolecular NMR. , 00327-00329 (2020).
- Gao, J., et al. Automated NMR Fragment Based Screening Identified a Novel Interface Blocker to the LARG/RhoA Complex. PLoS One. 9, 88098 (2014).
- Peng, C., et al. Fast and Efficient Fragment-Based Lead Generation by Fully Automated Processing and Analysis of Ligand-Observed NMR Binding Data. Journal of Medicinal Chemistry. 59, 3303-3310 (2016).
- Cox, O. B., et al. A poised fragment library enables rapid synthetic expansion yielding the first reported inhibitors of PHIP(2), an atypical bromodomain. Chemical Science. 7, 2322-2330 (2016).
- Hwang, T. L., Shaka, A. J. Water Suppression That Works. Excitation Sculpting Using Arbitrary Wave-Forms and Pulsed-Field Gradients. Journal of Magnetic Resonance, Series A. 112, 275-279 (1995).
- Gossert, A. D., Jahnke, W. NMR in drug discovery: A practical guide to identification and validation of ligands interacting with biological macromolecules. Progress in Nuclear Magnetic Resonance Spectroscopy. 97, 82-125 (2016).
- Sklenar, V., Bax, A. A new water suppression technique for generating pure-phase spectra with equal excitation over a wide bandwidth. Journal of Magnetic Resonance. 75, 378-383 (1987).
- Binas, O., et al. 19F NMR-Based Fragment Screening for 14 Different Biologically Active RNAs and 10 DNA and Protein Counter-Screens. ChemBioChem. , (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados