Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Identificación de empalme alternativo y poliadenilación en datos de RNA-seq
En este artículo
Resumen
El empalme alternativo (AS) y la poliadenilación alternativa (APA) amplían la diversidad de isoformas de transcripción y sus productos. Aquí, describimos protocolos bioinformáticos para analizar RNA-seq a granel y ensayos de secuenciación final 3' para detectar y visualizar AS y APA que varían según las condiciones experimentales.
Resumen
Además del análisis típico de RNA-Seq para medir la expresión génica diferencial (DGE) en condiciones experimentales / biológicas, los datos de RNA-seq también se pueden utilizar para explorar otros mecanismos reguladores complejos a nivel de exón. El empalme alternativo y la poliadenilación juegan un papel crucial en la diversidad funcional de un gen al generar diferentes isoformas para regular la expresión génica a nivel post-transcripcional, y limitar los análisis a todo el nivel del gen puede pasar por alto esta importante capa reguladora. Aquí, demostramos análisis detallados paso a paso para la identificación y visualización del uso diferencial del exón y el sitio de poliadenilación en todas las condiciones, utilizando Bioconductor y otros paquetes y funciones, incluidos DEXSeq, diffSplice del paquete Limma y rMATS.
Introducción
RNA-seq ha sido ampliamente utilizado a lo largo de los años, generalmente para estimar la expresión génica diferencial y el descubrimiento de genes1. Además, también se puede utilizar para estimar el uso variable del nivel de exón debido a que los genes expresan diferentes isoformas, lo que contribuye a una mejor comprensión de la regulación génica a nivel post-transcripcional. La mayoría de los genes eucariotas generan diferentes isoformas mediante empalme alternativo (AS) para aumentar la diversidad de la expresión del ARNm. Los eventos AS se pueden dividir en diferentes patrones: salto de exones completos (SE) donde un exón ("casete") se elimina completamente de la transcripción junto con sus intrones flanqueantes; selección alternativa (donante) del sitio de empalme de 5' (A5SS) y selección alternativa 3' (aceptor) del sitio de empalme (A3SS) cuando dos o más sitios de empalme están presentes en cada extremo de un exón; retención de intrones (RI) cuando un intrón se retiene dentro de la transcripción de ARNm maduro y exclusión mutua del uso de exones (MXE) donde solo uno de los dos exones disponibles puede ser retenido a la vez 2,3. La poliadenilación alternativa (APA) también juega un papel importante en la regulación de la expresión génica utilizando sitios alternativos de poli (A) para generar múltiples isoformas de ARNm a partir de una sola transcripción4. La mayoría de los sitios de poliadenilación (pAs) se encuentran en la región 3' no traducida (3' UTRs), generando isoformas de ARNm con diversas longitudes 3' UTR. Como el 3' UTR es el eje central para reconocer elementos reguladores, diferentes longitudes 3' UTR pueden afectar la localización, estabilidad y traducción del ARNm5. Hay una clase de ensayos de secuenciación final 3' optimizados para detectar APA que difieren en los detalles del protocolo6. La canalización descrita aquí está diseñada para PolyA-seq, pero se puede adaptar para otros protocolos como se describe.
En este estudio, presentamos una tubería de métodos de análisis de exones diferenciales7,8 (Figura 1), que se pueden dividir en dos grandes categorías: basados en exones (DEXSeq9, diffSplice10) y basados en eventos (replicate Multivariate Analysis of Transcript Splicing (rMATS)11). Los métodos basados en exones comparan el cambio de pliegue a través de las condiciones de los exones individuales, contra una medida del cambio general del pliegue genético para llamar al uso de exones expresado diferencialmente, y a partir de eso calcular una medida a nivel de gen de la actividad de AS. Los métodos basados en eventos utilizan lecturas de unión de expansión exón-intrón para detectar y clasificar eventos de empalme específicos, como la omisión de exón o la retención de intrones, y distinguir estos tipos de AS en la salida3. Por lo tanto, estos métodos proporcionan vistas complementarias para un análisis completo de la EA12,13. Se seleccionaron DEXSeq (basado en el paquete DESeq214 DGE) y diffSplice (basado en el paquete Limma10 DGE) para el estudio, ya que se encuentran entre los paquetes más utilizados para el análisis de empalme diferencial. rMATS fue elegido como un método popular para el análisis basado en eventos. Otro método popular basado en eventos es MISO (mezcla de isoformas)1. Para APA adaptamos el enfoque basado en exones.
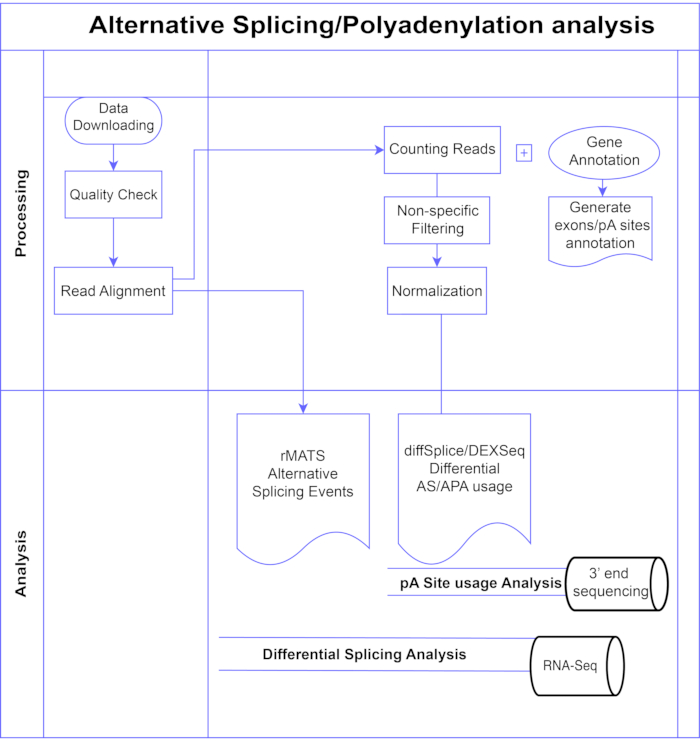
Figura 1. Pipeline de análisis. Diagrama de flujo de los pasos utilizados en el análisis. Los pasos incluyen: obtener los datos, realizar controles de calidad y alineación de lecturas seguidas de contar lecturas utilizando anotaciones para exones conocidos, intrones y sitios pA, filtrado para eliminar recuentos bajos y normalización. Los datos de PolyA-seq se analizaron para sitios de pA alternativos utilizando métodos diffSplice/DEXSeq, RNA-Seq a granel se analizaron para splicing alternativo a nivel de exón con métodos diffSplice/DEXseq, y los eventos de AS se analizaron con rMATS. Haga clic aquí para ver una versión más grande de esta figura.
Los datos de RNA-seq utilizados en este estudio fueron adquiridos de Gene Expression Omnibus (GEO) (GSE138691)15. Utilizamos datos de ARN-seq de ratón de este estudio con dos grupos de condiciones: tipo salvaje (WT) y knockout tipo 1 similar a Muscleblind (Mbnl1 KO) con tres réplicas cada uno. Para demostrar el análisis diferencial de uso del sitio de poliadenilación, obtuvimos datos de PolyA-seq de fibroblastos embrionarios de ratón (MEF) (GEO Accesion GSE60487)16. Los datos tienen cuatro grupos de condiciones: tipo salvaje (WT), tipo ciego muscular tipo 1 / tipo 2 doble knockout (Mbnl1/2 DKO), Mbnl 1/2 DKO con derribo Mbnl3 (KD) y Mbnl1/2 DKO con control Mbnl3 (Ctrl). Cada grupo de condición consta de dos réplicas.
| Adhesión al GEO | Número de ejecución de SRA | Nombre de la muestra | Condición | Replicar | Tejido | Secuenciación | Longitud de lectura | |
| RNA-Seq | GSM4116218 | SRR10261601 | Mbnl1KO_Thymus_1 | Mbnl1 nocaut | Rep 1 | Timo | Extremo emparejado | 100 pb |
| GSM4116219 | SRR10261602 | Mbnl1KO_Thymus_2 | Mbnl1 nocaut | Rep 2 | Timo | Extremo emparejado | 100 pb | |
| GSM4116220 | SRR10261603 | Mbnl1KO_Thymus_3 | Mbnl1 nocaut | Rep 3 | Timo | Extremo emparejado | 100 pb | |
| GSM4116221 | SRR10261604 | WT_Thymus_1 | Tipo salvaje | Rep 1 | Timo | Extremo emparejado | 100 pb | |
| GSM4116222 | SRR10261605 | WT_Thymus_2 | Tipo salvaje | Rep 2 | Timo | Extremo emparejado | 100 pb | |
| GSM4116223 | SRR10261606 | WT_Thymus_3 | Tipo salvaje | Rep 3 | Timo | Extremo emparejado | 100 pb | |
| 3P-seq | GSM1480973 | SRR1553129 | WT_1 | Tipo salvaje (WT) | Rep 1 | Fibroblastos embrionarios de ratón (MEF) | Extremo único | 40 pb |
| GSM1480974 | SRR1553130 | WT_2 | Tipo salvaje (WT) | Rep 2 | Fibroblastos embrionarios de ratón (MEF) | Extremo único | 40 pb | |
| GSM1480975 | SRR1553131 | DKO_1 | Mbnl 1/2 doble knockout (DKO) | Rep 1 | Fibroblastos embrionarios de ratón (MEF) | Extremo único | 40 pb | |
| GSM1480976 | SRR1553132 | DKO_2 | Mbnl 1/2 doble knockout (DKO) | Rep 2 | Fibroblastos embrionarios de ratón (MEF) | Extremo único | 40 pb | |
| GSM1480977 | SRR1553133 | DKOsiRNA_1 | Mbnl 1/2 doble knockout con Mbnl 3 siRNA (KD) | Rep 1 | Fibroblastos embrionarios de ratón (MEF) | Extremo único | 40 pb | |
| GSM1480978 | SRR1553134 | DKOsiRNA_2 | Mbnl 1/2 doble knockout con Mbnl 3 siRNA (KD) | Rep 2 | Fibroblastos embrionarios de ratón (MEF) | Extremo único | 36 pb | |
| GSM1480979 | SRR1553135 | DKONTsiRNA_1 | Mbnl 1/2 doble knockout con siRNA no dirigido (Ctrl) | Rep 1 | Fibroblastos embrionarios de ratón (MEF) | Extremo único | 40 pb | |
| GSM1480980 | SRR1553136 | DKONTsiRNA_2 | Mbnl 1/2 doble knockout con siRNA no dirigido (Ctrl) | Rep 2 | Fibroblastos embrionarios de ratón (MEF) | Extremo único | 40 pb |
Tabla 1. Resumen de los conjuntos de datos RNA-Seq y PolyA-seq utilizados para el análisis.
Protocolo
1. Instalación de herramientas y paquetes R utilizados en el análisis
- Conda es un administrador de paquetes popular y flexible que permite la instalación conveniente de paquetes con sus dependencias en todas las plataformas. Use 'Anaconda' (administrador de paquetes conda) para instalar 'conda' que se puede usar para instalar las herramientas / paquetes necesarios para el análisis.
- Descargue 'Anaconda' de acuerdo con los requisitos del sistema de https://www.anaconda.com/products/individual#Downloads e instálelo siguiendo las instrucciones en el instalador gráfico. Instale todos los paquetes necesarios usando 'conda' escribiendo lo siguiente en la línea de comandos de Linux.
conda install -c daler sratoolkit
conda install -c conda-forge parallel
conda install -c bioconda star bowtie fastqc rmats rmats2sashimiplot samtools fasterq-dump cutadapt bedtools deeptools - Para descargar todos los paquetes de R utilizados en el protocolo, escriba el código siguiente en la consola de R (iniciada en la línea de comandos de Linux escribiendo 'R') o en la consola de Rstudio.
bioc_packages<- c("DEXSeq", "Rsubread", "EnhancedVolcano", "edgeR", "limma", "maser","GenomicRanges")
packages<- c("magrittr", "rtracklayer", "tidyverse", "openxlsx", "BiocManager")
#Install if not already installed
installed_packages<-packages%in% rownames(installed.packages())
installed_bioc_packages<-bioc_packages%in% rownames(installed.packages())
if(any(installed_packages==FALSE)) {
install.packages(packages[!installed_packages],dependencies=TRUE)
BiocManager::install(packages[!installed_bioc_packages], dependencies=TRUE)
}
NOTA: En este protocolo computacional, los comandos se darán como archivos de R Notebook (archivos con extensión ". Rmd"), archivos de código R (archivos con extensión ". R"), o scripts de shell Bash de Linux (archivos con extensión ".sh"). Los archivos de R Notebook (Rmd) deben abrirse en RStudio usando File| Abra Archivo..., y los fragmentos de código individuales (que pueden ser comandos de R o comandos de shell Bash) se ejecutan interactivamente haciendo clic en la flecha verde en la parte superior derecha. Los archivos de código R se pueden ejecutar abriendo en RStudio, o en la línea de comandos de Linux anteponiendo "Rscript", por ejemplo, Rscript example.R. Los scripts de shell se ejecutan en la línea de comandos de Linux anteponiendo el script con el comando "sh", por ejemplo.sh example.sh.
2. Análisis de empalme alternativo (AS) utilizando RNA-seq
- Descarga y preprocesamiento de datos
NOTA: Los fragmentos de código anotados a continuación están disponibles en el archivo de código suplementario "AS_analysis_RNASeq.rmd", para seguir los pasos individuales de forma interactiva, y también se proporcionan como un script bash para ejecutarse por lotes en la línea de comandos de Linux (sh downloading_data_preprocessing.sh).- Descarga de los datos sin procesar.
- Descargue los datos sin procesar de Sequence Read Archive (SRA) utilizando el comando 'prefetch' del kit de herramientas SRA (v2.10.8)17. Proporcione los ID de acceso de SRA en secuencia en el siguiente comando para descargarlos en paralelo utilizando la utilidad paralelaGNU 18. Para descargar archivos SRA de identificadores de acceso de SRR10261601 a SRR10261606 en paralelo, use lo siguiente en la línea de comandos de Linux.
seq 10261601 10261606 | parallel prefetch SRR{} - Extraiga los archivos fastq del archivo utilizando la función 'fastq-dump' del kit de herramientas SRA. Utilice GNU paralelo y dé los nombres de todos los archivos SRA juntos.
parallel -j 3 fastq-dump --gzip --skip-technical --read-filter pass --dumpbase --split-e --clip --origfmt {} ::: - Descargue el genoma de referencia y las anotaciones para Mouse (ensamblaje del genoma GRCm39) desde www.ensembl.org utilizando lo siguiente en la línea de comandos de Linux.
wget -nv -O annotation.gtf.gz http://ftp.ensembl.org/pub/release-103/gtf/mus_musculus/Mus_musculus.GRCm39.103.gtf.gz \ && gunzip -f annotation.gtf.gz
wget -nv -O genome.fa.gz http://ftp.ensembl.org/pub/release-103/fasta/mus_musculus/dna/Mus_musculus.GRCm39.dna.primary_assembly.fa.gz \ && gunzip -f genome.fa.gz
GTF=$(readlink -f annotation.gtf)
GENOME=$(readlink -f genome.fa)
- Descargue los datos sin procesar de Sequence Read Archive (SRA) utilizando el comando 'prefetch' del kit de herramientas SRA (v2.10.8)17. Proporcione los ID de acceso de SRA en secuencia en el siguiente comando para descargarlos en paralelo utilizando la utilidad paralelaGNU 18. Para descargar archivos SRA de identificadores de acceso de SRR10261601 a SRR10261606 en paralelo, use lo siguiente en la línea de comandos de Linux.
- Preprocesamiento y mapeo de lecturas al ensamblaje del genoma
- Control de calidad. Evalúe la calidad de las lecturas sin procesar con FASTQC (FASTQ Quality Check v0.11.9)19. Cree una carpeta de salida y ejecute fastqc con paralelo en varios archivos fasta de entrada. Este paso generará un informe de calidad para cada muestra. Examine los informes para asegurarse de que la calidad de las lecturas es aceptable antes de realizar un análisis adicional. (Consulte el manual del usuario para comprender los informes en https://www.bioinformatics.babraham.ac.uk/projects/fastqc/)
mkdir fastqc_out
parallel "fastqc {} -o fastqc_out" ::: $RAW_DATA/*.fastq.gz
NOTA: Si es necesario, realice el recorte del adaptador con 'cutadapt'20 o 'trimmomatic'21 para eliminar la secuenciación en los adaptadores de flanqueo, que varía según el tamaño del fragmento de ARN y la longitud de lectura. En este análisis nos saltamos este paso ya que la fracción de lecturas afectadas fue mínima. - Leer alineación. El siguiente paso en el preprocesamiento incluye el mapeo de las lecturas al genoma de referencia. En primer lugar, construya el índice para el genoma de referencia utilizando la función 'genomeGenerate' de STAR22 y luego alinee las lecturas sin procesar con la referencia (alternativamente, los índices preconstruidos están disponibles en el sitio web de STAR y se pueden usar directamente para la alineación). Ejecute los siguientes comandos en la línea de comandos de Linux.
#Build STAR index
GDIR=STAR_indices
mkdir $GDIR
STAR --runMode genomeGenerate --genomeFastaFiles $GENOME --sjdbGTFfile $GTF --runThreadN 8 --genomeDir $GDIR
ODIR=results/mapping
mkdir -p $ODIR
#Align reads to the genome
for fq1 in $RAW_DATA/*R1.fastq.gz;
do
fq2=$(echo $fq1| sed 's/1.fastq.gz/2.fastq.gz/g');
OUTPUT=$(basename ${fq1}| sed 's/R1.fastq.gz//g');
STAR --genomeDir $GDIR \
--runThreadN 12 \
--readFilesCommand zcat \
--readFilesIn ${fq1}${fq2}\
--outFileNamePrefix $ODIR\/${OUTPUT} \
--outSAMtype BAM SortedByCoordinate \
--outSAMunmapped Within \
--outSAMattributes Standard
Done
NOTA: El alineador STAR generará y ordenará archivos BAM (mapa de alineación binaria) para cada muestra después de la alineación de lectura. Los archivos Bam deben ordenarse antes de continuar con los pasos adicionales.
- Control de calidad. Evalúe la calidad de las lecturas sin procesar con FASTQC (FASTQ Quality Check v0.11.9)19. Cree una carpeta de salida y ejecute fastqc con paralelo en varios archivos fasta de entrada. Este paso generará un informe de calidad para cada muestra. Examine los informes para asegurarse de que la calidad de las lecturas es aceptable antes de realizar un análisis adicional. (Consulte el manual del usuario para comprender los informes en https://www.bioinformatics.babraham.ac.uk/projects/fastqc/)
- Descarga de los datos sin procesar.
- Preparación de anotaciones Exon.
- Ejecute el archivo de código complementario "prepare_mm_exon_annotation. R" con la anotación descargada en formato GTF (formato de transferencia génica) para preparar las anotaciones. Para ejecutarlo, escriba lo siguiente en la línea de comandos de Linux.
Rscript prepare_mm_exon_annotation.R annotation.gtf
NOTA: El archivo GTF contiene varias entradas de exón para diferentes isoformas. Este archivo se utiliza para "contraer" los múltiples ID de transcripción para cada exón. Es un paso importante definir los contenedores de conteo de exones.
- Ejecute el archivo de código complementario "prepare_mm_exon_annotation. R" con la anotación descargada en formato GTF (formato de transferencia génica) para preparar las anotaciones. Para ejecutarlo, escriba lo siguiente en la línea de comandos de Linux.
- El conteo se lee. El siguiente paso es contar el número de lecturas asignadas a diferentes transcripciones/exones. Ver archivo complementario: "AS_analysis_RNASeq.Rmd".
- Cargar las bibliotecas necesarias:
packages<- c("Rsubread","tidyverse", "magrittr", "EnhancedVolcano", "edgeR","openxlsx")
invisible(lapply(packages, library, character.only=TRUE)) - Cargue el archivo de anotación procesado obtenido del paso anterior (2.2).
load("mm_exon_anno.RData") - Lea todos los archivos bam obtenidos en el paso 2.2.2 como entrada para que 'featureCounts' cuente las lecturas. Lea la carpeta que contiene los archivos bam enumerando primero cada archivo del directorio que termina con .bam. Utilice 'featureCounts' del paquete Rsubread que toma archivos bam y anotación GTF procesada (referencia) como entrada para generar una matriz de recuentos asociados con cada característica con filas que representan exones (características) y columnas que representan muestras.
countData <- dir("bams", pattern=".bam$", full.names=T) %>%
featureCounts(annot.ext=anno,
isGTFAnnotationFile=FALSE,
minMQS=0,useMetaFeatures=FALSE,
allowMultiOverlap=TRUE,
largestOverlap=TRUE,
countMultiMappingReads=FALSE,
primaryOnly=TRUE,
isPairedEnd=TRUE,
nthreads=12) - A continuación, realice un filtrado no específico para eliminar exones poco expresados ("no específico" indica que la información de la condición experimental no se utiliza en el filtrado, para evitar sesgos de selección). Transforme los datos de escala sin procesar a recuentos por millón (cpm) utilizando la función cpm del paquete 'edgeR'23 y mantenga exones con recuentos superiores a un umbral configurable (para este conjunto de datos se usa un cpm) en al menos tres muestras. También eliminan genes con un solo exón.
# Non-specific filtering: Remove the exons with low counts
isexpr<- rownames(countData$counts)[rowSums(cpm(countData$counts)>1) >=3]
countData$counts<-countData$counts[rownames(countData$counts) %in%isexpr, ]
anno<-anno%>% filter(GeneID%in% rownames(countData$counts))
# Remove genes with only 1 site and NA in geneIDs
dn<-anno%>%group_by(GeneID)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(GeneID))
anno<-anno%>% filter(GeneID%in%dn$GeneID)
countData$counts<-countData$counts[rownames(countData$counts) %in%anno$GeneID, ]
NOTA: Compruebe los parámetros necesarios para featureCounts cuando utilice datos diferentes, por ejemplo, para lecturas de un solo extremo, establezca 'isPairedEnd = FALSE'. Consulte la guía del usuario de RSubread para elegir las opciones para sus datos y consulte la sección Discusión a continuación.
- Cargar las bibliotecas necesarias:
- Empalme diferencial y análisis de uso de exones. Describimos dos alternativas para este paso: DEXSeq y DiffSplice. Cualquiera de los dos puede ser utilizado y dar resultados similares. Para mantener la coherencia, seleccione DEXSeq si prefiere un paquete DESeq2 para DGE y utilice DiffSplice para un análisis DGE basado en Limma. Ver ficha complementaria: "AS_analysis_RNASeq.rmd".
- Uso del paquete DEXSeq para el análisis diferencial de exones.
- Cargue la biblioteca y cree una tabla de ejemplo para definir el diseño experimental.
library(DEXSeq)
sampleTable<-data.frame(row.names= c("Mbnl1KO_Thymus_1", "Mbnl1KO_Thymus_2", "Mbnl1KO_Thymus_3", "WT_Thymus_1", "WT_Thymus_2", "WT_Thymus_3"), condition= rep(c("Mbnl1_KO", "WT"),c(3,3)), libType= rep(c("paired-end")))
Nota : los nombres de fila deben ser coherentes con los nombres de archivo bam utilizados por featureCounts para contar las lecturas. sampleTable consta de detalles de cada muestra que incluye: tipo de biblioteca y condición. Esto es necesario para definir los contrastes o el grupo de prueba para detectar el uso diferencial. - Prepare el archivo de información de exón. La información de exón en forma de objetos GRanges (rangos genómicos) (https://bioconductor.org/packages/release/bioc/html/GenomicRanges.html) se requiere como entrada para crear el objeto DEXSeq en el siguiente paso. Haga coincidir los identificadores de genes con los recuentos de lectura para crear el objeto exoninfo.
exoninfo<-anno[anno$GeneID%in% rownames(countData$counts),]
exoninfo<-GRanges(seqnames=anno$Chr,
ranges=IRanges(start=anno$Start, end=anno$End, width=anno$Width),strand=Rle(anno$Strand))
mcols(exoninfo)$TranscriptIDs<-anno$TranscriptIDs
mcols(exoninfo)$Ticker<-anno$Ticker
mcols(exoninfo)$ExonID<-anno$ExonID
mcols(exoninfo)$n<-anno$n
mcols(exoninfo)$GeneID<-anno$GeneID
transcripts_l= strsplit(exoninfo$TranscriptIDs, "\\,")
save(countData, sampleTable, exoninfo, transcripts_l, file="AS_countdata.RData") - Crear objeto DEXSeq mediante la función DEXSeqDataSet. El objeto DEXSeq recopila recuentos de lectura, información de características de exón e información de ejemplo. Utilice los recuentos de lectura generados en el paso 3 y la información de exón obtenida del paso anterior para crear el objeto DEXSeq a partir de la matriz de recuento. El argumento sampleData toma una entrada de marco de datos que define las muestras (y sus atributos: tipo de biblioteca y condición), 'design' usa sampleData para generar una matriz de diseño para la prueba diferencial utilizando la notación de fórmula de modelo. Tenga en cuenta que un término de interacción significativo, condición: exón, indica que la fracción de lecturas sobre un gen que cae en un exón particular depende de la condición experimental, es decir, hay AS. Consulte la documentación de DEXSeq para obtener una descripción completa de la configuración de la fórmula del modelo para diseños experimentales más complejos. Para obtener información sobre las características, se requieren identificadores de exón, genes correspondientes y transcripciones.
dxd<-DEXSeqDataSet(countData$counts,sampleData=sampleTable, design=~sample+exon+condition:exon,featureID=exoninfo$ExonID,groupID=exoninfo$GeneID,featureRanges=exoninfo, transcripts=transcripts_l) - Normalización y estimación de dispersión. A continuación, realice la normalización entre muestras y estime la varianza de los datos, debido tanto al ruido de conteo de Poisson de la naturaleza discreta de RNA-seq como a la variabilidad biológica, utilizando los siguientes comandos.
dxd %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - Prueba de uso diferencial. Después de la estimación de la variación, pruebe el uso diferencial del exón para cada gen y genere los resultados.
dxd%<>%testForDEU%>%estimateExonFoldChanges(fitExpToVar=
"condition")#Estimate fold changes
dxr=DEXSeqResults(dxd) - Visualice los eventos de empalme para los genes seleccionados mediante el siguiente comando.
plotDEXSeq(dxr,"Wnk1", displayTranscripts=TRUE, splicing=TRUE,legend
=TRUE,cex.axis=1.2,cex=1.3,lwd=2)
Examine el archivo de R Notebook "AS_analysis_RNASeq.Rmd" para generar gráficos adicionales para genes de interés y para generar gráficos de volcanes en diferentes umbrales.
- Cargue la biblioteca y cree una tabla de ejemplo para definir el diseño experimental.
- Uso de diffSplice de Limma para identificar el empalme diferencial. Siga el archivo de R Notebook "AS_analysis_RNASeq.rmd". Asegúrese de que se han seguido los pasos 2.1-2.3 para preparar los archivos de entrada antes de continuar.
- Cargar bibliotecas
library(limma)
library(edgeR) - Filtrado no específico. Extraer la matriz de recuentos de lectura obtenidos en 2.3. Cree una lista de entidades utilizando la función 'DGEList' del paquete edgeR, donde las filas representan genes y las columnas representan muestras.
mycounts=countData$counts
#Change the rownames of the countdata to exon Ids instead of genes for unique rownames.
rownames(mycounts) = exoninfo$ExonID
dge<-DGEList(counts=mycounts)
#Filtering
isexpr<- rowSums(cpm(dge)>1) >=3
dge<-dge[isexpr,,keep.lib.sizes=FALSE]
#Extract the exon annotations for only transcripts meeting non-specific filter
exoninfo=anno%>% filter(ExonID%in% rownames(dge$counts))
#Convert the exoninfo into GRanges object
exoninfo1<-GRanges(seqnames=exoninfo$Chr,
ranges=IRanges(start=exoninfo$Start, end=exoninfo$End, width=exoninfo$Width),strand=Rle(exoninfo$Strand))
mcols(exoninfo1)$TranscriptIDs<-exoninfo$TranscriptIDs
mcols(exoninfo1)$Ticker<-exoninfo$Ticker
mcols(exoninfo1)$ExonID<-exoninfo$ExonID
mcols(exoninfo1)$n<-exoninfo$n
mcols(exoninfo1)$GeneID<-exoninfo$GeneID
transcripts_l= strsplit(exoninfo1$TranscriptIDs, "\\,")
NOTA: Como paso de filtrado no específico, los recuentos se filtran por cpm < 1 en x de n muestras, donde x es el número mínimo de réplicas en cualquier condición. n = 6 y x = 3 para estos datos de ejemplo. - Normalice los recuentos entre muestras, con la función 'calcNormFactors' del paquete 'edgeR' utilizando la media recortada de los valores M (método de normalización TMM)24 Calculará factores de escala para ajustar los tamaños de la biblioteca.
dge<-calcNormFactors(dge) - Utilice sampleTable como se generó en el paso 2.4.1.1 y cree la matriz de diseño. La matriz de diseño caracteriza el diseño. Consulte los capítulos 8 y 9 de la Guía del usuario de Limma (https://www.bioconductor.org/packages/devel/bioc/vignettes/limma/inst/doc/usersguide.pdf) para obtener detalles sobre las matrices de diseño para diseños experimentales más avanzados.
Treat<- factor(sampleTable$condition)
design<- model.matrix(~0+Treat)
colnames(design) <- levels(Treat) - Ajuste un modelo lineal por exón. Ejecute la función 'voom' del paquete 'limma' para procesar datos de RNA-seq para estimar la varianza y generar pesos de precisión para corregir el ruido del recuento de Poisson, y transformar los recuentos a nivel de exón en log2 recuentos por millón (logCPM). A continuación, ejecute el modelado lineal utilizando la función 'lmfit' para ajustar los modelos lineales a los datos de expresión de cada exón. Calcule estadísticas empíricas de Bayes para el modelo ajustado utilizando la función 'eBayes' para detectar la expresión diferencial del exón. A continuación, defina una matriz de contraste para las comparaciones experimentales de interés. Utilice 'contrasts.fit' para obtener coeficientes y errores estándar para cada par de comparación.
v<-voom(dge,design,plot=FALSE)
fit<-lmFit(v,design)
fit<-eBayes(fit)
colnames(fit)
cont.matrix<-makeContrasts(
Mbnl1_KO_WT=Mbnl1_KO-WT,
levels=design)
fit2<-contrasts.fit(fit,cont.matrix) - Análisis de empalme diferencial. Ejecute 'diffSplice' en el modelo ajustado para probar las diferencias en el uso de exones de genes entre wild-type y knockout y explore los resultados mejor clasificados usando la función 'topSplice': test = "t" da una clasificación de los exones AS, test = "simes" da una clasificación de genes.
ex<-diffSplice(fit2,geneid=exoninfo$GeneID,exonid=exoninfo$ExonID)
ts<-topSplice(ex,n=Inf,FDR=0.1, test="t", sort.by="logFC")
tg<-topSplice(ex,n=Inf,FDR=0.1, test="simes") - Visualización. Trazar los resultados con la función 'plotSplice', dando al gen de interés en el argumento geneid. Guarde los resultados principales ordenados por registro Pliegue el cambio en un objeto y genere un diagrama de volcán para exhibir los exones.
plotSplice(ex,geneid="Wnk1", FDR=0.1)
#Volcano plot
EnhancedVolcano(ts,lab=ts$ExonID,selectLab= head((ts$ExonID),2000), xlab= bquote(~Log[2]~'fold change'), x='logFC', y='P.Value', title='Volcano Plot', subtitle='Mbnl1_KO vs WT (Limma_diffSplice)', FCcutoff=2, labSize=4,legendPosition="right", caption= bquote(~Log[2]~"Fold change cutoff, 2; FDR 10%"))
- Cargar bibliotecas
- Uso de rMATS
- Asegúrese de que la última versión de rMATS v4.1.1 (también conocida como rMATS turbo debido al tiempo de procesamiento reducido y menos requisitos de memoria) se instala utilizando conda o github (https://github.com/Xinglab/rmats-turbo/releases/download/v4.1.1/rmats_turbo_v4_1_1.tar.gz) en el directorio de trabajo. Siga la sección 4.3 en "AS_analysis_RNASeq.Rmd".
- Vaya a la carpeta que contiene los archivos bam obtenidos después de asignar y prepare los archivos de texto, según lo requiera rMATS, para las dos condiciones copiando el nombre de los archivos bam (junto con la ruta) separados por ','. Los siguientes comandos deben ejecutarse en la línea de comandos de Linux:
mkdir rMATS_analysis
cd bams/
ls -pd "$PWD"/*| grep "WT"| tr '\n'','> Wt.txt
ls -pd "$PWD"/*| grep "Mb"| tr '\n'','> KO.txt
mv *.txt ../rMATS_analysis - Ejecute rmats.py con los dos archivos de entrada generados en el paso anterior, junto con el archivo GTF obtenido en 2.1.1.3. Esto generará una carpeta de salida 'rmats_out' que contiene archivos de texto que describen estadísticas (valores p y niveles de inclusión) para cada evento de empalme por separado.
python rmats-turbo/rmats.py --b1 KO.txt --b2 Wt.txt --gtf annotation.gtf -t paired --readLength 50 --nthread 8 --od rmats_out/ --tmp rmats_tmp --task pos
NOTA: La anotación de referencia en forma de archivo GTF también es necesaria. Compruebe los parámetros si los datos son de un solo extremo y cambie la opción -t en consecuencia. - Explorando los resultados de rMATS. Utilice el paquete Bioconductor 'maser'25 para explorar los resultados de rMATS. Cargue los archivos de texto de conteo de uniones y exones (JCEC) en el objeto 'maser' y filtre el resultado en función de la cobertura incluyendo al menos cinco lecturas promedio por evento de empalme.
library(maser)
mbnl1<-maser("/rmats_out/", c("WT","Mbnl1_KO"), ftype="JCEC")
#Filtering out events by coverage
mbnl1_filt<-filterByCoverage(mbnl1,avg_reads=5) - Visualización de los resultados de rMATS. Seleccione los eventos de empalme significativos a la tasa de descubrimiento falso (FDR) del 10% y el cambio mínimo del 10% en el porcentaje empalmado (deltaPSI) utilizando la función 'topEvents' del paquete 'maser'. A continuación, verifique los eventos genéticos para genes individuales de interés (gen de muestra-Wnk1) y trace los valores de PSI para cada evento de empalme de ese gen. Genere un diagrama de volcán especificando el tipo de evento.
#Top splicing events at 10% FDR
mbnl1_top<-topEvents(mbnl1_filt,fdr=0.1, deltaPSI=0.1)
mbnl1_top
#Check the gene events for a particular gene
mbnl1_wnk1<-geneEvents(mbnl1_filt,geneS="Wnk1", fdr=0.1, deltaPSI=0.1)
maser::display(mbnl1_wnk1,"SE")
plotGenePSI(mbnl1_wnk1,type="SE", show_replicates
=TRUE)
volcano(mbnl1_filt,fdr=0.1, deltaPSI=0.1,type="SE")
+xlab("deltaPSI")+ylab("Log10 Adj. Pvalue")+ggtitle("Volcano Plot of exon skipping events") - Genere gráficos de Sashimi para el resultado de eventos de empalme obtenidos con rMATS en forma de archivos de texto utilizando el paquete 'rmats2shahimiplot'. Ejecute el script de Python en la línea de comandos de Linux.
python ./src/rmats2sashimiplot/rmats2sashimiplot.py --b1 ../bams/WT_Thymus_1.bam,../bams/WT_Thymus_2.bam,../bams/WT_Thymus_3.bam --b2 ../bams/Mbnl1KO_Thymus_1.bam,../bams/Mbnl1KO_Thymus_2.bam,../bams/Mbnl1KO_Thymus_3.bam -t SE -e ../rMATS_analysis/rmats_out/SE.MATS.JC.txt --l1 WT --l2 Mbnl1_KO --exon_s 1 --intron_s 5 -o ../rMATS_analysis/rmats2shasmi_output
NOTA: Este proceso puede llevar mucho tiempo, ya que generará el gráfico de Sashimi para todos los resultados en el archivo de eventos. Elija los resultados principales (nombres de genes y exones) como se muestra en la función topEvents de 'maser' y visualice el gráfico de Sashimi correspondiente.
- Uso del paquete DEXSeq para el análisis diferencial de exones.
3. Análisis alternativo de poliadenilación (APA) utilizando secuenciación de extremo 3'
- Descarga y preprocesamiento de datos
NOTA: Consulte el archivo suplementario de R Notebook "APA_analysis_3PSeq_notebook. Rmd" para los comandos completos para los pasos de descarga y preprocesamiento de datos, o ejecute el archivo bash suplementario "APA_data_downloading_preprocessing.sh" en la línea de comandos de Linux.- Descargar datos de SRA con los ID de Acceso (1553129 a 1553136).
- Adaptadores de recorte y complemento inverso para obtener la secuencia de hebras de detección.
NOTA: Este paso es específico para el ensayo PolyA-seq utilizado. - Lecturas de mapa para el ensamblaje del genoma del ratón usando el alineador de pajarita26.
- Preparación de anotaciones de sitios pA.
NOTA: El procesamiento del archivo de anotación del sitio pA se realiza en primer lugar utilizando el archivo suplementario de R Notebook "APA_analysis_3PSeq_notebook. Rmd" (2.1 - 2.6), y luego usando el archivo bash "APA_annotation_preparation.sh".- Descargar anotación de sitios pA de la base de datos PolyASite 2.06.
- Seleccione anotaciones de sitio pA para retener sitios pA de región no traducida (UTR) de 3', que se anotan como Exón terminal (TE) o 1000 nt aguas abajo de un exón terminal anotado (DS) para el análisis descendente.
- Obtener picos de sitio pA. Ancla en cada sitio de escisión pA y visualiza la cobertura de lectura promedio usando herramientas de cama y herramientas profundas27,28. Los resultados mostraron que los picos de las lecturas mapeadas se dispersaron principalmente dentro de ~ 60 pb aguas arriba de los sitios de escisión (figura 5 y figura complementaria 5). Por lo tanto, las coordenadas de los sitios pA se extendieron desde el archivo de anotación a 60 pb aguas arriba de sus sitios de escisión. Dependiendo del protocolo específico de secuenciación final de 3' utilizado, este paso deberá optimizarse para ensayos que no sean PolyA-seq.
- Lecturas de conteo
- Prepare el archivo de anotación de sitios pA.
anno<- read.table(file= "flanking60added.pA_annotation.bed",
stringsAsFactors=FALSE, check.names=FALSE, header=FALSE, sep="")
colnames(anno) <- c("chrom", "chromStart", "chromEnd", "name", "score", "strand", "rep", "annotation", "gene_name", "gene_id")
anno<- dplyr::select(anno,name,chrom, chromStart,chromEnd, strand,gene_id,gene_name,rep)
colnames(anno) <- c("GeneID", "Chr", "Start", "End", "Strand", "Ensembl", "Symbol", "repID") - Aplique 'featureCounts' para adquirir recuentos sin procesar. Guarde la tabla de recuento como el archivo RData "APA_countData.Rdata" para el análisis APA utilizando diferentes herramientas.
countData<- dir("bamfiles", pattern="sorted.bam$", full.names=TRUE) %>%
# Read all bam files as input for featureCounts
featureCounts(annot.ext=anno, isGTFAnnotationFile= FALSE,minMQS=0,useMetaFeatures= TRUE,allowMultiOverlap=TRUE, largestOverlap= TRUE,strandSpecific=1, countMultiMappingReads =TRUE,primaryOnly= TRUE,isPairedEnd= FALSE,nthreads=12)%T>%
save(file="APA_countData.Rdata")
NOTA: Tenga en cuenta que debe cambiar cualquiera de los parámetros enumerados en la función 'featureCounts'. Modifique el parámetro 'strandSpecific' para asegurarse de que sea consistente con la dirección de secuenciación del ensayo de secuenciación final 3' utilizado (empíricamente, visualizar los datos en un navegador del genoma sobre los genes en las hebras más y menos aclarará esto). - Aplicar filtrado no específico de countData. El filtrado puede mejorar significativamente la solidez estadística en las pruebas de uso diferencial del sitio pA. Primero, eliminamos esos genes con un solo sitio pA, en el que no se puede definir diferencialmente el uso del sitio pA. En segundo lugar, aplicamos un filtrado no específico basado en la cobertura: los recuentos se filtran por cpm menos de 1 en x de n muestras, donde x es el número mínimo de réplicas en cualquier condición. N = 8 y x = 2 para estos datos de ejemplo.
load(file= "APA_countData.Rdata")# Skip this step if already loaded
# Non-specific filtering: Remove the pA sites not differentially expressed in the samples
countData<-countData$counts%>%as.data.frame%>% .[rowSums(edgeR::cpm(.)>1) >=2, ]
anno%<>% .[.$GeneID%in% rownames(countData), ]
# Remove genes with only 1 site and NA in geneIDs
dnsites<-anno%>%group_by(Symbol)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(Symbol))
anno<-anno%>% filter(Symbol%in%dnsites$Symbol)
countData<-countData[rownames(countData) %in%anno$GeneID, ]
- Prepare el archivo de anotación de sitios pA.
- Análisis diferencial de uso de sitios de poliadenilación utilizando tuberías DEXSeq y diffSplice.
- Uso del paquete DEXSeq
NOTA: Como no se puede definir una matriz de contraste para la tubería DEXSeq, el análisis APA diferencial de cada dos condiciones experimentales debe realizarse por separado. El análisis diferencial APA de la condición WT y la condición DKO se realiza como ejemplo para explicar el procedimiento. Consulte el archivo complementario "APA_analysis_3PSeq_notebook. RMD" para el flujo de trabajo paso a paso de esta sección y el análisis diferencial APA de otros contrastes.- Cargue la biblioteca y cree una tabla de ejemplo para definir el diseño experimental.
c("DEXSeq", "GenomicRanges") %>% lapply(library, character.only=TRUE) %>%invisible
sampleTable1<- data.frame(row.names= c("WT_1","WT_2","DKO_1","DKO_2"),
condition= c(rep("WT", 2), rep("DKO", 2)),
libType= rep("single-end", 4)) - Prepare el archivo de información de sitios pA utilizando el paquete Bioconductor GRanges.
# Prepare the GRanges object for DEXSeqDataSet object construction
PASinfo <- GRanges(seqnames = anno$Chr,
ranges = IRanges(start = anno$Start, end = anno$End),strand = Rle(anno$Strand))
mcols(PASinfo)$PASID<-anno$repID
mcols(PASinfo)$GeneEns<-anno$Ensembl
mcols(PASinfo)$GeneID<-anno$Symbol
# Prepare the new feature IDs, replace the strand information with letters to match the current pA site clusterID
new.featureID <- anno$Strand %>% as.character %>% replace(. %in% "+", "F") %>% replace(. %in% "-", "R") %>% paste0(as.character(anno$repID), .) - Utilice los recuentos de lectura generados en el paso 3.3 y la información del sitio pA obtenida del paso anterior para crear el objeto DEXSeq.
# Select the read counts of the condition WT and DKO
countData1<- dplyr::select(countData, SRR1553129.sorted.bam, SRR1553130.sorted.bam, SRR1553131.sorted.bam, SRR1553132.sorted.bam)
# Rename the columns of countData using sample names in sampleTable
colnames(countData1) <- rownames(sampleTable1)
dxd1<-DEXSeqDataSet(countData=countData1,
sampleData=sampleTable1,
design=~sample+exon+condition:exon,
featureID=new.featureID,
groupID=anno$Symbol,
featureRanges=PASinfo) - Defina el par de contraste definiendo los niveles de condiciones en el objeto DEXSeq.
dxd1$condition<- factor(dxd1$condition, levels= c("WT", "DKO"))
# The contrast pair will be "DKO - WT" - Normalización y estimación de dispersión. Similar a los datos de RNA-seq, para los datos de secuenciación final de 3' realice la normalización entre muestras (mediana de proporciones en columna para cada muestra) utilizando la función 'estimateSizeFactors', y estime la variación de los datos usando con la función 'estimateDispersions', luego visualice el resultado de la estimación de dispersión usando la función 'plotDispEsts'.
dxd1 %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - Prueba diferencial de uso del sitio pA para cada gen usando la función 'testForDEU', luego estime el cambio de pliegue del uso del sitio pA usando la función 'estimateExonFoldChanges'. Verifique los resultados utilizando la función 'DEXSeqResults' y establezca 'FDR < 10%' como criterio para sitios de pA significativamente diferenciales.
dxd1 %<>% testForDEU %>% estimateExonFoldChanges(fitExpToVar = "condition")
dxr1 <- DEXSeqResults(dxd1)
dxr1
mcols(dxr1)$description
table(dxr1$padj<0.1) # Check the number of differential pA sites (FDR < 0.1)
table(tapply(dxr1$padj<0.1, dxr1$groupID, any)) # Check the number of gene overlapped with differential pA site - La visualización de los resultados de uso diferencial del sitio pA utilizando gráficos APA diferenciales generados por la función 'plotDEXSeq' y gráficos de volcán por la función 'EnhancedVolcano'.
# Select the top 100 significant differential pA sites ranked by FDR
topdiff.PAS<- dxr1%>%as.data.frame%>%rownames_to_column%>%arrange(padj)%$%groupID[1:100]
# Apply plotDEXSeq for the visualization of differential polyA usage
plotDEXSeq(dxr1,"S100a7a", legend=TRUE, expression=FALSE,splicing=TRUE, cex.axis=1.2, cex=1.3,lwd=2)
# Apply perGeneQValue to check the top genes with differential polyA site usage
dxr1%<>% .[!is.na(.$padj), ]
dgene<- data.frame(perGeneQValue= perGeneQValue (dxr1)) %>%rownames_to_column("groupID")
dePAS_sig1<-dxr1%>% data.frame() %>%
dplyr::select(-matches("dispersion|stat|countData|genomicData"))%>%
inner_join(dgene)%>%arrange(perGeneQValue)%>%distinct()%>%
filter(padj<0.1)
# Apply EnhancedVolcano package to visualise differential polyA site usage
"EnhancedVolcano"%>% lapply(library, character.only=TRUE) %>%invisible
EnhancedVolcano(dePAS_sig1, lab=dePAS_sig1$groupID, x='log2fold_DKO_WT',
y='pvalue',title='Volcano Plot',subtitle='DKO vs WT',
FCcutoff=1,labSize=4, legendPosition="right",
caption= bquote(~Log[2]~"Fold change cutoff, 1; FDR 10%"))
- Cargue la biblioteca y cree una tabla de ejemplo para definir el diseño experimental.
- Usando el paquete diffSplice. Consulte el archivo complementario de R Notebook "APA_analysis_3PSeq_notebook. Rmd" para el flujo de trabajo paso a paso de esta sección.
- Definir contrastes de interés para el análisis diferencial de uso de pA.
Nota : este paso debe realizarse después de la construcción y el procesamiento del objeto DGEList, que se incluye en el archivo de R Notebook "APA_analysis_3PSeq_notebook. Rmd".contrast.matrix<-makeContrasts(DKO_vs_WT=DKO-
WT,Ctrl_vs_DKO=Ctrl-DKO,
KD_vs_Ctrl=KD-Ctrl,KD_vs_DKO=KD-DKO,levels=design)
fit2<-fit%>%contrasts.fit(contrast.matrix)%>%eBayes
summary(decideTests(fit2))
ex<-diffSplice(fit2,geneid=anno$Symbol,exonid=new.featureID)
topSplice(ex) #Check the top significant results with topSplice - Visualice el resultado de contrastes de interés (aquí "DKO - WT") utilizando gráficos APA diferenciales mediante la función 'plotSplice' y gráficos de volcanes con la función 'EnhancedVolcano'. Consulte el archivo de R Notebook "APA_analysis_3PSeq_notebook. Rmd" 4.2.7 - 4.2.9 para la visualización de otros pares de contraste.
sig1<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="t", sort.by="logFC")
sig1.genes<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="simes")
plotSplice(ex, coef=1,geneid="S100a7a", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Tpm1", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Smc6", FDR = 0.1)
EnhancedVolcano(sig1, lab=sig1$GeneID,xlab= bquote(~Log[2]~'fold change'),
x='logFC', y='P.Value', title='Volcano Plot', subtitle='DKO vs WT',
FCcutoff=1, labSize=6, legendPosition="right")
- Definir contrastes de interés para el análisis diferencial de uso de pA.
- Uso del paquete DEXSeq
Resultados
Después de ejecutar el flujo de trabajo paso a paso anterior, los resultados del análisis AS y APA y los resultados representativos se presentan en forma de tablas y gráficos de datos, generados de la siguiente manera.
COMO:
El resultado principal del análisis AS (Tabla suplementaria 1 para diffSplice; Tabla 2 para DEXSeq) es una lista de exones que muestran el uso diferencial entre condiciones, y una lista de genes que muestran una act...
Discusión
En este estudio, evaluamos enfoques basados en exones y eventos para detectar AS y APA en datos masivos de RNA-Seq y secuenciación final 3'. Los enfoques de EA basados en exones producen tanto una lista de exones expresados diferencialmente como una clasificación a nivel de gen ordenada por la significación estadística de la actividad general de empalme diferencial a nivel de gen (Tablas 1-2, 4-5). Para el paquete diffSplice, el uso diferencial se determina ajustando modelos lineales ponderados a niv...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este estudio fue apoyado por una beca futura del Consejo Australiano de Investigación (ARC) (FT16010043) y ANU Futures Scheme.
Materiales
| Name | Company | Catalog Number | Comments |
| Not relevent for computational study |
Referencias
- Katz, Y., Wang, E. T., Airoldi, E. M., Burge, C. B. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods. 7 (12), 1009-1015 (2010).
- Wang, Y., et al. Mechanism of alternative splicing and its regulation. Biomedical Reports. 3 (2), 152-158 (2015).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in Bioinformatics. 21 (6), 2052-2065 (2020).
- Movassat, M., et al. Coupling between alternative polyadenylation and alternative splicing is limited to terminal introns. RNA Biology. 13 (7), 646-655 (2016).
- Tian, B., Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature Reviews Molecular Cell Biology. 18 (1), 18-30 (2017).
- Herrmann, C. J., et al. PolyASite 2.0: a consolidated atlas of polyadenylation sites from 3' end sequencing. Nucleic Acids Research. 48 (1), 174-179 (2020).
- Liu, R., Loraine, A. E., Dickerson, J. A. Comparisons of computational methods for differential alternative splicing detection using RNA-seq in plant systems. BMC Bioinformatics. 15 (1), 364 (2014).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (1), 13 (2016).
- Anders, S., Reyes, A., Huber, W. Detecting differential usage of exons from RNA-seq data. Genome Research. 22 (10), 2008-2017 (2012).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2014).
- Shen, S., et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data. Proceedings of the National Academy of Sciences. 111 (51), 5593-5601 (2014).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in bioinformatics. 21 (6), 2052-2065 (2020).
- Kanitz, A., et al. Comparative assessment of methods for the computational inference of transcript isoform abundance from RNA-seq data. Genome biology. 16 (1), 1-26 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Sznajder, L. J., et al. Loss of MBNL1 induces RNA misprocessing in the thymus and peripheral blood. Nature Communications. 11, 1-11 (2020).
- Batra, R., et al. Loss of MBNL leads to disruption of developmentally regulated alternative polyadenylation in RNA-mediated disease. Molecular Cell. 56 (2), 311-322 (2014).
- Leinonen, R., Sugawara, H., Shumway, M., et al. The sequence read archive. Nucleic acids research. 39, 19-21 (2010).
- Tange, O. . GNU parallel-the command-line power tool. 36, 42-47 (2011).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), 25 (2010).
- Veiga, D. F. T. maser: Mapping Alternative Splicing Events to pRoteins. R package version 1.4.0. , (2019).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (13), 25 (2009).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ramírez, F., Dündar, F., Diehl, S., Grüning, B. A., Manke, T. deepTools: a flexible platform for exploring deep-sequencing data. Nucleic acids research. 42 (1), 187-191 (2014).
- Merino, G. A., Conesa, A., Fernández, E. A. A benchmarking of workflows for detecting differential splicing and differential expression at isoform level in human RNA-seq studies. Briefings in bioinformatics. 20 (2), 471-481 (2019).
- Chhangawala, S., Rudy, G., Mason, C. E., Rosenfeld, J. A. The impact of read length on quantification of differentially expressed genes and splice junction detection. Genome biology. 16 (1), 1-10 (2015).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biol. 17, 13 (2016).
- Trapnell, C., et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 7 (3), 562-578 (2012).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 12, 323 (2011).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat Biotechnol. 34 (5), 525-527 (2016).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados