Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Identifizierung von alternativem Spleißen und Polyadenylierung in RNA-seq-Daten
In diesem Artikel
Zusammenfassung
Alternative Spleißen (AS) und alternative Polyadenylierung (APA) erweitern die Vielfalt der Transkriptisoformen und ihrer Produkte. Hier beschreiben wir bioinformatische Protokolle zur Analyse von Bulk-RNA-seq und 3'-Endsequenzierungsassays, um AS und APA zu erkennen und zu visualisieren, die unter experimentellen Bedingungen variieren.
Zusammenfassung
Neben der typischen Analyse von RNA-Seq zur Messung der differentiellen Genexpression (DGE) unter experimentellen/biologischen Bedingungen können RNA-seq-Daten auch verwendet werden, um andere komplexe regulatorische Mechanismen auf Exon-Ebene zu erforschen. Alternative Spleißung und Polyadenylierung spielen eine entscheidende Rolle für die funktionelle Vielfalt eines Gens, indem sie verschiedene Isoformen erzeugen, um die Genexpression auf posttranskriptioneller Ebene zu regulieren, und die Beschränkung der Analysen auf die gesamte Genebene kann diese wichtige regulatorische Schicht übersehen. Hier demonstrieren wir detaillierte Schritt-für-Schritt-Analysen zur Identifizierung und Visualisierung der differentiellen Exon- und Polyadenylierungsstellennutzung über Bedingungen hinweg, wobei Bioconductor und andere Pakete und Funktionen wie DEXSeq, diffSplice aus dem Limma-Paket und rMATES verwendet werden.
Einleitung
RNA-seq wurde im Laufe der Jahre häufig verwendet, typischerweise zur Schätzung der differentiellen Genexpression und Genentdeckung1. Darüber hinaus kann es auch verwendet werden, um die unterschiedliche Nutzung auf Exon-Ebene aufgrund der Genexprimierung verschiedener Isoformen abzuschätzen, was zu einem besseren Verständnis der Genregulation auf posttranskriptioneller Ebene beiträgt. Die Mehrheit der eukaryotischen Gene erzeugt verschiedene Isoformen durch alternatives Spleißen (AS), um die Vielfalt der mRNA-Expression zu erhöhen. AS-Ereignisse können in verschiedene Muster unterteilt werden: Überspringen vollständiger Exons (SE), bei denen ein ("Kassetten") Exon zusammen mit seinen flankierenden Introns vollständig aus dem Transkript entfernt wird; alternative (Donor) 5'-Spleißstellenauswahl (A5SS) und alternative 3' (Akzeptor) Spleißstellenauswahl (A3SS), wenn zwei oder mehr Spleißstellen an beiden Enden eines Exons vorhanden sind; Beibehaltung von Introns (RI), wenn ein Intron innerhalb des reifen mRNA-Transkripts beibehalten wird, und gegenseitiger Ausschluss der Exon-Nutzung (MXE), wobei nur eines der beiden verfügbaren Exons gleichzeitig beibehalten werden kann 2,3. Die alternative Polyadenylierung (APA) spielt auch eine wichtige Rolle bei der Regulierung der Genexpression unter Verwendung alternativer Poly(A)-Stellen, um mehrere mRNA-Isoformen aus einem einzigen Transkriptzu erzeugen 4. Die meisten Polyadenylierungsstellen (pAs) befinden sich in der 3' untranslatierten Region (3' UTRs) und erzeugen mRNA-Isoformen mit unterschiedlichen 3' UTR-Längen. Da die 3' UTR die zentrale Drehscheibe für die Erkennung regulatorischer Elemente ist, können unterschiedliche 3' UTR-Längen die mRNA-Lokalisierung, Stabilität und Translation beeinflussen5. Es gibt eine Klasse von 3'-Endsequenzierungsassays, die für den Nachweis von APA optimiert sind und sich in den Details des Protokolls6 unterscheiden. Die hier beschriebene Pipeline ist für PolyA-seq ausgelegt, kann aber wie beschrieben für andere Protokolle angepasst werden.
In dieser Studie stellen wir eine Pipeline von differentiellen Exon-Analysemethoden7,8 (Abbildung 1) vor, die in zwei große Kategorien unterteilt werden können: exon-basiert (DEXSeq9, diffSplice 10) und ereignisbasiert (replicate Multivariate Analysis of Transcript Splicing (rMATS)11). Die Exon-basierten Methoden vergleichen die Faltenänderung über die Bedingungen einzelner Exons hinweg mit einem Maß für die gesamte Genfaltenänderung, um differentiell exprimierte Exon-Nutzung zu nennen, und berechnen daraus ein Maß für die AS-Aktivität auf Genebene. Ereignisbasierte Methoden verwenden Exon-Intron-Spanning-Junction-Lesevorgänge, um bestimmte Spleißereignisse wie Exon-Skipping oder Beibehaltung von Introns zu erkennen und zu klassifizieren und diese AS-Typen in Ausgabe3 zu unterscheiden. Somit bieten diese Methoden komplementäre Sichtweisen für eine vollständige Analyse von AS12,13. Wir haben DEXSeq (basierend auf dem DESeq214 DGE-Paket) und diffSplice (basierend auf dem Limma10 DGE-Paket) für die Studie ausgewählt, da sie zu den am häufigsten verwendeten Paketen für die differentielle Spleißanalyse gehören. rMATS wurde als beliebte Methode für die ereignisbasierte Analyse ausgewählt. Eine weitere beliebte ereignisbasierte Methode ist MISO (Mixture of Isoforms)1. Für APA adaptieren wir den Exon-basierten Ansatz.
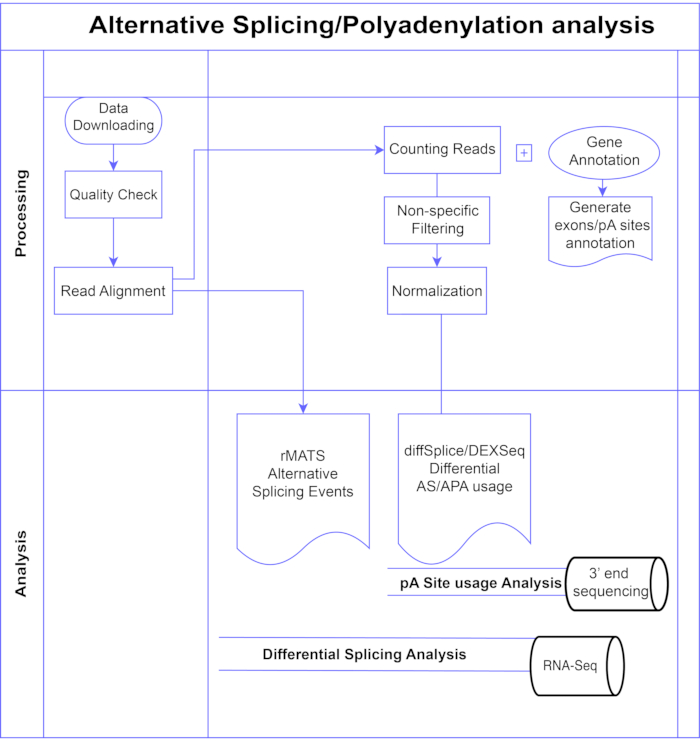
Abbildung 1. Analyse-Pipeline. Flussdiagramm der in der Analyse verwendeten Schritte. Zu den Schritten gehören: Abrufen der Daten, Durchführen von Qualitätsprüfungen und Leseausrichtung, gefolgt von Zählen von Lesevorgängen unter Verwendung von Anmerkungen für bekannte Exons, Introns und pA-Stellen, Filtern zur Entfernung niedriger Zählungen und Normalisierung. PolyA-seq-Daten wurden für alternative pA-Stellen mit diffSplice/DEXSeq-Methoden analysiert, Bulk-RNA-Seq wurde auf alternative Spleißung auf Exon-Ebene mit diffSplice/DEXseq-Methoden analysiert und AS-Ereignisse mit rMATS analysiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Die in dieser Umfrage verwendeten RNA-seq-Daten stammen von Gene Expression Omnibus (GEO) (GSE138691)15. Wir verwendeten Maus-RNA-seq-Daten aus dieser Studie mit zwei Bedingungsgruppen: Wildtyp (WT) und Muskelblind-ähnlicher Typ-1-Knockout (Mbnl1 KO) mit jeweils drei Replikaten. Um die Analyse der differentiellen Polyadenylierungsstandortnutzung zu demonstrieren, erhielten wir PolyA-seq-Daten von Mausembryo-Fibroblasten (MEFs) (GEO Accession GSE60487)16. Die Daten haben vier Bedingungsgruppen: Wild-Typ (WT), Muskelblind-Typ 1/Typ 2 Doppel-Knockout (Mbnl1/2 DKO), Mbnl 1/2 DKO mit Mbnl3-Knockdown (KD) und Mbnl1/2 DKO mit Mbnl3-Kontrolle (Strg). Jede Bedingungsgruppe besteht aus zwei Replikaten.
| GEO-Beitritt | SRA-Ausführungsnummer | Name des Beispiels | Zustand | Replizieren | Gewebe | Sequenzierung | Leselänge | |
| RNA-Seq | GSM4116218 | SRR10261601 | Mbnl1KO_Thymus_1 | Mbnl1 Knockout | Wiederholung 1 | Thymus | Gepaartes Ende | 100 bp |
| GSM4116219 | SRR10261602 | Mbnl1KO_Thymus_2 | Mbnl1 Knockout | Wiederholung 2 | Thymus | Gepaartes Ende | 100 bp | |
| GSM4116220 | SRR10261603 | Mbnl1KO_Thymus_3 | Mbnl1 Knockout | Wiederholung 3 | Thymus | Gepaartes Ende | 100 bp | |
| GSM4116221 | SRR10261604 | WT_Thymus_1 | Wildtyp | Wiederholung 1 | Thymus | Gepaartes Ende | 100 bp | |
| GSM4116222 | SRR10261605 | WT_Thymus_2 | Wildtyp | Wiederholung 2 | Thymus | Gepaartes Ende | 100 bp | |
| GSM4116223 | SRR10261606 | WT_Thymus_3 | Wildtyp | Wiederholung 3 | Thymus | Gepaartes Ende | 100 bp | |
| 3P-Seq | GSM1480973 | SRR1553129 | WT_1 | Wildtyp (WT) | Wiederholung 1 | Embryonale Fibroblasten (MEFs) der Maus | Single-End | 40 bp |
| GSM1480974 | SRR1553130 | WT_2 | Wildtyp (WT) | Wiederholung 2 | Embryonale Fibroblasten (MEFs) der Maus | Single-End | 40 bp | |
| GSM1480975 | SRR1553131 | DKO_1 | Mbnl 1/2 Doppel-Knockout (DKO) | Wiederholung 1 | Embryonale Fibroblasten (MEFs) der Maus | Single-End | 40 bp | |
| GSM1480976 | SRR1553132 | DKO_2 | Mbnl 1/2 Doppel-Knockout (DKO) | Wiederholung 2 | Embryonale Fibroblasten (MEFs) der Maus | Single-End | 40 bp | |
| GSM1480977 | SRR1553133 | DKOsiRNA_1 | Mbnl 1/2 Doppel-Knockout mit Mbnl 3 siRNA (KD) | Wiederholung 1 | Embryonale Fibroblasten (MEFs) der Maus | Single-End | 40 bp | |
| GSM1480978 | SRR1553134 | DKOsiRNA_2 | Mbnl 1/2 Doppel-Knockout mit Mbnl 3 siRNA (KD) | Wiederholung 2 | Embryonale Fibroblasten (MEFs) der Maus | Single-End | 36 bp | |
| GSM1480979 | SRR1553135 | DKONTsiRNA_1 | Mbnl 1/2 Doppel-Knockout mit nicht-targetender siRNA (Ctrl) | Wiederholung 1 | Embryonale Fibroblasten (MEFs) der Maus | Single-End | 40 bp | |
| GSM1480980 | SRR1553136 | DKONTsiRNA_2 | Mbnl 1/2 Doppel-Knockout mit nicht-targetender siRNA (Ctrl) | Wiederholung 2 | Embryonale Fibroblasten (MEFs) der Maus | Single-End | 40 bp |
Tabelle 1. Zusammenfassung der RNA-Seq- und PolyA-seq-Datensätze, die für die Analyse verwendet wurden.
Protokoll
1. Installation von Tools und R-Paketen, die in der Analyse verwendet werden
- Conda ist ein beliebter und flexibler Paketmanager, der eine komfortable Installation von Paketen mit ihren Abhängigkeiten über alle Plattformen hinweg ermöglicht. Verwenden Sie 'Anaconda' (conda-Paketmanager), um 'conda' zu installieren, mit dem die für die Analyse erforderlichen Tools/Pakete installiert werden können.
- Laden Sie 'Anaconda' gemäß den Systemanforderungen von https://www.anaconda.com/products/individual#Downloads herunter und installieren Sie es, indem Sie den Anweisungen im grafischen Installationsprogramm folgen. Installieren Sie alle erforderlichen Pakete mit 'conda', indem Sie Folgendes in die Linux-Befehlszeile eingeben.
conda install -c daler sratoolkit
conda install -c conda-forge parallel
conda install -c bioconda star bowtie fastqc rmats rmats2sashimiplot samtools fasterq-dump cutadapt bedtools deeptools - Um alle im Protokoll verwendeten R-Pakete herunterzuladen, geben Sie den folgenden Code in die R-Konsole (gestartet in der Linux-Befehlszeile durch Eingabe von 'R') oder Rstudio-Konsole ein.
bioc_packages<- c("DEXSeq", "Rsubread", "EnhancedVolcano", "edgeR", "limma", "maser","GenomicRanges")
packages<- c("magrittr", "rtracklayer", "tidyverse", "openxlsx", "BiocManager")
#Install if not already installed
installed_packages<-packages%in% rownames(installed.packages())
installed_bioc_packages<-bioc_packages%in% rownames(installed.packages())
if(any(installed_packages==FALSE)) {
install.packages(packages[!installed_packages],dependencies=TRUE)
BiocManager::install(packages[!installed_bioc_packages], dependencies=TRUE)
}
HINWEIS: In diesem Berechnungsprotokoll werden Befehle entweder als R-Notebook-Dateien (Dateien mit der Erweiterung " angegeben. Rmd"), R-Codedateien (Dateien mit der Erweiterung ". R") oder Linux Bash Shell-Skripte (Dateien mit der Erweiterung ".sh"). R-Notebook-Dateien (Rmd) sollten in RStudio mit Datei| Öffnen Sie Datei..., und einzelne Codeblöcke (die R-Befehle oder Bash-Shell-Befehle sein können) werden dann interaktiv ausgeführt, indem Sie auf den grünen Pfeil oben rechts klicken. R-Code-Dateien können durch Öffnen in RStudio oder auf der Linux-Befehlszeile durch Voranstellen von "Rscript" ausgeführt werden, z.B. Rscript example.R. Shell-Skripte werden auf der Linux-Befehlszeile ausgeführt, indem dem Skript der Befehl "sh" vorangestellt wird.sh example.sh.
2. Alternative Spleißanalyse (AS) mittels RNA-seq
- Herunterladen und Vorverarbeiten von Daten
HINWEIS: Die unten kommentierten Codeausschnitte sind in der ergänzenden Codedatei "AS_analysis_RNASeq.Rmd", um die einzelnen Schritte interaktiv zu befolgen, und werden auch als Bash-Skript bereitgestellt, das im Batch auf der Linux-Befehlszeile ausgeführt werden kann (sh downloading_data_preprocessing.sh).- Herunterladen der Rohdaten.
- Laden Sie die Rohdaten aus dem Sequence Read Archive (SRA) mit dem Befehl 'prefetch' aus dem SRA-Toolkit (v2.10.8)17 herunter. Geben Sie die SRA-Zugangs-IDs im folgenden Befehl nacheinander an, um sie parallel mit dem GNU Parallel-Dienstprogramm18 herunterzuladen. Um SRA-Dateien der Beitritts-IDs von SRR10261601 auf SRR10261606 parallel herunterzuladen, verwenden Sie die folgenden Schritte in der Linux-Befehlszeile.
seq 10261601 10261606 | parallel prefetch SRR{} - Extrahieren Sie die fastq-Dateien aus dem Archiv mit der Funktion 'fastq-dump' aus dem SRA-Toolkit. Verwenden Sie GNU parallel und geben Sie die Namen aller SRA-Dateien zusammen an.
parallel -j 3 fastq-dump --gzip --skip-technical --read-filter pass --dumpbase --split-e --clip --origfmt {} ::: - Laden Sie das Referenzgenom und die Anmerkungen für Maus (Genomassembly GRCm39) von www.ensembl.org herunter, indem Sie die folgenden Schritte in der Linux-Befehlszeile verwenden.
wget -nv -O annotation.gtf.gz http://ftp.ensembl.org/pub/release-103/gtf/mus_musculus/Mus_musculus.GRCm39.103.gtf.gz \ && gunzip -f annotation.gtf.gz
wget -nv -O genome.fa.gz http://ftp.ensembl.org/pub/release-103/fasta/mus_musculus/dna/Mus_musculus.GRCm39.dna.primary_assembly.fa.gz \ && gunzip -f genome.fa.gz
GTF=$(readlink -f annotation.gtf)
GENOME=$(readlink -f genome.fa)
- Laden Sie die Rohdaten aus dem Sequence Read Archive (SRA) mit dem Befehl 'prefetch' aus dem SRA-Toolkit (v2.10.8)17 herunter. Geben Sie die SRA-Zugangs-IDs im folgenden Befehl nacheinander an, um sie parallel mit dem GNU Parallel-Dienstprogramm18 herunterzuladen. Um SRA-Dateien der Beitritts-IDs von SRR10261601 auf SRR10261606 parallel herunterzuladen, verwenden Sie die folgenden Schritte in der Linux-Befehlszeile.
- Vorverarbeitung und Zuordnung von Lesevorgängen zur Genomassemblierung
- Qualitätskontrolle. Bewerten Sie die Qualität von Rohlesevorgängen mit FASTQC (FASTQ Quality Check v0.11.9)19. Erstellen Sie einen Ausgabeordner und führen Sie fastqc parallel auf mehreren Eingabe-Fasta-Dateien aus. In diesem Schritt wird für jede Probe ein Qualitätsbericht erstellt. Überprüfen Sie die Berichte, um sicherzustellen, dass die Qualität der Lesevorgänge akzeptabel ist, bevor Sie weitere Analysen durchführen. (Informationen zu den Berichten finden Sie im Benutzerhandbuch unter https://www.bioinformatics.babraham.ac.uk/projects/fastqc/)
mkdir fastqc_out
parallel "fastqc {} -o fastqc_out" ::: $RAW_DATA/*.fastq.gz
HINWEIS: Führen Sie bei Bedarf ein Adaptertrimmen mit 'cutadapt'20 oder 'trimmomatic'21 durch, um die Sequenzierung in flankierende Adapter zu entfernen, die je nach RNA-Fragmentgröße und Leselänge variiert. In dieser Analyse haben wir diesen Schritt übersprungen, da der Anteil der betroffenen Lesevorgänge minimal war. - Leseausrichtung. Der nächste Schritt in der Vorverarbeitung umfasst die Zuordnung der Lesevorgänge zum Referenzgenom. Erstellen Sie zunächst den Index für das Referenzgenom mit der Funktion "genomeGenerate" von STAR22 und richten Sie dann die Rohlesevorgänge an der Referenz aus (alternativ sind vorgefertigte Indizes auf der STAR-Website verfügbar und können direkt für die Ausrichtung verwendet werden). Führen Sie die folgenden Befehle in der Linux-Befehlszeile aus.
#Build STAR index
GDIR=STAR_indices
mkdir $GDIR
STAR --runMode genomeGenerate --genomeFastaFiles $GENOME --sjdbGTFfile $GTF --runThreadN 8 --genomeDir $GDIR
ODIR=results/mapping
mkdir -p $ODIR
#Align reads to the genome
for fq1 in $RAW_DATA/*R1.fastq.gz;
do
fq2=$(echo $fq1| sed 's/1.fastq.gz/2.fastq.gz/g');
OUTPUT=$(basename ${fq1}| sed 's/R1.fastq.gz//g');
STAR --genomeDir $GDIR \
--runThreadN 12 \
--readFilesCommand zcat \
--readFilesIn ${fq1}${fq2}\
--outFileNamePrefix $ODIR\/${OUTPUT} \
--outSAMtype BAM SortedByCoordinate \
--outSAMunmapped Within \
--outSAMattributes Standard
Done
HINWEIS: Der STAR Aligner generiert und sortiert BAM-Dateien (Binary Alignment Map) für jedes Sample nach dem Leseausrichtung. BAM-Dateien müssen sortiert werden, bevor Sie mit weiteren Schritten fortfahren.
- Qualitätskontrolle. Bewerten Sie die Qualität von Rohlesevorgängen mit FASTQC (FASTQ Quality Check v0.11.9)19. Erstellen Sie einen Ausgabeordner und führen Sie fastqc parallel auf mehreren Eingabe-Fasta-Dateien aus. In diesem Schritt wird für jede Probe ein Qualitätsbericht erstellt. Überprüfen Sie die Berichte, um sicherzustellen, dass die Qualität der Lesevorgänge akzeptabel ist, bevor Sie weitere Analysen durchführen. (Informationen zu den Berichten finden Sie im Benutzerhandbuch unter https://www.bioinformatics.babraham.ac.uk/projects/fastqc/)
- Herunterladen der Rohdaten.
- Vorbereiten von Exon-Anmerkungen.
- Führen Sie die ergänzende Codedatei "prepare_mm_exon_annotation. R" mit der heruntergeladenen Annotation im GTF-Format (Gene transfer format), um die Annotationen vorzubereiten. Geben Sie zum Ausführen Folgendes in die Linux-Befehlszeile ein.
Rscript prepare_mm_exon_annotation.R annotation.gtf
HINWEIS: Die GTF-Datei enthält mehrere Exon-Einträge für verschiedene Isoformen. Diese Datei wird verwendet, um die mehreren Transkript-IDs für jedes Exon zu "reduzieren". Es ist ein wichtiger Schritt, Exon-Zählbehälter zu definieren.
- Führen Sie die ergänzende Codedatei "prepare_mm_exon_annotation. R" mit der heruntergeladenen Annotation im GTF-Format (Gene transfer format), um die Annotationen vorzubereiten. Geben Sie zum Ausführen Folgendes in die Linux-Befehlszeile ein.
- Lesevorgänge zählen. Der nächste Schritt besteht darin, die Anzahl der Lesevorgänge zu zählen, die verschiedenen Transkripten / Exons zugeordnet sind. Siehe Zusatzdatei: "AS_analysis_RNASeq.Rmd".
- Erforderliche Bibliotheken laden:
packages<- c("Rsubread","tidyverse", "magrittr", "EnhancedVolcano", "edgeR","openxlsx")
invisible(lapply(packages, library, character.only=TRUE)) - Laden Sie die verarbeitete Anmerkungsdatei, die Sie aus dem vorherigen Schritt (2.2) erhalten haben.
load("mm_exon_anno.RData") - Lesen Sie alle in Schritt 2.2.2 erhaltenen BAM-Dateien als Eingabe für 'featureCounts', um Lesevorgänge zu zählen. Lesen Sie den Ordner, der die BAM-Dateien enthält, indem Sie zuerst jede Datei aus dem Verzeichnis auflisten, das mit .bam endet. Verwenden Sie 'featureCounts' aus dem Rsubread-Paket, das bam-Dateien und verarbeitete GTF-Anmerkungen (Referenz) als Eingabe verwendet, um eine Matrix von Zählungen zu generieren, die jedem Feature zugeordnet sind, mit Zeilen, die Exons (Features) und Spalten darstellen, die Beispiele darstellen.
countData <- dir("bams", pattern=".bam$", full.names=T) %>%
featureCounts(annot.ext=anno,
isGTFAnnotationFile=FALSE,
minMQS=0,useMetaFeatures=FALSE,
allowMultiOverlap=TRUE,
largestOverlap=TRUE,
countMultiMappingReads=FALSE,
primaryOnly=TRUE,
isPairedEnd=TRUE,
nthreads=12) - Führen Sie als Nächstes eine unspezifische Filterung durch, um schwach exprimierte Exons zu entfernen ("unspezifisch" zeigt an, dass die experimentellen Zustandsinformationen nicht in der Filterung verwendet werden, um Selektionsverzerrungen zu vermeiden). Transformieren Sie die Daten von der Rohskala in Counts per Million (cpm) mit der cpm-Funktion aus dem Paket 'edgeR'23 und halten Sie Exons mit Zählungen über einem einstellbaren Schwellenwert (für diesen Datensatz wird ein cpm verwendet) in mindestens drei Stichproben. Entfernen Sie auch Gene mit nur einem Exon.
# Non-specific filtering: Remove the exons with low counts
isexpr<- rownames(countData$counts)[rowSums(cpm(countData$counts)>1) >=3]
countData$counts<-countData$counts[rownames(countData$counts) %in%isexpr, ]
anno<-anno%>% filter(GeneID%in% rownames(countData$counts))
# Remove genes with only 1 site and NA in geneIDs
dn<-anno%>%group_by(GeneID)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(GeneID))
anno<-anno%>% filter(GeneID%in%dn$GeneID)
countData$counts<-countData$counts[rownames(countData$counts) %in%anno$GeneID, ]
HINWEIS: Überprüfen Sie die erforderlichen Parameter für featureCounts, wenn Sie andere Daten verwenden, z. B. für Single-End-Lesevorgänge, setzen Sie 'isPairedEnd = FALSE'. Lesen Sie das RSubread-Benutzerhandbuch, um die Optionen für Ihre Daten auszuwählen, und lesen Sie den Abschnitt Diskussion unten.
- Erforderliche Bibliotheken laden:
- Differentielles Spleißen und Exon-Nutzungsanalyse. Wir beschreiben zwei Alternativen für diesen Schritt: DEXSeq und DiffSplice. Beide können verwendet werden und ähnliche Ergebnisse liefern. Wählen Sie aus Gründen der Konsistenz DEXSeq aus, wenn Sie ein DESeq2-Paket für DGE bevorzugen, und verwenden Sie DiffSplice für eine Limma-basierte DGE-Analyse. Siehe Zusatzdatei: "AS_analysis_RNASeq.Rmd".
- Verwendung des DEXSeq-Pakets für die differentielle Exon-Analyse.
- Laden Sie die Bibliothek und erstellen Sie eine Beispieltabelle, um den Versuchsentwurf zu definieren.
library(DEXSeq)
sampleTable<-data.frame(row.names= c("Mbnl1KO_Thymus_1", "Mbnl1KO_Thymus_2", "Mbnl1KO_Thymus_3", "WT_Thymus_1", "WT_Thymus_2", "WT_Thymus_3"), condition= rep(c("Mbnl1_KO", "WT"),c(3,3)), libType= rep(c("paired-end")))
Hinweis: Die Zeilennamen sollten mit den BAM-Dateinamen übereinstimmen, die von featureCounts zum Zählen der Lesevorgänge verwendet werden. sampleTable besteht aus Details jedes Beispiels, darunter: Bibliothekstyp und -bedingung. Dies ist erforderlich, um die Kontraste oder die Testgruppe zum Erkennen der differentiellen Verwendung zu definieren. - Bereiten Sie die Exon-Informationsdatei vor. Exon-Informationen in Form von GRanges (genomic ranges) Objekten (https://bioconductor.org/packages/release/bioc/html/GenomicRanges.html) werden als Eingabe benötigt, um das DEXSeq-Objekt im nächsten Schritt zu erstellen. Ordnen Sie die Gen-IDs den Lesezahlen zu, um das exoninfo-Objekt zu erstellen.
exoninfo<-anno[anno$GeneID%in% rownames(countData$counts),]
exoninfo<-GRanges(seqnames=anno$Chr,
ranges=IRanges(start=anno$Start, end=anno$End, width=anno$Width),strand=Rle(anno$Strand))
mcols(exoninfo)$TranscriptIDs<-anno$TranscriptIDs
mcols(exoninfo)$Ticker<-anno$Ticker
mcols(exoninfo)$ExonID<-anno$ExonID
mcols(exoninfo)$n<-anno$n
mcols(exoninfo)$GeneID<-anno$GeneID
transcripts_l= strsplit(exoninfo$TranscriptIDs, "\\,")
save(countData, sampleTable, exoninfo, transcripts_l, file="AS_countdata.RData") - Erstellen Sie das DEXSeq-Objekt mithilfe der DEXSeqDataSet-Funktion. Das DEXSeq-Objekt sammelt Leseanzahl, Exon-Feature-Informationen und Beispielinformationen. Verwenden Sie die in Schritt 3 generierten Lesezähler und die Exon-Informationen aus dem vorherigen Schritt, um das DEXSeq-Objekt aus der Zählmatrix zu erstellen. Das sampleData-Argument verwendet eine Datenrahmeneingabe, die die Beispiele (und ihre Attribute: Bibliothekstyp und -bedingung) definiert, 'design' verwendet sampleData, um eine Entwurfsmatrix für den differentiellen Test unter Verwendung der Modellformelnotation zu generieren. Beachten Sie, dass ein signifikanter Interaktionsterm, condition:exon, angibt, dass der Anteil der Reads über ein Gen, das auf ein bestimmtes Exon fällt, von der experimentellen Bedingung abhängt, dh es gibt AS. Eine vollständige Beschreibung des Festlegens der Modellformel für komplexere Versuchspläne finden Sie in der DEXSeq-Dokumentation. Für Merkmalsinformationen sind Exon-IDs, entsprechende Gene und Transkripte erforderlich.
dxd<-DEXSeqDataSet(countData$counts,sampleData=sampleTable, design=~sample+exon+condition:exon,featureID=exoninfo$ExonID,groupID=exoninfo$GeneID,featureRanges=exoninfo, transcripts=transcripts_l) - Normalisierung und Dispersionsschätzung. Führen Sie als Nächstes eine Normalisierung zwischen den Proben durch und schätzen Sie die Varianz der Daten aufgrund des Poisson-Zählrauschens aufgrund der diskreten Natur der RNA-seq und der biologischen Variabilität mit den folgenden Befehlen.
dxd %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - Testen Sie auf unterschiedliche Nutzung. Nach der Abschätzung der Variation testen Sie die differentielle Exon-Nutzung für jedes Gen und generieren die Ergebnisse.
dxd%<>%testForDEU%>%estimateExonFoldChanges(fitExpToVar=
"condition")#Estimate fold changes
dxr=DEXSeqResults(dxd) - Visualisieren Sie Spleißereignisse für ausgewählte Gene mit dem folgenden Befehl.
plotDEXSeq(dxr,"Wnk1", displayTranscripts=TRUE, splicing=TRUE,legend
=TRUE,cex.axis=1.2,cex=1.3,lwd=2)
Untersuchen Sie die R-Notebook-Datei "AS_analysis_RNASeq.Rmd", um zusätzliche Diagramme für Gene von Interesse zu generieren und Vulkandiagramme mit unterschiedlichen Schwellenwerten zu generieren.
- Laden Sie die Bibliothek und erstellen Sie eine Beispieltabelle, um den Versuchsentwurf zu definieren.
- Verwendung von diffSplice von Limma zur Identifizierung von differentiellem Spleißen. Folgen Sie der R-Notebook-Datei "AS_analysis_RNASeq.Rmd". Stellen Sie sicher, dass die Schritte 2.1-2.3 befolgt wurden, um Eingabedateien vorzubereiten, bevor Sie fortfahren.
- Bibliotheken laden
library(limma)
library(edgeR) - Unspezifische Filterung. Extrahieren Sie die Matrix der Lesezähler, die in 2.3 erhalten wurden. Erstellen Sie eine Liste von Features mit der Funktion 'DGEList' aus dem edgeR-Paket, wobei Zeilen Gene und Spalten Proben darstellen.
mycounts=countData$counts
#Change the rownames of the countdata to exon Ids instead of genes for unique rownames.
rownames(mycounts) = exoninfo$ExonID
dge<-DGEList(counts=mycounts)
#Filtering
isexpr<- rowSums(cpm(dge)>1) >=3
dge<-dge[isexpr,,keep.lib.sizes=FALSE]
#Extract the exon annotations for only transcripts meeting non-specific filter
exoninfo=anno%>% filter(ExonID%in% rownames(dge$counts))
#Convert the exoninfo into GRanges object
exoninfo1<-GRanges(seqnames=exoninfo$Chr,
ranges=IRanges(start=exoninfo$Start, end=exoninfo$End, width=exoninfo$Width),strand=Rle(exoninfo$Strand))
mcols(exoninfo1)$TranscriptIDs<-exoninfo$TranscriptIDs
mcols(exoninfo1)$Ticker<-exoninfo$Ticker
mcols(exoninfo1)$ExonID<-exoninfo$ExonID
mcols(exoninfo1)$n<-exoninfo$n
mcols(exoninfo1)$GeneID<-exoninfo$GeneID
transcripts_l= strsplit(exoninfo1$TranscriptIDs, "\\,")
HINWEIS: Als unspezifischer Filterschritt werden die Zählungen nach cpm < 1 in x aus n Stichproben gefiltert, wobei x die minimale Anzahl von Replikaten in einer beliebigen Bedingung ist. n = 6 und x = 3 für diese Beispieldaten. - Normalisieren Sie die Anzahl über Stichproben hinweg, mit der Funktion 'calcNormFactors' aus dem Paket 'edgeR' unter Verwendung des getrimmten Mittelwerts von M-Werten (TMM-Normalisierungsmethode)24 Es werden Skalierungsfaktoren berechnet, um Bibliotheksgrößen anzupassen.
dge<-calcNormFactors(dge) - Verwenden Sie sampleTable wie in Schritt 2.4.1.1 generiert, und erstellen Sie die Designmatrix. Die Designmatrix prägt das Design. Weitere Informationen zu Designmatrizen für fortgeschrittenere experimentelle Designs finden Sie im Limma User Guide (https://www.bioconductor.org/packages/devel/bioc/vignettes/limma/inst/doc/usersguide.pdf) Kapitel 8 & 9.
Treat<- factor(sampleTable$condition)
design<- model.matrix(~0+Treat)
colnames(design) <- levels(Treat) - Passen Sie ein lineares Modell pro Exon an. Führen Sie die 'voom'-Funktion des 'limma'-Pakets aus, um RNA-seq-Daten zu verarbeiten, um die Varianz zu schätzen und Präzisionsgewichte zur Korrektur des Poisson-Zählrauschens zu generieren, und transformieren Sie die Exon-Level-Zählungen in log2-counts per million (logCPM). Führen Sie dann eine lineare Modellierung mit der Funktion 'lmfit' aus, um lineare Modelle an die Ausdrucksdaten für jedes Exon anzupassen. Berechnen Sie empirische Bayes-Statistiken für das angepasste Modell mit der Funktion 'eBayes', um differentielle Exon-Expressionen zu erkennen. Als nächstes definieren Sie eine Kontrastmatrix für die experimentellen Vergleiche von Interesse. Verwenden Sie 'contrasts.fit', um Koeffizienten und Standardfehler für jedes Vergleichspaar zu erhalten.
v<-voom(dge,design,plot=FALSE)
fit<-lmFit(v,design)
fit<-eBayes(fit)
colnames(fit)
cont.matrix<-makeContrasts(
Mbnl1_KO_WT=Mbnl1_KO-WT,
levels=design)
fit2<-contrasts.fit(fit,cont.matrix) - Differentielle Spleißanalyse. Führen Sie 'diffSplice' auf dem angepassten Modell aus, um die Unterschiede in der Exon-Nutzung von Genen zwischen Wildtyp und Knockout zu testen und die Top-Ranking-Ergebnisse mit der 'topSplice'-Funktion zu untersuchen: test="t" ergibt eine Rangfolge von AS-Exons, test="simes" gibt eine Rangfolge von Genen.
ex<-diffSplice(fit2,geneid=exoninfo$GeneID,exonid=exoninfo$ExonID)
ts<-topSplice(ex,n=Inf,FDR=0.1, test="t", sort.by="logFC")
tg<-topSplice(ex,n=Inf,FDR=0.1, test="simes") - Visualisierung. Zeichnen Sie die Ergebnisse mit der Funktion 'plotSplice', wodurch das Gen von Interesse im Genid-Argument entsteht. Speichern Sie die Top-Ergebnisse sortiert nach Log Fold in ein Objekt und generieren Sie ein Vulkandiagramm, um die Exons auszustellen.
plotSplice(ex,geneid="Wnk1", FDR=0.1)
#Volcano plot
EnhancedVolcano(ts,lab=ts$ExonID,selectLab= head((ts$ExonID),2000), xlab= bquote(~Log[2]~'fold change'), x='logFC', y='P.Value', title='Volcano Plot', subtitle='Mbnl1_KO vs WT (Limma_diffSplice)', FCcutoff=2, labSize=4,legendPosition="right", caption= bquote(~Log[2]~"Fold change cutoff, 2; FDR 10%"))
- Bibliotheken laden
- Verwendung von rMATS
- Stellen Sie sicher, dass die neueste Version von rMATS v4.1.1 (aufgrund der reduzierten Verarbeitungszeit und des geringeren Speicherbedarfs auch als rMATS turbo bezeichnet) entweder mit conda oder github (https://github.com/Xinglab/rmats-turbo/releases/download/v4.1.1/rmats_turbo_v4_1_1.tar.gz) im Arbeitsverzeichnis installiert ist. Folgen Sie Abschnitt 4.3 in "AS_analysis_RNASeq.Rmd".
- Wechseln Sie zu dem Ordner, der die BAM-Dateien enthält, die nach dem Mapping abgerufen wurden, und bereiten Sie Textdateien, wie von rMATES gefordert, für die beiden Bedingungen vor, indem Sie den Namen der BAM-Dateien (zusammen mit dem Pfad) getrennt durch ',' kopieren. Die folgenden Befehle sollten in der Linux-Befehlszeile ausgeführt werden:
mkdir rMATS_analysis
cd bams/
ls -pd "$PWD"/*| grep "WT"| tr '\n'','> Wt.txt
ls -pd "$PWD"/*| grep "Mb"| tr '\n'','> KO.txt
mv *.txt ../rMATS_analysis - Führen Sie rmats.py mit den beiden im vorherigen Schritt generierten Eingabedateien zusammen mit der in 2.1.1.3 erhaltenen GTF-Datei aus. Dadurch wird ein Ausgabeordner 'rmats_out' generiert, der Textdateien enthält, die Statistiken (p-Werte und Einschlussstufen) für jedes Spleißereignis separat beschreiben.
python rmats-turbo/rmats.py --b1 KO.txt --b2 Wt.txt --gtf annotation.gtf -t paired --readLength 50 --nthread 8 --od rmats_out/ --tmp rmats_tmp --task pos
HINWEIS: Die Referenzanmerkung in Form einer GTF-Datei ist ebenfalls erforderlich. Überprüfen Sie die Parameter, wenn die Daten Single-End sind, und ändern Sie die Option -t entsprechend. - Untersuchen von rMATS-Ergebnissen. Verwenden Sie das Bioconductor-Paket 'maser'25 , um die rMATS-Ergebnisse zu untersuchen. Laden Sie die JCEC-Textdateien (Junction and Exon Counts) in das Maser-Objekt und filtern Sie das Ergebnis basierend auf der Abdeckung, indem Sie mindestens fünf durchschnittliche Lesevorgänge pro Spleißereignis einbeziehen.
library(maser)
mbnl1<-maser("/rmats_out/", c("WT","Mbnl1_KO"), ftype="JCEC")
#Filtering out events by coverage
mbnl1_filt<-filterByCoverage(mbnl1,avg_reads=5) - Visualisierung von rMATS-Ergebnissen. Wählen Sie die signifikanten Spleißereignisse mit der False Discovery Rate (FDR) 10% und der minimalen Änderung von 10% in Percent Spliced In (deltaPSI) mit der Funktion 'topEvents' aus dem Paket 'maser' aus. Als nächstes überprüfen Sie die Genereignisse für einzelne Gene von Interesse (Probengen-Wnk1) und zeichnen PSI-Werte für jedes Spleißereignis dieses Gens. Generieren Sie ein Vulkandiagramm, indem Sie den Ereignistyp angeben.
#Top splicing events at 10% FDR
mbnl1_top<-topEvents(mbnl1_filt,fdr=0.1, deltaPSI=0.1)
mbnl1_top
#Check the gene events for a particular gene
mbnl1_wnk1<-geneEvents(mbnl1_filt,geneS="Wnk1", fdr=0.1, deltaPSI=0.1)
maser::display(mbnl1_wnk1,"SE")
plotGenePSI(mbnl1_wnk1,type="SE", show_replicates
=TRUE)
volcano(mbnl1_filt,fdr=0.1, deltaPSI=0.1,type="SE")
+xlab("deltaPSI")+ylab("Log10 Adj. Pvalue")+ggtitle("Volcano Plot of exon skipping events") - Generieren Sie Sashimi-Plots für das mit rMATS erhaltene Spleißereignisergebnis in Form von Textdateien mit dem Paket 'rmats2shahimiplot'. Führen Sie das Python-Skript über die Linux-Befehlszeile aus.
python ./src/rmats2sashimiplot/rmats2sashimiplot.py --b1 ../bams/WT_Thymus_1.bam,../bams/WT_Thymus_2.bam,../bams/WT_Thymus_3.bam --b2 ../bams/Mbnl1KO_Thymus_1.bam,../bams/Mbnl1KO_Thymus_2.bam,../bams/Mbnl1KO_Thymus_3.bam -t SE -e ../rMATS_analysis/rmats_out/SE.MATS.JC.txt --l1 WT --l2 Mbnl1_KO --exon_s 1 --intron_s 5 -o ../rMATS_analysis/rmats2shasmi_output
HINWEIS: Dieser Prozess kann zeitaufwändig sein, da er das Sashimi-Diagramm für alle Ergebnisse in der Ereignisdatei generiert. Wählen Sie die Top-Ergebnisse (Gennamen und Exons), wie sie von der topEvents-Funktion aus 'maser' angezeigt werden, und visualisieren Sie das entsprechende Sashimi-Diagramm.
- Verwendung des DEXSeq-Pakets für die differentielle Exon-Analyse.
3. Alternative Polyadenylierung (APA) Analyse mittels 3'-Endsequenzierung
- Herunterladen und Vorverarbeiten von Daten
HINWEIS: Weitere Informationen finden Sie in der ergänzenden R-Notebook-Datei "APA_analysis_3PSeq_notebook. Rmd" für die vollständigen Befehle zum Herunterladen und Vorverarbeiten von Daten oder führen Sie die ergänzende Bash-Datei "APA_data_downloading_preprocessing.sh" auf der Linux-Befehlszeile aus.- Laden Sie Daten von SRA mit den Beitritts-IDs (1553129 bis 1553136) herunter.
- Trimmadapter und umgekehrtes Komplement, um die Sensorstrangsequenz zu erhalten.
HINWEIS: Dieser Schritt ist spezifisch für den verwendeten PolyA-seq-Assay. - Die Karte liest die Genomassemblierung der Maus mit dem Bowtie Aligner26.
- Vorbereiten von pA-Site-Anmerkungen.
HINWEIS: Die Verarbeitung der pA-Site-Annotationsdatei erfolgt zunächst unter Verwendung der ergänzenden R-Notebook-Datei "APA_analysis_3PSeq_notebook. Rmd" (2.1 - 2.6) und dann die Bash-Datei "APA_annotation_preparation.sh".- Laden Sie die pA-Site-Anmerkungen aus der PolyASite 2.0-Datenbankherunter 6.
- Wählen Sie pA-Site-Annotationen aus, um 3'-untranslatierte Region (UTR)-pA-Sites beizubehalten, die als Terminal Exon (TE) oder 1000 nt stromabwärts eines annotierten Terminal-Exons (DS) für die Downstream-Analyse annotiert sind.
- Ermitteln Sie pA-Site-Peaks. Ankern Sie an jeder pA-Spaltstelle und visualisieren Sie die durchschnittliche Leseabdeckung mit Bettwerkzeugen und Deeptools27,28. Die Ergebnisse zeigten, dass die Peaks der kartierten Lesevorgänge hauptsächlich innerhalb von ~60 bp stromaufwärts der Spaltstellen verteilt waren (Abbildung 5 und ergänzende Abbildung 5). Daher wurden die Koordinaten von pA-Standorten aus der Annotationsdatei auf 60 bp stromaufwärts ihrer Spaltstellen erweitert. Abhängig vom verwendeten spezifischen 3'-Endsequenzierungsprotokoll muss dieser Schritt für andere Assays als PolyA-seq optimiert werden.
- Zählen von Lesevorgängen
- Bereiten Sie die Anmerkungsdatei für pA-Sites vor.
anno<- read.table(file= "flanking60added.pA_annotation.bed",
stringsAsFactors=FALSE, check.names=FALSE, header=FALSE, sep="")
colnames(anno) <- c("chrom", "chromStart", "chromEnd", "name", "score", "strand", "rep", "annotation", "gene_name", "gene_id")
anno<- dplyr::select(anno,name,chrom, chromStart,chromEnd, strand,gene_id,gene_name,rep)
colnames(anno) <- c("GeneID", "Chr", "Start", "End", "Strand", "Ensembl", "Symbol", "repID") - Wenden Sie 'featureCounts' an, um Rohzählungen zu erfassen. Speichern Sie die Zähltabelle als RData-Datei "APA_countData.Rdata" für die APA-Analyse mit verschiedenen Tools.
countData<- dir("bamfiles", pattern="sorted.bam$", full.names=TRUE) %>%
# Read all bam files as input for featureCounts
featureCounts(annot.ext=anno, isGTFAnnotationFile= FALSE,minMQS=0,useMetaFeatures= TRUE,allowMultiOverlap=TRUE, largestOverlap= TRUE,strandSpecific=1, countMultiMappingReads =TRUE,primaryOnly= TRUE,isPairedEnd= FALSE,nthreads=12)%T>%
save(file="APA_countData.Rdata")
HINWEIS: Achten Sie darauf, einen der in der Funktion "featureCounts" aufgeführten Parameter zu ändern. Modifizieren Sie den Parameter 'strandSpecific', um sicherzustellen, dass er mit der Sequenzierungsrichtung des verwendeten 3'-Endsequenzierungsassays übereinstimmt (empirisch wird dies durch die Visualisierung der Daten in einem Genombrowser über Gene auf Plus- und Minussträngen verdeutlicht). - Wenden Sie eine unspezifische Filterung von countData an. Die Filterung kann die statistische Robustheit in differentiellen pA-Site-Nutzungstests erheblich verbessern. Zuerst entfernten wir jene Gene mit nur einer pA-Site, auf der die differentielle pA-Site-Nutzung nicht definiert werden kann. Zweitens wenden wir eine unspezifische Filterung basierend auf der Abdeckung an: Die Anzahl wird nach cpm gefiltert, die kleiner als 1 in x aus n Stichproben ist, wobei x die minimale Anzahl von Replikaten unter jeder Bedingung ist. N = 8 und x = 2 für diese Beispieldaten.
load(file= "APA_countData.Rdata")# Skip this step if already loaded
# Non-specific filtering: Remove the pA sites not differentially expressed in the samples
countData<-countData$counts%>%as.data.frame%>% .[rowSums(edgeR::cpm(.)>1) >=2, ]
anno%<>% .[.$GeneID%in% rownames(countData), ]
# Remove genes with only 1 site and NA in geneIDs
dnsites<-anno%>%group_by(Symbol)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(Symbol))
anno<-anno%>% filter(Symbol%in%dnsites$Symbol)
countData<-countData[rownames(countData) %in%anno$GeneID, ]
- Bereiten Sie die Anmerkungsdatei für pA-Sites vor.
- Nutzungsanalyse der differentiellen Polyadenylierungsstelle mit DEXSeq- und diffSplice-Pipelines.
- Verwenden des DEXSeq-Pakets
HINWEIS: Da für die DEXSeq-Pipeline keine Kontrastmatrix definiert werden kann, muss die differentielle APA-Analyse von jeweils zwei experimentellen Bedingungen separat durchgeführt werden. Die differentielle APA-Analyse der Bedingung WT und der Bedingung DKO wird beispielhaft zur Erläuterung des Verfahrens durchgeführt. Siehe ergänzende Datei "APA_analysis_3PSeq_notebook. Rmd" für den Schritt-für-Schritt-Workflow dieses Abschnitts und die differentielle APA-Analyse anderer Kontraste.- Laden Sie die Bibliothek und erstellen Sie eine Beispieltabelle, um den Versuchsentwurf zu definieren.
c("DEXSeq", "GenomicRanges") %>% lapply(library, character.only=TRUE) %>%invisible
sampleTable1<- data.frame(row.names= c("WT_1","WT_2","DKO_1","DKO_2"),
condition= c(rep("WT", 2), rep("DKO", 2)),
libType= rep("single-end", 4)) - Bereiten Sie die Informationsdatei für pA-Standorte mit dem Bioconductor-Paket GRanges vor.
# Prepare the GRanges object for DEXSeqDataSet object construction
PASinfo <- GRanges(seqnames = anno$Chr,
ranges = IRanges(start = anno$Start, end = anno$End),strand = Rle(anno$Strand))
mcols(PASinfo)$PASID<-anno$repID
mcols(PASinfo)$GeneEns<-anno$Ensembl
mcols(PASinfo)$GeneID<-anno$Symbol
# Prepare the new feature IDs, replace the strand information with letters to match the current pA site clusterID
new.featureID <- anno$Strand %>% as.character %>% replace(. %in% "+", "F") %>% replace(. %in% "-", "R") %>% paste0(as.character(anno$repID), .) - Verwenden Sie die in Schritt 3.3 generierten Lesezähler und die pA-Siteinformationen aus dem vorherigen Schritt, um das DEXSeq-Objekt zu erstellen.
# Select the read counts of the condition WT and DKO
countData1<- dplyr::select(countData, SRR1553129.sorted.bam, SRR1553130.sorted.bam, SRR1553131.sorted.bam, SRR1553132.sorted.bam)
# Rename the columns of countData using sample names in sampleTable
colnames(countData1) <- rownames(sampleTable1)
dxd1<-DEXSeqDataSet(countData=countData1,
sampleData=sampleTable1,
design=~sample+exon+condition:exon,
featureID=new.featureID,
groupID=anno$Symbol,
featureRanges=PASinfo) - Definieren Sie das Kontrastpaar, indem Sie die Ebenen der Bedingungen im DEXSeq-Objekt definieren.
dxd1$condition<- factor(dxd1$condition, levels= c("WT", "DKO"))
# The contrast pair will be "DKO - WT" - Normalisierung und Dispersionsschätzung. Ähnlich wie bei RNA-seq-Daten führen 3'-Endsequenzierungsdaten eine Normalisierung zwischen Proben (spaltenweiser Median der Verhältnisse für jede Probe) mit der Funktion "estimateSizeFactors" durch und schätzen die Variation der Daten mit der Funktion "estimateDispersions" und visualisieren dann das Ergebnis der Dispersionsschätzung mit der Funktion "plotDispEsts".
dxd1 %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - Differentielle pA-Site-Nutzungstests für jedes Gen mit der Funktion 'testForDEU', dann Schätzung der Faltenänderung der pA-Site-Nutzung mit der Funktion 'estimateExonFoldChanges'. Überprüfen Sie die Ergebnisse mit der Funktion 'DEXSeqResults' und legen Sie 'FDR < 10%' als Kriterium für signifikant differentielle pA-Standorte fest.
dxd1 %<>% testForDEU %>% estimateExonFoldChanges(fitExpToVar = "condition")
dxr1 <- DEXSeqResults(dxd1)
dxr1
mcols(dxr1)$description
table(dxr1$padj<0.1) # Check the number of differential pA sites (FDR < 0.1)
table(tapply(dxr1$padj<0.1, dxr1$groupID, any)) # Check the number of gene overlapped with differential pA site - Visualisierung der differentiellen pA-Site-Nutzungsergebnisse mit differentiellen APA-Diagrammen, die von der Funktion 'plotDEXSeq' und volcano plot von der Funktion 'EnhancedVolcano' generiert werden.
# Select the top 100 significant differential pA sites ranked by FDR
topdiff.PAS<- dxr1%>%as.data.frame%>%rownames_to_column%>%arrange(padj)%$%groupID[1:100]
# Apply plotDEXSeq for the visualization of differential polyA usage
plotDEXSeq(dxr1,"S100a7a", legend=TRUE, expression=FALSE,splicing=TRUE, cex.axis=1.2, cex=1.3,lwd=2)
# Apply perGeneQValue to check the top genes with differential polyA site usage
dxr1%<>% .[!is.na(.$padj), ]
dgene<- data.frame(perGeneQValue= perGeneQValue (dxr1)) %>%rownames_to_column("groupID")
dePAS_sig1<-dxr1%>% data.frame() %>%
dplyr::select(-matches("dispersion|stat|countData|genomicData"))%>%
inner_join(dgene)%>%arrange(perGeneQValue)%>%distinct()%>%
filter(padj<0.1)
# Apply EnhancedVolcano package to visualise differential polyA site usage
"EnhancedVolcano"%>% lapply(library, character.only=TRUE) %>%invisible
EnhancedVolcano(dePAS_sig1, lab=dePAS_sig1$groupID, x='log2fold_DKO_WT',
y='pvalue',title='Volcano Plot',subtitle='DKO vs WT',
FCcutoff=1,labSize=4, legendPosition="right",
caption= bquote(~Log[2]~"Fold change cutoff, 1; FDR 10%"))
- Laden Sie die Bibliothek und erstellen Sie eine Beispieltabelle, um den Versuchsentwurf zu definieren.
- Verwenden des diffSplice-Pakets. Weitere Informationen finden Sie in der ergänzenden R Notebook-Datei "APA_analysis_3PSeq_notebook. Rmd" für den schrittweisen Workflow dieses Abschnitts.
- Definieren Sie Kontraste, die für die Analyse der differentiellen pA-Nutzung von Interesse sind.
Hinweis: Dieser Schritt sollte nach der Konstruktion und Verarbeitung des DGEList-Objekts ausgeführt werden, das in der R-Notebook-Datei "APA_analysis_3PSeq_notebook. Rmd".contrast.matrix<-makeContrasts(DKO_vs_WT=DKO-
WT,Ctrl_vs_DKO=Ctrl-DKO,
KD_vs_Ctrl=KD-Ctrl,KD_vs_DKO=KD-DKO,levels=design)
fit2<-fit%>%contrasts.fit(contrast.matrix)%>%eBayes
summary(decideTests(fit2))
ex<-diffSplice(fit2,geneid=anno$Symbol,exonid=new.featureID)
topSplice(ex) #Check the top significant results with topSplice - Visualisieren Sie das Ergebnis von Kontrasten von Interesse (hier "DKO - WT") mit differentiellen APA-Diagrammen mit der Funktion 'plotSplice' und Vulkandiagrammen mit der Funktion 'EnhancedVolcano'. Weitere Informationen finden Sie in der R-Notebook-Datei "APA_analysis_3PSeq_notebook. Rmd" 4.2.7 - 4.2.9 zur Visualisierung anderer Kontrastpaare.
sig1<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="t", sort.by="logFC")
sig1.genes<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="simes")
plotSplice(ex, coef=1,geneid="S100a7a", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Tpm1", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Smc6", FDR = 0.1)
EnhancedVolcano(sig1, lab=sig1$GeneID,xlab= bquote(~Log[2]~'fold change'),
x='logFC', y='P.Value', title='Volcano Plot', subtitle='DKO vs WT',
FCcutoff=1, labSize=6, legendPosition="right")
- Definieren Sie Kontraste, die für die Analyse der differentiellen pA-Nutzung von Interesse sind.
- Verwenden des DEXSeq-Pakets
Ergebnisse
Nach dem Ausführen des obigen Schritt-für-Schritt-Workflows werden die AS- und APA-Analyseausgaben und repräsentativen Ergebnisse in Form von Tabellen und Datendiagrammen erstellt, die wie folgt generiert werden.
WIE:
Die Hauptergebnisse der AS-Analyse (Zusatztabelle 1 für diffSplice; Tabelle 2 für DEXSeq) ist eine Liste von Exons, die eine unterschiedliche Nutzung über Bedingungen hinweg zeigen, und eine Liste von Genen, die eine sig...
Diskussion
In dieser Studie evaluierten wir Exon-basierte und ereignisbasierte Ansätze zum Nachweis von AS und APA in Bulk-RNA-Seq- und 3'-Endsequenzierungsdaten. Die Exon-basierten AS-Ansätze erzeugen sowohl eine Liste differentiell exprimierter Exons als auch ein Ranking auf Genebene, geordnet nach der statistischen Signifikanz der gesamten differentiellen Spleißaktivität auf Genebene (Tabellen 1-2, 4-5). Für das diffSplice-Paket wird die differentielle Nutzung bestimmt, indem gewichtete lineare Modelle auf ...
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Diese Studie wurde von einem Australian Research Council (ARC) Future Fellowship (FT16010043) und ANU Futures Scheme unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Not relevent for computational study |
Referenzen
- Katz, Y., Wang, E. T., Airoldi, E. M., Burge, C. B. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods. 7 (12), 1009-1015 (2010).
- Wang, Y., et al. Mechanism of alternative splicing and its regulation. Biomedical Reports. 3 (2), 152-158 (2015).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in Bioinformatics. 21 (6), 2052-2065 (2020).
- Movassat, M., et al. Coupling between alternative polyadenylation and alternative splicing is limited to terminal introns. RNA Biology. 13 (7), 646-655 (2016).
- Tian, B., Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature Reviews Molecular Cell Biology. 18 (1), 18-30 (2017).
- Herrmann, C. J., et al. PolyASite 2.0: a consolidated atlas of polyadenylation sites from 3' end sequencing. Nucleic Acids Research. 48 (1), 174-179 (2020).
- Liu, R., Loraine, A. E., Dickerson, J. A. Comparisons of computational methods for differential alternative splicing detection using RNA-seq in plant systems. BMC Bioinformatics. 15 (1), 364 (2014).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (1), 13 (2016).
- Anders, S., Reyes, A., Huber, W. Detecting differential usage of exons from RNA-seq data. Genome Research. 22 (10), 2008-2017 (2012).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2014).
- Shen, S., et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data. Proceedings of the National Academy of Sciences. 111 (51), 5593-5601 (2014).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in bioinformatics. 21 (6), 2052-2065 (2020).
- Kanitz, A., et al. Comparative assessment of methods for the computational inference of transcript isoform abundance from RNA-seq data. Genome biology. 16 (1), 1-26 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Sznajder, L. J., et al. Loss of MBNL1 induces RNA misprocessing in the thymus and peripheral blood. Nature Communications. 11, 1-11 (2020).
- Batra, R., et al. Loss of MBNL leads to disruption of developmentally regulated alternative polyadenylation in RNA-mediated disease. Molecular Cell. 56 (2), 311-322 (2014).
- Leinonen, R., Sugawara, H., Shumway, M., et al. The sequence read archive. Nucleic acids research. 39, 19-21 (2010).
- Tange, O. . GNU parallel-the command-line power tool. 36, 42-47 (2011).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), 25 (2010).
- Veiga, D. F. T. maser: Mapping Alternative Splicing Events to pRoteins. R package version 1.4.0. , (2019).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (13), 25 (2009).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ramírez, F., Dündar, F., Diehl, S., Grüning, B. A., Manke, T. deepTools: a flexible platform for exploring deep-sequencing data. Nucleic acids research. 42 (1), 187-191 (2014).
- Merino, G. A., Conesa, A., Fernández, E. A. A benchmarking of workflows for detecting differential splicing and differential expression at isoform level in human RNA-seq studies. Briefings in bioinformatics. 20 (2), 471-481 (2019).
- Chhangawala, S., Rudy, G., Mason, C. E., Rosenfeld, J. A. The impact of read length on quantification of differentially expressed genes and splice junction detection. Genome biology. 16 (1), 1-10 (2015).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biol. 17, 13 (2016).
- Trapnell, C., et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 7 (3), 562-578 (2012).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 12, 323 (2011).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat Biotechnol. 34 (5), 525-527 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten