È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Identificazione di splicing alternativo e poliadenilazione in dati di RNA-seq
In questo articolo
Riepilogo
Lo splicing alternativo (AS) e la poliadenilazione alternativa (APA) espandono la diversità delle isoforme di trascrizione e dei loro prodotti. Qui, descriviamo i protocolli bioinformatici per analizzare i saggi di sequenziamento di massa dell'RNA-seq e 3' per rilevare e visualizzare AS e APA che variano in base alle condizioni sperimentali.
Abstract
Oltre all'analisi tipica dell'RNA-Seq per misurare l'espressione genica differenziale (DGE) in condizioni sperimentali/biologiche, i dati di RNA-seq possono anche essere utilizzati per esplorare altri complessi meccanismi regolatori a livello di esone. Lo splicing alternativo e la poliadenilazione svolgono un ruolo cruciale nella diversità funzionale di un gene generando diverse isoforme per regolare l'espressione genica a livello post-trascrizionale e limitando le analisi all'intero livello genico può mancare questo importante strato regolatore. Qui, dimostriamo analisi dettagliate passo dopo passo per l'identificazione e la visualizzazione dell'utilizzo differenziale del sito di esone e poliadenilazione in tutte le condizioni, utilizzando Bioconductor e altri pacchetti e funzioni, tra cui DEXSeq, diffSplice dal pacchetto Limma e rMATS.
Introduzione
L'RNA-seq è stato ampiamente utilizzato nel corso degli anni, tipicamente per stimare l'espressione genica differenziale e la scopertagenica 1. Inoltre, può anche essere utilizzato per stimare l'uso variabile del livello di esone a causa del gene che esprime diverse isoforme, contribuendo così a una migliore comprensione della regolazione genica a livello post-trascrizionale. La maggior parte dei geni eucariotici genera diverse isoforme mediante splicing alternativo (AS) per aumentare la diversità dell'espressione dell'mRNA. Gli eventi AS possono essere suddivisi in diversi modelli: salto di esoni completi (SE) in cui un esone ("cassetta") viene completamente rimosso dalla trascrizione insieme ai suoi introni laterali; selezione alternativa (donatore) del sito di giunzione 5' (A5SS) e selezione alternativa del sito di giunzione 3' (accettore) (A3SS) quando due o più siti di giunzione sono presenti su entrambe le estremità di un esone; ritenzione degli introni (RI) quando un introne viene trattenuto all'interno del trascritto dell'mRNA maturo e mutua esclusione dell'uso dell'esone (MXE) in cui solo uno dei due esoni disponibili può essere trattenuto alla volta 2,3. La poliadenilazione alternativa (APA) svolge anche un ruolo importante nella regolazione dell'espressione genica utilizzando siti di poli alternativi (A) per generare più isoforme di mRNA da una singola trascrizione4. La maggior parte dei siti di poliadenilazione (pAs) si trovano nella regione 3' non tradotta (3' UTR), generando isoforme di mRNA con diverse lunghezze UTR 3'. Poiché l'UTR 3' è l'hub centrale per il riconoscimento degli elementi regolatori, diverse lunghezze UTR 3' possono influenzare la localizzazione, la stabilità e la traduzione dell'mRNA5. Esistono saggi di sequenziamento finale di classe 3' ottimizzati per rilevare APA che differiscono nei dettagli del protocollo6. La pipeline qui descritta è progettata per PolyA-seq, ma può essere adattata per altri protocolli come descritto.
In questo studio, presentiamo una pipeline di metodi di analisi differenziale degli esoni7,8 (Figura 1), che possono essere suddivisi in due grandi categorie: basati sull'esone (DEXSeq9, diffSplice 10) e basati sugli eventi (replicate Multivariate Analysis of Transcript Splicing (rMATS)11). I metodi basati sull'esone confrontano il cambiamento di piega tra le condizioni dei singoli esoni, contro una misura del cambiamento complessivo della piega genica per chiamare l'uso differenziale dell'esone, e da ciò calcolare una misura a livello genetico dell'attività AS. I metodi basati su eventi utilizzano letture di giunzione esone-introne per rilevare e classificare eventi di splicing specifici come il salto dell'esone o la ritenzione di introni e distinguere questi tipi di AS nell'output3. Pertanto, questi metodi forniscono punti di vista complementari per un'analisi completa di AS12,13. Abbiamo selezionato DEXSeq (basato sul pacchetto DESeq214 DGE) e diffSplice (basato sul pacchetto Limma10 DGE) per lo studio in quanto sono tra i pacchetti più utilizzati per l'analisi di splicing differenziale. rMATS è stato scelto come metodo popolare per l'analisi basata sugli eventi. Un altro metodo popolare basato su eventi è MISO (Mixture of Isoforms)1. Per l'APA adattiamo l'approccio basato sull'esone.
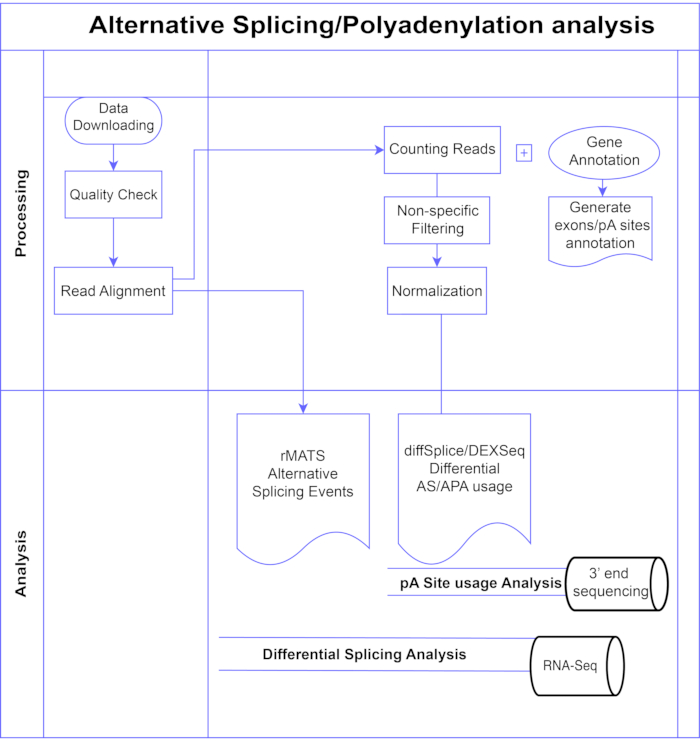
Figura 1. Pipeline di analisi. Diagramma di flusso dei passaggi utilizzati nell'analisi. I passaggi includono: ottenere i dati, eseguire controlli di qualità e allineamento delle letture seguito dal conteggio delle letture utilizzando annotazioni per esoni, introni e siti pA noti, filtraggio per rimuovere conteggi bassi e normalizzazione. I dati di PolyA-seq sono stati analizzati per siti pA alternativi utilizzando metodi diffSplice/DEXSeq, RNA-Seq di massa sono stati analizzati per lo splicing alternativo a livello di esone con metodi diffSplice/DEXseq e gli eventi AS analizzati con rMATS. Fare clic qui per visualizzare una versione ingrandita di questa figura.
I dati RNA-seq utilizzati in questa indagine sono stati acquisiti da Gene Expression Omnibus (GEO) (GSE138691)15. Abbiamo utilizzato i dati RNA-seq di topo di questo studio con due gruppi di condizioni: wild-type (WT) e Muscleblind-like type 1 knockout (Mbnl1 KO) con tre repliche ciascuno. Per dimostrare l'analisi differenziale dell'utilizzo del sito di poliadenilazione, abbiamo ottenuto dati PolyA-seq di fibroblasti embrionali di topo (MEF) (GEO Accession GSE60487)16. I dati hanno quattro gruppi di condizioni: Wild-type (WT), Muscleblind-like type1/type 2 double knockout (Mbnl1/2 DKO), Mbnl 1/2 DKO con Mbnl3 knockdown (KD) e Mbnl1/2 DKO con controllo Mbnl3 (Ctrl). Ogni gruppo di condizioni è costituito da due repliche.
| Adesione GEO | Numero di esecuzione SRA | Nome del campione | Condizione | Replicare | Fazzoletto | Sequenziamento | Lunghezza di lettura | |
| RNA-Seq | GSM4116218 | SRR10261601 | Mbnl1KO_Thymus_1 | Mbnl1 knockout | Rappresentante 1 | Timo | Estremità accoppiata | 100 pb |
| GSM4116219 | SRR10261602 | Mbnl1KO_Thymus_2 | Mbnl1 knockout | Rappresentante 2 | Timo | Estremità accoppiata | 100 pb | |
| GSM4116220 | SRR10261603 | Mbnl1KO_Thymus_3 | Mbnl1 knockout | Rappresentante 3 | Timo | Estremità accoppiata | 100 pb | |
| GSM4116221 | SRR10261604 | WT_Thymus_1 | Tipo selvaggio | Rappresentante 1 | Timo | Estremità accoppiata | 100 pb | |
| GSM4116222 | SRR10261605 | WT_Thymus_2 | Tipo selvaggio | Rappresentante 2 | Timo | Estremità accoppiata | 100 pb | |
| GSM4116223 | SRR10261606 | WT_Thymus_3 | Tipo selvaggio | Rappresentante 3 | Timo | Estremità accoppiata | 100 pb | |
| 3P-Seq | GSM1480973 | SRR1553129 | WT_1 | Tipo selvatico (WT) | Rappresentante 1 | Fibroblasti embrionali di topo (MEF) | Estremità singola | 40 pb |
| GSM1480974 | SRR1553130 | WT_2 | Tipo selvatico (WT) | Rappresentante 2 | Fibroblasti embrionali di topo (MEF) | Estremità singola | 40 pb | |
| GSM1480975 | SRR1553131 | DKO_1 | Mbnl 1/2 doppio knockout (DKO) | Rappresentante 1 | Fibroblasti embrionali di topo (MEF) | Estremità singola | 40 pb | |
| GSM1480976 | SRR1553132 | DKO_2 | Mbnl 1/2 doppio knockout (DKO) | Rappresentante 2 | Fibroblasti embrionali di topo (MEF) | Estremità singola | 40 pb | |
| GSM1480977 | SRR1553133 | DKOsiRNA_1 | Mbnl 1/2 doppio knockout con Mbnl 3 siRNA (KD) | Rappresentante 1 | Fibroblasti embrionali di topo (MEF) | Estremità singola | 40 pb | |
| GSM1480978 | SRR1553134 | DKOsiRNA_2 | Mbnl 1/2 doppio knockout con Mbnl 3 siRNA (KD) | Rappresentante 2 | Fibroblasti embrionali di topo (MEF) | Estremità singola | 36 pb | |
| GSM1480979 | SRR1553135 | DKONTsiRNA_1 | Mbnl 1/2 doppio knockout con siRNA non mirato (Ctrl) | Rappresentante 1 | Fibroblasti embrionali di topo (MEF) | Estremità singola | 40 pb | |
| GSM1480980 | SRR1553136 | DKONTsiRNA_2 | Mbnl 1/2 doppio knockout con siRNA non mirato (Ctrl) | Rappresentante 2 | Fibroblasti embrionali di topo (MEF) | Estremità singola | 40 pb |
Tabella 1. Riepilogo dei set di dati RNA-Seq e PolyA-seq utilizzati per l'analisi.
Protocollo
1. Installazione di strumenti e pacchetti R utilizzati nell'analisi
- Conda è un gestore di pacchetti popolare e flessibile che consente una comoda installazione dei pacchetti con le loro dipendenze su tutte le piattaforme. Utilizzare 'Anaconda' (conda package manager) per installare 'conda' che può essere utilizzato per installare gli strumenti/pacchetti necessari per l'analisi.
- Scarica 'Anaconda' secondo i requisiti di sistema da https://www.anaconda.com/products/individual#Downloads e installalo seguendo le istruzioni nell'installatore grafico. Installa tutti i pacchetti richiesti usando 'conda' digitando quanto segue nella riga di comando di Linux.
conda install -c daler sratoolkit
conda install -c conda-forge parallel
conda install -c bioconda star bowtie fastqc rmats rmats2sashimiplot samtools fasterq-dump cutadapt bedtools deeptools - Per scaricare tutti i pacchetti R utilizzati nel protocollo, digitare il seguente codice nella console R (avviata sulla riga di comando di Linux digitando 'R') o nella console Rstudio.
bioc_packages<- c("DEXSeq", "Rsubread", "EnhancedVolcano", "edgeR", "limma", "maser","GenomicRanges")
packages<- c("magrittr", "rtracklayer", "tidyverse", "openxlsx", "BiocManager")
#Install if not already installed
installed_packages<-packages%in% rownames(installed.packages())
installed_bioc_packages<-bioc_packages%in% rownames(installed.packages())
if(any(installed_packages==FALSE)) {
install.packages(packages[!installed_packages],dependencies=TRUE)
BiocManager::install(packages[!installed_bioc_packages], dependencies=TRUE)
}
NOTA: in questo protocollo computazionale, i comandi verranno forniti come file R Notebook (file con estensione ". Rmd"), file di codice R (file con estensione ". R"), o script di shell Linux Bash (file con estensione ".sh"). I file R Notebook (Rmd) devono essere aperti in RStudio utilizzando File| Apri File... e singoli blocchi di codice (che possono essere comandi R o comandi della shell Bash) vengono quindi eseguiti in modo interattivo facendo clic sulla freccia verde in alto a destra. I file di codice R possono essere eseguiti aprendo in RStudio o sulla riga di comando di Linux premettendo "Rscript", ad esempio Rscript example.R. Gli script della shell vengono eseguiti sulla riga di comando di Linux anteponendo lo script con il comando "sh", ad esempio .sh example.sh.
2. Analisi di splicing alternativo (AS) mediante RNA-seq
- Download e pre-elaborazione dei dati
NOTA: i frammenti di codice annotati di seguito sono disponibili nel file di codice supplementare "AS_analysis_RNASeq.Rmd", per seguire i singoli passaggi in modo interattivo, e sono forniti anche come script bash da eseguire in batch sulla riga di comando di Linux (sh downloading_data_preprocessing.sh).- Download dei dati grezzi.
- Scarica i dati grezzi da Sequence Read Archive (SRA) utilizzando il comando 'prefetch' dal toolkit SRA (v2.10.8)17. Fornire gli ID di adesione SRA in sequenza nel seguente comando per scaricarli in parallelo utilizzando l'utilità parallela GNU18. Per scaricare i file SRA degli ID di adesione da SRR10261601 a SRR10261606 in parallelo, utilizzare quanto segue nella riga di comando di Linux.
seq 10261601 10261606 | parallel prefetch SRR{} - Estrarre i file fastq dall'archivio utilizzando la funzione 'fastq-dump' dal toolkit SRA. Usa GNU parallelamente e dai i nomi di tutti i file SRA insieme.
parallel -j 3 fastq-dump --gzip --skip-technical --read-filter pass --dumpbase --split-e --clip --origfmt {} ::: - Scaricare il genoma di riferimento e le annotazioni per Mouse (Genome assembly GRCm39) da www.ensembl.org utilizzando quanto segue nella riga di comando di Linux.
wget -nv -O annotation.gtf.gz http://ftp.ensembl.org/pub/release-103/gtf/mus_musculus/Mus_musculus.GRCm39.103.gtf.gz \ && gunzip -f annotation.gtf.gz
wget -nv -O genome.fa.gz http://ftp.ensembl.org/pub/release-103/fasta/mus_musculus/dna/Mus_musculus.GRCm39.dna.primary_assembly.fa.gz \ && gunzip -f genome.fa.gz
GTF=$(readlink -f annotation.gtf)
GENOME=$(readlink -f genome.fa)
- Scarica i dati grezzi da Sequence Read Archive (SRA) utilizzando il comando 'prefetch' dal toolkit SRA (v2.10.8)17. Fornire gli ID di adesione SRA in sequenza nel seguente comando per scaricarli in parallelo utilizzando l'utilità parallela GNU18. Per scaricare i file SRA degli ID di adesione da SRR10261601 a SRR10261606 in parallelo, utilizzare quanto segue nella riga di comando di Linux.
- Pre-elaborazione e mappatura delle letture nell'assemblaggio del genoma
- Controllo qualità. Valuta la qualità delle letture non elaborate con FASTQC (FASTQ Quality Check v0.11.9)19. Creare una cartella di output ed eseguire fastqc con parallelo su più file fasta di input. Questo passaggio genererà un rapporto sulla qualità per ogni campione. Esaminare i report per assicurarsi che la qualità delle letture sia accettabile prima di eseguire ulteriori analisi. (Fare riferimento al manuale dell'utente per comprendere i report in https://www.bioinformatics.babraham.ac.uk/projects/fastqc/)
mkdir fastqc_out
parallel "fastqc {} -o fastqc_out" ::: $RAW_DATA/*.fastq.gz
NOTA: se necessario, eseguire il taglio dell'adattatore con 'cutadapt'20 o 'trimmomatic'21 per rimuovere il sequenziamento negli adattatori laterali, che varia in base alla dimensione del frammento di RNA e alla lunghezza di lettura. In questa analisi abbiamo saltato questo passaggio poiché la frazione di letture interessate era minima. - Leggi l'allineamento. Il passaggio successivo nella pre-elaborazione include la mappatura delle letture al genoma di riferimento. In primo luogo, costruire l'indice per il genoma di riferimento utilizzando la funzione 'genomeGenerate' di STAR22 e quindi allineare le letture grezze al riferimento (in alternativa gli indici precostruiti sono disponibili sul sito web STAR e possono essere utilizzati direttamente per l'allineamento). Eseguire i seguenti comandi dalla riga di comando di Linux.
#Build STAR index
GDIR=STAR_indices
mkdir $GDIR
STAR --runMode genomeGenerate --genomeFastaFiles $GENOME --sjdbGTFfile $GTF --runThreadN 8 --genomeDir $GDIR
ODIR=results/mapping
mkdir -p $ODIR
#Align reads to the genome
for fq1 in $RAW_DATA/*R1.fastq.gz;
do
fq2=$(echo $fq1| sed 's/1.fastq.gz/2.fastq.gz/g');
OUTPUT=$(basename ${fq1}| sed 's/R1.fastq.gz//g');
STAR --genomeDir $GDIR \
--runThreadN 12 \
--readFilesCommand zcat \
--readFilesIn ${fq1}${fq2}\
--outFileNamePrefix $ODIR\/${OUTPUT} \
--outSAMtype BAM SortedByCoordinate \
--outSAMunmapped Within \
--outSAMattributes Standard
Done
NOTA: l'allineatore STAR genererà e ordinerà i file BAM (Binary Alignment Map) per ogni campione dopo l'allineamento di lettura. I file Bam devono essere ordinati prima di procedere a ulteriori passaggi.
- Controllo qualità. Valuta la qualità delle letture non elaborate con FASTQC (FASTQ Quality Check v0.11.9)19. Creare una cartella di output ed eseguire fastqc con parallelo su più file fasta di input. Questo passaggio genererà un rapporto sulla qualità per ogni campione. Esaminare i report per assicurarsi che la qualità delle letture sia accettabile prima di eseguire ulteriori analisi. (Fare riferimento al manuale dell'utente per comprendere i report in https://www.bioinformatics.babraham.ac.uk/projects/fastqc/)
- Download dei dati grezzi.
- Preparazione delle annotazioni Exon.
- Eseguire il file di codice supplementare "prepare_mm_exon_annotation. R" con l'annotazione scaricata in formato GTF (Gene transfer format) per preparare le annotazioni. Per eseguire, digitare quanto segue nella riga di comando di Linux.
Rscript prepare_mm_exon_annotation.R annotation.gtf
NOTA: il file GTF contiene più voci di esone per diverse isoforme. Questo file viene utilizzato per "comprimere" gli ID di trascrizione multipli per ciascun esone. È un passo importante definire i contenitori per il conteggio degli esoni.
- Eseguire il file di codice supplementare "prepare_mm_exon_annotation. R" con l'annotazione scaricata in formato GTF (Gene transfer format) per preparare le annotazioni. Per eseguire, digitare quanto segue nella riga di comando di Linux.
- Letture di conteggio. Il passo successivo è contare il numero di letture mappate su diverse trascrizioni / esoni. Vedere File supplementare: "AS_analysis_RNASeq.Rmd".
- Caricare le librerie richieste:
packages<- c("Rsubread","tidyverse", "magrittr", "EnhancedVolcano", "edgeR","openxlsx")
invisible(lapply(packages, library, character.only=TRUE)) - Caricare il file di annotazione elaborato ottenuto dal passaggio precedente (2.2).
load("mm_exon_anno.RData") - Leggere tutti i file bam ottenuti nel passaggio 2.2.2 come input per 'featureCounts' per contare le letture. Leggere la cartella contenente i file bam elencando prima ogni file dalla directory che termina con .bam. Usa 'featureCounts' dal pacchetto Rsubread che prende i file bam e l'annotazione GTF elaborata (riferimento) come input per generare una matrice di conteggi associati a ciascuna funzionalità con righe che rappresentano esoni (caratteristiche) e colonne che rappresentano campioni.
countData <- dir("bams", pattern=".bam$", full.names=T) %>%
featureCounts(annot.ext=anno,
isGTFAnnotationFile=FALSE,
minMQS=0,useMetaFeatures=FALSE,
allowMultiOverlap=TRUE,
largestOverlap=TRUE,
countMultiMappingReads=FALSE,
primaryOnly=TRUE,
isPairedEnd=TRUE,
nthreads=12) - Successivamente, eseguire il filtraggio non specifico per rimuovere gli esoni poco espressi ("non specifico" indica che le informazioni sulla condizione sperimentale non vengono utilizzate nel filtraggio, per evitare distorsioni di selezione). Trasformare i dati da scala grezza a conteggi per milione (cpm) utilizzando la funzione cpm dal pacchetto 'edgeR'23 e mantenere gli esoni con conteggi superiori a una soglia impostabile (per questo set di dati viene utilizzato un cpm) in almeno tre campioni. Rimuovere anche i geni con un solo esone.
# Non-specific filtering: Remove the exons with low counts
isexpr<- rownames(countData$counts)[rowSums(cpm(countData$counts)>1) >=3]
countData$counts<-countData$counts[rownames(countData$counts) %in%isexpr, ]
anno<-anno%>% filter(GeneID%in% rownames(countData$counts))
# Remove genes with only 1 site and NA in geneIDs
dn<-anno%>%group_by(GeneID)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(GeneID))
anno<-anno%>% filter(GeneID%in%dn$GeneID)
countData$counts<-countData$counts[rownames(countData$counts) %in%anno$GeneID, ]
NOTA: controllare i parametri obbligatori per featureCounts quando si utilizzano dati diversi, ad esempio, per letture a estremità singola, impostare 'isPairedEnd = FALSE'. Fare riferimento alla guida utente di RSubread per scegliere le opzioni per i dati e vedere la sezione Discussione di seguito.
- Caricare le librerie richieste:
- Splicing differenziale e analisi dell'utilizzo dell'esone. Descriviamo due alternative per questo passaggio: DEXSeq e DiffSplice. Entrambi possono essere utilizzati e dare risultati simili. Per coerenza, selezionare DEXSeq se si preferisce un pacchetto DESeq2 per DGE e utilizzare DiffSplice per un'analisi DGE basata su Limma. Vedi scheda supplementare: "AS_analysis_RNASeq.Rmd".
- Utilizzo del pacchetto DEXSeq per l'analisi differenziale degli esoni.
- Caricare la libreria e creare una tabella di esempio per definire il progetto sperimentale.
library(DEXSeq)
sampleTable<-data.frame(row.names= c("Mbnl1KO_Thymus_1", "Mbnl1KO_Thymus_2", "Mbnl1KO_Thymus_3", "WT_Thymus_1", "WT_Thymus_2", "WT_Thymus_3"), condition= rep(c("Mbnl1_KO", "WT"),c(3,3)), libType= rep(c("paired-end")))
Nota : i nomi di riga devono essere coerenti con i nomi di file bam utilizzati da featureCounts per contare le letture. sampleTable è costituito dai dettagli di ogni esempio che include: tipo di libreria e condizione. Questo è necessario per definire i contrasti o il gruppo di test per rilevare l'utilizzo differenziale. - Preparare il file di informazioni sull'esone. Le informazioni sugli esoni sotto forma di oggetti GRanges (genomic ranges) (https://bioconductor.org/packages/release/bioc/html/GenomicRanges.html) sono necessarie come input per creare l'oggetto DEXSeq nel passaggio successivo. Abbina gli ID genetici con i conteggi di lettura per creare l'oggetto exoninfo.
exoninfo<-anno[anno$GeneID%in% rownames(countData$counts),]
exoninfo<-GRanges(seqnames=anno$Chr,
ranges=IRanges(start=anno$Start, end=anno$End, width=anno$Width),strand=Rle(anno$Strand))
mcols(exoninfo)$TranscriptIDs<-anno$TranscriptIDs
mcols(exoninfo)$Ticker<-anno$Ticker
mcols(exoninfo)$ExonID<-anno$ExonID
mcols(exoninfo)$n<-anno$n
mcols(exoninfo)$GeneID<-anno$GeneID
transcripts_l= strsplit(exoninfo$TranscriptIDs, "\\,")
save(countData, sampleTable, exoninfo, transcripts_l, file="AS_countdata.RData") - Creare un oggetto DEXSeq utilizzando la funzione DEXSeqDataSet. L'oggetto DEXSeq raccoglie insieme i conteggi di lettura, le informazioni sulle feature dell'esone e le informazioni di esempio. Utilizzare i conteggi di lettura generati nel passaggio 3 e le informazioni sull'esone ottenute dal passaggio precedente per creare l'oggetto DEXSeq dalla matrice di conteggio. L'argomento sampleData accetta un input di frame di dati che definisce i campioni (e i relativi attributi: tipo di libreria e condizione), 'design' utilizza sampleData per generare una matrice di progettazione per il test differenziale utilizzando la notazione della formula del modello. Si noti che un termine di interazione significativo, condizione:esone, indica che la frazione di letture su un gene che cade su un particolare esone dipende dalla condizione sperimentale, cioè c'è AS. Vedere la documentazione di DEXSeq per una descrizione completa dell'impostazione della formula del modello per progetti sperimentali più complessi. Per le informazioni sulle caratteristiche, sono necessari gli ID dell'esone, il gene corrispondente e le trascrizioni.
dxd<-DEXSeqDataSet(countData$counts,sampleData=sampleTable, design=~sample+exon+condition:exon,featureID=exoninfo$ExonID,groupID=exoninfo$GeneID,featureRanges=exoninfo, transcripts=transcripts_l) - Normalizzazione e stima della dispersione. Successivamente, eseguire la normalizzazione tra i campioni e stimare la varianza dei dati, dovuta sia al rumore del conteggio di Poisson dalla natura discreta dell'RNA-seq che alla variabilità biologica, utilizzando i seguenti comandi.
dxd %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - Test per l'utilizzo differenziale. Dopo la stima della variazione, testare l'uso differenziale dell'esone per ciascun gene e generare i risultati.
dxd%<>%testForDEU%>%estimateExonFoldChanges(fitExpToVar=
"condition")#Estimate fold changes
dxr=DEXSeqResults(dxd) - Visualizza gli eventi di splicing per i geni selezionati usando il seguente comando.
plotDEXSeq(dxr,"Wnk1", displayTranscripts=TRUE, splicing=TRUE,legend
=TRUE,cex.axis=1.2,cex=1.3,lwd=2)
Esaminare il file R Notebook "AS_analysis_RNASeq.Rmd" per generare grafici aggiuntivi per i geni di interesse e per generare grafici vulcanici a soglie diverse.
- Caricare la libreria e creare una tabella di esempio per definire il progetto sperimentale.
- Utilizzo di diffSplice di Limma per identificare lo splicing differenziale. Segui il file R Notebook "AS_analysis_RNASeq.Rmd". Assicurarsi che siano stati seguiti i passaggi 2.1-2.3 per preparare i file di input prima di procedere ulteriormente.
- Caricare le librerie
library(limma)
library(edgeR) - Filtraggio non specifico. Estrarre la matrice dei conteggi di lettura ottenuti in 2.3. Crea un elenco di funzionalità usando la funzione 'DGEList' dal pacchetto edgeR, dove le righe rappresentano i geni e le colonne rappresentano i campioni.
mycounts=countData$counts
#Change the rownames of the countdata to exon Ids instead of genes for unique rownames.
rownames(mycounts) = exoninfo$ExonID
dge<-DGEList(counts=mycounts)
#Filtering
isexpr<- rowSums(cpm(dge)>1) >=3
dge<-dge[isexpr,,keep.lib.sizes=FALSE]
#Extract the exon annotations for only transcripts meeting non-specific filter
exoninfo=anno%>% filter(ExonID%in% rownames(dge$counts))
#Convert the exoninfo into GRanges object
exoninfo1<-GRanges(seqnames=exoninfo$Chr,
ranges=IRanges(start=exoninfo$Start, end=exoninfo$End, width=exoninfo$Width),strand=Rle(exoninfo$Strand))
mcols(exoninfo1)$TranscriptIDs<-exoninfo$TranscriptIDs
mcols(exoninfo1)$Ticker<-exoninfo$Ticker
mcols(exoninfo1)$ExonID<-exoninfo$ExonID
mcols(exoninfo1)$n<-exoninfo$n
mcols(exoninfo1)$GeneID<-exoninfo$GeneID
transcripts_l= strsplit(exoninfo1$TranscriptIDs, "\\,")
NOTA: come passaggio di filtraggio non specifico, i conteggi vengono filtrati in base a cpm < 1 in x su n campioni, dove x è il numero minimo di repliche in qualsiasi condizione. n = 6 e x = 3 per questi dati di esempio. - Normalizzare i conteggi tra i campioni, con la funzione 'calcNormFactors' dal pacchetto 'edgeR' utilizzando i valori Trimmed Mean of M (metodo di normalizzazione TMM)24 Calcolerà i fattori di scala per regolare le dimensioni della libreria.
dge<-calcNormFactors(dge) - Utilizzare sampleTable come generato nel passaggio 2.4.1.1 e creare la matrice di progettazione. La matrice di progettazione caratterizza il design. Vedere la Guida per l'utente di Limma (https://www.bioconductor.org/packages/devel/bioc/vignettes/limma/inst/doc/usersguide.pdf) capitoli 8 e 9 per i dettagli sulle matrici di progettazione per progetti sperimentali più avanzati.
Treat<- factor(sampleTable$condition)
design<- model.matrix(~0+Treat)
colnames(design) <- levels(Treat) - Montare un modello lineare per esone. Eseguire la funzione 'voom' del pacchetto 'limma' per elaborare i dati RNA-seq per stimare la varianza e generare pesi di precisione per correggere il rumore del conteggio di Poisson e trasformare i conteggi a livello di esone in log2-counts per milione (logCPM). Quindi eseguire la modellazione lineare utilizzando la funzione 'lmfit' per adattare i modelli lineari ai dati di espressione per ciascun esone. Calcola le statistiche empiriche di Bayes per il modello adattato utilizzando la funzione 'eBayes' per rilevare l'espressione differenziale dell'esone. Quindi, definire una matrice di contrasto per i confronti sperimentali di interesse. Usa 'contrasts.fit' per ottenere coefficienti ed errori standard per ogni coppia di confronti.
v<-voom(dge,design,plot=FALSE)
fit<-lmFit(v,design)
fit<-eBayes(fit)
colnames(fit)
cont.matrix<-makeContrasts(
Mbnl1_KO_WT=Mbnl1_KO-WT,
levels=design)
fit2<-contrasts.fit(fit,cont.matrix) - Analisi di splicing differenziale. Esegui 'diffSplice' sul modello adattato per testare le differenze nell'uso degli esoni dei geni tra wild-type e knockout ed esplorare i risultati migliori usando la funzione 'topSplice': test="t" fornisce una classificazione degli esoni AS, test="simes" fornisce una classificazione dei geni.
ex<-diffSplice(fit2,geneid=exoninfo$GeneID,exonid=exoninfo$ExonID)
ts<-topSplice(ex,n=Inf,FDR=0.1, test="t", sort.by="logFC")
tg<-topSplice(ex,n=Inf,FDR=0.1, test="simes") - Visualizzazione. Tracciare i risultati con la funzione 'plotSplice', dando il gene di interesse nell'argomento geneide. Salva i risultati principali ordinati per log Piega la modifica in un oggetto e genera un grafico vulcanico per mostrare gli esoni.
plotSplice(ex,geneid="Wnk1", FDR=0.1)
#Volcano plot
EnhancedVolcano(ts,lab=ts$ExonID,selectLab= head((ts$ExonID),2000), xlab= bquote(~Log[2]~'fold change'), x='logFC', y='P.Value', title='Volcano Plot', subtitle='Mbnl1_KO vs WT (Limma_diffSplice)', FCcutoff=2, labSize=4,legendPosition="right", caption= bquote(~Log[2]~"Fold change cutoff, 2; FDR 10%"))
- Caricare le librerie
- Utilizzo di rMATS
- Assicurarsi che l'ultima versione di rMATS v4.1.1 (nota anche come rMATS turbo a causa del tempo di elaborazione ridotto e dei minori requisiti di memoria) sia installata utilizzando conda o github (https://github.com/Xinglab/rmats-turbo/releases/download/v4.1.1/rmats_turbo_v4_1_1.tar.gz) nella directory di lavoro. Seguire il paragrafo 4.3 in "AS_analysis_RNASeq.Rmd".
- Passare alla cartella contenente i file bam ottenuti dopo il mapping e preparare i file di testo, come richiesto da rMATS, per le due condizioni copiando il nome dei file bam (insieme al percorso) separati da ','. I seguenti comandi devono essere eseguiti dalla riga di comando di Linux:
mkdir rMATS_analysis
cd bams/
ls -pd "$PWD"/*| grep "WT"| tr '\n'','> Wt.txt
ls -pd "$PWD"/*| grep "Mb"| tr '\n'','> KO.txt
mv *.txt ../rMATS_analysis - Eseguire rmats.py con i due file di input generati nel passaggio precedente, insieme al file GTF ottenuto nella versione 2.1.1.3. Questo genererà una cartella di output 'rmats_out' contenente file di testo che descrivono le statistiche (valori p e livelli di inclusione) per ogni evento di giunzione separatamente.
python rmats-turbo/rmats.py --b1 KO.txt --b2 Wt.txt --gtf annotation.gtf -t paired --readLength 50 --nthread 8 --od rmats_out/ --tmp rmats_tmp --task pos
Nota : è richiesta anche l'annotazione di riferimento sotto forma di file GTF. Controllare i parametri se i dati sono single-end e modificare l'opzione -t di conseguenza. - Esplorazione dei risultati di rMATS. Utilizzare il pacchetto Bioconductor 'maser'25 per esplorare i risultati di rMATS. Carica i file di testo JCEC (Junction and exon counts) nell'oggetto 'maser' e filtra il risultato in base alla copertura includendo almeno cinque letture medie per evento di giunzione.
library(maser)
mbnl1<-maser("/rmats_out/", c("WT","Mbnl1_KO"), ftype="JCEC")
#Filtering out events by coverage
mbnl1_filt<-filterByCoverage(mbnl1,avg_reads=5) - Visualizzazione dei risultati rMATS. Selezionare gli eventi di splicing significativi al 10% del tasso di falsa scoperta (FDR) e la variazione minima del 10% della percentuale di giunzione in (deltaPSI) utilizzando la funzione 'topEvents' dal pacchetto 'maser'. Quindi, controllare gli eventi genetici per i singoli geni di interesse (gene campione-Wnk1) e tracciare i valori PSI per ogni evento di splicing di quel gene. Generare un grafico a vulcano specificando il tipo di evento.
#Top splicing events at 10% FDR
mbnl1_top<-topEvents(mbnl1_filt,fdr=0.1, deltaPSI=0.1)
mbnl1_top
#Check the gene events for a particular gene
mbnl1_wnk1<-geneEvents(mbnl1_filt,geneS="Wnk1", fdr=0.1, deltaPSI=0.1)
maser::display(mbnl1_wnk1,"SE")
plotGenePSI(mbnl1_wnk1,type="SE", show_replicates
=TRUE)
volcano(mbnl1_filt,fdr=0.1, deltaPSI=0.1,type="SE")
+xlab("deltaPSI")+ylab("Log10 Adj. Pvalue")+ggtitle("Volcano Plot of exon skipping events") - Genera grafici Sashimi per il risultato degli eventi di giunzione ottenuti con rMATS sotto forma di file di testo utilizzando il pacchetto 'rmats2shahimiplot'. Eseguire lo script python dalla riga di comando di Linux.
python ./src/rmats2sashimiplot/rmats2sashimiplot.py --b1 ../bams/WT_Thymus_1.bam,../bams/WT_Thymus_2.bam,../bams/WT_Thymus_3.bam --b2 ../bams/Mbnl1KO_Thymus_1.bam,../bams/Mbnl1KO_Thymus_2.bam,../bams/Mbnl1KO_Thymus_3.bam -t SE -e ../rMATS_analysis/rmats_out/SE.MATS.JC.txt --l1 WT --l2 Mbnl1_KO --exon_s 1 --intron_s 5 -o ../rMATS_analysis/rmats2shasmi_output
NOTA: questo processo può richiedere molto tempo in quanto genererà il grafico Sashimi per tutti i risultati nel file degli eventi. Scegli i risultati migliori (nomi di geni ed esoni) come visualizzati dalla funzione topEvents da 'maser' e visualizza il grafico del Sashimi corrispondente.
- Utilizzo del pacchetto DEXSeq per l'analisi differenziale degli esoni.
3. Analisi di poliadenilazione alternativa (APA) mediante sequenziamento finale 3'
- Download e pre-elaborazione dei dati
Nota : fare riferimento al file supplementare R Notebook "APA_analysis_3PSeq_notebook. Rmd" per i comandi completi per il download dei dati e le fasi di pre-elaborazione, oppure eseguire il file bash supplementare "APA_data_downloading_preprocessing.sh" sulla riga di comando di Linux.- Scaricare i dati dall'SRA con gli ID di adesione (da 1553129 a 1553136).
- Adattatori trim e complemento inverso per ottenere la sequenza del filo di senso.
NOTA: questo passaggio è specifico per il test PolyA-seq utilizzato. - La mappa legge l'assemblaggio del genoma del mouse utilizzando l'allineatore per papillon26.
- Preparazione delle annotazioni dei siti pA.
NOTA: l'elaborazione del file di annotazione del sito pA viene eseguita in primo luogo utilizzando il file supplementare R Notebook "APA_analysis_3PSeq_notebook. Rmd" (2.1 - 2.6), quindi utilizzando il file bash "APA_annotation_preparation.sh".- Scarica l'annotazione dei siti pA dal database PolyASite 2.06.
- Selezionare le annotazioni del sito pA per conservare i siti pA della regione non tradotta (UTR) 3', annotati come esone terminale (TE) o 1000 nt a valle di un esone terminale annotato (DS) per l'analisi a valle.
- Ottieni picchi del sito pA. Ancoraggio in ogni sito di scissione pA e visualizzare la copertura media di lettura utilizzando bedtools e deeptools27,28. I risultati hanno mostrato che i picchi delle letture mappate erano principalmente dispersi entro ~ 60 bp a monte dei siti di scissione (figura 5 e figura supplementare 5). Pertanto, le coordinate dei siti pA sono state estese dal file di annotazione a 60 bp a monte dei loro siti di scissione. A seconda del protocollo di sequenziamento finale 3' specifico, questo passaggio dovrà essere ottimizzato per saggi diversi da PolyA-seq.
- Letture di conteggio
- Preparare il file di annotazione dei siti pA.
anno<- read.table(file= "flanking60added.pA_annotation.bed",
stringsAsFactors=FALSE, check.names=FALSE, header=FALSE, sep="")
colnames(anno) <- c("chrom", "chromStart", "chromEnd", "name", "score", "strand", "rep", "annotation", "gene_name", "gene_id")
anno<- dplyr::select(anno,name,chrom, chromStart,chromEnd, strand,gene_id,gene_name,rep)
colnames(anno) <- c("GeneID", "Chr", "Start", "End", "Strand", "Ensembl", "Symbol", "repID") - Applica 'featureCounts' per acquisire conteggi non elaborati. Salvare la tabella di conteggio come file RData "APA_countData.Rdata" per l'analisi APA utilizzando strumenti diversi.
countData<- dir("bamfiles", pattern="sorted.bam$", full.names=TRUE) %>%
# Read all bam files as input for featureCounts
featureCounts(annot.ext=anno, isGTFAnnotationFile= FALSE,minMQS=0,useMetaFeatures= TRUE,allowMultiOverlap=TRUE, largestOverlap= TRUE,strandSpecific=1, countMultiMappingReads =TRUE,primaryOnly= TRUE,isPairedEnd= FALSE,nthreads=12)%T>%
save(file="APA_countData.Rdata")
NOTA: tenere presente di modificare uno qualsiasi dei parametri elencati nella funzione 'featureCounts'. Modificare il parametro 'strandSpecific' per assicurarsi che sia coerente con la direzione di sequenziamento del saggio di sequenziamento finale 3' utilizzato (empiricamente, visualizzare i dati in un browser del genoma sui geni sui filamenti più e meno chiarirà questo). - Applicare un filtro non specifico di countData. Il filtraggio può migliorare significativamente la robustezza statistica nei test differenziali di utilizzo del sito pA. In primo luogo, abbiamo rimosso quei geni con un solo sito pA, su cui l'utilizzo differenziale del sito pA non può essere definito. In secondo luogo, applichiamo un filtraggio non specifico basato sulla copertura: i conteggi sono filtrati da cpm meno di 1 su x su n campioni, dove x è il numero minimo di repliche in qualsiasi condizione. N = 8 e x = 2 per questi dati di esempio.
load(file= "APA_countData.Rdata")# Skip this step if already loaded
# Non-specific filtering: Remove the pA sites not differentially expressed in the samples
countData<-countData$counts%>%as.data.frame%>% .[rowSums(edgeR::cpm(.)>1) >=2, ]
anno%<>% .[.$GeneID%in% rownames(countData), ]
# Remove genes with only 1 site and NA in geneIDs
dnsites<-anno%>%group_by(Symbol)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(Symbol))
anno<-anno%>% filter(Symbol%in%dnsites$Symbol)
countData<-countData[rownames(countData) %in%anno$GeneID, ]
- Preparare il file di annotazione dei siti pA.
- Analisi differenziale dell'utilizzo del sito di poliadenilazione mediante pipeline DEXSeq e diffSplice.
- Utilizzo del pacchetto DEXSeq
NOTA: Poiché non è possibile definire una matrice di contrasto per la pipeline DEXSeq, l'analisi APA differenziale di ciascuna due condizioni sperimentali deve essere eseguita separatamente. L'analisi APA differenziale della condizione WT e della condizione DKO viene eseguita come esempio per spiegare la procedura. Fare riferimento al file supplementare "APA_analysis_3PSeq_notebook. RMD ·" per il flusso di lavoro passo-passo di questa sezione e l'analisi APA differenziale di altri contrasti.- Caricare la libreria e creare una tabella di esempio per definire il progetto sperimentale.
c("DEXSeq", "GenomicRanges") %>% lapply(library, character.only=TRUE) %>%invisible
sampleTable1<- data.frame(row.names= c("WT_1","WT_2","DKO_1","DKO_2"),
condition= c(rep("WT", 2), rep("DKO", 2)),
libType= rep("single-end", 4)) - Preparare il file di informazioni sui siti pA utilizzando il pacchetto Bioconductor GRanges.
# Prepare the GRanges object for DEXSeqDataSet object construction
PASinfo <- GRanges(seqnames = anno$Chr,
ranges = IRanges(start = anno$Start, end = anno$End),strand = Rle(anno$Strand))
mcols(PASinfo)$PASID<-anno$repID
mcols(PASinfo)$GeneEns<-anno$Ensembl
mcols(PASinfo)$GeneID<-anno$Symbol
# Prepare the new feature IDs, replace the strand information with letters to match the current pA site clusterID
new.featureID <- anno$Strand %>% as.character %>% replace(. %in% "+", "F") %>% replace(. %in% "-", "R") %>% paste0(as.character(anno$repID), .) - Utilizzare i conteggi di lettura generati nel passaggio 3.3 e le informazioni sul sito pA ottenute dal passaggio precedente per creare l'oggetto DEXSeq.
# Select the read counts of the condition WT and DKO
countData1<- dplyr::select(countData, SRR1553129.sorted.bam, SRR1553130.sorted.bam, SRR1553131.sorted.bam, SRR1553132.sorted.bam)
# Rename the columns of countData using sample names in sampleTable
colnames(countData1) <- rownames(sampleTable1)
dxd1<-DEXSeqDataSet(countData=countData1,
sampleData=sampleTable1,
design=~sample+exon+condition:exon,
featureID=new.featureID,
groupID=anno$Symbol,
featureRanges=PASinfo) - Definire la coppia di contrasto definendo i livelli delle condizioni nell'oggetto DEXSeq.
dxd1$condition<- factor(dxd1$condition, levels= c("WT", "DKO"))
# The contrast pair will be "DKO - WT" - Normalizzazione e stima della dispersione. Analogamente ai dati RNA-seq, per 3' i dati di sequenziamento finale eseguire la normalizzazione tra i campioni (mediana dei rapporti per colonna per ciascun campione) utilizzando la funzione 'estimateSizeFactors' e stimare la variazione dei dati utilizzando la funzione 'estimateDispersions', quindi visualizzare il risultato della stima della dispersione utilizzando la funzione 'plotDispEsts'.
dxd1 %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - Test differenziale di utilizzo del sito pA per ciascun gene utilizzando la funzione 'testForDEU', quindi stimare il cambiamento di piega dell'utilizzo del sito pA utilizzando la funzione 'estimateExonFoldChanges'. Controllare i risultati utilizzando la funzione 'DEXSeqResults' e impostare 'FDR < 10%' come criterio per siti pA significativamente differenziali.
dxd1 %<>% testForDEU %>% estimateExonFoldChanges(fitExpToVar = "condition")
dxr1 <- DEXSeqResults(dxd1)
dxr1
mcols(dxr1)$description
table(dxr1$padj<0.1) # Check the number of differential pA sites (FDR < 0.1)
table(tapply(dxr1$padj<0.1, dxr1$groupID, any)) # Check the number of gene overlapped with differential pA site - Visualizzazione dei risultati dell'utilizzo differenziale del sito pA utilizzando grafici APA differenziali generati dalla funzione 'plotDEXSeq' e diagramma vulcano dalla funzione 'EnhancedVolcano'.
# Select the top 100 significant differential pA sites ranked by FDR
topdiff.PAS<- dxr1%>%as.data.frame%>%rownames_to_column%>%arrange(padj)%$%groupID[1:100]
# Apply plotDEXSeq for the visualization of differential polyA usage
plotDEXSeq(dxr1,"S100a7a", legend=TRUE, expression=FALSE,splicing=TRUE, cex.axis=1.2, cex=1.3,lwd=2)
# Apply perGeneQValue to check the top genes with differential polyA site usage
dxr1%<>% .[!is.na(.$padj), ]
dgene<- data.frame(perGeneQValue= perGeneQValue (dxr1)) %>%rownames_to_column("groupID")
dePAS_sig1<-dxr1%>% data.frame() %>%
dplyr::select(-matches("dispersion|stat|countData|genomicData"))%>%
inner_join(dgene)%>%arrange(perGeneQValue)%>%distinct()%>%
filter(padj<0.1)
# Apply EnhancedVolcano package to visualise differential polyA site usage
"EnhancedVolcano"%>% lapply(library, character.only=TRUE) %>%invisible
EnhancedVolcano(dePAS_sig1, lab=dePAS_sig1$groupID, x='log2fold_DKO_WT',
y='pvalue',title='Volcano Plot',subtitle='DKO vs WT',
FCcutoff=1,labSize=4, legendPosition="right",
caption= bquote(~Log[2]~"Fold change cutoff, 1; FDR 10%"))
- Caricare la libreria e creare una tabella di esempio per definire il progetto sperimentale.
- Utilizzo del pacchetto diffSplice. Fare riferimento al file supplementare R Notebook "APA_analysis_3PSeq_notebook. Rmd" per il flusso di lavoro dettagliato di questa sezione.
- Definire i contrasti di interesse per l'analisi dell'utilizzo differenziale di pA.
Nota : questo passaggio deve essere eseguito dopo la costruzione e l'elaborazione dell'oggetto DGEList, incluso nel file R Notebook "APA_analysis_3PSeq_notebook. Rmd".contrast.matrix<-makeContrasts(DKO_vs_WT=DKO-
WT,Ctrl_vs_DKO=Ctrl-DKO,
KD_vs_Ctrl=KD-Ctrl,KD_vs_DKO=KD-DKO,levels=design)
fit2<-fit%>%contrasts.fit(contrast.matrix)%>%eBayes
summary(decideTests(fit2))
ex<-diffSplice(fit2,geneid=anno$Symbol,exonid=new.featureID)
topSplice(ex) #Check the top significant results with topSplice - Visualizza il risultato dei contrasti di interesse (qui "DKO - WT") utilizzando grafici APA differenziali con la funzione 'plotSplice' e grafici vulcanologici con la funzione 'EnhancedVolcano'. Fare riferimento al file R Notebook "APA_analysis_3PSeq_notebook. Rmd" 4.2.7 - 4.2.9 per la visualizzazione di altre coppie di contrasto.
sig1<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="t", sort.by="logFC")
sig1.genes<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="simes")
plotSplice(ex, coef=1,geneid="S100a7a", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Tpm1", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Smc6", FDR = 0.1)
EnhancedVolcano(sig1, lab=sig1$GeneID,xlab= bquote(~Log[2]~'fold change'),
x='logFC', y='P.Value', title='Volcano Plot', subtitle='DKO vs WT',
FCcutoff=1, labSize=6, legendPosition="right")
- Definire i contrasti di interesse per l'analisi dell'utilizzo differenziale di pA.
- Utilizzo del pacchetto DEXSeq
Risultati
Dopo aver eseguito il flusso di lavoro dettagliato di cui sopra, i risultati dell'analisi AS e APA e i risultati rappresentativi sono sotto forma di tabelle e grafici di dati, generati come segue.
COME:
Il principale risultato dell'analisi AS (Tabella supplementare 1 per diffSplice; La tabella 2 per DEXSeq) è un elenco di esoni che mostrano un uso differenziale tra le condizioni e un elenco di geni che mostrano una significativa attività ...
Discussione
In questo studio, abbiamo valutato approcci basati su esoni e basati su eventi per rilevare AS e APA in massa RNA-Seq e 3' dati di sequenziamento finale. Gli approcci AS basati sugli esoni producono sia un elenco di esoni differenzialmente espressi sia una classificazione a livello genico ordinata in base alla significatività statistica dell'attività complessiva di splicing differenziale a livello genetico (Tabelle 1-2, 4-5). Per il pacchetto diffSplice, l'uso differenziale è determinato adattando mod...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo studio è stato supportato da una Future Fellowship dell'Australian Research Council (ARC) (FT16010043) e da ANU Futures Scheme.
Materiali
| Name | Company | Catalog Number | Comments |
| Not relevent for computational study |
Riferimenti
- Katz, Y., Wang, E. T., Airoldi, E. M., Burge, C. B. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods. 7 (12), 1009-1015 (2010).
- Wang, Y., et al. Mechanism of alternative splicing and its regulation. Biomedical Reports. 3 (2), 152-158 (2015).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in Bioinformatics. 21 (6), 2052-2065 (2020).
- Movassat, M., et al. Coupling between alternative polyadenylation and alternative splicing is limited to terminal introns. RNA Biology. 13 (7), 646-655 (2016).
- Tian, B., Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature Reviews Molecular Cell Biology. 18 (1), 18-30 (2017).
- Herrmann, C. J., et al. PolyASite 2.0: a consolidated atlas of polyadenylation sites from 3' end sequencing. Nucleic Acids Research. 48 (1), 174-179 (2020).
- Liu, R., Loraine, A. E., Dickerson, J. A. Comparisons of computational methods for differential alternative splicing detection using RNA-seq in plant systems. BMC Bioinformatics. 15 (1), 364 (2014).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (1), 13 (2016).
- Anders, S., Reyes, A., Huber, W. Detecting differential usage of exons from RNA-seq data. Genome Research. 22 (10), 2008-2017 (2012).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2014).
- Shen, S., et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data. Proceedings of the National Academy of Sciences. 111 (51), 5593-5601 (2014).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in bioinformatics. 21 (6), 2052-2065 (2020).
- Kanitz, A., et al. Comparative assessment of methods for the computational inference of transcript isoform abundance from RNA-seq data. Genome biology. 16 (1), 1-26 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Sznajder, L. J., et al. Loss of MBNL1 induces RNA misprocessing in the thymus and peripheral blood. Nature Communications. 11, 1-11 (2020).
- Batra, R., et al. Loss of MBNL leads to disruption of developmentally regulated alternative polyadenylation in RNA-mediated disease. Molecular Cell. 56 (2), 311-322 (2014).
- Leinonen, R., Sugawara, H., Shumway, M., et al. The sequence read archive. Nucleic acids research. 39, 19-21 (2010).
- Tange, O. . GNU parallel-the command-line power tool. 36, 42-47 (2011).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), 25 (2010).
- Veiga, D. F. T. maser: Mapping Alternative Splicing Events to pRoteins. R package version 1.4.0. , (2019).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (13), 25 (2009).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ramírez, F., Dündar, F., Diehl, S., Grüning, B. A., Manke, T. deepTools: a flexible platform for exploring deep-sequencing data. Nucleic acids research. 42 (1), 187-191 (2014).
- Merino, G. A., Conesa, A., Fernández, E. A. A benchmarking of workflows for detecting differential splicing and differential expression at isoform level in human RNA-seq studies. Briefings in bioinformatics. 20 (2), 471-481 (2019).
- Chhangawala, S., Rudy, G., Mason, C. E., Rosenfeld, J. A. The impact of read length on quantification of differentially expressed genes and splice junction detection. Genome biology. 16 (1), 1-10 (2015).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biol. 17, 13 (2016).
- Trapnell, C., et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 7 (3), 562-578 (2012).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 12, 323 (2011).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat Biotechnol. 34 (5), 525-527 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon