Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
RNA-seq Verilerinde Alternatif Ekleme ve Poliadenilasyonun Tanımlanması
Bu Makalede
Özet
Alternatif ekleme (AS) ve alternatif poliadenilasyon (APA), transkript izoformlarının ve ürünlerinin çeşitliliğini genişletir. Burada, deneysel koşullar arasında değişen AS ve APA'yı tespit etmek ve görselleştirmek için toplu RNA-seq ve 3' uç dizileme testlerini analiz etmek için biyoinformatik protokolleri açıklıyoruz.
Özet
Deneysel / biyolojik koşullar boyunca diferansiyel gen ekspresyonunu (DGE) ölçmek için RNA-Seq'in tipik analizinin yanı sıra, RNA-seq verileri ekzon seviyesindeki diğer karmaşık düzenleyici mekanizmaları keşfetmek için de kullanılabilir. Alternatif ekleme ve poliadenilasyon, transkripsiyon sonrası seviyede gen ekspresyonunu düzenlemek için farklı izoformlar üreterek bir genin fonksiyonel çeşitliliğinde çok önemli bir rol oynar ve analizleri tüm gen seviyesine sınırlamak bu önemli düzenleyici katmanı kaçırabilir. Burada, Bioconductor ve DEXSeq, Limma paketinden diffSplice ve rMATS dahil olmak üzere diğer paketleri ve fonksiyonları kullanarak, koşullar arasında diferansiyel ekzon ve poliadenilasyon sahası kullanımının tanımlanması ve görselleştirilmesi için ayrıntılı adım adım analizler gösteriyoruz.
Giriş
RNA-seq, yıllar boyunca tipik olarak diferansiyel gen ekspresyonunu ve gen keşfini tahmin etmek için yaygın olarak kullanılmıştır1. Ek olarak, farklı izoformları ifade eden gen nedeniyle değişen ekzon seviyesi kullanımını tahmin etmek için de kullanılabilir, böylece transkripsiyon sonrası seviyede gen düzenlemesinin daha iyi anlaşılmasına katkıda bulunur. Ökaryotik genlerin çoğunluğu, mRNA ekspresyonunun çeşitliliğini arttırmak için alternatif ekleme (AS) ile farklı izoformlar üretir. AS olayları farklı kalıplara ayrılabilir: bir ("kaset") ekzonun yan tarafındaki intronlarla birlikte transkriptten tamamen çıkarıldığı tam ekzonların (SE) atlanması; ekzonun her iki ucunda iki veya daha fazla ekleme bölgesi bulunduğunda alternatif (donör) 5' ekleme yeri seçimi (A5SS) ve alternatif 3' (alıcı) ekleme yeri seçimi (A3SS); Bir intron olgun mRNA transkriptinde tutulduğunda intronların (RI) tutulması ve mevcut iki ekzondan sadece birininbir seferde tutulabildiği ekzon kullanımının (MXE) karşılıklı dışlanması 2,3. Alternatif poliadenilasyon (APA), tek bir transkript4'ten çoklu mRNA izoformları üretmek için alternatif poli (A) bölgeleri kullanarak gen ekspresyonunun düzenlenmesinde de önemli bir rol oynar. Çoğu poliadenilasyon bölgesi (pAs), 3' çevrilmemiş bölgede (3' UTR'ler) bulunur ve çeşitli 3' UTR uzunluklarına sahip mRNA izoformları üretir. 3' UTR, düzenleyici unsurları tanımak için merkezi merkez olduğundan, farklı 3' UTR uzunlukları mRNA lokalizasyonunu, kararlılığını ve translasyonunu etkileyebilir5. Protokol6'nın ayrıntılarında farklılık gösteren APA'yı tespit etmek için optimize edilmiş bir 3' uç sıralama tahlilleri sınıfı vardır. Burada açıklanan boru hattı PolyA-seq için tasarlanmıştır, ancak açıklandığı gibi diğer protokoller için uyarlanabilir.
Bu çalışmada, ekzon bazlı (DEXSeq9, diffSplice10) ve olay tabanlı (Multivariate Analysis of Transcript Splicing (rMATS)11) olmak üzere iki geniş kategoriye ayrılabilen diferansiyel ekzon analiz yöntemleri 7,8 (Şekil 1) boru hattını sunuyoruz. Ekzon tabanlı yöntemler, bireysel ekzonların koşulları arasındaki kıvrım değişimini, farklı şekilde ifade edilen ekzon kullanımını çağırmak için genel gen kıvrım değişiminin bir ölçüsüyle karşılaştırır ve bundan AS aktivitesinin gen düzeyinde bir ölçüsünü hesaplar. Olay tabanlı yöntemler, ekzon atlama veya intronların tutulması gibi belirli ekleme olaylarını algılamak ve sınıflandırmak için ekzon intronunu kapsayan bağlantı okumalarını kullanır ve çıktı3'teki bu AS türlerini ayırt eder. Bu nedenle, bu yöntemler AS12,13'ün tam bir analizi için tamamlayıcı görüşler sağlar. Diferansiyel ekleme analizi için en yaygın kullanılan paketler arasında yer aldıkları için çalışma için DEXSeq (DESeq214 DGE paketine dayanarak) ve diffSplice (Limma10 DGE paketine dayanarak) seçtik. rMATS, olay tabanlı analiz için popüler bir yöntem olarak seçildi. Bir başka popüler olay tabanlı yöntem MISO (İzoform Karışımı)1'dir. APA için ekzon tabanlı yaklaşımı uyarlıyoruz.
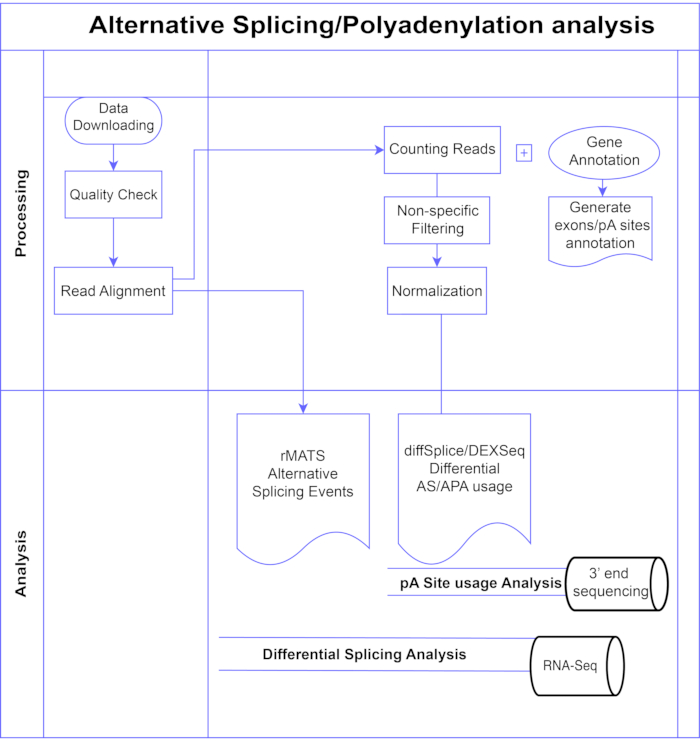
Şekil 1. Analiz işlem hattı. Analizde kullanılan adımların akış şeması. Adımlar şunları içerir: verileri elde etmek, kalite kontrolleri yapmak ve okuma hizalaması, ardından bilinen ekzonlar, intronlar ve pA siteleri için ek açıklamalar kullanarak okumaları saymak, düşük sayıları kaldırmak için filtreleme ve normalleştirme. PolyA-seq verileri diffSplice/DEXSeq yöntemleri kullanılarak alternatif pA bölgeleri için, bulk RNA-Seq diffSplice/DEXseq yöntemleri ile ekzon düzeyinde alternatif ekleme için ve AS olayları rMATS ile analiz edilmiştir. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
Bu araştırmada kullanılan RNA-seq verileri, Gen İfade Omnibus'undan (GEO) (GSE138691)15 elde edilmiştir. Bu çalışmadan elde edilen fare RNA-seq verilerini iki koşul grubuyla kullandık: vahşi tip (WT) ve her biri üç kopya ile Kas körü benzeri tip 1 nakavt (Mbnl1 KO). Diferansiyel poliadenilasyon alanı kullanım analizini göstermek için, fare embriyo fibroblastları (MEF'ler) PoliA-seq verilerini elde ettik (GEO Katılımı GSE60487)16. Verilerin dört koşul grubu vardır: Wild-type (WT), Kas körü benzeri tip1/tip 2 çift nakavt (Mbnl1/2 DKO), Mbnl3 knockdown (KD) ile Mbnl 1/2 DKO ve Mbnl3 kontrollü Mbnl1/2 DKO (Ctrl). Her koşul grubu iki çoğaltmadan oluşur.
| GEO Katılımı | SRA Çalıştırma numarası | Örnek adı | Koşul | Çoğaltmak | Doku | Sıralama | Okuma uzunluğu | |
| RNA-Seq | GSM4116218 | SRR10261601 | Mbnl1KO_Thymus_1 | Mbnl1 nakavt | Temsilci 1 | Timus | Eşleştirilmiş uç | 100 bp |
| GSM4116219 | SRR10261602 | Mbnl1KO_Thymus_2 | Mbnl1 nakavt | Temsilci 2 | Timus | Eşleştirilmiş uç | 100 bp | |
| GSM4116220 göster | SRR10261603 | Mbnl1KO_Thymus_3 | Mbnl1 nakavt | Temsilci 3 | Timus | Eşleştirilmiş uç | 100 bp | |
| GSM4116221 | SRR10261604 | WT_Thymus_1 | Vahşi tip | Temsilci 1 | Timus | Eşleştirilmiş uç | 100 bp | |
| GSM4116222 | SRR10261605 | WT_Thymus_2 | Vahşi tip | Temsilci 2 | Timus | Eşleştirilmiş uç | 100 bp | |
| GSM4116223 | SRR10261606 | WT_Thymus_3 | Vahşi tip | Temsilci 3 | Timus | Eşleştirilmiş uç | 100 bp | |
| 3P-Seks | GSM1480973 | SRR1553129 | WT_1 | Vahşi tip (WT) | Temsilci 1 | Fare embriyonik Fibroblastları (MEF'ler) | Tek uçlu | 40 bp |
| GSM1480974 | SRR1553130 | WT_2 | Vahşi tip (WT) | Temsilci 2 | Fare embriyonik Fibroblastları (MEF'ler) | Tek uçlu | 40 bp | |
| GSM1480975 | SRR1553131 | DKO_1 | Mbnl 1/2 çift nakavt (DKO) | Temsilci 1 | Fare embriyonik Fibroblastları (MEF'ler) | Tek uçlu | 40 bp | |
| GSM1480976 | SRR1553132 | DKO_2 | Mbnl 1/2 çift nakavt (DKO) | Temsilci 2 | Fare embriyonik Fibroblastları (MEF'ler) | Tek uçlu | 40 bp | |
| GSM1480977 göster | SRR1553133 | DKOsiRNA_1 | Mbnl 3 siRNA (KD) ile Mbnl 1/2 çift nakavt | Temsilci 1 | Fare embriyonik Fibroblastları (MEF'ler) | Tek uçlu | 40 bp | |
| GSM1480978 | SRR1553134 | DKOsiRNA_2 | Mbnl 3 siRNA (KD) ile Mbnl 1/2 çift nakavt | Temsilci 2 | Fare embriyonik Fibroblastları (MEF'ler) | Tek uçlu | 36 bg | |
| GSM1480979 | SRR1553135 | DKONTsiRNA_1 | Mbnl 1/2 çift nakavt, hedeflemeyen siRNA (Ctrl) ile | Temsilci 1 | Fare embriyonik Fibroblastları (MEF'ler) | Tek uçlu | 40 bp | |
| GSM1480980 | SRR1553136 | DKONTsiRNA_2 | Mbnl 1/2 çift nakavt, hedeflemeyen siRNA (Ctrl) ile | Temsilci 2 | Fare embriyonik Fibroblastları (MEF'ler) | Tek uçlu | 40 bp |
Tablo 1. Analiz için kullanılan RNA-Seq ve PolyA-seq veri setlerinin özeti.
Protokol
1. Analizde kullanılan aletlerin ve R paketlerinin kurulumu
- Conda, paketlerin tüm platformlardaki bağımlılıklarıyla birlikte kolayca kurulmasını sağlayan popüler ve esnek bir paket yöneticisidir. Analiz için gerekli araçları/paketleri yüklemek için kullanılabilecek 'conda'yı yüklemek için 'Anaconda' (conda paket yöneticisi) kullanın.
- https://www.anaconda.com/products/individual#Downloads'dan sistem gereksinimlerine göre 'Anaconda'yı indirin ve grafik yükleyicideki istemleri izleyerek yükleyin. Linux komut satırına aşağıdakileri yazarak 'conda' kullanarak gerekli tüm paketleri yükleyin.
conda install -c daler sratoolkit
conda install -c conda-forge parallel
conda install -c bioconda star bowtie fastqc rmats rmats2sashimiplot samtools fasterq-dump cutadapt bedtools deeptools - Protokolde kullanılan tüm R paketlerini indirmek için, R konsoluna (Linux komut satırında 'R' yazarak başlatılır) veya Rstudio konsoluna aşağıdaki kodu yazın.
bioc_packages<- c("DEXSeq", "Rsubread", "EnhancedVolcano", "edgeR", "limma", "maser","GenomicRanges")
packages<- c("magrittr", "rtracklayer", "tidyverse", "openxlsx", "BiocManager")
#Install if not already installed
installed_packages<-packages%in% rownames(installed.packages())
installed_bioc_packages<-bioc_packages%in% rownames(installed.packages())
if(any(installed_packages==FALSE)) {
install.packages(packages[!installed_packages],dependencies=TRUE)
BiocManager::install(packages[!installed_bioc_packages], dependencies=TRUE)
}
NOT: Bu hesaplama protokolünde, komutlar R Notebook dosyaları (uzantılı dosyalar) olarak verilecektir. Rmd"), R kod dosyaları (" uzantılı dosyalar. R") veya Linux Bash kabuk betikleri (".sh" uzantılı dosyalar). R Notebook (Rmd) dosyaları RStudio'da Dosya | Dosya...'yı açın ve tek tek kod parçaları (R komutları veya Bash kabuk komutları olabilir) sağ üstteki yeşil oka tıklayarak etkileşimli olarak çalıştırın. R kod dosyaları RStudio'da açılarak veya Linux komut satırında "Rscript" ile önaçı yapılarak çalıştırılabilir, örneğin Rscript example.R. Shell betikleri, Linux komut satırında komut dosyasının önüne "sh" komutu ile başlayarak çalıştırılır.sh example.sh.
2. RNA-seq kullanarak alternatif ekleme (AS) analizi
- Veri indirme ve ön işleme
NOT: Aşağıda ek açıklama yapılan kod parçacıkları ek kod dosyasında mevcuttur "AS_analysis_RNASeq.Rmd", bireysel adımları etkileşimli olarak takip etmek için ve ayrıca Linux komut satırında toplu olarak çalıştırılacak bir bash betiği olarak sağlanır (sh downloading_data_preprocessing.sh).- Ham verileri indirme.
- SRA araç seti (v2.10.8)17'deki 'prefetch' komutunu kullanarak ham verileri Sıra Okuma Arşivi'nden (SRA) indirin. GNU paralel yardımcı programı18'i kullanarak paralel olarak indirmek için SRA Katılım Kimliklerini aşağıdaki komutta sırayla verin. Katılım kimliklerinin SRA dosyalarını SRR10261601'den SRR10261606'ya paralel olarak indirmek için Linux komut satırında aşağıdakileri kullanın.
seq 10261601 10261606 | parallel prefetch SRR{} - SRA araç setinden 'fastq-dump' işlevini kullanarak fastq dosyalarını arşivden ayıklayın. GNU paralelini kullanın ve tüm SRA dosyalarının adlarını birlikte verin.
parallel -j 3 fastq-dump --gzip --skip-technical --read-filter pass --dumpbase --split-e --clip --origfmt {} ::: - Linux komut satırında aşağıdakileri kullanarak www.ensembl.org'dan Fare (Genom derlemesi GRCm39) için referans genomu ve ek açıklamaları indirin.
wget -nv -O annotation.gtf.gz http://ftp.ensembl.org/pub/release-103/gtf/mus_musculus/Mus_musculus.GRCm39.103.gtf.gz \ && gunzip -f annotation.gtf.gz
wget -nv -O genome.fa.gz http://ftp.ensembl.org/pub/release-103/fasta/mus_musculus/dna/Mus_musculus.GRCm39.dna.primary_assembly.fa.gz \ && gunzip -f genome.fa.gz
GTF=$(readlink -f annotation.gtf)
GENOME=$(readlink -f genome.fa)
- SRA araç seti (v2.10.8)17'deki 'prefetch' komutunu kullanarak ham verileri Sıra Okuma Arşivi'nden (SRA) indirin. GNU paralel yardımcı programı18'i kullanarak paralel olarak indirmek için SRA Katılım Kimliklerini aşağıdaki komutta sırayla verin. Katılım kimliklerinin SRA dosyalarını SRR10261601'den SRR10261606'ya paralel olarak indirmek için Linux komut satırında aşağıdakileri kullanın.
- Ön işleme ve haritalama genom derlemesine okunur
- Kalite Kontrol. FASTQC (FASTQ Kalite Kontrolü v0.11.9)19 ile ham okumaların kalitesini değerlendirin. Bir çıktı klasörü oluşturun ve fastqc'yi birden fazla giriş fasta dosyasında paralel olarak çalıştırın. Bu adım, her örnek için bir kalite raporu oluşturur. Daha fazla analiz yapmadan önce okumaların kalitesinin kabul edilebilir olduğundan emin olmak için raporları inceleyin. ( https://www.bioinformatics.babraham.ac.uk/projects/fastqc/'daki raporları anlamak için kullanım kılavuzuna bakın)
mkdir fastqc_out
parallel "fastqc {} -o fastqc_out" ::: $RAW_DATA/*.fastq.gz
NOT: Gerekirse, RNA parça boyutuna ve okuma uzunluğuna bağlı olarak değişen yan adaptörlerdeki sıralamayı kaldırmak için 'cutadapt'20 veya 'trimmomatic'21 ile adaptör kırpma işlemi gerçekleştirin. Bu analizde, etkilenen okumaların oranı minimum olduğu için bu adımı atladık. - Okuma hizalaması. Ön işlemedeki bir sonraki adım, okumaların referans genomla eşlenmesini içerir. İlk olarak, STAR22'nin 'genomeGenerate' işlevini kullanarak referans genom için dizin oluşturun ve ardından ham okumaları referansla hizalayın (alternatif olarak önceden oluşturulmuş dizinler STAR web sitesinden edinilebilir ve doğrudan hizalama için kullanılabilir). Linux komut satırında aşağıdaki komutları çalıştırın.
#Build STAR index
GDIR=STAR_indices
mkdir $GDIR
STAR --runMode genomeGenerate --genomeFastaFiles $GENOME --sjdbGTFfile $GTF --runThreadN 8 --genomeDir $GDIR
ODIR=results/mapping
mkdir -p $ODIR
#Align reads to the genome
for fq1 in $RAW_DATA/*R1.fastq.gz;
do
fq2=$(echo $fq1| sed 's/1.fastq.gz/2.fastq.gz/g');
OUTPUT=$(basename ${fq1}| sed 's/R1.fastq.gz//g');
STAR --genomeDir $GDIR \
--runThreadN 12 \
--readFilesCommand zcat \
--readFilesIn ${fq1}${fq2}\
--outFileNamePrefix $ODIR\/${OUTPUT} \
--outSAMtype BAM SortedByCoordinate \
--outSAMunmapped Within \
--outSAMattributes Standard
Done
NOT: STAR hizalayıcı, okuma hizalamasından sonra her örnek için BAM (İkili Hizalama Haritası) dosyaları oluşturur ve sıralar. Sonraki adımlara geçmeden önce Bam dosyaları sıralanmalıdır.
- Kalite Kontrol. FASTQC (FASTQ Kalite Kontrolü v0.11.9)19 ile ham okumaların kalitesini değerlendirin. Bir çıktı klasörü oluşturun ve fastqc'yi birden fazla giriş fasta dosyasında paralel olarak çalıştırın. Bu adım, her örnek için bir kalite raporu oluşturur. Daha fazla analiz yapmadan önce okumaların kalitesinin kabul edilebilir olduğundan emin olmak için raporları inceleyin. ( https://www.bioinformatics.babraham.ac.uk/projects/fastqc/'daki raporları anlamak için kullanım kılavuzuna bakın)
- Ham verileri indirme.
- Exon ek açıklamalarını hazırlama.
- Ek kod dosyasını çalıştırın "prepare_mm_exon_annotation. R" ile indirilen ek açıklamayı GTF (Gen transfer formatı) formatında hazırlayarak ek açıklamaları hazırlar. Çalıştırmak için, Linux komut satırına aşağıdakileri yazın.
Rscript prepare_mm_exon_annotation.R annotation.gtf
NOT: GTF dosyası farklı izoformlar için birden çok ekzon girdisi içerir. Bu dosya, her ekzon için birden fazla transkript kimliğini "daraltmak" için kullanılır. Ekzon sayım kutularını tanımlamak önemli bir adımdır.
- Ek kod dosyasını çalıştırın "prepare_mm_exon_annotation. R" ile indirilen ek açıklamayı GTF (Gen transfer formatı) formatında hazırlayarak ek açıklamaları hazırlar. Çalıştırmak için, Linux komut satırına aşağıdakileri yazın.
- Okuma Sayma. Bir sonraki adım, farklı transkriptlere / ekzonlara eşlenen okuma sayısını saymaktır. Bkz. Ek dosya: "AS_analysis_RNASeq.Rmd".
- Gerekli kitaplıkları yükleyin:
packages<- c("Rsubread","tidyverse", "magrittr", "EnhancedVolcano", "edgeR","openxlsx")
invisible(lapply(packages, library, character.only=TRUE)) - Önceki adımdan (2.2) elde edilen işlenmiş ek açıklama dosyasını yükleyin.
load("mm_exon_anno.RData") - Adım 2.2.2'de elde edilen tüm bam dosyalarını, okunanları saymak için 'featureCounts' için giriş olarak okuyun. Önce .bam ile biten dizindeki her dosyayı listeleyerek bam dosyalarını içeren klasörü okuyun. Bam dosyalarını alan ve GTF ek açıklamasını (referans) giriş olarak işleyen Rsubread paketinden 'featureCounts'u kullanarak, ekzonları (özellikleri) temsil eden satırlar ve örnekleri temsil eden sütunlarla her bir özellikle ilişkili bir sayım matrisi oluşturun.
countData <- dir("bams", pattern=".bam$", full.names=T) %>%
featureCounts(annot.ext=anno,
isGTFAnnotationFile=FALSE,
minMQS=0,useMetaFeatures=FALSE,
allowMultiOverlap=TRUE,
largestOverlap=TRUE,
countMultiMappingReads=FALSE,
primaryOnly=TRUE,
isPairedEnd=TRUE,
nthreads=12) - Ardından, düşük ifade edilen ekzonları kaldırmak için spesifik olmayan filtreleme yapın ("spesifik olmayan", seçim yanlılıklarını önlemek için deneysel koşul bilgilerinin filtrelemede kullanılmadığını gösterir). 'edgeR' paketi23'teki cpm işlevini kullanarak verileri ham ölçekten milyon başına sayıma (cpm) dönüştürün ve en az üç örnekte ayarlanabilir bir eşikten daha büyük sayımlara sahip ekzonları (bu veri kümesi için bir cpm kullanılır) tutun. Ayrıca sadece bir ekzon ile genleri çıkarın.
# Non-specific filtering: Remove the exons with low counts
isexpr<- rownames(countData$counts)[rowSums(cpm(countData$counts)>1) >=3]
countData$counts<-countData$counts[rownames(countData$counts) %in%isexpr, ]
anno<-anno%>% filter(GeneID%in% rownames(countData$counts))
# Remove genes with only 1 site and NA in geneIDs
dn<-anno%>%group_by(GeneID)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(GeneID))
anno<-anno%>% filter(GeneID%in%dn$GeneID)
countData$counts<-countData$counts[rownames(countData$counts) %in%anno$GeneID, ]
NOT: Farklı veriler kullanırken featureCounts için gerekli parametreleri kontrol edin, örneğin tek uçlu okumalar için 'isPairedEnd = FALSE' değerini ayarlayın. Verilerinize ilişkin seçenekleri belirlemek için RSubread kullanım kılavuzuna bakın ve aşağıdaki Tartışma bölümüne bakın.
- Gerekli kitaplıkları yükleyin:
- Diferansiyel ekleme ve ekzon kullanım analizi. Bu adım için iki alternatif açıklıyoruz: DEXSeq ve DiffSplice. Her ikisi de kullanılabilir ve benzer sonuçlar verebilir. Tutarlılık için, DGE için bir DESeq2 paketini tercih ediyorsanız DEXSeq'u seçin ve Limma tabanlı DGE analizi için DiffSplice kullanın. Ek dosyaya bakınız: "AS_analysis_RNASeq.Rmd".
- Diferansiyel ekzon analizi için DEXSeq paketini kullanma.
- Kitaplığı yükleyin ve deneysel tasarımı tanımlamak için örnek bir tablo oluşturun.
library(DEXSeq)
sampleTable<-data.frame(row.names= c("Mbnl1KO_Thymus_1", "Mbnl1KO_Thymus_2", "Mbnl1KO_Thymus_3", "WT_Thymus_1", "WT_Thymus_2", "WT_Thymus_3"), condition= rep(c("Mbnl1_KO", "WT"),c(3,3)), libType= rep(c("paired-end")))
NOT: Satır adları, okumaları saymak için featureCounts tarafından kullanılan bam dosya adlarıyla tutarlı olmalıdır. sampleTable, aşağıdakileri içeren her bir örneğin ayrıntılarından oluşur: kitaplık türü ve durumu. Bu, diferansiyel kullanımı algılamak için kontrastları veya test grubunu tanımlamak için gereklidir. - Ekzon bilgi dosyasını hazırlayın. Bir sonraki adımda DEXSeq nesnesini oluşturmak için girdi olarak GRanges (genomik aralıklar) nesneleri (https://bioconductor.org/packages/release/bioc/html/GenomicRanges.html) biçimindeki ekzon bilgisi gereklidir. Exoninfo nesnesi oluşturmak için gen kimliklerini okuma sayılarıyla eşleştirin.
exoninfo<-anno[anno$GeneID%in% rownames(countData$counts),]
exoninfo<-GRanges(seqnames=anno$Chr,
ranges=IRanges(start=anno$Start, end=anno$End, width=anno$Width),strand=Rle(anno$Strand))
mcols(exoninfo)$TranscriptIDs<-anno$TranscriptIDs
mcols(exoninfo)$Ticker<-anno$Ticker
mcols(exoninfo)$ExonID<-anno$ExonID
mcols(exoninfo)$n<-anno$n
mcols(exoninfo)$GeneID<-anno$GeneID
transcripts_l= strsplit(exoninfo$TranscriptIDs, "\\,")
save(countData, sampleTable, exoninfo, transcripts_l, file="AS_countdata.RData") - DEXSeqDataSet işlevini kullanarak DEXSeq nesnesi oluşturun. DEXSeq nesnesi okuma sayılarını, ekzon özellik bilgilerini ve örnek bilgileri birlikte toplar. Sayı matrisinden DEXSeq nesnesi oluşturmak için adım 3'te oluşturulan okuma sayılarını ve önceki adımdan elde edilen ekzon bilgilerini kullanın. sampleData bağımsız değişkeni, örnekleri (ve özniteliklerini: kitaplık türü ve koşulu) tanımlayan bir veri çerçevesi girişi alır, 'design', model formülü gösterimini kullanarak diferansiyel test için bir tasarım matrisi oluşturmak üzere sampleData'yı kullanır. Önemli bir etkileşim terimi olan condition:exon'un, belirli bir ekzona düşen bir gen üzerindeki okumaların fraksiyonunun deneysel duruma bağlı olduğunu, yani AS olduğunu gösterdiğini unutmayın. Daha karmaşık deneysel tasarımlar için model formülünü ayarlamanın tam açıklaması için DEXSeq belgelerine bakın. Özellik bilgisi için, ekzon kimlikleri, karşılık gelen gen ve transkriptler gereklidir.
dxd<-DEXSeqDataSet(countData$counts,sampleData=sampleTable, design=~sample+exon+condition:exon,featureID=exoninfo$ExonID,groupID=exoninfo$GeneID,featureRanges=exoninfo, transcripts=transcripts_l) - Normalizasyon ve dağılım tahmini. Daha sonra, örnekler arasında normalleştirme yapın ve aşağıdaki komutları kullanarak hem RNA-seq'in ayrık doğasından hem de biyolojik değişkenlikten kaynaklanan Poisson sayım gürültüsü nedeniyle verilerin varyansını tahmin edin.
dxd %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - Diferansiyel kullanım testi. Varyasyon tahmininden sonra, her gen için diferansiyel ekzon kullanımını test edin ve sonuçları üretin.
dxd%<>%testForDEU%>%estimateExonFoldChanges(fitExpToVar=
"condition")#Estimate fold changes
dxr=DEXSeqResults(dxd) - Aşağıdaki komutu kullanarak seçili genler için ekleme olaylarını görselleştirin.
plotDEXSeq(dxr,"Wnk1", displayTranscripts=TRUE, splicing=TRUE,legend
=TRUE,cex.axis=1.2,cex=1.3,lwd=2)
İlgilenilen genler için ek grafikler oluşturmak ve farklı eşiklerde volkan grafikleri oluşturmak için R Notebook dosyasını "AS_analysis_RNASeq.Rmd" dosyasını inceleyin.
- Kitaplığı yükleyin ve deneysel tasarımı tanımlamak için örnek bir tablo oluşturun.
- Diferansiyel eklemeyi tanımlamak için Limma'dan diffSplice kullanma. R Notebook dosyasını takip edin "AS_analysis_RNASeq.Rmd". Daha fazla ilerlemeden önce giriş dosyalarını hazırlamak için 2.1-2.3 arasındaki adımların izlendiğinden emin olun.
- Kitaplıkları yükleme
library(limma)
library(edgeR) - Spesifik olmayan filtreleme. 2.3'te elde edilen okuma sayılarının matrisini ayıklayın. Satırların genleri ve sütunların örnekleri temsil ettiği edgeR paketinden 'DGEList' işlevini kullanarak özelliklerin bir listesini oluşturun.
mycounts=countData$counts
#Change the rownames of the countdata to exon Ids instead of genes for unique rownames.
rownames(mycounts) = exoninfo$ExonID
dge<-DGEList(counts=mycounts)
#Filtering
isexpr<- rowSums(cpm(dge)>1) >=3
dge<-dge[isexpr,,keep.lib.sizes=FALSE]
#Extract the exon annotations for only transcripts meeting non-specific filter
exoninfo=anno%>% filter(ExonID%in% rownames(dge$counts))
#Convert the exoninfo into GRanges object
exoninfo1<-GRanges(seqnames=exoninfo$Chr,
ranges=IRanges(start=exoninfo$Start, end=exoninfo$End, width=exoninfo$Width),strand=Rle(exoninfo$Strand))
mcols(exoninfo1)$TranscriptIDs<-exoninfo$TranscriptIDs
mcols(exoninfo1)$Ticker<-exoninfo$Ticker
mcols(exoninfo1)$ExonID<-exoninfo$ExonID
mcols(exoninfo1)$n<-exoninfo$n
mcols(exoninfo1)$GeneID<-exoninfo$GeneID
transcripts_l= strsplit(exoninfo1$TranscriptIDs, "\\,")
NOT: Belirli olmayan bir filtreleme adımı olarak, sayımlar cpm tarafından filtrelenir < n örnekten x'te 1'e kadar x, burada x, herhangi bir koşulda minimum çoğaltma sayısıdır. Bu örnek veriler için n = 6 ve x = 3. - Kesilmiş Ortalama M değerlerini kullanarak 'edgeR' paketinden 'calcNormFactors' işleviyle örnekler arasındaki sayımları normalleştirin (TMM normalleştirme yöntemi)24 Kitaplık boyutlarını ayarlamak için ölçeklendirme faktörlerini hesaplar.
dge<-calcNormFactors(dge) - sampleTable'ı adım 2.4.1.1'de oluşturulan şekilde kullanın ve tasarım matrisini oluşturun. Tasarım matrisi tasarımı karakterize eder. Daha gelişmiş deneysel tasarımlar için tasarım matrisleri hakkında ayrıntılar için Limma Kullanım Kılavuzu (https://www.bioconductor.org/packages/devel/bioc/vignettes/limma/inst/doc/usersguide.pdf) bölüm 8 ve 9'a bakın.
Treat<- factor(sampleTable$condition)
design<- model.matrix(~0+Treat)
colnames(design) <- levels(Treat) - Ekzon başına doğrusal bir model takın. Varyansı tahmin etmek ve Poisson sayım gürültüsünü düzeltmek için hassas ağırlıklar oluşturmak üzere RNA-seq verilerini işlemek ve ekzon düzeyindeki sayımları milyonda log2 sayımlarına (logCPM) dönüştürmek için 'limma' paketinin 'voom' işlevini çalıştırın. Ardından, doğrusal modelleri her ekzonun ifade verilerine sığdırmak için 'lmfit' işlevini kullanarak doğrusal modellemeyi çalıştırın. Diferansiyel ekzon ifadesini algılamak için 'eBayes' işlevini kullanarak takılı model için ampirik Bayes istatistiklerini hesaplayın. Ardından, ilgilenilen deneysel karşılaştırmalar için bir kontrast matrisi tanımlayın. Her karşılaştırma çifti için katsayılar ve standart hatalar elde etmek üzere 'contrasts.fit' kullanın.
v<-voom(dge,design,plot=FALSE)
fit<-lmFit(v,design)
fit<-eBayes(fit)
colnames(fit)
cont.matrix<-makeContrasts(
Mbnl1_KO_WT=Mbnl1_KO-WT,
levels=design)
fit2<-contrasts.fit(fit,cont.matrix) - Diferansiyel ekleme analizi. Vahşi tip ve nakavt arasındaki genlerin ekzon kullanımındaki farklılıkları test etmek için uygun modelde 'diffSplice' komutunu çalıştırın ve 'topSplice' işlevini kullanarak en üst sıradaki sonuçları keşfedin: test = "t" AS ekzonlarının bir sıralamasını verir, test = "simes" genlerin bir sıralamasını verir.
ex<-diffSplice(fit2,geneid=exoninfo$GeneID,exonid=exoninfo$ExonID)
ts<-topSplice(ex,n=Inf,FDR=0.1, test="t", sort.by="logFC")
tg<-topSplice(ex,n=Inf,FDR=0.1, test="simes") - Görsel -leştirme. Sonuçları 'plotSplice' işleviyle çizin ve geneid argümanına ilgi duyan geni verin. Günlüğe göre sıralanmış en iyi sonuçları kaydedin Değişikliği bir nesneye katlayın ve ekzonları sergilemek için bir volkan grafiği oluşturun.
plotSplice(ex,geneid="Wnk1", FDR=0.1)
#Volcano plot
EnhancedVolcano(ts,lab=ts$ExonID,selectLab= head((ts$ExonID),2000), xlab= bquote(~Log[2]~'fold change'), x='logFC', y='P.Value', title='Volcano Plot', subtitle='Mbnl1_KO vs WT (Limma_diffSplice)', FCcutoff=2, labSize=4,legendPosition="right", caption= bquote(~Log[2]~"Fold change cutoff, 2; FDR 10%"))
- Kitaplıkları yükleme
- rMATS'ı kullanma
- rMATS v4.1.1'in en son sürümünün (daha kısa işlem süresi ve daha az bellek gereksinimi nedeniyle rMATS turbo olarak da bilinir) çalışma dizininde conda veya github (https://github.com/Xinglab/rmats-turbo/releases/download/v4.1.1/rmats_turbo_v4_1_1.tar.gz) kullanılarak yüklendiğinden emin olun. "AS_analysis_RNASeq.Rmd" bölümündeki 4.3 bölümünü izleyin.
- Eşlemeden sonra elde edilen bam dosyalarını içeren klasöre gidin ve rMATS gerektirdiği şekilde, bam dosyalarının adını (yol ile birlikte) ',' ile ayırarak kopyalayarak iki koşul için metin dosyaları hazırlayın. Linux komut satırında aşağıdaki komutlar çalıştırılmalıdır:
mkdir rMATS_analysis
cd bams/
ls -pd "$PWD"/*| grep "WT"| tr '\n'','> Wt.txt
ls -pd "$PWD"/*| grep "Mb"| tr '\n'','> KO.txt
mv *.txt ../rMATS_analysis - rmats.py, önceki adımda oluşturulan iki giriş dosyasıyla ve 2.1.1.3'te elde edilen GTF dosyasıyla çalıştırın. Bu, her ekleme olayı için ayrı ayrı istatistikleri (p değerleri ve Dahil etme düzeyleri) açıklayan metin dosyalarını içeren bir çıktı klasörü 'rmats_out' oluşturur.
python rmats-turbo/rmats.py --b1 KO.txt --b2 Wt.txt --gtf annotation.gtf -t paired --readLength 50 --nthread 8 --od rmats_out/ --tmp rmats_tmp --task pos
NOT: GTF dosyası biçimindeki referans ek açıklaması da gereklidir. Verilerin tek uçlu olup olmadığını kontrol edin ve -t seçeneğini buna göre değiştirin. - rMATS sonuçlarını keşfetme. rMATS sonuçlarını keşfetmek için Bioconductor paketi 'maser'25'i kullanın. Junction and exon counts (JCEC) metin dosyalarını 'maser' nesnesine yükleyin ve ekleme olayı başına en az beş ortalama okuma ekleyerek sonucu kapsama göre filtreleyin.
library(maser)
mbnl1<-maser("/rmats_out/", c("WT","Mbnl1_KO"), ftype="JCEC")
#Filtering out events by coverage
mbnl1_filt<-filterByCoverage(mbnl1,avg_reads=5) - rMATS sonuçlarını görselleştirme. 'maser' paketinden 'topEvents' işlevini kullanarak False Discovery Rate (FDR) %10 ve Yüzde Eklenmiş Dilim'de (deltaPSI) en az %10 değişiklik ile önemli ekleme olaylarını seçin. Daha sonra, ilgilenilen bireysel genler için gen olaylarını kontrol edin (örnek gen-Wnk1) ve bu genin her bir ekleme olayı için PSI değerlerini çizin. Olay türünü belirterek bir volkan grafiği oluşturun.
#Top splicing events at 10% FDR
mbnl1_top<-topEvents(mbnl1_filt,fdr=0.1, deltaPSI=0.1)
mbnl1_top
#Check the gene events for a particular gene
mbnl1_wnk1<-geneEvents(mbnl1_filt,geneS="Wnk1", fdr=0.1, deltaPSI=0.1)
maser::display(mbnl1_wnk1,"SE")
plotGenePSI(mbnl1_wnk1,type="SE", show_replicates
=TRUE)
volcano(mbnl1_filt,fdr=0.1, deltaPSI=0.1,type="SE")
+xlab("deltaPSI")+ylab("Log10 Adj. Pvalue")+ggtitle("Volcano Plot of exon skipping events") - 'rmats2shahimiplot' paketini kullanarak metin dosyaları biçiminde rMATS ile elde edilen ekleme olayları sonucu için Sashimi grafikleri oluşturun. Python betiğini Linux komut satırında çalıştırın.
python ./src/rmats2sashimiplot/rmats2sashimiplot.py --b1 ../bams/WT_Thymus_1.bam,../bams/WT_Thymus_2.bam,../bams/WT_Thymus_3.bam --b2 ../bams/Mbnl1KO_Thymus_1.bam,../bams/Mbnl1KO_Thymus_2.bam,../bams/Mbnl1KO_Thymus_3.bam -t SE -e ../rMATS_analysis/rmats_out/SE.MATS.JC.txt --l1 WT --l2 Mbnl1_KO --exon_s 1 --intron_s 5 -o ../rMATS_analysis/rmats2shasmi_output
NOT: Bu işlem, olaylar dosyasındaki tüm sonuçlar için Sashimi grafiğini oluşturacağından zaman alıcı olabilir. 'maser'den topEvents fonksiyonu tarafından görüntülenen en iyi sonuçları (gen adları ve ekzonlar) seçin ve ilgili Sashimi grafiğini görselleştirin.
- Diferansiyel ekzon analizi için DEXSeq paketini kullanma.
3. 3' uç dizileme kullanarak alternatif poliadenilasyon (APA) analizi
- Veri indirme ve ön işleme
NOT: Ek R Notebook dosyasına bakın "APA_analysis_3PSeq_notebook. Veri indirme ve ön işleme adımları için komutların tamamı için "Rmd" veya Linux komut satırında ek bash dosyası "APA_data_downloading_preprocessing.sh" çalıştırın.- SRA'dan Accession Ids (1553129 to 1553136) ile veri indirin.
- Algılama ipliği dizisini elde etmek için adaptörleri ve ters tamamlayıcıyı kırpın.
NOT: Bu adım, kullanılan PolyA-seq testine özgüdür. - Harita, papyon hizalayıcı26 kullanılarak fare genom montajına okunur.
- pA siteleri ek açıklamalarını hazırlama.
NOT: pA site ek açıklama dosyasının işlenmesi öncelikle ek R Notebook dosyası "APA_analysis_3PSeq_notebook kullanılarak gerçekleştirilir. Rmd" (2,1 - 2,6) ve ardından bash dosyası "APA_annotation_preparation.sh" kullanarak.- PolyASite 2.0 veritabanı6'dan pA siteleri ek açıklamasını indirin.
- Aşağı akış analizi için Terminal Exon (TE) veya açıklamalı terminal ekzonunun (DS) 1000 nt aşağı akışı olarak ek açıklama eklenmiş 3'-çevrilmemiş bölge (UTR) pA sitelerini korumak için pA sitesi ek açıklamalarını seçin.
- pA sitesi zirvelerini elde edin. Her pA bölünme alanına demirleyin ve yatak aletleri ve derin aletler kullanarak ortalama okuma kapsamını görselleştirin27,28. Sonuçlar, haritalanan okumaların zirvelerinin esas olarak bölünme bölgelerinin ~ 60 bp yukarı yönünde dağıldığını göstermiştir (şekil 5 ve ek şekil 5). Bu nedenle, pA sitelerinin koordinatları ek açıklama dosyasından bölünme alanlarının 60 bp yukarı akışına genişletildi. Kullanılan belirli 3' uç sıralama protokolüne bağlı olarak, bu adımın PolyA-seq dışındaki tahliller için optimize edilmesi gerekecektir.
- Okumaları sayma
- pA siteleri ek açıklama dosyasını hazırlayın.
anno<- read.table(file= "flanking60added.pA_annotation.bed",
stringsAsFactors=FALSE, check.names=FALSE, header=FALSE, sep="")
colnames(anno) <- c("chrom", "chromStart", "chromEnd", "name", "score", "strand", "rep", "annotation", "gene_name", "gene_id")
anno<- dplyr::select(anno,name,chrom, chromStart,chromEnd, strand,gene_id,gene_name,rep)
colnames(anno) <- c("GeneID", "Chr", "Start", "End", "Strand", "Ensembl", "Symbol", "repID") - Ham sayımlar elde etmek için 'featureCounts' komutunu uygulayın. Farklı araçlar kullanarak APA analizi için sayı tablosunu RData dosyası "APA_countData.Rdata" olarak kaydedin.
countData<- dir("bamfiles", pattern="sorted.bam$", full.names=TRUE) %>%
# Read all bam files as input for featureCounts
featureCounts(annot.ext=anno, isGTFAnnotationFile= FALSE,minMQS=0,useMetaFeatures= TRUE,allowMultiOverlap=TRUE, largestOverlap= TRUE,strandSpecific=1, countMultiMappingReads =TRUE,primaryOnly= TRUE,isPairedEnd= FALSE,nthreads=12)%T>%
save(file="APA_countData.Rdata")
NOT: 'featureCounts' işlevinde listelenen parametrelerden herhangi birini değiştirmek için bilinçli olun. Kullanılan 3' uç dizileme testinin dizileme yönüyle tutarlı olduğundan emin olmak için 'strandSpecific' parametresini değiştirin (ampirik olarak, bir genom tarayıcısındaki verileri artı ve eksi iplikçiklerdeki genler üzerinde görselleştirmek bunu açıklığa kavuşturacaktır). - countData'nın spesifik olmayan filtresini uygulayın. Filtreleme, diferansiyel pA site kullanım testlerinde istatistiksel sağlamlığı önemli ölçüde artırabilir. İlk olarak, bu genleri, farklı pA sitesi kullanımının tanımlanamadığı tek bir pA bölgesi ile çıkardık. İkinci olarak, kapsama alanına göre spesifik olmayan filtreleme uygularız: sayımlar, n örneklemden x'te 1'den daha az olan cpm ile filtrelenir, burada x, herhangi bir koşulda minimum çoğaltma sayısıdır. Bu örnek veriler için N = 8 ve x = 2.
load(file= "APA_countData.Rdata")# Skip this step if already loaded
# Non-specific filtering: Remove the pA sites not differentially expressed in the samples
countData<-countData$counts%>%as.data.frame%>% .[rowSums(edgeR::cpm(.)>1) >=2, ]
anno%<>% .[.$GeneID%in% rownames(countData), ]
# Remove genes with only 1 site and NA in geneIDs
dnsites<-anno%>%group_by(Symbol)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(Symbol))
anno<-anno%>% filter(Symbol%in%dnsites$Symbol)
countData<-countData[rownames(countData) %in%anno$GeneID, ]
- pA siteleri ek açıklama dosyasını hazırlayın.
- DEXSeq ve diffSplice boru hatlarını kullanarak diferansiyel poliadenilasyon sahası kullanım analizi.
- DEXSeq paketini kullanma
NOT: DEXSeq boru hattı için bir kontrast matrisi tanımlanamadığından, her iki deneysel koşulun diferansiyel APA analizi ayrı ayrı gerçekleştirilmelidir. WT ve koşul DKO'nun diferansiyel APA analizi, prosedürü açıklamak için örnek olarak gerçekleştirilir. Ek dosyaya bakın "APA_analysis_3PSeq_notebook. Rmd" Bu bölümün adım adım iş akışı ve diğer kontrastların diferansiyel APA analizi için.- Kitaplığı yükleyin ve deneysel tasarımı tanımlamak için örnek bir tablo oluşturun.
c("DEXSeq", "GenomicRanges") %>% lapply(library, character.only=TRUE) %>%invisible
sampleTable1<- data.frame(row.names= c("WT_1","WT_2","DKO_1","DKO_2"),
condition= c(rep("WT", 2), rep("DKO", 2)),
libType= rep("single-end", 4)) - Bioconductor package GRanges kullanarak pA siteleri bilgi dosyasını hazırlayın.
# Prepare the GRanges object for DEXSeqDataSet object construction
PASinfo <- GRanges(seqnames = anno$Chr,
ranges = IRanges(start = anno$Start, end = anno$End),strand = Rle(anno$Strand))
mcols(PASinfo)$PASID<-anno$repID
mcols(PASinfo)$GeneEns<-anno$Ensembl
mcols(PASinfo)$GeneID<-anno$Symbol
# Prepare the new feature IDs, replace the strand information with letters to match the current pA site clusterID
new.featureID <- anno$Strand %>% as.character %>% replace(. %in% "+", "F") %>% replace(. %in% "-", "R") %>% paste0(as.character(anno$repID), .) - DEXSeq nesnesini oluşturmak için adım 3.3'te oluşturulan okuma sayılarını ve önceki adımdan elde edilen pA site bilgilerini kullanın.
# Select the read counts of the condition WT and DKO
countData1<- dplyr::select(countData, SRR1553129.sorted.bam, SRR1553130.sorted.bam, SRR1553131.sorted.bam, SRR1553132.sorted.bam)
# Rename the columns of countData using sample names in sampleTable
colnames(countData1) <- rownames(sampleTable1)
dxd1<-DEXSeqDataSet(countData=countData1,
sampleData=sampleTable1,
design=~sample+exon+condition:exon,
featureID=new.featureID,
groupID=anno$Symbol,
featureRanges=PASinfo) - DEXSeq nesnesindeki koşul düzeylerini tanımlayarak karşıtlık çiftini tanımlayın.
dxd1$condition<- factor(dxd1$condition, levels= c("WT", "DKO"))
# The contrast pair will be "DKO - WT" - Normalizasyon ve dağılım tahmini. RNA-seq verilerine benzer şekilde, 3' uç dizileme verileri için 'estimateSizeFactors' işlevini kullanarak numuneler arasında normalleştirme (her numune için sütun bazında oran medyanı) gerçekleştirir ve 'estimateDispersions' işlevini kullanarak verilerin varyasyonunu tahmin eder, ardından 'plotDispEsts' işlevini kullanarak dağılım tahmin sonucunu görselleştirir.
dxd1 %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - 'testForDEU' işlevini kullanarak her gen için diferansiyel pA site kullanım testi, ardından 'estimateExonFoldChanges' işlevini kullanarak pA site kullanımının katlama değişimini tahmin edin. 'DEXSeqResults' fonksiyonunu kullanarak sonuçları kontrol edin ve önemli ölçüde farklı pA siteleri için ölçüt olarak 'FDR <% 10' olarak ayarlayın.
dxd1 %<>% testForDEU %>% estimateExonFoldChanges(fitExpToVar = "condition")
dxr1 <- DEXSeqResults(dxd1)
dxr1
mcols(dxr1)$description
table(dxr1$padj<0.1) # Check the number of differential pA sites (FDR < 0.1)
table(tapply(dxr1$padj<0.1, dxr1$groupID, any)) # Check the number of gene overlapped with differential pA site - Diferansiyel pA sahası kullanımının görselleştirilmesi, 'plotDEXSeq' fonksiyonu tarafından oluşturulan diferansiyel APA grafikleri ve 'EnhancedVolcano' fonksiyonu tarafından volkan grafiği kullanılarak elde edilir.
# Select the top 100 significant differential pA sites ranked by FDR
topdiff.PAS<- dxr1%>%as.data.frame%>%rownames_to_column%>%arrange(padj)%$%groupID[1:100]
# Apply plotDEXSeq for the visualization of differential polyA usage
plotDEXSeq(dxr1,"S100a7a", legend=TRUE, expression=FALSE,splicing=TRUE, cex.axis=1.2, cex=1.3,lwd=2)
# Apply perGeneQValue to check the top genes with differential polyA site usage
dxr1%<>% .[!is.na(.$padj), ]
dgene<- data.frame(perGeneQValue= perGeneQValue (dxr1)) %>%rownames_to_column("groupID")
dePAS_sig1<-dxr1%>% data.frame() %>%
dplyr::select(-matches("dispersion|stat|countData|genomicData"))%>%
inner_join(dgene)%>%arrange(perGeneQValue)%>%distinct()%>%
filter(padj<0.1)
# Apply EnhancedVolcano package to visualise differential polyA site usage
"EnhancedVolcano"%>% lapply(library, character.only=TRUE) %>%invisible
EnhancedVolcano(dePAS_sig1, lab=dePAS_sig1$groupID, x='log2fold_DKO_WT',
y='pvalue',title='Volcano Plot',subtitle='DKO vs WT',
FCcutoff=1,labSize=4, legendPosition="right",
caption= bquote(~Log[2]~"Fold change cutoff, 1; FDR 10%"))
- Kitaplığı yükleyin ve deneysel tasarımı tanımlamak için örnek bir tablo oluşturun.
- diffSplice paketini kullanma. Ek R Notebook dosyasına bakın "APA_analysis_3PSeq_notebook. Rmd", bu bölümün adım adım iş akışı için.
- Diferansiyel pA kullanım analizi için ilgilenilen karşıtlıkları tanımlayın.
NOT: Bu adım, R Notebook dosyası "APA_analysis_3PSeq_notebook bulunan DGEList nesnesinin oluşturulmasından ve işlenmesinden sonra gerçekleştirilmelidir. Rmd".contrast.matrix<-makeContrasts(DKO_vs_WT=DKO-
WT,Ctrl_vs_DKO=Ctrl-DKO,
KD_vs_Ctrl=KD-Ctrl,KD_vs_DKO=KD-DKO,levels=design)
fit2<-fit%>%contrasts.fit(contrast.matrix)%>%eBayes
summary(decideTests(fit2))
ex<-diffSplice(fit2,geneid=anno$Symbol,exonid=new.featureID)
topSplice(ex) #Check the top significant results with topSplice - İlgilenilen kontrastların sonucunu (burada "DKO - WT") 'plotSplice' işleviyle diferansiyel APA grafiklerini ve 'EnhancedVolcano' işlevine sahip volkan grafiklerini kullanarak görselleştirin. R Notebook dosyasına bakın "APA_analysis_3PSeq_notebook. Diğer kontrast çiftlerinin görselleştirilmesi için Rmd" 4.2.7 - 4.2.9.
sig1<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="t", sort.by="logFC")
sig1.genes<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="simes")
plotSplice(ex, coef=1,geneid="S100a7a", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Tpm1", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Smc6", FDR = 0.1)
EnhancedVolcano(sig1, lab=sig1$GeneID,xlab= bquote(~Log[2]~'fold change'),
x='logFC', y='P.Value', title='Volcano Plot', subtitle='DKO vs WT',
FCcutoff=1, labSize=6, legendPosition="right")
- Diferansiyel pA kullanım analizi için ilgilenilen karşıtlıkları tanımlayın.
- DEXSeq paketini kullanma
Sonuçlar
Yukarıdaki adım adım iş akışını çalıştırdıktan sonra, AS ve APA analiz çıktıları ve temsili sonuçlar, aşağıdaki gibi oluşturulan tablolar ve veri grafikleri şeklindedir.
GİBİ:
AS analizinin ana çıktısı (diffSplice için Ek Tablo 1; DEXSeq için Tablo 2), koşullar arasında diferansiyel kullanımı gösteren ekzonların bir listesi ve istatistiksel anlamlılığa göre sıralanmış, bir veya daha fazla kurucu e...
Tartışmalar
Bu çalışmada, toplu RNA-Seq ve 3' uç dizileme verilerinde AS ve APA'yı saptamak için ekzon tabanlı ve olay tabanlı yaklaşımlar değerlendirildi. Ekzon tabanlı AS yaklaşımları, hem diferansiyel olarak eksprese edilen ekzonların bir listesini hem de genel gen seviyesi diferansiyel ekleme aktivitesinin istatistiksel önemine göre sıralanmış bir gen seviyesi sıralaması üretir (Tablo 1-2, 4-5). Diferansiyel kullanım, bir ekzonun diferansiyel log kıvrım değişimini aynı gen içindeki...
Açıklamalar
Yazarların açıklayacak hiçbir şeyleri yoktur.
Teşekkürler
Bu çalışma, Avustralya Araştırma Konseyi (ARC) Future Fellowship (FT16010043) ve ANU Futures Scheme tarafından desteklenmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Not relevent for computational study |
Referanslar
- Katz, Y., Wang, E. T., Airoldi, E. M., Burge, C. B. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods. 7 (12), 1009-1015 (2010).
- Wang, Y., et al. Mechanism of alternative splicing and its regulation. Biomedical Reports. 3 (2), 152-158 (2015).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in Bioinformatics. 21 (6), 2052-2065 (2020).
- Movassat, M., et al. Coupling between alternative polyadenylation and alternative splicing is limited to terminal introns. RNA Biology. 13 (7), 646-655 (2016).
- Tian, B., Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature Reviews Molecular Cell Biology. 18 (1), 18-30 (2017).
- Herrmann, C. J., et al. PolyASite 2.0: a consolidated atlas of polyadenylation sites from 3' end sequencing. Nucleic Acids Research. 48 (1), 174-179 (2020).
- Liu, R., Loraine, A. E., Dickerson, J. A. Comparisons of computational methods for differential alternative splicing detection using RNA-seq in plant systems. BMC Bioinformatics. 15 (1), 364 (2014).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (1), 13 (2016).
- Anders, S., Reyes, A., Huber, W. Detecting differential usage of exons from RNA-seq data. Genome Research. 22 (10), 2008-2017 (2012).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2014).
- Shen, S., et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data. Proceedings of the National Academy of Sciences. 111 (51), 5593-5601 (2014).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in bioinformatics. 21 (6), 2052-2065 (2020).
- Kanitz, A., et al. Comparative assessment of methods for the computational inference of transcript isoform abundance from RNA-seq data. Genome biology. 16 (1), 1-26 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Sznajder, L. J., et al. Loss of MBNL1 induces RNA misprocessing in the thymus and peripheral blood. Nature Communications. 11, 1-11 (2020).
- Batra, R., et al. Loss of MBNL leads to disruption of developmentally regulated alternative polyadenylation in RNA-mediated disease. Molecular Cell. 56 (2), 311-322 (2014).
- Leinonen, R., Sugawara, H., Shumway, M., et al. The sequence read archive. Nucleic acids research. 39, 19-21 (2010).
- Tange, O. . GNU parallel-the command-line power tool. 36, 42-47 (2011).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), 25 (2010).
- Veiga, D. F. T. maser: Mapping Alternative Splicing Events to pRoteins. R package version 1.4.0. , (2019).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (13), 25 (2009).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ramírez, F., Dündar, F., Diehl, S., Grüning, B. A., Manke, T. deepTools: a flexible platform for exploring deep-sequencing data. Nucleic acids research. 42 (1), 187-191 (2014).
- Merino, G. A., Conesa, A., Fernández, E. A. A benchmarking of workflows for detecting differential splicing and differential expression at isoform level in human RNA-seq studies. Briefings in bioinformatics. 20 (2), 471-481 (2019).
- Chhangawala, S., Rudy, G., Mason, C. E., Rosenfeld, J. A. The impact of read length on quantification of differentially expressed genes and splice junction detection. Genome biology. 16 (1), 1-10 (2015).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biol. 17, 13 (2016).
- Trapnell, C., et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 7 (3), 562-578 (2012).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 12, 323 (2011).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat Biotechnol. 34 (5), 525-527 (2016).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır