JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
RNA-seq 데이터에서 대체 스플라이싱 및 폴리아데닐화 식별
요약
대체 스플라이싱(AS) 및 대체 폴리아데닐화(APA)는 전사체 이소형과 그 생성물의 다양성을 확장합니다. 여기에서는 실험 조건에 따라 다양한 AS 및 APA를 검출하고 시각화하기 위해 벌크 RNA-seq 및 3' 말단 시퀀싱 분석을 분석하는 생물정보학 프로토콜을 설명합니다.
초록
실험/생물학적 조건에서 차등 유전자 발현(DGE)을 측정하기 위한 RNA-Seq의 일반적인 분석뿐만 아니라 RNA-seq 데이터를 활용하여 엑손 수준에서 다른 복잡한 조절 메커니즘을 탐색할 수도 있습니다. 대체 스플라이싱 및 폴리아데닐화는 전사 후 수준에서 유전자 발현을 조절하기 위해 다양한 이소형을 생성하여 유전자의 기능적 다양성에 중요한 역할을 하며, 분석을 전체 유전자 수준으로 제한하면 이 중요한 조절 층을 놓칠 수 있습니다. 여기에서는 바이오컨덕터와 DEXSeq, Limma 패키지의 diffSplice 및 rMATS를 포함한 기타 패키지 및 기능을 사용하여 조건에 따른 차등 엑손 및 폴리아데닐화 부위 사용의 식별 및 시각화를 위한 자세한 단계별 분석을 시연합니다.
서문
RNA-seq는 일반적으로 차등 유전자 발현 및유전자 발견을 추정하기 위해 수년에 걸쳐 널리 사용되어 왔습니다1. 또한 다양한 이소 형을 발현하는 유전자로 인해 다양한 엑손 수준 사용량을 추정하는 데 활용할 수 있으므로 전사 후 수준에서 유전자 조절을 더 잘 이해하는 데 기여할 수 있습니다. 대부분의 진핵생물 유전자는 mRNA 발현의 다양성을 증가시키기 위해 대안적 스플라이싱(AS)에 의해 상이한 이소형을 생성한다. AS 이벤트는 다른 패턴으로 나눌 수 있습니다 : ( "카세트") 엑손이 측면 인트론과 함께 전사체에서 완전히 제거되는 완전한 엑손 (SE)의 건너 뛰기; 대안 (공여체) 5' 스플라이스 부위 선택 (A5SS) 및 대안 3' (수용체) 스플라이스 부위 선택 (A3SS) 2개 이상의 스플라이스 부위가 엑손의 양쪽 말단에 존재할 때; 인트론이 성숙한 mRNA 전사체 내에 유지되는 경우 인트론(RI)의 유지 및 한 번에 두 개의 사용 가능한 엑손 중 하나만 보유할 수 있는 엑손 사용(MXE)의 상호 배제 2,3. 대안적 폴리아데닐화 (APA)는 또한 단일 전사체로부터 다수의 mRNA 이소형을 생성하기 위해 대안적인 폴리 (A) 부위를 사용하여 유전자 발현을 조절하는데 중요한 역할을한다4. 대부분의 폴리아데닐화 부위 (pAs)는 3' 비번역 영역 (3' UTR) 내에 위치하여, 다양한 3' UTR 길이를 갖는 mRNA 이소형을 생성한다. 3' UTR이 조절 요소를 인식하기 위한 중앙 허브이기 때문에, 상이한 3' UTR 길이는 mRNA 국소화, 안정성 및 번역(5)에 영향을 미칠 수 있다. 프로토콜6의 세부 사항에서 다른 APA를 검출하도록 최적화 된 3 '최종 시퀀싱 분석의 클래스가 있습니다. 여기에 설명된 파이프라인은 PolyA-seq용으로 설계되었지만 설명된 대로 다른 프로토콜에 맞게 조정할 수 있습니다.
이 연구에서는 차등 엑손 분석 방법7,8(그림 1)의 파이프라인을 제시하며, 이는 엑손 기반(DEXSeq9, diffSplice 10)과 이벤트 기반(전사체 접합의 복제 다변량 분석(rMATS)11)의 두 가지 범주로 나눌 수 있습니다. 엑손 기반 방법은 개별 엑손의 조건에 따른 폴드 변화를 차등적으로 발현된 엑손 사용량을 호출하기 위한 전체 유전자 폴드 변화의 척도와 비교하고, 그로부터 AS 활성의 유전자 수준 측정을 계산합니다. 이벤트 기반 방법은 엑손 인트론 스패닝 접합 읽기를 사용하여 엑손 건너뛰기 또는 인트론 유지와 같은 특정 스플라이싱 이벤트를 감지 및 분류하고 출력3에서 이러한 AS 유형을 구별합니다. 따라서 이러한 방법은 AS12,13의 완전한 분석을위한 보완적인 견해를 제공합니다. DEXSeq(DESeq214 DGE 패키지 기반)와 diffSplice(Limma10 DGE 패키지 기반)는 차동 접합 분석에 가장 널리 사용되는 패키지 중 하나이기 때문에 연구를 위해 선택했습니다. rMATS는 이벤트 기반 분석에 널리 사용되는 방법으로 선택되었습니다. 또 다른 인기 있는 이벤트 기반 방법은 MISO(Mix of Isoforms)1입니다. APA의 경우 엑손 기반 접근 방식을 적용합니다.
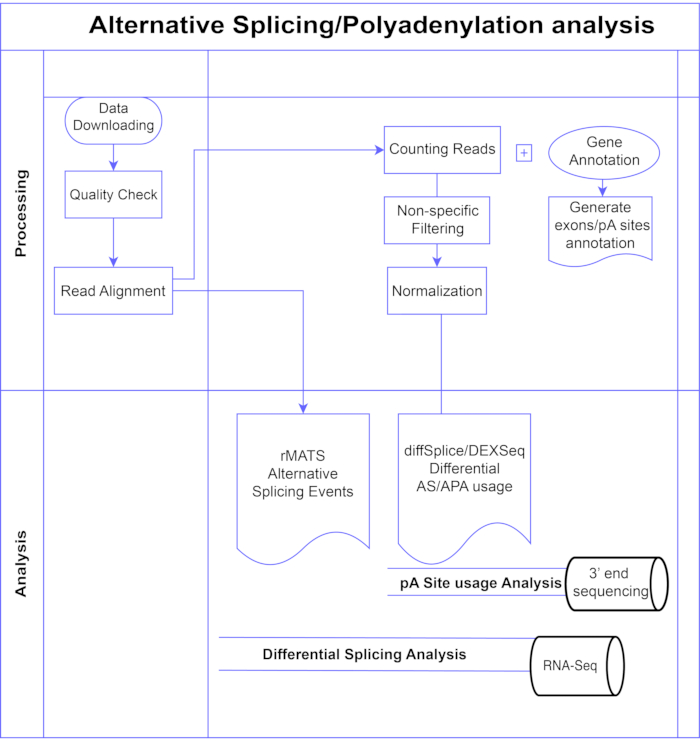
그림 1. 분석 파이프라인. 분석에 사용된 단계의 순서도입니다. 단계에는 데이터 획득, 품질 검사 수행 및 읽기 정렬 수행 후 알려진 엑손, 인트론 및 pA 사이트에 대한 주석을 사용하여 읽기 계산, 낮은 카운트 제거 및 정규화를 위한 필터링이 포함됩니다. PolyA-seq 데이터는 diffSplice/DEXSeq 방법을 사용하여 대체 pA 부위에 대해 분석되었고, 벌크 RNA-Seq는 diffSplice/DEXseq 방법을 사용하여 엑손 수준에서 대체 스플라이싱에 대해 분석되었으며, AS 이벤트는 rMATS로 분석되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
이 조사에 사용된 RNA-seq 데이터는 유전자 발현 옴니버스(GEO)(GSE138691)15에서 획득한 것이다. 우리는 이 연구의 마우스 RNA-seq 데이터를 야생형(WT) 및 근맹 유사 유형 1 녹아웃(Mbnl1 KO)의 두 가지 조건 그룹과 함께 각각 3개의 반복으로 사용했습니다. 차등 폴리아데닐화 부위 사용 분석을 입증하기 위해, 마우스 배아 섬유아세포(MEF) PolyA-seq 데이터(GEO Accession GSE60487)16를 얻었다. 데이터에는 야생형(WT), 근맹형 유형 1/유형 2 이중 녹아웃(Mbnl1/2 DKO), Mbnl3 녹다운(KD)이 있는 Mbnl 1/2 DKO 및 Mbnl3 대조군이 있는 Mbnl1/2 DKO(Ctrl)의 네 가지 조건 그룹이 있습니다. 각 조건 그룹은 두 번의 반복실험으로 구성됩니다.
| 지역 가입 | SRA 실행 번호 | 샘플 이름 | 조건 | 복제 | 조직 | 시퀀싱 | 읽기 길이 | |
| RNA-서열 | GSM4116218 | SRR10261601 | Mbnl1KO_Thymus_1 | Mbnl1 녹아웃 | 담당자 1 | 흉선 | 페어링 엔드 | 100 bp |
| GSM4116219 | SRR10261602 | Mbnl1KO_Thymus_2 | Mbnl1 녹아웃 | 담당자 2 | 흉선 | 페어링 엔드 | 100 bp | |
| GSM4116220 | SRR10261603 | Mbnl1KO_Thymus_3 | Mbnl1 녹아웃 | 담당자 3 | 흉선 | 페어링 엔드 | 100 bp | |
| GSM4116221 | SRR10261604 | WT_Thymus_1 | 와일드 타입 | 담당자 1 | 흉선 | 페어링 엔드 | 100 bp | |
| GSM4116222 | SRR10261605 | WT_Thymus_2 | 와일드 타입 | 담당자 2 | 흉선 | 페어링 엔드 | 100 bp | |
| GSM4116223 | SRR10261606 | WT_Thymus_3 | 와일드 타입 | 담당자 3 | 흉선 | 페어링 엔드 | 100 bp | |
| 3P-시퀀스 | GSM1480973 | SRR1553129 | WT_1 | 와일드 타입 (WT) | 담당자 1 | 마우스 배아 섬유아세포(MEF) | 단일 종단 | 40 bp |
| GSM1480974 | SRR1553130 | WT_2 | 와일드 타입 (WT) | 담당자 2 | 마우스 배아 섬유아세포(MEF) | 단일 종단 | 40 bp | |
| GSM1480975 | SRR1553131 | DKO_1 | Mbnl 1/2 더블 녹아웃 (DKO) | 담당자 1 | 마우스 배아 섬유아세포(MEF) | 단일 종단 | 40 bp | |
| GSM1480976 | SRR1553132 | DKO_2 | Mbnl 1/2 더블 녹아웃 (DKO) | 담당자 2 | 마우스 배아 섬유아세포(MEF) | 단일 종단 | 40 bp | |
| GSM1480977 | SRR1553133 | DKOsiRNA_1 | Mbnl 3 siRNA (KD)를 사용한 Mbnl 1/2 이중 녹아웃 | 담당자 1 | 마우스 배아 섬유아세포(MEF) | 단일 종단 | 40 bp | |
| GSM1480978 | SRR1553134 | DKOsiRNA_2 | Mbnl 3 siRNA (KD)를 사용한 Mbnl 1/2 이중 녹아웃 | 담당자 2 | 마우스 배아 섬유아세포(MEF) | 단일 종단 | 36 bp | |
| GSM1480979 | SRR1553135 | DKONTsiRNA_1 | 비표적 siRNA를 사용한 Mbnl 1/2 이중 녹아웃(Ctrl) | 담당자 1 | 마우스 배아 섬유아세포(MEF) | 단일 종단 | 40 bp | |
| GSM1480980 | SRR1553136 | DKONTsiRNA_2 | 비표적 siRNA를 사용한 Mbnl 1/2 이중 녹아웃(Ctrl) | 담당자 2 | 마우스 배아 섬유아세포(MEF) | 단일 종단 | 40 bp |
표 1. 분석에 사용된 RNA-Seq 및 PolyA-seq 데이터 세트의 요약.
프로토콜
1. 분석에 사용되는 도구 및 R 패키지 설치
- Conda는 모든 플랫폼에서 종속성이 있는 패키지를 편리하게 설치할 수 있는 인기 있고 유연한 패키지 관리자입니다. 'Anaconda'(conda 패키지 관리자)를 사용하여 분석에 필요한 도구 / 패키지를 설치하는 데 사용할 수있는 'conda'를 설치하십시오.
- https://www.anaconda.com/products/individual#Downloads 에서 시스템 요구 사항에 따라 'Anaconda'를 다운로드하고 그래픽 설치 프로그램의 지시에 따라 설치하십시오. Linux 명령줄에 다음을 입력하여 'conda' 를 사용하여 필요한 모든 패키지를 설치합니다.
conda install -c daler sratoolkit
conda install -c conda-forge parallel
conda install -c bioconda star bowtie fastqc rmats rmats2sashimiplot samtools fasterq-dump cutadapt bedtools deeptools - 프로토콜에 사용 된 모든 R 패키지를 다운로드 하려면 R 콘솔 (Linux 명령줄에서 'R'을 입력 하 여 시작) 또는 Rstudio 콘솔에서 다음 코드를 입력 합니다.
bioc_packages<- c("DEXSeq", "Rsubread", "EnhancedVolcano", "edgeR", "limma", "maser","GenomicRanges")
packages<- c("magrittr", "rtracklayer", "tidyverse", "openxlsx", "BiocManager")
#Install if not already installed
installed_packages<-packages%in% rownames(installed.packages())
installed_bioc_packages<-bioc_packages%in% rownames(installed.packages())
if(any(installed_packages==FALSE)) {
install.packages(packages[!installed_packages],dependencies=TRUE)
BiocManager::install(packages[!installed_bioc_packages], dependencies=TRUE)
}
참고: 이 계산 프로토콜에서 명령은 R Notebook 파일(확장자가 "인 파일)로 제공됩니다. Rmd"), R 코드 파일(확장자가 "인 파일". R") 또는 Linux Bash 셸 스크립트(확장자가 ".sh"인 파일). R 노트북 (Rmd) 파일은 파일 | 파일 열기..., 개별 코드 청크(R 명령 또는 Bash 셸 명령일 수 있음)를 클릭한 다음 오른쪽 위에 있는 녹색 화살표를 클릭하여 대화형으로 실행합니다. R 코드 파일은 RStudio에서 열거나 Linux 명령줄에서 앞에 "Rscript"(예: Rscript example)를 사용하여 실행할 수 있습니다. 셸 스크립트는 스크립트 앞에 "sh" 명령(예: 예: .sh example.sh)을 추가하여 Linux 명령줄에서 실행됩니다.
2. RNA-seq를 이용한 대체 접합(AS) 분석
- 데이터 다운로드 및 전처리
참고: 아래 주석이 달린 코드 조각은 보충 코드 파일 "AS_analysis_RNASeq.RMD", 대화식으로 개별 단계를 따르며 Linux 명령 줄에서 일괄 적으로 실행되는 bash 스크립트로도 제공됩니다 (쉬 downloading_data_preprocessing.sh).- 원시 데이터 다운로드.
- SRA 툴킷(v2.10.8)17의 '프리페치' 명령을 사용하여 시퀀스 읽기 아카이브(SRA)에서 원시 데이터를 다운로드합니다. 다음 명령에서 SRA 가입 ID를 순서대로 제공하여 GNU 병렬 유틸리티18을 사용하여 병렬로 다운로드합니다. SRR10261601에서 SRR10261606으로 등록 ID의 SRA 파일을 병렬로 다운로드하려면 Linux 명령줄에서 다음을 사용합니다.
seq 10261601 10261606 | parallel prefetch SRR{} - SRA 툴킷에서 'fastq-dump'기능을 사용하여 아카이브에서 fastq 파일을 추출합니다. GNU 병렬을 사용하고 모든 SRA 파일의 이름을 함께 지정하십시오.
parallel -j 3 fastq-dump --gzip --skip-technical --read-filter pass --dumpbase --split-e --clip --origfmt {} ::: - Linux 명령줄에서 다음을 사용하여 마우스(게놈 어셈블리 GRCm39)에 대한 참조 게놈 및 주석을 www.ensembl.org 에서 다운로드합니다.
wget -nv -O annotation.gtf.gz http://ftp.ensembl.org/pub/release-103/gtf/mus_musculus/Mus_musculus.GRCm39.103.gtf.gz \ && gunzip -f annotation.gtf.gz
wget -nv -O genome.fa.gz http://ftp.ensembl.org/pub/release-103/fasta/mus_musculus/dna/Mus_musculus.GRCm39.dna.primary_assembly.fa.gz \ && gunzip -f genome.fa.gz
GTF=$(readlink -f annotation.gtf)
GENOME=$(readlink -f genome.fa)
- SRA 툴킷(v2.10.8)17의 '프리페치' 명령을 사용하여 시퀀스 읽기 아카이브(SRA)에서 원시 데이터를 다운로드합니다. 다음 명령에서 SRA 가입 ID를 순서대로 제공하여 GNU 병렬 유틸리티18을 사용하여 병렬로 다운로드합니다. SRR10261601에서 SRR10261606으로 등록 ID의 SRA 파일을 병렬로 다운로드하려면 Linux 명령줄에서 다음을 사용합니다.
- 전처리 및 매핑은 게놈 어셈블리에 대한 판독
- 품질 관리. FASTQC(FASTQ 품질 검사 v0.11.9)19를 사용하여 원시 판독값의 품질을 평가합니다. 출력 폴더를 만들고 여러 입력 fasta 파일에서 병렬로 fastqc를 실행합니다. 이 단계에서는 각 샘플에 대한 품질 보고서를 생성합니다. 추가 분석을 수행하기 전에 보고서를 검사하여 읽기 품질이 허용되는지 확인합니다. ( https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ 의 보고서를 이해하려면 사용 설명서를 참조하십시오.)
mkdir fastqc_out
parallel "fastqc {} -o fastqc_out" ::: $RAW_DATA/*.fastq.gz
참고: 필요한 경우 'cutadapt'20 또는 'trimmomatic'21로 어댑터 트리밍을 수행하여 RNA 단편 크기 및 판독 길이에 따라 달라지는 측면 어댑터로의 시퀀싱을 제거합니다. 이 분석에서는 영향을 받는 읽기 비율이 최소화되었기 때문에 이 단계를 건너뛰었습니다. - 읽기 정렬. 전처리의 다음 단계에는 판독값을 참조 게놈에 매핑하는 작업이 포함됩니다. 먼저 STAR22 의 'genomeGenerate' 기능을 사용하여 참조 게놈에 대한 인덱스를 구축한 다음 원시 판독값을 참조에 정렬합니다(또는 STAR 웹사이트에서 사전 빌드된 인덱스를 사용할 수 있으며 정렬에 직접 사용할 수 있음). Linux 명령줄에서 다음 명령을 실행합니다.
#Build STAR index
GDIR=STAR_indices
mkdir $GDIR
STAR --runMode genomeGenerate --genomeFastaFiles $GENOME --sjdbGTFfile $GTF --runThreadN 8 --genomeDir $GDIR
ODIR=results/mapping
mkdir -p $ODIR
#Align reads to the genome
for fq1 in $RAW_DATA/*R1.fastq.gz;
do
fq2=$(echo $fq1| sed 's/1.fastq.gz/2.fastq.gz/g');
OUTPUT=$(basename ${fq1}| sed 's/R1.fastq.gz//g');
STAR --genomeDir $GDIR \
--runThreadN 12 \
--readFilesCommand zcat \
--readFilesIn ${fq1}${fq2}\
--outFileNamePrefix $ODIR\/${OUTPUT} \
--outSAMtype BAM SortedByCoordinate \
--outSAMunmapped Within \
--outSAMattributes Standard
Done
참고: STAR 얼라이너는 읽기 정렬 후 각 샘플에 대한 BAM(이진 정렬 맵) 파일을 생성하고 정렬합니다. 추가 단계를 진행하기 전에 Bam 파일을 정렬해야 합니다.
- 품질 관리. FASTQC(FASTQ 품질 검사 v0.11.9)19를 사용하여 원시 판독값의 품질을 평가합니다. 출력 폴더를 만들고 여러 입력 fasta 파일에서 병렬로 fastqc를 실행합니다. 이 단계에서는 각 샘플에 대한 품질 보고서를 생성합니다. 추가 분석을 수행하기 전에 보고서를 검사하여 읽기 품질이 허용되는지 확인합니다. ( https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ 의 보고서를 이해하려면 사용 설명서를 참조하십시오.)
- 원시 데이터 다운로드.
- 엑손 주석 준비.
- 보조 코드 파일 "prepare_mm_exon_annotation. R"을 GTF(유전자 전달 포맷) 포맷으로 다운로드한 주석과 함께 주석을 준비한다. 실행하려면 Linux 명령줄에 다음을 입력합니다.
Rscript prepare_mm_exon_annotation.R annotation.gtf
참고 : GTF 파일에는 서로 다른 이소 폼에 대한 여러 엑손 항목이 포함되어 있습니다. 이 파일은 각 엑손에 대한 여러 전사체 ID를 "축소"하는 데 사용됩니다. 엑손 카운팅 빈을 정의하는 것은 중요한 단계입니다.
- 보조 코드 파일 "prepare_mm_exon_annotation. R"을 GTF(유전자 전달 포맷) 포맷으로 다운로드한 주석과 함께 주석을 준비한다. 실행하려면 Linux 명령줄에 다음을 입력합니다.
- 읽기 계산. 다음 단계는 다른 전사체/엑손에 매핑된 읽기 수를 계산하는 것입니다. 보조 파일 "AS_analysis_RNASeq.Rmd"를 참조하십시오.
- 필요한 라이브러리 로드:
packages<- c("Rsubread","tidyverse", "magrittr", "EnhancedVolcano", "edgeR","openxlsx")
invisible(lapply(packages, library, character.only=TRUE)) - 이전 단계(2.2)에서 가져온 처리된 주석 파일을 로드합니다.
load("mm_exon_anno.RData") - 2.2.2단계에서 얻은 모든 bam 파일을 읽기 횟수를 계산하기 위한 'featureCounts' 입력으로 읽습니다. 먼저 .bam으로 끝나는 디렉터리에서 각 파일을 나열하여 bam 파일이 포함된 폴더를 읽습니다. bam 파일을 사용하고 GTF 주석 (참조)을 입력으로 처리하는 Rsubread 패키지의 'featureCounts'를 사용하여 엑손 (기능)을 나타내는 행과 샘플을 나타내는 열이있는 각 기능과 관련된 카운트 행렬을 생성합니다.
countData <- dir("bams", pattern=".bam$", full.names=T) %>%
featureCounts(annot.ext=anno,
isGTFAnnotationFile=FALSE,
minMQS=0,useMetaFeatures=FALSE,
allowMultiOverlap=TRUE,
largestOverlap=TRUE,
countMultiMappingReads=FALSE,
primaryOnly=TRUE,
isPairedEnd=TRUE,
nthreads=12) - 다음으로, 비특이적 필터링을 수행하여 낮게 발현된 엑손을 제거한다("비특이적"은 선택 편향을 피하기 위해 실험 조건 정보가 필터링에 사용되지 않음을 나타냄). 'edgeR'패키지23 의 cpm 함수를 사용하여 원시 스케일에서 백만 당 카운트 (cpm)로 데이터를 변환하고 최소 3 개의 샘플에서 설정 가능한 임계 값 (이 데이터 세트의 경우 1 cpm이 사용됨)보다 큰 카운트를 가진 엑손을 유지합니다. 또한 하나의 엑손 만 가진 유전자를 제거하십시오.
# Non-specific filtering: Remove the exons with low counts
isexpr<- rownames(countData$counts)[rowSums(cpm(countData$counts)>1) >=3]
countData$counts<-countData$counts[rownames(countData$counts) %in%isexpr, ]
anno<-anno%>% filter(GeneID%in% rownames(countData$counts))
# Remove genes with only 1 site and NA in geneIDs
dn<-anno%>%group_by(GeneID)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(GeneID))
anno<-anno%>% filter(GeneID%in%dn$GeneID)
countData$counts<-countData$counts[rownames(countData$counts) %in%anno$GeneID, ]
참고: 기능 개수에 필요한 매개 변수를 확인합니다(예: 단일 끝 읽기의 경우) 다른 데이터를 사용하는 경우 'isPairedEnd = FALSE'를 설정합니다. RSubread 사용자 가이드를 참조하여 데이터에 대한 옵션을 선택하고 아래의 토론 섹션을 참조하십시오.
- 필요한 라이브러리 로드:
- 차등 접합 및 엑손 사용 분석. 이 단계에 대한 두 가지 대안인 DEXSeq 및 DiffSplice에 대해 설명합니다. 어느 쪽이든 사용할 수 있으며 비슷한 결과를 얻을 수 있습니다. DGE용 DESeq2 패키지를 선호하는 경우 일관성을 위해 DEXSeq를 선택하고 Limma 기반 DGE 해석에 DiffSplice를 사용합니다. 보충 파일 참조: "AS_analysis_RNASeq.RMD".
- 차동 엑손 분석을 위해 DEXSeq 패키지 사용.
- 라이브러리를 로드하고 샘플 테이블을 작성하여 실험 설계를 정의합니다.
library(DEXSeq)
sampleTable<-data.frame(row.names= c("Mbnl1KO_Thymus_1", "Mbnl1KO_Thymus_2", "Mbnl1KO_Thymus_3", "WT_Thymus_1", "WT_Thymus_2", "WT_Thymus_3"), condition= rep(c("Mbnl1_KO", "WT"),c(3,3)), libType= rep(c("paired-end")))
참고: 행 이름은 읽기 횟수를 계산하기 위해 featureCounts에서 사용하는 bam 파일 이름과 일치해야 합니다. sampleTable은 라이브러리 유형 및 조건을 포함하는 각 샘플의 세부 정보로 구성됩니다. 이는 차등 사용을 감지하기 위한 대비 또는 테스트 그룹을 정의하는 데 필요합니다. - 엑손 정보 파일을 준비합니다. GRanges(게놈 범위) 객체(https://bioconductor.org/packages/release/bioc/html/GenomicRanges.html) 형태의 엑손 정보는 다음 단계에서 DEXSeq 객체를 생성하기 위한 입력으로 필요합니다. 유전자 ID를 읽기 횟수와 일치시켜 exoninfo 개체를 만듭니다.
exoninfo<-anno[anno$GeneID%in% rownames(countData$counts),]
exoninfo<-GRanges(seqnames=anno$Chr,
ranges=IRanges(start=anno$Start, end=anno$End, width=anno$Width),strand=Rle(anno$Strand))
mcols(exoninfo)$TranscriptIDs<-anno$TranscriptIDs
mcols(exoninfo)$Ticker<-anno$Ticker
mcols(exoninfo)$ExonID<-anno$ExonID
mcols(exoninfo)$n<-anno$n
mcols(exoninfo)$GeneID<-anno$GeneID
transcripts_l= strsplit(exoninfo$TranscriptIDs, "\\,")
save(countData, sampleTable, exoninfo, transcripts_l, file="AS_countdata.RData") - DEXSeqDataSet 함수를 사용하여 DEXSeq 객체를 만듭니다. DEXSeq 객체는 읽기 횟수, 엑손 특징 정보 및 샘플 정보를 함께 수집합니다. 3단계에서 생성된 읽기 횟수와 이전 단계에서 얻은 엑손 정보를 사용하여 개수 행렬에서 DEXSeq 개체를 만듭니다. sampleData 인수는 샘플(및 해당 속성: 라이브러리 유형 및 조건)을 정의하는 데이터 프레임 입력을 사용하고, 'design'은 sampleData를 사용하여 모델 공식 표기법을 사용하여 차등 테스트를 위한 설계 매트릭스를 생성합니다. 유의한 상호작용 용어인 condition:exon은 특정 엑손에 속하는 유전자에 대한 판독값의 비율이 실험 조건, 즉 AS가 있음에 따라 달라진다는 것을 나타냅니다. 더 복잡한 실험 설계를 위한 모델 공식 설정에 대한 전체 설명은 DEXSeq 설명서를 참조하십시오. 특징 정보를 위해서는 엑손 ID, 해당 유전자 및 전사체가 필요합니다.
dxd<-DEXSeqDataSet(countData$counts,sampleData=sampleTable, design=~sample+exon+condition:exon,featureID=exoninfo$ExonID,groupID=exoninfo$GeneID,featureRanges=exoninfo, transcripts=transcripts_l) - 정규화 및 분산 추정. 다음으로, 다음 명령을 사용하여 샘플 간의 정규화를 수행하고 RNA-seq의 이산 특성으로 인한 포아송 카운트 잡음과 생물학적 변동성으로 인한 데이터의 분산을 추정합니다.
dxd %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - 차등 사용을 테스트합니다. 변이 추정 후, 각 유전자에 대한 차등 엑손 사용량을 테스트하고 결과를 생성한다.
dxd%<>%testForDEU%>%estimateExonFoldChanges(fitExpToVar=
"condition")#Estimate fold changes
dxr=DEXSeqResults(dxd) - 다음 명령을 사용하여 선택된 유전자에 대한 접합 이벤트를 시각화합니다.
plotDEXSeq(dxr,"Wnk1", displayTranscripts=TRUE, splicing=TRUE,legend
=TRUE,cex.axis=1.2,cex=1.3,lwd=2)
R Notebook 파일 "AS_analysis_RNASeq.Rmd"를 검사하여 관심 유전자에 대한 추가 플롯을 생성하고 다른 임계값에서 화산 플롯을 생성합니다.
- 라이브러리를 로드하고 샘플 테이블을 작성하여 실험 설계를 정의합니다.
- Limma의 diffSplice를 사용하여 차등 접합을 식별합니다. R 노트북 파일을 따르십시오 "AS_analysis_RNASeq.RMD". 계속 진행하기 전에 입력 파일을 준비하기 위해 2.1-2.3단계를 수행했는지 확인합니다.
- 라이브러리 로드
library(limma)
library(edgeR) - 불특정 필터링. 2.3에서 얻은 읽기 횟수의 행렬을 추출합니다. edgeR 패키지의 'DGEList'함수를 사용하여 기능 목록을 만듭니다.이 경우 행은 유전자를 나타내고 열은 샘플을 나타냅니다.
mycounts=countData$counts
#Change the rownames of the countdata to exon Ids instead of genes for unique rownames.
rownames(mycounts) = exoninfo$ExonID
dge<-DGEList(counts=mycounts)
#Filtering
isexpr<- rowSums(cpm(dge)>1) >=3
dge<-dge[isexpr,,keep.lib.sizes=FALSE]
#Extract the exon annotations for only transcripts meeting non-specific filter
exoninfo=anno%>% filter(ExonID%in% rownames(dge$counts))
#Convert the exoninfo into GRanges object
exoninfo1<-GRanges(seqnames=exoninfo$Chr,
ranges=IRanges(start=exoninfo$Start, end=exoninfo$End, width=exoninfo$Width),strand=Rle(exoninfo$Strand))
mcols(exoninfo1)$TranscriptIDs<-exoninfo$TranscriptIDs
mcols(exoninfo1)$Ticker<-exoninfo$Ticker
mcols(exoninfo1)$ExonID<-exoninfo$ExonID
mcols(exoninfo1)$n<-exoninfo$n
mcols(exoninfo1)$GeneID<-exoninfo$GeneID
transcripts_l= strsplit(exoninfo1$TranscriptIDs, "\\,")
참고: 비특이적 필터링 단계로서 카운트는 n개 샘플 중 cpm < 1/x 1로 필터링되며, 여기서 x는 모든 조건에서 최소 반복실험 횟수입니다. 이 예제 데이터의 경우 n = 6 및 x = 3입니다. - M 값의 절사 평균(TMM 정규화 방법)24을 사용하여 'edgeR' 패키지의 'calcNormFactors' 함수를 사용하여 샘플 전체의 개수를 정규화합니다.24 라이브러리 크기를 조정하기 위해 배율 인수를 계산합니다.
dge<-calcNormFactors(dge) - 2.4.1.1단계에서 생성된 sampleTable을 사용하여 디자인 매트릭스를 만듭니다. 설계 매트릭스는 설계를 특징짓습니다. Limma User Guide (https://www.bioconductor.org/packages/devel/bioc/vignettes/limma/inst/doc/usersguide.pdf) 장 8 & 9 장에서 고급 실험 설계를위한 설계 매트릭스에 대한 자세한 내용을 참조하십시오.
Treat<- factor(sampleTable$condition)
design<- model.matrix(~0+Treat)
colnames(design) <- levels(Treat) - 엑손당 선형 모델을 피팅합니다. 'limma' 패키지의 'voom' 함수를 실행하여 RNA-seq 데이터를 처리하여 분산을 추정하고 포아송 카운트 잡음을 보정하기 위한 정밀 가중치를 생성하고 엑손 레벨 카운트를 백만당 log2-counts(logCPM)로 변환합니다. 그런 다음 'lmfit' 함수를 사용하여 선형 모델링을 실행하여 선형 모델을 각 엑손의 발현 데이터에 피팅합니다. 미분 엑손 발현을 탐지하기 위해 'eBayes' 함수를 사용하여 적합 모델에 대한 경험적 Bayes 통계량을 계산합니다. 다음으로, 관심 있는 실험적 비교를 위한 대비 행렬을 정의합니다. 'contrasts.fit'을 사용하여 각 비교 쌍에 대한 계수와 표준 오차를 얻을 수 있습니다.
v<-voom(dge,design,plot=FALSE)
fit<-lmFit(v,design)
fit<-eBayes(fit)
colnames(fit)
cont.matrix<-makeContrasts(
Mbnl1_KO_WT=Mbnl1_KO-WT,
levels=design)
fit2<-contrasts.fit(fit,cont.matrix) - 차동 접합 분석. 적합된 모델에서 'diffSplice'를 실행하여 야생형과 녹아웃 간 유전자의 엑손 사용 차이를 테스트하고 'topSplice' 함수를 사용하여 최상위 결과를 탐색합니다. test="t"는 AS 엑손의 순위를 제공하고 test="simes"는 유전자의 순위를 제공합니다.
ex<-diffSplice(fit2,geneid=exoninfo$GeneID,exonid=exoninfo$ExonID)
ts<-topSplice(ex,n=Inf,FDR=0.1, test="t", sort.by="logFC")
tg<-topSplice(ex,n=Inf,FDR=0.1, test="simes") - 시각화. 'plotSplice' 함수로 결과를 플로팅하여 geneid 인수에 관심 유전자를 제공합니다. 로그별로 정렬된 상위 결과 저장 변경 사항을 개체로 접고 엑손을 나타내는 화산 플롯을 생성합니다.
plotSplice(ex,geneid="Wnk1", FDR=0.1)
#Volcano plot
EnhancedVolcano(ts,lab=ts$ExonID,selectLab= head((ts$ExonID),2000), xlab= bquote(~Log[2]~'fold change'), x='logFC', y='P.Value', title='Volcano Plot', subtitle='Mbnl1_KO vs WT (Limma_diffSplice)', FCcutoff=2, labSize=4,legendPosition="right", caption= bquote(~Log[2]~"Fold change cutoff, 2; FDR 10%"))
- 라이브러리 로드
- rMATS 사용
- 작업 디렉터리에 conda 또는 github(https://github.com/Xinglab/rmats-turbo/releases/download/v4.1.1/rmats_turbo_v4_1_1.tar.gz)를 사용하여 최신 버전의 rMATS v4.1.1(처리 시간 단축 및 메모리 요구 사항 감소로 인해 rMATS 터보라고도 함)이 설치되어 있는지 확인합니다. "AS_analysis_RNASeq.Rmd"의 섹션 4.3을 따르십시오.
- 매핑 후 얻은 bam 파일이 포함된 폴더로 이동하고 rMATS의 필요에 따라 bam 파일의 이름(경로와 함께)을 ','로 구분하여 두 조건에 대한 텍스트 파일을 준비합니다. Linux 명령줄에서 다음 명령을 실행해야 합니다.
mkdir rMATS_analysis
cd bams/
ls -pd "$PWD"/*| grep "WT"| tr '\n'','> Wt.txt
ls -pd "$PWD"/*| grep "Mb"| tr '\n'','> KO.txt
mv *.txt ../rMATS_analysis - 2.1.1.3에서 가져온 GTF 파일과 함께 이전 단계에서 생성된 두 개의 입력 파일을 사용하여 rmats.py 실행합니다. 이렇게 하면 각 접합 이벤트에 대한 통계(p-값 및 포함 수준)를 개별적으로 설명하는 텍스트 파일이 포함된 출력 폴더 'rmats_out'가 생성됩니다.
python rmats-turbo/rmats.py --b1 KO.txt --b2 Wt.txt --gtf annotation.gtf -t paired --readLength 50 --nthread 8 --od rmats_out/ --tmp rmats_tmp --task pos
참고: GTF 파일 형식의 참조 주석도 필요합니다. 데이터가 단일 끝인 경우 매개 변수를 확인하고 그에 따라 -t 옵션을 변경합니다. - rMATS 결과 탐색. 바이오컨덕터 패키지 'maser'25 를 사용하여 rMATS 결과를 탐색하십시오. 접합 및 엑손 카운트(JCEC) 텍스트 파일을 'maser' 객체에 로드하고 스플라이싱 이벤트당 평균 읽기 횟수를 5개 이상 포함하여 적용 범위를 기준으로 결과를 필터링합니다.
library(maser)
mbnl1<-maser("/rmats_out/", c("WT","Mbnl1_KO"), ftype="JCEC")
#Filtering out events by coverage
mbnl1_filt<-filterByCoverage(mbnl1,avg_reads=5) - rMATS 결과 시각화. 'maser'패키지의 'topEvents'함수를 사용하여 거짓 발견 속도 (FDR) 10 % 및 스플 라이스 인 비율 (deltaPSI)의 최소 10 % 변화에서 중요한 스플라이싱 이벤트를 선택하십시오. 다음으로, 관심 유전자(샘플 gene-Wnk1)에 대한 유전자 이벤트를 확인하고 해당 유전자의 각 스플라이싱 이벤트에 대한 PSI 값을 플로팅합니다. 사건 유형을 지정하여 화산 플롯을 생성합니다.
#Top splicing events at 10% FDR
mbnl1_top<-topEvents(mbnl1_filt,fdr=0.1, deltaPSI=0.1)
mbnl1_top
#Check the gene events for a particular gene
mbnl1_wnk1<-geneEvents(mbnl1_filt,geneS="Wnk1", fdr=0.1, deltaPSI=0.1)
maser::display(mbnl1_wnk1,"SE")
plotGenePSI(mbnl1_wnk1,type="SE", show_replicates
=TRUE)
volcano(mbnl1_filt,fdr=0.1, deltaPSI=0.1,type="SE")
+xlab("deltaPSI")+ylab("Log10 Adj. Pvalue")+ggtitle("Volcano Plot of exon skipping events") - rMATS로 얻은 접합 이벤트 결과에 대한 사시미 플롯을 'rmats2shahimiplot' 패키지를 사용하여 텍스트 파일 형식으로 생성합니다. 리눅스 명령줄에서 파이썬 스크립트를 실행합니다.
python ./src/rmats2sashimiplot/rmats2sashimiplot.py --b1 ../bams/WT_Thymus_1.bam,../bams/WT_Thymus_2.bam,../bams/WT_Thymus_3.bam --b2 ../bams/Mbnl1KO_Thymus_1.bam,../bams/Mbnl1KO_Thymus_2.bam,../bams/Mbnl1KO_Thymus_3.bam -t SE -e ../rMATS_analysis/rmats_out/SE.MATS.JC.txt --l1 WT --l2 Mbnl1_KO --exon_s 1 --intron_s 5 -o ../rMATS_analysis/rmats2shasmi_output
참고: 이 프로세스는 이벤트 파일의 모든 결과에 대한 사시미 플롯을 생성하므로 시간이 많이 걸릴 수 있습니다. 'maser'에서 topEvents 함수에 의해 표시되는 상위 결과(유전자 이름 및 엑손)를 선택하고 해당 사시미 플롯을 시각화합니다.
- 차동 엑손 분석을 위해 DEXSeq 패키지 사용.
3. 3' 말단 시퀀싱을 사용한 대체 폴리아데닐화(APA) 분석
- 데이터 다운로드 및 전처리
참고: 보충 R 노트북 파일 "APA_analysis_3PSeq_notebook. Rmd"를 사용하여 데이터 다운로드 및 전처리 단계를 위한 전체 명령을 사용하거나 Linux 명령줄에서 보충 bash 파일 "APA_data_downloading_preprocessing.sh"를 실행합니다.- 등록 ID(1553129 - 1553136)를 사용하여 SRA에서 데이터를 다운로드합니다.
- 어댑터를 다듬고 리버스 보수를 수행하여 감지 스트랜드 시퀀스를 얻습니다.
참고: 이 단계는 사용된 PolyA-seq 분석에만 해당됩니다. - 지도는 나비 넥타이 얼라이너26을 사용하여 마우스 게놈 어셈블리를 읽습니다.
- pA 사이트 주석 준비.
참고: pA 사이트 주석 파일의 처리는 먼저 보충 R 노트북 파일 "APA_analysis_3PSeq_notebook. Rmd"(2.1 - 2.6)를 사용한 다음 bash 파일 "APA_annotation_preparation.sh"을 사용합니다.- PolyASite 2.0 데이터베이스에서 pA 사이트 주석 다운로드6.
- 다운스트림 분석을 위해 주석이 달린 말단 엑손(DS)의 터미널 엑손(TE) 또는 1000nt 다운스트림으로 주석이 달린 3'-비번역 영역(UTR) pA 사이트를 유지하기 위해 pA 사이트 주석을 선택합니다.
- pA 사이트 피크를 가져옵니다. 각 pA 절단 부위에 고정하고, 베드툴 및 딥툴(27,28)을 사용하여 평균 판독 커버리지를 시각화한다. 결과는 매핑된 판독값의 피크가 주로 절단 부위의 상류에서 ~60bp 이내에 분산되어 있음을 보여주었습니다(그림 5 및 보충 그림 5). 따라서, pA 부위의 좌표는 주석 파일로부터 그들의 분열 부위의 60bp 상류까지 확장되었다. 사용된 특정 3' 말단 시퀀싱 프로토콜에 따라 이 단계는 PolyA-seq 이외의 분석에 대해 최적화되어야 합니다.
- 읽기 횟수 계산
- pA 사이트 주석 파일을 준비합니다.
anno<- read.table(file= "flanking60added.pA_annotation.bed",
stringsAsFactors=FALSE, check.names=FALSE, header=FALSE, sep="")
colnames(anno) <- c("chrom", "chromStart", "chromEnd", "name", "score", "strand", "rep", "annotation", "gene_name", "gene_id")
anno<- dplyr::select(anno,name,chrom, chromStart,chromEnd, strand,gene_id,gene_name,rep)
colnames(anno) <- c("GeneID", "Chr", "Start", "End", "Strand", "Ensembl", "Symbol", "repID") - '기능 개수'를 적용하여 원시 개수를 획득합니다. 다른 도구를 사용하여 APA 분석을 위해 카운트 테이블을 RData 파일 "APA_countData.Rdata"로 저장합니다.
countData<- dir("bamfiles", pattern="sorted.bam$", full.names=TRUE) %>%
# Read all bam files as input for featureCounts
featureCounts(annot.ext=anno, isGTFAnnotationFile= FALSE,minMQS=0,useMetaFeatures= TRUE,allowMultiOverlap=TRUE, largestOverlap= TRUE,strandSpecific=1, countMultiMappingReads =TRUE,primaryOnly= TRUE,isPairedEnd= FALSE,nthreads=12)%T>%
save(file="APA_countData.Rdata")
참고: 'featureCounts' 기능에 나열된 매개변수를 변경해야 합니다. 'strandSpecific' 매개변수를 수정하여 사용된 3' 말단 시퀀싱 분석의 시퀀싱 방향과 일치하도록 합니다(경험적으로 플러스 및 마이너스 가닥의 유전자에 대해 게놈 브라우저에서 데이터를 시각화하면 이를 명확히 할 수 있습니다). - countData의 불특정 필터링을 적용합니다. 필터링은 차등 pA 사이트 사용 테스트에서 통계적 견고성을 크게 향상시킬 수 있습니다. 첫째, 우리는 차등 적으로 pA 부위 사용을 정의 할 수없는 단 하나의 pA 부위로 이들 유전자를 제거했다. 둘째, 커버리지를 기반으로 비특이적 필터링을 적용합니다: 카운트는 n개의 샘플 중 x에서 1보다 작은 cpm으로 필터링되며, 여기서 x는 모든 조건에서 최소 반복 횟수입니다. 이 예제 데이터의 경우 N = 8 및 x = 2입니다.
load(file= "APA_countData.Rdata")# Skip this step if already loaded
# Non-specific filtering: Remove the pA sites not differentially expressed in the samples
countData<-countData$counts%>%as.data.frame%>% .[rowSums(edgeR::cpm(.)>1) >=2, ]
anno%<>% .[.$GeneID%in% rownames(countData), ]
# Remove genes with only 1 site and NA in geneIDs
dnsites<-anno%>%group_by(Symbol)%>%summarise(nsites=n())%>% filter(nsites>1&!is.na(Symbol))
anno<-anno%>% filter(Symbol%in%dnsites$Symbol)
countData<-countData[rownames(countData) %in%anno$GeneID, ]
- pA 사이트 주석 파일을 준비합니다.
- DEXSeq 및 diffSplice 파이프라인을 사용한 차등 폴리아데닐화 부위 사용량 분석.
- DEXSeq 패키지 사용
참고: DEXSeq 파이프라인에 대해 대비 매트릭스를 정의할 수 없으므로 각 두 실험 조건의 차동 APA 분석을 별도로 수행해야 합니다. WT 조건과 DKO 조건의 시차 APA 분석을 예로 들어 절차를 설명합니다. 보충 파일 참조 "APA_analysis_3PSeq_notebook. 증권 시세 표시기" 이 섹션의 단계별 워크 플로우와 다른 대비의 차등 APA 분석.- 라이브러리를 로드하고 샘플 테이블을 작성하여 실험 설계를 정의합니다.
c("DEXSeq", "GenomicRanges") %>% lapply(library, character.only=TRUE) %>%invisible
sampleTable1<- data.frame(row.names= c("WT_1","WT_2","DKO_1","DKO_2"),
condition= c(rep("WT", 2), rep("DKO", 2)),
libType= rep("single-end", 4)) - 바이오컨덕터 패키지 GRanges를 사용하여 pA 사이트 정보 파일을 준비합니다.
# Prepare the GRanges object for DEXSeqDataSet object construction
PASinfo <- GRanges(seqnames = anno$Chr,
ranges = IRanges(start = anno$Start, end = anno$End),strand = Rle(anno$Strand))
mcols(PASinfo)$PASID<-anno$repID
mcols(PASinfo)$GeneEns<-anno$Ensembl
mcols(PASinfo)$GeneID<-anno$Symbol
# Prepare the new feature IDs, replace the strand information with letters to match the current pA site clusterID
new.featureID <- anno$Strand %>% as.character %>% replace(. %in% "+", "F") %>% replace(. %in% "-", "R") %>% paste0(as.character(anno$repID), .) - 3.3단계에서 생성된 읽기 횟수와 이전 단계에서 얻은 pA 사이트 정보를 사용하여 DEXSeq 개체를 만듭니다.
# Select the read counts of the condition WT and DKO
countData1<- dplyr::select(countData, SRR1553129.sorted.bam, SRR1553130.sorted.bam, SRR1553131.sorted.bam, SRR1553132.sorted.bam)
# Rename the columns of countData using sample names in sampleTable
colnames(countData1) <- rownames(sampleTable1)
dxd1<-DEXSeqDataSet(countData=countData1,
sampleData=sampleTable1,
design=~sample+exon+condition:exon,
featureID=new.featureID,
groupID=anno$Symbol,
featureRanges=PASinfo) - DEXSeq 객체의 조건 수준을 정의하여 대비 쌍을 정의합니다.
dxd1$condition<- factor(dxd1$condition, levels= c("WT", "DKO"))
# The contrast pair will be "DKO - WT" - 정규화 및 분산 추정. RNA-seq 데이터와 유사하게, 3' 말단 시퀀싱 데이터에 대해 'estimateSizeFactors' 함수를 사용하여 샘플 간 정규화(각 샘플에 대한 비율의 컬럼별 중앙값)를 수행하고 'estimateDispersions' 함수를 사용하여 데이터의 변동을 추정한 다음 'plotDispEsts' 함수를 사용하여 분산 추정 결과를 시각화합니다.
dxd1 %<>% estimateSizeFactors %>% estimateDispersions %T>% plotDispEsts - 'testForDEU' 함수를 사용하여 각 유전자에 대한 차등 pA 부위 사용량 검사를 수행한 다음 'estimateExonFoldChanges' 함수를 사용하여 pA 부위 사용량의 폴드 변화를 추정합니다. 'DEXSeqResults' 기능을 사용하여 결과를 확인하고 유의하게 차이가 있는 pA 부위에 대한 기준으로 'FDR < 10%'를 설정합니다.
dxd1 %<>% testForDEU %>% estimateExonFoldChanges(fitExpToVar = "condition")
dxr1 <- DEXSeqResults(dxd1)
dxr1
mcols(dxr1)$description
table(dxr1$padj<0.1) # Check the number of differential pA sites (FDR < 0.1)
table(tapply(dxr1$padj<0.1, dxr1$groupID, any)) # Check the number of gene overlapped with differential pA site - 'plotDEXSeq' 함수에 의해 생성된 차등 APA 플롯과 'EnhancedVolcano' 함수에 의해 생성된 화산 플롯을 사용하여 차등 pA 사이트 사용 결과를 시각화합니다.
# Select the top 100 significant differential pA sites ranked by FDR
topdiff.PAS<- dxr1%>%as.data.frame%>%rownames_to_column%>%arrange(padj)%$%groupID[1:100]
# Apply plotDEXSeq for the visualization of differential polyA usage
plotDEXSeq(dxr1,"S100a7a", legend=TRUE, expression=FALSE,splicing=TRUE, cex.axis=1.2, cex=1.3,lwd=2)
# Apply perGeneQValue to check the top genes with differential polyA site usage
dxr1%<>% .[!is.na(.$padj), ]
dgene<- data.frame(perGeneQValue= perGeneQValue (dxr1)) %>%rownames_to_column("groupID")
dePAS_sig1<-dxr1%>% data.frame() %>%
dplyr::select(-matches("dispersion|stat|countData|genomicData"))%>%
inner_join(dgene)%>%arrange(perGeneQValue)%>%distinct()%>%
filter(padj<0.1)
# Apply EnhancedVolcano package to visualise differential polyA site usage
"EnhancedVolcano"%>% lapply(library, character.only=TRUE) %>%invisible
EnhancedVolcano(dePAS_sig1, lab=dePAS_sig1$groupID, x='log2fold_DKO_WT',
y='pvalue',title='Volcano Plot',subtitle='DKO vs WT',
FCcutoff=1,labSize=4, legendPosition="right",
caption= bquote(~Log[2]~"Fold change cutoff, 1; FDR 10%"))
- 라이브러리를 로드하고 샘플 테이블을 작성하여 실험 설계를 정의합니다.
- diffSplice 패키지 사용. 보충 R 노트북 파일 "APA_analysis_3PSeq_notebook. Rmd"를 사용하여 이 섹션의 단계별 워크플로를 참조하십시오.
- 차등 pA 사용량 분석에 대한 관심 대비를 정의합니다.
참고: 이 단계는 R 노트북 파일 "APA_analysis_3PSeq_notebook"에 포함된 DGEList 개체를 생성하고 처리한 후에 수행해야 합니다. Rmd".contrast.matrix<-makeContrasts(DKO_vs_WT=DKO-
WT,Ctrl_vs_DKO=Ctrl-DKO,
KD_vs_Ctrl=KD-Ctrl,KD_vs_DKO=KD-DKO,levels=design)
fit2<-fit%>%contrasts.fit(contrast.matrix)%>%eBayes
summary(decideTests(fit2))
ex<-diffSplice(fit2,geneid=anno$Symbol,exonid=new.featureID)
topSplice(ex) #Check the top significant results with topSplice - 'plotSplice' 함수에 의한 미분 APA 플롯과 'EnhancedVolcano' 함수로 화산 플롯을 사용하여 관심 대비 결과(여기서는 "DKO - WT")를 시각화합니다. R 노트북 파일 "APA_analysis_3PSeq_notebook. Rmd" 4.2.7 - 4.2.9 다른 대비 쌍의 시각화를 위해.
sig1<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="t", sort.by="logFC")
sig1.genes<-topSplice(ex,n=Inf,FDR=0.1,coef=1, test="simes")
plotSplice(ex, coef=1,geneid="S100a7a", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Tpm1", FDR = 0.1)
plotSplice(ex,coef=1,geneid="Smc6", FDR = 0.1)
EnhancedVolcano(sig1, lab=sig1$GeneID,xlab= bquote(~Log[2]~'fold change'),
x='logFC', y='P.Value', title='Volcano Plot', subtitle='DKO vs WT',
FCcutoff=1, labSize=6, legendPosition="right")
- 차등 pA 사용량 분석에 대한 관심 대비를 정의합니다.
- DEXSeq 패키지 사용
결과
위의 단계별 워크플로우를 실행한 후 AS 및 APA 분석 출력과 대표 결과는 다음과 같이 생성된 테이블 및 데이터 플롯 형태입니다.
만큼:
AS 분석의 주요 출력(diffSplice에 대한 보충 표 1; DEXSeq)에 대한 표 2는 조건에 따른 차등 용법을 나타내는 엑손의 목록이고, 통계적 유의성에 의해 순위가 매겨진 하나 이상의 구성 엑손의 유의한 전체 스?...
토론
이 연구에서는 대량 RNA-Seq 및 3' 말단 시퀀싱 데이터에서 AS 및 APA를 검출하기 위한 엑손 기반 및 이벤트 기반 접근 방식을 평가했습니다. 엑손 기반 AS 접근법은 차등적으로 발현된 엑손의 목록과 전체 유전자 수준의 차등 스플라이싱 활성의 통계적 유의성에 따라 정렬된 유전자 수준 순위를 모두 생성합니다(표 1-2, 4-5). diffSplice 패키지의 경우, 차등 사용은 엑손 수준에서 가중 선형 모?...
공개
저자는 공개 할 것이 없습니다.
감사의 말
이 연구는 호주 연구위원회 (ARC) 미래 펠로우십 (FT16010043) 및 ANU 선물 계획의 지원을 받았습니다.
자료
| Name | Company | Catalog Number | Comments |
| Not relevent for computational study |
참고문헌
- Katz, Y., Wang, E. T., Airoldi, E. M., Burge, C. B. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods. 7 (12), 1009-1015 (2010).
- Wang, Y., et al. Mechanism of alternative splicing and its regulation. Biomedical Reports. 3 (2), 152-158 (2015).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in Bioinformatics. 21 (6), 2052-2065 (2020).
- Movassat, M., et al. Coupling between alternative polyadenylation and alternative splicing is limited to terminal introns. RNA Biology. 13 (7), 646-655 (2016).
- Tian, B., Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature Reviews Molecular Cell Biology. 18 (1), 18-30 (2017).
- Herrmann, C. J., et al. PolyASite 2.0: a consolidated atlas of polyadenylation sites from 3' end sequencing. Nucleic Acids Research. 48 (1), 174-179 (2020).
- Liu, R., Loraine, A. E., Dickerson, J. A. Comparisons of computational methods for differential alternative splicing detection using RNA-seq in plant systems. BMC Bioinformatics. 15 (1), 364 (2014).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (1), 13 (2016).
- Anders, S., Reyes, A., Huber, W. Detecting differential usage of exons from RNA-seq data. Genome Research. 22 (10), 2008-2017 (2012).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2014).
- Shen, S., et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data. Proceedings of the National Academy of Sciences. 111 (51), 5593-5601 (2014).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in bioinformatics. 21 (6), 2052-2065 (2020).
- Kanitz, A., et al. Comparative assessment of methods for the computational inference of transcript isoform abundance from RNA-seq data. Genome biology. 16 (1), 1-26 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Sznajder, L. J., et al. Loss of MBNL1 induces RNA misprocessing in the thymus and peripheral blood. Nature Communications. 11, 1-11 (2020).
- Batra, R., et al. Loss of MBNL leads to disruption of developmentally regulated alternative polyadenylation in RNA-mediated disease. Molecular Cell. 56 (2), 311-322 (2014).
- Leinonen, R., Sugawara, H., Shumway, M., et al. The sequence read archive. Nucleic acids research. 39, 19-21 (2010).
- Tange, O. . GNU parallel-the command-line power tool. 36, 42-47 (2011).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), 25 (2010).
- Veiga, D. F. T. maser: Mapping Alternative Splicing Events to pRoteins. R package version 1.4.0. , (2019).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (13), 25 (2009).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ramírez, F., Dündar, F., Diehl, S., Grüning, B. A., Manke, T. deepTools: a flexible platform for exploring deep-sequencing data. Nucleic acids research. 42 (1), 187-191 (2014).
- Merino, G. A., Conesa, A., Fernández, E. A. A benchmarking of workflows for detecting differential splicing and differential expression at isoform level in human RNA-seq studies. Briefings in bioinformatics. 20 (2), 471-481 (2019).
- Chhangawala, S., Rudy, G., Mason, C. E., Rosenfeld, J. A. The impact of read length on quantification of differentially expressed genes and splice junction detection. Genome biology. 16 (1), 1-10 (2015).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biol. 17, 13 (2016).
- Trapnell, C., et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 7 (3), 562-578 (2012).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 12, 323 (2011).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat Biotechnol. 34 (5), 525-527 (2016).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유