Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Reconstitución de la actividad de extracción de Msp1 con componentes totalmente purificados
En este artículo
Resumen
Aquí, presentamos un protocolo detallado para la reconstitución de la actividad de extracción de Msp1 con componentes totalmente purificados en proteoliposomas definidos.
Resumen
Como centro de fosforilación oxidativa y regulación apoptótica, las mitocondrias desempeñan un papel vital en la salud humana. La función mitocondrial adecuada depende de un sistema de control de calidad robusto para mantener la homeostasis de proteínas (proteostasis). Las disminuciones en la proteostasis mitocondrial se han relacionado con el cáncer, el envejecimiento, la neurodegeneración y muchas otras enfermedades. Msp1 es una AAA+ ATPasa anclada en la membrana mitocondrial externa que mantiene la proteostasis mediante la eliminación de proteínas mal localizadas ancladas en la cola. Utilizando componentes purificados reconstituidos en proteoliposomas, hemos demostrado que Msp1 es necesario y suficiente para extraer una proteína modelo anclada a la cola de una bicapa lipídica. Nuestro sistema reconstituido simplificado supera varias de las barreras técnicas que han obstaculizado el estudio detallado de la extracción de proteínas de membrana. Aquí, proporcionamos métodos detallados para la generación de liposomas, la reconstitución de proteínas de membrana y el ensayo de extracción Msp1.
Introducción
La función celular adecuada depende de un proceso llamado proteostasis, que asegura que las proteínas funcionales estén en la concentración y ubicación celularcorrectas 1. Las fallas en la proteostasis conducen a una función orgánulo comprometida y se asocian con muchas enfermedades neurodegenerativas2,3,4. Las proteínas de membrana presentan desafíos únicos para la red de proteostasis, ya que deben dirigirse a la membrana correcta, evitando al mismo tiempo la agregación de los dominios transmembrana hidrofóbicos (TMD)5. En consecuencia, la maquinaria especializada ha evolucionado para proteger el TMD hidrofóbico del citosol y facilitar la orientación y la inserción en la membrana celular adecuada6,7,8,9,10,11,12,13,14,15.
Las mitocondrias son el centro metabólico de la célula y están involucradas en numerosos procesos celulares esenciales como: fosforilación oxidativa, generación de cúmulos de hierro-azufre y regulación apoptótica16,17. Estos orgánulos endosimbióticos contienen dos membranas, conocidas como la membrana mitocondrial interna (IMM) y la membrana mitocondrial externa (OMM). Más del 99% de las 1.500 proteínas mitocondriales humanas están codificadas en el genoma nuclear y necesitan ser translocadas a través de una o dos membranas diferentes18,19. Por lo tanto, la función mitocondrial adecuada depende de una red de proteostasis robusta para corregir cualquier error en la orientación o translocación de proteínas.
Nuestro laboratorio se centra en un subconjunto de proteínas de membrana mitocondrial llamadas proteínas ancladas en la cola (TA), que tienen un solo dominio transmembrana en el extremo C20,21,22,23,24. Las proteínas TA están involucradas en una serie de procesos esenciales, como la apoptosis, el transporte de vesículas y la translocación de proteínas25. La topología única de las proteínas TA requiere la inserción post-traduccional, que ocurre en el retículo endoplásmico (ER) por la entrada guiada de las vías del complejo proteico de membrana (EMC) anclada en la cola (GET) o en el retículo endoplásmico (EMC) o en el OMM por una vía mal caracterizada20,26,27,28. Las propiedades biofísicas del TMD son necesarias y suficientes para guiar las proteínas TA a la membrana correcta29. El reconocimiento de características biofísicas en lugar de un motivo de secuencia definido limita la fidelidad de las vías de orientación5. Por lo tanto, la localización errónea de las proteínas TA es un estrés común para las redes de proteostasis. El estrés celular, como la inhibición de la vía GET, causa un aumento en la mala localización de proteínas a la OMM y disfunción mitocondrial a menos que estas proteínas se eliminen rápidamente30,31.
Un tema común en la proteostasis de membrana es el uso de proteínas AAA+(ATPasa Associada con calidades Acelulares) para eliminar proteínas viejas, dañadas o mal localizadas de la bicapa lipídica1,32,33,34,35,36,37,38 . Las proteínas AAA+ son motores moleculares que forman anillos hexaméricos y sufren movimientos dependientes de ATP para remodelar un sustrato, a menudo por translocación a través de un estrecho poro axial39,40. Aunque se ha dedicado un gran esfuerzo a estudiar la extracción de proteínas de membrana por AAA+ ATPasas, las reconstituciones son complejas o implican una mezcla de lípidos y detergente41,42,lo que limita el poder experimental para examinar el mecanismo de extracción del sustrato a partir de la bicapa lipídica.
Msp1 es una AAA+ ATPasa altamente conservada anclada en la OMM y los peroxisomas que desempeña un papel crítico en la proteostasis de membrana al eliminar las proteínas TA mal localizadas43,44,45,46,47. Msp1 también se demostró recientemente que alivia el estrés de importación de proteínas mitocondriales al eliminar las proteínas de membrana que se estancan durante la translocación a través de la OMM48. La pérdida de Msp1 o el homólogo humano ATAD1 resulta en fragmentación mitocondrial, fallas en la fosforilación oxidativa, convulsiones, aumento de lesiones después de un accidente cerebrovascular y muerte prematura31,49,50,51,52,53,54,55,56.
Hemos demostrado que es posible co-reconstituir proteínas TA con Msp1 y detectar la extracción de la bicapa lipídica57. Este sistema simplificado utiliza proteínas totalmente purificadas reconstituidas en liposomas definidos que imitan el OMM(Figura 1)58,59. Este nivel de control experimental puede abordar cuestiones mecanicistas detalladas de extracción de sustrato que son experimentalmente intratables con reconstituciones más complejas que involucran otras proteínas AAA +. Aquí, proporcionamos protocolos experimentales que detallan nuestros métodos para la preparación de liposomas, la reconstitución de proteínas de membrana y el ensayo de extracción. Esperamos que estos detalles experimentales faciliten un mayor estudio del proceso esencial pero poco comprendido de la proteostasis de la membrana.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Preparación de liposomas
- Combine las reservas de cloroformo de lípidos en proporciones apropiadas para imitar la membrana mitocondrial externa.
- Preparar 25 mg de mezcla lipídica. Utilizamos una mezcla previamente establecida de lípidos que imitan las membranas mitocondriales, que consiste en una proporción molar de 48:28:10:10:4 de fosfatidilcolina de huevo de gallina (PC), fosfatidiletanolamina (PE) de huevo de gallina, fosfatidil inositol hepático bovino (PI), sintético 1,2-dioleoil-sn-glicero-3-fosfo-L-serina (DOPS) y sintético 1',3'-bis[1,2-dioleoil-sn-glicero-3-fosfo]-glicerol (TOCL)58,59. Los cálculos de muestra se muestran en la Tabla 1.
- Lleve todas las reservas de lípidos a temperatura ambiente antes de abrirlas, ya que esto limitará la condensación. Como la mayoría de los laboratorios no tienen una forma precisa de medir la concentración de los lípidos, cualquier agua absorbida por el stock de cloroformo cambiará la concentración del stock de lípidos y, por lo tanto, la proporción de lípidos utilizados en el ensayo.
- Como las existencias de lípidos vienen en ampollas de vidrio, transfiera la cantidad requerida de lípidos a un vial de vidrio usando una jeringa de 1 ml. Añadir 2 mg de ditiotreitol (TDT) al vial para prevenir la oxidación lipídica. Trabaje rápidamente ya que la evaporación del cloroformo cambiará la concentración del lípido.
- Transfiera cualquier lípido restante a un vial de vidrio separado y encaje con un septo PFTE. Añadir 2 mg de TDT al vial, envolver con parafilm y conservar a -20 °C para evitar la oxidación lipídica. Trate de usar los lípidos dentro de los 3 meses posteriores a la transferencia a los viales. Para evitar la posible contaminación de las existencias de cloroformo por escorrentía de marcadores, transfiera las pegatinas de las ampollas originales a los viales de vidrio en lugar de etiquetarlas con marcador.
- Evapore el cloroformo bajo una corriente muy suave de nitrógeno mientras gira el vial de vidrio continuamente a mano en una campana de humos, actuando esencialmente como una rotovap manual. Gire el vial a una velocidad constante (20-40 rpm) a mano para mantener los lípidos en movimiento. El objetivo es evaporar todo el cloroformo y obtener una capa uniforme de lípidos sobre todo el vial de vidrio.
PRECAUCIÓN: El cloroformo es neurotóxico, y este paso debe realizarse en la campana de humos.- Conecte una pipeta Pasteur fresca al tubo de nitrógeno. No deje que ninguno de los lípidos salpique del vial o de la pipeta Pasteur. Apunte la punta a la parte inferior del vial para que el aire rebote en la parte inferior y empuje los lípidos hacia el centro del vial. Obtenga un recubrimiento uniforme sobre todo el vial mientras evita cualquier acumulación en las esquinas o por la tapa. Todo este proceso dura unos 5 minutos.
- A medida que la mezcla se espesa en una "cuenta" de lípidos, guíala hacia el centro del vial cambiando el ángulo del vial. Una vez que la cuenta comience a hacerse más pequeña, suba ligeramente la corriente de nitrógeno para dispersar la cuenta, asegurándose de que ninguno de los lípidos salga del vial.
- Retire el cloroformo restante al vacío.
- Coloque el vial de vidrio en una bomba de vacío o de diafragma de la casa durante 1 hora para eliminar la mayoría del cloroformo residual. Estos vacíos generalmente no son lo suficientemente fuertes como para eliminar todo el cloroformo, pero pueden tolerar pequeñas cantidades de disolvente mejor que los vacíos de vano rotativo.
- Coloque el vial en un vacío fuerte (<1 mTorr) durante 12-16 h para eliminar todo el cloroformo residual. Asegúrese de evitar golpes en el vial durante este proceso.
- Resuspend los lípidos en 1,25 mL de tampón liposómico (50 mM HEPES KOH pH 7,5, 15% glicerol, 1 mM TDT). Como comenzamos con 25 mg de lípidos, esto resulta en una concentración de 20 mg / ml. Resuspenda completamente los lípidos sin trozos visibles. Si los lípidos se agrupan en la esquina del vial, este puede ser un proceso largo.
- Vórtice el vial vigorosamente hasta que la muestra esté suave como la leche. Encontramos que si la evaporación del cloroformo se realizó correctamente la noche anterior, este proceso toma alrededor de 5-10 minutos.
- Para garantizar la resuspensión completa de los lípidos, gire sobre una rueda a temperatura ambiente durante 3 horas a ~ 80 rpm. Retire el vial de la rueda una vez cada hora durante 1 minuto de vórtice para garantizar una mezcla uniforme.
- Transfiera los lípidos cuidadosamente a un tubo de microcentrífuga limpio de 1,5 ml. Realice 5 ciclos de congelación-descongelación utilizando nitrógeno líquido para congelar y un bloque de calor de 30 °C para descongelar. Este paso ayuda a convertir las vesículas multilamelares en vesículas unilamelares.
- Extruir los lípidos.
- Durante los ciclos de descongelación por congelación, prepare la miniextrusora. Ensamble la miniextrusora con soportes de filtro de 10 mm y una membrana de policarbonato del tamaño de poro deseado (utilizamos 200 nm). El tamaño del filtro afectará el tamaño del liposoma, lo que afectará la concentración de proteínas requeridas para la reconstitución (Paso 2.3).
- Coloque la miniextrusora sobre una placa caliente y lleve la temperatura de la extrusora hasta 60 °C.
- Extraiga los lípidos en una jeringa de vidrio hermética al gas de 1 ml y colóquelos cuidadosamente en un extremo de la miniextrusora. Coloque la jeringa hermética al gas vacía en el otro lado de la miniextrusora. Deje que los lípidos se equilibren a la temperatura del soporte del extrusor durante 5-10 minutos.
- Transfiera los lípidos a la jeringa alternativa empujando suavemente el émbolo de la jeringa llena. Empuje la solución de la jeringa alternativa hacia la jeringa original. Repita este proceso de ida y vuelta 15 veces, de modo que al 15paso los lípidos terminen en la jeringa alternativa. Controle el volumen en cada pasada para asegurarse de que no haya fugas.
- Preparar alícuotas de un solo uso de los lípidos, congelar flash en nitrógeno líquido y almacenar a -80 °C. Los liposomas son estables a -80 °C durante varios meses. Las reconstituciones requieren 10 μL de liposomas a la vez (Paso 2.3.4), por lo que es conveniente preparar alícuotas de 10 μL o 20 μL.
2 Reconstitución de msp1 y proteína modelo TA
- Preparar el tampón de reconstitución: 50 mM HEPES pH 7.5, 200 mM acetato de potasio, 7 mM acetato de magnesio, 2 mM TDT, 10% sacarosa, 0.01% azida de sodio, 0.2-0.8% Deoxy Big Chaps (DBC).
- Optimizar las condiciones de reconstitución para el nuevo lote de liposomas. La concentración de DBC y bioperlas requeridas para una reconstitución óptima varía según el lote de liposomas utilizados. Para limitar la variabilidad de la preparación a la preparación, use el mismo lote de DBC para todos los experimentos. Cuando cambie muchos DBC, repita el proceso de optimización.
- Establezca una serie de reconstituciones con diferentes concentraciones de DBC (0.2% - 0.8%) y bioperlas (25 mg - 100 mg) cada vez que se prepare un nuevo lote de liposomas. Es importante no dejar caer el DBC por debajo de la concentración crítica de micelas (CMC) de ~ 0.12%. Una vez que se optimizan las condiciones, recomendamos recopilar todos los datos utilizando las mismas condiciones de preparación y reconstitución de liposomas.
- Evaluar la eficacia de las diversas condiciones de reconstitución mediante el ensayo de extracción descrito en el Paso 3.
- Preparar bioperlas a una concentración final de 250 mg/ml.
- Pesar 2,7 g de bioperlas secas y resuspend en un tubo centrífugo de 50 ml de metanol al 100% (aproximadamente 45 ml) para humedecer las perlas. Inicialmente humedezca las bioperlas en metanol para evitar que el aire quede atrapado en los poros de las perlas. Una vez en metanol, mantenga las bioperlas húmedas, ya que cualquier aire atrapado por las bioperlas alterará su capacidad para absorber el detergente.
- Retire el metanol lavando las perlas 8x con aproximadamente 45 ml de agua ultrapura (18,2 mΩ), en lo sucesivo denominada ddH2O. Perlas de pellets girando a 3.200 x g durante 1 minuto. Decantar el líquido y resuspend en ddH2O.
- Después del lavado, resuspendir en 10 ml de ddH2O con azida de sodio al 0,02% y conservar a 4 °C. Las bioperlas se pueden almacenar a 4 °C durante varios meses. Este caldo es de 250 mg / ml, ya que se supone que se pierden ~ 0.2 g durante los pasos de lavado.
- Calcular el tamaño del liposoma y el número deseado de moléculas de proteína TA y Msp1 por liposoma. Esto determinará la concentración de proteína Msp1 y TA requerida para la reconstitución.
- Primero, calcule el número de moléculas lipídicas por liposoma unilamelar(Ntotal)utilizando la ecuación
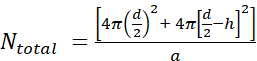 donde d es el diámetro del liposoma, h es el grosor de la bicapa y a es el área del grupo de cabeza lipídico.
donde d es el diámetro del liposoma, h es el grosor de la bicapa y a es el área del grupo de cabeza lipídico.- Mida el diámetro del liposoma por DLS. En nuestro ejemplo, se obtuvo un valor de 70 nm para el diámetro del liposoma (d).
- Utilice un valor de 5 nm para h y 0,71 nm2 para a, que es el tamaño del grupo de cabeza para la fosfatidilcolina. En esta situación particular, Ntotal es de 37.610.
- A continuación, calcule la concentración molar de lípido mlípido utilizando el peso molecular promedio de los lípidos en la mezcla. En este ejemplo, la concentración de lípidos es de 20 mg/ml (Paso 1.4) y el peso molecular promedio de los lípidos es de 810 g/mol (Tabla 1). Esto da como resultado un valor de 0,0247 M para MLipid.
- A continuación, calcule la concentración molar de liposomas, MLiposoma,utilizando la ecuación
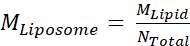 donde M Lipid es la concentración molar de lípidos del Paso 2.3.2,y Ntotal es el número total de lípidos por liposoma calculado en el Paso 2.3.1. En este ejemplo, la concentración de 20 mg/ml de liposomas es de aproximadamente 660 nM.
donde M Lipid es la concentración molar de lípidos del Paso 2.3.2,y Ntotal es el número total de lípidos por liposoma calculado en el Paso 2.3.1. En este ejemplo, la concentración de 20 mg/ml de liposomas es de aproximadamente 660 nM. - Calcule la cantidad de proteína Msp1 y TA requerida para una reacción de reconstitución de 100 μL.
NOTA: La concentración final de lípidos en la reconstitución es de 2 mg/ml, que es una dilución 10x del stock de liposomas. Esto da una concentración final de liposomas de 66 nM. La concentración final de Msp1 es de 792 nM, lo que da un promedio de 12 copias totales (2 hexámeros funcionales) por liposoma. La concentración final de proteína TA es de 660 nM, lo que da un promedio de 10 copias por liposoma.
- Primero, calcule el número de moléculas lipídicas por liposoma unilamelar(Ntotal)utilizando la ecuación
- En un tubo de PCR, mezcle Msp1 purificado, proteína TA y liposomas en tampón de reconstitución. El orden de adición es tampón, proteínas y liposomas últimos. El volumen total es de 100 μL. Deje que la mezcla repose sobre hielo durante 10 minutos. Purificar la proteína Msp1 y TA como se describió anteriormente57.
- Utilice la proteína modelo TA bien caracterizada His-Flag-Sumo-Sec22 como sustrato de control positivo al establecer por primera vez el ensayo. Esta construcción tiene una etiqueta His para una fácil purificación, una etiqueta 3x-Flag para la detección por western blot, un dominio Sumo para una mayor solubilidad y el TMD de la proteína ER-TA Sec22 para la reconstitución y el reconocimiento por Msp1.
- Asegúrese de que las soluciones madre de Msp1 y la proteína TA sean de aproximadamente 100 μM para minimizar el efecto de N-dodecil β-D-maltósido (DDM) de la purificación de proteínas en la reconstitución. Msp1 purificado está en 20 mM HEPES pH 7.5, 100 mM cloruro de sodio, 0.1 mM Tris(2-carboxietilo)clorhidrato de fosfina (TCEP), 0.05% DDM mientras que la proteína TA purificada está en 50 mM Tris pH 7.4, 150 mM cloruro de sodio, 10 mM cloruro de magnesio, 5 mM β-mercaptoetanol, 10% glicerol, 0.1% DDM. Las soluciones madre de aproximadamente 100 μM de proteína TA y monómero Msp1 aseguran que estos componentes constituirán < 5% del volumen de reconstitución final, lo que resulta en una dilución de DDM por debajo del CMC.
- Agregue la cantidad deseada de bioperlas a la muestra para eliminar el detergente.
- Corte la punta de una punta de pipeta p200 a aproximadamente1/8 de pulgada de diámetro para que las cuentas puedan caber a través de la punta. Vórtice el tubo de bioperlas a fondo para obtener una mezcla uniforme y retire rápidamente la tapa y la pipeta hacia arriba del volumen antes de que las bioperlas se asienten. Transfiera las bioperlas a un tubo de PCR vacío.
- Cuando la reconstitución haya terminado su incubación de 10 minutos sobre hielo, use una punta de pipeta sin cortar para eliminar todo el líquido de las bioperlas. Luego transfiera la reconstitución de 100 μL al tubo con las bioperlas. Esto debe hacerse rápidamente para que las bioperlas no atrapen el aire, lo que hará que las perlas floten.
- Permita que la reconstitución gire sobre una rueda a ~80 rpm durante 16 horas a 4 °C.
- Retire el material reconstituido de las bioperlas. Haga un giro rápido en un picofuge para peletizar las bioperlas, y luego use una punta de pipeta sin cortar para transferir el material reconstituido a un tubo de PCR limpio. Repita este proceso 1-2 veces hasta que no queden bioperlas en la muestra. Mantenga la reconstitución en hielo.
- Limpie previamente el material reconstituido para eliminar cualquier proteína que no haya podido reconstituirse en los liposomas.
- Prepare el tampón de extracción: 50 mM HEPES pH 7.5, 200 mM de acetato de potasio, 7 mM de acetato de magnesio, 2 mM de TDT, 100 nM de cloruro de calcio.
- Equilibre las columnas de espín de glutatión con Extraction Buffer de acuerdo con las instrucciones del fabricante. Esto generalmente implica 3 rondas de lavado con 400 μL de tampón y luego centrifugar a 700 x g durante 2 minutos a temperatura ambiente para eliminar el tampón.
- Añadir 5 μM de cada chaperona (GST-SGTA y GST-Calmodulina) al material reconstituido. Estas chaperonas se unirán al TMD de cualquier proteína que no se haya reconstituido en los liposomas. La purificación de chaperonas fue descrita anteriormente6,57.
NOTA: Estas chaperonas están disponibles comercialmente, pero preferimos purificarlas en casa para controlar el costo y la calidad. Ambas proteínas están en 20 mM Tris pH 7.5, 100 mM de cloruro de sodio y 0.1 mM TCEP, y tienen una concentración de stock alrededor de 160 μM. SGTA reconoce sustratos con un TMD altamente hidrófobo mientras que Calmodulin se une a TMD con hidrofobicidad moderada6,29. Juntos, este cóctel chaperone puede reconocer una amplia gama de sustratos. - Añadir 100 μL de tampón de extracción al material reconstituido, elevando el volumen hasta 200 μL. Agregue esto a las columnas de espín de glutatión equilibradas. Tenga en cuenta que las columnas de espín de glutatión proporcionan la mayor recuperación de la muestra cuando el volumen de pre-compensación es de 200 a 400 μL.
- Enchufe las columnas de centrifugado y gire a ~80 rpm a 4 °C durante 30 minutos para permitir que las chaperonas se unan a la resina.
- Gire las columnas a 700 x g durante 2 minutos a temperatura ambiente. El flujo a través es material pre-limpiado que se agota de proteínas agregadas. Mantenga el material en hielo y proceda directamente con el ensayo de extracción.
3. Ensayo de extracción
- Preparar tubos para el análisis SDS PAGE. Cada reacción tendrá 4 tubos: INPUT (I), FLOW THROUGH (FT), WASH (W) y ELUTE (E).
- Añadir 45 μL de muestra de ddH2O al tubo INPUT, 40 μL de ddH2O al tubo FLOW THROUGH y 0 μL a los tubos WASH y ELUTE.
NOTA: La mejor relación señal/ruido en este ensayo se obtiene cuando las muestras WASH y ELUTE se concentran 5 veces en relación con las muestras INPUT y FLOW THROUGH. Debido a las diluciones durante el ensayo, esto requiere tomar 5 μL de muestra para la muestra INPUT, 10 μL de muestra para la muestra FLOW THROUGH y 50 μL de muestra para las muestras WASH y ELUTE. - Agregue 16.6 μL de 4x SDS PAGE Loading Buffer a cada tubo. El volumen total de cada muestra es de 50 μL antes del Búfer de carga SDS PAGE. El volumen final es de 66,6 μL (muestra de 50 μL + 16,6 μL de 4x SDS PAGE Loading Buffer).
- Añadir 45 μL de muestra de ddH2O al tubo INPUT, 40 μL de ddH2O al tubo FLOW THROUGH y 0 μL a los tubos WASH y ELUTE.
- Montar el ensayo de extracción.
- Preparar la reacción de extracción que contenga 60 μL de proteoliposomas pre-aclarados, 5 μM de GST-SGTA, 5 μM de GST-Calmodulina y 2 mM de ATP. Combine todos los reactivos excepto el ATP, que se utiliza para iniciar la reacción. Llevar a un volumen final de 200 μL con Buffer de extracción.
NOTA: Como se utilizan 60 μL de muestra para cada ensayo de extracción, se puede utilizar una reconstitución para tres ensayos de extracción diferentes. Realizar controles positivos y negativos (+ATP y -ATP) sobre material de la misma reconstitución. - Ensayo de extracción precaliente en bloque térmico a 30 °C durante 2 minutos.
- Preparar la reacción de extracción que contenga 60 μL de proteoliposomas pre-aclarados, 5 μM de GST-SGTA, 5 μM de GST-Calmodulina y 2 mM de ATP. Combine todos los reactivos excepto el ATP, que se utiliza para iniciar la reacción. Llevar a un volumen final de 200 μL con Buffer de extracción.
- Inicie el ensayo de extracción agregando ATP a la concentración final de 2 mM y al temporizador de inicio.
- Dé un giro de 5 segundos en un picofuge para mezclar ATP en la reacción. Incubar la reacción a 30 °C durante 30 minutos.
- Durante la incubación, tome 5 μL de la reacción y agréguela al tubo de ENTRADA. El momento de esto es flexible.
- Durante este período de incubación, equilibre una columna de espín de glutatión para cada muestra en el ensayo de extracción.
- Realizar pull down sobre chaperonas para aislar el material extraído.
- Una vez finalizada la incubación de 30 minutos, agregue 200 μl de tampón de extracción al tubo para llevar el volumen total a 400 μL. Añadir a la resina de glutatión equilibrada y dejar unir en la rueda a 4° C durante 30 minutos.
- Gire las columnas a 700 x g durante 2 minutos a temperatura ambiente para recoger el flujo. Tome 10 μL para el tubo FLOW THROUGH. Esta muestra contiene sustratos que todavía están integrados en la bicapa lipídica.
- Lave la resina dos veces con 400 μL de tampón de extracción, descartando el flujo. En el tercer lavado, mantenga el flujo y tome 50 μL para el tubo WASH.
- Prepare 5 ml de tampón de elución agregando glutatión reducido a una concentración final de 5 mM en el tampón de extracción. Prepare este búfer fresco cada vez.
- Agregue 200 μL de búfer de elución a la columna de centrifugado. Incubar a temperatura ambiente durante 5 minutos. Girar a 700 x g durante 2 minutos a temperatura ambiente para eluir. Mantenga el flujo a través. Repita el proceso una segunda vez para que el volumen total de elución sea de 400 μL.
- Tome 50 μL de muestra de la muestra de Elution y agréguela al tubo ELUTE.
- Analice la actividad de extracción utilizando SDS-PAGE y western blot.
NOTA: Como un western blot es un procedimiento bastante estándar, se proporciona un protocolo básico que destaca algunos de los detalles únicos de este ensayo.- Cargue las muestras en un gel de poliacrilamida sin manchas (4% de apilamiento, 15% de separación) y corra a 200 V durante 50 minutos en el tampón Tris-glycine. Si el espacio lo permite, use una escalera sin manchas y una escalera manchada para permitir la visualización en un generador de imágenes de gel sin manchas y la transferencia a la membrana de PVDF, respectivamente. El gel libre de manchas permite la visualización cuantitativa de las proteínas que contienen triptófano tras la activación con luz ultravioleta, al tiempo que permite que el gel se use para un western blot.
- Imagine el gel sin manchas para confirmar que hay una carga igual en todas las muestras. Este es un control esencial que garantiza que cualquier cambio en la señal detectada por Western Blot no sea el resultado de una carga variable de proteínas. Solo debe haber bandas visibles para las chaperonas (GST-calmodulina y GST-SGTA) en las muestras INPUT y ELUTION. Recordemos que la muestra ELUTE estará más concentrada que la muestra INPUT.
- Montar un cassette western blot utilizando una membrana de PVDF de 45 μm. Transfiera a una corriente constante de 300 mA durante 60 minutos.
- Después de bloquear la membrana, únase al anticuerpo primario durante 16 h a 4 ° C con una agitación suave ~ 15 rpm. Blot para sustrato con conejo Anti-FLAG en una dilución 1:1.000.
NOTA: Los anticuerpos primarios y secundarios utilizados serán específicos del sustrato y es posible que sea necesario optimizar la concentración para su uso. - Lavar la membrana e incubar con anticuerpo secundario, anti-conejo de cabra a una dilución de 1:10.000, con agitación suave durante 1 hora a temperatura ambiente.
- Lavar membrana e imagen para análisis utilizando el agente de detección western blotting.
Access restricted. Please log in or start a trial to view this content.
Resultados
Para interpretar correctamente los resultados, el gel libre de manchas y la mancha occidental deben verse juntos. El gel sin manchas garantiza una carga igual en todas las muestras. Al ver el gel libre de manchas, las chaperonas (GST-calmodulin y GST-SGTA) serán visibles en los carriles INPUT (I) y ELUTE (E). Verifique que la intensidad de estas bandas sea uniforme en todas las muestras input. Del mismo modo, asegúrese de que la intensidad sea uniforme en todas las muestras eluticas. El ELUTE es 5 veces más concentrad...
Access restricted. Please log in or start a trial to view this content.
Discusión
La función mitocondrial adecuada depende de un sistema robusto de control de calidad de proteínas. Debido a los límites inherentes en la fidelidad de las vías de orientación de la proteína TA, las proteínas TA mal localizadas son una fuente constante de estrés para las mitocondrias. Un componente clave de la red de proteostasis mitocondrial es Msp1, que es una AAA+ ATPasa anclada a la membrana que elimina las proteínas TA mal localizadas de la OMM. Aquí, hemos descrito cómo preparar proteoliposomas, co-reconst...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Ninguno
Agradecimientos
MLW desarrolló parte de este protocolo durante sus estudios postdoctorales con el Dr. Robert Keenan en la Universidad de Chicago.
Este trabajo está financiado por la subvención 1R35GM137904-01 de los NIH a MLW.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Biobeads | Bio-Rad | 1523920 | |
| Bovine liver phosphatidyl inositol | Avanti | 840042C | PI |
| Chicken egg phosphatidyl choline | Avanti | 840051C | PC |
| Chicken egg phosphatidyl ethanolamine | Avanti | 840021C | PE |
| ECL Select western blotting detection reagent | GE | RPN2235 | |
| Filter supports | Avanti | 610014 | |
| Glass vial | VWR | 60910L-1 | |
| Glutathione spin column | Thermo Fisher | PI16103 | |
| Goat anti-rabbit | Thermo Fisher | NC1050917 | |
| Mini-Extruder | Avanti | 610020 | |
| Polycarbonate membrane | Avanti | 610006 | 200 nM |
| PVDF membrane | Thermo Fisher | 88518 | 45 µM |
| Rabbit anti-FLAG | Sigma-Aldrich | F7245 | |
| Synthetic 1,2-dioleoyl-sn-glycero-3-phospho-L-serine | Avanti | 840035C | DOPS |
| Synthetic 1',3'-bis[1,2-dioleoyl-sn-glycero-3-phospho]-glycerol | Avanti | 710335C | TOCL |
| Syringe, 1 mL | Norm-Ject | 53548-001 | |
| Syringe, 1 mL, gas-tight | Avanti | 610017 |
Referencias
- Song, J., Herrmann, J. M., Becker, T. Quality control of the mitochondrial proteome. Nature Reviews Molecular Cell Biology. 22, 54-70 (2021).
- Phillips, B. P., Miller, E. A. Membrane protein folding and quality control. Current Opinion in Structural Biology. 69, 50-54 (2021).
- Jiang, H. Quality control pathways of tail-anchored proteins. Biochimica et Biophysica Acta - Molecular Cell Research. 1868, 118922(2020).
- McKenna, M. J., et al. The endoplasmic reticulum P5A-ATPase is a transmembrane helix dislocase. Science. 369, New York, N.Y. (2020).
- Hegde, R. S., Zavodszky, E. Recognition and Degradation of Mislocalized Proteins in Health and Disease. Cold Spring Harbor Perspectives in Biology. 11, 033902(2019).
- Shao, S., Hegde, R. S. A calmodulin-dependent translocation pathway for small secretory proteins. Cell. 147, 1576-1588 (2011).
- Samuelson, J. C., et al. YidC mediates membrane protein insertion in bacteria. Nature. 406, 637-641 (2000).
- Anghel, S. A., McGilvray, P. T., Hegde, R. S., Keenan, R. J. Identification of Oxa1 Homologs Operating in the Eukaryotic Endoplasmic Reticulum. Cell Reports. 21, 3708-3716 (2017).
- Aviram, N., et al. The SND proteins constitute an alternative targeting route to the endoplasmic reticulum. Nature. 540, 134-138 (2016).
- Voorhees, R. M., Hegde, R. S. Structure of the Sec61 channel opened by a signal sequence. Science. 351, New York, NY. 88-91 (2016).
- Cichocki, B. A., Krumpe, K., Vitali, D. G., Rapaport, D. Pex19 is involved in importing dually targeted tail-anchored proteins to both mitochondria and peroxisomes. Traffic. 19, Copenhagen, Denmark. 770-785 (2018).
- Mateja, A., et al. Protein targeting. Structure of the Get3 targeting factor in complex with its membrane protein cargo. Science. 347, New York, NY. 1152-1155 (2015).
- Chacinska, A., Koehler, C. M., Milenkovic, D., Lithgow, T., Pfanner, N. Importing mitochondrial proteins: machineries and mechanisms. Cell. 138, 628-644 (2009).
- Chitwood, P. J., Hegde, R. S. An intramembrane chaperone complex facilitates membrane protein biogenesis. Nature. , (2020).
- Chitwood, P. J., Juszkiewicz, S., Guna, A., Shao, S., Hegde, R. S. EMC Is Required to Initiate Accurate Membrane Protein Topogenesis. Cell. 175, 1-30 (2018).
- Bock, F. J., Tait, S. W. G. Mitochondria as multifaceted regulators of cell death. Nature Reviews Molecular Cell Biology. 21, 85-100 (2020).
- Pfanner, N., Warscheid, B., Wiedemann, N. Mitochondrial proteins: from biogenesis to functional networks. Nature Reviews Molecular Cell Biology. 20, (2019).
- Bykov, Y. S., Rapaport, D., Herrmann, J. M., Schuldiner, M. Cytosolic Events in the Biogenesis of Mitochondrial Proteins. Trends in Biochemical Sciences. 45, 650-667 (2020).
- Pfanner, N., Warscheid, B., Wiedemann, N. Mitochondrial proteins: from biogenesis to functional networks. Nature Reviews Molecular Cell Biology. 427, 1135(2019).
- Borgese, N., Coy-Vergara, J., Colombo, S. F., Schwappach, B. The Ways of Tails: the GET Pathway and more. The Protein Journal. , 1-17 (2019).
- Mateja, A., Keenan, R. J. A structural perspective on tail-anchored protein biogenesis by the GET pathway. Current Opinion in Structural Biology. 51, 195-202 (2018).
- Chio, U. S., Cho, H., Shan, S. Mechanisms of Tail-Anchored Membrane Protein Targeting and Insertion. Annual review of cell and developmental biology. 33, 417-438 (2017).
- Denic, V. A portrait of the GET pathway as a surprisingly complicated young man. Trends in biochemical sciences. , (2012).
- Hegde, R. S., Keenan, R. J. Tail-anchored membrane protein insertion into the endoplasmic reticulum. Nature Reviews Molecular Cell Biology. 12, 787-798 (2011).
- Kalbfleisch, T., Cambon, A., Wattenberg, B. W. A bioinformatics approach to identifying tail-anchored proteins in the human genome. Traffic. 8, Copenhagen, Denmark. 1687-1694 (2007).
- Doan, K. N., et al. The Mitochondrial Import Complex MIM Functions as Main Translocase for α-Helical Outer Membrane Proteins. Cell Reports. 31, (2020).
- McDowell, M. A., et al. Structural Basis of Tail-Anchored Membrane Protein Biogenesis by the GET Insertase Complex. Molecular Cell. 80, (2020).
- Guna, A., Volkmar, N., Christianson, J. C., Hegde, R. S. The ER membrane protein complex is a transmembrane domain insertase. Science. 591, New York, NY. 3099(2017).
- Rao, M., et al. Multiple selection filters ensure accurate tail-anchored membrane protein targeting. eLife. 5, 21301(2016).
- Schuldiner, M., et al. The GET complex mediates insertion of tail-anchored proteins into the ER membrane. Cell. 134, 634-645 (2008).
- Chen, Y. -C., et al. Msp1/ATAD1 maintains mitochondrial function by facilitating the degradation of mislocalized tail-anchored proteins. The EMBO journal. 33, 1548-1564 (2014).
- Wu, X., Rapoport, T. A. Translocation of Proteins through a Distorted Lipid Bilayer. Trends in Cell Biology. , (2021).
- Phillips, B. P., Gomez-Navarro, N., Miller, E. A. Protein quality control in the endoplasmic reticulum. Current Opinion in Cell Biology. 65, 96-102 (2020).
- van de Weijer, M. L., et al. Quality Control of ER Membrane Proteins by the RNF185/Membralin Ubiquitin Ligase Complex. Molecular Cell. 79, (2020).
- Weir, N. R., Kamber, R. A., Martenson, J. S., Denic, V. The AAA protein Msp1 mediates clearance of excess tail-anchored proteins from the peroxisomal membrane. eLife. 6, 28507(2017).
- Gardner, B. M., et al. The peroxisomal AAA-ATPase Pex1/Pex6 unfolds substrates by processive threading. Nature communications. 9, 135(2018).
- Puchades, C., et al. Unique Structural Features of the Mitochondrial AAA+ Protease AFG3L2 Reveal the Molecular Basis for Activity in Health and Disease. Molecular Cell. , (2019).
- Castanzo, D. T., LaFrance, B., Martin, A. The AAA+ ATPase Msp1 is a processive protein translocase with robust unfoldase activity. Proceedings of the National Academy of Sciences of the United States of America. 117, 14970-14977 (2020).
- Wang, L., Myasnikov, A., Pan, X., Walter, P. Structure of the AAA protein Msp1 reveals mechanism of mislocalized membrane protein extraction. eLife. 9, (2020).
- Puchades, C., Sandate, C. R., Lander, G. C. The molecular principles governing the activity and functional diversity of AAA+ proteins. Nature Reviews Molecular Cell Biology. , 1-16 (2019).
- Yang, Y., et al. Folding-Degradation Relationship of a Membrane Protein Mediated by the Universally Conserved ATP-Dependent Protease FtsH. Journal of the American Chemical Society. , 10(2018).
- Baldridge, R. D., Rapoport, T. A. Autoubiquitination of the Hrd1 Ligase Triggers Protein Retrotranslocation in ERAD. Cell. 166, 394-407 (2016).
- Fresenius, H. L., Wohlever, M. L. Sorting out how Msp1 maintains mitochondrial membrane proteostasis. Mitochondrion. 49, 128-134 (2019).
- Wang, L., Walter, P. Msp1/ATAD1 in Protein Quality Control and Regulation of Synaptic Activities. Annual Review of Cell and Developmental Biology. 36, 1-24 (2020).
- Dederer, V., et al. Cooperation of mitochondrial and ER factors in quality control of tail-anchored proteins. eLife. 8, 1126(2019).
- Matsumoto, S., et al. Msp1 Clears Mistargeted Proteins by Facilitating Their Transfer from Mitochondria to the ER. Molecular Cell. , (2019).
- Li, L., Zheng, J., Wu, X., Jiang, H. Mitochondrial AAA-ATPase Msp1 detects mislocalized tail-anchored proteins through a dual-recognition mechanism. EMBO Reports. 20, (2019).
- Weidberg, H., Amon, A. MitoCPR - a surveillance pathway that protects mitochondria in response to protein import stress. Science. 360, New York, NY. (2018).
- Okreglak, V., Walter, P. The conserved AAA-ATPase Msp1 confers organelle specificity to tail-anchored proteins. Proceedings of the National Academy of Sciences of the United States of America. 111, (2014).
- Piard, J., et al. A homozygous ATAD1 mutation impairs postsynaptic AMPA receptor trafficking and causes a lethal encephalopathy. Brain. , (2018).
- Zhang, J., et al. The AAA+ ATPase Thorase regulates AMPA receptor-dependent synaptic plasticity and behavior. Cell. 145, 284-299 (2011).
- Prendergast, J., et al. Ganglioside regulation of AMPA receptor trafficking. The Journal of Neuroscience. 34, 13246-13258 (2014).
- Umanah, G. K. E., et al. Thorase variants are associated with defects in glutamatergic neurotransmission that can be rescued by Perampanel. Science Translational Medicine. 9, 4985(2017).
- Pignatelli, M., et al. Synaptic Plasticity onto Dopamine Neurons Shapes Fear Learning. Neuron. 93, 425-440 (2017).
- Zhang, J., et al. The AAA Thorase is neuroprotective against ischemic injury. Journal of Cerebral Blood Flow and Metabolism. , 271678(2018).
- Umanah, G. K. E., et al. AMPA Receptor Surface Expression Is Regulated by S-Nitrosylation of Thorase and Transnitrosylation of NSF. Cell Reports. 33, 108329(2020).
- Wohlever, M. L., Mateja, A., McGilvray, P. T., Day, K. J., Keenan, R. J. Msp1 Is a Membrane Protein Dislocase for Tail-Anchored Proteins. Molecular Cell. 67, 194-202 (2017).
- Lovell, J. F., et al. Membrane binding by tBid initiates an ordered series of events culminating in membrane permeabilization by Bax. Cell. 135, 1074-1084 (2008).
- Leshchiner, E. S., Braun, C. R., Bird, G. H., Walensky, L. D. Direct activation of full-length proapoptotic BAK. Proceedings of the National Academy of Sciences of the United States of America. 110, 986-995 (2013).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados