Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Msp1 Ayıklama Etkinliğinin Tamamen Saflaştırılmış Bileşenlerle Yeniden Uzlaştırılması
Bu Makalede
Özet
Burada, tanımlanmış proteolipozomlarda tamamen saflaştırılmış bileşenlerle Msp1 ekstraksiyon aktivitesinin yeniden uzlaştırılması için ayrıntılı bir protokol sunuyoruz.
Özet
Oksidatif fosforilasyon ve apoptotik regülasyon merkezi olarak mitokondri insan sağlığında hayati bir rol oynar. Uygun mitokondriyal fonksiyon, protein homeostazının (proteostaz) korunması için sağlam bir kalite kontrol sistemine bağlıdır. Mitokondriyal proteostazdaki düşüşler kanser, yaşlanma, nörodejenerasyon ve diğer birçok hastalıkla bağlantılıdır. Msp1, yanlış hesaplanmış kuyruk bağlantılı proteinleri çıkararak proteostaz koruyan dış mitokondriyal membrana tutturulmuş bir AAA+ ATPazdır. Proteolipozomlara yeniden inşa edilen saflaştırılmış bileşenleri kullanarak, Msp1'in bir lipid bilayerden model kuyruk bağlantılı bir protein çıkarmak için gerekli ve yeterli olduğunu gösterdik. Basitleştirilmiş yeniden inşa edilmiş sistemimiz, membran protein ekstraksiyonunun ayrıntılı çalışmasını engelleyen teknik engellerin birkaçını aşıyor. Burada lipozomların üretimi, membran proteini rekonsupsiyonu ve MSP1 ekstraksiyon tahlilleri için detaylı yöntemler sunuyoruz.
Giriş
Doğru hücresel fonksiyon, fonksiyonel proteinlerin doğru konsantrasyonda ve hücresel konumda olmasını sağlayan proteostaz adı verilen bir işleme bağlıdır1. Proteostazdaki başarısızlıklar organel fonksiyonlarının tehlikeye atmasına yol açar ve birçok nörodejeneratifhastalıklailişkilidir 2,3,4. Membran proteinleri, hidrofobik transmembran etki alanlarından (TMD'ler) toplanmaktan kaçınırken doğru zara hedeflenmeleri gerektiğinden proteostaz ağına benzersiz zorluklar sunar5. Sonuç olarak, özel makine hidrofobik TMD'yi sitozodan korumak ve uygun hücresel membran 6 , 7 , 8,9, 10,11,12,13,14,15'ehedeflemeyi ve yerleştirmeyi kolaylaştırmak için evrimleşmiştir.
Mitokondri hücrenin metabolik merkezidir ve oksidatif fosforilasyon, demir-kükürt kümesi üretimi ve apoptotik düzenleme16,17gibi çok sayıda temel hücresel işlemde yer almaktadır. Bu endosimbiyotik organeller, iç mitokondriyal membran (İbb) ve dış mitokondriyal membran (OMM) olarak adlandırılan iki membran içerir. 1.500 insan mitokondriyal proteininin %99'undan fazlası nükleer genomda kodlanmıştır ve bir veya iki farklı zarda yer değiştirmeleri gerekir18,19. Bu nedenle uygun mitokondriyal fonksiyon, protein hedefleme veya translokasyondaki hataları düzeltmek için sağlam bir proteostaz ağına bağlıdır.
Laboratuvarımız, çok C-terminus 20,21,22,23,24'tetek bir transmembranetki alanına sahip kuyruk bağlantılı (TA) proteinleri adı verilen mitokondriyal membran proteinlerinin bir alt kümesine odaklanmaktadır. TA proteinleri, apoptoz, vezikül taşınması ve protein translokasyonu25gibi bir dizi temel işlemde yer almaktadır. TA proteinlerinin benzersiz topolojisi, endoplazmik retikulumda (ER) Kuyruk bağlantılı (GET) veya Endoplazmik retikül Membran protein Kompleksi (EMC) yollarının Rehberli Girişi ile veya kötü karakterize edilmiş bir yol olan 20 , 26 ,27,28ile OMM'ye giren çeviri sonrası ekleme gerektirir. TMD'nin biyofiziksel özellikleri, TA proteinlerini doğru zara yönlendirmek için gerekli ve yeterlidir29. Tanımlanmış bir dizi motifi yerine biyofiziksel özelliklerin tanınması, hedefleme yollarının doğruluğunu sınırlar5. Bu nedenle, TA proteinlerinin yanlış hesaplanması proteostaz ağları için yaygın bir strestir. GET yolunun inhibisyonu gibi hücresel stres, bu proteinler derhal çıkarılmadığı sürece OMM'ye protein yanlış sapkalizasyonunda ve mitokondriyal disfonksiyonda artışa neden olur30,31.
Membran proteostazında ortak bir tema, lipid bilayer 1 , 32, 33 , 34 , 35 ,36,37,38'den eski, hasarlı veya yanlış tanımlanmış proteinleri çıkarmak için AAA+ (hücresel A eğilimleri ile ssociated bir TPase A)proteinlerinin kullanılmasıdır. . AAA+ proteinleri, altıgen halkalar oluşturan ve genellikle dar bir eksenel gözenek yoluyla translokasyon yoluyla bir substratı yeniden şekillendirmek için ATP bağımlı hareketlerinden geçen moleküler motorlardır39,40. AAA+ ATPases tarafından membran proteinlerinin ekstraksiyonunu incelemeye büyük çaba sarf edilmiş olsa da, rekonstrüzyonlar karmaşıktır veya lipid bilayerden substrat ekstraksiyon mekanizmasını incelemek için deneysel gücü sınırlayan lipit ve deterjan41,42karışımını içerir.
Msp1, OMM'ye bağlı yüksek oranda korunmuş bir AAA+ ATPaz ve yanlış tanımlanmış TA proteinleri43 , 44 , 45,46,47'yiçıkararak membran proteostazında kritik rol oynayan peroksiomlardır. Msp1'in ayrıca yakın zamanda OMM48'detranslokasyon sırasında duran membran proteinlerini çıkararak mitokondriyal protein ithalat stresini hafifletdiği gösterilmiştir. Msp1 veya insan homolog ATAD1 kaybı mitokondriyal parçalanma, oksidatif fosforilasyonda başarısızlıklar, nöbetler, inme sonrasında yaralanma artışı ve erken ölüm31,49,50 , 51,52,53,54,55,56ile sonuçlanır.
TA proteinlerini Msp1 ile birlikte yeniden inşa etmenin ve lipid bilayer57'denekstraksiyonu tespit etmenin mümkün olduğunu gösterdik. Bu basitleştirilmiş sistem, OMM'yi taklit eden tanımlanmış lipozomlara yeniden inşa edilmiş tamamen saflaştırılmış proteinler kullanır (Şekil 1)58,59. Bu deneysel kontrol seviyesi, diğer AAA+ proteinlerini içeren daha karmaşık rekonstrüzyonlarla deneysel olarak zor olan substrat ekstraksiyonunun ayrıntılı mekanistik sorularını ele alabilir. Burada, lipozom hazırlama, membran proteini rekonsupsiyonu ve ekstraksiyon tahlil yöntemlerini ayrıntılı olarak açıklayan deneysel protokoller sunuyoruz. Bu deneysel detayların membran proteostazının temel ama iyi anlaşılmayan sürecinin daha fazla incelenmesini kolaylaştıracağını umuyoruz.
Access restricted. Please log in or start a trial to view this content.
Protokol
1. Lipozom Hazırlığı
- Dış mitokondriyal zarı taklit etmek için lipitlerin kloroform stoklarını uygun oranlarda birleştirin.
- 25 mg lipit karışımı hazırlayın. Mitokondriyal membranları taklit eden daha önce kurulmuş bir lipit karışımı kullanıyoruz, 48:28:10:10:4 azı dişi tavuk yumurtası fosfatidyl kolin (PC), tavuk yumurtası fosfatidil etanolamin (PE), sığır karaciğer fosfatidil inositol (PI), sentetik 1,2-dioleoyl-s'den oluşur ve sentetik 1',3'-bis[1,2-dioleoyl-sn-glisero-3-fosfor]-gliserol (TOCL)58,59. Örnek hesaplamalar Tablo 1'de gösterilmiştir.
- Yoğuşma sınırlaması yapacağı için açılmadan önce tüm lipit stoklarını oda sıcaklığına getirin. Çoğu laboratuvarın lipitlerin konsantrasyonunu ölçmek için kesin bir yolu olmadığı için, kloroform stoğu tarafından emilen herhangi bir su lipit stoğunun konsantrasyonunu ve dolayısıyla testte kullanılan lipitlerin oranını değiştirecektir.
- Lipid stokları cam ampullerde geldiğinden, gerekli miktarda lipid 1 mL şırınç kullanarak bir cam şişeye aktarın. Lipid oksidasyonunu önlemek için şişeye 2 mg dithiothreitol (DTT) ekleyin. Kloroformun buharlaşması lipit konsantrasyonunu değiştireceğinden hızlı bir şekilde çalışın.
- Kalan lipidleri ayrı bir cam şişeye aktarın ve PFTE septa ile takin. Şişeye 2 mg DTT ekleyin, parafilm ile sarın ve lipid oksidasyonunu önlemek için -20 °C'de saklayın. Lipitleri şişelere transfer edildiklerinden sonraki 3 ay içinde kullanmaya çalışın. Kloroform stoklarının işaretlenerek potansiyel kirlenmesini önlemek için çıkartmaları işaretleyici ile etiketlemek yerine orijinal ampullerden cam şişelere aktarın.
- Cam şişeyi bir duman kaputunda elle sürekli döndürürken çok nazik bir azot akışı altında kloroform buharlaştırarak, esasen manuel bir rotovap görevi görerek buharlaştırın. Lipitlerin hareket etmesini sağlamak için şişeyi elle tutarlı bir hızda (20-40 rpm) döndürün. Amaç, tüm kloroformu buharlaştırmak ve tüm cam şişenin üzerinde eşit bir lipit kaplaması elde etmektir.
DİkKAT: Kloroform nörotoksiktir ve bu adım duman kaputunda yapılmalıdır.- Azot tüpüne taze bir Pasteur pipet takın. Lipitlerin hiçbirinin şişeden veya Pasteur pipetine sıçramasına izin vermeyin. Ucu şişenin altına nişan alın, böylece hava alttan sıçrar ve lipitleri şişenin ortasına doğru itir. Köşelerde veya kapakta herhangi bir yığılma olmamasını önlerken tüm şişenin üzerinde eşit bir kaplama elde edin. Tüm bu işlem yaklaşık 5 dakika sürer.
- Karışım bir "boncuk" haline gelince, şişenin açısını değiştirerek şişenin ortasına yönlendirin. Boncuk küçülmeye başladığında, besağı dağıtmak için azot akışını hafifçe açın ve lipitlerin hiçbirinin şişeden patlamamasını sağlayın.
- Kalan kloroformları vakum altında çıkarın.
- Kalan kloroformun çoğunu çıkarmak için cam şişeyi bir ev vakumu veya diyafram vakum pompasına 1 saat boyunca koyun. Bu vakumlar genellikle tüm kloroformu kaldıracak kadar güçlü değildir, ancak küçük miktarlarda çözücüyü döner boşuna vakumlardan daha iyi tolere edebilir.
- Tüm kalıntı kloroformları çıkarmak için şişeyi 12-16 saat boyunca güçlü bir vakuma (<1 mTorr) koyun. Bu işlem sırasında şişenin çarpmasını önleyin.
- Lipitleri 1,25 mL lipozom tamponunda yeniden biriktirin (50 mM HEPES KOH pH 7,5, %15 gliserol, 1 mM DTT). 25 mg lipit ile başladığımız gibi, bu 20 mg / mL konsantrasyon ile sonuçlanır. Lipitleri görünür parçalar olmadan tamamen yeniden diriler. Lipitler şişenin köşesinde birikmişse, bu uzun bir işlem olabilir.
- Numune sütlü pürüzsüz olana kadar şişeyi kuvvetlice vorteksleyin. Kloroform buharlaşmasının bir gece önce düzgün bir şekilde yapılması durumunda, bu işlemin yaklaşık 5-10 dakika sürdüğünü görüyoruz.
- Lipitlerin tam olarak yeniden depolanabilmesini sağlamak için oda sıcaklığında bir tekerlek üzerinde ~80 rpm'de 3 saat döndürün. Karıştırmayı bile sağlamak için 1 dakikalık girdap için her saat bir kez şişeyi tekerlekten çıkarın.
- Lipitleri dikkatlice temiz bir 1,5 mL mikrosantrifüj tüpüne aktarın. Donmak için sıvı nitrojen ve çözülmek için 30 °C ısı bloğu kullanarak 5 donma-çözülme döngüsü gerçekleştirin. Bu adım, çokmeller veziklin unilamellar vesicles'e dönüştürülmesine yardımcı olur.
- Lipitleri ekstrüzyona sok.
- Donma çözülme döngüleri sırasında mini ekstrüderi hazırlayın. Mini ekstrüderi 10 mm filtre destekleri ve istenen gözenek boyutunda bir polikarbonat membran ile monte edin (200 nm kullanıyoruz). Filtrenin büyüklüğü lipozom boyutunu etkileyecektir, bu da yeniden yapılanma için gerekli proteinlerin konsantrasyonlarını etkileyecektir (Adım 2.3).
- Mini ekstrüderi sıcak bir tabağa yerleştirin ve ekstrüder sıcaklığını 60 °C'ye kadar getirin.
- Lipitleri 1 mL gaz geçirmez cam şırınnaya çekin ve mini ekstrüderin bir ucuna dikkatlice yerleştirin. Boş gaz geçirmez şırınnayı mini ekstrüderin diğer tarafına yerleştirin. Lipitlerin ekstrüder stand sıcaklığına 5-10 dakika boyunca aşındırmasını bekleyin.
- Dolgulu şırınnanın pistonunun pistonunun hafifçe itilerek lipitleri alternatif şırınna aktarın. Çözeltiyi alternatif şırınnadan orijinal şırınna itin. Bu ileri geri işlemi 15 kez tekrarlayın, böylece15. Sızıntı olmadığından emin olmak için her geçişteki sesi izleyin.
- Lipitlerin tek kullanımlık aliquotlarını hazırlayın, sıvı nitrojende flaş dondurun ve -80 °C'de saklayın. Lipozomlar birkaç ay boyunca -80 ° C'de stabildir. Rekonstallar bir seferde 10 μL lipozom gerektirir (Adım 2.3.4), bu nedenle 10 μL veya 20 μL aliquots hazırlamak uygundur.
2 Msp1 ve Model TA proteininin yeniden uzlaştırılması
- Rekonsiyel tamponunu hazırlayın: 50 mM HEPES pH 7.5, 200 mM potasyum asetat, 7 mM magnezyum asetat, 2 mM DTT, %10 sakkaroz, %0.01 sodyum azit, %0.2-0.8 Deoksi Big Chaps (DBC).
- Yeni lipozom grubu için yeniden yapılandırım koşullarını optimize edin. Optimal rekonstal için gerekli olan DBC ve biyobead konsantrasyonu, kullanılan lipozomların grubuna bağlı olarak değişir. Hazırlığı hazırlık değişkenliğiyle sınırlamak için tüm denemeler için aynı miktarda DBC kullanın. Çok sayıda DBC değiştirirken, iyileştirme işlemini tekrarlayın.
- Her yeni lipozom grubu hazırlandığında farklı DBC (%0,2 - %0,8) ve biyobead (25 mg - 100 mg) konsantrasyonlarında bir dizi yeniden yapılanma ayarlayın. DBC'yi ~%0,12'lik kritik misel konsantrasyonunun (CMC) altına düşürmemek önemlidir. Koşullar optimize edildikten sonra, tüm verilerin aynı lipozom hazırlığı ve yeniden yapılandırım koşullarını kullanarak toplanmış olmanızı öneririz.
- Adım 3'teaçıklanan ekstraksiyon testini kullanarak çeşitli yeniden yapılandırı koşullarının etkinliğini test edin.
- Biyobeadları 250 mg/mL'lik son konsantrasyonda hazırlayın.
- Boncukları ıslatmak için 2,7 g kurutulmuş biyobead tartın ve % 100 metanol (yaklaşık 45 mL) 50 mL santrifüj tüpünde yeniden ıslatın. Başlangıçta, havanın boncukların gözeneklerine sıkışmasını önlemek için biyobeadları metanolde ıslatın. Metanol olduktan sonra, biyobeadlar tarafından sıkışan herhangi bir hava deterjan emme yeteneklerini değiştireceği için biyobeadları ıslak tutun.
- Boncukları yaklaşık 45 mL ultra saf suyla (18,2 mΩ) 8x yıkayarak metanol çıkarın, bundan sonra 1 dakika boyunca3.200x g'da dönerek ddH 2 O. Pelet boncukları olarak adlandırılır. Sıvıyı demle ve ddH2O'da yeniden sulan.
- Yıkadıktan sonra, % 0.02 sodyum azit ile10ml ddH 2 O'da yeniden depolayın ve 4 °C'de saklayın. Biyobeadlar birkaç ay boyunca 4 °C'de saklanabilir. Yıkama adımları sırasında ~0,2 g kaybolduğu varsayılsın diye bu stok 250 mg/mL'dir.
- Lipozom boyutunu ve lipozom başına TA proteini ve Msp1 moleküllerinin istenen sayısını hesaplayın. Bu, yeniden yapılanma için gerekli olan Msp1 ve TA proteininin konsantrasyonunun belirlenmesini sağlayacaktır.
- İlk olarak, d lipozom çapı, h bilayer kalınlığı ve a lipid kafa grubu alanı olduğu denklemi kullanarak teklimeller lipozom başına lipit moleküllerinin sayısını(Ntoplam)
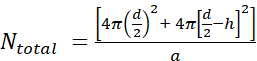 hesaplayın.
hesaplayın. - Lipozom çapını DLS ile ölçün. Örneğimizde lipozom çapı (d) için 70 nm değerinde bir değer elde edildi.
- H için 5 nm ve fosfattidylcholine için kafa grubu boyutu olan biriçin 0,71 nm2 değerini kullanın. Bu özel durumda, Ntoplamı 37.610'dur.
- Daha sonra, karışımdaki lipitlerin ortalama moleküler ağırlığını kullanarak lipid MLipid'in azı dişi konsantrasyonunun hesaplayın. Bu örnekte, lipitlerin konsantrasyonu 20 mg/mL (Adım 1.4) ve lipitlerin ortalama moleküler ağırlığı 810 g/mol'dür( Tablo 1 ). Bu, MLipidiçin 0.0247 M değeri ile sonuçlanır.
- Daha sonra, lipozomların azı dişi konsantrasyonu hesaplayın, M
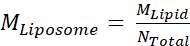 Lipid'in Adım 2.3.2'denlipitin azı dişi konsantrasyonu olduğu denklemi kullanarak ve Ntoplamı Adım 2.3.1'dehesaplanan lipozom başına toplam lipit sayısıdır. Bu örnekte lipozomların 20 mg/mL stok konsantrasyonu yaklaşık 660 nM'dir.
Lipid'in Adım 2.3.2'denlipitin azı dişi konsantrasyonu olduğu denklemi kullanarak ve Ntoplamı Adım 2.3.1'dehesaplanan lipozom başına toplam lipit sayısıdır. Bu örnekte lipozomların 20 mg/mL stok konsantrasyonu yaklaşık 660 nM'dir. - 100 μL rekonsup reaksiyon için gereken Msp1 ve TA protein miktarını hesaplayın.
NOT: Rekonsitteki son lipit konsantrasyonu, lipozom stoğunun 10x seyreltilmesi olan 2 mg / mL'dir. Bu, 66 nM'lik son bir lipozom konsantrasyonu verir. Msp1'in son konsantrasyonu 792 nM'dir, bu da lipozom başına ortalama 12 toplam kopya (2 fonksiyonel altıgen) verir. TA proteininin son konsantrasyonu 660 nM'dir ve lipozom başına ortalama 10 kopya verir.
- İlk olarak, d lipozom çapı, h bilayer kalınlığı ve a lipid kafa grubu alanı olduğu denklemi kullanarak teklimeller lipozom başına lipit moleküllerinin sayısını(Ntoplam)
- Bir PCR tüpünde, saflaştırılmış Msp1, TA proteini ve lipozomları reconstitution tamponunda karıştırın. Ekleme sırası tampon, proteinler ve lipozomlardır. Toplam hacim 100 μL'dir. Karışımın 10 dakika boyunca buzun üzerinde oturmasına izin verin. Daha önce açıklandığı gibi Msp1 ve TA proteinini arındırın57.
- Test ilk kurulurken iyi karakterize model TA protein His-Flag-Sumo-Sec22 pozitif kontrol substratı olarak kullanın. Bu yapı, kolay arınma için bir His etiketine, batı blot tarafından tespit için 3x-Flag etiketine, artan çözünürlük için bir Sumo etki alanına ve MSP1 tarafından yeniden yapılanma ve tanıma için ER-TA proteini Sec22'nin TMD'sine sahiptir.
- N-Dodecyl β-D-maltoside'ın (DDM) protein saflaştırmasının rekonstrüt üzerindeki etkisini en aza indirmek için hem Msp1 hem de TA proteininin stok çözeltilerinin yaklaşık 100 μM olduğundan emin olun. Saflaştırılmış Msp1 20 mM HEPES pH 7.5, 100 mM sodyum klorür, 0.1 mM Tris (2-karboksitil)fosfin hidroklorür (TCEP), % 0.05 DDM'de bulunurken, saflaştırılmış TA proteini 50 mM Tris pH 7.4, 150 mM sodyum klorür, 10 mM magnezyum klorür, 5 mM β-mercaptoethanol, % 10 gliserol, % 0.1 DDM. Yaklaşık 100 μM TA proteini ve Msp1 monomer stok çözümleri, bu bileşenlerin nihai rekonsyon hacminin% 5'< oluşturmasını sağlar ve bu da CMC'nin altında DDM seyreltilmesine neden olur.
- Deterjanı çıkarmak için numuneye istediğiniz miktarda biyobead ekleyin.
- Boncukların ucuna sığabilmesi için p200 pipet ucunun ucunu yaklaşık1/8 inç çapa kesin. Düzgün bir karışım elde etmek için biyobead tüpünü iyice vorteks edin ve biyobeadlar yerleşmeden önce kapağı ve pipetleri hızla çıkarın. Biyobeadları boş bir PCR tüpüne aktarın.
- Yeniden yapılanma buz üzerinde 10 dakikalık inkübasyonunu bitirdiğinde, tüm sıvıyı biyobeadlardan çıkarmak için kesilmemiş bir pipet ucu kullanın. Daha sonra 100 μL rekonsesi biyobeadlarla tüpe aktarın. Bu hızlı bir şekilde yapılmalıdır, böylece biyobeadlar havayı hapsetmez, bu da boncukların yüzmesine neden olur.
- Yeniden yapılanma işlemine ~80 rpm'de bir tekerlek üzerinde 4 °C'de 16 saat boyunca dönmeleri için izin verin.
- Yeniden inşa edilen malzemeyi biyobeadlardan çıkarın. Biyobeadları peletmek için bir piofuge hızlı bir dönüş yapın ve ardından yeniden inşa edilen malzemeyi temiz bir PCR tüpüne aktarmak için kesilmemiş bir boru ucu kullanın. Numunede biyobead kalana kadar bu işlemi 1-2 kez tekrarlayın. Buzun üzerinde yeniden yapılandırıya devam et.
- Lipozomlara yeniden inşa edemeyen proteinleri çıkarmak için yeniden inşa edilen malzemeyi önceden temizleyin.
- Ekstraksiyon Tamponu Hazırlayın: 50 mM HEPES pH 7.5, 200 mM potasyum asetat, 7 mM magnezyum asetat, 2 mM DTT, 100 nM kalsiyum klorür.
- Glutatyon spin sütunlarını üreticinin talimatlarına göre Ekstraksiyon Tamponu ile dengeler. Bu tipik olarak 400 μL tampon ile 3 tur yıkamayı ve ardından tamponu çıkarmak için oda sıcaklığında 2 dakika boyunca 700 x g'da santrifüj yapmayı içerir.
- Yeniden inşa edilen malzemeye her refakatçiden (GST-SGTA ve GST-Calmodulin) 5 μM ekleyin. Bu refakatçiler lipozomlara yeniden inşa edemeyen proteinlerin TMD'sine bağlanacaklar. Refakatçilerin saflaştırılması daha önce6,57olarak tanımlanmıştır.
NOT: Bu refakatçiler ticari olarak mevcuttur, ancak maliyet ve kaliteyi kontrol etmek için evde saflaştırmayı tercih ediyoruz. Her iki protein de 20 mM Tris pH 7.5, 100 mM sodyum klorür ve 0.1 mM TCEP'tedir ve 160 μM civarında stok konsantrasyonuna sahiptir. SGTA, substratları yüksek hidrofobik bir TMD ile tanırken, Calmodulin TMD'leri orta derecede hidrofobik6,29ile bağlar. Birlikte, bu refakatçi kokteyli çok çeşitli substratları tanıyabilir. - Yeniden inşa edilen malzemeye 100 μL ekstraksiyon tamponu ekleyerek hacmi 200 μL'ye kadar getirin. Bunu dengelenmiş glutatyon spin sütunlarına ekleyin. Glutatyon spin sütunlarının ön temizleme hacmi 200 – 400 μL olduğunda en yüksek numune geri kazanımını sağladığını unutmayın.
- Döndürme sütunlarını takın ve refakatçilerin reçineye bağlanmasını sağlamak için 30 dakika boyunca 4 °C'de ~80 rpm'de döndürün.
- Kolonları oda sıcaklığında 2 dakika boyunca 700 x g'da döndürün. Akış, agrega proteinlerinin tükendiğı önceden temizlenmiş malzemedir. Malzemeyi buzda tutun ve doğrudan ekstraksiyon testiyle devam edin.
3. Ekstraksiyon Tahlil
- Tüpleri SDS PAGE analizi için hazırlayın. Her reaksiyon 4 tüpe sahip olacaktır: Gİrİş (I), FLOW THROUGH (FT), WASH (W) ve ELUTE (E).
- Gİrİş tüpüne 45 μL ddH2O numunesi, FLOW THROUGH tüpüne 40 μL ddH2O ve WASH ve ELUTE tüplerine 0 μL ekleyin.
NOT: Bu testteki en iyi sinyal, WASH ve ELUTE numuneleri Gİrİş ve FLOW THROUGH örneklerine göre 5 kat yoğunlaştığında elde edilir. Test sırasındaki seyreltmeler nedeniyle, bu, INPUT numunesi için 5 μL numune, FLOW THROUGH örneği için 10 μL numune ve WASH ve ELUTE numuneleri için 50 μL numune almayı gerektirir. - Her tüpe 16,6 μL 4x SDS PAGE Yükleme Tamponu ekleyin. SDS PAGE Yükleme Tamponu'ndan önce her numunenin toplam hacmi 50 μL'dir. Son hacim 66,6 μL'dir (50 μL numune + 16,6 μL 4x SDS PAGE Yükleme Tamponu).
- Gİrİş tüpüne 45 μL ddH2O numunesi, FLOW THROUGH tüpüne 40 μL ddH2O ve WASH ve ELUTE tüplerine 0 μL ekleyin.
- Çıkarma testini birleştirin.
- Önceden temizlenmiş 60 μL proteolipozom, 5 μM GST-SGTA, 5 μM GST-Calmodulin ve 2 mM ATP içeren ekstraksiyon reaksiyonu hazırlayın. Reaksiyonun başlatılmasında kullanılan ATP dışındaki tüm reaktifleri birleştirin. Ekstraksiyon Tamponu ile 200 μL'lik son hacme getirin.
NOT: Her ekstraksiyon tahlil için 60 μL numune kullanıldığından, üç farklı ekstraksiyon tahlilleri için bir rekonsepsiyon kullanılabilir. Aynı yeniden yapılandırıdaki malzeme üzerinde pozitif ve negatif kontroller (+ATP ve -ATP) gerçekleştirin. - 2 dakika boyunca 30 °C ısı bloğunda önceden ılık ekstraksiyon tahlilleri yapın.
- Önceden temizlenmiş 60 μL proteolipozom, 5 μM GST-SGTA, 5 μM GST-Calmodulin ve 2 mM ATP içeren ekstraksiyon reaksiyonu hazırlayın. Reaksiyonun başlatılmasında kullanılan ATP dışındaki tüm reaktifleri birleştirin. Ekstraksiyon Tamponu ile 200 μL'lik son hacme getirin.
- 2 mM'lik son konsantrasyona ATP ekleyerek ekstraksiyon testini başlatın ve zamanlayıcıyı başlatın.
- ATP'yi reaksiyona karıştırmak için bir picofuge'da 5 saniyelik bir dönüş verin. Reaksiyon 30 °C'de 30 dakika boyunca kuluçkaya yaslanın.
- Kuluçka sırasında, reaksiyonun 5 μL'sini alın ve Gİrİş tüpüne ekleyin. Bunun zamanlaması esnektir.
- Bu inkübasyon döneminde, ekstraksiyon testinde her örnek için bir glutatyon spin sütununu aşındırın.
- Çıkarılan malzemeyi izole etmek için refakatçileri aşağı çekme gerçekleştirin.
- 30 dakikalık inkübasyon tamamlandıktan sonra, toplam hacmi 400 μL'ye getirmek için tüpe 200 μl ekstraksiyon tamponu ekleyin. Dengelenmiş glutatyon reçinesine ekleyin ve 4° C'de tekerlek üzerinde 30 dakika boyunca bağlanmaya izin verin.
- Akışı toplamak için kolonları oda sıcaklığında 2 dakika boyunca 700 x g'da döndürün. FLOW THROUGH tüpü için 10 μL alın. Bu örnek, lipid bilayer'e hala entegre olan substratlar içerir.
- Reçineyi 400 μL ekstraksiyon tamponu ile iki kez yıkayın ve akışı atın. Üçüncü yıkamada, akışı tutun ve WASH tüpü için 50 μL alın.
- Ekstraksiyon Tamponunda 5 mM'lik son konsantrasyona azaltılmış glutatyon ekleyerek 5 mL Elution Buffer hazırlayın. Bu arabelleği her seferinde taze hazırlayın.
- Spin sütununa 200 μL Elution Buffer ekleyin. Oda sıcaklığında 5 dakika kuluçkaya yatır. 700 x g'da 2 dakika boyunca oda sıcaklığında döndürün. Akışı devam ettir. Toplam elution hacminin 400 μL olması için işlemi ikinci kez tekrarlayın.
- Elution örneğinden 50 μL numune alın ve ELUTE tüpüne ekleyin.
- SDS-PAGE ve batı blot kullanarak ayıklama etkinliğini analiz edin.
NOT: Batı lekesi oldukça standart bir prosedür olduğundan, bu teste özgü ayrıntılardan birkaçını vurgulayan temel bir protokol sağlanır.- Numuneleri lekesiz bir poliakrilamid jeline yükleyin (%4 istifleme, %15 ayırma) ve Tris glisin tamponunda 50 dakika boyunca 200 V'ta çalıştırın. Alan izin verirse, lekesiz jel görüntüleyicide görselleştirmeye izin vermek ve sırasıyla PVDF membranlarına aktarmak için hem lekesiz hem de lekeli bir merdiven kullanın. Lekesiz jel, ultraviyole ışıkla aktivasyon üzerine protein içeren triptofanın nicel görselleştirilmesine izin verirken, jelin batı lekesi için kullanılmasına izin verir.
- Tüm numunelerde eşit yükleme olduğunu onaylamak için lekesiz jeli görüntüleyin. Bu, batı lekesi tarafından tespit edilen sinyaldeki herhangi bir değişikliğin değişken protein yüklemesinin bir sonucu olmamasını sağlayan önemli bir kontroldür. INPUT ve ELUTION örneklerinde sadece refakatçiler (GST-calmodulin ve GST-SGTA) için görünür bantlar olmalıdır. ELUTE örneğinin INPUT örneğinden daha konsantre olacağını hatırlayın.
- 45 μm PVDF membran kullanarak batı blot kasetini monte edin. 60 dakika boyunca 300 mA sabit akımda aktarın.
- Zarı bloke ettikten sonra, hafif sallama ~15 rpm ile 4 °C'de 16 saat boyunca birincil antikora bağlayın. 1:1.000 seyreltmede tavşan Anti-FLAG ile substrat için leke.
NOT: Kullanılan birincil ve ikincil antikorlar substrat spesifik olacaktır ve kullanım konsantrasyonu optimize edilmelidir. - Membran yıkayın ve ikincil antikor, keçi anti-tavşanı 1:10.000 seyreltmede, oda sıcaklığında 1 saat hafif sallanarak kuluçkaya yatırın.
- Batı şişkinlik algılama maddesini kullanarak analiz için membran ve görüntüyü yıkayın.
Access restricted. Please log in or start a trial to view this content.
Sonuçlar
Sonuçları düzgün bir şekilde yorumlamak için lekesiz jel ve batı lekesi birlikte görülmelidir. Lekesiz jel, tüm numunelerde eşit yükleme sağlar. Lekesiz jeli görüntülerken, refakatçiler (GST-calmodulin ve GST-SGTA) INPUT (I) ve ELUTE (E) şeritlerinde görülebilir. Bu bantların yoğunluğunun tüm INPUT örneklerinde tekdüze olup olmadığını iki kez kontrol edin. Aynı şekilde, yoğunluğun ELUTE numuneleri boyunca düzgün olduğundan emin olun. ELUTE, Gİrİş'ten 5 kat daha konsantredir ve bu y...
Access restricted. Please log in or start a trial to view this content.
Tartışmalar
Uygun mitokondriyal fonksiyon sağlam bir protein kalite kontrol sistemine bağlıdır. TA protein hedefleme yollarının doğruluğundaki doğal sınırlar nedeniyle, yanlış hesaplanan TA proteinleri mitokondri için sürekli bir stres kaynağıdır. Mitokondriyal proteostaz ağının önemli bir bileşeni, yanlış etiketlenmiş TA proteinlerini OMM'den uzaklaştıran bir membran bağlantılı AAA + ATPase olan Msp1'dir. Burada, proteolipozomların nasıl hazırlanacağını, Msp1 ve bir model TA proteinini nasıl ye...
Access restricted. Please log in or start a trial to view this content.
Açıklamalar
Hiç kimse
Teşekkürler
MLW, chicago Üniversitesi'nde Dr. Robert Keenan ile yaptığı doktora sonrası çalışmalar sırasında bu protokolün bir parçasını geliştirdi.
Bu çalışma MLW'ye NIH hibe 1R35GM137904-01 tarafından finanse edilir.
Access restricted. Please log in or start a trial to view this content.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Biobeads | Bio-Rad | 1523920 | |
| Bovine liver phosphatidyl inositol | Avanti | 840042C | PI |
| Chicken egg phosphatidyl choline | Avanti | 840051C | PC |
| Chicken egg phosphatidyl ethanolamine | Avanti | 840021C | PE |
| ECL Select western blotting detection reagent | GE | RPN2235 | |
| Filter supports | Avanti | 610014 | |
| Glass vial | VWR | 60910L-1 | |
| Glutathione spin column | Thermo Fisher | PI16103 | |
| Goat anti-rabbit | Thermo Fisher | NC1050917 | |
| Mini-Extruder | Avanti | 610020 | |
| Polycarbonate membrane | Avanti | 610006 | 200 nM |
| PVDF membrane | Thermo Fisher | 88518 | 45 µM |
| Rabbit anti-FLAG | Sigma-Aldrich | F7245 | |
| Synthetic 1,2-dioleoyl-sn-glycero-3-phospho-L-serine | Avanti | 840035C | DOPS |
| Synthetic 1',3'-bis[1,2-dioleoyl-sn-glycero-3-phospho]-glycerol | Avanti | 710335C | TOCL |
| Syringe, 1 mL | Norm-Ject | 53548-001 | |
| Syringe, 1 mL, gas-tight | Avanti | 610017 |
Referanslar
- Song, J., Herrmann, J. M., Becker, T. Quality control of the mitochondrial proteome. Nature Reviews Molecular Cell Biology. 22, 54-70 (2021).
- Phillips, B. P., Miller, E. A. Membrane protein folding and quality control. Current Opinion in Structural Biology. 69, 50-54 (2021).
- Jiang, H. Quality control pathways of tail-anchored proteins. Biochimica et Biophysica Acta - Molecular Cell Research. 1868, 118922(2020).
- McKenna, M. J., et al. The endoplasmic reticulum P5A-ATPase is a transmembrane helix dislocase. Science. 369, New York, N.Y. (2020).
- Hegde, R. S., Zavodszky, E. Recognition and Degradation of Mislocalized Proteins in Health and Disease. Cold Spring Harbor Perspectives in Biology. 11, 033902(2019).
- Shao, S., Hegde, R. S. A calmodulin-dependent translocation pathway for small secretory proteins. Cell. 147, 1576-1588 (2011).
- Samuelson, J. C., et al. YidC mediates membrane protein insertion in bacteria. Nature. 406, 637-641 (2000).
- Anghel, S. A., McGilvray, P. T., Hegde, R. S., Keenan, R. J. Identification of Oxa1 Homologs Operating in the Eukaryotic Endoplasmic Reticulum. Cell Reports. 21, 3708-3716 (2017).
- Aviram, N., et al. The SND proteins constitute an alternative targeting route to the endoplasmic reticulum. Nature. 540, 134-138 (2016).
- Voorhees, R. M., Hegde, R. S. Structure of the Sec61 channel opened by a signal sequence. Science. 351, New York, NY. 88-91 (2016).
- Cichocki, B. A., Krumpe, K., Vitali, D. G., Rapaport, D. Pex19 is involved in importing dually targeted tail-anchored proteins to both mitochondria and peroxisomes. Traffic. 19, Copenhagen, Denmark. 770-785 (2018).
- Mateja, A., et al. Protein targeting. Structure of the Get3 targeting factor in complex with its membrane protein cargo. Science. 347, New York, NY. 1152-1155 (2015).
- Chacinska, A., Koehler, C. M., Milenkovic, D., Lithgow, T., Pfanner, N. Importing mitochondrial proteins: machineries and mechanisms. Cell. 138, 628-644 (2009).
- Chitwood, P. J., Hegde, R. S. An intramembrane chaperone complex facilitates membrane protein biogenesis. Nature. , (2020).
- Chitwood, P. J., Juszkiewicz, S., Guna, A., Shao, S., Hegde, R. S. EMC Is Required to Initiate Accurate Membrane Protein Topogenesis. Cell. 175, 1-30 (2018).
- Bock, F. J., Tait, S. W. G. Mitochondria as multifaceted regulators of cell death. Nature Reviews Molecular Cell Biology. 21, 85-100 (2020).
- Pfanner, N., Warscheid, B., Wiedemann, N. Mitochondrial proteins: from biogenesis to functional networks. Nature Reviews Molecular Cell Biology. 20, (2019).
- Bykov, Y. S., Rapaport, D., Herrmann, J. M., Schuldiner, M. Cytosolic Events in the Biogenesis of Mitochondrial Proteins. Trends in Biochemical Sciences. 45, 650-667 (2020).
- Pfanner, N., Warscheid, B., Wiedemann, N. Mitochondrial proteins: from biogenesis to functional networks. Nature Reviews Molecular Cell Biology. 427, 1135(2019).
- Borgese, N., Coy-Vergara, J., Colombo, S. F., Schwappach, B. The Ways of Tails: the GET Pathway and more. The Protein Journal. , 1-17 (2019).
- Mateja, A., Keenan, R. J. A structural perspective on tail-anchored protein biogenesis by the GET pathway. Current Opinion in Structural Biology. 51, 195-202 (2018).
- Chio, U. S., Cho, H., Shan, S. Mechanisms of Tail-Anchored Membrane Protein Targeting and Insertion. Annual review of cell and developmental biology. 33, 417-438 (2017).
- Denic, V. A portrait of the GET pathway as a surprisingly complicated young man. Trends in biochemical sciences. , (2012).
- Hegde, R. S., Keenan, R. J. Tail-anchored membrane protein insertion into the endoplasmic reticulum. Nature Reviews Molecular Cell Biology. 12, 787-798 (2011).
- Kalbfleisch, T., Cambon, A., Wattenberg, B. W. A bioinformatics approach to identifying tail-anchored proteins in the human genome. Traffic. 8, Copenhagen, Denmark. 1687-1694 (2007).
- Doan, K. N., et al. The Mitochondrial Import Complex MIM Functions as Main Translocase for α-Helical Outer Membrane Proteins. Cell Reports. 31, (2020).
- McDowell, M. A., et al. Structural Basis of Tail-Anchored Membrane Protein Biogenesis by the GET Insertase Complex. Molecular Cell. 80, (2020).
- Guna, A., Volkmar, N., Christianson, J. C., Hegde, R. S. The ER membrane protein complex is a transmembrane domain insertase. Science. 591, New York, NY. 3099(2017).
- Rao, M., et al. Multiple selection filters ensure accurate tail-anchored membrane protein targeting. eLife. 5, 21301(2016).
- Schuldiner, M., et al. The GET complex mediates insertion of tail-anchored proteins into the ER membrane. Cell. 134, 634-645 (2008).
- Chen, Y. -C., et al. Msp1/ATAD1 maintains mitochondrial function by facilitating the degradation of mislocalized tail-anchored proteins. The EMBO journal. 33, 1548-1564 (2014).
- Wu, X., Rapoport, T. A. Translocation of Proteins through a Distorted Lipid Bilayer. Trends in Cell Biology. , (2021).
- Phillips, B. P., Gomez-Navarro, N., Miller, E. A. Protein quality control in the endoplasmic reticulum. Current Opinion in Cell Biology. 65, 96-102 (2020).
- van de Weijer, M. L., et al. Quality Control of ER Membrane Proteins by the RNF185/Membralin Ubiquitin Ligase Complex. Molecular Cell. 79, (2020).
- Weir, N. R., Kamber, R. A., Martenson, J. S., Denic, V. The AAA protein Msp1 mediates clearance of excess tail-anchored proteins from the peroxisomal membrane. eLife. 6, 28507(2017).
- Gardner, B. M., et al. The peroxisomal AAA-ATPase Pex1/Pex6 unfolds substrates by processive threading. Nature communications. 9, 135(2018).
- Puchades, C., et al. Unique Structural Features of the Mitochondrial AAA+ Protease AFG3L2 Reveal the Molecular Basis for Activity in Health and Disease. Molecular Cell. , (2019).
- Castanzo, D. T., LaFrance, B., Martin, A. The AAA+ ATPase Msp1 is a processive protein translocase with robust unfoldase activity. Proceedings of the National Academy of Sciences of the United States of America. 117, 14970-14977 (2020).
- Wang, L., Myasnikov, A., Pan, X., Walter, P. Structure of the AAA protein Msp1 reveals mechanism of mislocalized membrane protein extraction. eLife. 9, (2020).
- Puchades, C., Sandate, C. R., Lander, G. C. The molecular principles governing the activity and functional diversity of AAA+ proteins. Nature Reviews Molecular Cell Biology. , 1-16 (2019).
- Yang, Y., et al. Folding-Degradation Relationship of a Membrane Protein Mediated by the Universally Conserved ATP-Dependent Protease FtsH. Journal of the American Chemical Society. , 10(2018).
- Baldridge, R. D., Rapoport, T. A. Autoubiquitination of the Hrd1 Ligase Triggers Protein Retrotranslocation in ERAD. Cell. 166, 394-407 (2016).
- Fresenius, H. L., Wohlever, M. L. Sorting out how Msp1 maintains mitochondrial membrane proteostasis. Mitochondrion. 49, 128-134 (2019).
- Wang, L., Walter, P. Msp1/ATAD1 in Protein Quality Control and Regulation of Synaptic Activities. Annual Review of Cell and Developmental Biology. 36, 1-24 (2020).
- Dederer, V., et al. Cooperation of mitochondrial and ER factors in quality control of tail-anchored proteins. eLife. 8, 1126(2019).
- Matsumoto, S., et al. Msp1 Clears Mistargeted Proteins by Facilitating Their Transfer from Mitochondria to the ER. Molecular Cell. , (2019).
- Li, L., Zheng, J., Wu, X., Jiang, H. Mitochondrial AAA-ATPase Msp1 detects mislocalized tail-anchored proteins through a dual-recognition mechanism. EMBO Reports. 20, (2019).
- Weidberg, H., Amon, A. MitoCPR - a surveillance pathway that protects mitochondria in response to protein import stress. Science. 360, New York, NY. (2018).
- Okreglak, V., Walter, P. The conserved AAA-ATPase Msp1 confers organelle specificity to tail-anchored proteins. Proceedings of the National Academy of Sciences of the United States of America. 111, (2014).
- Piard, J., et al. A homozygous ATAD1 mutation impairs postsynaptic AMPA receptor trafficking and causes a lethal encephalopathy. Brain. , (2018).
- Zhang, J., et al. The AAA+ ATPase Thorase regulates AMPA receptor-dependent synaptic plasticity and behavior. Cell. 145, 284-299 (2011).
- Prendergast, J., et al. Ganglioside regulation of AMPA receptor trafficking. The Journal of Neuroscience. 34, 13246-13258 (2014).
- Umanah, G. K. E., et al. Thorase variants are associated with defects in glutamatergic neurotransmission that can be rescued by Perampanel. Science Translational Medicine. 9, 4985(2017).
- Pignatelli, M., et al. Synaptic Plasticity onto Dopamine Neurons Shapes Fear Learning. Neuron. 93, 425-440 (2017).
- Zhang, J., et al. The AAA Thorase is neuroprotective against ischemic injury. Journal of Cerebral Blood Flow and Metabolism. , 271678(2018).
- Umanah, G. K. E., et al. AMPA Receptor Surface Expression Is Regulated by S-Nitrosylation of Thorase and Transnitrosylation of NSF. Cell Reports. 33, 108329(2020).
- Wohlever, M. L., Mateja, A., McGilvray, P. T., Day, K. J., Keenan, R. J. Msp1 Is a Membrane Protein Dislocase for Tail-Anchored Proteins. Molecular Cell. 67, 194-202 (2017).
- Lovell, J. F., et al. Membrane binding by tBid initiates an ordered series of events culminating in membrane permeabilization by Bax. Cell. 135, 1074-1084 (2008).
- Leshchiner, E. S., Braun, C. R., Bird, G. H., Walensky, L. D. Direct activation of full-length proapoptotic BAK. Proceedings of the National Academy of Sciences of the United States of America. 110, 986-995 (2013).
Access restricted. Please log in or start a trial to view this content.
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır