Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Восстановление экстракции Msp1 с полностью очищенными компонентами
В этой статье
Резюме
Здесь мы представляем подробный протокол восстановления экстракционной активности Msp1 с полностью очищенными компонентами в определенных протеолипосомах.
Аннотация
Являясь центром окислительного фосфорилирования и апоптотической регуляции, митохондрии играют жизненно важную роль в здоровье человека. Правильная функция митохондрий зависит от надежной системы контроля качества для поддержания белкового гомеостаза (протеостаза). Снижение митохондриального протеостаза было связано с раком, старением, нейродегенерацией и многими другими заболеваниями. Msp1 представляет собой АТФазу AAA+, закрепленную во внешней митохондриальной мембране, которая поддерживает протеостаз путем удаления неправильно локализованных хвостовых белков. Используя очищенные компоненты, преобразованные в протеолипосомы, мы показали, что Msp1 необходим и достаточен для извлечения модельного хвостового белка из липидного бислоя. Наша упрощенная восстановленная система преодолевает несколько технических барьеров, которые препятствовали детальному изучению экстракции мембранного белка. Здесь мы предоставляем подробные методы генерации липосом, восстановления мембранного белка и экстракционного анализа Msp1.
Введение
Правильная клеточная функция зависит от процесса, называемого протеостазом, который гарантирует, что функциональные белки находятся в правильной концентрации и клеточном расположении1. Сбои в протеостазе приводят к нарушению функции органелл и связаны со многими нейродегенеративными заболеваниями2,3,4. Мембранные белки представляют собой уникальные проблемы для сети протеостаза, поскольку они должны быть нацелены на правильную мембрану, избегая при этом агрегации из гидрофобных трансмембранных доменов (TMD)5. Следовательно, специализированные механизмы эволюционировали, чтобы защитить гидрофобную ВНЧС от цитозоля и облегчить нацеливание и введение в правильную клеточную мембрану6,7,8,9,10,11,12,13,14,15.
Митохондрии являются метаболическим центром клетки и участвуют в многочисленных важных клеточных процессах, таких как: окислительное фосфорилирование, генерация железо-серного кластера и апоптотическая регуляция16,17. Эти эндосимбиотические органеллы содержат две мембраны, называемые внутренней митохондриальной мембраной (IMM) и внешней митохондриальной мембраной (OMM). Более 99% из 1 500 митохондриальных белков человека кодируются в ядерном геноме и должны быть перемещены через одну или две различные мембраны18,19. Таким образом, правильная функция митохондрий зависит от надежной сети протеостаза для исправления любых ошибок в нацеливании на белок или транслокации.
Наша лаборатория фокусируется на подмножестве белков митохондриальной мембраны, называемых белками хвостового якоря (TA), которые имеют один трансмембранный домен на самом С-конце20,21,22,23,24. Белки TA участвуют в ряде важных процессов, таких как апоптоз, транспорт пузырьков и транслокация белка25. Уникальная топология белков ТА требует посттрансляционной вставки, которая происходит в эндоплазматическом ретикулуме (ER) по направляющему входу хвостового якорного (GET) или эндоплазматического ретикулярного мембранного белкового комплекса (EMC) или в ОММ по плохо охарактеризованному пути20,26,27,28. Биофизические свойства ВНЧС необходимы и достаточны для направления белков ТА к правильной мембране29. Распознавание биофизических характеристик, а не определенного мотива последовательности, ограничивает точность путей нацеливания5. Таким образом, неправильная локализация белков ТА является общим стрессом для сетей протеостаза. Клеточный стресс, такой как ингибирование пути GET, вызывает увеличение неправильной локализации белка к ОММ и митохондриальную дисфункцию, если эти белки не будут быстро удалены30,31.
Общей темой в мембранном протеостазе является использование белков AAA+(ATPase A,ссоциированная с клеточными активами A)для удаления старых, поврежденных или неправильно локализованных белков из липидного бислоя1,32,33,34,35,36,37,38 . Белки AAA+ представляют собой молекулярные двигатели, которые образуют гексамерные кольца и подвергаются АТФ-зависимым движениям для ремоделирования субстрата, часто путем транслокации через узкую осевую пору39,40. Хотя большие усилия были посвящены изучению экстракции мембранных белков AAA+ ATPases, реконструкции являются сложными или включают смесь липидов и моющего средства41,42,что ограничивает экспериментальную способность исследовать механизм экстракции субстрата из липидного бислоя.
Msp1 представляет собой высококонсервативную ААА+ АТФазу, закрепленную в ОММ и пероксисомах, которая играет решающую роль в мембранном протеостазе путем удаления неправильно локализованных белков ТА43,44,45,46,47. Недавно было также показано, что Msp1 облегчает стресс импорта митохондриальных белков путем удаления мембранных белков, которые останавливаются во время транслокации через OMM48. Потеря Msp1 или человеческого гомолога ATAD1 приводит к фрагментации митохондрий, сбоям в окислительном фосфорилировании, судорогам, увеличению травматизма после инсульта и ранней смерти31,49,50,51,52,53,54,55,56.
Мы показали, что можно совместно воссоздать белки ТА с Msp1 и обнаружить экстракцию из липидного бислоя57. Эта упрощенная система использует полностью очищенные белки, преобразованные в определенные липосомы, которые имитируют OMM(Рисунок 1)58,59. Этот уровень экспериментального контроля может решить подробные механистические вопросы экстракции субстрата, которые экспериментально трудноразрешимы с более сложными восстановлениями с участием других белков AAA+. Здесь мы предоставляем экспериментальные протоколы, подробно описывающие наши методы получения липосом, восстановления мембранного белка и экстракционного анализа. Мы надеемся, что эти экспериментальные детали облегчат дальнейшее изучение существенного, но плохо изученного процесса мембранного протеостаза.
протокол
1. Подготовка липосом
- Объедините запасы хлороформа липидов в соответствующих соотношениях, чтобы имитировать внешнюю митохондриальную мембрану.
- Приготовить 25 мг липидной смеси. Мы используем ранее установленную смесь липидов, имитирующих митохондриальные мембраны, состоящую из 48:28:10:10:4 молярного соотношения фосфатидилхолина куриного яйца (ПК), фосфатидилэтаноламина куриного яйца (ПЭ), фосфатидилидозитола печени крупного рогатого скота (PI), синтетического 1,2-диолеоил-sn-глицеро-3-фосфо-L-серина (ДОФС) и синтетического 1',3'-бис[1,2-диолеоил-сна-глицеро-3-фосфо]-глицерина (TOCL)58,59. Примеры расчетов приведены в таблице 1.
- Доведите все запасы липидов до комнатной температуры перед открытием, так как это ограничит конденсацию. Поскольку большинство лабораторий не имеют точного способа измерения концентрации липидов, любая вода, поглощенная запасом хлороформа, изменит концентрацию липидного запаса и, следовательно, соотношение липидов, используемых в анализе.
- Поскольку запасы липидов поступают в стеклянных ампулах, перенесите необходимое количество липидов в стеклянный флакон с помощью шприца 1 мл. Добавьте 2 мг дитиотрейтола (DTT) во флакон, чтобы предотвратить окисление липидов. Работают быстро, так как испарение хлороформа изменит концентрацию липидов.
- Переложите оставшиеся липиды в отдельный стеклянный флакон и поместите в него перегородки PFTE. Добавьте 2 мг DTT во флакон, оберните парапленкой и храните при -20 °C, чтобы предотвратить окисление липидов. Старайтесь использовать липиды в течение 3 месяцев после переноса во флаконы. Чтобы предотвратить потенциальное загрязнение запасов хлороформа маркерным стоком, перенесите наклейки с оригинальных ампул на стеклянные флаконы, а не на этикетку с маркером.
- Испаряйте хлороформ под очень мягким потоком азота, непрерывно вращая стеклянный флакон вручную в вытяжном шкафу, по сути, действуя как ручной ротовап. Вращайте флакон с постоянной скоростью (20-40 оборотов в минуту) вручную, чтобы липиды двигались. Цель состоит в том, чтобы испарить весь хлороформ и получить равномерное покрытие липидов по всему стеклянному флакону.
ВНИМАНИЕ: Хлороформ является нейротоксичным, и этот этап должен выполняться в вытяжном шкафу.- Прикрепите свежую пипетку Пастера к азотной трубке. Не позволяйте липидам выплескиваться из флакона или на пипетку Пастера. Направьте наконечник на дно флакона, чтобы воздух отскакивал от дна и подталкивал липиды вверх к центру флакона. Получите равномерное покрытие по всему флакону, избегая при этом каких-либо скоплений в углах или у колпачка. Весь этот процесс занимает около 5 минут.
- По мере того, как смесь сгущается в «шарик» липидов, направляйте ее в центр флакона, изменяя угол флакона. Как только шарик начнет становиться меньше, слегка поднимите поток азота, чтобы рассеять шарик, гарантируя, что ни один из липидов не выдуется из флакона.
- Удалите оставшийся хлороформ под вакуумом.
- Поместите стеклянный флакон на домашний вакуумный или мембранный вакуумный насос на 1 час, чтобы удалить большую часть остаточного хлороформа. Эти вакуумы, как правило, недостаточно прочны, чтобы удалить весь хлороформ, но они могут переносить небольшое количество растворителя лучше, чем роторно-вакуумные пылесосы.
- Поставьте флакон на сильный вакуум (<1 мТорр) на 12-16 ч, чтобы удалить весь остаточный хлороформ. Обязательно избегайте натыкания на флакон во время этого процесса.
- Повторное суспендирование липидов в 1,25 мл липосомного буфера (50 мМ HEPES KOH pH 7,5, 15% глицерин, 1 мМ DTT). Поскольку мы начали с 25 мг липидов, это приводит к концентрации 20 мг / мл. Полностью повторно суспендировать липиды без видимых кусков. Если липиды скапливаются в углу флакона, это может быть длительным процессом.
- Энергично вращайте флакон до тех пор, пока образец не станет молочно-гладким. Мы находим, что если испарение хлороформа было сделано правильно накануне вечером, этот процесс занимает около 5-10 минут.
- Чтобы обеспечить полное повторное суспендирование липидов, вращайте на колесе при комнатной температуре в течение 3 часов при ~80 об/мин. Снимайте флакон с колеса один раз в час на 1 минуту вихря, чтобы обеспечить равномерное перемешивание.
- Осторожно перенесите липиды в чистую микроцентрифужную трубку объемом 1,5 мл. Выполните 5 циклов замораживания-оттаивания, используя жидкий азот для замораживания и тепловой блок 30 °C для оттаивания. Этот шаг помогает преобразовать многоламеллярные везикулы в одноламеллярные везикулы.
- Выдавливают липиды.
- Во время циклов замораживания оттаивайте мини-экструдер. Соберите мини-экструдер с 10 мм фильтрующими опорами и поликарбонатной мембраной нужного размера пор (мы используем 200 нм). Размер фильтра будет влиять на размер липосомы, что повлияет на концентрацию белков, необходимых для восстановления(шаг 2.3).
- Поместите мини-экструдер на конфорку и доведите температуру экструдера до 60 °C.
- Вытяните липиды в газонепроницаемый стеклянный шприц объемом 1 мл и аккуратно поместите в один конец мини-экструдера. Поместите пустой газонепроницаемый шприц в другую сторону мини-экструдера. Дайте липидам уравновеситься до температуры экструдера в течение 5-10 минут.
- Перенесите липиды на альтернативный шприц, осторожно толкая поршень заполненного шприца. Вставьте раствор из альтернативного шприца в исходный шприц. Повторите этот процесс взад-вперед 15 раз, чтобы на15-м проходе липиды заканчивались в альтернативном шприце. Контролируйте громкость на каждом проходе, чтобы убедиться в отсутствии утечек.
- Готовят одноразовые аликвоты липидов, замораживают в жидком азоте и хранят при -80 °C. Липосомы стабильны при -80 °C в течение нескольких месяцев. Для восстановления требуется 10 мкл липосом за один раз(шаг 2.3.4),поэтому удобно готовить 10 мкл или 20 мкл аликвот.
2 Восстановление белка Msp1 и модели TA
- Подготовьте буфер восстановления: 50 мМ HEPES pH 7,5, 200 мМ ацетата калия, 7 мМ ацетата магния, 2 мМ DTT, 10% сахарозы, 0,01% азида натрия, 0,2-0,8% Deoxy Big Chaps (DBC).
- Оптимизировать условия восстановления новой партии липосом. Концентрация DBC и биошариков, необходимая для оптимального восстановления, варьируется в зависимости от партии используемых липосом. Чтобы ограничить вариативность подготовки к подготовке, используйте один и тот же лот DBC для всех экспериментов. При смене большого количества DBC повторите процесс оптимизации.
- Установите серию реконструкций с различными концентрациями DBC (0,2% - 0,8%) и биошариков (25 мг - 100 мг) каждый раз, когда готовится новая партия липосом. Важно не опускать DBC ниже критической концентрации мицелл (CMC) ~ 0,12%. После оптимизации условий мы рекомендуем собирать все данные, используя одни и те же условия подготовки и восстановления липосом.
- Анализ эффективности различных условий восстановления с помощью экстракционного анализа, описанного в Шаге 3.
- Готовят биошарики в конечной концентрации 250 мг/мл.
- Взвесьте 2,7 г высушенных биошариков и повторно суспендируйте в центрифужной трубке объемом 50 мл из 100% метанола (около 45 мл), чтобы намочить шарики. Первоначально смачивайте биошарики метанолом, чтобы предотвратить попадание воздуха в поры бусин. Попав в метанол, держите биошарики влажными, так как любой воздух, захваченный биошариками, изменит их способность поглощать моющее средство.
- Удалите метанол, промыв шарики 8x примерно 45 мл сверхчистой воды (18,2 мОм), далее именуемые ddH2O. Гранулы путем прядения при 3 200 х г в течение 1 минуты. Декантировать жидкость и повторно суспендировать в ddH2O.
- После промывки повторно суспендируют в 10 мл ddH2Oс 0,02% азидом натрия и хранят при 4 °C. Биошарики могут храниться при 4 °C в течение нескольких месяцев. Этот запас составляет 250 мг / мл, так как предполагается, что ~ 0,2 г теряется во время этапов стирки.
- Рассчитайте размер липосомы и желаемое количество молекул белка ТА и Msp1 на липосому. Это определит концентрацию белка Msp1 и TA, необходимую для восстановления.
- Во-первых, рассчитайте количество липидных молекул на одноламеллярную липосому(N total),используя уравнение,
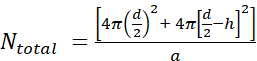 где d — диаметр липосомы, h — толщина бислоя, а a — площадь липидной головной группы.
где d — диаметр липосомы, h — толщина бислоя, а a — площадь липидной головной группы.- Измерьте диаметр липосом с помощью DLS. В нашем примере было получено значение 70 нм для диаметра липосомы(d).
- Используйте значение 5 нм для h и 0,71 нм2 для a , что является размером головнойгруппы для фосфатидилхолина. В этой конкретной ситуации общее число N составляет 37 610.
- Далее рассчитывают молярную концентрацию липида MLipid, используя среднюю молекулярную массу липидов в смеси. В этом примере концентрация липидов составляет 20 мг/мл(этап 1.4),а средняя молекулярная масса липидов составляет 810 г/моль(таблица 1). Это приводит к значению 0,0247 M для MLipid.
- Затем рассчитайте молярную концентрацию липосом, MLiposome,используя уравнение,
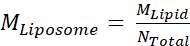 где M Lipid - молярная концентрация липидов из Шага 2.3.2,а Ntotal - общее количество липидов на липосому, рассчитанное на Шаге 2.3.1. В этом примере концентрация липосом в 20 мг/мл составляет приблизительно 660 нМ.
где M Lipid - молярная концентрация липидов из Шага 2.3.2,а Ntotal - общее количество липидов на липосому, рассчитанное на Шаге 2.3.1. В этом примере концентрация липосом в 20 мг/мл составляет приблизительно 660 нМ. - Рассчитайте количество белка Msp1 и TA, необходимое для реакции восстановления 100 мкл.
ПРИМЕЧАНИЕ: Конечная концентрация липидов в восстановлении составляет 2 мг/мл, что представляет собой 10-кратное разведение липосомного запаса. Это дает конечную концентрацию липосом 66 нМ. Конечная концентрация Msp1 составляет 792 нМ, что дает в среднем 12 суммарных копий (2 функциональных гексамера) на липосому. Конечная концентрация белка ТА составляет 660 нМ, что дает в среднем 10 копий на липосому.
- Во-первых, рассчитайте количество липидных молекул на одноламеллярную липосому(N total),используя уравнение,
- В пробирке ПЦР смешайте очищенный Msp1, белок TA и липосомы в буфере восстановления. Порядок добавления — буфер, белки и липосомы последние. Общий объем составляет 100 мкл. Дайте смеси постоять на льду в течение 10 минут. Очистите белок Msp1 и TA, какописано ранее 57.
- Используйте хорошо охарактеризованную модель TA белка His-Flag-Sumo-Sec22 в качестве положительного контрольного субстрата при первом установлении анализа. Эта конструкция имеет His-tag для легкой очистки, тег 3x-Flag для обнаружения западным пятном, домен Sumo для повышения растворимости и TMD белка ER-TA Sec22 для восстановления и распознавания Msp1.
- Убедитесь, что исходные растворы как Msp1, так и белка TA составляют приблизительно 100 мкМ, чтобы свести к минимуму влияние N-додецил-β-D-мальтозида (DDM) от очистки белка на восстановление. Очищенный Msp1 содержит 20 мМ HEPES pH 7,5, 100 мМ хлорида натрия, 0,1 мМ Tris(2-карбоксиэтил)фосфин гидрохлорида (TCEP), 0,05% DDM, тогда как очищенный белок TA находится в 50 мМ Tris pH 7,4, 150 мМ хлорида натрия, 10 мМ хлорида магния, 5 мМ β-меркаптоэтанола, 10% глицерина, 0,1% DDM. Исходные растворы примерно 100 мкМ белка TA и мономера Msp1 гарантируют, что эти компоненты будут составлять < 5% от окончательного объема восстановления, что приведет к разбавлению DDM ниже CMC.
- Добавьте нужное количество биошариков в образец, чтобы удалить моющее средство.
- Вырежьте наконечник пипетки p200 примерно до1/8 дюйма в диаметре, чтобы бусины могли проходить через наконечник. Тщательно вихрьте трубку биошариков, чтобы получить однородную смесь и быстро снимите крышку и пипетку вверх по объему, прежде чем биошарики осядут. Переложите биошарики в пустую ПЦР-трубку.
- Когда восстановление закончит свою 10-минутную инкубацию на льду, используйте неразрезанный наконечник пипетки, чтобы удалить всю жидкость из биошариков. Затем переложите восстановление 100 мкл в пробирку с биошариками. Это нужно сделать быстро, чтобы биошарики не задерживали воздух, который заставит бусины плавать.
- Позвольте восстановлению вращаться на колесе со скоростью ~80 об/мин в течение 16 часов при 4 °C.
- Удалите восстановленный материал из биошариков. Сделайте быстрый отжим в пикофуге, чтобы гранулировать биошарики, а затем используйте неразрезанный наконечник пипетки, чтобы перенести восстановленный материал в чистую трубку ПЦР. Повторяйте этот процесс 1-2 раза, пока в образце не останется биогранул. Держите восстановление на льду.
- Предварительно очистите восстановленный материал, чтобы удалить любые белки, которые не смогли восстановиться в липосомах.
- Подготовьте экстракционный буфер: 50 мМ HEPES pH 7,5, 200 мМ ацетат калия, 7 мМ ацетата магния, 2 мМ DTT, 100 нМ хлорида кальция.
- Уравновешивайте спиновые колонны глутатиона с помощью экстракционного буфера в соответствии с указаниями производителя. Это обычно включает в себя 3 раунда промывки с 400 мкл буфера, а затем центрифугирование при 700 х г в течение 2 минут при комнатной температуре для удаления буфера.
- Добавьте 5 мкМ каждого шаперона (GST-SGTA и GST-Calmodulin) к восстановленному материалу. Эти шапероны будут связываться с ВНЧС любых белков, которые не смогли восстановиться в липосомах. Очищение шаперонов было описаноранее 6,57.
ПРИМЕЧАНИЕ: Эти сопровождающие коммерчески доступны, но мы предпочитаем очищать дома, чтобы контролировать стоимость и качество. Оба белка находятся в 20 мМ Tris pH 7,5, 100 мМ хлорида натрия и 0,1 мМ TCEP и имеют концентрацию запаса около 160 мкМ. SGTA распознает субстраты с высокогидрофобной ВНЧС, тогда как Калмодулин связывает ВНЧС с умеренной гидрофобностью6,29. Вместе этот коктейль-шаперон может распознавать широкий спектр субстратов. - Добавьте 100 мкл экстракционного буфера к восстановленному материалу, доведя объем до 200 мкл. Добавьте это к уравновешенным спиновым колонкам глутатиона. Обратите внимание, что спиновые колонны глутатиона обеспечивают максимальное извлечение образца при объеме предварительной очистки 200 – 400 мкл.
- Заткните спиновые колонны и вращайте со скоростью ~80 об/мин при 4 °C в течение 30 минут, чтобы позволить шаперонам связываться со смолой.
- Вращайте колонны при 700 х г в течение 2 минут при комнатной температуре. Поток представляет собой предварительно очищенный материал, который обеднен агрегированными белками. Держите материал на льду и приступайте непосредственно к экстракционному анализу.
3. Экстракционный анализ
- Подготовьте трубки для анализа SDS PAGE. Каждая реакция будет иметь 4 трубки: ВХОДНАЯ (I), ПРОТОЧНАЯ (FT), WASH (W) и ELUTE (E).
- Добавьте 45 мкл образца ddH2O во ВХОДНУЮ трубку, 40 мкл ddH2O в пробирку FLOW THROUGH и 0 мкл в трубки WASH и ELUTE.
ПРИМЕЧАНИЕ: Наилучшее соотношение сигнал/шум в этом анализе получается, когда образцы WASH и ELUTE в 5 раз сконцентрированы относительно образцов INPUT и FLOW THROUGH. Из-за разбавлений во время анализа требуется взять 5 мкл образца для образца INPUT, 10 мкл образца для образца FLOW THROUGH и 50 мкл образца для образцов WASH и ELUTE. - Добавьте 16,6 мкл 4x SDS PAGE Loading Buffer на каждую трубку. Общий объем каждого образца составляет 50 мкл до загрузки буфера SDS PAGE. Конечный объем составляет 66,6 мкл (образец 50 мкл + 16,6 мкл 4x SDS PAGE Loading Buffer).
- Добавьте 45 мкл образца ddH2O во ВХОДНУЮ трубку, 40 мкл ddH2O в пробирку FLOW THROUGH и 0 мкл в трубки WASH и ELUTE.
- Соберите экстракционный анализ.
- Готовят реакцию экстракции, содержащую 60 мкл предварительно очищенных протеолипосом, 5 мкМ GST-SGTA, 5 мкМ GST-кальмодулина и 2 мМ АТФ. Объединить все реагенты, кроме АТФ, который используется для инициирования реакции. Доведите до конечного объема 200 мкл с помощью буфера извлечения.
ПРИМЕЧАНИЕ: Поскольку для каждого экстракционного анализа используется 60 мкл образца, одно восстановление может быть использовано для трех различных экстракционных анализов. Выполнение положительного и отрицательного контроля (+АТФ и -АТФ) на материале из одного и того же восстановления. - Предварительно теплый экстракционный анализ в термоблоке при температуре 30 °C в течение 2 минут.
- Готовят реакцию экстракции, содержащую 60 мкл предварительно очищенных протеолипосом, 5 мкМ GST-SGTA, 5 мкМ GST-кальмодулина и 2 мМ АТФ. Объединить все реагенты, кроме АТФ, который используется для инициирования реакции. Доведите до конечного объема 200 мкл с помощью буфера извлечения.
- Инициируют экстракционный анализ путем добавления АТФ к конечной концентрации 2 мМ и таймера запуска.
- Дайте 5-секундный спин в пикофуге, чтобы смешать АТФ в реакции. Инкубационная реакция при 30 °C в течение 30 минут.
- Во время инкубации берут 5 мкл реакции и добавляют во входную трубку. Сроки этого являются гибкими.
- В течение этого инкубационного периода уравновешивайте одну спиновую колонку глутатиона для каждого образца в экстракционном анализе.
- Выполните вытягивание шаперонов, чтобы изолировать извлеченный материал.
- После завершения 30-минутной инкубации добавьте 200 мкл экстракционного буфера в трубку, чтобы довести общий объем до 400 мкл. Добавьте к уравновешенной глутатионовой смоле и дайте связать на колесе при 4°С в течение 30 минут.
- Вращайте колонны при 700 х г в течение 2 минут при комнатной температуре, чтобы собрать поток. Возьмите 10 мкл для проточной трубки. Этот образец содержит субстраты, которые все еще интегрированы в липидный бислой.
- Дважды промывайте смолу 400 мкл экстракционного буфера, отбрасывая поток насквозь. На третьей промывке поддерживайте поток и возьмите 50 мкл для трубки WASH.
- Приготовьте 5 мл элюционного буфера, добавив восстановленный глутатион к конечной концентрации 5 мМ в буфере экстракции. Каждый раз подготавливайте этот буфер свежим.
- Добавьте 200 мкл буфера элюирования в спиновой столбец. Инкубировать при комнатной температуре в течение 5 минут. Вращайте при 700 х г в течение 2 минут при комнатной температуре до элюирования. Держите поток насквозь. Повторите процесс второй раз, чтобы общий объем элюирования составил 400 мкл.
- Возьмите 50 мкл образца из образца Elution и добавьте его в пробирку ELUTE.
- Анализируйте активность извлечения с помощью SDS-PAGE и вестерн-блоттинга.
ПРИМЕЧАНИЕ: Поскольку вестерн-блот является довольно стандартной процедурой, предоставляется базовый протокол, который выделяет некоторые детали, уникальные для этого анализа.- Загрузите образцы в полиакриламидный гель без пятен (4% укладка, 15% разделение) и работайте при 200 В в течение 50 минут в трис-глициновом буфере. Если позволяет пространство, используйте как неокрашенную, так и окрашенную лестницу, чтобы обеспечить визуализацию на гелевом тепловизоре без пятен и перенести на мембрану PVDF соответственно. Гель без пятен позволяет количественно визуализировать триптофансодержащие белки при активации ультрафиолетовым светом, в то же время позволяя использовать гель для вестерн-блоттинга.
- Визуализируйте гель без пятен, чтобы убедиться, что во всех образцах наблюдается одинаковая нагрузка. Это важный контроль, который гарантирует, что любые изменения сигнала, обнаруженные западным блоттом, не являются результатом переменной нагрузки на белок. В образцах INPUT и ELUTION должны быть видны только полосы для шаперонов (GST-кальмодулин и GST-SGTA). Напомним, что образец ELUTE будет более концентрированным, чем образец INPUT.
- Соберите вестерн-блот-кассету, используя 45-мкм PVDF-мембрану. Передача при постоянном токе 300 мА в течение 60 минут.
- После блокирования мембраны связывают с первичным антителом в течение 16 ч при 4 °C с легким встряхиванием ~15 об/мин. Пятно для субстрата с кроликом Anti-FLAG в разведении 1:1000.
ПРИМЕЧАНИЕ: Используемые первичные и вторичные антитела будут специфичными для субстрата, и концентрацию для использования, возможно, потребуется оптимизировать. - Промыть мембрану и инкубировать вторичным антителом, козьим анти-кроликом в разведении 1:10 000, с нежным встряхиванием в течение 1 часа при комнатной температуре.
- Промывка мембраны и изображение для анализа с использованием западного блоттингового детектирующего агента.
Результаты
Чтобы правильно интерпретировать результаты, гель без пятен и вестерн-блот должны рассматриваться вместе. Гель без пятен обеспечивает одинаковую нагрузку на все образцы. При просмотре геля без пятен шапероны (GST-calmodulin и GST-SGTA) будут видны в полосах INPUT (I) и ELUTE (E). Дважды проверьте, что инте...
Обсуждение
Правильная функция митохондрий зависит от надежной системы контроля качества белка. Из-за присущих ограничений в точности путей таргетирования белка TA, неправильно локализованные белки TA являются постоянным источником стресса для митохондрий. Ключевым компонентом сети митохондриал...
Раскрытие информации
Никакой
Благодарности
MLW разработал часть этого протокола во время своих постдокторских исследований с доктором Робертом Кинаном в Чикагском университете.
Эта работа финансируется грантом NIH 1R35GM137904-01 для MLW.
Материалы
| Name | Company | Catalog Number | Comments |
| Biobeads | Bio-Rad | 1523920 | |
| Bovine liver phosphatidyl inositol | Avanti | 840042C | PI |
| Chicken egg phosphatidyl choline | Avanti | 840051C | PC |
| Chicken egg phosphatidyl ethanolamine | Avanti | 840021C | PE |
| ECL Select western blotting detection reagent | GE | RPN2235 | |
| Filter supports | Avanti | 610014 | |
| Glass vial | VWR | 60910L-1 | |
| Glutathione spin column | Thermo Fisher | PI16103 | |
| Goat anti-rabbit | Thermo Fisher | NC1050917 | |
| Mini-Extruder | Avanti | 610020 | |
| Polycarbonate membrane | Avanti | 610006 | 200 nM |
| PVDF membrane | Thermo Fisher | 88518 | 45 µM |
| Rabbit anti-FLAG | Sigma-Aldrich | F7245 | |
| Synthetic 1,2-dioleoyl-sn-glycero-3-phospho-L-serine | Avanti | 840035C | DOPS |
| Synthetic 1',3'-bis[1,2-dioleoyl-sn-glycero-3-phospho]-glycerol | Avanti | 710335C | TOCL |
| Syringe, 1 mL | Norm-Ject | 53548-001 | |
| Syringe, 1 mL, gas-tight | Avanti | 610017 |
Ссылки
- Song, J., Herrmann, J. M., Becker, T. Quality control of the mitochondrial proteome. Nature Reviews Molecular Cell Biology. 22, 54-70 (2021).
- Phillips, B. P., Miller, E. A. Membrane protein folding and quality control. Current Opinion in Structural Biology. 69, 50-54 (2021).
- Jiang, H. Quality control pathways of tail-anchored proteins. Biochimica et Biophysica Acta - Molecular Cell Research. 1868, 118922 (2020).
- McKenna, M. J., et al. The endoplasmic reticulum P5A-ATPase is a transmembrane helix dislocase. Science. 369, (2020).
- Hegde, R. S., Zavodszky, E. Recognition and Degradation of Mislocalized Proteins in Health and Disease. Cold Spring Harbor Perspectives in Biology. 11, 033902 (2019).
- Shao, S., Hegde, R. S. A calmodulin-dependent translocation pathway for small secretory proteins. Cell. 147, 1576-1588 (2011).
- Samuelson, J. C., et al. YidC mediates membrane protein insertion in bacteria. Nature. 406, 637-641 (2000).
- Anghel, S. A., McGilvray, P. T., Hegde, R. S., Keenan, R. J. Identification of Oxa1 Homologs Operating in the Eukaryotic Endoplasmic Reticulum. Cell Reports. 21, 3708-3716 (2017).
- Aviram, N., et al. The SND proteins constitute an alternative targeting route to the endoplasmic reticulum. Nature. 540, 134-138 (2016).
- Voorhees, R. M., Hegde, R. S. Structure of the Sec61 channel opened by a signal sequence. Science. 351, 88-91 (2016).
- Cichocki, B. A., Krumpe, K., Vitali, D. G., Rapaport, D. Pex19 is involved in importing dually targeted tail-anchored proteins to both mitochondria and peroxisomes. Traffic. 19, 770-785 (2018).
- Mateja, A., et al. Protein targeting. Structure of the Get3 targeting factor in complex with its membrane protein cargo. Science. 347, 1152-1155 (2015).
- Chacinska, A., Koehler, C. M., Milenkovic, D., Lithgow, T., Pfanner, N. Importing mitochondrial proteins: machineries and mechanisms. Cell. 138, 628-644 (2009).
- Chitwood, P. J., Hegde, R. S. An intramembrane chaperone complex facilitates membrane protein biogenesis. Nature. , (2020).
- Chitwood, P. J., Juszkiewicz, S., Guna, A., Shao, S., Hegde, R. S. EMC Is Required to Initiate Accurate Membrane Protein Topogenesis. Cell. 175, 1-30 (2018).
- Bock, F. J., Tait, S. W. G. Mitochondria as multifaceted regulators of cell death. Nature Reviews Molecular Cell Biology. 21, 85-100 (2020).
- Pfanner, N., Warscheid, B., Wiedemann, N. Mitochondrial proteins: from biogenesis to functional networks. Nature Reviews Molecular Cell Biology. 20, (2019).
- Bykov, Y. S., Rapaport, D., Herrmann, J. M., Schuldiner, M. Cytosolic Events in the Biogenesis of Mitochondrial Proteins. Trends in Biochemical Sciences. 45, 650-667 (2020).
- Pfanner, N., Warscheid, B., Wiedemann, N. Mitochondrial proteins: from biogenesis to functional networks. Nature Reviews Molecular Cell Biology. 427, 1135 (2019).
- Borgese, N., Coy-Vergara, J., Colombo, S. F., Schwappach, B. The Ways of Tails: the GET Pathway and more. The Protein Journal. , 1-17 (2019).
- Mateja, A., Keenan, R. J. A structural perspective on tail-anchored protein biogenesis by the GET pathway. Current Opinion in Structural Biology. 51, 195-202 (2018).
- Chio, U. S., Cho, H., Shan, S. Mechanisms of Tail-Anchored Membrane Protein Targeting and Insertion. Annual review of cell and developmental biology. 33, 417-438 (2017).
- Denic, V. A portrait of the GET pathway as a surprisingly complicated young man. Trends in biochemical sciences. , (2012).
- Hegde, R. S., Keenan, R. J. Tail-anchored membrane protein insertion into the endoplasmic reticulum. Nature Reviews Molecular Cell Biology. 12, 787-798 (2011).
- Kalbfleisch, T., Cambon, A., Wattenberg, B. W. A bioinformatics approach to identifying tail-anchored proteins in the human genome. Traffic. 8, 1687-1694 (2007).
- Doan, K. N., et al. The Mitochondrial Import Complex MIM Functions as Main Translocase for α-Helical Outer Membrane Proteins. Cell Reports. 31, (2020).
- McDowell, M. A., et al. Structural Basis of Tail-Anchored Membrane Protein Biogenesis by the GET Insertase Complex. Molecular Cell. 80, (2020).
- Guna, A., Volkmar, N., Christianson, J. C., Hegde, R. S. The ER membrane protein complex is a transmembrane domain insertase. Science. 591, 3099 (2017).
- Rao, M., et al. Multiple selection filters ensure accurate tail-anchored membrane protein targeting. eLife. 5, 21301 (2016).
- Schuldiner, M., et al. The GET complex mediates insertion of tail-anchored proteins into the ER membrane. Cell. 134, 634-645 (2008).
- Chen, Y. -. C., et al. Msp1/ATAD1 maintains mitochondrial function by facilitating the degradation of mislocalized tail-anchored proteins. The EMBO journal. 33, 1548-1564 (2014).
- Wu, X., Rapoport, T. A. Translocation of Proteins through a Distorted Lipid Bilayer. Trends in Cell Biology. , (2021).
- Phillips, B. P., Gomez-Navarro, N., Miller, E. A. Protein quality control in the endoplasmic reticulum. Current Opinion in Cell Biology. 65, 96-102 (2020).
- van de Weijer, M. L., et al. Quality Control of ER Membrane Proteins by the RNF185/Membralin Ubiquitin Ligase Complex. Molecular Cell. 79, (2020).
- Weir, N. R., Kamber, R. A., Martenson, J. S., Denic, V. The AAA protein Msp1 mediates clearance of excess tail-anchored proteins from the peroxisomal membrane. eLife. 6, 28507 (2017).
- Gardner, B. M., et al. The peroxisomal AAA-ATPase Pex1/Pex6 unfolds substrates by processive threading. Nature communications. 9, 135 (2018).
- Puchades, C., et al. Unique Structural Features of the Mitochondrial AAA+ Protease AFG3L2 Reveal the Molecular Basis for Activity in Health and Disease. Molecular Cell. , (2019).
- Castanzo, D. T., LaFrance, B., Martin, A. The AAA+ ATPase Msp1 is a processive protein translocase with robust unfoldase activity. Proceedings of the National Academy of Sciences of the United States of America. 117, 14970-14977 (2020).
- Wang, L., Myasnikov, A., Pan, X., Walter, P. Structure of the AAA protein Msp1 reveals mechanism of mislocalized membrane protein extraction. eLife. 9, (2020).
- Puchades, C., Sandate, C. R., Lander, G. C. The molecular principles governing the activity and functional diversity of AAA+ proteins. Nature Reviews Molecular Cell Biology. , 1-16 (2019).
- Yang, Y., et al. Folding-Degradation Relationship of a Membrane Protein Mediated by the Universally Conserved ATP-Dependent Protease FtsH. Journal of the American Chemical Society. , 10 (2018).
- Baldridge, R. D., Rapoport, T. A. Autoubiquitination of the Hrd1 Ligase Triggers Protein Retrotranslocation in ERAD. Cell. 166, 394-407 (2016).
- Fresenius, H. L., Wohlever, M. L. Sorting out how Msp1 maintains mitochondrial membrane proteostasis. Mitochondrion. 49, 128-134 (2019).
- Wang, L., Walter, P. Msp1/ATAD1 in Protein Quality Control and Regulation of Synaptic Activities. Annual Review of Cell and Developmental Biology. 36, 1-24 (2020).
- Dederer, V., et al. Cooperation of mitochondrial and ER factors in quality control of tail-anchored proteins. eLife. 8, 1126 (2019).
- Matsumoto, S., et al. Msp1 Clears Mistargeted Proteins by Facilitating Their Transfer from Mitochondria to the ER. Molecular Cell. , (2019).
- Li, L., Zheng, J., Wu, X., Jiang, H. Mitochondrial AAA-ATPase Msp1 detects mislocalized tail-anchored proteins through a dual-recognition mechanism. EMBO Reports. 20, (2019).
- Weidberg, H., Amon, A. MitoCPR - a surveillance pathway that protects mitochondria in response to protein import stress. Science. 360, (2018).
- Okreglak, V., Walter, P. The conserved AAA-ATPase Msp1 confers organelle specificity to tail-anchored proteins. Proceedings of the National Academy of Sciences of the United States of America. 111, (2014).
- Piard, J., et al. A homozygous ATAD1 mutation impairs postsynaptic AMPA receptor trafficking and causes a lethal encephalopathy. Brain. , (2018).
- Zhang, J., et al. The AAA+ ATPase Thorase regulates AMPA receptor-dependent synaptic plasticity and behavior. Cell. 145, 284-299 (2011).
- Prendergast, J., et al. Ganglioside regulation of AMPA receptor trafficking. The Journal of Neuroscience. 34, 13246-13258 (2014).
- Umanah, G. K. E., et al. Thorase variants are associated with defects in glutamatergic neurotransmission that can be rescued by Perampanel. Science Translational Medicine. 9, 4985 (2017).
- Pignatelli, M., et al. Synaptic Plasticity onto Dopamine Neurons Shapes Fear Learning. Neuron. 93, 425-440 (2017).
- Zhang, J., et al. The AAA Thorase is neuroprotective against ischemic injury. Journal of Cerebral Blood Flow and Metabolism. , 271678 (2018).
- Umanah, G. K. E., et al. AMPA Receptor Surface Expression Is Regulated by S-Nitrosylation of Thorase and Transnitrosylation of NSF. Cell Reports. 33, 108329 (2020).
- Wohlever, M. L., Mateja, A., McGilvray, P. T., Day, K. J., Keenan, R. J. Msp1 Is a Membrane Protein Dislocase for Tail-Anchored Proteins. Molecular Cell. 67, 194-202 (2017).
- Lovell, J. F., et al. Membrane binding by tBid initiates an ordered series of events culminating in membrane permeabilization by Bax. Cell. 135, 1074-1084 (2008).
- Leshchiner, E. S., Braun, C. R., Bird, G. H., Walensky, L. D. Direct activation of full-length proapoptotic BAK. Proceedings of the National Academy of Sciences of the United States of America. 110, 986-995 (2013).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены