需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
用完全纯化的组分重构Msp1提取活性
摘要
在这里,我们提出了一个详细的方案,用于在定义的蛋白脂质体中用完全纯化的组分重建Msp1提取活性。
摘要
作为氧化磷酸化和凋亡调节的中心,线粒体在人体健康中起着至关重要的作用。适当的线粒体功能取决于强大的质量控制系统来维持蛋白质稳态(蛋白质稳态)。线粒体蛋白质平衡的下降与癌症,衰老,神经变性和许多其他疾病有关。Msp1是一种锚定在外线粒体膜中的AAA + ATP酶,通过去除错位的尾锚定蛋白来维持蛋白质平衡。使用重组成蛋白脂质体的纯化组分,我们已经证明Msp1是必要且足以从脂质双层中提取模型尾锚定蛋白的。我们简化的重组系统克服了阻碍膜蛋白提取详细研究的几个技术障碍。在这里,我们提供了脂质体生成,膜蛋白重建和Msp1提取测定的详细方法。
引言
适当的细胞功能取决于称为蛋白质稳态的过程,该过程确保功能蛋白处于正确的浓度和细胞位置1。蛋白质稳态的失败导致细胞器功能受损,并与许多神经退行性疾病2,3,4有关。膜蛋白对蛋白质稳态网络提出了独特的挑战,因为它们必须靶向正确的膜,同时避免疏水性跨膜结构域(TMDs)聚集5。因此,专门的机器已经发展到可以保护疏水性TMD免受细胞质基质的影响,并促进靶向和插入适当的细胞膜6,7,8,9,10,11,12,13,14,15。
线粒体是细胞的代谢中心,参与许多必要的细胞过程,例如:氧化磷酸化,铁硫簇生成和凋亡调节16,17。这些内共生细胞器包含两个膜,称为内线粒体膜(IMM)和外线粒体膜(OMM)。超过99%的1,500种人类线粒体蛋白编码在核基因组中,需要在一个或两个不同的膜上易位18,19。因此,正确的线粒体功能取决于强大的蛋白质稳态网络来纠正蛋白质靶向或易位中的任何错误。
我们的实验室专注于称为尾锚(TA)蛋白的线粒体膜蛋白的子集,它们在C端20,21,22,23,24处具有单个跨膜结构域。TA蛋白参与许多基本过程,例如细胞凋亡,囊泡转运和蛋白质易位25。TA蛋白的独特拓扑需要翻译后插入,其发生在内质网(ER)中,通过尾锚(GET)或内质网膜蛋白复合物(EMC)途径的引导进入或通过表征不良的途径进入OMM 20,26,27,28。TMD的生物物理特性是必要且足以将TA蛋白引导到正确的膜29。对生物物理特征而不是定义的序列基序的识别限制了靶向途径的保真度5。因此,TA蛋白的错位是蛋白质稳态网络的常见压力。细胞应激,例如GET途径的抑制,导致OMM和线粒体功能障碍的蛋白质错位增加,除非这些蛋白质被迅速去除30,31。
膜蛋白平衡的一个共同主题是使用AAA+(ATP酶A与细胞Activities结合)蛋白从脂质双层中去除旧的,受损的或错位的蛋白质1,32,33,34,35,36,37,38 .AAA +蛋白是形成六聚环并经历ATP依赖性运动以重塑底物的分子马达,通常通过狭窄的轴向孔39,40易位。尽管在研究AAA+ATPases提取膜蛋白方面投入了大量精力,但重构是复杂的或涉及脂质和洗涤剂41,42的混合物,这限制了从脂质双层中提取底物的机制的实验能力。
Msp1是一种高度保守的AAA + ATP酶,锚定在OMM和过氧化物酶体中,通过去除错位的TA蛋白43,44,45,46,47在膜蛋白平衡中起关键作用。最近还显示,Msp1通过去除在OMM48易位期间停滞的膜蛋白来缓解线粒体蛋白导入应激。失去Msp1或人类同源物ATAD1导致线粒体碎片化,氧化磷酸化失败,癫痫发作,中风后损伤增加和早期死亡31,49,50,51,52,53,54,55,56。
我们已经证明,有可能与Msp1共同重建TA蛋白并检测从脂质双层57中提取。这个简化的系统使用完全纯化的蛋白质重组成定义的脂质体,模仿OMM(图1)58,59。这种水平的实验控制可以解决底物提取的详细机制问题,这些问题在实验上难以解决,涉及其他AAA +蛋白的更复杂的重构。在这里,我们提供了实验方案,详细介绍了我们的脂质体制备,膜蛋白重构和提取测定的方法。我们希望这些实验细节将有助于进一步研究膜蛋白平衡的基本但知之甚少的过程。
研究方案
1. 脂质体制备
- 以适当的比例混合脂质的氯仿储备,以模拟外线粒体膜。
- 准备25毫克脂质混合物。我们使用先前建立的模仿线粒体膜的脂质混合物,由48:28:10:10:4摩尔比的鸡蛋磷脂酰胆碱(PC),鸡蛋磷脂酰乙醇胺(PE),牛肝磷脂酰肌醇(PI),合成1,2-二油酰基-sn-甘油-3-磷酸-L-丝氨酸(DOPS)和合成1',3'-双[1,2-二油酰基-sn-甘油-3-磷酸]-甘油(TOCL)58,59组成。示例计算如表1所示。
- 开封前将所有脂质储备置于室温,因为这会限制冷凝。由于大多数实验室没有精确的方法来测量脂质的浓度,因此氯仿原液吸收的任何水都会改变脂质储备的浓度,从而改变测定中使用的脂质比例。
- 当脂质储备装在玻璃安瓿中时,使用1 mL注射器将所需量的脂质转移到玻璃小瓶中。向小瓶中加入2mg二硫代甲状腺素(DTT)以防止脂质氧化。快速工作,因为氯仿的蒸发会改变脂质的浓度。
- 将任何剩余的脂质转移到单独的玻璃瓶中,并与PFTE隔膜合合。向小瓶中加入2mg DTT,用石蜡膜包裹并储存在-20°C以防止脂质氧化。尝试在转移到小瓶后的3个月内使用脂质。为了防止标记径流对氯仿库存的潜在污染,请将贴纸从原始安瓿转移到玻璃小瓶上,而不是用标记标记标记。
- 在非常温和的氮气流下蒸发氯仿,同时在通风橱中用手连续旋转玻璃小瓶,基本上充当手动旋转蒸发器。用手以一致的速度(20-40 rpm)旋转小瓶,以保持脂质移动。目标是蒸发所有的氯仿,并在整个玻璃瓶上均匀地涂覆脂质。
注意:氯仿具有神经毒性,此步骤应在通风橱中进行。- 将新鲜的巴斯德移液器连接到氮气管上。不要让任何脂质从小瓶中溅出或溅到巴斯德移液器上。将尖端对准小瓶的底部,使空气从底部反弹并将脂质向上推向小瓶的中心。在整个小瓶上均匀涂覆,同时避免角落或盖子上积聚。整个过程大约需要5分钟。
- 当混合物变稠成脂质的"珠子"时,通过改变小瓶的角度将其引导到小瓶的中心。一旦珠子开始变小,稍微调高氮气流以分散珠子,确保没有脂质从小瓶中吹出。
- 在真空下除去任何剩余的氯仿。
- 将玻璃瓶放在室内真空或隔膜真空泵上1小时,以除去大部分残留的氯仿。这些真空通常不够强,无法去除所有的氯仿,但它们可以比旋转真空更好地耐受少量溶剂。
- 将小瓶放在强真空(<1mTorr)上12-16小时,以除去所有残留的氯仿。在此过程中,请务必避免小瓶磕碰。
- 将脂质重悬于1.25mL脂质体缓冲液(50mM HEPES KOH pH 7.5,15%甘油,1mM DTT)中。当我们从25毫克脂质开始时,这导致浓度为20毫克/ mL。完全重悬脂质,没有可见的块。如果脂质聚集在小瓶的角落,这可能是一个漫长的过程。
- 用力涡旋小瓶,直到样品呈乳白色光滑。我们发现,如果氯仿蒸发在前一天晚上正确完成,这个过程大约需要5-10分钟。
- 为确保脂质完全重悬,请在室温下以~80 rpm的速度在砂轮上旋转3小时。每小时从砂轮上取出一次小瓶,持续1分钟的涡旋,以确保均匀混合。
- 将脂质小心地转移到干净的1.5 mL微量离心管中。使用液氮冷冻和30°C加热块解冻进行5次冻融循环。此步骤有助于将多层囊泡转化为单层囊泡。
- 挤出脂质。
- 在冻融循环期间,准备小型挤出机。用10 mm过滤器支架和所需孔径的聚碳酸酯膜组装微型挤出机(我们使用200nm)。过滤器的大小会影响脂质体的大小,这将影响重组所需的蛋白质的浓度(步骤2.3)。
- 将小型挤出机放在热板上,使挤出机温度高达60°C。
- 将脂质吸入1 mL气密玻璃注射器中,并小心地放入小型挤出机的一端。将空的气密注射器放入小型挤出机的另一侧。让脂质平衡到挤出机静置温度5-10分钟。
- 通过轻轻推动填充注射器的柱塞,将脂质转移到备用注射器上。将溶液从备用注射器推入原始注射器中。重复这个来回过程15次,以便在第15次 通过时,脂质末端在备用注射器中。监控每次通过中的体积,以确保没有泄漏。
- 制备脂质的一次性等分试样,在液氮中快速冷冻并储存在-80°C。 脂质体在-80°C下稳定数月。重构需要一次10μL脂质体(步骤2.3.4),因此制备10μL或20μL等分试样很方便。
2 Msp1和TA型蛋白的重建
- 准备复原缓冲液:50 mM HEPES pH 7.5,200 mM乙酸钾,7 mM乙酸镁,2 mM DTT,10%蔗糖,0.01%叠氮化钠,0.2-0.8%脱氧大瓶(DBC)。
- 优化新一批脂质体的重构条件。最佳重构所需的DBC和生物珠的浓度因所使用的脂质体批次而异。要将制备限制为制备变异性,请对所有实验使用相同批次的 DBC。更改大量 DBC 时,请重复优化过程。
- 每次制备新一批脂质体时,用不同浓度的DBC(0.2%-0.8%)和生物珠(25mg-100mg)建立一系列重构。重要的是不要将DBC降至~0.12%的临界胶束浓度(CMC)以下。一旦条件得到优化,我们建议使用相同的脂质体制备和重建条件收集所有数据。
- 通过使用 步骤3中描述的提取测定法测定各种重构条件的有效性。
- 以250mg / mL的终浓度制备生物珠。
- 称出2.7g干燥的生物豆,并重悬于50 mL的100%甲醇(约45 mL)离心管中以润湿微珠。最初将生物珠润湿甲醇,以防止空气被困在珠子的孔隙中。一旦进入甲醇,保持生物珠湿,因为生物珠捕获的任何空气都会改变它们吸收洗涤剂的能力。
- 通过用约45 mL超纯水(18.2 mΩ)洗涤珠子8x来除去甲醇,以下称为ddH2O.沉淀珠,以3,200×g旋转1分钟。倾析液体并重悬于ddH2O中。
- 洗涤后,用0.02%叠氮化钠重悬于10ml ddH 2 O中并储存在4°C。 生物珠可以在4°C下储存数月。该储备液为250 mg / mL,因为假设在洗涤步骤中损失约0.2g。
- 计算脂质体的大小以及每个脂质体所需的TA蛋白和Msp1分子数。这将决定重建所需的Msp1和TA蛋白的浓度。
- 首先,使用以下等式计算每个单层脂质体的脂质分子数量(N总数
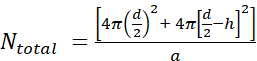 ),其中d是脂质体的直径,h是双层的厚度,a是脂质头群面积。
),其中d是脂质体的直径,h是双层的厚度,a是脂质头群面积。- 通过DLS测量脂质体直径。在我们的例子中,脂质体直径(d)的值为70nm。
- 使用 5 nm 的值表示 h,0.71 nm2 表示 a,这是磷脂酰胆碱的头基大小。在这种特殊情况下 ,N总数 为 37,610。
- 接下来,使用混合物中脂质的平均分子量计算脂质M脂质的摩尔浓度。在这个例子中,脂质的浓度是20毫克/毫升(步骤1.4),脂质的平均分子量是810克/摩尔(表1)。这导致M脂质的值为0.0247 M。
- 接下来,计算脂质体的摩尔浓度 ,M脂质体,使用等式
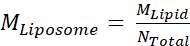 ,其中 M脂质 是 步骤2.3.2中脂质的摩尔浓度 ,N总 数是 步骤2.3.1中计算的每个脂质体的脂质总数。在本例中,脂质体的20mg / mL储备浓度约为660 nM。
,其中 M脂质 是 步骤2.3.2中脂质的摩尔浓度 ,N总 数是 步骤2.3.1中计算的每个脂质体的脂质总数。在本例中,脂质体的20mg / mL储备浓度约为660 nM。 - 计算100μL重构反应所需的Msp1和TA蛋白的量。
注意:重构中脂质的最终浓度为2mg / mL,这是脂质体原液的10倍稀释。这使得最终脂质体浓度为66 nM。Msp1的最终浓度为792 nM,平均每个脂质体总共12个拷贝(2个功能六聚体)。TA蛋白的最终浓度为660 nM,平均每个脂质体10个拷贝。
- 首先,使用以下等式计算每个单层脂质体的脂质分子数量(N总数
- 在PCR管中,将纯化的Msp1,TA蛋白和脂质体混合在复原缓冲液中。添加顺序是缓冲液,蛋白质和脂质体最后。总体积为100 μL。让混合物在冰上静置10分钟。如前所述纯化Msp1和TA蛋白57。
- 首次建立测定时,使用表征良好的模型TA蛋白His-Flag-Sumo-Sec22作为阳性对照底物。该结构具有易于纯化的His标签,用于通过蛋白质印迹检测的3x-Flag标签,用于增加溶解度的Sumo结构域,以及用于重建和识别Msp1的ER-TA蛋白Sec22的TMD。
- 确保Msp1和TA蛋白的储备溶液约为100μM,以尽量减少蛋白质纯化中N-十二烷基β-D-麦芽糖苷(DDM)对重建的影响。纯化的Msp1为20 mM HEPES pH 7.5,100mM氯化钠,0.1mM Tris(2-羧乙基)膦盐酸盐(TCEP),0.05%DDM,而纯化的TA蛋白在50mM Tris pH 7.4,150mM氯化钠,10 mM氯化镁,5mM β巯基乙醇,10%甘油,0.1%DDM中。约100μM TA蛋白和Msp1单体的储备溶液确保这些组分将占最终重构体积的 <5%,从而在CMC以下稀释DDM。
- 向样品中加入所需量的生物珠以除去洗涤剂。
- 将 p200 移液器吸头的吸头切成直径约1/8 英寸,以便磁珠可以穿过吸头。彻底涡旋生物珠管以获得均匀的混合物,并迅速取下盖子并在生物珠沉降之前移取体积。将生物珠转移到空的PCR管中。
- 当重构在冰上完成10分钟的孵育后,使用未切割的移液器吸头从生物珠中除去所有液体。然后将100μL重构转移到带有生物珠的管中。必须快速完成此操作,以便生物珠子不会捕获空气,这将导致珠子漂浮。
- 允许重构在4°C下以~80 rpm的速度在车轮上旋转16小时。
- 从生物珠中去除重组材料。在皮微中快速旋转以沉淀生物珠,然后使用未切割的移液管尖端将重构的材料转移到干净的PCR管中。重复此过程1-2次,直到样品中没有生物珠子。在冰上保持重建。
- 预先清除重组的材料,以除去任何未能重组到脂质体中的蛋白质。
- 准备提取缓冲液:50 mM HEPES pH 7.5,200 mM乙酸钾,7 mM乙酸镁,2 mM DTT,100 nM氯化钙。
- 根据制造商的说明,用提取缓冲液平衡谷胱甘肽离心柱。这通常涉及用400μL缓冲液洗涤3轮,然后在室温下以700×g离心2分钟以除去缓冲液。
- 将每个伴侣(GST-SGTA和GST-钙调蛋白)的5μM添加到重组材料中。这些伴侣将与任何未能重建到脂质体中的蛋白质的TMD结合。伴护者的纯化在前面6、57中描述过。
注意:这些伴侣是市售的,但我们更喜欢在内部净化,以控制成本和质量。两种蛋白质均为20 mM Tris pH 7.5,100 mM氯化钠和0.1mM TCEP,并且具有约160μM的储备浓度。SGTA识别具有高度疏水性TMD的底物,而Calmodulin以中等疏水性结合TMD6,29。总之,这种伴侣鸡尾酒可以识别广泛的底物。 - 向重组材料中加入100μL提取缓冲液,使体积达到200μL。将其添加到平衡的谷胱甘肽离心柱中。请注意,当预清除体积为200 – 400μL时,谷胱甘肽离心柱可提供最高的样品回收率。
- 插入离心柱,在4°C下以~80rpm旋转30分钟,以使伴侣与树脂结合。
- 在室温下以700×g旋转色谱柱2分钟。流经的是预先清除的物质,其聚集的蛋白质已经耗尽。将材料放在冰上,然后直接进行提取测定。
3. 提取测定
- 准备用于 SDS 页面分析的试管。每个反应将有4个管:输入(I),流过(FT),洗涤(W)和洗脱(E)。
- 将45μLdH2O样品加入输入管中,将40μLdH2O样品加入FLOW THROUGH管中,0μL加入WASH和洗脱管中。
注意:本测定中的最佳信噪比是当WASH和洗脱样品相对于输入和流过样品的浓度为5倍时获得的。由于测定过程中的稀释,这需要为输入样品取5μL样品,为FLOW THROUGH样品取10μL样品,为WASH和洗脱样品取50μL样品。 - 向每个试管中加入16.6μL的4x SDS PAGE上样缓冲液。在SDS PAGE上样缓冲液之前,每个样品的总体积为50μL。最终体积为66.6μL(50μL样品+ 16.6μL 4x SDS PAGE上样缓冲液)。
- 将45μLdH2O样品加入输入管中,将40μLdH2O样品加入FLOW THROUGH管中,0μL加入WASH和洗脱管中。
- 组装提取测定。
- 准备含有60μL预清除的蛋白脂质体,5μMGST-SGTA,5μMGST-钙调蛋白和2mM ATP的提取反应。结合除ATP以外的所有试剂,ATP用于引发反应。用提取缓冲液使最终体积达到200μL。
注意:由于每次提取测定使用60μL样品,因此一次重构可用于三种不同的提取测定。对来自相同重构的材料进行阳性和阴性对照(+ATP和-ATP)。 - 在30°C加热块中预热提取测定2分钟。
- 准备含有60μL预清除的蛋白脂质体,5μMGST-SGTA,5μMGST-钙调蛋白和2mM ATP的提取反应。结合除ATP以外的所有试剂,ATP用于引发反应。用提取缓冲液使最终体积达到200μL。
- 通过将ATP加入到2mM的最终浓度并开始计时器来启动提取测定。
- 在皮微中旋转5秒,将ATP混合到反应中。在30°C下孵育反应30分钟。
- 在孵育过程中,取5μL反应并加入INPUT管中。这样做的时机是灵活的。
- 在此孵育期间,在提取测定中为每个样品平衡一个谷胱甘肽离心柱。
- 对伴侣执行下拉以分离提取的材料。
- 一旦30分钟的孵育完成,向管中加入200μl提取缓冲液,使总体积达到400μL。加入平衡谷胱甘肽树脂中,在4°C的砂轮上结合30分钟。
- 在室温下以700×g旋转色谱柱2分钟以收集流经。取10μL用于流经管。该样品含有仍然集成在脂质双层中的底物。
- 用400μL提取缓冲液洗涤树脂两次,丢弃流经的液体。在第三次洗涤时,保持流量,并取50μL用于WASH管。
- 通过将还原谷胱甘肽加入提取缓冲液中5 mM的终浓度来制备5 mL洗脱缓冲液。每次都准备新鲜的此缓冲液。
- 向离心柱中加入200μL洗脱缓冲液。在室温下孵育5分钟。在室温下以700 x g 旋转2分钟以洗脱。保持流程通过。重复该过程第二次,使总洗脱体积为400μL。
- 从洗脱样品中取50μL样品并将其加入洗脱管中。
- 使用 SDS-PAGE 和蛋白质印迹分析提取活性。
注意:由于蛋白质印迹是一种相当标准的程序,因此提供了一个基本方案,突出了该测定的一些独特细节。- 将样品加载到无污渍的聚丙烯酰胺凝胶中(4%堆叠,15%分离),并在Tris-甘氨酸缓冲液中以200 V运行50分钟。如果空间允许,请使用未染色和染色的梯子,以便在无污渍凝胶成像仪上进行可视化,并分别转移到PVDF膜上。无染色凝胶允许在用紫外线活化时定量可视化含有色氨酸的蛋白质,同时仍然允许凝胶用于蛋白质印迹。
- 对无污渍凝胶进行成像,以确认所有样品的上样量相等。这是一项基本控制措施,可确保蛋白质印迹检测到的任何信号变化都不是可变蛋白质上样的结果。在输入和洗脱样品中,只能有伴随体(GST-钙调蛋白和GST-SGTA)的可见条带。回想一下,洗脱样品将比输入样品更浓缩。
- 使用45μm PVDF膜组装蛋白质印迹盒。以 300 mA 的恒定电流传输 60 分钟。
- 阻断膜后,在4°C下与一抗结合16小时,轻轻摇动〜15rpm。用兔抗 FLAG 以 1:1,000 稀释度进行底物印迹。
注意:使用的一抗和二抗将是底物特异性的,并且可能需要优化使用的浓度。 - 洗净膜并用二抗孵育,山羊抗兔,以1:10,000稀释,在室温下轻轻摇动1小时。
- 使用蛋白质印迹检测剂洗涤膜和图像进行分析。
结果
为了正确解释结果,必须同时观察无污渍凝胶和蛋白质印迹。无污渍凝胶可确保所有样品的上样量相等。当观察无污渍凝胶时,陪同人员(GST-钙调蛋白和GST-SGTA)将在输入(I)和洗脱(E)通道中可见。仔细检查这些波段的强度在所有 INPUT 样本中是否均匀。同样,确保洗脱样品的强度均匀。洗脱液的浓度是INPUT的5倍,这种强度差异在凝胶中是可见的。
使用无?...
讨论
正确的线粒体功能取决于强大的蛋白质质量控制系统。由于TA蛋白靶向途径的保真度的固有限制,错位的TA蛋白是线粒体的持续应激源。线粒体蛋白质平衡网络的一个关键组成部分是Msp1,它是一种膜锚定的AAA + ATP酶,可从OMM中去除错位的TA蛋白。在这里,我们描述了如何制备蛋白脂质体,共同重建Msp1和模型TA蛋白,并进行提取测定。我们之前使用该测定来证明Msp1直接识别错位的TA蛋白,并且能够在...
披露声明
没有
致谢
MLW在芝加哥大学与Robert Keenan博士的博士后研究期间开发了该协议的一部分。
这项工作由NIH资助1R35GM137904-01给MLW。
材料
| Name | Company | Catalog Number | Comments |
| Biobeads | Bio-Rad | 1523920 | |
| Bovine liver phosphatidyl inositol | Avanti | 840042C | PI |
| Chicken egg phosphatidyl choline | Avanti | 840051C | PC |
| Chicken egg phosphatidyl ethanolamine | Avanti | 840021C | PE |
| ECL Select western blotting detection reagent | GE | RPN2235 | |
| Filter supports | Avanti | 610014 | |
| Glass vial | VWR | 60910L-1 | |
| Glutathione spin column | Thermo Fisher | PI16103 | |
| Goat anti-rabbit | Thermo Fisher | NC1050917 | |
| Mini-Extruder | Avanti | 610020 | |
| Polycarbonate membrane | Avanti | 610006 | 200 nM |
| PVDF membrane | Thermo Fisher | 88518 | 45 µM |
| Rabbit anti-FLAG | Sigma-Aldrich | F7245 | |
| Synthetic 1,2-dioleoyl-sn-glycero-3-phospho-L-serine | Avanti | 840035C | DOPS |
| Synthetic 1',3'-bis[1,2-dioleoyl-sn-glycero-3-phospho]-glycerol | Avanti | 710335C | TOCL |
| Syringe, 1 mL | Norm-Ject | 53548-001 | |
| Syringe, 1 mL, gas-tight | Avanti | 610017 |
参考文献
- Song, J., Herrmann, J. M., Becker, T. Quality control of the mitochondrial proteome. Nature Reviews Molecular Cell Biology. 22, 54-70 (2021).
- Phillips, B. P., Miller, E. A. Membrane protein folding and quality control. Current Opinion in Structural Biology. 69, 50-54 (2021).
- Jiang, H. Quality control pathways of tail-anchored proteins. Biochimica et Biophysica Acta - Molecular Cell Research. 1868, 118922 (2020).
- McKenna, M. J., et al. The endoplasmic reticulum P5A-ATPase is a transmembrane helix dislocase. Science. 369, (2020).
- Hegde, R. S., Zavodszky, E. Recognition and Degradation of Mislocalized Proteins in Health and Disease. Cold Spring Harbor Perspectives in Biology. 11, 033902 (2019).
- Shao, S., Hegde, R. S. A calmodulin-dependent translocation pathway for small secretory proteins. Cell. 147, 1576-1588 (2011).
- Samuelson, J. C., et al. YidC mediates membrane protein insertion in bacteria. Nature. 406, 637-641 (2000).
- Anghel, S. A., McGilvray, P. T., Hegde, R. S., Keenan, R. J. Identification of Oxa1 Homologs Operating in the Eukaryotic Endoplasmic Reticulum. Cell Reports. 21, 3708-3716 (2017).
- Aviram, N., et al. The SND proteins constitute an alternative targeting route to the endoplasmic reticulum. Nature. 540, 134-138 (2016).
- Voorhees, R. M., Hegde, R. S. Structure of the Sec61 channel opened by a signal sequence. Science. 351, 88-91 (2016).
- Cichocki, B. A., Krumpe, K., Vitali, D. G., Rapaport, D. Pex19 is involved in importing dually targeted tail-anchored proteins to both mitochondria and peroxisomes. Traffic. 19, 770-785 (2018).
- Mateja, A., et al. Protein targeting. Structure of the Get3 targeting factor in complex with its membrane protein cargo. Science. 347, 1152-1155 (2015).
- Chacinska, A., Koehler, C. M., Milenkovic, D., Lithgow, T., Pfanner, N. Importing mitochondrial proteins: machineries and mechanisms. Cell. 138, 628-644 (2009).
- Chitwood, P. J., Hegde, R. S. An intramembrane chaperone complex facilitates membrane protein biogenesis. Nature. , (2020).
- Chitwood, P. J., Juszkiewicz, S., Guna, A., Shao, S., Hegde, R. S. EMC Is Required to Initiate Accurate Membrane Protein Topogenesis. Cell. 175, 1-30 (2018).
- Bock, F. J., Tait, S. W. G. Mitochondria as multifaceted regulators of cell death. Nature Reviews Molecular Cell Biology. 21, 85-100 (2020).
- Pfanner, N., Warscheid, B., Wiedemann, N. Mitochondrial proteins: from biogenesis to functional networks. Nature Reviews Molecular Cell Biology. 20, (2019).
- Bykov, Y. S., Rapaport, D., Herrmann, J. M., Schuldiner, M. Cytosolic Events in the Biogenesis of Mitochondrial Proteins. Trends in Biochemical Sciences. 45, 650-667 (2020).
- Pfanner, N., Warscheid, B., Wiedemann, N. Mitochondrial proteins: from biogenesis to functional networks. Nature Reviews Molecular Cell Biology. 427, 1135 (2019).
- Borgese, N., Coy-Vergara, J., Colombo, S. F., Schwappach, B. The Ways of Tails: the GET Pathway and more. The Protein Journal. , 1-17 (2019).
- Mateja, A., Keenan, R. J. A structural perspective on tail-anchored protein biogenesis by the GET pathway. Current Opinion in Structural Biology. 51, 195-202 (2018).
- Chio, U. S., Cho, H., Shan, S. Mechanisms of Tail-Anchored Membrane Protein Targeting and Insertion. Annual review of cell and developmental biology. 33, 417-438 (2017).
- Denic, V. A portrait of the GET pathway as a surprisingly complicated young man. Trends in biochemical sciences. , (2012).
- Hegde, R. S., Keenan, R. J. Tail-anchored membrane protein insertion into the endoplasmic reticulum. Nature Reviews Molecular Cell Biology. 12, 787-798 (2011).
- Kalbfleisch, T., Cambon, A., Wattenberg, B. W. A bioinformatics approach to identifying tail-anchored proteins in the human genome. Traffic. 8, 1687-1694 (2007).
- Doan, K. N., et al. The Mitochondrial Import Complex MIM Functions as Main Translocase for α-Helical Outer Membrane Proteins. Cell Reports. 31, (2020).
- McDowell, M. A., et al. Structural Basis of Tail-Anchored Membrane Protein Biogenesis by the GET Insertase Complex. Molecular Cell. 80, (2020).
- Guna, A., Volkmar, N., Christianson, J. C., Hegde, R. S. The ER membrane protein complex is a transmembrane domain insertase. Science. 591, 3099 (2017).
- Rao, M., et al. Multiple selection filters ensure accurate tail-anchored membrane protein targeting. eLife. 5, 21301 (2016).
- Schuldiner, M., et al. The GET complex mediates insertion of tail-anchored proteins into the ER membrane. Cell. 134, 634-645 (2008).
- Chen, Y. -. C., et al. Msp1/ATAD1 maintains mitochondrial function by facilitating the degradation of mislocalized tail-anchored proteins. The EMBO journal. 33, 1548-1564 (2014).
- Wu, X., Rapoport, T. A. Translocation of Proteins through a Distorted Lipid Bilayer. Trends in Cell Biology. , (2021).
- Phillips, B. P., Gomez-Navarro, N., Miller, E. A. Protein quality control in the endoplasmic reticulum. Current Opinion in Cell Biology. 65, 96-102 (2020).
- van de Weijer, M. L., et al. Quality Control of ER Membrane Proteins by the RNF185/Membralin Ubiquitin Ligase Complex. Molecular Cell. 79, (2020).
- Weir, N. R., Kamber, R. A., Martenson, J. S., Denic, V. The AAA protein Msp1 mediates clearance of excess tail-anchored proteins from the peroxisomal membrane. eLife. 6, 28507 (2017).
- Gardner, B. M., et al. The peroxisomal AAA-ATPase Pex1/Pex6 unfolds substrates by processive threading. Nature communications. 9, 135 (2018).
- Puchades, C., et al. Unique Structural Features of the Mitochondrial AAA+ Protease AFG3L2 Reveal the Molecular Basis for Activity in Health and Disease. Molecular Cell. , (2019).
- Castanzo, D. T., LaFrance, B., Martin, A. The AAA+ ATPase Msp1 is a processive protein translocase with robust unfoldase activity. Proceedings of the National Academy of Sciences of the United States of America. 117, 14970-14977 (2020).
- Wang, L., Myasnikov, A., Pan, X., Walter, P. Structure of the AAA protein Msp1 reveals mechanism of mislocalized membrane protein extraction. eLife. 9, (2020).
- Puchades, C., Sandate, C. R., Lander, G. C. The molecular principles governing the activity and functional diversity of AAA+ proteins. Nature Reviews Molecular Cell Biology. , 1-16 (2019).
- Yang, Y., et al. Folding-Degradation Relationship of a Membrane Protein Mediated by the Universally Conserved ATP-Dependent Protease FtsH. Journal of the American Chemical Society. , 10 (2018).
- Baldridge, R. D., Rapoport, T. A. Autoubiquitination of the Hrd1 Ligase Triggers Protein Retrotranslocation in ERAD. Cell. 166, 394-407 (2016).
- Fresenius, H. L., Wohlever, M. L. Sorting out how Msp1 maintains mitochondrial membrane proteostasis. Mitochondrion. 49, 128-134 (2019).
- Wang, L., Walter, P. Msp1/ATAD1 in Protein Quality Control and Regulation of Synaptic Activities. Annual Review of Cell and Developmental Biology. 36, 1-24 (2020).
- Dederer, V., et al. Cooperation of mitochondrial and ER factors in quality control of tail-anchored proteins. eLife. 8, 1126 (2019).
- Matsumoto, S., et al. Msp1 Clears Mistargeted Proteins by Facilitating Their Transfer from Mitochondria to the ER. Molecular Cell. , (2019).
- Li, L., Zheng, J., Wu, X., Jiang, H. Mitochondrial AAA-ATPase Msp1 detects mislocalized tail-anchored proteins through a dual-recognition mechanism. EMBO Reports. 20, (2019).
- Weidberg, H., Amon, A. MitoCPR - a surveillance pathway that protects mitochondria in response to protein import stress. Science. 360, (2018).
- Okreglak, V., Walter, P. The conserved AAA-ATPase Msp1 confers organelle specificity to tail-anchored proteins. Proceedings of the National Academy of Sciences of the United States of America. 111, (2014).
- Piard, J., et al. A homozygous ATAD1 mutation impairs postsynaptic AMPA receptor trafficking and causes a lethal encephalopathy. Brain. , (2018).
- Zhang, J., et al. The AAA+ ATPase Thorase regulates AMPA receptor-dependent synaptic plasticity and behavior. Cell. 145, 284-299 (2011).
- Prendergast, J., et al. Ganglioside regulation of AMPA receptor trafficking. The Journal of Neuroscience. 34, 13246-13258 (2014).
- Umanah, G. K. E., et al. Thorase variants are associated with defects in glutamatergic neurotransmission that can be rescued by Perampanel. Science Translational Medicine. 9, 4985 (2017).
- Pignatelli, M., et al. Synaptic Plasticity onto Dopamine Neurons Shapes Fear Learning. Neuron. 93, 425-440 (2017).
- Zhang, J., et al. The AAA Thorase is neuroprotective against ischemic injury. Journal of Cerebral Blood Flow and Metabolism. , 271678 (2018).
- Umanah, G. K. E., et al. AMPA Receptor Surface Expression Is Regulated by S-Nitrosylation of Thorase and Transnitrosylation of NSF. Cell Reports. 33, 108329 (2020).
- Wohlever, M. L., Mateja, A., McGilvray, P. T., Day, K. J., Keenan, R. J. Msp1 Is a Membrane Protein Dislocase for Tail-Anchored Proteins. Molecular Cell. 67, 194-202 (2017).
- Lovell, J. F., et al. Membrane binding by tBid initiates an ordered series of events culminating in membrane permeabilization by Bax. Cell. 135, 1074-1084 (2008).
- Leshchiner, E. S., Braun, C. R., Bird, G. H., Walensky, L. D. Direct activation of full-length proapoptotic BAK. Proceedings of the National Academy of Sciences of the United States of America. 110, 986-995 (2013).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。