É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Reconstituição da Atividade de Extração de Msp1 com Componentes Totalmente Purificados
Neste Artigo
Resumo
Aqui, apresentamos um protocolo detalhado para a reconstituição da atividade extração de Msp1 com componentes totalmente purificados em proteoliposomes definidos.
Resumo
Como centro de fosforilação oxidativa e regulação apoptótica, as mitocôndrias desempenham um papel vital na saúde humana. A função mitocondrial adequada depende de um sistema robusto de controle de qualidade para manter a homeostase proteica (proteostase). Declínios na proteostase mitocondrial têm sido ligados ao câncer, envelhecimento, neurodegeneração e muitas outras doenças. Msp1 é um AAA+ ATPase ancorado na membrana mitocondrial externa que mantém a proteostase removendo proteínas ancoradas na cauda deslocalizadas. Utilizando componentes purificados reconstituídos em proteoliposomes, mostramos que o Msp1 é necessário e suficiente para extrair um modelo de proteína ancorada na cauda de uma bicamada lipídica. Nosso sistema simplificado reconstituído supera várias das barreiras técnicas que têm dificultado o estudo detalhado da extração de proteínas de membrana. Aqui, fornecemos métodos detalhados para a geração de lipossomos, reconstituição de proteínas de membrana e o ensaio de extração de Msp1.
Introdução
A função celular adequada depende de um processo chamado proteostase, que garante que as proteínas funcionais estejam na concentração correta e localização celular1. Falhas na proteostase levam à função organela comprometida e estão associadas a muitas doenças neurodegenerativas2,3,4. As proteínas de membrana apresentam desafios únicos à rede de proteostase, pois devem ser direcionadas para a membrana correta, evitando a agregação dos domínios hidrofóbicos transmembrano (TMDs)5. Consequentemente, o maquinário especializado evoluiu para proteger o TMD hidrofóbico do citosol e facilitar a segmentação e inserção na membrana celular adequada6,7,8,9,10,11,12,13,14,15.
Mitocôndrias são o centro metabólico da célula e estão envolvidas em inúmeros processos celulares essenciais, tais como: fosforilação oxidativa, geração de aglomerados ferro-enxofre e regulação apoptótica16,17. Essas organelas endossímbiticas contêm duas membranas, conhecidas como a membrana mitocondrial interna (IMM) e a membrana mitocondrial externa (OMM). Mais de 99% das 1.500 proteínas mitocondriais humanas estão codificadas no genoma nuclear e precisam ser translocadas através de uma ou duas membranas diferentes18,19. A função mitocondrial adequada depende, portanto, de uma rede robusta de proteostase para corrigir quaisquer erros na segmentação ou translocação de proteínas.
Nosso laboratório se concentra em um subconjunto de proteínas de membrana mitocondrial chamadas proteínas ancoradas na cauda (TA), que têm um único domínio transmembrano no próprio C-terminus20,21,22,23,24. As proteínas TA estão envolvidas em uma série de processos essenciais, como apoptose, transporte de vesículas e translocação de proteínas25. A topologia única das proteínas TA requer a inserção pós-translacional, que ocorre no ânticulo endoplasmático (ER) pela Entrada Guiada de Extremidades Ancoradas (GET) ou Complexo de Proteína de Membrana Ticúrica Endoplasmática (EMC) ou no OMM por uma via mal caracterizada20,26,27,28. As propriedades biofísicas do TMD são necessárias e suficientes para guiar as proteínas TA para a membrana correta29. O reconhecimento de características biofísicas em vez de um motivo de sequência definida limita a fidelidade das vias de segmentação5. Assim, a málocalização das proteínas TA é um estresse comum para as redes de proteostase. O estresse celular, como a inibição da via GET, causa um aumento da mislocalização da proteína à OMM e disfunção mitocondrial, a menos que essas proteínas sejam prontamente removidas30,31.
Um tema comum na proteostase da membrana é o uso de proteínas AAA+ (ATPase Associated com ctivities celular A)proteínas para remover proteínas antigas, danificadas ou mislocalizadas da bicamada lipídica1,32,33,34,35,36,37,38 . As proteínas AAA+ são motores moleculares que formam anéis hexamericos e sofrem movimentos dependentes de ATP para remodelar um substrato, muitas vezes por translocação através de um poro axial estreito39,40. Embora grande esforço tenha sido dedicado ao estudo da extração de proteínas de membrana por ATPases AAA+, as reconstituições são complexas ou envolvem uma mistura de lipídios e detergente41,42, que limita o poder experimental de examinar o mecanismo de extração de substrato a partir da bicamada lipídica.
Msp1 é um ATPase AAA+ altamente conservado ancorado no OMM e peroxisoos que desempenha um papel crítico na proteostase da membrana, removendo proteínas TA mal localizadas43,44,45,46,47. O MSP1 também foi recentemente mostrado para aliviar o estresse de importação de proteínas mitocondriais, removendo proteínas de membrana que param durante a translocação através da OMM48. A perda de Msp1 ou o homólogo humano ATAD1 resulta em fragmentação mitocondrial, falhas na fosforilação oxidativa, convulsões, aumento da lesão após acidente vascular cerebral e morte precoce31,49,50,51,52,53,54,55,56.
Mostramos que é possível co-reconstituir proteínas TA com Msp1 e detectar a extração da bicamadas lipídica57. Este sistema simplificado utiliza proteínas totalmente purificadas reconstituídas em lipossomos definidos que imitam o OMM (Figura 1)58,59. Esse nível de controle experimental pode abordar questões mecanicistas detalhadas da extração de substratos que são experimentalmente intratáveis com reconstituições mais complexas envolvendo outras proteínas AAA+. Aqui, fornecemos protocolos experimentais detalhando nossos métodos de preparação de lipossomos, reconstituição de proteínas de membrana e o ensaio de extração. Esperamos que esses detalhes experimentais facilitem um estudo mais aprofundado do processo essencial, mas mal compreendido, da proteostase da membrana.
Protocolo
1. Preparação liposomial
- Combine estoques de clorofórmio de lipídios em proporções apropriadas para imitar a membrana mitocondrial externa.
- Prepare 25 mg de mistura lipídica. Usamos uma mistura previamente estabelecida de lipídios que imitam membranas mitocondriais, constituído por uma razão molar de 48:28:10:10:4 de cerline de ovo de galinha fosfartidil (PC), ovo de frango fosfatidyl etanolamina (PE), inositol de fosfatidil hepático bovino (PI), sintético 1,2-dioleoyl-sn-glycero-3-phospho-L-serine (DOPS), e sintético 1',3'-bis[1,2-dioleoyl-sn-glycero-3-phospho]-glicerol (TOCL)58,59. Os cálculos amostrais são mostrados na Tabela 1.
- Leve todos os estoques lipídes para a temperatura ambiente antes de abrir, pois isso limitará a condensação. Como a maioria dos laboratórios não tem uma maneira precisa de medir a concentração dos lipídios, qualquer água absorvida pelo estoque de clorofórmio mudará a concentração do caldo lipíduo e, portanto, a razão de lipídios usados no ensaio.
- Como os estoques lipídicos vêm em ampolas de vidro, transfira a quantidade necessária de lipídio para um frasco de vidro usando uma seringa de 1 mL. Adicione 2 mg de dithiothreitol (DTT) ao frasco para evitar a oxidação lipídica. Trabalhe rapidamente, pois a evaporação do clorofórmio mudará a concentração do lipídio.
- Transfira qualquer lipídio restante para um frasco de vidro separado e encaixe com um septa PFTE. Adicione 2 mg de DTT ao frasco, enrole com parafilme e armazene a -20 °C para evitar a oxidação lipídica. Tente usar os lipídios dentro de 3 meses após a transferência para os frascos. Para evitar a contaminação potencial dos estoques de clorofórmio por escoamento de marcadores, transfira os adesivos das ampolas originais para os frascos de vidro em vez de rotular com marcador.
- Evaporar clorofórmio sob um fluxo muito suave de nitrogênio enquanto gira o frasco de vidro continuamente à mão em um capô de fumaça, essencialmente agindo como um rotovap manual. Gire o frasco a uma velocidade consistente (20-40 rpm) à mão para manter os lipídios em movimento. O objetivo é evaporar todo o clorofórmio e obter um revestimento uniforme de lipídios sobre todo o frasco de vidro.
ATENÇÃO: O clorofórmio é neurotóxico, e este passo deve ser realizado no capô da fumaça.- Conecte uma pipeta pasteur fresca ao tubo de nitrogênio. Não deixe nenhum dos lipídios espirrar para fora do frasco ou para a pipeta Pasteur. Aponte a ponta para a parte inferior do frasco para que o ar salte por baixo e empurre os lipídios para o centro do frasco. Obtenha um revestimento uniforme sobre todo o frasco, evitando qualquer acúmulo nos cantos ou pela tampa. Todo esse processo leva cerca de 5 minutos.
- À medida que a mistura engrossa em uma "pérola" de lipídios, guie-a para o centro do frasco alterando o ângulo do frasco. Uma vez que a conta começa a ficar menor, aumente ligeiramente o fluxo de nitrogênio para dispersar a conta, garantindo que nenhum dos lipídios exploda para fora do frasco.
- Remova qualquer clorofórmio restante sob vácuo.
- Coloque o frasco de vidro em uma bomba de vácuo de casa ou diafragma por 1 hora para remover a maioria do clorofórmio residual. Estes vácuos geralmente não são fortes o suficiente para remover todo o clorofórmio, mas eles podem tolerar pequenas quantidades de solvente melhor do que vácuos rotativos-vaidosos.
- Coloque o frasco em um vácuo forte (<1 mTorr) por 12-16 h para remover todo o clorofórmio residual. Certifique-se de evitar o colisão do frasco durante este processo.
- Resuspende os lipídios em 1,25 mL de tampão lipossomo (50 mM HEPES KOH pH 7,5, 15% glicerol, 1 mM DTT). Como começamos com 25 mg de lipídios, isso resulta em uma concentração de 20 mg/mL. Resuspende totalmente os lipídios sem pedaços visíveis. Se os lipídios se agruparam no canto do frasco, este pode ser um processo demorado.
- Vórtice o frasco vigorosamente até que a amostra esteja leitosa suave. Descobrimos que se a evaporação do clorofórmio foi feita corretamente na noite anterior, esse processo leva cerca de 5-10 minutos.
- Para garantir a ressuspensão completa dos lipídios, gire em uma roda à temperatura ambiente por 3 horas a ~80 rpm. Remova o frasco da roda uma vez a cada hora durante 1 minuto de vórtice para garantir a mistura uniforme.
- Transfira os lipídios cuidadosamente para um tubo de microcentrífugo limpo de 1,5 mL. Realize 5 ciclos de degelo congelado usando nitrogênio líquido para congelar e um bloco de calor de 30 °C para descongelar. Esta etapa ajuda a converter vesículas multilamellar em vesículas unilamellar.
- Extrude os lipídios.
- Durante os ciclos de congelamento, prepare o mini-extrusor. Monte o mini-extrusor com suportes de filtro de 10 mm e uma membrana de policarbonato do tamanho do poro desejado (usamos 200 nm). O tamanho do filtro afetará o tamanho do lipossomo, o que afetará a concentração de proteínas necessárias para a reconstituição (Passo 2.3).
- Coloque o mini-extrusor em uma placa quente e leve a temperatura da extrusora até 60 °C.
- Desenhe os lipídios em uma seringa de vidro de 1 mL e coloque cuidadosamente em uma extremidade da mini-extrusora. Coloque a seringa vazia e apertada no outro lado da mini-extrusora. Deixe que os lipídios se equilibrem até a temperatura do suporte da extrusora por 5-10 minutos.
- Transfira os lipídios para a seringa alternativa empurrando suavemente o êmbolo da seringa encheu. Empurre a solução da seringa alternativa para a seringa original. Repita este processo de ida e volta 15 vezes, de modo que no15º passem os lipídios terminam na seringa alternativa. Monitore o volume em cada passe para ter certeza de que não há vazamentos.
- Prepare alíquotas de uso único dos lipídios, congele em nitrogênio líquido e armazene a -80 °C. Os lipossomos estão estáveis a -80 °C por vários meses. As reconstituiçãos exigem 10 μL de lipossomos por vez (Passo 2.3.4), por isso é conveniente preparar 10 μL ou 20 μL alíquotas.
2 Reconstituição da proteína Msp1 e Modelo TA
- Prepare o tampão de reconstituição: 50 mM HEPES pH 7,5, 200 mM de acetato de potássio, acetato de magnésio de 7 mM, 2 mM DTT, 10% sacarose, 0,01% azide de sódio, 0,2-0,8% Deoxy Big Chaps (DBC).
- Otimize as condições de reconstituição para o novo lote de lipossomos. A concentração de DBC e bioesferas necessárias para a reconstituição ideal varia dependendo do lote de lipossomos utilizados. Para limitar a preparação para a variabilidade de preparação, use o mesmo lote de DBC para todos os experimentos. Ao alterar muito dBC, repita o processo de otimização.
- Estabeleceu uma série de reconstituiçãos com diferentes concentrações de DBC (0,2% - 0,8%) e bioesferas (25 mg - 100 mg) cada vez que um novo lote de lipossomos é preparado. É importante não deixar o DBC abaixo da concentração crítica de micelas (CMC) de ~0,12%. Uma vez que as condições sejam otimizadas, recomendamos coletar todos os dados usando as mesmas condições de preparação e reconstituição liposso.
- Ensaio a eficácia das várias condições de reconstituição utilizando o ensaio de extração descrito no Passo 3.
- Prepare bioesferas em uma concentração final de 250 mg/mL.
- Pesar 2,7 g de bioesferas secas e resuspend em um tubo centrífuga de 50 mL de 100% metanol (cerca de 45 mL) para molhar as contas. Inicialmente molhe as bioesferas em metanol para evitar que o ar fique preso nos poros das contas. Uma vez no metanol, mantenha as bioesferas molhadas como qualquer ar preso pelas bioesferas alterará sua capacidade de absorver detergente.
- Remova o metanol lavando as contas 8x com cerca de 45 mL de água ultrapura (18,2 mΩ), daqui em diante referidas como ddH2O. Contas de pelota girando a 3.200 x g por 1 minuto. Decante o líquido e resuspend em ddH2O.
- Após a lavagem, resuspense em 10 ml de ddH2O com azida de sódio de 0,02% e armazene a 4 °C. As bioesferas podem ser armazenadas a 4 °C por vários meses. Este estoque é de 250 mg/mL, pois presume-se que ~0,2 g é perdido durante as etapas de lavagem.
- Calcule o tamanho do lipossomo e o número desejado de moléculas de proteína TA e Msp1 por lipossomo. Isso determinará a concentração de proteína Msp1 e TA necessária para a reconstituição.
- Primeiro, calcule o número de moléculas lipídicas por lipóculos unilamellar(Ntotal) usando a equação
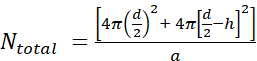 onde d é o diâmetro do lipossomo, h é a espessura da bicamada, e a é a área do grupo de cabeça lipídica.
onde d é o diâmetro do lipossomo, h é a espessura da bicamada, e a é a área do grupo de cabeça lipídica.- Meça o diâmetro do lipossomo por DLS. Em nosso exemplo, obteve-se um valor de 70 nm para o diâmetro lipossomo(d).
- Use um valor de 5 nm para h e 0,71 nm2 para um, que é o tamanho do headgroup para fosfattidylcholina. Nesta situação em particular, ntotal é de 37.610.
- Em seguida, calcule a concentração molar de lipídio mlipídio usando o peso molecular médio dos lipídios na mistura. Neste exemplo, a concentração de lipídios é de 20 mg/mL (Passo 1.4) e o peso molecular médio dos lipídios é de 810 g/mol(Tabela 1). Isso resulta em um valor de 0,0247 M para MLipid.
- Em seguida, calcule a concentração molar de lipossomos, MLiposome, utilizando a equação
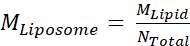 onde M Lipid é a concentração molar de lipídio da Etapa 2.3.2, e Ntotal é o número total de lipídios por liposome calculado na Etapa 2.3.1. Neste exemplo, a concentração de 20 mg/mL de lipossomos é de aproximadamente 660 nM.
onde M Lipid é a concentração molar de lipídio da Etapa 2.3.2, e Ntotal é o número total de lipídios por liposome calculado na Etapa 2.3.1. Neste exemplo, a concentração de 20 mg/mL de lipossomos é de aproximadamente 660 nM. - Calcule a quantidade de proteína Msp1 e TA necessária para uma reação de reconstituição de 100 μL.
NOTA: A concentração final de lipídios na reconstituição é de 2 mg/mL, que é uma diluição de 10x do caldo lipossomo. Isso dá uma concentração liposemática final de 66 nM. A concentração final de Msp1 é de 792 nM, o que dá uma média de 12 cópias totais (2 hexamers funcionais) por liposesomo. A concentração final da proteína TA é de 660 nM, o que dá uma média de 10 cópias por lipososom.
- Primeiro, calcule o número de moléculas lipídicas por lipóculos unilamellar(Ntotal) usando a equação
- Em um tubo PCR, misture msp1 purificado, proteína TA e lipossomos em tampão de reconstituição. A ordem de adição é tampão, proteínas e lipossomos duram. O volume total é de 100 μL. Deixe a mistura sentar no gelo por 10 minutos. Purifique a proteína Msp1 e TA como descrito anteriormente57.
- Use a bem caracterizada proteína TA modelo His-Flag-Sumo-Sec22 como um substrato de controle positivo ao estabelecer o ensaio pela primeira vez. Esta construção tem uma tag His-tag para fácil purificação, tag 3x-Flag para detecção por mancha ocidental, um domínio de Sumô para maior solubilidade, e o TMD da proteína ER-TA Sec22 para reconstituição e reconhecimento por Msp1.
- Certifique-se de que as soluções de estoque tanto de Msp1 quanto da proteína TA são de aproximadamente 100 μM para minimizar o efeito do N-Dodecyl β-D-maltoside (DDM) da purificação de proteínas na reconstituição. O Msp1 purificado está em 20 mM HEPES pH 7.5, 100 mM cloreto de sódio, 0,1 mM Tris (2-carboxyethyl)cloridrato de fosfina (TCEP), 0,05% DDM enquanto a proteína TA purificada está em 50 mM Tris pH 7.4, Cloreto de sódio de 150 mM, cloreto de magnésio de 10 mM, 5 mM β-mercaptoetanol, 10% glicerol, 0,1% DDM. Soluções de estoque de aproximadamente 100 μM de proteína TA e monômero Msp1 garantem que esses componentes comporão < 5% do volume final de reconstituição, resultando em uma diluição do DDM abaixo do CMC.
- Adicione a quantidade desejada de bioesferas à amostra para remover o detergente.
- Corte a ponta de uma ponta de pipeta p200 para cerca de 1/8de polegada de diâmetro para que as contas possam caber através da ponta. Vórtice o tubo bioesfera para obter uma mistura uniforme e remover rapidamente a tampa e pipeta até o volume antes que as bioesferas se instalem. Transfira as bioesferas para um tubo PCR vazio.
- Quando a reconstituição terminar sua incubação de 10 minutos no gelo, use uma ponta de pipeta sem cortes para remover todo o líquido das bioesferas. Em seguida, transfira a reconstituição de 100 μL para o tubo com as bioesferas. Isso deve ser feito rapidamente para que as bioesferas não presençam o ar, o que fará com que as contas flutuem.
- Deixe a reconstituição girar em uma roda a ~80 rpm por 16 horas a 4 °C.
- Remova o material reconstituído das bioesferas. Faça um giro rápido em um picofuge para pelotar as bioesferas e, em seguida, use uma ponta de tubulação sem cortes para transferir o material reconstituído para um tubo PCR limpo. Repita este processo 1-2 vezes até que não haja bioesferas na amostra. Mantenha a reconstituição no gelo.
- Pré-limpe o material reconstituído para remover quaisquer proteínas que não se reconstituíssem nos lipossomos.
- Prepare o tampão de extração: 50 mM HEPES pH 7.5, 200 mM de acetato de potássio, acetato de magnésio de 7 mM, 2 mM DTT, cloreto de cálcio de 100 nM.
- Equilibre as colunas de giro de glutationa com buffer de extração de acordo com as instruções do fabricante. Isso normalmente envolve 3 rodadas de lavagem com 400 μL de tampão e, em seguida, centrifugação a 700 x g por 2 minutos em temperatura ambiente para remover o buffer.
- Adicione 5 μM de cada acompanhante (GST-SGTA e GST-Calmodulin) ao material reconstituído. Esses acompanhantes se ligarão ao TMD de quaisquer proteínas que não se reconstituíssem nos lipossomos. A purificação dos acompanhantes foi descrita anteriormente6,57.
NOTA: Esses acompanhantes estão disponíveis comercialmente, mas preferimos purificar em casa para controlar o custo e a qualidade. Ambas as proteínas estão em 20 mM Tris pH 7.5, 100 mM cloreto de sódio, e 0,1 mM TCEP, e têm uma concentração de estoque em torno de 160 μM. O SGTA reconhece substratos com TMD altamente hidrofóbico, enquanto calmodulin une TMDs com hidrofóbica moderada6,29. Juntos, este coquetel de acompanhantes pode reconhecer uma ampla gama de substratos. - Adicione 100 μL de tampão de extração ao material reconstituído, elevando o volume para 200 μL. Adicione isso às colunas de rotação de glutationa equilibradas. Observe que as colunas de spin de glutationa fornecem a maior recuperação amostral quando o volume de pré-compensação é de 200 – 400 μL.
- Conecte as colunas de giro e gire a ~80 rpm a 4 °C por 30 minutos para permitir que acompanhantes se liguem à resina.
- Gire as colunas a 700 x g por 2 minutos em temperatura ambiente. O fluxo através é material pré-desmatado que é esgotado de proteínas agregadas. Mantenha o material no gelo e prossiga diretamente com o ensaio de extração.
3. Ensaio de extração
- Prepare tubos para análise de PAGE SDS. Cada reação terá 4 tubos: INPUT (I), FLOW THROUGH (FT), WASH (W) e ELUTE (E).
- Adicione 45 μL de amostra ddH2O ao tubo de entrada, 40 μL de ddH2O ao tubo FLOW THROUGH e 0 μL aos tubos WASH e ELUTE.
NOTA: A melhor relação sinal/ruído neste ensaio é obtida quando as amostras de WASH e ELUTE estão 5x concentradas em relação ao INPUT e FLOW ATRAVÉS DE AMOSTRAS. Devido às diluições durante o ensaio, isso requer a coleta de 5 μL de amostra para a amostra de INSUMO, 10 μL de amostra para a amostra FLOW THROUGH e 50 μL de amostra para as amostras de WASH e ELUTE. - Adicione 16,6 μL de 4x SDS PAGE Loading Buffer em cada tubo. O volume total de cada amostra é de 50 μL antes do buffer de carregamento de PÁGINA do SDS. O volume final é de 66,6 μL (amostra de 50 μL + 16,6 μL de 4x SDS PAGE Loading Buffer).
- Adicione 45 μL de amostra ddH2O ao tubo de entrada, 40 μL de ddH2O ao tubo FLOW THROUGH e 0 μL aos tubos WASH e ELUTE.
- Monte o ensaio de extração.
- Prepare a reação de extração contendo 60 μL de proteoliposomes pré-limpos, 5 μM de GST-SGTA, 5 μM de GST-Calmodulin e ATP de 2 mM. Combine todos os reagentes, exceto ATP, que é usado para iniciar a reação. Leve para um volume final de 200 μL com buffer de extração.
NOTA: Como 60 μL de amostra são usados para cada ensaio de extração, uma reconstituição pode ser usada para três ensaios de extração diferentes. Realizar controles positivos e negativos (+ATP e -ATP) em material da mesma reconstituição. - Ensaio de extração pré-quente em bloco de calor de 30 °C por 2 minutos.
- Prepare a reação de extração contendo 60 μL de proteoliposomes pré-limpos, 5 μM de GST-SGTA, 5 μM de GST-Calmodulin e ATP de 2 mM. Combine todos os reagentes, exceto ATP, que é usado para iniciar a reação. Leve para um volume final de 200 μL com buffer de extração.
- Inicie o ensaio de extração adicionando ATP à concentração final de 2 mM e temporizador inicial.
- Dê uma volta de 5 segundos em um picofuge para misturar ATP na reação. Incubar a reação a 30 °C por 30 minutos.
- Durante a incubação, tome 5 μL da reação e adicione ao tubo de entrada. O momento é flexível.
- Durante este período de incubação, equilibre uma coluna de spin de glutationa para cada amostra no ensaio de extração.
- Faça puxar para baixo em acompanhantes para isolar o material extraído.
- Uma vez concluída a incubação de 30 minutos, adicione 200 μl de tampão de extração ao tubo para aumentar o volume total para 400 μL. Adicione à resina de glutationa equilibrada e deixe ligar a roda a 4° C por 30 minutos.
- Gire as colunas a 700 x g por 2 minutos em temperatura ambiente para coletar o fluxo. Tome 10 μL para o tubo FLOW THROUGH. Esta amostra contém substratos que ainda estão integrados na bicamadas lipídicas.
- Lave a resina duas vezes com 400 μL de tampão de extração, descartando o fluxo através. Na terceira lavagem, mantenha o fluxo e leve 50 μL para o tubo WASH.
- Prepare 5 mL de Tampão de Elução adicionando glutationa reduzida a uma concentração final de 5 mM no Buffer de Extração. Prepare este buffer fresco cada vez.
- Adicione 200 μL de Tampão de Elução à coluna de giro. Incubar em temperatura ambiente por 5 minutos. Gire a 700 x g por 2 minutos em temperatura ambiente até eluto. Mantenha o fluxo. Repita o processo uma segunda vez para que o volume total de eluição seja de 400 μL.
- Pegue 50 μL de amostra da amostra de eluição e adicione-a ao tubo ELUTE.
- Analise a atividade de extração usando SDS-PAGE e mancha ocidental.
NOTA: Como uma mancha ocidental é um procedimento bastante padrão, é fornecido um protocolo básico que destaca alguns dos detalhes exclusivos deste ensaio.- Carregue amostras em um gel de poliacrilamida livre de manchas (4% empilhamento, 15% separando) e execute a 200 V por 50 minutos no buffer tris-glicina. Se o espaço permitir, use uma escada manchada e manchada para permitir a visualização do imager de gel livre de manchas e transferir para a membrana PVDF, respectivamente. O gel livre de manchas permite visualização quantitativa de triptofano contendo proteínas após ativação com luz ultravioleta, ao mesmo tempo em que permite que o gel seja usado para uma mancha ocidental.
- Imagem o gel livre de manchas para confirmar que há carga igual em todas as amostras. Este é um controle essencial que garante que quaisquer alterações no sinal detectadas pela mancha ocidental não sejam resultado do carregamento variável de proteínas. Só devem haver faixas visíveis para os acompanhantes (GST-calmodulin e GST-SGTA) nas amostras de INSUMO e ELUTION. Lembre-se que a amostra ELUTE será mais concentrada do que a amostra INPUT.
- Monte um de mancha ocidental usando uma membrana PVDF de 45 μm. Transfira a uma corrente constante de 300 mA por 60 minutos.
- Depois de bloquear a membrana, ligue ao anticorpo primário por 16 h a 4 °C com agitação suave ~15 rpm. Blot para substrato com coelho Anti-BANDEIRA em uma diluição de 1:1.000.
NOTA: Os anticorpos primários e secundários utilizados serão específicos do substrato e a concentração para uso pode precisar ser otimizada. - Lave a membrana e incubar com anticorpos secundários, anti-coelho de cabra a uma diluição de 1:10.000, com agitação suave por 1 hora à temperatura ambiente.
- Lave a membrana e a imagem para análise usando o agente de detecção de manchas ocidentais.
Resultados
Para interpretar corretamente os resultados, o gel livre de manchas e a mancha ocidental devem ser vistos juntos. O gel livre de manchas garante o carregamento igual em todas as amostras. Ao visualizar o gel livre de manchas, os acompanhantes (GST-calmodulin e GST-SGTA) serão visíveis nas pistas INPUT (I) e ELUTE (E). Verifique duas vezes se a intensidade dessas bandas é uniforme em todas as amostras de ENTRADA. Da mesma forma, certifique-se de que a intensidade é uniforme em todas as amostras de ELUTE. O ELUTE é 5x...
Discussão
A função mitocondrial adequada depende de um sistema robusto de controle de qualidade de proteína. Devido aos limites inerentes à fidelidade das vias de direcionamento da proteína TA, as proteínas T deslocalizadas são uma fonte constante de estresse para mitocôndrias. Um componente-chave da rede de proteostase mitocondrial é o Msp1, que é um ATPase AAA+ ancorado em membrana que remove proteínas TA mal localizadas do OMM. Aqui, descrevemos como preparar proteoliposomes, co-reconstituir Msp1 e uma proteína TA m...
Divulgações
Nenhum
Agradecimentos
MLW desenvolveu parte deste protocolo durante seus estudos de pós-doutorado com o Dr. Robert Keenan na Universidade de Chicago.
Este trabalho é financiado pela concessão do NIH 1R35GM137904-01 à MLW.
Materiais
| Name | Company | Catalog Number | Comments |
| Biobeads | Bio-Rad | 1523920 | |
| Bovine liver phosphatidyl inositol | Avanti | 840042C | PI |
| Chicken egg phosphatidyl choline | Avanti | 840051C | PC |
| Chicken egg phosphatidyl ethanolamine | Avanti | 840021C | PE |
| ECL Select western blotting detection reagent | GE | RPN2235 | |
| Filter supports | Avanti | 610014 | |
| Glass vial | VWR | 60910L-1 | |
| Glutathione spin column | Thermo Fisher | PI16103 | |
| Goat anti-rabbit | Thermo Fisher | NC1050917 | |
| Mini-Extruder | Avanti | 610020 | |
| Polycarbonate membrane | Avanti | 610006 | 200 nM |
| PVDF membrane | Thermo Fisher | 88518 | 45 µM |
| Rabbit anti-FLAG | Sigma-Aldrich | F7245 | |
| Synthetic 1,2-dioleoyl-sn-glycero-3-phospho-L-serine | Avanti | 840035C | DOPS |
| Synthetic 1',3'-bis[1,2-dioleoyl-sn-glycero-3-phospho]-glycerol | Avanti | 710335C | TOCL |
| Syringe, 1 mL | Norm-Ject | 53548-001 | |
| Syringe, 1 mL, gas-tight | Avanti | 610017 |
Referências
- Song, J., Herrmann, J. M., Becker, T. Quality control of the mitochondrial proteome. Nature Reviews Molecular Cell Biology. 22, 54-70 (2021).
- Phillips, B. P., Miller, E. A. Membrane protein folding and quality control. Current Opinion in Structural Biology. 69, 50-54 (2021).
- Jiang, H. Quality control pathways of tail-anchored proteins. Biochimica et Biophysica Acta - Molecular Cell Research. 1868, 118922 (2020).
- McKenna, M. J., et al. The endoplasmic reticulum P5A-ATPase is a transmembrane helix dislocase. Science. 369, (2020).
- Hegde, R. S., Zavodszky, E. Recognition and Degradation of Mislocalized Proteins in Health and Disease. Cold Spring Harbor Perspectives in Biology. 11, 033902 (2019).
- Shao, S., Hegde, R. S. A calmodulin-dependent translocation pathway for small secretory proteins. Cell. 147, 1576-1588 (2011).
- Samuelson, J. C., et al. YidC mediates membrane protein insertion in bacteria. Nature. 406, 637-641 (2000).
- Anghel, S. A., McGilvray, P. T., Hegde, R. S., Keenan, R. J. Identification of Oxa1 Homologs Operating in the Eukaryotic Endoplasmic Reticulum. Cell Reports. 21, 3708-3716 (2017).
- Aviram, N., et al. The SND proteins constitute an alternative targeting route to the endoplasmic reticulum. Nature. 540, 134-138 (2016).
- Voorhees, R. M., Hegde, R. S. Structure of the Sec61 channel opened by a signal sequence. Science. 351, 88-91 (2016).
- Cichocki, B. A., Krumpe, K., Vitali, D. G., Rapaport, D. Pex19 is involved in importing dually targeted tail-anchored proteins to both mitochondria and peroxisomes. Traffic. 19, 770-785 (2018).
- Mateja, A., et al. Protein targeting. Structure of the Get3 targeting factor in complex with its membrane protein cargo. Science. 347, 1152-1155 (2015).
- Chacinska, A., Koehler, C. M., Milenkovic, D., Lithgow, T., Pfanner, N. Importing mitochondrial proteins: machineries and mechanisms. Cell. 138, 628-644 (2009).
- Chitwood, P. J., Hegde, R. S. An intramembrane chaperone complex facilitates membrane protein biogenesis. Nature. , (2020).
- Chitwood, P. J., Juszkiewicz, S., Guna, A., Shao, S., Hegde, R. S. EMC Is Required to Initiate Accurate Membrane Protein Topogenesis. Cell. 175, 1-30 (2018).
- Bock, F. J., Tait, S. W. G. Mitochondria as multifaceted regulators of cell death. Nature Reviews Molecular Cell Biology. 21, 85-100 (2020).
- Pfanner, N., Warscheid, B., Wiedemann, N. Mitochondrial proteins: from biogenesis to functional networks. Nature Reviews Molecular Cell Biology. 20, (2019).
- Bykov, Y. S., Rapaport, D., Herrmann, J. M., Schuldiner, M. Cytosolic Events in the Biogenesis of Mitochondrial Proteins. Trends in Biochemical Sciences. 45, 650-667 (2020).
- Pfanner, N., Warscheid, B., Wiedemann, N. Mitochondrial proteins: from biogenesis to functional networks. Nature Reviews Molecular Cell Biology. 427, 1135 (2019).
- Borgese, N., Coy-Vergara, J., Colombo, S. F., Schwappach, B. The Ways of Tails: the GET Pathway and more. The Protein Journal. , 1-17 (2019).
- Mateja, A., Keenan, R. J. A structural perspective on tail-anchored protein biogenesis by the GET pathway. Current Opinion in Structural Biology. 51, 195-202 (2018).
- Chio, U. S., Cho, H., Shan, S. Mechanisms of Tail-Anchored Membrane Protein Targeting and Insertion. Annual review of cell and developmental biology. 33, 417-438 (2017).
- Denic, V. A portrait of the GET pathway as a surprisingly complicated young man. Trends in biochemical sciences. , (2012).
- Hegde, R. S., Keenan, R. J. Tail-anchored membrane protein insertion into the endoplasmic reticulum. Nature Reviews Molecular Cell Biology. 12, 787-798 (2011).
- Kalbfleisch, T., Cambon, A., Wattenberg, B. W. A bioinformatics approach to identifying tail-anchored proteins in the human genome. Traffic. 8, 1687-1694 (2007).
- Doan, K. N., et al. The Mitochondrial Import Complex MIM Functions as Main Translocase for α-Helical Outer Membrane Proteins. Cell Reports. 31, (2020).
- McDowell, M. A., et al. Structural Basis of Tail-Anchored Membrane Protein Biogenesis by the GET Insertase Complex. Molecular Cell. 80, (2020).
- Guna, A., Volkmar, N., Christianson, J. C., Hegde, R. S. The ER membrane protein complex is a transmembrane domain insertase. Science. 591, 3099 (2017).
- Rao, M., et al. Multiple selection filters ensure accurate tail-anchored membrane protein targeting. eLife. 5, 21301 (2016).
- Schuldiner, M., et al. The GET complex mediates insertion of tail-anchored proteins into the ER membrane. Cell. 134, 634-645 (2008).
- Chen, Y. -. C., et al. Msp1/ATAD1 maintains mitochondrial function by facilitating the degradation of mislocalized tail-anchored proteins. The EMBO journal. 33, 1548-1564 (2014).
- Wu, X., Rapoport, T. A. Translocation of Proteins through a Distorted Lipid Bilayer. Trends in Cell Biology. , (2021).
- Phillips, B. P., Gomez-Navarro, N., Miller, E. A. Protein quality control in the endoplasmic reticulum. Current Opinion in Cell Biology. 65, 96-102 (2020).
- van de Weijer, M. L., et al. Quality Control of ER Membrane Proteins by the RNF185/Membralin Ubiquitin Ligase Complex. Molecular Cell. 79, (2020).
- Weir, N. R., Kamber, R. A., Martenson, J. S., Denic, V. The AAA protein Msp1 mediates clearance of excess tail-anchored proteins from the peroxisomal membrane. eLife. 6, 28507 (2017).
- Gardner, B. M., et al. The peroxisomal AAA-ATPase Pex1/Pex6 unfolds substrates by processive threading. Nature communications. 9, 135 (2018).
- Puchades, C., et al. Unique Structural Features of the Mitochondrial AAA+ Protease AFG3L2 Reveal the Molecular Basis for Activity in Health and Disease. Molecular Cell. , (2019).
- Castanzo, D. T., LaFrance, B., Martin, A. The AAA+ ATPase Msp1 is a processive protein translocase with robust unfoldase activity. Proceedings of the National Academy of Sciences of the United States of America. 117, 14970-14977 (2020).
- Wang, L., Myasnikov, A., Pan, X., Walter, P. Structure of the AAA protein Msp1 reveals mechanism of mislocalized membrane protein extraction. eLife. 9, (2020).
- Puchades, C., Sandate, C. R., Lander, G. C. The molecular principles governing the activity and functional diversity of AAA+ proteins. Nature Reviews Molecular Cell Biology. , 1-16 (2019).
- Yang, Y., et al. Folding-Degradation Relationship of a Membrane Protein Mediated by the Universally Conserved ATP-Dependent Protease FtsH. Journal of the American Chemical Society. , 10 (2018).
- Baldridge, R. D., Rapoport, T. A. Autoubiquitination of the Hrd1 Ligase Triggers Protein Retrotranslocation in ERAD. Cell. 166, 394-407 (2016).
- Fresenius, H. L., Wohlever, M. L. Sorting out how Msp1 maintains mitochondrial membrane proteostasis. Mitochondrion. 49, 128-134 (2019).
- Wang, L., Walter, P. Msp1/ATAD1 in Protein Quality Control and Regulation of Synaptic Activities. Annual Review of Cell and Developmental Biology. 36, 1-24 (2020).
- Dederer, V., et al. Cooperation of mitochondrial and ER factors in quality control of tail-anchored proteins. eLife. 8, 1126 (2019).
- Matsumoto, S., et al. Msp1 Clears Mistargeted Proteins by Facilitating Their Transfer from Mitochondria to the ER. Molecular Cell. , (2019).
- Li, L., Zheng, J., Wu, X., Jiang, H. Mitochondrial AAA-ATPase Msp1 detects mislocalized tail-anchored proteins through a dual-recognition mechanism. EMBO Reports. 20, (2019).
- Weidberg, H., Amon, A. MitoCPR - a surveillance pathway that protects mitochondria in response to protein import stress. Science. 360, (2018).
- Okreglak, V., Walter, P. The conserved AAA-ATPase Msp1 confers organelle specificity to tail-anchored proteins. Proceedings of the National Academy of Sciences of the United States of America. 111, (2014).
- Piard, J., et al. A homozygous ATAD1 mutation impairs postsynaptic AMPA receptor trafficking and causes a lethal encephalopathy. Brain. , (2018).
- Zhang, J., et al. The AAA+ ATPase Thorase regulates AMPA receptor-dependent synaptic plasticity and behavior. Cell. 145, 284-299 (2011).
- Prendergast, J., et al. Ganglioside regulation of AMPA receptor trafficking. The Journal of Neuroscience. 34, 13246-13258 (2014).
- Umanah, G. K. E., et al. Thorase variants are associated with defects in glutamatergic neurotransmission that can be rescued by Perampanel. Science Translational Medicine. 9, 4985 (2017).
- Pignatelli, M., et al. Synaptic Plasticity onto Dopamine Neurons Shapes Fear Learning. Neuron. 93, 425-440 (2017).
- Zhang, J., et al. The AAA Thorase is neuroprotective against ischemic injury. Journal of Cerebral Blood Flow and Metabolism. , 271678 (2018).
- Umanah, G. K. E., et al. AMPA Receptor Surface Expression Is Regulated by S-Nitrosylation of Thorase and Transnitrosylation of NSF. Cell Reports. 33, 108329 (2020).
- Wohlever, M. L., Mateja, A., McGilvray, P. T., Day, K. J., Keenan, R. J. Msp1 Is a Membrane Protein Dislocase for Tail-Anchored Proteins. Molecular Cell. 67, 194-202 (2017).
- Lovell, J. F., et al. Membrane binding by tBid initiates an ordered series of events culminating in membrane permeabilization by Bax. Cell. 135, 1074-1084 (2008).
- Leshchiner, E. S., Braun, C. R., Bird, G. H., Walensky, L. D. Direct activation of full-length proapoptotic BAK. Proceedings of the National Academy of Sciences of the United States of America. 110, 986-995 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados