Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Rekonstitution der Msp1-Extraktionsaktivität mit vollständig gereinigten Komponenten
In diesem Artikel
Zusammenfassung
Hier stellen wir ein detailliertes Protokoll zur Rekonstitution der Msp1-Extraktionsaktivität mit vollständig gereinigten Komponenten in definierten Proteoliposomen vor.
Zusammenfassung
Als Zentrum für oxidative Phosphorylierung und apoptotische Regulation spielen Mitochondrien eine wichtige Rolle für die menschliche Gesundheit. Die richtige mitochondriale Funktion hängt von einem robusten Qualitätskontrollsystem ab, um die Proteinhomöostase (Proteostase) aufrechtzuerhalten. Der Rückgang der mitochondrialen Proteostase wurde mit Krebs, Alterung, Neurodegeneration und vielen anderen Krankheiten in Verbindung gebracht. Msp1 ist eine AAA+ ATPase, die in der äußeren mitochondrialen Membran verankert ist und die Proteostase aufrechterhält, indem sie falsch lokalisierte schwanzverankerte Proteine entfernt. Unter Verwendung von gereinigten Komponenten, die zu Proteoliposomen rekonstituiert wurden, haben wir gezeigt, dass Msp1 notwendig und ausreichend ist, um ein modellhaftes schwanzverankertes Protein aus einer Lipiddoppelschicht zu extrahieren. Unser vereinfachtes rekonstituiertes System überwindet mehrere der technischen Barrieren, die eine detaillierte Untersuchung der Membranproteinextraktion behindert haben. Hier stellen wir detaillierte Methoden zur Generierung von Liposomen, zur Membranproteinrekonstitution und zum Msp1-Extraktionsassay zur Verfügung.
Einleitung
Die richtige zelluläre Funktion hängt von einem Prozess namens Proteostase ab, der sicherstellt, dass funktionelle Proteine in der richtigen Konzentration und zellulären Position sind1. Versagen in der Proteostase führen zu einer beeinträchtigten Organellenfunktion und sind mit vielen neurodegenerativen Erkrankungen verbunden2,3,4. Membranproteine stellen einzigartige Herausforderungen an das Proteostasenetzwerk dar, da sie auf die richtige Membran ausgerichtet werden müssen, während die Aggregation aus den hydrophoben Transmembrandomänen (TMDs) vermieden wird5. Folglich hat sich eine spezielle Maschinerie entwickelt, um die hydrophobe TMD vom Zytosol abzuschirmen und das Zielen und Einsetzen in die richtige Zellmembran zu erleichtern6,7,8,9,10,11,12,13,14,15.
Mitochondrien sind das metabolische Zentrum der Zelle und an zahlreichen essentiellen zellulären Prozessen beteiligt, wie zum Beispiel: oxidative Phosphorylierung, Eisen-Schwefel-Cluster-Erzeugung und apoptotische Regulation16,17. Diese endosymbiotischen Organellen enthalten zwei Membranen, die als innere mitochondriale Membran (IMM) und äußere mitochondriale Membran (OMM) bezeichnet werden. Über 99% der 1.500 menschlichen mitochondrialen Proteine sind im Kerngenom kodiert und müssen über eine oder zwei verschiedene Membranen transloziert werden18,19. Die richtige mitochondriale Funktion hängt daher von einem robusten Proteostasenetzwerk ab, um Fehler beim Protein-Targeting oder bei der Translokation zu korrigieren.
Unser Labor konzentriert sich auf eine Untergruppe von mitochondrialen Membranproteinen, die als schwanzverankerte (TA) Proteine bezeichnet werden und eine einzelne Transmembrandomäne am C-Terminus20,21,22,23,24haben. TA-Proteine sind an einer Reihe von essentiellen Prozessen beteiligt, wie Apoptose, Vesikeltransport und Proteintranslokation25. Die einzigartige Topologie von TA-Proteinen erfordert eine posttranslationale Insertion, die im endoplasmatischen Retikulum (ER) durch den geführten Eintritt von Tail-verankerten (GET) oder endoplasmatischen Retikulummembranproteinkomplex (EMC) -Signalwegen oder in das OMM durch einen schlecht charakterisierten Wegauftritt 20,26,27,28. Die biophysikalischen Eigenschaften des TMD sind notwendig und ausreichend, um TA-Proteine zur richtigen Membran zu führen29. Die Erkennung biophysikalischer Merkmale anstelle eines definierten Sequenzmotivs schränkt die Genauigkeit der Targeting-Pfade ein5. Daher ist die Fehllokalisierung von TA-Proteinen ein häufiger Stress für die Proteostase-Netzwerke. Zellulärer Stress, wie die Hemmung des GET-Signalwegs, verursacht eine Zunahme der Proteinfehllokalisierung zum OMM und mitochondriale Dysfunktion, es sei denn, diese Proteine werden sofort entfernt30,31.
Ein häufiges Thema in der Membranproteostase ist die Verwendung von AAA+(ATPase Associated with cellular Activities) Proteinen, um alte, beschädigte oder fehllokalisierte Proteine aus der Lipiddoppelschicht1,32,33,34,35,36,37,38 zu entfernen . AAA+-Proteine sind molekulare Motoren, die hexamere Ringe bilden und ATP-abhängigen Bewegungen unterzogen werden, um ein Substrat umzugestalten, oft durch Translokation durch eine schmale axiale Pore39,40. Obwohl große Anstrengungen unternommen wurden, um die Extraktion von Membranproteinen durch AAA + ATPasen zu untersuchen, sind die Rekonstitutionen komplex oder beinhalten eine Mischung aus Lipiden und Waschmittel41,42, was die experimentelle Kraft begrenzt, den Mechanismus der Substratextraktion aus der Lipiddoppelschicht zu untersuchen.
Msp1 ist eine hochkonservierte AAA+ ATPase, die im OMM und in den Peroxisomen verankert ist und eine entscheidende Rolle bei der Membranproteostase spielt, indem sie fehllokalisierte TA-Proteine43,44,45,46,47entfernt. Es wurde kürzlich auch gezeigt, dass Msp1 den mitochondrialen Proteinimportstress lindert, indem es Membranproteine entfernt, die während der Translokation über das OMM48zum Stillstand kommen. Der Verlust von Msp1 oder des menschlichen Homologs ATAD1 führt zu mitochondrialer Fragmentierung, Versagen bei der oxidativen Phosphorylierung, Krampfanfällen, erhöhten Verletzungen nach Schlaganfall und frühem Tod31,49,50,51,52,53,54,55,56.
Wir haben gezeigt, dass es möglich ist, TA-Proteine mit Msp1 ko-rekonstituieren und die Extraktion aus der Lipiddoppelschicht57nachzuweisen. Dieses vereinfachte System verwendet vollständig gereinigte Proteine, die zu definierten Liposomen rekonstituiert sind, die das OMM nachahmen (Abbildung 1)58,59. Diese Ebene der experimentellen Kontrolle kann detaillierte mechanistische Fragen der Substratextraktion angehen, die experimentell unlösbar sind, mit komplexeren Rekonstitutionen, an denen andere AAA+ Proteine beteiligt sind. Hier stellen wir experimentelle Protokolle zur Verfügung, die unsere Methoden zur Liposomenpräparation, zur Membranproteinrekonstitution und zum Extraktionsassay beschreiben. Wir hoffen, dass diese experimentellen Details die weitere Untersuchung des wesentlichen, aber wenig verstandenen Prozesses der Membranproteostase erleichtern werden.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Liposomen-Vorbereitung
- Kombinieren Sie Chloroform-Bestände von Lipiden in geeigneten Verhältnissen, um die äußere mitochondriale Membran nachzuahmen.
- Bereiten Sie 25 mg Lipidmischung vor. Wir verwenden eine zuvor etablierte Mischung von Lipiden, die mitochondriale Membranen nachahmen, bestehend aus einem molaren Verhältnis von 48:28:10:10:4 aus Hühnerei-Phosphatidylcholin (PC), Hühnerei-Phosphatidylethanolamin (PE), Rinderleberphosphatidyl-Inositol (PI), synthetischem 1,2-Dioleoyl-sn-glycero-3-phospho-L-Serin (DOPS) und synthetischem 1',3'-Bis[1,2-Dioleoyl-sn-glycero-3-phospho]-glycerin (TOCL)58,59. Beispielrechnungen sind in Tabelle 1 dargestellt.
- Bringen Sie alle Lipidvorräte vor dem Öffnen auf Raumtemperatur, da dies die Kondensation begrenzt. Da die meisten Labore keine genaue Möglichkeit haben, die Konzentration der Lipide zu messen, ändert jedes Wasser, das vom Chloroform-Stamm absorbiert wird, die Konzentration des Lipidstocks und damit das Verhältnis der im Assay verwendeten Lipide.
- Da Lipidvorräte in Glasampullen vorliegen, übertragen Sie die erforderliche Menge an Lipid mit einer 1 ml Spritze in eine Glasfläschchen. Geben Sie 2 mg Dithiothreitol (DTT) in die Durchstechflasche, um eine Lipidoxidation zu verhindern. Arbeiten Sie schnell, da die Verdampfung von Chloroform die Konzentration des Lipids verändert.
- Übertragen Sie das verbleibende Lipid auf eine separate Glasfläschchen und passen Sie sie mit einem PFTE-Septum an. 2 mg DTT in die Durchstechflasche geben, mit Parafilm umwickeln und bei -20 °C lagern, um eine Lipidoxidation zu verhindern. Versuchen Sie, die Lipide innerhalb von 3 Monaten nach dem Transfer zu den Fläschchen zu verwenden. Um eine mögliche Kontamination der Chloroformvorräte durch Markerabfluss zu verhindern, übertragen Sie die Aufkleber von den ursprünglichen Ampullen auf die Glasfläschchen, anstatt sie mit Marker zu etikettieren.
- Verdampfen Sie Chloroform unter einem sehr sanften Stickstoffstrom, während Sie die Glasfläschchen kontinuierlich von Hand in einem Abzug drehen, der im Wesentlichen als manueller Rotovap wirkt. Drehen Sie die Durchstechflasche mit einer konstanten Geschwindigkeit (20-40 U / min) von Hand, um die Lipide in Bewegung zu halten. Ziel ist es, das gesamte Chloroform zu verdampfen und eine gleichmäßige Lipidschicht über das gesamte Glasfläschchen zu erhalten.
VORSICHT: Chloroform ist neurotoxisch, und dieser Schritt sollte im Abzug durchgeführt werden.- Befestigen Sie eine frische Pasteurpipette am Stickstoffröhrchen. Lassen Sie keines der Lipide aus der Durchstechflasche oder auf die Pasteur-Pipette spritzen. Richten Sie die Spitze auf den Boden der Durchstechflasche, so dass die Luft vom Boden abprallt und die Lipide in Richtung der Mitte der Durchstechflasche drückt. Erhalten Sie eine gleichmäßige Beschichtung über die gesamte Durchstechflasche, während Sie eine Ansammlung in den Ecken oder an der Kappe vermeiden. Dieser gesamte Vorgang dauert ca. 5 Minuten.
- Wenn sich die Mischung zu einer "Perle" von Lipiden verdickt, führen Sie sie in die Mitte der Durchstechflasche, indem Sie den Winkel der Durchstechflasche ändern. Sobald die Perle kleiner wird, drehen Sie den Stickstoffstrom leicht nach oben, um die Perle zu verteilen, und stellen Sie sicher, dass keines der Lipide aus der Durchstechflasche ausbläst.
- Entfernen Sie alle verbleibenden Chloroformen unter Vakuum.
- Legen Sie die Glasfläschchen für 1 Stunde auf eine Hausvakuum- oder Membran-Vakuumpumpe, um den Großteil des Restchloroforms zu entfernen. Diese Staubsauger sind im Allgemeinen nicht stark genug, um das gesamte Chloroform zu entfernen, aber sie können kleine Mengen an Lösungsmittel besser vertragen als rotierende eitle Staubsauger.
- Legen Sie die Durchstechflasche für 12-16 h auf ein starkes Vakuum (<1 mTorr), um alle Restchloroform zu entfernen. Achten Sie darauf, das Stoßen der Durchstechflasche während dieses Vorgangs zu vermeiden.
- Resuspendieren Sie die Lipide in 1,25 ml Liposomenpuffer (50 mM HEPES KOH pH 7,5, 15% Glycerin, 1 mM DTT). Da wir mit 25 mg Lipiden begonnen haben, ergibt dies eine Konzentration von 20 mg / ml. Resuspendieren Sie die Lipide vollständig ohne sichtbare Brocken. Wenn sich die Lipide in der Ecke der Durchstechflasche sammeln, kann dies ein langwieriger Prozess sein.
- Die Durchstechflasche kräftig schwenken, bis die Probe milchig glatt ist. Wir stellen fest, dass, wenn die Chloroform-Verdampfung in der Nacht zuvor richtig durchgeführt wurde, dieser Prozess etwa 5-10 Minuten dauert.
- Um eine vollständige Rückgewinnung der Lipide zu gewährleisten, drehen Sie sich auf einem Rad bei Raumtemperatur für 3 Stunden bei ~ 80 U / min. Entfernen Sie die Durchstechflasche einmal pro Stunde für 1 Minute Vom Rad, um eine gleichmäßige Vermischung zu gewährleisten.
- Lipide vorsichtig in ein sauberes 1,5 ml Mikrozentrifugenröhrchen übertragen. Führen Sie 5 Gefrier-Tau-Zyklen mit flüssigem Stickstoff zum Einfrieren und einem 30 °C Hitzeblock zum Auftauen durch. Dieser Schritt hilft, mehrschalige Vesikel in einlamellare Vesikel umzuwandeln.
- Extrudieren Sie die Lipide.
- Bereiten Sie während der Gefrierauftauzyklen den Mini-Extruder vor. Montieren Sie den Mini-Extruder mit 10 mm Filterträgern und einer Polycarbonatmembran der gewünschten Porengröße (wir verwenden 200 nm). Die Größe des Filters beeinflusst die Größe des Liposoms, was sich auf die Konzentration der für die Rekonstitution erforderlichen Proteine auswirkt (Schritt 2.3).
- Stellen Sie den Mini-Extruder auf eine Heizplatte und bringen Sie die Extrudertemperatur auf bis zu 60 °C.
- Ziehen Sie die Lipide in eine 1 ml gasdichte Glasspritze und legen Sie sie vorsichtig in ein Ende des Miniextruders. Legen Sie die leere gasdichte Spritze in die andere Seite des Mini-Extruders. Lassen Sie die Lipide 5-10 Minuten lang auf die Extruderstandtemperatur ausgleichen.
- Übertragen Sie die Lipide auf die alternative Spritze, indem Sie vorsichtig den Kolben der gefüllten Spritze drücken. Drücken Sie die Lösung aus der Alternativspritze in die Originalspritze. Wiederholen Sie diesen Hin- und Her-Vorgang 15 Mal, so dass beim15. Durchgang die Lipide in der alternativen Spritze enden. Überwachen Sie die Lautstärke in jedem Durchgang, um sicherzustellen, dass keine Lecks vorhanden sind.
- Einweg-Aliquots der Lipide herstellen, in flüssigem Stickstoff schockgefrieren und bei -80 °C lagern. Die Liposomen sind bei -80 °C über mehrere Monate stabil. Die Rekonstitutionen erfordern jeweils 10 μL Liposomen (Schritt 2.3.4), so dass es zweckmäßig ist, 10 μL oder 20 μL Aliquots herzustellen.
2 Rekonstitution von Msp1 und Model TA Protein
- Bereiten Sie den Rekonstitutionspuffer vor: 50 mM HEPES pH 7,5, 200 mM Kaliumacetat, 7 mM Magnesiumacetat, 2 mM DTT, 10% Saccharose, 0,01% Natriumazid, 0,2-0,8% Deoxy Big Chaps (DBC).
- Optimieren Sie die Rekonstitutionsbedingungen für die neue Charge von Liposomen. Die Konzentration von DBC und Biokügelchen, die für eine optimale Rekonstitution erforderlich sind, variiert je nach charge der verwendeten Liposomen. Um die Prep-Variation auf die Prep-Variabilität zu beschränken, verwenden Sie für alle Experimente die gleiche Menge DBC. Wenn Sie viele DBC ändern, wiederholen Sie den Optimierungsprozess.
- Richten Sie eine Reihe von Rekonstitutionen mit unterschiedlichen Konzentrationen von DBC (0,2% - 0,8%) und Biokügelchen (25 mg - 100 mg) jedes Mal ein, wenn eine neue Charge von Liposomen hergestellt wird. Es ist wichtig, den DBC nicht unter die kritische Mizellbildungskonzentration (CMC) von ~0,12% fallen zu lassen. Sobald die Bedingungen optimiert sind, empfehlen wir, alle Daten unter Verwendung derselben Liposomenvorbereitungs- und Rekonstitutionsbedingungen zu sammeln.
- Die Wirksamkeit der verschiedenen Rekonstitutionsbedingungen unter Verwendung des in Schritt 3 beschriebenen Extraktionsassays zu untersuchen.
- Biokügelchen in einer Endkonzentration von 250 mg/ml zubereiten.
- 2,7 g getrocknete Biokügelchen abwiegen und in einem 50 ml Zentrifugenröhrchen aus 100% Methanol (ca. 45 ml) resuspendieren, um die Kügelchen zu benetzen. Befeuchten Sie die Biokügelchen zunächst mit Methanol, um zu verhindern, dass Luft in den Poren der Kügelchen eingeschlossen wird. Halten Sie die Biokügelchen in Methanol nass, da die von den Biokügelchen eingeschlossene Luft ihre Fähigkeit, Reinigungsmittel aufzunehmen, verändert.
- Entfernen Sie Methanol, indem Sie die Kügelchen 8x mit ca. 45 mL Reinstwasser (18,2 mΩ), im Folgenden ddH2O genannt, waschen. Pelletperlen durch Drehen bei 3.200 x g für 1 Minute. Die Flüssigkeit dekantieren und inddH2Oresuspendieren.
- Nach dem Waschen in 10 ml ddH2O mit0,02%Natriumazid resuspend und bei 4 °C lagern. Biokügelchen können mehrere Monate bei 4 °C gelagert werden. Dieser Bestand beträgt 250 mg / ml, da angenommen wird, dass während der Waschschritte ~ 0,2 g verloren gehen.
- Berechnen Sie die Größe des Liposoms und die gewünschte Anzahl von Molekülen des TA-Proteins und Msp1 pro Liposom. Dies bestimmt die Konzentration von Msp1- und TA-Protein, die für die Rekonstitution erforderlich ist.
- Berechnen Sie zunächst die Anzahl der Lipidmoleküle pro unilamellares Liposom (Ninsgesamt) unter Verwendung der Gleichung,
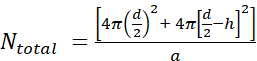 wobei d der Durchmesser des Liposoms, h die Dicke der Doppelschicht und a der Lipidkopfgruppenbereich ist.
wobei d der Durchmesser des Liposoms, h die Dicke der Doppelschicht und a der Lipidkopfgruppenbereich ist.- Messen Sie den Liposomendurchmesser mittels DLS. In unserem Beispiel wurde ein Wert von 70 nm für den Liposomendurchmesser (d) erhalten.
- Verwenden Sie einen Wert von 5 nm für h und 0,71 nm2 für a, was die Kopfgruppengröße für Phosphatidylcholin ist. In dieser besonderen Situation beträgt Ninsgesamt 37.610.
- Als nächstes berechnen Sie die molare Konzentration von Lipid MLipid unter Verwendung des durchschnittlichen Molekulargewichts der Lipide in der Mischung. In diesem Beispiel beträgt die Konzentration der Lipide 20 mg/ml (Schritt 1.4) und das mittlere Molekulargewicht der Lipide 810 g/mol (Tabelle 1). Daraus ergibt sich ein Wert von 0,0247 M für MLipid.
- Als nächstes berechnen Sie die molare Konzentration von Liposomen, M-Liposom,unter Verwendung der
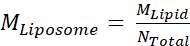 Gleichung, wobei MLipid die molare Konzentration von Lipid aus Schritt 2.3.2und Ninsgesamt die Gesamtzahl der Lipide pro Liposom ist, die in Schritt 2.3.1berechnet wird. In diesem Beispiel beträgt die 20 mg/ml Stammkonzentration von Liposomen etwa 660 nM.
Gleichung, wobei MLipid die molare Konzentration von Lipid aus Schritt 2.3.2und Ninsgesamt die Gesamtzahl der Lipide pro Liposom ist, die in Schritt 2.3.1berechnet wird. In diesem Beispiel beträgt die 20 mg/ml Stammkonzentration von Liposomen etwa 660 nM. - Berechnen Sie die Menge an Msp1- und TA-Protein, die für eine 100-μL-Rekonstitutionsreaktion erforderlich ist.
HINWEIS: Die Endkonzentration der Lipide in der Rekonstitution beträgt 2 mg/ml, was einer 10-fachen Verdünnung des Liposomenstocks entspricht. Dies ergibt eine endgültige Liposomenkonzentration von 66 nM. Die Endkonzentration von Msp1 beträgt 792 nM, was durchschnittlich 12 Gesamtkopien (2 funktionelle Hexamere) pro Liposom ergibt. Die Endkonzentration des TA-Proteins beträgt 660 nM, was durchschnittlich 10 Kopien pro Liposom ergibt.
- Berechnen Sie zunächst die Anzahl der Lipidmoleküle pro unilamellares Liposom (Ninsgesamt) unter Verwendung der Gleichung,
- Mischen Sie in einem PCR-Röhrchen gereinigtes Msp1, TA-Protein und Liposomen in Rekonstitutionspuffer. Die Reihenfolge der Addition ist Puffer, Proteine und Liposomen zuletzt. Das Gesamtvolumen beträgt 100 μL. Lassen Sie die Mischung 10 Minuten auf Eis sitzen. Reinigen Sie das Msp1- und TA-Protein wie zuvor beschrieben57.
- Verwenden Sie das gut charakterisierte Modell-TA-Protein His-Flag-Sumo-Sec22 als Positivkontrollsubstrat bei der ersten Etablierung des Assays. Dieses Konstrukt hat ein His-Tag für eine einfache Reinigung, ein 3x-Flag-Tag für den Nachweis durch Western Blot, eine Sumo-Domäne für erhöhte Löslichkeit und die TMD des ER-TA-Proteins Sec22 für die Rekonstitution und Erkennung durch Msp1.
- Stellen Sie sicher, dass die Stammlösungen sowohl von Msp1 als auch des TA-Proteins etwa 100 μM betragen, um die Wirkung von N-Dodecyl-β-D-Maltosid (DDM) aus der Proteinreinigung auf die Rekonstitution zu minimieren. Gereinigtes Msp1 ist in 20 mM HEPES pH 7,5, 100 mM Natriumchlorid, 0,1 mM Tris(2-carboxyethyl)phosphinhydrochlorid (TCEP), 0,05% DDM, während das gereinigte TA-Protein in 50 mM Tris pH 7,4, 150 mM Natriumchlorid, 10 mM Magnesiumchlorid, 5 mM β-Mercaptoethanol, 10% Glycerin, 0,1% DDM liegt. Stammlösungen von etwa 100 μM TA-Protein und Msp1-Monomer stellen sicher, dass diese Komponenten < 5% des endgültigen Rekonstitutionsvolumens ausmachen, was zu einer Verdünnung von DDM unterhalb des CMC führt.
- Fügen Sie der Probe die gewünschte Menge an Biokügelchen hinzu, um das Reinigungsmittel zu entfernen.
- Schneiden Sie die Spitze einer p200-Pipettenspitze auf einen Durchmesser von etwa1/8 Zoll, damit Perlen durch die Spitze passen. Wirbeln Sie das Bioperlenröhrchen gründlich vor, um eine gleichmäßige Mischung zu erhalten, entfernen Sie schnell den Deckel und pipettieren Sie das Volumen, bevor sich die Bioperlen absetzen. Übertragen Sie die Biokügelchen in ein leeres PCR-Röhrchen.
- Wenn die Rekonstitution ihre 10-minütige Inkubation auf Eis beendet hat, verwenden Sie eine ungeschnittene Pipettenspitze, um die gesamte Flüssigkeit aus den Biokügelchen zu entfernen. Anschließend wird die 100 μL Rekonstitution mit den Biokügelchen in das Röhrchen überführt. Dies muss schnell geschehen, damit die Biokügelchen keine Luft einfangen, wodurch die Kügelchen schwimmen.
- Lassen Sie die Rekonstitution 16 Stunden bei 4 °C auf einem Rad mit ~80 U/min drehen.
- Rekonstituiertes Material aus Biokügelchen entfernen. Machen Sie eine schnelle Drehung in einem Picofuge, um die Biokügelchen zu pelletieren, und verwenden Sie dann eine ungeschnittene Pipettspitze, um das rekonstituierte Material in ein sauberes PCR-Röhrchen zu übertragen. Wiederholen Sie diesen Vorgang 1-2 Mal, bis keine Biokügelchen mehr in der Probe vorhanden sind. Rekonstitution auf Eis aufbewahren.
- Das rekonstituierte Material vorab entfernen, um Proteine zu entfernen, die sich nicht in den Liposomen rekonstituieren konnten.
- Extraktionspuffer vorbereiten: 50 mM HEPES pH 7,5, 200 mM Kaliumacetat, 7 mM Magnesiumacetat, 2 mM DTT, 100 nM Calciumchlorid.
- Gleichsetzen Sie die Glutathion-Spinsäulen mit Extraction Buffer gemäß den Anweisungen des Herstellers. Dies beinhaltet typischerweise 3 Runden Waschen mit 400 μL Puffer und dann Zentrifugieren bei 700 x g für 2 Minuten bei Raumtemperatur, um den Puffer zu entfernen.
- Fügen Sie 5 μM jedes Chaperons (GST-SGTA und GST-Calmodulin) zu dem rekonstituierten Material hinzu. Diese Chaperone binden an die TMD aller Proteine, die sich nicht in den Liposomen rekonstituieren konnten. Die Reinigung von Chaperonen wurde zuvor beschrieben6,57.
HINWEIS: Diese Chaperone sind im Handel erhältlich, aber wir ziehen es vor, im Haus zu reinigen, um Kosten und Qualität zu kontrollieren. Beide Proteine sind in 20 mM Tris pH 7,5, 100 mM Natriumchlorid und 0,1 mM TCEP und haben eine Stammkonzentration um 160 μM. SGTA erkennt Substrate mit einer hochhydrophoben TMD, während Calmodulin TMDs mit moderater Hydrophobiebindet 6,29. Zusammen kann dieser Chaperon-Cocktail eine breite Palette von Substraten erkennen. - Fügen Sie dem rekonstituierten Material 100 μL Extraktionspuffer hinzu, wodurch das Volumen auf 200 μL steigt. Fügen Sie dies zu den gleichgewichteten Glutathion-Spin-Spalten hinzu. Beachten Sie, dass die Glutathion-Spinsäulen die höchste Probenausbeute bieten, wenn das Vorreinigungsvolumen 200 – 400 μL beträgt.
- Stecken Sie die Spinsäulen ein und drehen Sie sie bei ~ 80 U / min bei 4 ° C für 30 Minuten, damit chaperone sich an Harz binden können.
- Drehen Sie die Säulen bei 700 x g für 2 Minuten bei Raumtemperatur. Der Durchfluss ist vorgereinigtes Material, das an aggregierten Proteinen erschöpft ist. Bewahren Sie das Material auf Eis auf und fahren Sie direkt mit dem Extraktionsassay fort.
3. Extraktionsassay
- Bereiten Sie Röhrchen für die SDB-PAGE-Analyse vor. Jede Reaktion hat 4 Röhren: INPUT (I), FLOW THROUGH (FT), WASH (W) und ELUTE (E).
- Geben Sie 45 μL ddH2O Probe in das INPUT-Röhrchen, 40 μL ddH2O in das FLOW THROUGH-Rohr und 0 μL in das WASH- und ELUTE-Röhrchen.
HINWEIS: Das beste Signal-Rausch-Verhältnis in diesem Assay wird erzielt, wenn die WASH- und ELUTE-Proben im Verhältnis zu den INPUT- und FLOW THROUGH-Proben 5x konzentriert sind. Aufgrund der Verdünnungen während des Assays erfordert dies die Entnahme von 5 μL Probe für die INPUT-Probe, 10 μL Probe für die FLOW THROUGH-Probe und 50 μL Probe für die WASH- und ELUTE-Proben. - Fügen Sie 16,6 μL 4x SDS PAGE Loading Buffer zu jedem Rohr hinzu. Das Gesamtvolumen jeder Probe beträgt 50 μL vor dem SDS PAGE Loading Buffer. Das Endvolumen beträgt 66,6 μL (50 μL Probe + 16,6 μL 4x SDS PAGE Loading Buffer).
- Geben Sie 45 μL ddH2O Probe in das INPUT-Röhrchen, 40 μL ddH2O in das FLOW THROUGH-Rohr und 0 μL in das WASH- und ELUTE-Röhrchen.
- Stellen Sie den Extraktionsassay zusammen.
- Bereiten Sie die Extraktionsreaktion mit 60 μL vorgereichten Proteoliposomen, 5 μM GST-SGTA, 5 μM GST-Calmodulin und 2 mM ATP vor. Kombinieren Sie alle Reagenzien mit Ausnahme von ATP, das zur Einleitung der Reaktion verwendet wird. Mit Extraction Buffer auf ein Endvolumen von 200 μL bringen.
HINWEIS: Da für jeden Extraktionsassay 60 μL Probe verwendet werden, kann eine Rekonstitution für drei verschiedene Extraktionsassays verwendet werden. Führen Sie Positiv- und Negativkontrollen (+ATP und -ATP) an Material aus derselben Rekonstitution durch. - Vorwarmextraktionsassay in 30 °C Hitzeblock für 2 Minuten.
- Bereiten Sie die Extraktionsreaktion mit 60 μL vorgereichten Proteoliposomen, 5 μM GST-SGTA, 5 μM GST-Calmodulin und 2 mM ATP vor. Kombinieren Sie alle Reagenzien mit Ausnahme von ATP, das zur Einleitung der Reaktion verwendet wird. Mit Extraction Buffer auf ein Endvolumen von 200 μL bringen.
- Beginnen Sie mit dem Extraktionsassay, indem Sie ATP in die Endkonzentration von 2 mM geben und den Timer starten.
- Geben Sie einen 5-Sekunden-Spin in einem Picofuge, um ATP in die Reaktion zu mischen. Inkubation der Reaktion bei 30 °C für 30 Minuten.
- Nehmen Sie während der Inkubation 5 μL der Reaktion und geben Sie sie in das INPUT-Röhrchen. Der Zeitpunkt dafür ist flexibel.
- Während dieser Inkubationszeit ist eine Glutathion-Spin-Säule für jede Probe im Extraktionsassay auszugleichen.
- Führen Sie einen Pulldown auf Chaperone durch, um extrahiertes Material zu isolieren.
- Sobald die 30-minütige Inkubation abgeschlossen ist, fügen Sie 200 μl Extraktionspuffer in das Röhrchen hinzu, um das Gesamtvolumen auf 400 μL zu bringen. Zu äquilibriertem Glutathionharz geben und 30 Minuten auf dem Rad bei 4° C binden lassen.
- Drehen Sie die Säulen bei 700 x g für 2 Minuten bei Raumtemperatur, um den Durchfluss zu sammeln. Nehmen Sie 10 μL für das FLOW THROUGH-Rohr. Diese Probe enthält Substrate, die noch in der Lipiddoppelschicht integriert sind.
- Harz zweimal mit 400 μL Extraktionspuffer waschen und den Durchfluss verwerfen. Bei der dritten Wäsche den Durchfluss beibehalten und 50 μL für das WASH-Röhrchen nehmen.
- Bereiten Sie 5 ml Elutionspuffer vor, indem Sie reduziertes Glutathion zu einer Endkonzentration von 5 mM im Extraktionspuffer hinzufügen. Bereiten Sie diesen Puffer jedes Mal frisch vor.
- Fügen Sie 200 μL Elution Buffer zur Spin-Spalte hinzu. Bei Raumtemperatur 5 Minuten inkubieren. Bei 700 x g für 2 Minuten bei Raumtemperatur drehen, um zu eluieren. Halten Sie den Fluss durch. Wiederholen Sie den Vorgang ein zweites Mal, so dass das gesamte Elutionsvolumen 400 μL beträgt.
- Nehmen Sie 50 μL Probe aus der Elutionsprobe und geben Sie sie in das ELUTE-Röhrchen.
- Analysieren Sie die Extraktionsaktivität mit SDS-PAGE und Western Blot.
HINWEIS: Da ein Western Blot ein ziemlich standardmäßiges Verfahren ist, wird ein grundlegendes Protokoll bereitgestellt, das einige der für diesen Assay einzigartigen Details hervorhebt.- Laden Sie die Proben in ein fleckenfreies Polyacrylamid-Gel (4% Stapelung, 15% Trennung) und laufen Sie bei 200 V für 50 Minuten in Tris-Glycin-Puffer. Wenn es der Platz erlaubt, verwenden Sie sowohl eine ungefärbte als auch eine gebeizte Leiter, um die Visualisierung auf einem fleckenfreien Gel-Imager zu ermöglichen und auf die PVDF-Membran zu übertragen. Das fleckenfreie Gel ermöglicht die quantitative Visualisierung von Tryptophan-haltigen Proteinen bei Aktivierung mit ultraviolettem Licht, während das Gel weiterhin für einen Western Blot verwendet werden kann.
- Stellen Sie sich das fleckenfreie Gel vor, um zu bestätigen, dass alle Proben gleich belastet sind. Dies ist eine wesentliche Kontrolle, die sicherstellt, dass Änderungen des Signals, die durch Western Blot erkannt werden, nicht auf eine variable Proteinbeladung zurückzuführen sind. Es sollten nur sichtbare Bänder für die Chaperone (GST-Calmodulin und GST-SGTA) in den INPUT- und ELUTION-Proben vorhanden sein. Erinnern Sie sich daran, dass die ELUTE-Probe konzentrierter ist als die INPUT-Probe.
- Montieren Sie eine Western-Blot-Kassette mit einer 45-μm-PVDF-Membran. Übertragung bei einem konstanten Strom von 300 mA für 60 Minuten.
- Nach dem Blockieren der Membran binden Sie 16 h bei 4 °C mit sanftem Schütteln ~15 U/min an den primären Antikörper. Blot für Substrat mit Kaninchen Anti-FLAG bei einer Verdünnung von 1:1.000.
HINWEIS: Die verwendeten primären und sekundären Antikörper sind substratspezifisch und die Konzentration für die Verwendung muss möglicherweise optimiert werden. - Membran waschen und mit sekundärem Antikörper, Ziegen-Anti-Kaninchen in einer Verdünnung von 1:10.000, mit sanftem Schütteln für 1 Stunde bei Raumtemperatur inkubieren.
- Waschen Sie Membran und Bild für die Analyse mit Western Blotting Detection Agent.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Um die Ergebnisse richtig zu interpretieren, müssen das fleckenfreie Gel und der Western Blot zusammen betrachtet werden. Das fleckenfreie Gel sorgt für eine gleichmäßige Belastung aller Proben. Bei der Betrachtung des fleckenfreien Gels sind die Chaperone (GST-Calmodulin und GST-SGTA) in den Spuren INPUT (I) und ELUTE (E) sichtbar. Überprüfen Sie, ob die Intensität dieser Bänder in allen INPUT-Samples einheitlich ist. Stellen Sie außerdem sicher, dass die Intensität in den ELUTE-Proben gleichmäßig ist. Der E...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Die richtige mitochondriale Funktion hängt von einem robusten Proteinqualitätskontrollsystem ab. Aufgrund inhärenter Grenzen in der Genauigkeit der TA-Protein-Targeting-Pfade sind falsch lokalisierte TA-Proteine eine ständige Stressquelle für Mitochondrien. Eine Schlüsselkomponente des mitochondrialen Proteostasenetzwerks ist Msp1, eine membranverankerte AAA + ATPase, die fehllokalisierte TA-Proteine aus dem OMM entfernt. Hier haben wir beschrieben, wie man Proteoliposomen herstellt, Msp1 und ein Modell-TA-Protein ...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Nichts
Danksagungen
MLW entwickelte einen Teil dieses Protokolls während seines Postdoc-Studiums bei Dr. Robert Keenan an der University of Chicago.
Diese Arbeit wird durch den NIH-Zuschuss 1R35GM137904-01 an MLW finanziert.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Biobeads | Bio-Rad | 1523920 | |
| Bovine liver phosphatidyl inositol | Avanti | 840042C | PI |
| Chicken egg phosphatidyl choline | Avanti | 840051C | PC |
| Chicken egg phosphatidyl ethanolamine | Avanti | 840021C | PE |
| ECL Select western blotting detection reagent | GE | RPN2235 | |
| Filter supports | Avanti | 610014 | |
| Glass vial | VWR | 60910L-1 | |
| Glutathione spin column | Thermo Fisher | PI16103 | |
| Goat anti-rabbit | Thermo Fisher | NC1050917 | |
| Mini-Extruder | Avanti | 610020 | |
| Polycarbonate membrane | Avanti | 610006 | 200 nM |
| PVDF membrane | Thermo Fisher | 88518 | 45 µM |
| Rabbit anti-FLAG | Sigma-Aldrich | F7245 | |
| Synthetic 1,2-dioleoyl-sn-glycero-3-phospho-L-serine | Avanti | 840035C | DOPS |
| Synthetic 1',3'-bis[1,2-dioleoyl-sn-glycero-3-phospho]-glycerol | Avanti | 710335C | TOCL |
| Syringe, 1 mL | Norm-Ject | 53548-001 | |
| Syringe, 1 mL, gas-tight | Avanti | 610017 |
Referenzen
- Song, J., Herrmann, J. M., Becker, T. Quality control of the mitochondrial proteome. Nature Reviews Molecular Cell Biology. 22, 54-70 (2021).
- Phillips, B. P., Miller, E. A. Membrane protein folding and quality control. Current Opinion in Structural Biology. 69, 50-54 (2021).
- Jiang, H. Quality control pathways of tail-anchored proteins. Biochimica et Biophysica Acta - Molecular Cell Research. 1868, 118922(2020).
- McKenna, M. J., et al. The endoplasmic reticulum P5A-ATPase is a transmembrane helix dislocase. Science. 369, New York, N.Y. (2020).
- Hegde, R. S., Zavodszky, E. Recognition and Degradation of Mislocalized Proteins in Health and Disease. Cold Spring Harbor Perspectives in Biology. 11, 033902(2019).
- Shao, S., Hegde, R. S. A calmodulin-dependent translocation pathway for small secretory proteins. Cell. 147, 1576-1588 (2011).
- Samuelson, J. C., et al. YidC mediates membrane protein insertion in bacteria. Nature. 406, 637-641 (2000).
- Anghel, S. A., McGilvray, P. T., Hegde, R. S., Keenan, R. J. Identification of Oxa1 Homologs Operating in the Eukaryotic Endoplasmic Reticulum. Cell Reports. 21, 3708-3716 (2017).
- Aviram, N., et al. The SND proteins constitute an alternative targeting route to the endoplasmic reticulum. Nature. 540, 134-138 (2016).
- Voorhees, R. M., Hegde, R. S. Structure of the Sec61 channel opened by a signal sequence. Science. 351, New York, NY. 88-91 (2016).
- Cichocki, B. A., Krumpe, K., Vitali, D. G., Rapaport, D. Pex19 is involved in importing dually targeted tail-anchored proteins to both mitochondria and peroxisomes. Traffic. 19, Copenhagen, Denmark. 770-785 (2018).
- Mateja, A., et al. Protein targeting. Structure of the Get3 targeting factor in complex with its membrane protein cargo. Science. 347, New York, NY. 1152-1155 (2015).
- Chacinska, A., Koehler, C. M., Milenkovic, D., Lithgow, T., Pfanner, N. Importing mitochondrial proteins: machineries and mechanisms. Cell. 138, 628-644 (2009).
- Chitwood, P. J., Hegde, R. S. An intramembrane chaperone complex facilitates membrane protein biogenesis. Nature. , (2020).
- Chitwood, P. J., Juszkiewicz, S., Guna, A., Shao, S., Hegde, R. S. EMC Is Required to Initiate Accurate Membrane Protein Topogenesis. Cell. 175, 1-30 (2018).
- Bock, F. J., Tait, S. W. G. Mitochondria as multifaceted regulators of cell death. Nature Reviews Molecular Cell Biology. 21, 85-100 (2020).
- Pfanner, N., Warscheid, B., Wiedemann, N. Mitochondrial proteins: from biogenesis to functional networks. Nature Reviews Molecular Cell Biology. 20, (2019).
- Bykov, Y. S., Rapaport, D., Herrmann, J. M., Schuldiner, M. Cytosolic Events in the Biogenesis of Mitochondrial Proteins. Trends in Biochemical Sciences. 45, 650-667 (2020).
- Pfanner, N., Warscheid, B., Wiedemann, N. Mitochondrial proteins: from biogenesis to functional networks. Nature Reviews Molecular Cell Biology. 427, 1135(2019).
- Borgese, N., Coy-Vergara, J., Colombo, S. F., Schwappach, B. The Ways of Tails: the GET Pathway and more. The Protein Journal. , 1-17 (2019).
- Mateja, A., Keenan, R. J. A structural perspective on tail-anchored protein biogenesis by the GET pathway. Current Opinion in Structural Biology. 51, 195-202 (2018).
- Chio, U. S., Cho, H., Shan, S. Mechanisms of Tail-Anchored Membrane Protein Targeting and Insertion. Annual review of cell and developmental biology. 33, 417-438 (2017).
- Denic, V. A portrait of the GET pathway as a surprisingly complicated young man. Trends in biochemical sciences. , (2012).
- Hegde, R. S., Keenan, R. J. Tail-anchored membrane protein insertion into the endoplasmic reticulum. Nature Reviews Molecular Cell Biology. 12, 787-798 (2011).
- Kalbfleisch, T., Cambon, A., Wattenberg, B. W. A bioinformatics approach to identifying tail-anchored proteins in the human genome. Traffic. 8, Copenhagen, Denmark. 1687-1694 (2007).
- Doan, K. N., et al. The Mitochondrial Import Complex MIM Functions as Main Translocase for α-Helical Outer Membrane Proteins. Cell Reports. 31, (2020).
- McDowell, M. A., et al. Structural Basis of Tail-Anchored Membrane Protein Biogenesis by the GET Insertase Complex. Molecular Cell. 80, (2020).
- Guna, A., Volkmar, N., Christianson, J. C., Hegde, R. S. The ER membrane protein complex is a transmembrane domain insertase. Science. 591, New York, NY. 3099(2017).
- Rao, M., et al. Multiple selection filters ensure accurate tail-anchored membrane protein targeting. eLife. 5, 21301(2016).
- Schuldiner, M., et al. The GET complex mediates insertion of tail-anchored proteins into the ER membrane. Cell. 134, 634-645 (2008).
- Chen, Y. -C., et al. Msp1/ATAD1 maintains mitochondrial function by facilitating the degradation of mislocalized tail-anchored proteins. The EMBO journal. 33, 1548-1564 (2014).
- Wu, X., Rapoport, T. A. Translocation of Proteins through a Distorted Lipid Bilayer. Trends in Cell Biology. , (2021).
- Phillips, B. P., Gomez-Navarro, N., Miller, E. A. Protein quality control in the endoplasmic reticulum. Current Opinion in Cell Biology. 65, 96-102 (2020).
- van de Weijer, M. L., et al. Quality Control of ER Membrane Proteins by the RNF185/Membralin Ubiquitin Ligase Complex. Molecular Cell. 79, (2020).
- Weir, N. R., Kamber, R. A., Martenson, J. S., Denic, V. The AAA protein Msp1 mediates clearance of excess tail-anchored proteins from the peroxisomal membrane. eLife. 6, 28507(2017).
- Gardner, B. M., et al. The peroxisomal AAA-ATPase Pex1/Pex6 unfolds substrates by processive threading. Nature communications. 9, 135(2018).
- Puchades, C., et al. Unique Structural Features of the Mitochondrial AAA+ Protease AFG3L2 Reveal the Molecular Basis for Activity in Health and Disease. Molecular Cell. , (2019).
- Castanzo, D. T., LaFrance, B., Martin, A. The AAA+ ATPase Msp1 is a processive protein translocase with robust unfoldase activity. Proceedings of the National Academy of Sciences of the United States of America. 117, 14970-14977 (2020).
- Wang, L., Myasnikov, A., Pan, X., Walter, P. Structure of the AAA protein Msp1 reveals mechanism of mislocalized membrane protein extraction. eLife. 9, (2020).
- Puchades, C., Sandate, C. R., Lander, G. C. The molecular principles governing the activity and functional diversity of AAA+ proteins. Nature Reviews Molecular Cell Biology. , 1-16 (2019).
- Yang, Y., et al. Folding-Degradation Relationship of a Membrane Protein Mediated by the Universally Conserved ATP-Dependent Protease FtsH. Journal of the American Chemical Society. , 10(2018).
- Baldridge, R. D., Rapoport, T. A. Autoubiquitination of the Hrd1 Ligase Triggers Protein Retrotranslocation in ERAD. Cell. 166, 394-407 (2016).
- Fresenius, H. L., Wohlever, M. L. Sorting out how Msp1 maintains mitochondrial membrane proteostasis. Mitochondrion. 49, 128-134 (2019).
- Wang, L., Walter, P. Msp1/ATAD1 in Protein Quality Control and Regulation of Synaptic Activities. Annual Review of Cell and Developmental Biology. 36, 1-24 (2020).
- Dederer, V., et al. Cooperation of mitochondrial and ER factors in quality control of tail-anchored proteins. eLife. 8, 1126(2019).
- Matsumoto, S., et al. Msp1 Clears Mistargeted Proteins by Facilitating Their Transfer from Mitochondria to the ER. Molecular Cell. , (2019).
- Li, L., Zheng, J., Wu, X., Jiang, H. Mitochondrial AAA-ATPase Msp1 detects mislocalized tail-anchored proteins through a dual-recognition mechanism. EMBO Reports. 20, (2019).
- Weidberg, H., Amon, A. MitoCPR - a surveillance pathway that protects mitochondria in response to protein import stress. Science. 360, New York, NY. (2018).
- Okreglak, V., Walter, P. The conserved AAA-ATPase Msp1 confers organelle specificity to tail-anchored proteins. Proceedings of the National Academy of Sciences of the United States of America. 111, (2014).
- Piard, J., et al. A homozygous ATAD1 mutation impairs postsynaptic AMPA receptor trafficking and causes a lethal encephalopathy. Brain. , (2018).
- Zhang, J., et al. The AAA+ ATPase Thorase regulates AMPA receptor-dependent synaptic plasticity and behavior. Cell. 145, 284-299 (2011).
- Prendergast, J., et al. Ganglioside regulation of AMPA receptor trafficking. The Journal of Neuroscience. 34, 13246-13258 (2014).
- Umanah, G. K. E., et al. Thorase variants are associated with defects in glutamatergic neurotransmission that can be rescued by Perampanel. Science Translational Medicine. 9, 4985(2017).
- Pignatelli, M., et al. Synaptic Plasticity onto Dopamine Neurons Shapes Fear Learning. Neuron. 93, 425-440 (2017).
- Zhang, J., et al. The AAA Thorase is neuroprotective against ischemic injury. Journal of Cerebral Blood Flow and Metabolism. , 271678(2018).
- Umanah, G. K. E., et al. AMPA Receptor Surface Expression Is Regulated by S-Nitrosylation of Thorase and Transnitrosylation of NSF. Cell Reports. 33, 108329(2020).
- Wohlever, M. L., Mateja, A., McGilvray, P. T., Day, K. J., Keenan, R. J. Msp1 Is a Membrane Protein Dislocase for Tail-Anchored Proteins. Molecular Cell. 67, 194-202 (2017).
- Lovell, J. F., et al. Membrane binding by tBid initiates an ordered series of events culminating in membrane permeabilization by Bax. Cell. 135, 1074-1084 (2008).
- Leshchiner, E. S., Braun, C. R., Bird, G. H., Walensky, L. D. Direct activation of full-length proapoptotic BAK. Proceedings of the National Academy of Sciences of the United States of America. 110, 986-995 (2013).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten