Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Aislamiento de células endoteliales retinianas murinas para secuenciación de próxima generación
En este artículo
Resumen
Este protocolo describe un método para el aislamiento de células endoteliales retinianas postnatales murinas optimizadas para el rendimiento, la pureza y la viabilidad celular. Estas células son adecuadas para los enfoques de secuenciación de próxima generación.
Resumen
Las mejoras recientes en la secuenciación de próxima generación han avanzado el conocimiento de los investigadores de la biología molecular y celular, con varios estudios que revelan nuevos paradigmas en biología vascular. La aplicación de estos métodos a modelos de desarrollo vascular requiere la optimización de técnicas de aislamiento celular a partir de tejidos embrionarios y postnatales. El rendimiento, la viabilidad y la pureza de la célula deben ser máximos para obtener resultados precisos y reproducibles de los enfoques de secuenciación de próxima generación. El modelo de vascularización retiniana neonatal de ratón es utilizado por los investigadores para estudiar los mecanismos del desarrollo vascular. Los investigadores han utilizado este modelo para investigar los mecanismos de la angiogénesis y la especificación del destino arterial-venoso durante la formación y maduración de los vasos sanguíneos. La aplicación de técnicas de secuenciación de próxima generación para estudiar el modelo de desarrollo vascular retiniano requiere la optimización de un método para el aislamiento de las células endoteliales de la retina que maximice el rendimiento, la viabilidad y la pureza celular. Este protocolo describe un método para el aislamiento, digestión y purificación del tejido retiniano murino mediante clasificación celular activada por fluorescencia (FACS). Los resultados indican que la población de células endoteliales CD31+/CD45- purificada por FACS está altamente enriquecida para la expresión génica de las células endoteliales y no presenta cambios en la viabilidad durante 60 minutos después del FACS. Se incluyen resultados representativos de enfoques de secuenciación de próxima generación en células endoteliales aisladas utilizando este método, incluida la secuenciación de ARN a granel y la secuenciación de ARN de células individuales, lo que demuestra que este método para el aislamiento de células endoteliales de la retina es compatible con las aplicaciones de secuenciación de próxima generación. Este método de aislamiento de células endoteliales retinianas permitirá técnicas avanzadas de secuenciación para revelar nuevos mecanismos de desarrollo vascular.
Introducción
La capacidad de alto rendimiento de la secuenciación de ácidos nucleicos a través de enfoques de secuenciación de próxima generación ha avanzado enormemente el conocimiento de los investigadores de biología molecular y celular. Estas técnicas avanzadas incluyen la secuenciación de ARN del transcriptoma completo, la secuenciación del ADN de regiones específicas para identificar polimorfismos de nucleótido único (SNP), la secuenciación del ADN de factores de transcripción unidos en la secuenciación de inmunoprecipitación de cromatina (ChIP) o regiones de cromatina abierta en el ensayo para la secuenciación de cromatina accesible por transposasa (ATAC) y la secuenciación de ARN unicelular1 . En biología vascular, estos avances han permitido a los investigadores dilucidar mecanismos complicados de desarrollo y enfermedad, junto con la distinción de patrones de expresión génica a lo largo de un continuo de fenotipos variables 2,3. Los experimentos futuros pueden definir aún más mecanismos complejos combinando la secuenciación de próxima generación con modelos evaluados de desarrollo vascular, pero los métodos para la preparación de muestras deben ser compatibles con las técnicas avanzadas de secuenciación.
La calidad, precisión y reproducibilidad de los enfoques de secuenciación de próxima generación dependen del método de preparación de la muestra. Al aislar un subconjunto de células o generar suspensiones unicelulares a partir de tejidos, los métodos óptimos de digestión y purificación son esenciales para maximizar el número celular, la viabilidad y la pureza de la población celular 4,5. Esto requiere un equilibrio en el método de digestión: la digestión fuerte es necesaria para liberar células del tejido y obtener suficientes células para los enfoques posteriores, pero la viabilidad celular se verá afectada negativamente si la digestión es demasiado fuerte 6,7. Además, la pureza de la población celular es necesaria para obtener resultados sólidos y un análisis preciso de los datos, que se puede lograr a través de FACS. Esto resalta la importancia de optimizar los métodos de aislamiento celular para aplicar la secuenciación de próxima generación a modelos establecidos de desarrollo vascular.
Un modelo bien caracterizado para investigar el desarrollo vascular es el modelo murino de desarrollo vascular retiniano. La vasculatura retiniana murina se desarrolla postnatalmente en un plexo superficial bidimensional, con brotación angiogénica inicial del nervio óptico visible en el día postnatal (P)3, frente angiogénico con células del tallo y de la punta y maduración inicial del vaso visible en P6, y maduración del plexo vascular visible después de P9 8,9. Durante la remodelación del plexo vascular inicial, las células endoteliales se especifican hacia fenotipos arteriales, capilares y venosos en diferentes vasos para generar una red circulatoria10,11. Por lo tanto, este método permite a los investigadores visualizar la formación del plexo vascular angiogénico y la especificación y maduración arterial-venosa endotelial en varios puntos de tiempo durante el desarrollo9. Además, este modelo proporciona un método para investigar los efectos de la manipulación transgénica sobre la angiogénesis y el desarrollo del plexo vascular, que se ha aplicado para la investigación del desarrollo vascular, malformaciones arteriales-venosas y neovascularización inducida por oxígeno 12,13,14,15,16 . Para combinar los enfoques de secuenciación de próxima generación con el modelo de desarrollo vascular retiniano murino, es necesario un protocolo optimizado para el aislamiento de las células endoteliales del tejido retiniano.
Este protocolo describe un método optimizado para digerir el tejido retiniano de ratones en P6 para maximizar el rendimiento celular, la pureza y la viabilidad. El tejido retiniano se aísla de ratones P6, se digiere durante 20 minutos, se inmunotiñe para CD31 y CD45 y se purifica a través de FACS para aislar una suspensión unicelular de células endoteliales en aproximadamente 2,5 h (Figura 1A). Se encontró que estas células endoteliales mantienen una alta viabilidad durante 60 minutos después del aislamiento17, lo que permite la preparación de la biblioteca para los métodos de secuenciación de próxima generación. Además, se proporcionan resultados representativos para la activación del sistema de control de control de calidad y el sistema de control de calidad de dos métodos separados de secuenciación de próxima generación que utilizan este protocolo de aislamiento: secuenciación de ARN del transcriptoma completo y secuenciación de ARN unicelular. Este método permite que los enfoques de secuenciación de próxima generación se utilicen junto con el modelo de vascularización retiniana para dilucidar nuevos mecanismos de desarrollo vascular.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Los Comités Institucionales de Cuidado y Uso de Animales de la Universidad de Yale y la Universidad de Virginia aprobaron todos los experimentos con animales enumerados en este protocolo.
1. Obtener ojos de ratón para aislamiento retiniano
- Prepare 1x PBS helado y agregue 500 μL a cada pocillo de una placa de 48 pocillos.
- Eutanasia de ratones neonatos en el sexto día postnatal (P6) de acuerdo con las directrices institucionales aprobadas. Para este experimento, camadas de aproximadamente 4-8 ratones neonatos son sacrificados en P6 a través de la inhalación de isoflurano durante al menos tres minutos después del paro respiratorio, seguido de la decapitación.
- Retire los ojos de cada uno de los ratones. Corte la piel y la membrana sobre el ojo cortando perpendicular al párpado con tijeras de disección. Luego, use fórceps para presionar suavemente hacia abajo por encima y por debajo del ojo para que el ojo salga de la cavidad.
- Pellizque cuidadosamente debajo del ojo con los fórceps y corte el nervio óptico que mantiene el ojo unido. Luego, coloque cada ojo en la placa de 48 pocillos en el PBS preparado 1x helado hasta que termine de cosechar.
NOTA: Utilice un pocillo por ratón, con ambos ojos de un solo ratón en el mismo pozo.
- Pellizque cuidadosamente debajo del ojo con los fórceps y corte el nervio óptico que mantiene el ojo unido. Luego, coloque cada ojo en la placa de 48 pocillos en el PBS preparado 1x helado hasta que termine de cosechar.
2. Aislar el tejido retiniano del ratón
- Llene una placa de Petri, forrada con una almohadilla de disección en la parte inferior, con 500 μL de PBS helado 1x para sumergir los ojos y colóquela bajo un microscopio de disección ajustado a un aumento de 4.0x.
- Suspenda los ojos en la almohadilla de disección con una pipeta de transferencia con una punta ancha para no dañar los ojos.
NOTA: Asegúrese de que los ojos estén completamente cubiertos con PBS.
- Suspenda los ojos en la almohadilla de disección con una pipeta de transferencia con una punta ancha para no dañar los ojos.
- Usando dos pinzas de disección finas, sostenga el nervio óptico con uno de los fórceps para la estabilización, y con el segundo fórceps perfore un orificio a través de la cámara anterior donde se conectan la córnea y la esclerótica (Figura 1B). Rasgue el agujero en un círculo alrededor del 75% del camino alrededor de la córnea.
- Mientras aún sostiene el nervio óptico con uno de los fórceps, use el segundo fórceps para arrancar suavemente la esclerótica del tejido retiniano.
- Como se mencionó anteriormente, use los segundos fórceps para arrancar suavemente el cuerpo vítreo del tejido retiniano.
- Nuevamente, usando el segundo fórceps, retire suavemente el cristalino y los vasos del plexo hialoide que no se desprendieron cuando se retiró el cuerpo vítreo. Para hacer esto, busque la retina con fórceps abiertos hasta casi tocar la retina, cierre las pinzas para agarrar la lente y los vasos del plexo hialoide, y saque las pinzas de la retina. Esto se puede repetir hasta que se retiren todos los vasos.
NOTA: El plexo hialoideo se asemeja a una estructura clara, similar a una red.
- Llene 2 ml de tubos de microcentrífuga con 500 μL de 1x PBS helado y coloque las retinas en los tubos. Aísle todas las retinas de los ojos y colóquelas en PBS helado antes de continuar.
NOTA: Utilice un tubo por ratón, con dos retinas del mismo ratón compartiendo un tubo. Si el tejido de la retina está desgarrado, transfiéralo en varias piezas. El tiempo total de disección del tejido retiniano para una camada de ratones debe tomar de 15 a 60 minutos, comenzando con la eutanasia y terminando con el aislamiento final del tejido retiniano.
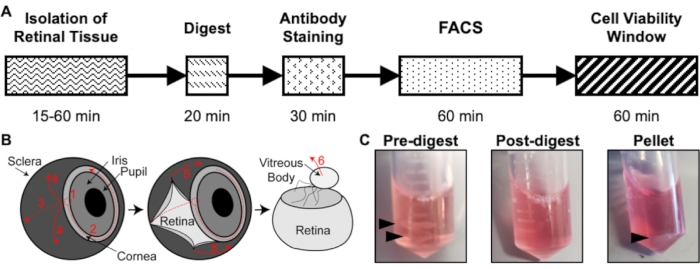
Figura 1: Descripción general del protocolo de aislamiento. (A) Esquema de la línea de tiempo de aislamiento con un tiempo estimado para cada paso: Aislamiento del tejido retiniano, digesto, tinción de anticuerpos, FACS y ventana de viabilidad celular. (B) Guía paso a paso para el aislamiento del tejido retiniano del ojo, con pasos de disección numerados: 1) perforar la córnea, 2) lárnea lagrimal, 3) esclerótica lagrimal, 4) eliminar la esclerótica de la retina, 5) eliminar aún más la esclerótica y el tejido de conexión de la retina, 6) eliminar el cuerpo vítreo y los vasos vítreos de la retina. (C) Imágenes representativas del tejido retiniano durante varios pasos de digestión: Pre-digestión, Post-digestión, Pellet (las flechas negras resaltan el tejido retiniano o la gránula celular). Republicado con permiso de Chavkin et al. publicado en S. Karger AG, Basilea17. Haga clic aquí para ver una versión más grande de esta figura.
3. Digerir el tejido retiniano en una suspensión de una sola célula
- Preparar una solución de digestión de 500 μL por cada dos retinas aisladas. Agregue FBS y colagenasa tipo II a DMEM a una concentración final de 10% FBS y 1 mg / ml de colagenasa tipo II. Mezclar y calentar la solución a 37 °C al baño maría.
- Elimine el exceso de PBS de los tubos de microcentrífuga de 2 ml pipeteando cuidadosamente. Deje suficiente PBS para cubrir completamente el tejido retiniano, aproximadamente 100 μL en cada tubo.
- Añadir 500 μL de la solución digestiva a cada tubo con tejido retiniano (Figura 1C).
- Use una pipeta P1000 y una punta de pipeta para pipetear hacia arriba y hacia abajo del tejido retiniano en la solución de digestión cinco veces.
- Incubar la mezcla de digestión en un baño maría a 37 °C durante 20 min.
- Utilice una pipeta P1000 y una punta de pipeta para pipetear la mezcla de digestión hacia arriba y hacia abajo cada 5 minutos. Después de la incubación, el tejido retiniano se ha disuelto en una suspensión de una sola célula, por lo que la mezcla de digestión debe estar turbia (Figura 1C).
4. Cuenta celdas
- Coloque una centrífuga de mesa a 4 °C y coloque los tubos de mezcla de digestión en su interior. Granular la mezcla de digestión por centrifugación a 375 x g durante 5 min.
- Retire con cuidado la solución de digestión sobrenadante mediante pipeteo. No perturbe el pellet celular (Figura 1C). Vuelva a suspender el pellet en 500 μL de 1x PBS helado mezclando suavemente hacia arriba y hacia abajo con la pipeta.
- Cuente las células con un hemocitómetro bajo un microscopio.
NOTA: Los recuentos celulares son aproximadamente 1 x 106 células por dos retinas. - Asegúrese de que las células se resuspendan correctamente mediante una mezcla suave, y luego alícuota 20 μL de suspensión celular en tres tubos para la tinción de control (tubo 1: control de IgG, tubo 2: control de CD31 y tubo 3: control de CD45), como se describió y utilizó anteriormente17,18.
- Coloque una centrífuga de sobremesa a 4 °C y coloque los tubos que contengan células en su interior. Granular las células por centrifugación a 375 x g durante 5 min.
5. Inmunoteñir las células con anticuerpos
- Preparar 100 μL de tampón de tinción por cada dos retinas teñidas más cuatro tubos de control. Agregue FBS, HEPES y D-glucosa al tampón HBSS a una concentración final de 10% FBS, 10 mM HEPES y 1 mg / ml D-glucosa.
- Obtener anticuerpos conjugados fluorescentes contra CD31 y CD45. Agregue los anticuerpos en una dilución 1:100 en el tampón de tinción (tubo 1: solo anticuerpo IgG, tubo 2: solo anticuerpo CD31 y tubo 3: solo anticuerpo CD45). Añadir 1 μL de anticuerpo por 100 μL del tampón de tinción para una concentración final de anticuerpos de 2 μg/ml.
- Retire con cuidado el PBS del pellet de celda lavada mediante pipeteo.
- Resuspender el pellet en 100 μL de solución de tinción de anticuerpos por 0,5 x 106 células.
- Incubar la suspensión unicelular con la solución de tinción de anticuerpos durante 30 minutos sobre hielo en la oscuridad. Golpee los tubos cada 10 minutos para mezclar suavemente las células.
6. Prepárese para la clasificación celular activada por fluorescencia
- Coloque una centrífuga de sobremesa a 4 °C y coloque los tubos que contienen células en su interior. Células de pellet por centrifugación a 375 x g durante 5 min.
- Retire el sobrenadante y resuspenda estas células en 500 μL de 1x PBS para lavar.
- Coloque una centrífuga de sobremesa a 4 °C y coloque los tubos que contienen células en su interior. Células de pellet por centrifugación a 375 x g durante 5 min
- Preparar 1,5 ml de buffer FACS (1% FBS en 1x PBS).
- Retire el sobrenadante y vuelva a suspender el pellet celular en 300 μL de tampón FACS mezclando suavemente con un pipete.
- Añadir yoduro de propidio (PI) a los tubos de muestra que contienen el tampón FACS hasta una concentración final de 0,5 μg/ml. PI se utiliza como marcador de viabilidad.
- Transfiera y combine las suspensiones celulares en tubos de ensayo de 5 ml a través de una tapa de presión del filtro celular. La tapa del filtro de la celda debe contener un filtro de 35 μm, y las suspensiones de la celda se pueden pipetear directamente sobre el filtro.
- Mantenga los tubos FACS en hielo en la oscuridad y transfiéralos al clasificador de celdas.
7. Configurar el instrumento FACS
- Instale una boquilla de 100 μm en un instrumento FACS.
NOTA: Esta boquilla de tamaño minimiza el volumen de recolección y maximiza la densidad celular aislada. - Preparar 250 μL de 1x PBS en un tubo de microcentrífuga de 1,5 ml como tubo de recolección para instalarlo en el instrumento FACS.
8. Aislar células endoteliales viables a través de FACS
- Encienda el instrumento FACS y el ordenador. Haga clic en el icono del software FACS para abrir el software FACS en la computadora para ejecutar y operar el instrumento FACS. Realizar pruebas rutinarias de control de calidad.
- Cargue las muestras teñidas por control en el instrumento FACS para ajustar los ejes. Comience con células de anticuerpos IgG solo para generar y ajustar FSC y SSC, luego con anticuerpos CD31 solo para generar y ajustar CD31, y luego con anticuerpos CD45 solo para generar y ajustar CD45. Registre datos de muestras de control.
- Cargue las células de muestra teñidas en el instrumento FACS y ejecute la muestra.
- Gatee las celdas según los parámetros de dispersión directa y dispersión lateral (FSC-A y SSC-A, respectivamente) (Figura 2A).
NOTA: Los parámetros FSC-A y SSC-A se utilizan para seleccionar celdas en función del tamaño, la densidad, la granularidad, las propiedades de la superficie y el índice de refracción. - Celdas de puerta por FSC-A y FSC-H para identificar dobletes celulares y recolectar solo celdas individuales (Figura 2A).
NOTA: Los parámetros FSC-A y FSC-H se utilizan para seleccionar celdas individuales basándose en el principio de que durante la dispersión hacia adelante, los dobletes de celda serán gotas que contienen una mayor relación área-altura. - Celdas de compuerta por PI y SSC-A para identificar celdas viables (Figura 2A).
NOTA: Las células viables serán PI-negativas. - Haga una puerta CD31+/CD45- con controles y celdas de puerta por CD31 y CD45 (Figura 2A,B).
- Insertar un tubo de recogida con 250 μL de tampón FACS.
- Comience a clasificar las células que son CD31-positivo/CD45-negativo en el tubo de recolección instalado.
- Mantenga los tubos de recolección en hielo para su posterior análisis. Las muestras se pueden procesar para aplicaciones de secuenciación de próxima generación en este paso1.
9. Realizar ensayo de viabilidad
- Mezclar 10 μL de células con 10 μL de solución de azul de tripano al 0,4% para evaluar la viabilidad celular.
- Cuente las células viables pipeteando la mezcla teñida en un portaobjetos del hemocitómetro. Coloque el portaobjetos bajo un microscopio para obtener recuentos de viabilidad precisos. Cuente las celdas azules como no viables y cuente las celdas claras como viables.
- Divida el recuento de células viables por el recuento total de células para calcular el porcentaje de viabilidad.
10. Realizar ensayo de expresión génica
- Obtener 10.000-20.000 células por muestra y realizar el aislamiento de ARN para analizar la expresión génica de las células endoteliales de la retina murina. El aislamiento de ARN se realiza utilizando un kit disponible comercialmente que utiliza precipitación de ARN a base de etanol, y luego centrifugación y columnas de membrana para lavar y eluyer el ARN purificado del lisado celular. Las muestras se pueden procesar para algunas aplicaciones de secuenciación de próxima generación en este paso1.
- Convertir el ARN en ADNc utilizando la enzima transcriptasa inversa y reactivos de soporte19.
- Cuantificar utilizando un colorante de unión al ADN y una máquina de PCR cuantitativa (qPCR)20. Cargue muestras de ADNc en una placa de qPCR de 384 pocillos con cebadores para amplificar genes de interés. El volumen de reacción debe ascender a 10 μL, incluido el colorante de unión al ADN, que se utilizará para cuantificar la amplificación de genes.
- Use CD31, VE-Cadherin, CD45 y cebadores directos e inversos de β-actina para medir la expresión génica.
- Usar el ejemplo para aplicaciones de secuenciación de próxima generación1.
Access restricted. Please log in or start a trial to view this content.
Resultados
La digestión del tejido retiniano y la inmunotinción para CD31 y CD45 dan como resultado una población identificable de células endoteliales CD31 + / CD45- después de la activación de células, células individuales y viabilidad (Figura 2A). La inmunotinción CD45 es necesaria para eliminar las células CD31+/CD45+, que incluyen plaquetas y algunos leucocitos21. Se deben realizar controles para cada experimento para mostrar la especificidad de anticuerpos y guia...
Access restricted. Please log in or start a trial to view this content.
Discusión
Este protocolo describe un método para el aislamiento de células endoteliales del tejido retiniano murino postnatal que se ha optimizado para un alto número de células, pureza y viabilidad. La pureza celular se obtiene mediante el aislamiento FACS de poblaciones de células endoteliales de la suspensión unicelular digerida mediante inmunotinción CD31+/CD45-. La calidad del aislamiento se cuantifica en ensayos de viabilidad mediante tinción con azul de tripano y expresión génica por qPCR para CD31, CD45 y VE-cadh...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen divulgaciones relevantes.
Agradecimientos
Gracias a Yale Flow Cytometry Facility, University of Virginia Flow Cytometry Core Facility, Yale Center for Genomic Analysis y University of Virginia Genome Analysis and Technology Core por su esfuerzo, experiencia y asesoramiento para contribuir a los experimentos presentados. Este estudio fue financiado por subvenciones de los NIH a N.W.C. (T32 HL007224, T32 HL007284), S.C. (T32 HL007284), K.W. (R01 HL142650) y K.K.H. (R01 HL146056, R01 DK118728, UH3 EB025765).
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 2 mL Eppendorf safe-lock tubes | USA Scientific | 4036-3352 | |
| 5 ml Falcon Test Tubes with Cell Strainer Snap Cap | Corning | 352235 | |
| 60 mm Non TC-treated Culture Dish | Corning | 430589 | |
| APC Rat Anti-Mouse CD31 | BD Biosciences | 551262 | |
| APC Rat IgG2a κ Isotype Control | BD Biosciences | 553932 | |
| BD FACSChorus Software | BD Biosciences | FACSCHORUS | |
| BD FACSMelody Cell Sorter | BD Biosciences | FACSMELODY | |
| Collagenase Type II | Sigma-Aldrich | 234115 | |
| Costar 48-well Clear TC-treated Multiple Well Plates, Individually Wrapped, Sterile | Corning | 3548 | |
| D-Glucose | Gibco | A2494001 | |
| Disposable Graduated Transfer Pipettes | Fisher Scientific | 12-711-9AM | |
| Dissecting Pan Wax | Carolina | 629100 | |
| Dissection scissors | Fine Science Tools | 14085-08 | |
| Dissection Stereo Microscope M165 FC | Leica | M165FC | |
| Dulbecco's Modified Eagle Medium (DMEM) | Gibco | 11965-052 | |
| Dulbecco’s Phosphate Buffered Saline (PBS) | Gibco | 14190144 | |
| Eppendorf Flex-Tubes Microcentrifuge Tubes 1.5 mL | Sigma-Aldrich | 22364120 | |
| Fetal Bovine Serum (FBS) | Gemini Bio | 100-106 | |
| Fine dissection forceps | Fine Science Tools | 11250-00 | |
| Hank's Buffered Salt Solution (HBSS) | Gibco | 14175095 | |
| HEPES (1M) | Gibco | 15630130 | |
| iScript cDNA Synthesis Kit | Bio-Rad | 1708890 | |
| Isoflurane, USP | Covetrus | 11695067772 | |
| Isotemp General Purpose Deluxe Water Bath | Fisher Scientific | FSGPD20 | |
| Primer: ActB_Forward: 5’- agagggaaatcgtgcgtgac -3’ | Integrated DNA Technologies | N/A | |
| Primer: ActB_Reverse: 5’- caatagtgatgacctggccgt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD31_Forward: 5’- gagcccaatcacgtttcagttt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD31_Reverse: 5’- tccttcctgcttcttgctagct -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD45_Forward: 5’- gggttgttctgtgccttgtt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD45_Reverse: 5’- ctggacggacacagttagca -3’ | Integrated DNA Technologies | N/A | |
| Primer: VE-Cadherin_Forward: 5’- tcctctgcatcctcactatcaca -3’ | Integrated DNA Technologies | N/A | |
| Primer: VE-Cadherin_Reverse: 5’- gtaagtgaccaactgctcgtgaat -3’ | Integrated DNA Technologies | N/A | |
| Propidium iodide | Sigma-Aldrich | P4864 | |
| RNeasy Plus Mini Kit | Qiagen | 74134 | |
| Sorvall Legend Micro 21R Centrifuge, Refrigerated | ThermoFisher | 75002477 | |
| SYBR-Green iTaq Universal SYBR Green Supermix | Bio-Rad | 172-5120 | |
| Trypan Blue Solution | ThermoFisher | 15250061 | |
| V450 Rat Anti-Mouse CD45 | BD Biosciences | 560501 | |
| V450 Rat IgG2b, κ Isotype Control | BD Biosciences | 560457 |
Referencias
- Slatko, B. E., Gardner, A. F., Ausubel, F. M. Overview of next-generation sequencing technologies. Current Protocols in Molecular Biology. 122 (1), 59(2018).
- Chavkin, N. W., Hirschi, K. K. Single cell analysis in vascular biology. Frontiers in Cardiovascular Medicine. 7, 42(2020).
- Ma, F., Hernandez, G. E., Romay, M., Iruela-Arispe, M. L. Single-cell RNA sequencing to study vascular diversity and function. Current Opinion in Hematology. 28 (3), 221-229 (2021).
- Potter, A. S., Potter, S. S., S, Dissociation of tissues for single-cell analysis. Methods in Molecular Biology. 1926, 55-62 (2019).
- Braga, F. A. V., Miragaia, R. J. Tissue handling and dissociation for single-cell RNA-seq. Single Cell Methods: Methods in Molecular Biology. 1979, 9-21 (2019).
- Brink, S. C., et al. Single-cell sequencing reveals dissociatin-induced gene expression in tissue subpopulations. Nature Methods. 14 (10), 935-936 (2017).
- Skulska, K., Wegrzyn, A. S., Chelmonska-Soyta, A., Chodaczek, G. Impact of tissue enzymatic digestion on analysis of immune cells in mouse reproductive mucosa with a focus on gammadelta T cells. Journal of Immunological Methods. 474, 112665(2019).
- Connolly, S., Hores, T., Smith, L., D'Amore, P. Characterization of vascular development in the mouse retina. Microvascular Research. 36 (3), 275-290 (1988).
- Crist, A., Young, C., Meadows, S. Characterization of arteriovenous identity in the developing neonate mouse retina. Gene Expression Patterns: GEP. 23-24, 22-31 (2017).
- dela Paz, N. G., D'Amore, P. A. Arterial versus venous endothelial cells. Cell and Tissue Research. 335 (1), 5-16 (2009).
- Fang, J. S., Hirschi, K. K. Molecular regulation of arteriovenous endothelial cell specification. F1000Res. 8, (2019).
- Smith, L. E., et al. Oxygen-induced retinopathy in the mouse. Investigative Ophthalmology and Visual Science. 35 (1), 101-111 (1994).
- Pitulescu, M. E., Schmidt, I., Benedito, R., Adams, R. H. Inducible gene targeting in the neonatal vasculature and analysis of retinal angiogenesis in mice. Nature Protocols. 5 (9), 1518-1534 (2010).
- Ruiz, S., et al. A mouse model of hereditary hemorrhagic telangiectasia generated by transmammary-delivered immunoblocking of BMP9 and BMP10. Scientific Reports. 6, 37366(2016).
- Fang, J. S., et al. Shear-induced Notch-Cx37-p27 axis arrests endothelial cell cycle to enable arterial specification. Nature Communication. 8 (1), 2149(2017).
- Ola, R., et al. SMAD4 prevents flow induced arterial-venous malformations by inhibiting Casein Kinase 2. Circulation. 138 (21), 2379-2394 (2018).
- Chavkin, N. W., Walsh, K., Hirschi, K. K. Isolation of highly purified and viable retinal endothelial cells. Journal of Vascular Research. 58 (1), 49-57 (2020).
- Hulspas, R., O'Gorman, M. R. G., Wood, B. L., Gratama, J. W., Sutherland, D. R. Considerations for the control of background fluorescence in clinical flow cytometry. Cytometry PartB: Clinical Cytometry. 76 (6), 355-364 (2009).
- Bachman, J. Reverse-transcription PCR (RT-PCR). Methods in Enzymology. 530, 67-74 (2013).
- Green, M. R., Sambrook, J. Quantification of RNA by real-time Reverse Transcription-Polymerase Chain Reaction (RT-PCR). Cold Spring Harbour Protocols. 2018 (10), (2018).
- Liu, L., Shi, G. P. CD31: beyond a marker for endothelial cells. Cardiovascular Research. 94 (1), 3-5 (2012).
- Zarkada, G., et al. Specialized endothelial tip cells guide neuroretina vascularization and blood-retina-barrier formation. Developmental Cell. 56 (15), 2237-2251 (2021).
- Su, X., Sorenson, C. M., Sheibani, N. Isolation and characterization of murine retinal endothelial cells. Molecular Vision. 9, 171-178 (2003).
- Benedito, R., et al. The notch ligands Dll4 and Jagged1 have opposing effects on angiogenesis. Cell. 137 (6), 1124-1135 (2009).
- Daneman, R., et al. Wnt/beta-catenin signaling is required for CNS, but not non-CNS, angiogenesis. Proceedings of the National Academy of Sciences of the United States of America. 106 (2), 641-646 (2009).
- Okabe, K., et al. Neurons limit angiogenesis by titrating VEGF in retina. Cell. 159 (3), 584-596 (2014).
- Crist, A. M., Lee, A. R., Patel, N. R., Westhoff, D. E., Meadows, S. M. Vascular deficiency of Smad4 causes arteriovenous malformations: a mouse model of Hereditary Hemorrhagic Telangiectasia. Angiogenesis. 21 (2), 363-380 (2018).
- Kim, Y. H., Choe, S. W., Chae, M. Y., Hong, S., Oh, S. P. SMAD4 deficiency leads to development of arteriovenous malformations in neonatal and adult mice. Journal of the American Heart Association. 7 (21), 009514(2018).
- Ma, W., et al. Absence of TGFbeta signaling in retinal microglia induces retinal degeneration and exacerbates choroidal neovascularization. eLife. 8, 42049(2019).
- Luo, W., et al. Arterialization requires the timely suppression of cell growth. Nature. 589 (7842), 437-441 (2021).
- Lawson, N. D., Vogel, A. M., Weinstein, B. M. sonic hedgehog and vascular endothelial growth factor act upstream of the Notch pathway during arterial endothelial differentiation. Developmental Cell. 3 (1), 127-136 (2002).
- Larrivee, B., et al. ALK1 signaling inhibits angiogenesis by cooperating with the Notch pathway. Developmental Cell. 22 (3), 489-500 (2012).
- Wythe, J. D., et al. ETS factors regulate Vegf-dependent arterial specification. Developmental Cell. 26 (1), 45-58 (2013).
- Davis, D. M., Purvis, J. E. Computational analysis of signaling patterns in single cells. Seminars in Cell and Developmental Biology. 0, 35-43 (2015).
- Gaudet, S., Miller-Jensen, K. Redefining signaling pathways with an expanding single-cell toolbox. Trends in Biotechnology. 34 (6), 458-469 (2016).
- Aibar, S., et al. SCENIC: single-cell regulatory network inference and clustering. Nature Methods. 14 (11), 1083-1086 (2017).
- Trapnell, C., et al. The dynamics and regulators of cell fate decisions are revealed by pseudotemporal ordering of single cells. Nature Biotechnology. 32 (4), 381-386 (2014).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados