JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
차세대 염기서열분석을 위한 뮤린 망막 내피 세포의 분리
요약
이 프로토콜은 세포 수율, 순도 및 생존성에 최적화된 뮤린 출생후 망막 내피 세포의 단리를 위한 방법을 설명한다. 이 세포는 차세대 시퀀싱 접근법에 적합합니다.
초록
차세대 시퀀싱의 최근 개선은 분자 및 세포 생물학에 대한 연구자의 지식을 향상 시켰으며 여러 연구에서 혈관 생물학의 새로운 패러다임을 밝혀 냈습니다. 이러한 방법을 혈관 발달 모델에 적용하려면 배아 및 출생 후 조직에서 세포 분리 기술을 최적화해야합니다. 세포 수율, 생존력 및 순도는 모두 차세대 염기서열분석 접근법에서 정확하고 재현 가능한 결과를 얻기 위해 최대여야 합니다. 신생아 마우스 망막 혈관 형성 모델은 연구자들이 혈관 발달 메커니즘을 연구하는 데 사용됩니다. 연구원들은 이 모델을 사용하여 혈관 형성 및 성숙 동안 혈관신생 및 동맥-정맥 운명 사양의 메커니즘을 조사했습니다. 망막 혈관 개발 모델을 연구하기 위해 차세대 시퀀싱 기술을 적용하려면 세포 수율, 생존력 및 순도를 극대화하는 망막 내피 세포의 분리 방법을 최적화해야 합니다. 이 프로토콜은 형광 활성화 세포 분류 (FACS)를 사용하여 쥐 망막 조직 분리, 소화 및 정제하는 방법을 설명합니다. 상기 결과는 FACS-정제된 CD31+/CD45-내피 세포 집단이 내피 세포 유전자 발현에 대해 고도로 농축되고, FACS 후 60분 동안 생존력의 변화를 나타내지 않음을 나타낸다. 여기에는 벌크 RNA 시퀀싱 및 단일 세포 RNA 시퀀싱을 포함하여 이 방법을 사용하여 분리된 내피 세포에 대한 차세대 시퀀싱 접근법의 대표적인 결과가 포함되어 망막 내피 세포 분리를 위한 이 방법이 차세대 시퀀싱 애플리케이션과 호환됨을 보여줍니다. 이 망막 내피 세포 분리 방법은 혈관 발달의 새로운 메커니즘을 밝히는 고급 시퀀싱 기술을 허용합니다.
서문
차세대 염기서열분석 접근법을 통한 핵산 염기서열분석의 높은 처리량은 분자 및 세포 생물학에 대한 연구자들의 지식을 크게 발전시켰습니다. 이러한 고급 기술에는 전체 전사체 RNA 시퀀싱, 단일 염기 다형성(SNP)을 식별하기 위한 표적 영역의 DNA 시퀀싱, 염색질 면역침전(ChIP) 시퀀싱에서 결합된 전사 인자의 DNA 시퀀싱 또는 트랜스포아제 접근 가능 염색질(ATAC) 시퀀싱을 위한 분석에서 열린 염색질 영역 및 단일 세포 RNA 시퀀싱1이 포함됩니다. . 혈관 생물학에서 이러한 발전으로 연구자들은 다양한 표현형 2,3의 연속체를 따라 유전자 발현 패턴을 구별하는 것과 함께 복잡한 발달 및 질병 메커니즘을 밝힐 수있었습니다. 향후 실험에서는 차세대 염기서열분석과 평가된 혈관 발달 모델을 결합하여 복잡한 메커니즘을 추가로 정의할 수 있지만, 시료 전처리 방법은 고급 염기서열분석 기법과 호환되어야 합니다.
차세대 염기서열분석 접근법의 품질, 정확성 및 재현성은 시료 전처리 방법에 따라 달라집니다. 세포의 하위 집합을 분리하거나 조직에서 단일 세포 현탁액을 생성할 때세포 집단의 세포 수, 생존력 및 순도를 최대화하기 위해서는 최적의 분해 및 정제 방법이 필수적입니다4,5. 이것은 소화 방법의 균형을 필요로합니다 : 조직에서 세포를 방출하고 다운 스트림 접근을위한 충분한 세포를 얻으려면 강한 소화가 필요하지만 소화가 너무 강하면 세포 생존율에 부정적인 영향을 미칩니다 6,7. 또한 FACS를 통해 달성할 수 있는 강력한 결과와 정확한 데이터 분석을 위해서는 세포 집단의 순도가 필요합니다. 이는 확립된 혈관 발달 모델에 차세대 염기서열분석을 적용하기 위해 세포 분리 방법을 최적화하는 것의 중요성을 강조합니다.
혈관 발달을 조사하기위한 잘 특성화 된 모델은 쥐 망막 혈관 발달 모델입니다. 뮤린 망막 혈관 구조는 출생 후 2 차원 표면 신경총에서 발생하며, 출생 후 일 (P) 3에 보이는 시신경에서 초기 혈관 신생 싹이 트고, 줄기 및 팁 세포가있는 혈관 신생 전선과 P6에서 볼 수있는 초기 혈관 성숙, P9 8,9 이후에 볼 수있는 혈관 신경총의 성숙. 초기 혈관 신경총의 리모델링 동안, 내피 세포는 순환 네트워크(10,11)를 생성하기 위해 상이한 혈관에서 동맥, 모세 혈관 및 정맥 표현형에 대한 사양을 거친다. 따라서, 이 방법을 통해연구자들은 발달 중 다양한 시점에서 혈관신생 혈관 신경총 형성 및 내피 동맥-정맥 사양 및 성숙을 시각화할 수 있습니다9. 또한, 이 모델은 혈관 발달, 동맥-정맥 기형 및 산소 유도 신생혈관의 조사에 적용된 혈관신생 및 혈관 신경총 발달에 대한 형질전환 조작의 효과를 조사하는 방법을 제공합니다 12,13,14,15,16 . 차세대 염기서열분석 접근법과 쥐 망막 혈관 개발 모델을 결합하려면 망막 조직에서 내피 세포를 분리하기 위한 최적화된 프로토콜이 필요합니다.
이 프로토콜은 P6에서 마우스의 망막 조직을 소화하여 세포 수율, 순도 및 생존력을 극대화하는 최적화된 방법을 설명합니다. 망막 조직을 P6 마우스로부터 분리하고, 20분 동안 소화시키고, CD31 및 CD45에 대해 면역염색하고, FACS를 통해 정제하여 약 2.5시간 내에 내피 세포의 단일 세포 현탁액을 분리한다(도 1A). 이러한 내피 세포는 분리 후 60분 동안 높은 생존력을 유지하는 것으로 밝혀졌으며17, 차세대 시퀀싱 방법을 위한 라이브러리 준비를 가능하게 합니다. 또한 FACS 게이팅 및 품질 관리 결과에 대한 대표 결과가 이 분리 프로토콜을 사용하는 두 가지 개별 차세대 시퀀싱 방법인 전체 전사체 RNA 시퀀싱 및 단일 세포 RNA 시퀀싱에서 제공됩니다. 이 방법을 사용하면 망막 혈관 형성 모델과 함께 차세대 시퀀싱 접근법을 사용하여 혈관 발달의 새로운 메커니즘을 밝힐 수 있습니다.
프로토콜
예일 대학교와 버지니아 대학교의 기관 동물 관리 및 사용위원회는이 프로토콜에 나열된 모든 동물 실험을 승인했습니다.
1. 망막 분리를 위한 마우스 눈 획득
- 1x 얼음처럼 차가운 PBS를 준비하고 48웰 플레이트의 각 웰에 500μL를 추가합니다.
- 승인된 기관 지침에 따라 출생 후 6일(P6)에 신생아 마우스를 안락사시킵니다. 이 실험을 위해, 약 4-8 신생아 마우스의 새끼는 호흡 정지 후 적어도 3 분 동안 이소 플루 란 흡입을 통해 P6에서 안락사 된 후 참수된다.
- 각 마우스에서 눈을 제거하십시오. 해부 가위를 사용하여 눈꺼풀에 수직으로 잘라 눈 위의 피부와 막을 잘라냅니다. 그런 다음 집게를 사용하여 눈 위와 아래를 부드럽게 눌러 눈이 소켓에서 나오도록 합니다.
- 집게로 눈 밑을 조심스럽게 꼬집고 눈을 붙여있는 시신경을 자릅니다. 그런 다음 수확이 완료될 때까지 준비된 1x 얼음처럼 차가운 PBS의 48웰 플레이트에 각 눈을 놓습니다.
참고: 마우스당 하나의 웰을 사용하고 동일한 웰에 있는 단일 마우스의 두 눈을 사용합니다.
- 집게로 눈 밑을 조심스럽게 꼬집고 눈을 붙여있는 시신경을 자릅니다. 그런 다음 수확이 완료될 때까지 준비된 1x 얼음처럼 차가운 PBS의 48웰 플레이트에 각 눈을 놓습니다.
2. 마우스 망막 조직 분리
- 바닥에 해부 패드가 늘어선 페트리 접시에 500μL의 얼음처럼 차가운 PBS 1개를 채워 눈을 담그고 4.0배 배율로 설정된 해부 현미경 아래에 놓습니다.
- 눈을 손상시키지 않도록 넓은 팁이있는 이송 피펫을 사용하여 해부 패드에 눈을 매달아 놓습니다.
알림: 눈이 PBS로 완전히 덮여 있는지 확인하십시오.
- 눈을 손상시키지 않도록 넓은 팁이있는 이송 피펫을 사용하여 해부 패드에 눈을 매달아 놓습니다.
- 두 개의 미세 해부 집게를 사용하여 안정화를 위해 집게 중 하나로 시신경을 잡고 두 번째 집게로 각막과 공막이 연결되는 전방을 통해 구멍을 뚫습니다 (그림 1B). 각막 주위의 약 75 %를 원으로 구멍을 뚫습니다.
- 집게 중 하나로 시신경을 유지하면서 두 번째 집게를 사용하여 망막 조직에서 공막을 부드럽게 찢습니다.
- 위와 같이 두 번째 집게를 사용하여 망막 조직의 유리체를 부드럽게 찢습니다.
- 다시, 두 번째 집게를 사용하여 유리체를 제거 할 때 벗겨지지 않은 렌즈와 히알로이드 신경총 혈관을 부드럽게 제거하십시오. 이렇게하려면 망막에 거의 닿을 때까지 열린 집게로 망막에 손을 뻗고 집게를 닫아 렌즈와 히알로이드 신경총 혈관을 잡고 망막에서 집게를 빼냅니다. 이것은 모든 혈관이 제거 될 때까지 반복 될 수 있습니다.
참고: 히알로이드 신경총은 명확하고 웹과 같은 구조와 유사합니다.
- 2mL 마이크로 원심분리 튜브에 500μL의 1x 얼음처럼 차가운 PBS를 채우고 망막을 튜브에 넣습니다. 계속 진행하기 전에 눈에서 모든 망막을 분리하고 얼음처럼 차가운 PBS에 넣으십시오.
참고: 마우스당 하나의 튜브를 사용하고 동일한 마우스의 망막 두 개가 튜브를 공유합니다. 망막 조직이 찢어진 경우 여러 조각으로 옮깁니다. 쥐의 깔짚에 대한 망막 조직 해부의 총 시간은 안락사로 시작하여 최종 망막 조직 분리로 끝나는 15-60 분이 소요됩니다.
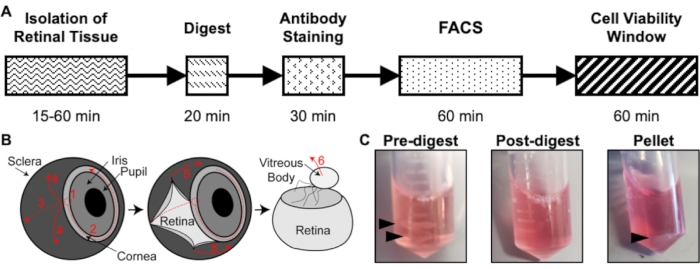
그림 1: 격리 프로토콜 개요. (A) 각 단계에 대한 예상 시간이 포함된 분리 타임라인의 개략도: 망막 조직 분리, 분해, 항체 염색, FACS 및 세포 생존율 창. (B) 번호가 매겨진 해부 단계와 함께 눈에서 망막 조직을 분리하기위한 단계별 가이드 : 1) 각막 관통, 2) 각막 파열, 3) 눈물 공막, 4) 망막에서 공막 제거, 5) 망막에서 공막 및 연결 조직 제거, 6) 망막에서 유리체 및 유리체 혈관 제거. (C) 다양한 소화 단계 동안 망막 조직의 대표 이미지 : 사전 소화, 사후 소화, 펠렛 (검은 색 화살표는 망막 조직 또는 세포 펠릿을 강조 표시). Chavkin et al.의 허가를 받아 재출판 S. Karger AG, Basel17. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
3. 망막 조직을 단일 세포 현탁액으로 소화
- 분리된 망막 2개마다 500μL 분해 용액을 준비합니다. FBS와 콜라게나제 유형 II를 DMEM에 추가하여 최종 농도 10% FBS 및 1mg/mL 콜라게나제 유형 II에 추가합니다. 혼합하고 수조를 사용하여 용액을 37°C로 가온한다.
- 조심스럽게 피펫팅하여 2mL 미세 원심분리 튜브에서 과도한 PBS를 제거합니다. 망막 조직을 완전히 덮을 수 있을 만큼 충분한 PBS를 각 튜브에 약 100μL씩 남겨 둡니다.
- 망막 조직이 있는 각 튜브에 500μL의 분해 용액을 추가합니다(그림 1C).
- P1000 피펫터와 피펫 팁을 사용하여 분해 용액의 망막 조직을 위아래로 5회 피펫팅합니다.
- 소화 혼합물을 37°C 수조에서 20분 동안 배양합니다.
- P1000 피펫터와 피펫 팁을 사용하여 5분마다 분해 혼합물을 위아래로 피펫팅합니다. 배양 후 망막 조직은 단일 세포 현탁액으로 용해되므로 소화 혼합물이 흐려야 합니다(그림 1C).
4. 셀 계산
- 탁상용 원심분리기를 4°C에 놓고 분해 혼합물 튜브를 내부에 넣습니다. 소화 혼합물을 375 x g 에서 5분 동안 원심분리하여 펠릿화합니다.
- 피펫팅으로 상청액 소화 용액을 조심스럽게 제거합니다. 셀 펠릿을 방해하지 마십시오(그림 1C). 피펫과 위아래로 부드럽게 혼합하여 500μL의 얼음처럼 차가운 1x PBS에 펠릿을 다시 현탁합니다.
- 현미경으로 혈구 측정기로 세포를 세십시오.
참고: 세포 수는 망막 2개당 약 1 x 106 세포입니다. - 세포가 온화한 혼합에 의해 적절하게 재현탁되었는지 확인한 다음, 이전에 설명되고 사용된 바와 같이 대조군 염색을 위해 20μL의 세포 현탁액을 3개의 튜브(튜브 1: IgG 대조군, 튜브 2: CD31 대조군 및 튜브 3: CD45 대조군)로 분취합니다(17,18).
- 탁상용 원심분리기를 4°C에 놓고 내부에 셀이 들어 있는 튜브를 놓습니다. 세포를 375 x g 에서 5분 동안 원심분리하여 펠릿화합니다.
5. 항체로 세포를 면역염색
- 염색된 망막 2개와 대조군 튜브 4개당 100μL의 염색 완충액을 준비합니다. FBS, HEPES 및 D-포도당을 HBSS 버퍼에 추가하여 최종 농도 10% FBS, 10mM hepes 및 1mg/mL D-포도당을 추가합니다.
- CD31 및 CD45에 대한 형광 접합 항체를 얻습니다. 항체를 1:100 희석액으로 염색 완충액에 추가합니다(튜브 1: IgG 항체만, 튜브 2: CD31 항체만, 튜브 3: CD45 항체만). 2μg/mL의 최종 항체 농도에 대해 염색 버퍼 100μL당 1μL의 항체를 추가합니다.
- 피펫팅으로 세척된 세포 펠릿에서 PBS를 조심스럽게 제거합니다.
- 펠렛을 0.5 x 106 세포당 100 μL의 항체 염색 용액에 재현탁시킨다.
- 단일 세포 현탁액을 항체 염색 용액과 함께 어두운 얼음 위에서 30분 동안 인큐베이션합니다. 10분마다 튜브를 두드려 세포를 부드럽게 섞습니다.
6. 형광 활성화 세포 분류 준비
- 탁상용 원심분리기를 4°C로 설정하고 내부에 세포가 들어 있는 튜브를 놓습니다. 펠렛 세포를 375 x g 에서 5분 동안 원심분리합니다.
- 상청액을 제거하고 이들 세포를 500 μL의 1x PBS에 재현탁시켜 세척한다.
- 탁상용 원심분리기를 4°C로 설정하고 내부에 세포가 들어 있는 튜브를 놓습니다. 375 x g 에서 5분 동안 원심분리하여 펠렛 세포
- 1.5mL의 FACS 버퍼(1x PBS 중 1% FBS)를 준비합니다.
- 상청액을 제거하고 피펫터와 부드럽게 혼합하여 300μL의 FACS 완충액에 세포 펠릿을 재현탁합니다.
- FACS 버퍼가 포함된 샘플 튜브에 요오드화프로피듐(PI)을 최종 농도 0.5μg/mL로 추가합니다. PI는 생존력 마커로 사용됩니다.
- 세포 여과기 스냅 캡을 통해 세포 현탁액을 5mL 시험관으로 옮기고 결합합니다. 셀 스트레이너 스냅 캡에는 35μm 필터가 포함되어야 하며 셀 현탁액을 필터에 직접 피펫팅할 수 있습니다.
- FACS 튜브를 어둠 속에서 얼음 위에 놓고 세포 분류기로 옮깁니다.
7. FACS 계측기 설정
- FACS 기기에 100μm 노즐을 설치합니다.
알림: 이 크기의 노즐은 포집량을 최소화하고 격리된 셀 밀도를 최대화합니다. - FACS 기기에 설치할 수집 튜브로 250μL의 1x PBS를 1.5mL 미세 원심분리 튜브에 준비합니다.
8. FACS 를 통해 생존 가능한 내피 세포 분리
- FACS 계측기와 컴퓨터를 켭니다. FACS 소프트웨어 아이콘을 클릭하여 컴퓨터에서 FACS 소프트웨어를 열어 FACS 기기를 실행하고 작동합니다. 일상적인 품질 관리 테스트를 수행합니다.
- 제어 염색 샘플을 FACS 기기에 로드하여 축을 조정합니다. IgG 항체 전용 세포로 시작하여 FSC 및 SSC를 생성 및 조정한 다음 CD31-항체만 생성하여 조정한 다음 CD45-항체만 생성하여 CD45를 생성하고 조정합니다. 대조군 샘플의 데이터를 기록합니다.
- 염색된 샘플 셀을 FACS 기기에 로드하고 샘플을 실행합니다.
- 전방 산란 및 측면 산란 (각각 FSC-A 및 SSC-A) 매개 변수를 기반으로 세포를 게이트합니다 (그림 2A).
참고: FSC-A 및 SSC-A 매개변수는 크기, 밀도, 입도, 표면 특성 및 굴절률을 기준으로 셀을 선택하는 데 사용됩니다. - FSC-A 및 FSC-H에 의한 게이트 셀은 셀 더블릿을 식별하고 단일 셀만 수집합니다(그림 2A).
참고: FSC-A 및 FSC-H 매개변수는 전방 산란 중에 셀 이중선이 더 큰 면적 대 높이 비율을 포함하는 액적이 된다는 원칙에 따라 단일 셀을 선택하는 데 사용됩니다. - 생존 세포를 확인하기 위해 PI 및 SSC-A에 의한 게이트 세포(도 2A).
참고: 생존 가능한 세포는 PI 음성입니다. - CD31+/CD45- 게이트를 대조군으로 만들고 CD31 및 CD45에 의해 세포를 게이트합니다(그림 2A,B).
- 250μL의 FACS 버퍼가 있는 수집 튜브를 삽입합니다.
- CD31 양성/CD45 음성인 세포를 설치된 수집 튜브로 분류하기 시작합니다.
- 추가 분석을 위해 수집 튜브를 얼음 위에 보관하십시오. 이단계 1에서 차세대 시퀀싱 애플리케이션을 위해 샘플을 처리할 수 있습니다.
9. 생존력 분석 수행
- 10μL의 세포와 10μL의 0.4% 트리판 블루 용액을 혼합하여 세포 생존율을 평가합니다.
- 염색된 혼합물을 혈구측정기 슬라이드에 피펫팅하여 생존 세포를 계수합니다. 정확한 생존력 계산을 위해 슬라이드를 현미경 아래에 놓습니다. 파란색 세포를 실행 불가능한 것으로 계산하고 명확한 세포를 실행 가능한 것으로 계산합니다.
- 생존 가능한 세포 수를 총 세포 수로 나누어 생존율을 계산합니다.
10. 유전자 발현 분석 수행
- 샘플당 10,000-20,000개의 세포를 얻고 RNA 분리를 수행하여 쥐 망막 내피 세포 유전자 발현을 분석합니다. RNA 분리는 에탄올 기반 RNA 침전을 사용하는 시판되는 키트를 사용하여 수행된 다음 원심분리 및 멤브레인 컬럼을 사용하여 세포 용해물에서 정제된 RNA를 세척하고 용리합니다. 이 단계1에서 일부 차세대 시퀀싱 애플리케이션을 위한 샘플을 처리할 수 있습니다.
- 역전사효소 및 지지 시약을 사용하여 RNA를 cDNA로 전환시킨다19.
- DNA 결합 염료 및 정량 PCR(qPCR) 기계(20)를 사용하여 정량한다. 관심 유전자를 증폭하기 위해 프라이머가 있는 384웰 qPCR 플레이트에 cDNA 샘플을 로드합니다. 반응 부피는 유전자 증폭을 정량화하는 데 사용될 DNA 결합 염료를 포함하여 총 10μL여야 합니다.
- CD31, VE-Cadherin, CD45 및 β-액틴 정방향 및 역방향 프라이머를 사용하여 유전자 발현을 측정합니다.
- 차세대 시퀀싱 어플리케이션에 샘플 사용1.
결과
망막 조직의 소화 및 CD31 및 CD45에 대한 면역염색은 세포, 단일 세포 및 생존력에 대한 게이팅 후 CD31+/CD45-내피 세포의 식별 가능한 집단을 초래합니다(그림 2A). CD45 면역염색은 혈소판 및 일부 백혈구를 포함하는 CD31+/CD45+ 세포를 제거하는 데 필요합니다21. 항체 특이성을 보여주고 게이팅 전략을 안내하기 위해 각 실험에 대해 대조군을 수행해야 합니다(
토론
이 프로토콜은 높은 세포 수, 순도 및 생존력에 최적화된 출생 후 뮤린 망막 조직으로부터 내피 세포를 분리하는 방법을 설명합니다. 세포 순도는 CD31+/CD45-면역염색에 의해 소화된 단일-세포 현탁액으로부터 내피 세포 집단의 FACS 단리에 의해 수득된다. 분리 품질은 트리판 블루 염색에 의한 생존력 분석 및 CD31, CD45 및 VE-카데린에 대한 qPCR에 의한 유전자 발현에 대한 분석에서 정량화됩니다(VE-카?...
공개
저자는 관련 공개가 없습니다.
감사의 말
Yale Flow Cytometry Facility, University of Virginia Flow Cytometry Core Facility, Yale Center for Genomic Analysis 및 University of Virginia Genome Analysis and Technology Core에 감사드립니다. 이 연구는 NUC (T32 HL007224, T32 HL007284), SC (T32 HL007284), KW (R01 HL142650) 및 K.K.H. (R01 HL146056, R01 DK118728, UH3 EB025765)에 대한 NIH 보조금으로 자금을 지원받았습니다.
자료
| Name | Company | Catalog Number | Comments |
| 2 mL Eppendorf safe-lock tubes | USA Scientific | 4036-3352 | |
| 5 ml Falcon Test Tubes with Cell Strainer Snap Cap | Corning | 352235 | |
| 60 mm Non TC-treated Culture Dish | Corning | 430589 | |
| APC Rat Anti-Mouse CD31 | BD Biosciences | 551262 | |
| APC Rat IgG2a κ Isotype Control | BD Biosciences | 553932 | |
| BD FACSChorus Software | BD Biosciences | FACSCHORUS | |
| BD FACSMelody Cell Sorter | BD Biosciences | FACSMELODY | |
| Collagenase Type II | Sigma-Aldrich | 234115 | |
| Costar 48-well Clear TC-treated Multiple Well Plates, Individually Wrapped, Sterile | Corning | 3548 | |
| D-Glucose | Gibco | A2494001 | |
| Disposable Graduated Transfer Pipettes | Fisher Scientific | 12-711-9AM | |
| Dissecting Pan Wax | Carolina | 629100 | |
| Dissection scissors | Fine Science Tools | 14085-08 | |
| Dissection Stereo Microscope M165 FC | Leica | M165FC | |
| Dulbecco's Modified Eagle Medium (DMEM) | Gibco | 11965-052 | |
| Dulbecco’s Phosphate Buffered Saline (PBS) | Gibco | 14190144 | |
| Eppendorf Flex-Tubes Microcentrifuge Tubes 1.5 mL | Sigma-Aldrich | 22364120 | |
| Fetal Bovine Serum (FBS) | Gemini Bio | 100-106 | |
| Fine dissection forceps | Fine Science Tools | 11250-00 | |
| Hank's Buffered Salt Solution (HBSS) | Gibco | 14175095 | |
| HEPES (1M) | Gibco | 15630130 | |
| iScript cDNA Synthesis Kit | Bio-Rad | 1708890 | |
| Isoflurane, USP | Covetrus | 11695067772 | |
| Isotemp General Purpose Deluxe Water Bath | Fisher Scientific | FSGPD20 | |
| Primer: ActB_Forward: 5’- agagggaaatcgtgcgtgac -3’ | Integrated DNA Technologies | N/A | |
| Primer: ActB_Reverse: 5’- caatagtgatgacctggccgt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD31_Forward: 5’- gagcccaatcacgtttcagttt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD31_Reverse: 5’- tccttcctgcttcttgctagct -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD45_Forward: 5’- gggttgttctgtgccttgtt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD45_Reverse: 5’- ctggacggacacagttagca -3’ | Integrated DNA Technologies | N/A | |
| Primer: VE-Cadherin_Forward: 5’- tcctctgcatcctcactatcaca -3’ | Integrated DNA Technologies | N/A | |
| Primer: VE-Cadherin_Reverse: 5’- gtaagtgaccaactgctcgtgaat -3’ | Integrated DNA Technologies | N/A | |
| Propidium iodide | Sigma-Aldrich | P4864 | |
| RNeasy Plus Mini Kit | Qiagen | 74134 | |
| Sorvall Legend Micro 21R Centrifuge, Refrigerated | ThermoFisher | 75002477 | |
| SYBR-Green iTaq Universal SYBR Green Supermix | Bio-Rad | 172-5120 | |
| Trypan Blue Solution | ThermoFisher | 15250061 | |
| V450 Rat Anti-Mouse CD45 | BD Biosciences | 560501 | |
| V450 Rat IgG2b, κ Isotype Control | BD Biosciences | 560457 |
참고문헌
- Slatko, B. E., Gardner, A. F., Ausubel, F. M. Overview of next-generation sequencing technologies. Current Protocols in Molecular Biology. 122 (1), 59 (2018).
- Chavkin, N. W., Hirschi, K. K. Single cell analysis in vascular biology. Frontiers in Cardiovascular Medicine. 7, 42 (2020).
- Ma, F., Hernandez, G. E., Romay, M., Iruela-Arispe, M. L. Single-cell RNA sequencing to study vascular diversity and function. Current Opinion in Hematology. 28 (3), 221-229 (2021).
- Potter, A. S., Potter, S. S., S, Dissociation of tissues for single-cell analysis. Methods in Molecular Biology. 1926, 55-62 (2019).
- Braga, F. A. V., Miragaia, R. J. Tissue handling and dissociation for single-cell RNA-seq. Single Cell Methods: Methods in Molecular Biology. 1979, 9-21 (2019).
- Brink, S. C., et al. Single-cell sequencing reveals dissociatin-induced gene expression in tissue subpopulations. Nature Methods. 14 (10), 935-936 (2017).
- Skulska, K., Wegrzyn, A. S., Chelmonska-Soyta, A., Chodaczek, G. Impact of tissue enzymatic digestion on analysis of immune cells in mouse reproductive mucosa with a focus on gammadelta T cells. Journal of Immunological Methods. 474, 112665 (2019).
- Connolly, S., Hores, T., Smith, L., D'Amore, P. Characterization of vascular development in the mouse retina. Microvascular Research. 36 (3), 275-290 (1988).
- Crist, A., Young, C., Meadows, S. Characterization of arteriovenous identity in the developing neonate mouse retina. Gene Expression Patterns: GEP. 23-24, 22-31 (2017).
- dela Paz, N. G., D'Amore, P. A. Arterial versus venous endothelial cells. Cell and Tissue Research. 335 (1), 5-16 (2009).
- Fang, J. S., Hirschi, K. K. Molecular regulation of arteriovenous endothelial cell specification. F1000Res. 8, (2019).
- Smith, L. E., et al. Oxygen-induced retinopathy in the mouse. Investigative Ophthalmology and Visual Science. 35 (1), 101-111 (1994).
- Pitulescu, M. E., Schmidt, I., Benedito, R., Adams, R. H. Inducible gene targeting in the neonatal vasculature and analysis of retinal angiogenesis in mice. Nature Protocols. 5 (9), 1518-1534 (2010).
- Ruiz, S., et al. A mouse model of hereditary hemorrhagic telangiectasia generated by transmammary-delivered immunoblocking of BMP9 and BMP10. Scientific Reports. 6, 37366 (2016).
- Fang, J. S., et al. Shear-induced Notch-Cx37-p27 axis arrests endothelial cell cycle to enable arterial specification. Nature Communication. 8 (1), 2149 (2017).
- Ola, R., et al. SMAD4 prevents flow induced arterial-venous malformations by inhibiting Casein Kinase 2. Circulation. 138 (21), 2379-2394 (2018).
- Chavkin, N. W., Walsh, K., Hirschi, K. K. Isolation of highly purified and viable retinal endothelial cells. Journal of Vascular Research. 58 (1), 49-57 (2020).
- Hulspas, R., O'Gorman, M. R. G., Wood, B. L., Gratama, J. W., Sutherland, D. R. Considerations for the control of background fluorescence in clinical flow cytometry. Cytometry PartB: Clinical Cytometry. 76 (6), 355-364 (2009).
- Bachman, J. Reverse-transcription PCR (RT-PCR). Methods in Enzymology. 530, 67-74 (2013).
- Green, M. R., Sambrook, J. Quantification of RNA by real-time Reverse Transcription-Polymerase Chain Reaction (RT-PCR). Cold Spring Harbour Protocols. 2018 (10), (2018).
- Liu, L., Shi, G. P. CD31: beyond a marker for endothelial cells. Cardiovascular Research. 94 (1), 3-5 (2012).
- Zarkada, G., et al. Specialized endothelial tip cells guide neuroretina vascularization and blood-retina-barrier formation. Developmental Cell. 56 (15), 2237-2251 (2021).
- Su, X., Sorenson, C. M., Sheibani, N. Isolation and characterization of murine retinal endothelial cells. Molecular Vision. 9, 171-178 (2003).
- Benedito, R., et al. The notch ligands Dll4 and Jagged1 have opposing effects on angiogenesis. Cell. 137 (6), 1124-1135 (2009).
- Daneman, R., et al. Wnt/beta-catenin signaling is required for CNS, but not non-CNS, angiogenesis. Proceedings of the National Academy of Sciences of the United States of America. 106 (2), 641-646 (2009).
- Okabe, K., et al. Neurons limit angiogenesis by titrating VEGF in retina. Cell. 159 (3), 584-596 (2014).
- Crist, A. M., Lee, A. R., Patel, N. R., Westhoff, D. E., Meadows, S. M. Vascular deficiency of Smad4 causes arteriovenous malformations: a mouse model of Hereditary Hemorrhagic Telangiectasia. Angiogenesis. 21 (2), 363-380 (2018).
- Kim, Y. H., Choe, S. W., Chae, M. Y., Hong, S., Oh, S. P. SMAD4 deficiency leads to development of arteriovenous malformations in neonatal and adult mice. Journal of the American Heart Association. 7 (21), 009514 (2018).
- Ma, W., et al. Absence of TGFbeta signaling in retinal microglia induces retinal degeneration and exacerbates choroidal neovascularization. eLife. 8, 42049 (2019).
- Luo, W., et al. Arterialization requires the timely suppression of cell growth. Nature. 589 (7842), 437-441 (2021).
- Lawson, N. D., Vogel, A. M., Weinstein, B. M. sonic hedgehog and vascular endothelial growth factor act upstream of the Notch pathway during arterial endothelial differentiation. Developmental Cell. 3 (1), 127-136 (2002).
- Larrivee, B., et al. ALK1 signaling inhibits angiogenesis by cooperating with the Notch pathway. Developmental Cell. 22 (3), 489-500 (2012).
- Wythe, J. D., et al. ETS factors regulate Vegf-dependent arterial specification. Developmental Cell. 26 (1), 45-58 (2013).
- Davis, D. M., Purvis, J. E. Computational analysis of signaling patterns in single cells. Seminars in Cell and Developmental Biology. , 35-43 (2015).
- Gaudet, S., Miller-Jensen, K. Redefining signaling pathways with an expanding single-cell toolbox. Trends in Biotechnology. 34 (6), 458-469 (2016).
- Aibar, S., et al. SCENIC: single-cell regulatory network inference and clustering. Nature Methods. 14 (11), 1083-1086 (2017).
- Trapnell, C., et al. The dynamics and regulators of cell fate decisions are revealed by pseudotemporal ordering of single cells. Nature Biotechnology. 32 (4), 381-386 (2014).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유