È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Isolamento di cellule endoteliali retiniche murine per il sequenziamento di nuova generazione
In questo articolo
Riepilogo
Questo protocollo descrive un metodo per l'isolamento di cellule endoteliali retiniche postnatali murine ottimizzate per la resa, la purezza e la vitalità delle cellule. Queste cellule sono adatte per approcci di sequenziamento di prossima generazione.
Abstract
I recenti miglioramenti nel sequenziamento di prossima generazione hanno fatto progredire la conoscenza dei ricercatori sulla biologia molecolare e cellulare, con diversi studi che rivelano nuovi paradigmi nella biologia vascolare. L'applicazione di questi metodi a modelli di sviluppo vascolare richiede l'ottimizzazione delle tecniche di isolamento cellulare da tessuti embrionali e postnatali. La resa cellulare, la vitalità e la purezza devono essere tutte massime per ottenere risultati accurati e riproducibili dagli approcci di sequenziamento di prossima generazione. Il modello di vascolarizzazione retinica neonatale del topo viene utilizzato dai ricercatori per studiare i meccanismi dello sviluppo vascolare. I ricercatori hanno utilizzato questo modello per studiare i meccanismi dell'angiogenesi e la specifica del destino arterioso-venoso durante la formazione e la maturazione dei vasi sanguigni. L'applicazione di tecniche di sequenziamento di nuova generazione per studiare il modello di sviluppo vascolare retinico richiede l'ottimizzazione di un metodo per l'isolamento delle cellule endoteliali retiniche che massimizzi la resa, la vitalità e la purezza delle cellule. Questo protocollo descrive un metodo per l'isolamento, la digestione e la purificazione del tessuto retinico murino utilizzando la selezione cellulare attivata dalla fluorescenza (FACS). I risultati indicano che la popolazione di cellule endoteliali CD31+/CD45- purificata da FACS è altamente arricchita per l'espressione genica delle cellule endoteliali e non mostra alcun cambiamento nella vitalità per 60 minuti post-FACS. Sono inclusi risultati rappresentativi degli approcci di sequenziamento di nuova generazione su cellule endoteliali isolate con questo metodo, tra cui il sequenziamento dell'RNA di massa e il sequenziamento dell'RNA a singola cellula, dimostrando che questo metodo per l'isolamento delle cellule endoteliali retiniche è compatibile con le applicazioni di sequenziamento di prossima generazione. Questo metodo di isolamento delle cellule endoteliali retiniche consentirà tecniche avanzate di sequenziamento per rivelare nuovi meccanismi di sviluppo vascolare.
Introduzione
L'elevata capacità di sequenziamento degli acidi nucleici tramite approcci di sequenziamento di nuova generazione ha notevolmente migliorato la conoscenza dei ricercatori sulla biologia molecolare e cellulare. Queste tecniche avanzate includono il sequenziamento dell'RNA dell'intero trascrittoma, il sequenziamento del DNA di regioni mirate per identificare i polimorfismi a singolo nucleotide (SNP), il sequenziamento del DNA dei fattori di trascrizione legati nel sequenziamento dell'immunoprecipitazione della cromatina (ChIP) o le regioni aperte della cromatina nel sequenziamento della cromatina accessibile alla trasposasi (ATAC) e il sequenziamento dell'RNA a singola cellula1 . Nella biologia vascolare, questi progressi hanno permesso ai ricercatori di chiarire complicati meccanismi di sviluppo e malattia, insieme a distinguere i modelli di espressione genica lungo un continuum di fenotipi variabili 2,3. Gli esperimenti futuri possono definire ulteriormente meccanismi complessi combinando il sequenziamento di prossima generazione con modelli valutati di sviluppo vascolare, ma i metodi per la preparazione del campione devono essere compatibili con le tecniche avanzate di sequenziamento.
La qualità, l'accuratezza e la riproducibilità degli approcci di sequenziamento di nuova generazione dipendono dal metodo di preparazione del campione. Quando si isola un sottogruppo di cellule o si generano sospensioni monocellulari dai tessuti, metodi ottimali di digestione e purificazione sono essenziali per massimizzare il numero di cellule, la vitalità e la purezza della popolazione cellulare 4,5. Ciò richiede un equilibrio nel metodo di digestione: una forte digestione è necessaria per liberare le cellule dal tessuto e ottenere abbastanza cellule per gli approcci a valle, ma la vitalità cellulare sarà influenzata negativamente se la digestione è troppo forte 6,7. Inoltre, la purezza della popolazione cellulare è necessaria per risultati solidi e un'analisi accurata dei dati, che può essere realizzata tramite FACS. Ciò evidenzia l'importanza di ottimizzare i metodi di isolamento cellulare per applicare il sequenziamento di prossima generazione a modelli consolidati di sviluppo vascolare.
Un modello ben caratterizzato per studiare lo sviluppo vascolare è il modello di sviluppo vascolare retinico murino. La vascolarizzazione retinica murina si sviluppa postnatale in un plesso superficiale bidimensionale, con germinazione angiogenica iniziale dal nervo ottico visibile al giorno postnatale (P)3, fronte angiogenico con cellule a stelo e punta e maturazione iniziale dei vasi visibili a P6 e maturazione del plesso vascolare visibile dopo P9 8,9. Durante il rimodellamento del plesso vascolare iniziale, le cellule endoteliali subiscono una specifica verso fenotipi arteriosi, capillari e venosi in diversi vasi per generare una rete circolatoria10,11. Pertanto, questo metodo consente ai ricercatori di visualizzare la formazione del plesso vascolare angiogenico e la specifica e la maturazione endoteliale arterioso-venosa in vari punti temporali durante lo sviluppo9. Inoltre, questo modello fornisce un metodo per studiare gli effetti della manipolazione transgenica sull'angiogenesi e sullo sviluppo del plesso vascolare, che è stato applicato per lo studio dello sviluppo vascolare, delle malformazioni arterio-venose e della neovascolarizzazione indotta dall'ossigeno 12,13,14,15,16 . Al fine di combinare approcci di sequenziamento di nuova generazione con il modello di sviluppo vascolare retinico murino, è necessario un protocollo ottimizzato per l'isolamento delle cellule endoteliali dal tessuto retinico.
Questo protocollo descrive un metodo ottimizzato per digerire il tessuto retinico dai topi a P6 per massimizzare la resa, la purezza e la vitalità delle cellule. Il tessuto retinico viene isolato dai topi P6, digerito per 20 minuti, immunocolorato per CD31 e CD45 e purificato attraverso FACS per isolare una sospensione a singola cellula di cellule endoteliali in circa 2,5 ore (Figura 1A). Si è scoperto che queste cellule endoteliali mantengono un'elevata vitalità per 60 minuti dopo l'isolamento17, consentendo la preparazione della libreria per i metodi di sequenziamento di prossima generazione. Inoltre, vengono forniti risultati rappresentativi per i risultati del gating FACS e del controllo di qualità da due diversi metodi di sequenziamento di nuova generazione che utilizzano questo protocollo di isolamento: sequenziamento dell'RNA dell'intero trascrittoma e sequenziamento dell'RNA a singola cellula. Questo metodo consente di utilizzare approcci di sequenziamento di nuova generazione in combinazione con il modello di vascolarizzazione retinica per chiarire nuovi meccanismi di sviluppo vascolare.
Access restricted. Please log in or start a trial to view this content.
Protocollo
I comitati istituzionali per la cura e l'uso degli animali dell'Università di Yale e dell'Università della Virginia hanno approvato tutti gli esperimenti sugli animali elencati in questo protocollo.
1. Ottenere occhi di topo per l'isolamento della retina
- Preparare 1x PBS ghiacciato e aggiungere 500 μL a ciascun pozzetto di una piastra da 48 pozzetti.
- Eutanasia dei topi neonatali al sesto giorno postnatale (P6) secondo le linee guida istituzionali approvate. Per questo esperimento, cucciolate di circa 4-8 topi neonatali vengono eutanasizzati a P6 tramite inalazione di isoflurano per almeno tre minuti dopo l'arresto respiratorio, seguito dalla decapitazione.
- Rimuovi gli occhi da ciascuno dei topi. Tagliare via la pelle e la membrana sopra l'occhio tagliando perpendicolarmente alla palpebra usando le forbici da dissezione. Quindi, utilizzare una pinza per premere delicatamente sopra e sotto l'occhio in modo che l'occhio si muova fuori dalla presa.
- Pizzicare delicatamente sotto l'occhio con la pinza e tagliare il nervo ottico che tiene l'occhio attaccato. Quindi, posizionare ciascun occhio nella piastra a 48 pozzetti nel PBS ghiacciato 1x preparato fino al termine della raccolta.
NOTA: utilizzare un pozzetto per mouse, con entrambi gli occhi di un singolo mouse nello stesso pozzetto.
- Pizzicare delicatamente sotto l'occhio con la pinza e tagliare il nervo ottico che tiene l'occhio attaccato. Quindi, posizionare ciascun occhio nella piastra a 48 pozzetti nel PBS ghiacciato 1x preparato fino al termine della raccolta.
2. Isolare il tessuto retinico del topo
- Riempire una capsula di Petri, foderata con un tampone di dissezione sul fondo, con 500 μL di 1x PBS ghiacciato per immergere gli occhi e metterlo sotto un microscopio a dissezione impostato su un ingrandimento 4.0x.
- Sospendere gli occhi nel tampone di dissezione utilizzando una pipetta di trasferimento con una punta larga in modo da non danneggiare gli occhi.
NOTA: Assicurarsi che gli occhi siano completamente coperti di PBS.
- Sospendere gli occhi nel tampone di dissezione utilizzando una pipetta di trasferimento con una punta larga in modo da non danneggiare gli occhi.
- Usando due pinze da dissezione fine, tenere il nervo ottico con una delle pinze per la stabilizzazione e con la seconda pinza perforare un foro attraverso la camera anteriore dove la cornea e la sclera si collegano (Figura 1B). Strappare il buco in un cerchio circa il 75% del percorso intorno alla cornea.
- Mentre si tiene ancora il nervo ottico con una delle pinze, utilizzare la seconda pinza per strappare delicatamente la sclera dal tessuto retinico.
- Come sopra, utilizzare la seconda pinza per strappare delicatamente il corpo vitreo del tessuto retinico.
- Ancora una volta, usando la seconda pinza, rimuovere delicatamente la lente e i vasi del plesso ialoide che non si sono staccati quando il corpo vitreo è stato rimosso. Per fare questo, raggiungere la retina con una pinza aperta fino quasi a toccare la retina, chiudere la pinza per afferrare la lente e i vasi del plesso ialoide e estrarre il forcipe dalla retina. Questo può essere ripetuto fino a quando tutti i vasi non vengono rimossi.
NOTA: Il plesso ialoide assomiglia a una struttura chiara simile a una ragnatela.
- Riempire 2 mL di provette da microcentrifuga con 500 μL di 1x PBS ghiacciato e posizionare le retine nelle provette. Isolare tutte le retine dagli occhi e metterle in PBS ghiacciato prima di andare avanti.
NOTA: Utilizzare un tubo per mouse, con due retine dello stesso mouse che condividono un tubo. Se il tessuto retinico è strappato, trasferirlo in più pezzi. Il tempo totale di dissezione del tessuto retinico per una cucciolata di topi dovrebbe richiedere 15-60 minuti, iniziando con l'eutanasia e terminando con l'isolamento finale del tessuto retinico.
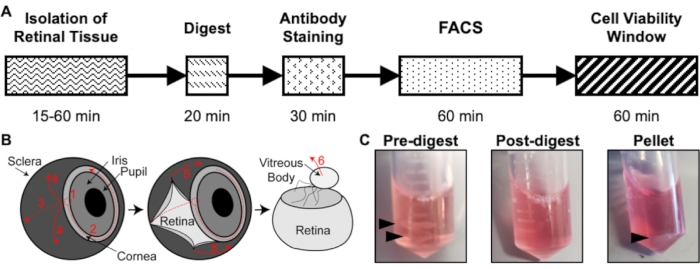
Figura 1: Panoramica del protocollo di isolamento. (A) Schema della sequenza temporale dell'isolamento con un tempo stimato per ogni fase: isolamento del tessuto retinico, digest, colorazione degli anticorpi, FACS e finestra di vitalità cellulare. (B) Guida passo-passo per l'isolamento del tessuto retinico dall'occhio, con fasi di dissezione numerate: 1) perforare la cornea, 2) lacrimare la cornea, 3) strappare la sclera, 4) rimuovere la sclera dalla retina, 5) rimuovere ulteriormente la sclera e il tessuto di collegamento dalla retina, 6) rimuovere il corpo vitreo e i vasi vitreali dalla retina. (C) Immagini rappresentative del tessuto retinico durante le varie fasi della digestione: Pre-digestione, Post-digestione, Pellet (le frecce nere evidenziano il tessuto retinico o il pellet cellulare). Ripubblicato con il permesso di Chavkin et al. pubblicato in S. Karger AG, Basilea17. Fare clic qui per visualizzare una versione ingrandita di questa figura.
3. Digerire il tessuto retinico in una sospensione monocellulare
- Preparare 500 μL di soluzione digestiva per ogni due retine isolate. Aggiungere FBS e collagenasi di tipo II a DMEM ad una concentrazione finale del 10% FBS e 1 mg/mL di collagenasi di tipo II. Mescolare e riscaldare la soluzione a 37 °C a bagnomaria.
- Rimuovere il PBS in eccesso dalle provette della microcentrifuga da 2 mL mediante pipettaggio accurato. Lasciare abbastanza PBS per coprire completamente il tessuto retinico, circa 100 μL in ogni tubo.
- Aggiungere 500 μL della soluzione digestiva a ciascuna provetta con tessuto retinico (Figura 1C).
- Utilizzare un pipettor P1000 e una punta per pipetta su e giù per il tessuto retinico nella soluzione di digestione cinque volte.
- Incubare la miscela di digestione a bagnomaria a 37 °C per 20 minuti.
- Utilizzare un pipettor P1000 e una punta per pipetta per pipettare la miscela di digestione su e giù ogni 5 minuti. Dopo l'incubazione, il tessuto retinico si è dissolto in una sospensione a singola cellula, quindi la miscela di digestione dovrebbe essere torbida (Figura 1C).
4. Contare le celle
- Impostare una centrifuga da tavolo a 4 °C e posizionare all'interno i tubi della miscela di digestione. Pellettare la miscela di digestione mediante centrifugazione a 375 x g per 5 min.
- Rimuovere con cautela la soluzione di digestione surnatante mediante pipettaggio. Non disturbare il pellet cellulare (Figura 1C). Risospendere il pellet in 500 μL di 1x PBS ghiacciato mescolando delicatamente su e giù con la pipetta.
- Contare le cellule con un emocitometro al microscopio.
NOTA: La conta delle cellule è di circa 1 x 106 cellule per due retine. - Assicurarsi che le cellule siano correttamente risospese mediante miscelazione delicata, quindi aliquote 20 μL di sospensione cellulare in tre provette per la colorazione di controllo (tubo 1: controllo IgG, tubo 2: controllo CD31 e tubo 3: controllo CD45), come descritto e usato in precedenza17,18.
- Impostare una centrifuga da tavolo a 4 °C e posizionare all'interno i tubi contenenti celle. Pellettare le celle mediante centrifugazione a 375 x g per 5 min.
5. Immunocolorare le cellule con anticorpi
- Preparare 100 μL di tampone colorante per due retine colorate più quattro tubi di controllo. Aggiungere FBS, HEPES e D-glucosio al tampone HBSS a una concentrazione finale del 10% FBS, 10 mM HEPES e 1 mg / mL D-glucosio.
- Ottenere anticorpi coniugati fluorescentmente contro CD31 e CD45. Aggiungere gli anticorpi ad una diluizione 1:100 nel tampone colorante (tubo 1: solo anticorpo IgG, tubo 2: solo anticorpo CD31 e tubo 3: solo anticorpo CD45). Aggiungere 1 μL di anticorpo per 100 μL del tampone colorante per una concentrazione finale di anticorpi di 2 μg/ml.
- Rimuovere con cautela il PBS dal pellet cellulare lavato mediante pipettaggio.
- Risospendere il pellet in 100 μL di soluzione anticorpale per 0,5 x 106 cellule.
- Incubare la sospensione unicellulare con la soluzione anticorpale per 30 minuti su ghiaccio al buio. Picchiettare i tubi ogni 10 minuti per mescolare delicatamente le cellule.
6. Prepararsi per la selezione cellulare attivata dalla fluorescenza
- Impostare una centrifuga da tavolo a 4 °C e posizionare all'interno i tubi contenenti celle. Celle a pellet per centrifugazione a 375 x g per 5 min.
- Rimuovere il surnatante e risospendere queste cellule in 500 μL di 1x PBS da lavare.
- Impostare una centrifuga da tavolo a 4 °C e posizionare all'interno i tubi contenenti celle. Celle a pellet per centrifugazione a 375 x g per 5 min
- Preparare 1,5 mL di tampone FACS (1% FBS in 1x PBS).
- Rimuovere il surnatante e risospendere il pellet cellulare in 300 μL di tampone FACS mescolando delicatamente con un pipettatore.
- Aggiungere lo ioduro di propidio (PI) alle provette contenenti il tampone FACS fino ad una concentrazione finale di 0,5 μg/ml. PI è usato come indicatore di vitalità.
- Trasferire e combinare le sospensioni cellulari in provette da 5 mL attraverso un tappo a scatto del filtro cellulare. Il tappo a scatto del filtro cellulare deve contenere un filtro da 35 μm e le sospensioni cellulari possono essere pipettate direttamente sul filtro.
- Tenere i tubi FACS sul ghiaccio al buio e trasferirli nella selezionatrice cellulare.
7. Impostare lo strumento FACS
- Installare un ugello da 100 μm in uno strumento FACS.
NOTA: questo ugello di dimensioni riduce al minimo il volume di raccolta e massimizza la densità delle celle isolate. - Preparare 250 μL di 1x PBS in una provetta da microcentrifuga da 1,5 mL come tubo di raccolta da installare nello strumento FACS.
8. Isolare le cellule endoteliali vitali tramite FACS
- Accendere lo strumento FACS e il computer. Fare clic sull'icona del software FACS per aprire il software FACS sul computer per eseguire e utilizzare lo strumento FACS. Eseguire test di controllo qualità di routine.
- Caricare i campioni colorati di controllo nello strumento FACS per regolare gli assi. Inizia con le sole cellule anticorpali IgG per generare e regolare FSC e SSC, quindi l'anticorpo CD31 solo per generare e regolare CD31 e quindi l'anticorpo CD45 solo per generare e regolare CD45. Registrare i dati dai campioni di controllo.
- Caricare le celle del campione colorato nello strumento FACS ed eseguire il campione.
- Gate le celle in base ai parametri forward-scatter e side-scatter (FSC-A e SSC-A, rispettivamente) (Figura 2A).
NOTA: i parametri FSC-A e SSC-A vengono utilizzati per selezionare le celle in base a dimensioni, densità, granularità, proprietà superficiali e indice di rifrazione. - Celle gate di FSC-A e FSC-H per identificare i doppietti cellulari e raccogliere solo singole celle (Figura 2A).
NOTA: I parametri FSC-A e FSC-H vengono utilizzati per selezionare singole celle in base al principio che durante la diffusione in avanti, i doppietti delle celle saranno goccioline contenenti un rapporto area-altezza maggiore. - Celle gate per PI e SSC-A per identificare le cellule vitali (Figura 2A).
NOTA: Le cellule vitali saranno PI-negative. - Crea un gate CD31+/CD45- con controlli e celle di gate di CD31 e CD45 (Figura 2A,B).
- Inserire una provetta di raccolta con 250 μL di tampone FACS.
- Iniziare a smistare le celle CD31-positive/CD45-negative nella provetta di raccolta installata.
- Tenere i tubi di raccolta sul ghiaccio per ulteriori analisi. I campioni possono essere elaborati per applicazioni di sequenziamento di nuova generazione in questa fase1.
9. Eseguire il test di vitalità
- Mescolare 10 μL di cellule con 10 μL di soluzione di tripano blu allo 0,4% per valutare la vitalità cellulare.
- Contare le cellule vitali pipettando la miscela colorata su un vetrino emocitometrico. Posizionare il vetrino al microscopio per conteggi accurati della vitalità. Conta le cellule blu come non vitali e conta le cellule chiare come vitali.
- Dividere il conteggio delle cellule vitali per il conteggio totale delle cellule per calcolare la percentuale di vitalità.
10. Eseguire il test di espressione genica
- Ottenere 10.000-20.000 cellule per campione ed eseguire l'isolamento dell'RNA per analizzare l'espressione genica delle cellule endoteliali retiniche murine. L'isolamento dell'RNA viene eseguito utilizzando un kit disponibile in commercio che utilizza la precipitazione dell'RNA a base di etanolo, quindi la centrifugazione e le colonne di membrana per lavare ed eluire l'RNA purificato dal lisato cellulare. I campioni possono essere elaborati per alcune applicazioni di sequenziamento di nuova generazione in questa fase1.
- Convertire l'RNA in cDNA usando l'enzima trascrittasi inversa e i reagenti di supporto19.
- Quantificare utilizzando un colorante legante il DNA e una macchina per PCR quantitativa (qPCR)20. Caricare campioni di cDNA in una piastra qPCR da 384 pozzetti con primer per amplificare i geni di interesse. Il volume di reazione dovrebbe ammontare a 10 μL, compreso il colorante legante il DNA, che verrà utilizzato per quantificare l'amplificazione genica.
- Utilizzare CD31, VE-caderina, CD45 e primer β-actina avanti e indietro per misurare l'espressione genica.
- Utilizzare l'esempio per le applicazioni di sequenziamento di nuova generazione1.
Access restricted. Please log in or start a trial to view this content.
Risultati
La digestione del tessuto retinico e l'immunocolorazione per CD31 e CD45 si traducono in una popolazione identificabile di cellule endoteliali CD31+/CD45- dopo il gating per cellule, singole cellule e vitalità (Figura 2A). L'immunocolorazione CD45 è necessaria per eliminare le cellule CD31+/CD45+, che includono piastrine e alcuni leucociti21. I controlli devono essere eseguiti per ogni esperimento per mostrare la specificità degli anticorpi e guidare la strategia d...
Access restricted. Please log in or start a trial to view this content.
Discussione
Questo protocollo descrive un metodo per l'isolamento delle cellule endoteliali dal tessuto retinico murino postnatale che è stato ottimizzato per un elevato numero di cellule, purezza e vitalità. La purezza cellulare è ottenuta mediante isolamento FACS di popolazioni di cellule endoteliali dalla sospensione monocellulare digerita mediante immunocolorazione CD31+/CD45-. La qualità dell'isolamento è quantificata nei saggi per la vitalità mediante colorazione blu di Trypan ed espressione genica mediante qPCR per CD31...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno divulgazioni rilevanti.
Riconoscimenti
Grazie alla Yale Flow Cytometry Facility, alla University of Virginia Flow Cytometry Core Facility, allo Yale Center for Genomic Analysis e alla University of Virginia Genome Analysis and Technology Core per il loro impegno, competenza e consulenza nel contribuire agli esperimenti presentati. Questo studio è stato finanziato da sovvenzioni NIH a N.W.C. (T32 HL007224, T32 HL007284), S.C. (T32 HL007284), K.W. (R01 HL142650) e K.K.H. (R01 HL146056, R01 DK118728, UH3 EB025765).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 2 mL Eppendorf safe-lock tubes | USA Scientific | 4036-3352 | |
| 5 ml Falcon Test Tubes with Cell Strainer Snap Cap | Corning | 352235 | |
| 60 mm Non TC-treated Culture Dish | Corning | 430589 | |
| APC Rat Anti-Mouse CD31 | BD Biosciences | 551262 | |
| APC Rat IgG2a κ Isotype Control | BD Biosciences | 553932 | |
| BD FACSChorus Software | BD Biosciences | FACSCHORUS | |
| BD FACSMelody Cell Sorter | BD Biosciences | FACSMELODY | |
| Collagenase Type II | Sigma-Aldrich | 234115 | |
| Costar 48-well Clear TC-treated Multiple Well Plates, Individually Wrapped, Sterile | Corning | 3548 | |
| D-Glucose | Gibco | A2494001 | |
| Disposable Graduated Transfer Pipettes | Fisher Scientific | 12-711-9AM | |
| Dissecting Pan Wax | Carolina | 629100 | |
| Dissection scissors | Fine Science Tools | 14085-08 | |
| Dissection Stereo Microscope M165 FC | Leica | M165FC | |
| Dulbecco's Modified Eagle Medium (DMEM) | Gibco | 11965-052 | |
| Dulbecco’s Phosphate Buffered Saline (PBS) | Gibco | 14190144 | |
| Eppendorf Flex-Tubes Microcentrifuge Tubes 1.5 mL | Sigma-Aldrich | 22364120 | |
| Fetal Bovine Serum (FBS) | Gemini Bio | 100-106 | |
| Fine dissection forceps | Fine Science Tools | 11250-00 | |
| Hank's Buffered Salt Solution (HBSS) | Gibco | 14175095 | |
| HEPES (1M) | Gibco | 15630130 | |
| iScript cDNA Synthesis Kit | Bio-Rad | 1708890 | |
| Isoflurane, USP | Covetrus | 11695067772 | |
| Isotemp General Purpose Deluxe Water Bath | Fisher Scientific | FSGPD20 | |
| Primer: ActB_Forward: 5’- agagggaaatcgtgcgtgac -3’ | Integrated DNA Technologies | N/A | |
| Primer: ActB_Reverse: 5’- caatagtgatgacctggccgt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD31_Forward: 5’- gagcccaatcacgtttcagttt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD31_Reverse: 5’- tccttcctgcttcttgctagct -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD45_Forward: 5’- gggttgttctgtgccttgtt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD45_Reverse: 5’- ctggacggacacagttagca -3’ | Integrated DNA Technologies | N/A | |
| Primer: VE-Cadherin_Forward: 5’- tcctctgcatcctcactatcaca -3’ | Integrated DNA Technologies | N/A | |
| Primer: VE-Cadherin_Reverse: 5’- gtaagtgaccaactgctcgtgaat -3’ | Integrated DNA Technologies | N/A | |
| Propidium iodide | Sigma-Aldrich | P4864 | |
| RNeasy Plus Mini Kit | Qiagen | 74134 | |
| Sorvall Legend Micro 21R Centrifuge, Refrigerated | ThermoFisher | 75002477 | |
| SYBR-Green iTaq Universal SYBR Green Supermix | Bio-Rad | 172-5120 | |
| Trypan Blue Solution | ThermoFisher | 15250061 | |
| V450 Rat Anti-Mouse CD45 | BD Biosciences | 560501 | |
| V450 Rat IgG2b, κ Isotype Control | BD Biosciences | 560457 |
Riferimenti
- Slatko, B. E., Gardner, A. F., Ausubel, F. M. Overview of next-generation sequencing technologies. Current Protocols in Molecular Biology. 122 (1), 59(2018).
- Chavkin, N. W., Hirschi, K. K. Single cell analysis in vascular biology. Frontiers in Cardiovascular Medicine. 7, 42(2020).
- Ma, F., Hernandez, G. E., Romay, M., Iruela-Arispe, M. L. Single-cell RNA sequencing to study vascular diversity and function. Current Opinion in Hematology. 28 (3), 221-229 (2021).
- Potter, A. S., Potter, S. S., S, Dissociation of tissues for single-cell analysis. Methods in Molecular Biology. 1926, 55-62 (2019).
- Braga, F. A. V., Miragaia, R. J. Tissue handling and dissociation for single-cell RNA-seq. Single Cell Methods: Methods in Molecular Biology. 1979, 9-21 (2019).
- Brink, S. C., et al. Single-cell sequencing reveals dissociatin-induced gene expression in tissue subpopulations. Nature Methods. 14 (10), 935-936 (2017).
- Skulska, K., Wegrzyn, A. S., Chelmonska-Soyta, A., Chodaczek, G. Impact of tissue enzymatic digestion on analysis of immune cells in mouse reproductive mucosa with a focus on gammadelta T cells. Journal of Immunological Methods. 474, 112665(2019).
- Connolly, S., Hores, T., Smith, L., D'Amore, P. Characterization of vascular development in the mouse retina. Microvascular Research. 36 (3), 275-290 (1988).
- Crist, A., Young, C., Meadows, S. Characterization of arteriovenous identity in the developing neonate mouse retina. Gene Expression Patterns: GEP. 23-24, 22-31 (2017).
- dela Paz, N. G., D'Amore, P. A. Arterial versus venous endothelial cells. Cell and Tissue Research. 335 (1), 5-16 (2009).
- Fang, J. S., Hirschi, K. K. Molecular regulation of arteriovenous endothelial cell specification. F1000Res. 8, (2019).
- Smith, L. E., et al. Oxygen-induced retinopathy in the mouse. Investigative Ophthalmology and Visual Science. 35 (1), 101-111 (1994).
- Pitulescu, M. E., Schmidt, I., Benedito, R., Adams, R. H. Inducible gene targeting in the neonatal vasculature and analysis of retinal angiogenesis in mice. Nature Protocols. 5 (9), 1518-1534 (2010).
- Ruiz, S., et al. A mouse model of hereditary hemorrhagic telangiectasia generated by transmammary-delivered immunoblocking of BMP9 and BMP10. Scientific Reports. 6, 37366(2016).
- Fang, J. S., et al. Shear-induced Notch-Cx37-p27 axis arrests endothelial cell cycle to enable arterial specification. Nature Communication. 8 (1), 2149(2017).
- Ola, R., et al. SMAD4 prevents flow induced arterial-venous malformations by inhibiting Casein Kinase 2. Circulation. 138 (21), 2379-2394 (2018).
- Chavkin, N. W., Walsh, K., Hirschi, K. K. Isolation of highly purified and viable retinal endothelial cells. Journal of Vascular Research. 58 (1), 49-57 (2020).
- Hulspas, R., O'Gorman, M. R. G., Wood, B. L., Gratama, J. W., Sutherland, D. R. Considerations for the control of background fluorescence in clinical flow cytometry. Cytometry PartB: Clinical Cytometry. 76 (6), 355-364 (2009).
- Bachman, J. Reverse-transcription PCR (RT-PCR). Methods in Enzymology. 530, 67-74 (2013).
- Green, M. R., Sambrook, J. Quantification of RNA by real-time Reverse Transcription-Polymerase Chain Reaction (RT-PCR). Cold Spring Harbour Protocols. 2018 (10), (2018).
- Liu, L., Shi, G. P. CD31: beyond a marker for endothelial cells. Cardiovascular Research. 94 (1), 3-5 (2012).
- Zarkada, G., et al. Specialized endothelial tip cells guide neuroretina vascularization and blood-retina-barrier formation. Developmental Cell. 56 (15), 2237-2251 (2021).
- Su, X., Sorenson, C. M., Sheibani, N. Isolation and characterization of murine retinal endothelial cells. Molecular Vision. 9, 171-178 (2003).
- Benedito, R., et al. The notch ligands Dll4 and Jagged1 have opposing effects on angiogenesis. Cell. 137 (6), 1124-1135 (2009).
- Daneman, R., et al. Wnt/beta-catenin signaling is required for CNS, but not non-CNS, angiogenesis. Proceedings of the National Academy of Sciences of the United States of America. 106 (2), 641-646 (2009).
- Okabe, K., et al. Neurons limit angiogenesis by titrating VEGF in retina. Cell. 159 (3), 584-596 (2014).
- Crist, A. M., Lee, A. R., Patel, N. R., Westhoff, D. E., Meadows, S. M. Vascular deficiency of Smad4 causes arteriovenous malformations: a mouse model of Hereditary Hemorrhagic Telangiectasia. Angiogenesis. 21 (2), 363-380 (2018).
- Kim, Y. H., Choe, S. W., Chae, M. Y., Hong, S., Oh, S. P. SMAD4 deficiency leads to development of arteriovenous malformations in neonatal and adult mice. Journal of the American Heart Association. 7 (21), 009514(2018).
- Ma, W., et al. Absence of TGFbeta signaling in retinal microglia induces retinal degeneration and exacerbates choroidal neovascularization. eLife. 8, 42049(2019).
- Luo, W., et al. Arterialization requires the timely suppression of cell growth. Nature. 589 (7842), 437-441 (2021).
- Lawson, N. D., Vogel, A. M., Weinstein, B. M. sonic hedgehog and vascular endothelial growth factor act upstream of the Notch pathway during arterial endothelial differentiation. Developmental Cell. 3 (1), 127-136 (2002).
- Larrivee, B., et al. ALK1 signaling inhibits angiogenesis by cooperating with the Notch pathway. Developmental Cell. 22 (3), 489-500 (2012).
- Wythe, J. D., et al. ETS factors regulate Vegf-dependent arterial specification. Developmental Cell. 26 (1), 45-58 (2013).
- Davis, D. M., Purvis, J. E. Computational analysis of signaling patterns in single cells. Seminars in Cell and Developmental Biology. 0, 35-43 (2015).
- Gaudet, S., Miller-Jensen, K. Redefining signaling pathways with an expanding single-cell toolbox. Trends in Biotechnology. 34 (6), 458-469 (2016).
- Aibar, S., et al. SCENIC: single-cell regulatory network inference and clustering. Nature Methods. 14 (11), 1083-1086 (2017).
- Trapnell, C., et al. The dynamics and regulators of cell fate decisions are revealed by pseudotemporal ordering of single cells. Nature Biotechnology. 32 (4), 381-386 (2014).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon