É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Isolamento de células endoteliais da retina murinas para sequenciamento de próxima geração
Neste Artigo
Resumo
Este protocolo descreve um método para o isolamento de células endoteliais da retina pós-natais murinas otimizado para o rendimento, pureza e viabilidade celular. Essas células são adequadas para abordagens de sequenciamento de próxima geração.
Resumo
Melhorias recentes no sequenciamento de próxima geração avançaram o conhecimento dos pesquisadores sobre biologia molecular e celular, com vários estudos revelando novos paradigmas em biologia vascular. A aplicação desses métodos a modelos de desenvolvimento vascular requer a otimização de técnicas de isolamento celular de tecidos embrionários e pós-natais. O rendimento, a viabilidade e a pureza das células precisam ser máximos para obter resultados precisos e reprodutíveis das abordagens de sequenciamento de próxima geração. O modelo de vascularização da retina de camundongos neonatais é usado por pesquisadores para estudar mecanismos de desenvolvimento vascular. Os pesquisadores usaram esse modelo para investigar mecanismos de angiogênese e especificação do destino arterial-venoso durante a formação e maturação dos vasos sanguíneos. A aplicação de técnicas de sequenciamento de próxima geração para estudar o modelo de desenvolvimento vascular da retina requer a otimização de um método para o isolamento de células endoteliais da retina que maximize o rendimento, a viabilidade e a pureza celular. Este protocolo descreve um método para isolamento, digestão e purificação do tecido retiniano murino usando a classificação celular ativada por fluorescência (FACS). Os resultados indicam que a população de células endoteliais CD31+/CD45- purificadas por CACS é altamente enriquecida para a expressão gênica de células endoteliais e não apresenta alteração na viabilidade por 60 min pós-CASC. Estão incluídos resultados representativos de abordagens de sequenciamento de próxima geração em células endoteliais isoladas usando este método, incluindo sequenciamento de RNA em massa e sequenciamento de RNA de célula única, demonstrando que este método para isolamento de células endoteliais da retina é compatível com aplicações de sequenciamento de próxima geração. Este método de isolamento das células endoteliais da retina permitirá técnicas avançadas de sequenciamento para revelar novos mecanismos de desenvolvimento vascular.
Introdução
A capacidade de alto rendimento do sequenciamento de ácidos nucleicos por meio de abordagens de sequenciamento de próxima geração avançou muito o conhecimento dos pesquisadores sobre biologia molecular e celular. Essas técnicas avançadas incluem sequenciamento de RNA de transcriptoma inteiro, sequenciamento de DNA de regiões-alvo para identificar polimorfismos de nucleotídeo único (SNPs), sequenciamento de DNA de fatores de transcrição ligados no sequenciamento de imunoprecipitação de cromatina (ChIP) ou regiões de cromatina abertas em ensaio para sequenciamento de cromatina acessível à transposase (ATAC) e sequenciamento de RNA de célula única1 . Na biologia vascular, esses avanços permitiram aos pesquisadores elucidar mecanismos complicados de desenvolvimento e doença, além de distinguir padrões de expressão gênica ao longo de um continuum de fenótipos variados 2,3. Experimentos futuros podem definir ainda mais mecanismos complexos, combinando o sequenciamento de próxima geração com modelos avaliados de desenvolvimento vascular, mas os métodos de preparação de amostras precisam ser compatíveis com as técnicas avançadas de sequenciamento.
A qualidade, precisão e reprodutibilidade das abordagens de sequenciamento de próxima geração dependem do método de preparação da amostra. Ao isolar um subconjunto de células ou gerar suspensões unicelulares a partir de tecidos, métodos ideais de digestão e purificação são essenciais para maximizar o número celular, a viabilidade e a pureza da população celular 4,5. Isso requer um equilíbrio no método de digestão: uma digestão forte é necessária para liberar células do tecido e obter células suficientes para abordagens a jusante, mas a viabilidade celular será afetada negativamente se a digestão for muito forte 6,7. Além disso, a pureza da população celular é necessária para resultados robustos e análise precisa dos dados, o que pode ser realizado através do FACS. Isso destaca a importância de otimizar os métodos de isolamento celular para aplicar o sequenciamento de próxima geração a modelos estabelecidos de desenvolvimento vascular.
Um modelo bem caracterizado para investigar o desenvolvimento vascular é o modelo de desenvolvimento vascular da retina murina. A vasculatura retiniana murina desenvolve-se no pós-natal em um plexo superficial bidimensional, com brotação angiogênica inicial do nervo óptico visível no dia pós-natal (P)3, frente angiogênica com células do caule e da ponta e maturação inicial dos vasos visível em P6, e maturação do plexo vascular visível após P9 8,9. Durante o remodelamento do plexo vascular inicial, as células endoteliais passam por especificação para fenótipos arteriais, capilares e venosos em diferentes vasos para gerar uma rede circulatória10,11. Portanto, esse método permite aos pesquisadores visualizar a formação do plexo vascular angiogênico e a especificação e maturação arterial-venosa endotelial em vários momentos durante o desenvolvimento9. Além disso, esse modelo fornece um método para investigar os efeitos da manipulação transgênica sobre a angiogênese e o desenvolvimento do plexo vascular, que tem sido aplicado para a investigação do desenvolvimento vascular, malformações arteriais-venosas e neovascularização induzida por oxigênio 12,13,14,15,16 . A fim de combinar abordagens de sequenciamento de próxima geração com o modelo de desenvolvimento vascular da retina murina, é necessário um protocolo otimizado para o isolamento de células endoteliais do tecido da retina.
Este protocolo descreve um método otimizado para digerir o tecido da retina de camundongos em P6 para maximizar o rendimento celular, a pureza e a viabilidade. O tecido da retina é isolado de camundongos P6, digerido por 20 min, imunocorado para CD31 e CD45 e purificado através de FACS para isolar uma única suspensão celular de células endoteliais em cerca de 2,5 h (Figura 1A). Verificou-se que essas células endoteliais mantêm alta viabilidade por 60 minutos após o isolamento17, permitindo preparações de biblioteca para métodos de sequenciamento de próxima geração. Além disso, resultados representativos são fornecidos para o controle de qualidade e os resultados do controle de qualidade do FACS de dois métodos separados de sequenciamento de próxima geração usando esse protocolo de isolamento: sequenciamento de RNA de transcriptoma inteiro e sequenciamento de RNA de célula única. Este método permite que abordagens de sequenciamento de próxima geração sejam usadas em conjunto com o modelo de vascularização da retina para elucidar novos mecanismos de desenvolvimento vascular.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Os Comitês Institucionais de Cuidado e Uso de Animais da Universidade de Yale e da Universidade da Virgínia aprovaram todos os experimentos em animais listados neste protocolo.
1. Obter olhos de rato para isolamento da retina
- Prepare 1x PBS gelado e adicione 500 μL a cada poço de uma placa de 48 poços.
- Eutanasiar camundongos neonatais no sexto dia pós-natal (P6) de acordo com as diretrizes institucionais aprovadas. Para este experimento, ninhadas de aproximadamente 4-8 camundongos neonatais são eutanasiadas em P6 via inalação de isoflurano por pelo menos três minutos após a parada respiratória, seguida de decapitação.
- Remova os olhos de cada um dos ratos. Corte a pele e a membrana sobre o olho, cortando perpendicularmente à pálpebra usando uma tesoura de dissecação. Em seguida, use fórceps para pressionar suavemente acima e abaixo do olho para que o olho se mova para fora da órbita.
- Aperte cuidadosamente sob o olho com a pinça e corte o nervo óptico que está mantendo o olho ligado. Em seguida, coloque cada olho na placa de 48 poços no PBS 1x gelado preparado até terminar a colheita.
NOTA: Use um poço por mouse, com os dois olhos de um único mouse no mesmo poço.
- Aperte cuidadosamente sob o olho com a pinça e corte o nervo óptico que está mantendo o olho ligado. Em seguida, coloque cada olho na placa de 48 poços no PBS 1x gelado preparado até terminar a colheita.
2. Isole o tecido da retina do rato
- Encha uma placa de Petri, forrada com uma almofada de dissecação na parte inferior, com 500 μL de 1x PBS gelado para submergir os olhos e colocá-lo sob um microscópio de dissecação ajustado para ampliação de 4,0x.
- Suspenda os olhos na almofada de dissecação usando uma pipeta de transferência com uma ponta larga para não danificar os olhos.
NOTA: Certifique-se de que os olhos estão completamente cobertos com PBS.
- Suspenda os olhos na almofada de dissecação usando uma pipeta de transferência com uma ponta larga para não danificar os olhos.
- Usando duas pinças de dissecção finas, segure o nervo óptico com uma das pinças para estabilização e, com a segunda pinça, perfure um orifício através da câmara anterior onde a córnea e a esclera se conectam (Figura 1B). Rasgue o buraco em um círculo de cerca de 75% do caminho ao redor da córnea.
- Enquanto ainda segura o nervo óptico com uma das pinças, use a segunda pinça para rasgar suavemente a esclera do tecido da retina.
- Como acima, use a segunda pinça para arrancar suavemente o corpo vítreo do tecido da retina.
- Novamente, usando a segunda pinça, remova suavemente a lente e os vasos do plexo hialoide que não saíram quando o corpo vítreo foi removido. Para fazer isso, alcance a retina com pinça aberta até quase tocar a retina, feche a pinça para agarrar a lente e os vasos do plexo hialoide e puxe a pinça para fora da retina. Isso pode ser repetido até que todos os vasos sejam removidos.
NOTA: O plexo hialoide assemelha-se a uma estrutura clara, semelhante a uma teia.
- Encha 2 mL de tubos de microcentrífuga com 500 μL de 1x PBS gelado e coloque as retinas nos tubos. Isole todas as retinas dos olhos e coloque-as em PBS gelado antes de seguir em frente.
NOTA: Use um tubo por mouse, com duas retinas do mesmo mouse compartilhando um tubo. Se o tecido da retina estiver rasgado, transfira-o em vários pedaços. O tempo total de dissecção do tecido da retina para uma ninhada de camundongos deve levar de 15 a 60 minutos, começando com a eutanásia e terminando com o isolamento final do tecido da retina.
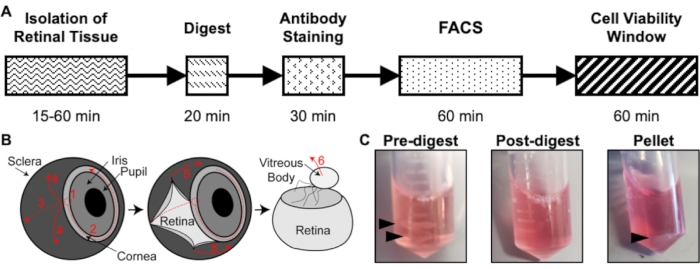
Figura 1: Visão geral do protocolo de isolamento. (A) Esquema da linha do tempo de isolamento com um tempo estimado para cada etapa: Isolamento do Tecido da Retina, Digest, Coloração de Anticorpos, FACS e Janela de Viabilidade Celular. (B) Guia passo-a-passo para isolamento do tecido da retina do olho, com etapas de dissecção numeradas: 1) perfurar a córnea, 2) lágrima da córnea, 3) esclera lacrimal, 4) remover a esclera da retina, 5) remover ainda mais a esclera e o tecido de conexão da retina, 6) remover o corpo vítreo e os vasos vítreos da retina. (C) Imagens representativas do tecido da retina durante várias etapas da digestão: Pré-digestão, Pós-digestão, Pellet (setas pretas destacam o tecido da retina ou pellet celular). Republicado com permissão de Chavkin et al. publicado em S. Karger AG, Basel17. Por favor, clique aqui para ver uma versão maior desta figura.
3. Digerir o tecido da retina em uma única suspensão celular
- Preparar uma solução de digestão de 500 μL para cada duas retinas isoladas. Adicionar FBS e colagenase tipo II ao DMEM a uma concentração final de 10% de FBS e 1 mg/mL de colagenase tipo II. Misture e aqueça a solução a 37 °C com banho-maria.
- Remova o excesso de PBS dos tubos de microcentrífuga de 2 mL por pipetagem cuidadosa. Deixe PBS suficiente para cobrir completamente o tecido da retina, cerca de 100 μL em cada tubo.
- Adicionar 500 μL da solução de digestão a cada tubo com tecido retiniano (Figura 1C).
- Use um pipetador P1000 e uma ponta de pipeta para pipetar para cima e para baixo o tecido da retina na solução de digestão cinco vezes.
- Incubar a mistura de digestão em banho-maria de 37 °C durante 20 min.
- Use um pipetador P1000 e uma ponta de pipeta para pipetar a mistura de digestão para cima e para baixo a cada 5 minutos. Após a incubação, o tecido da retina se dissolveu em uma única suspensão celular, de modo que a mistura de digestão deve estar turva (Figura 1C).
4. Conte células
- Coloque uma centrífuga de mesa a 4 °C e coloque os tubos de mistura de digestão no interior. Pellet a mistura de digestão por centrifugação a 375 x g durante 5 min.
- Remova cuidadosamente a solução de digestão sobrenadante por pipetagem. Não perturbe o pellet celular (Figura 1C). Ressuspeite o pellet em 500 μL de PBS 1x gelado misturando suavemente para cima e para baixo com a pipeta.
- Conte as células com um hemocitômetro sob um microscópio.
NOTA: A contagem de células é de aproximadamente 1 x 106 células por duas retinas. - Certifique-se de que as células sejam ressuspensas adequadamente por mistura suave e, em seguida, alíquota de 20 μL de suspensão celular em três tubos para coloração de controle (tubo 1: controle IgG, tubo 2: controle CD31 e tubo 3: controle CD45), conforme descrito e usado anteriormente17,18.
- Coloque uma centrífuga de mesa a 4 °C e coloque tubos contendo células no interior. Pellet as células por centrifugação a 375 x g durante 5 min.
5. Imunomanchar as células com anticorpos
- Prepare 100 μL de tampão de coloração por duas retinas coradas mais quatro tubos de controle. Adicione FBS, HEPES e D-Glucose ao tampão HBSS a uma concentração final de 10% FBS, 10 mM HEPES e 1 mg/mL de D-Glicose.
- Obter anticorpos conjugados fluorescentemente contra CD31 e CD45. Adicione os anticorpos a uma diluição de 1:100 no tampão de coloração (tubo 1: apenas anticorpo IgG, tubo 2: apenas anticorpo CD31 e tubo 3: apenas anticorpo CD45). Adicionar 1 μL de anticorpo por 100 μL do tampão corante para uma concentração final de anticorpos de 2 μg/mL.
- Remova cuidadosamente o PBS do pellet da célula lavada por pipeta.
- Ressuscite o pellet em 100 μL de solução de coloração de anticorpos por 0,5 x 106 células.
- Incubar a suspensão unicelular com a solução de coloração de anticorpos por 30 minutos no gelo no escuro. Bata nos tubos a cada 10 minutos para misturar suavemente as células.
6. Prepare-se para a classificação de células ativadas por fluorescência
- Coloque uma centrífuga de mesa a 4 °C e coloque os tubos contendo células no interior. Células de pellets por centrifugação a 375 x g durante 5 min.
- Remova o sobrenadante e ressuspenda essas células em 500 μL de 1x PBS para lavar.
- Coloque uma centrífuga de mesa a 4 °C e coloque os tubos contendo células no interior. Células de pellets por centrifugação a 375 x g durante 5 min
- Prepare 1,5 mL de tampão FACS (1% FBS em 1x PBS).
- Remova o sobrenadante e ressuspenda o pellet celular em 300 μL de tampão FACS misturando suavemente com um pipettor.
- Adicionar iodeto de propídio (PI) aos tubos de amostra que contêm o tampão FACS até uma concentração final de 0,5 μg/mL. O IP é usado como um marcador de viabilidade.
- Transfira e combine as suspensões celulares em tubos de ensaio de 5 mL através de uma tampa de encaixe de filtro celular. A tampa de encaixe do filtro celular deve conter um filtro de 35 μm, e as suspensões celulares podem ser pipetadas diretamente no filtro.
- Mantenha os tubos FACS no gelo no escuro e transfira-os para o classificador de células.
7. Configurar o instrumento FACS
- Instale um bocal de 100 μm em um instrumento FACS.
NOTA: Este bocal de tamanho minimiza o volume de coleta e maximiza a densidade de células isoladas. - Prepare 250 μL de 1x PBS em um tubo de microcentrífuga de 1,5 mL como um tubo de coleta a ser instalado no instrumento FACS.
8. Isolar células endoteliais viáveis via FACS
- Ligue o instrumento FACS e o computador. Clique no ícone do software FACS para abrir o software FACS no computador para executar e operar o instrumento FACS. Realizar testes de controle de qualidade de rotina.
- Carregue as amostras coradas pelo controle no instrumento FACS para ajustar os eixos. Comece com células apenas de anticorpos IgG para gerar e ajustar FSC e SSC, depois com anticorpos CD31 apenas para gerar e ajustar CD31 e, em seguida, com anticorpos CD45 apenas para gerar e ajustar CD45. Registre dados de amostras de controle.
- Carregar células de amostra coradas no instrumento FACS e executar a amostra.
- Porta das células com base nos parâmetros de dispersão frontal e dispersão lateral (FSC-A e SSC-A, respectivamente) (Figura 2A).
NOTA: Os parâmetros FSC-A e SSC-A são usados para selecionar células com base no tamanho, densidade, granularidade, propriedades da superfície e índice de refração. - Células gate por FSC-A e FSC-H para identificar duplicatas celulares e coletar apenas células simples (Figura 2A).
NOTA: Os parâmetros FSC-A e FSC-H são usados para selecionar células individuais com base no princípio de que, durante o espalhamento para a frente, os duplicados de células serão gotículas contendo uma maior relação área-altura. - Células gate por PI e SSC-A para identificar células viáveis (Figura 2A).
NOTA: As células viáveis serão PI-negativas. - Faça uma porta CD31+/CD45- com controles e células de porta por CD31 e CD45 (Figura 2A,B).
- Insira um tubo de coleta com 250 μL de tampão FACS.
- Comece a classificar as células que são CD31-positivo/CD45-negativo no tubo de coleta instalado.
- Mantenha os tubos de coleta no gelo para uma análise mais aprofundada. As amostras podem ser processadas para aplicativos de sequenciamento de próxima geração nesta etapa1.
9. Realizar ensaio de viabilidade
- Misture 10 μL de células com 10 μL de solução azul de tripano a 0,4% para avaliar a viabilidade celular.
- Conte as células viáveis pipetando a mistura corada em uma lâmina de hemocitômetro. Coloque a lâmina sob um microscópio para contagens precisas de viabilidade. Conte as células azuis como inviáveis e conte as células claras como viáveis.
- Divida a contagem de células viáveis pela contagem total de células para calcular a viabilidade percentual.
10. Realizar ensaio de expressão gênica
- Obter 10.000-20.000 células por amostra e realizar isolamento de RNA para analisar a expressão gênica das células endoteliais da retina murina. O isolamento de RNA é realizado usando um kit comercialmente disponível que usa precipitação de RNA à base de etanol e, em seguida, centrífuga e colunas de membrana para lavar e eluir o RNA purificado do lisado celular. As amostras podem ser processadas para alguns aplicativos de sequenciamento de próxima geração nesta etapa1.
- Converter o RNA em cDNA utilizando a enzima transcriptase reversa e reagentes de suporte19.
- Quantificar usando um corante de ligação ao DNA e uma máquina quantitativa de PCR (qPCR)20. Carregue amostras de cDNA em uma placa qPCR de 384 poços com primers para amplificar genes de interesse. O volume de reação deve totalizar 10 μL, incluindo o corante de ligação ao DNA, que será usado para quantificar a amplificação gênica.
- Use CD31, VE-Caderrin, CD45 e β-actina para frente e para trás para medir a expressão gênica.
- Use o exemplo para aplicativos de sequenciamento de próxima geração1.
Access restricted. Please log in or start a trial to view this content.
Resultados
A digestão do tecido retiniano e a imunocoloração para CD31 e CD45 resultam em uma população identificável de células endoteliais CD31+/CD45- após o gating para células, células únicas e viabilidade (Figura 2A). A imunocoloração CD45 é necessária para eliminar as células CD31+/CD45+, que incluem plaquetas e alguns leucócitos21. Controles devem ser realizados para cada experimento para mostrar a especificidade dos anticorpos e orientar a estratégia de...
Access restricted. Please log in or start a trial to view this content.
Discussão
Este protocolo descreve um método para o isolamento de células endoteliais do tecido retiniano murino pós-natal que foi otimizado para alto número de células, pureza e viabilidade. A pureza celular é obtida pelo isolamento FACS de populações de células endoteliais da suspensão unicelular digerida por imunocoloração CD31+/CD45-. A qualidade do isolamento é quantificada em ensaios de viabilidade pela coloração azul de tripano e expressão gênica por qPCR para CD31, CD45 e VE-Caderina (embora a VE-Caderrina ...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm divulgações relevantes.
Agradecimentos
Obrigado ao Yale Flow Cytometry Facility, ao University of Virginia Flow Cytometry Core Facility, ao Yale Center for Genomic Analysis e ao University of Virginia Genome Analysis and Technology Core por seu esforço, experiência e aconselhamento em contribuir para os experimentos apresentados. Este estudo foi financiado por subsídios do NIH para N.W.C. (T32 HL007224, T32 HL007284), S.C. (T32 HL007284), K.W. (R01 HL142650) e K.K.H. (R01 HL146056, R01 DK118728, UH3 EB025765).
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 2 mL Eppendorf safe-lock tubes | USA Scientific | 4036-3352 | |
| 5 ml Falcon Test Tubes with Cell Strainer Snap Cap | Corning | 352235 | |
| 60 mm Non TC-treated Culture Dish | Corning | 430589 | |
| APC Rat Anti-Mouse CD31 | BD Biosciences | 551262 | |
| APC Rat IgG2a κ Isotype Control | BD Biosciences | 553932 | |
| BD FACSChorus Software | BD Biosciences | FACSCHORUS | |
| BD FACSMelody Cell Sorter | BD Biosciences | FACSMELODY | |
| Collagenase Type II | Sigma-Aldrich | 234115 | |
| Costar 48-well Clear TC-treated Multiple Well Plates, Individually Wrapped, Sterile | Corning | 3548 | |
| D-Glucose | Gibco | A2494001 | |
| Disposable Graduated Transfer Pipettes | Fisher Scientific | 12-711-9AM | |
| Dissecting Pan Wax | Carolina | 629100 | |
| Dissection scissors | Fine Science Tools | 14085-08 | |
| Dissection Stereo Microscope M165 FC | Leica | M165FC | |
| Dulbecco's Modified Eagle Medium (DMEM) | Gibco | 11965-052 | |
| Dulbecco’s Phosphate Buffered Saline (PBS) | Gibco | 14190144 | |
| Eppendorf Flex-Tubes Microcentrifuge Tubes 1.5 mL | Sigma-Aldrich | 22364120 | |
| Fetal Bovine Serum (FBS) | Gemini Bio | 100-106 | |
| Fine dissection forceps | Fine Science Tools | 11250-00 | |
| Hank's Buffered Salt Solution (HBSS) | Gibco | 14175095 | |
| HEPES (1M) | Gibco | 15630130 | |
| iScript cDNA Synthesis Kit | Bio-Rad | 1708890 | |
| Isoflurane, USP | Covetrus | 11695067772 | |
| Isotemp General Purpose Deluxe Water Bath | Fisher Scientific | FSGPD20 | |
| Primer: ActB_Forward: 5’- agagggaaatcgtgcgtgac -3’ | Integrated DNA Technologies | N/A | |
| Primer: ActB_Reverse: 5’- caatagtgatgacctggccgt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD31_Forward: 5’- gagcccaatcacgtttcagttt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD31_Reverse: 5’- tccttcctgcttcttgctagct -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD45_Forward: 5’- gggttgttctgtgccttgtt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD45_Reverse: 5’- ctggacggacacagttagca -3’ | Integrated DNA Technologies | N/A | |
| Primer: VE-Cadherin_Forward: 5’- tcctctgcatcctcactatcaca -3’ | Integrated DNA Technologies | N/A | |
| Primer: VE-Cadherin_Reverse: 5’- gtaagtgaccaactgctcgtgaat -3’ | Integrated DNA Technologies | N/A | |
| Propidium iodide | Sigma-Aldrich | P4864 | |
| RNeasy Plus Mini Kit | Qiagen | 74134 | |
| Sorvall Legend Micro 21R Centrifuge, Refrigerated | ThermoFisher | 75002477 | |
| SYBR-Green iTaq Universal SYBR Green Supermix | Bio-Rad | 172-5120 | |
| Trypan Blue Solution | ThermoFisher | 15250061 | |
| V450 Rat Anti-Mouse CD45 | BD Biosciences | 560501 | |
| V450 Rat IgG2b, κ Isotype Control | BD Biosciences | 560457 |
Referências
- Slatko, B. E., Gardner, A. F., Ausubel, F. M. Overview of next-generation sequencing technologies. Current Protocols in Molecular Biology. 122 (1), 59(2018).
- Chavkin, N. W., Hirschi, K. K. Single cell analysis in vascular biology. Frontiers in Cardiovascular Medicine. 7, 42(2020).
- Ma, F., Hernandez, G. E., Romay, M., Iruela-Arispe, M. L. Single-cell RNA sequencing to study vascular diversity and function. Current Opinion in Hematology. 28 (3), 221-229 (2021).
- Potter, A. S., Potter, S. S., S, Dissociation of tissues for single-cell analysis. Methods in Molecular Biology. 1926, 55-62 (2019).
- Braga, F. A. V., Miragaia, R. J. Tissue handling and dissociation for single-cell RNA-seq. Single Cell Methods: Methods in Molecular Biology. 1979, 9-21 (2019).
- Brink, S. C., et al. Single-cell sequencing reveals dissociatin-induced gene expression in tissue subpopulations. Nature Methods. 14 (10), 935-936 (2017).
- Skulska, K., Wegrzyn, A. S., Chelmonska-Soyta, A., Chodaczek, G. Impact of tissue enzymatic digestion on analysis of immune cells in mouse reproductive mucosa with a focus on gammadelta T cells. Journal of Immunological Methods. 474, 112665(2019).
- Connolly, S., Hores, T., Smith, L., D'Amore, P. Characterization of vascular development in the mouse retina. Microvascular Research. 36 (3), 275-290 (1988).
- Crist, A., Young, C., Meadows, S. Characterization of arteriovenous identity in the developing neonate mouse retina. Gene Expression Patterns: GEP. 23-24, 22-31 (2017).
- dela Paz, N. G., D'Amore, P. A. Arterial versus venous endothelial cells. Cell and Tissue Research. 335 (1), 5-16 (2009).
- Fang, J. S., Hirschi, K. K. Molecular regulation of arteriovenous endothelial cell specification. F1000Res. 8, (2019).
- Smith, L. E., et al. Oxygen-induced retinopathy in the mouse. Investigative Ophthalmology and Visual Science. 35 (1), 101-111 (1994).
- Pitulescu, M. E., Schmidt, I., Benedito, R., Adams, R. H. Inducible gene targeting in the neonatal vasculature and analysis of retinal angiogenesis in mice. Nature Protocols. 5 (9), 1518-1534 (2010).
- Ruiz, S., et al. A mouse model of hereditary hemorrhagic telangiectasia generated by transmammary-delivered immunoblocking of BMP9 and BMP10. Scientific Reports. 6, 37366(2016).
- Fang, J. S., et al. Shear-induced Notch-Cx37-p27 axis arrests endothelial cell cycle to enable arterial specification. Nature Communication. 8 (1), 2149(2017).
- Ola, R., et al. SMAD4 prevents flow induced arterial-venous malformations by inhibiting Casein Kinase 2. Circulation. 138 (21), 2379-2394 (2018).
- Chavkin, N. W., Walsh, K., Hirschi, K. K. Isolation of highly purified and viable retinal endothelial cells. Journal of Vascular Research. 58 (1), 49-57 (2020).
- Hulspas, R., O'Gorman, M. R. G., Wood, B. L., Gratama, J. W., Sutherland, D. R. Considerations for the control of background fluorescence in clinical flow cytometry. Cytometry PartB: Clinical Cytometry. 76 (6), 355-364 (2009).
- Bachman, J. Reverse-transcription PCR (RT-PCR). Methods in Enzymology. 530, 67-74 (2013).
- Green, M. R., Sambrook, J. Quantification of RNA by real-time Reverse Transcription-Polymerase Chain Reaction (RT-PCR). Cold Spring Harbour Protocols. 2018 (10), (2018).
- Liu, L., Shi, G. P. CD31: beyond a marker for endothelial cells. Cardiovascular Research. 94 (1), 3-5 (2012).
- Zarkada, G., et al. Specialized endothelial tip cells guide neuroretina vascularization and blood-retina-barrier formation. Developmental Cell. 56 (15), 2237-2251 (2021).
- Su, X., Sorenson, C. M., Sheibani, N. Isolation and characterization of murine retinal endothelial cells. Molecular Vision. 9, 171-178 (2003).
- Benedito, R., et al. The notch ligands Dll4 and Jagged1 have opposing effects on angiogenesis. Cell. 137 (6), 1124-1135 (2009).
- Daneman, R., et al. Wnt/beta-catenin signaling is required for CNS, but not non-CNS, angiogenesis. Proceedings of the National Academy of Sciences of the United States of America. 106 (2), 641-646 (2009).
- Okabe, K., et al. Neurons limit angiogenesis by titrating VEGF in retina. Cell. 159 (3), 584-596 (2014).
- Crist, A. M., Lee, A. R., Patel, N. R., Westhoff, D. E., Meadows, S. M. Vascular deficiency of Smad4 causes arteriovenous malformations: a mouse model of Hereditary Hemorrhagic Telangiectasia. Angiogenesis. 21 (2), 363-380 (2018).
- Kim, Y. H., Choe, S. W., Chae, M. Y., Hong, S., Oh, S. P. SMAD4 deficiency leads to development of arteriovenous malformations in neonatal and adult mice. Journal of the American Heart Association. 7 (21), 009514(2018).
- Ma, W., et al. Absence of TGFbeta signaling in retinal microglia induces retinal degeneration and exacerbates choroidal neovascularization. eLife. 8, 42049(2019).
- Luo, W., et al. Arterialization requires the timely suppression of cell growth. Nature. 589 (7842), 437-441 (2021).
- Lawson, N. D., Vogel, A. M., Weinstein, B. M. sonic hedgehog and vascular endothelial growth factor act upstream of the Notch pathway during arterial endothelial differentiation. Developmental Cell. 3 (1), 127-136 (2002).
- Larrivee, B., et al. ALK1 signaling inhibits angiogenesis by cooperating with the Notch pathway. Developmental Cell. 22 (3), 489-500 (2012).
- Wythe, J. D., et al. ETS factors regulate Vegf-dependent arterial specification. Developmental Cell. 26 (1), 45-58 (2013).
- Davis, D. M., Purvis, J. E. Computational analysis of signaling patterns in single cells. Seminars in Cell and Developmental Biology. 0, 35-43 (2015).
- Gaudet, S., Miller-Jensen, K. Redefining signaling pathways with an expanding single-cell toolbox. Trends in Biotechnology. 34 (6), 458-469 (2016).
- Aibar, S., et al. SCENIC: single-cell regulatory network inference and clustering. Nature Methods. 14 (11), 1083-1086 (2017).
- Trapnell, C., et al. The dynamics and regulators of cell fate decisions are revealed by pseudotemporal ordering of single cells. Nature Biotechnology. 32 (4), 381-386 (2014).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados