Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Isolement de cellules endothéliales rétiniennes murines pour le séquençage de nouvelle génération
Dans cet article
Résumé
Ce protocole décrit une méthode d’isolement des cellules endothéliales rétiniennes postnatales murines optimisées pour le rendement, la pureté et la viabilité cellulaires. Ces cellules conviennent aux approches de séquençage de nouvelle génération.
Résumé
Les améliorations récentes du séquençage de nouvelle génération ont fait progresser les connaissances des chercheurs en biologie moléculaire et cellulaire, plusieurs études révélant de nouveaux paradigmes en biologie vasculaire. L’application de ces méthodes à des modèles de développement vasculaire nécessite l’optimisation des techniques d’isolement cellulaire à partir de tissus embryonnaires et postnataux. Le rendement, la viabilité et la pureté des cellules doivent tous être maximaux pour obtenir des résultats précis et reproductibles à partir d’approches de séquençage de nouvelle génération. Le modèle de vascularisation rétinienne de souris néonatale est utilisé par les chercheurs pour étudier les mécanismes du développement vasculaire. Les chercheurs ont utilisé ce modèle pour étudier les mécanismes de l’angiogenèse et la spécification du devenir artériel-veineux pendant la formation et la maturation des vaisseaux sanguins. L’application de techniques de séquençage de nouvelle génération pour étudier le modèle de développement vasculaire rétinien nécessite l’optimisation d’une méthode d’isolement des cellules endothéliales rétiniennes qui maximise le rendement, la viabilité et la pureté cellulaires. Ce protocole décrit une méthode d’isolement, de digestion et de purification des tissus murins de la rétine à l’aide du tri cellulaire activé par fluorescence (FACS). Les résultats indiquent que la population de cellules endothéliales CD31+/CD45- purifiées par FACS est fortement enrichie pour l’expression génique des cellules endothéliales et ne présente aucun changement de viabilité pendant 60 minutes après la FACS. Sont inclus les résultats représentatifs des approches de séquençage de nouvelle génération sur les cellules endothéliales isolées à l’aide de cette méthode, y compris le séquençage de l’ARN en vrac et le séquençage de l’ARN unicellulaire, démontrant que cette méthode d’isolement des cellules endothéliales rétiniennes est compatible avec les applications de séquençage de la prochaine génération. Cette méthode d’isolement des cellules endothéliales rétiniennes permettra des techniques de séquençage avancées pour révéler de nouveaux mécanismes de développement vasculaire.
Introduction
La capacité à haut débit du séquençage des acides nucléiques via des approches de séquençage de nouvelle génération a grandement fait progresser les connaissances des chercheurs en biologie moléculaire et cellulaire. Ces techniques avancées comprennent le séquençage de l’ARN du transcriptome entier, le séquençage de l’ADN des régions ciblées pour identifier les polymorphismes mononucléotidiques (SNP), le séquençage de l’ADN des facteurs de transcription liés dans le séquençage de l’immunoprécipitation de la chromatine (ChIP) ou les régions de chromatine ouvertes dans le dosage pour le séquençage de la chromatine accessible par la transposase (ATAC) et le séquençage de l’ARN unicellulaire1 . En biologie vasculaire, ces progrès ont permis aux chercheurs d’élucider des mécanismes complexes de développement et de maladie, ainsi que de distinguer les modèles d’expression génique le long d’un continuum de phénotypesvariés 2,3. Les expériences futures peuvent définir davantage des mécanismes complexes en combinant le séquençage de nouvelle génération avec des modèles évalués de développement vasculaire, mais les méthodes de préparation des échantillons doivent être compatibles avec les techniques de séquençage avancées.
La qualité, la précision et la reproductibilité des approches de séquençage de nouvelle génération dépendent de la méthode de préparation des échantillons. Lors de l’isolement d’un sous-ensemble de cellules ou de la génération de suspensions unicellulaires à partir de tissus, des méthodes optimales de digestion et de purification sont essentielles pour maximiser le nombre de cellules, la viabilité et la pureté de la population cellulaire 4,5. Cela nécessite un équilibre dans la méthode de digestion: une digestion forte est nécessaire pour libérer les cellules du tissu et obtenir suffisamment de cellules pour les approches en aval, mais la viabilité cellulaire sera affectée négativement si la digestion est trop forte 6,7. De plus, la pureté de la population cellulaire est nécessaire pour obtenir des résultats robustes et une analyse précise des données, ce qui peut être accompli via FACS. Cela souligne l’importance d’optimiser les méthodes d’isolement cellulaire pour appliquer le séquençage de nouvelle génération aux modèles établis de développement vasculaire.
Un modèle bien caractérisé pour étudier le développement vasculaire est le modèle de développement vasculaire murin de la rétine. Le système vasculaire murin de la rétine se développe postnatalement dans un plexus superficiel bidimensionnel, avec une germination angiogénique initiale du nerf optique visible au jour postnatal (P)3, un front angiogénique avec des cellules pédoncules et des pointes et une maturation initiale des vaisseaux visibles à P6, et une maturation du plexus vasculaire visible après P9 8,9. Lors du remodelage du plexus vasculaire initial, les cellules endothéliales subissent une spécification vers les phénotypes artériels, capillaires et veineux dans différents vaisseaux pour générer un réseau circulatoire10,11. Par conséquent, cette méthode permet aux chercheurs de visualiser la formation du plexus vasculaire angiogénique et la spécification et la maturation artério-veineuses endothéliales à différents moments du développement9. De plus, ce modèle fournit une méthode pour étudier les effets de la manipulation transgénique sur l’angiogenèse et le développement du plexus vasculaire, qui a été appliquée pour l’étude du développement vasculaire, des malformations artério-veineuses et de la néovascularisation induite par l’oxygène 12,13,14,15,16 . Afin de combiner les approches de séquençage de nouvelle génération avec le modèle de développement vasculaire murin de la rétine, un protocole optimisé pour l’isolement des cellules endothéliales du tissu rétinien est nécessaire.
Ce protocole décrit une méthode optimisée pour digérer le tissu rétinien de souris à P6 afin de maximiser le rendement, la pureté et la viabilité cellulaires. Le tissu rétinien est isolé chez des souris P6, digéré pendant 20 minutes, immunocoloré pour CD31 et CD45 et purifié par FACS pour isoler une suspension unicellulaire de cellules endothéliales en environ 2,5 heures (Figure 1A). Ces cellules endothéliales se sont avérées maintenir une viabilité élevée pendant 60 minutes après l’isolement17, ce qui a permis de préparer la bibliothèque pour les méthodes de séquençage de nouvelle génération. De plus, des résultats représentatifs sont fournis pour les résultats de contrôle et de contrôle de la qualité FACS provenant de deux méthodes distinctes de séquençage de nouvelle génération utilisant ce protocole d’isolation : le séquençage de l’ARN du transcriptome entier et le séquençage de l’ARN unicellulaire. Cette méthode permet d’utiliser des approches de séquençage de nouvelle génération en conjonction avec le modèle de vascularisation rétinienne pour élucider de nouveaux mécanismes de développement vasculaire.
Protocole
Les comités institutionnels de soin et d’utilisation des animaux de l’Université de Yale et de l’Université de Virginie ont approuvé toutes les expériences sur les animaux énumérées dans ce protocole.
1. Obtenir des yeux de souris pour l’isolement rétinien
- Préparez 1x PBS glacé et ajoutez 500 μL à chaque puits d’une plaque de 48 puits.
- Euthanasier les souris néonatales au sixième jour postnatal (P6) selon les directives approuvées de l’établissement. Pour cette expérience, des portées d’environ 4 à 8 souris néonatales sont euthanasiées à P6 par inhalation d’isoflurane pendant au moins trois minutes après l’arrêt respiratoire, suivi d’une décapitation.
- Retirez les yeux de chacune des souris. Coupez la peau et la membrane sur l’œil en coupant perpendiculairement à la paupière à l’aide de ciseaux à dissection. Ensuite, utilisez des pinces pour appuyer doucement au-dessus et au-dessous de l’œil afin que l’œil sorte de l’orbite.
- Pincez soigneusement sous l’œil avec la pince et coupez le nerf optique qui maintient l’œil attaché. Ensuite, placez chaque œil dans la plaque de 48 puits dans le PBS 1x glacé préparé jusqu’à la fin de la récolte.
REMARQUE: Utilisez un puits par souris, avec les deux yeux d’une seule souris dans le même puits.
- Pincez soigneusement sous l’œil avec la pince et coupez le nerf optique qui maintient l’œil attaché. Ensuite, placez chaque œil dans la plaque de 48 puits dans le PBS 1x glacé préparé jusqu’à la fin de la récolte.
2. Isoler le tissu rétinien de la souris
- Remplissez une boîte de Pétri, tapissée d’un tampon de dissection sur le fond, avec 500 μL de PBS 1x glacé pour submerger les yeux et placez-la sous un microscope à dissection réglé sur un grossissement de 4,0x.
- Suspendre les yeux dans le tampon de dissection à l’aide d’une pipette de transfert avec une pointe large afin de ne pas endommager les yeux.
REMARQUE: Assurez-vous que les yeux sont complètement couverts de PBS.
- Suspendre les yeux dans le tampon de dissection à l’aide d’une pipette de transfert avec une pointe large afin de ne pas endommager les yeux.
- À l’aide de deux pinces à dissection fine, tenez le nerf optique avec l’une des pinces pour la stabilisation et, avec la deuxième pince, percez un trou dans la chambre antérieure où la cornée et la sclérotique se connectent (Figure 1B). Déchirez le trou en cercle à environ 75% du chemin autour de la cornée.
- Tout en tenant le nerf optique avec l’une des pinces, utilisez la deuxième pince pour arracher doucement la sclérotique du tissu rétinien.
- Comme ci-dessus, utilisez la deuxième pince pour arracher doucement le corps vitré du tissu rétinien.
- Encore une fois, à l’aide de la deuxième pince, retirez doucement la lentille et les vaisseaux du plexus hyaloïde qui ne se sont pas détachés lorsque le corps vitré a été retiré. Pour ce faire, pénétrez dans la rétine avec une pince ouverte jusqu’à presque toucher la rétine, fermez les forceps pour saisir le cristallin et les vaisseaux du plexus hyaloïde et retirez les pinces de la rétine. Cela peut être répété jusqu’à ce que tous les vaisseaux soient retirés.
REMARQUE: Le plexus hyaloïde ressemble à une structure claire, semblable à une toile.
- Remplir 2 mL de tubes microcentrifugés avec 500 μL de 1x PBS glacé et placer les rétines dans les tubes. Isolez toutes les rétines des yeux et placez-les dans un PBS glacé avant de continuer.
REMARQUE: Utilisez un tube par souris, avec deux rétines de la même souris partageant un tube. Si le tissu rétinien est déchiré, transférez-le en plusieurs morceaux. Le temps total de dissection tissulaire rétinienne pour une portée de souris devrait prendre de 15 à 60 minutes, en commençant par l’euthanasie et en terminant par l’isolement final du tissu rétinien.
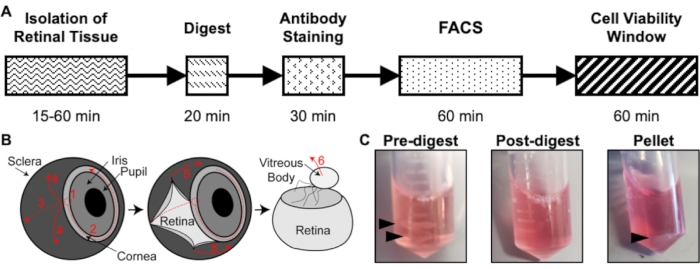
Figure 1 : Vue d’ensemble du protocole d’isolement. (A) Schéma de la chronologie de l’isolement avec une estimation du temps pour chaque étape : Isolement du tissu rétinien, digestion, coloration des anticorps, FACS et fenêtre de viabilité cellulaire. (B) Guide étape par étape pour l’isolement du tissu rétinien de l’œil, avec des étapes de dissection numérotées: 1) percer la cornée, 2) déchirer la cornée, 3) la sclérotique lacrymale, 4) retirer la sclérotique de la rétine, 5) enlever davantage la sclérotique et le tissu de connexion de la rétine, 6) retirer le corps vitré et les vaisseaux vitréens de la rétine. (C) Images représentatives du tissu rétinien au cours des différentes étapes de la digestion: pré-digestion, post-digestion, granulé (les flèches noires mettent en évidence le tissu rétinien ou la pastille cellulaire). Republié avec la permission de Chavkin et al. publié dans S. Karger AG, Basel17. Veuillez cliquer ici pour voir une version agrandie de cette figure.
3. Digérer le tissu rétinien en une seule suspension cellulaire
- Préparer 500 μL de solution de digestion pour deux rétines isolées. Ajouter le FBS et la collagénase de type II au DMEM à une concentration finale de 10 % de FBS et de 1 mg/mL de collagénase de type II. Mélanger et réchauffer la solution à 37 °C à l’aide d’un bain-marie.
- Retirer l’excès de PBS des tubes microcentrifugés de 2 mL en le pipetant soigneusement. Laissez suffisamment de PBS pour couvrir complètement le tissu rétinien, environ 100 μL dans chaque tube.
- Ajouter 500 μL de la solution de digestion dans chaque tube contenant du tissu rétinien (Figure 1C).
- Utilisez une pipette P1000 et l’embout de la pipette pour pipeter le tissu rétinien dans la solution de digestion cinq fois.
- Incuber le mélange de digestion dans un bain-marie à 37 °C pendant 20 min.
- Utilisez une pipette P1000 et l’embout de la pipette pour pipeter le mélange de digestion de haut en bas toutes les 5 minutes. Après l’incubation, le tissu rétinien s’est dissous en une seule suspension cellulaire, de sorte que le mélange de digestion doit être trouble (Figure 1C).
4. Compter les cellules
- Placez une centrifugeuse de table à 4 °C et placez les tubes de mélange de digestion à l’intérieur. Enduire le mélange de digestion par centrifugation à 375 x g pendant 5 min.
- Retirer délicatement la solution de digestion surnageante par pipetage. Ne pas déranger la pastille de la cellule (figure 1C). Re-suspendre la pastille dans 500 μL de 1x PBS glacé en mélangeant doucement de haut en bas avec la pipette.
- Comptez les cellules avec un hémocytomètre au microscope.
REMARQUE: Le nombre de cellules est d’environ 1 x 106 cellules pour deux rétines. - S’assurer que les cellules sont correctement remises en suspension par mélange doux, puis aliquote 20 μL de suspension cellulaire dans trois tubes pour la coloration de contrôle (tube 1: contrôle IgG, tube 2: contrôle CD31 et tube 3: contrôle CD45), comme décrit et utilisé précédemment17,18.
- Placez une centrifugeuse de table à 4 °C et placez des tubes contenant des cellules à l’intérieur. Enduire les cellules par centrifugation à 375 x g pendant 5 min.
5. Immunocolorer les cellules avec des anticorps
- Préparer 100 μL de tampon de coloration pour deux rétines colorées plus quatre tubes de contrôle. Ajouter FBS, HEPES et D-Glucose au tampon HBSS à une concentration finale de FBS 10%, 10 mM HEPES, et 1 mg / mL D-Glucose.
- Obtenir des anticorps conjugués par fluorescence contre CD31 et CD45. Ajouter les anticorps à une dilution de 1:100 dans le tampon de coloration (tube 1 : anticorps IgG uniquement, tube 2 : anticorps CD31 uniquement, et tube 3 : anticorps CD45 uniquement). Ajouter 1 μL d’anticorps par 100 μL de tampon de coloration pour une concentration finale d’anticorps de 2 μg/mL.
- Retirez délicatement le PBS de la pastille de cellule lavée par pipetage.
- Remettez la pastille en suspension dans 100 μL de solution de coloration d’anticorps pour 0,5 x 106 cellules.
- Incuber la suspension unicellulaire avec la solution de coloration d’anticorps pendant 30 minutes sur de la glace à l’obscurité. Tapotez les tubes toutes les 10 minutes pour mélanger doucement les cellules.
6. Préparez-vous au tri cellulaire activé par fluorescence
- Placez une centrifugeuse de table à 4 °C et placez les tubes contenant les cellules à l’intérieur. Cellules à granulés par centrifugation à 375 x g pendant 5 min.
- Retirez le surnageant et remettez ces cellules en suspension dans 500 μL de 1x PBS pour les laver.
- Placez une centrifugeuse de table à 4 °C et placez les tubes contenant les cellules à l’intérieur. Cellules à granulés par centrifugation à 375 x g pendant 5 min
- Préparer 1,5 mL de tampon FACS (1% FBS dans 1x PBS).
- Retirer le surnageant et remettre en suspension la pastille de cellule dans 300 μL de tampon FACS en mélangeant doucement avec un pipeteur.
- Ajouter de l’iodure de propidium (PI) aux tubes d’échantillon contenant le tampon FACS jusqu’à une concentration finale de 0,5 μg/mL. L’IP est utilisé comme marqueur de viabilité.
- Transférer et combiner les suspensions cellulaires dans des tubes à essai de 5 mL à travers un capuchon de crépine à cellules. Le capuchon de la crépine à cellules doit contenir un filtre de 35 μm et les suspensions cellulaires peuvent être pipetées directement sur le filtre.
- Gardez les tubes FACS sur de la glace dans l’obscurité et transférez-les dans le trieur de cellules.
7. Mettre en place l’instrument FACS
- Installez une buse de 100 μm dans un instrument FACS.
REMARQUE: Cette buse de taille minimise le volume de collecte et maximise la densité de cellules isolées. - Préparer 250 μL de 1x PBS dans un tube microcentrifuge de 1,5 mL comme tube de collecte à installer dans l’instrument FACS.
8. Isoler les cellules endothéliales viables via FACS
- Allumez l’instrument FACS et l’ordinateur. Cliquez sur l’icône du logiciel FACS pour ouvrir le logiciel FACS sur l’ordinateur afin d’exécuter et d’utiliser l’instrument FACS. Effectuer des tests de contrôle de la qualité de routine.
- Chargez les échantillons colorés de contrôle dans l’instrument FACS pour ajuster les axes. Commencez avec les cellules d’anticorps IgG uniquement pour générer et ajuster le FSC et le SSC, puis l’anticorps CD31 uniquement pour générer et ajuster CD31, puis l’anticorps CD45 uniquement pour générer et ajuster CD45. Enregistrer les données des échantillons témoins.
- Chargez les cellules d’échantillon colorées dans l’instrument FACS et exécutez l’échantillon.
- Grille des cellules en fonction des paramètres de diffusion vers l’avant et de diffusion latérale (FSC-A et SSC-A, respectivement) (Figure 2A).
REMARQUE : Les paramètres FSC-A et SSC-A sont utilisés pour sélectionner les cellules en fonction de leur taille, de leur densité, de leur granularité, de leurs propriétés de surface et de leur indice de réfraction. - Cellules portes FSC-A et FSC-H pour identifier les doublets cellulaires et ne collecter que des cellules individuelles (Figure 2A).
REMARQUE: Les paramètres FSC-A et FSC-H sont utilisés pour sélectionner des cellules individuelles en fonction du principe selon lequel, pendant la diffusion directe, les doublets de cellules seront des gouttelettes contenant un rapport surface/hauteur supérieur. - Cellules portes par IP et SSC-A pour identifier les cellules viables (Figure 2A).
REMARQUE: Les cellules viables seront PI-négatives. - Fabriquer une grille CD31+/CD45- avec des commandes et des cellules de grille par CD31 et CD45 (Figure 2A,B).
- Insérez un tube collecteur avec 250 μL de tampon FACS.
- Commencez à trier les cellules CD31-positives/CD45-négatives dans le tube de collecte installé.
- Gardez les tubes de collecte sur la glace pour une analyse plus approfondie. Les échantillons peuvent être traités pour les applications de séquençage de nouvelle génération à cette étape1.
9. Effectuer un essai de viabilité
- Mélanger 10 μL de cellules avec 10 μL de solution de bleu de trypan à 0,4% pour évaluer la viabilité cellulaire.
- Compter les cellules viables en pipetant le mélange coloré sur une lame d’hémocytomètre. Placez la lame sous un microscope pour obtenir un comptage précis de la viabilité. Comptez les cellules bleues comme non viables et les cellules claires comme viables.
- Divisez le nombre de cellules viables par le nombre total de cellules pour calculer le pourcentage de viabilité.
10. Effectuer un test d’expression génique
- Obtenir 10 000 à 20 000 cellules par échantillon et effectuer une isolation de l’ARN pour analyser l’expression des gènes des cellules endothéliales rétiniennes murines. L’isolement de l’ARN est effectué à l’aide d’un kit disponible dans le commerce qui utilise la précipitation de l’ARN à base d’éthanol, puis la centrifugation et les colonnes membranaires pour laver et éluer l’ARN purifié du lysat cellulaire. Les échantillons peuvent être traités pour certaines applications de séquençage de nouvelle génération à cette étape1.
- Convertir l’ARN en ADNc en utilisant l’enzyme de transcriptase inverse et les réactifsde soutien 19.
- Quantifier à l’aide d’un colorant de liaison à l’ADN et d’une machine de PCR quantitative (qPCR)20. Chargez des échantillons d’ADNc dans une plaque qPCR de 384 puits avec des amorces pour amplifier les gènes d’intérêt. Le volume de réaction devrait atteindre 10 μL, y compris le colorant de liaison à l’ADN, qui sera utilisé pour quantifier l’amplification des gènes.
- Utilisez CD31, VE-Cadhérin, CD45 et β-actine avant et arrière amorces pour mesurer l’expression génique.
- Utiliser l’exemple pour les applications de séquençage de nouvelle génération1.
Résultats
La digestion du tissu rétinien et l’immunomarquage pour CD31 et CD45 donnent lieu à une population identifiable de cellules endothéliales CD31+/CD45- après vérification des cellules, des cellules individuelles et de la viabilité (figure 2A). L’immunocoloration CD45 est nécessaire pour éliminer les cellules CD31+/CD45+, qui comprennent les plaquettes et certains leucocytes21. Des contrôles doivent être effectués pour chaque expérience afin de montrer la...
Discussion
Ce protocole décrit une méthode d’isolement des cellules endothéliales à partir du tissu rétinien murin postnatal qui a été optimisée pour un nombre élevé de cellules, une pureté et une viabilité. La pureté cellulaire est obtenue par isolement FACS de populations de cellules endothéliales à partir de la suspension unicellulaire digérée par immunomarquage CD31+/CD45-. La qualité de l’isolement est quantifiée dans les essais de viabilité par coloration au bleu de trypan et expression génique par qP...
Déclarations de divulgation
Les auteurs n’ont pas de divulgations pertinentes.
Remerciements
Merci au Yale Flow Cytometry Facility, au Flow Cytometry Core Facility de l’Université de Virginie, au Yale Center for Genomic Analysis et au Genome Analysis and Technology Core de l’Université de Virginie pour leurs efforts, leur expertise et leurs conseils dans la contribution aux expériences présentées. Cette étude a été financée par des subventions des NIH à N.W.C. (T32 HL007224, T32 HL007284), S.C. (T32 HL007284), K.W. (R01 HL142650) et K.K.H. (R01 HL146056, R01 DK118728, UH3 EB025765).
matériels
| Name | Company | Catalog Number | Comments |
| 2 mL Eppendorf safe-lock tubes | USA Scientific | 4036-3352 | |
| 5 ml Falcon Test Tubes with Cell Strainer Snap Cap | Corning | 352235 | |
| 60 mm Non TC-treated Culture Dish | Corning | 430589 | |
| APC Rat Anti-Mouse CD31 | BD Biosciences | 551262 | |
| APC Rat IgG2a κ Isotype Control | BD Biosciences | 553932 | |
| BD FACSChorus Software | BD Biosciences | FACSCHORUS | |
| BD FACSMelody Cell Sorter | BD Biosciences | FACSMELODY | |
| Collagenase Type II | Sigma-Aldrich | 234115 | |
| Costar 48-well Clear TC-treated Multiple Well Plates, Individually Wrapped, Sterile | Corning | 3548 | |
| D-Glucose | Gibco | A2494001 | |
| Disposable Graduated Transfer Pipettes | Fisher Scientific | 12-711-9AM | |
| Dissecting Pan Wax | Carolina | 629100 | |
| Dissection scissors | Fine Science Tools | 14085-08 | |
| Dissection Stereo Microscope M165 FC | Leica | M165FC | |
| Dulbecco's Modified Eagle Medium (DMEM) | Gibco | 11965-052 | |
| Dulbecco’s Phosphate Buffered Saline (PBS) | Gibco | 14190144 | |
| Eppendorf Flex-Tubes Microcentrifuge Tubes 1.5 mL | Sigma-Aldrich | 22364120 | |
| Fetal Bovine Serum (FBS) | Gemini Bio | 100-106 | |
| Fine dissection forceps | Fine Science Tools | 11250-00 | |
| Hank's Buffered Salt Solution (HBSS) | Gibco | 14175095 | |
| HEPES (1M) | Gibco | 15630130 | |
| iScript cDNA Synthesis Kit | Bio-Rad | 1708890 | |
| Isoflurane, USP | Covetrus | 11695067772 | |
| Isotemp General Purpose Deluxe Water Bath | Fisher Scientific | FSGPD20 | |
| Primer: ActB_Forward: 5’- agagggaaatcgtgcgtgac -3’ | Integrated DNA Technologies | N/A | |
| Primer: ActB_Reverse: 5’- caatagtgatgacctggccgt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD31_Forward: 5’- gagcccaatcacgtttcagttt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD31_Reverse: 5’- tccttcctgcttcttgctagct -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD45_Forward: 5’- gggttgttctgtgccttgtt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD45_Reverse: 5’- ctggacggacacagttagca -3’ | Integrated DNA Technologies | N/A | |
| Primer: VE-Cadherin_Forward: 5’- tcctctgcatcctcactatcaca -3’ | Integrated DNA Technologies | N/A | |
| Primer: VE-Cadherin_Reverse: 5’- gtaagtgaccaactgctcgtgaat -3’ | Integrated DNA Technologies | N/A | |
| Propidium iodide | Sigma-Aldrich | P4864 | |
| RNeasy Plus Mini Kit | Qiagen | 74134 | |
| Sorvall Legend Micro 21R Centrifuge, Refrigerated | ThermoFisher | 75002477 | |
| SYBR-Green iTaq Universal SYBR Green Supermix | Bio-Rad | 172-5120 | |
| Trypan Blue Solution | ThermoFisher | 15250061 | |
| V450 Rat Anti-Mouse CD45 | BD Biosciences | 560501 | |
| V450 Rat IgG2b, κ Isotype Control | BD Biosciences | 560457 |
Références
- Slatko, B. E., Gardner, A. F., Ausubel, F. M. Overview of next-generation sequencing technologies. Current Protocols in Molecular Biology. 122 (1), 59 (2018).
- Chavkin, N. W., Hirschi, K. K. Single cell analysis in vascular biology. Frontiers in Cardiovascular Medicine. 7, 42 (2020).
- Ma, F., Hernandez, G. E., Romay, M., Iruela-Arispe, M. L. Single-cell RNA sequencing to study vascular diversity and function. Current Opinion in Hematology. 28 (3), 221-229 (2021).
- Potter, A. S., Potter, S. S., S, Dissociation of tissues for single-cell analysis. Methods in Molecular Biology. 1926, 55-62 (2019).
- Braga, F. A. V., Miragaia, R. J. Tissue handling and dissociation for single-cell RNA-seq. Single Cell Methods: Methods in Molecular Biology. 1979, 9-21 (2019).
- Brink, S. C., et al. Single-cell sequencing reveals dissociatin-induced gene expression in tissue subpopulations. Nature Methods. 14 (10), 935-936 (2017).
- Skulska, K., Wegrzyn, A. S., Chelmonska-Soyta, A., Chodaczek, G. Impact of tissue enzymatic digestion on analysis of immune cells in mouse reproductive mucosa with a focus on gammadelta T cells. Journal of Immunological Methods. 474, 112665 (2019).
- Connolly, S., Hores, T., Smith, L., D'Amore, P. Characterization of vascular development in the mouse retina. Microvascular Research. 36 (3), 275-290 (1988).
- Crist, A., Young, C., Meadows, S. Characterization of arteriovenous identity in the developing neonate mouse retina. Gene Expression Patterns: GEP. 23-24, 22-31 (2017).
- dela Paz, N. G., D'Amore, P. A. Arterial versus venous endothelial cells. Cell and Tissue Research. 335 (1), 5-16 (2009).
- Fang, J. S., Hirschi, K. K. Molecular regulation of arteriovenous endothelial cell specification. F1000Res. 8, (2019).
- Smith, L. E., et al. Oxygen-induced retinopathy in the mouse. Investigative Ophthalmology and Visual Science. 35 (1), 101-111 (1994).
- Pitulescu, M. E., Schmidt, I., Benedito, R., Adams, R. H. Inducible gene targeting in the neonatal vasculature and analysis of retinal angiogenesis in mice. Nature Protocols. 5 (9), 1518-1534 (2010).
- Ruiz, S., et al. A mouse model of hereditary hemorrhagic telangiectasia generated by transmammary-delivered immunoblocking of BMP9 and BMP10. Scientific Reports. 6, 37366 (2016).
- Fang, J. S., et al. Shear-induced Notch-Cx37-p27 axis arrests endothelial cell cycle to enable arterial specification. Nature Communication. 8 (1), 2149 (2017).
- Ola, R., et al. SMAD4 prevents flow induced arterial-venous malformations by inhibiting Casein Kinase 2. Circulation. 138 (21), 2379-2394 (2018).
- Chavkin, N. W., Walsh, K., Hirschi, K. K. Isolation of highly purified and viable retinal endothelial cells. Journal of Vascular Research. 58 (1), 49-57 (2020).
- Hulspas, R., O'Gorman, M. R. G., Wood, B. L., Gratama, J. W., Sutherland, D. R. Considerations for the control of background fluorescence in clinical flow cytometry. Cytometry PartB: Clinical Cytometry. 76 (6), 355-364 (2009).
- Bachman, J. Reverse-transcription PCR (RT-PCR). Methods in Enzymology. 530, 67-74 (2013).
- Green, M. R., Sambrook, J. Quantification of RNA by real-time Reverse Transcription-Polymerase Chain Reaction (RT-PCR). Cold Spring Harbour Protocols. 2018 (10), (2018).
- Liu, L., Shi, G. P. CD31: beyond a marker for endothelial cells. Cardiovascular Research. 94 (1), 3-5 (2012).
- Zarkada, G., et al. Specialized endothelial tip cells guide neuroretina vascularization and blood-retina-barrier formation. Developmental Cell. 56 (15), 2237-2251 (2021).
- Su, X., Sorenson, C. M., Sheibani, N. Isolation and characterization of murine retinal endothelial cells. Molecular Vision. 9, 171-178 (2003).
- Benedito, R., et al. The notch ligands Dll4 and Jagged1 have opposing effects on angiogenesis. Cell. 137 (6), 1124-1135 (2009).
- Daneman, R., et al. Wnt/beta-catenin signaling is required for CNS, but not non-CNS, angiogenesis. Proceedings of the National Academy of Sciences of the United States of America. 106 (2), 641-646 (2009).
- Okabe, K., et al. Neurons limit angiogenesis by titrating VEGF in retina. Cell. 159 (3), 584-596 (2014).
- Crist, A. M., Lee, A. R., Patel, N. R., Westhoff, D. E., Meadows, S. M. Vascular deficiency of Smad4 causes arteriovenous malformations: a mouse model of Hereditary Hemorrhagic Telangiectasia. Angiogenesis. 21 (2), 363-380 (2018).
- Kim, Y. H., Choe, S. W., Chae, M. Y., Hong, S., Oh, S. P. SMAD4 deficiency leads to development of arteriovenous malformations in neonatal and adult mice. Journal of the American Heart Association. 7 (21), 009514 (2018).
- Ma, W., et al. Absence of TGFbeta signaling in retinal microglia induces retinal degeneration and exacerbates choroidal neovascularization. eLife. 8, 42049 (2019).
- Luo, W., et al. Arterialization requires the timely suppression of cell growth. Nature. 589 (7842), 437-441 (2021).
- Lawson, N. D., Vogel, A. M., Weinstein, B. M. sonic hedgehog and vascular endothelial growth factor act upstream of the Notch pathway during arterial endothelial differentiation. Developmental Cell. 3 (1), 127-136 (2002).
- Larrivee, B., et al. ALK1 signaling inhibits angiogenesis by cooperating with the Notch pathway. Developmental Cell. 22 (3), 489-500 (2012).
- Wythe, J. D., et al. ETS factors regulate Vegf-dependent arterial specification. Developmental Cell. 26 (1), 45-58 (2013).
- Davis, D. M., Purvis, J. E. Computational analysis of signaling patterns in single cells. Seminars in Cell and Developmental Biology. , 35-43 (2015).
- Gaudet, S., Miller-Jensen, K. Redefining signaling pathways with an expanding single-cell toolbox. Trends in Biotechnology. 34 (6), 458-469 (2016).
- Aibar, S., et al. SCENIC: single-cell regulatory network inference and clustering. Nature Methods. 14 (11), 1083-1086 (2017).
- Trapnell, C., et al. The dynamics and regulators of cell fate decisions are revealed by pseudotemporal ordering of single cells. Nature Biotechnology. 32 (4), 381-386 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon