Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Extracción y purificación de la proteína FAHD1 del riñón porcino y el hígado de ratón
En este artículo
Resumen
Este protocolo describe cómo extraer la proteína 1 que contiene el dominio fumarilacetato hidrolasa (FAHD1) del riñón porcino y el hígado de ratón. Los métodos enumerados pueden adaptarse a otras proteínas de interés y modificarse para otros tejidos.
Resumen
La proteína 1 que contiene el dominio de la fumarilacetoacetato hidrolasa (FAHD1) es el primer miembro identificado de la superfamilia FAH en eucariotas, actuando como oxaloacetato descarboxilasa en las mitocondrias. Este artículo presenta una serie de métodos para la extracción y purificación de FAHD1 a partir de riñón porcino e hígado de ratón. Los métodos cubiertos son la cromatografía de intercambio iónico con cromatografía líquida rápida de proteínas (FPLC), la filtración en gel preparativa y analítica con FPLC y los enfoques proteómicos. Después de la extracción total de proteínas, se exploró la precipitación de sulfato de amonio y la cromatografía de intercambio iónico, y se extrajo FAHD1 a través de una estrategia secuencial utilizando intercambio iónico y cromatografía de exclusión de tamaño. Este enfoque representativo puede adaptarse a otras proteínas de interés (expresadas a niveles significativos) y modificarse para otros tejidos. La proteína purificada del tejido puede apoyar el desarrollo de anticuerpos de alta calidad y/o inhibidores farmacológicos potentes y específicos.
Introducción
La proteína 1 que contiene el dominio faH eucariota (FAHD1) actúa como oxaloacetato bifuncional (OAA) descarboxilasa (ODx)1 y acilpiruvato hidrolasa (ApH)2. Se localiza en las mitocondrias2 y pertenece a la amplia superfamilia faH de enzimas 1,2,3,4,5,6. Mientras que su actividad apH es solo de menor relevancia, la actividad ODx de FAHD1 está involucrada en la regulación del flujo del ciclo TCA 1,7,8,9. La OAA no solo es necesaria para la reacción de la citrato sintasa central en el ciclo del ácido tricarboxílico, sino que también actúa como un inhibidor competitivo de la succinato deshidrogenasa como parte del sistema de transporte de electrones y como metabolito cataplerótico. La regulación a la baja de la expresión del gen FAHD1 en células endoteliales de la vena umbilical humana (HUVEC) resultó en una reducción significativa en la tasa de proliferación celular10, y una inhibición significativa del potencial de membrana mitocondrial, asociada con un cambio concomitante a la glucólisis. El modelo de trabajo se refiere a la disfunción mitocondrial asociada a la senescencia (MiDAS)11-como el fenotipo8, donde los niveles de OAA mitocondrial están estrechamente regulados por la actividad de FAHD1 1,8,9.
La proteína recombinante es más fácil de obtener a través de la expresión y purificación de la bacteria12 en lugar de a partir del tejido. Sin embargo, una proteína expresada en bacterias puede estar sesgada por la posible falta de modificaciones post-traduccionales, o simplemente puede ser problemática (es decir, debido a la pérdida de plásmidos, respuestas al estrés bacteriano, enlaces disulfuro distorsionados / no formados, secreción nula o deficiente, agregación de proteínas, escisión proteolítica, etc.). Para ciertas aplicaciones, la proteína debe obtenerse a partir de lisado celular o tejido, con el fin de incluir tales modificaciones y / o excluir posibles artefactos. La proteína purificada del tejido apoya el desarrollo de anticuerpos de alta calidad y/o inhibidores farmacológicos potentes y específicos para enzimas seleccionadas, como para FAHD113.
Este manuscrito presenta una serie de métodos para la extracción y purificación de FAHD1 a partir de riñón porcino e hígado de ratón. Los métodos descritos requieren cromatografía líquida rápida de proteínas (FPLC), pero por lo demás utilizan equipos de laboratorio comunes. Los métodos alternativos se pueden encontrar en otros lugares 14,15,16,17. Después de la extracción total de proteínas, el protocolo propuesto implica una fase de prueba, en la que se discuten los subprotocolos para la precipitación de sulfato de amonio y la cromatografía de intercambio iónico (Figura 1). Después de definir estos subprotos protocolos, la proteína de interés se extrae a través de una estrategia secuencial utilizando intercambio iónico y cromatografía de exclusión de tamaño con FPLC. Sobre la base de estas directrices, el protocolo final puede adaptarse individualmente para otras proteínas de interés.
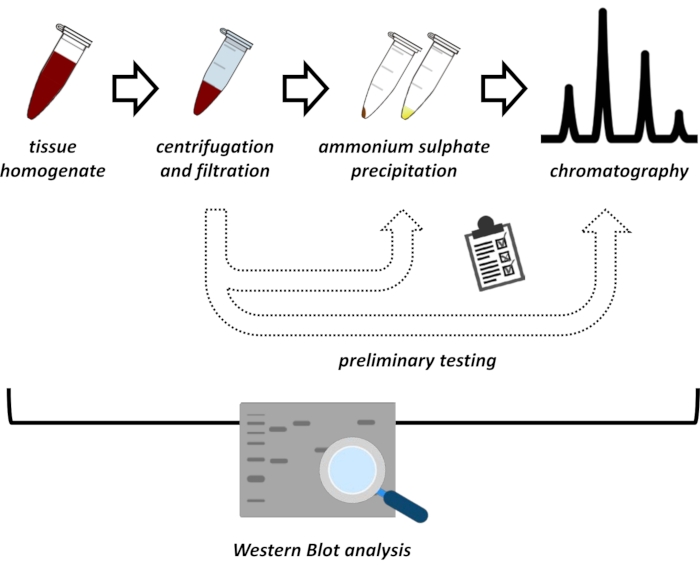
Figura 1: La estrategia general de este protocolo. De arriba a abajo: La proteína se extrae de los tejidos. El homogeneizado tisular se prepara, centrifuga y filtra. Para cada par de muestras sobrenadantes y derivadas de pellets, se deben realizar pruebas de precipitación de sulfato de amonio y cromatografía de intercambio iónico (FPLC) para sondear las condiciones óptimas. Después de establecer estos subprotos protocolos, la proteína se puede extraer a través de un procedimiento secuencial de precipitación de sulfato de amonio, cromatografía de intercambio iónico y cromatografía de exclusión de tamaño repetitivo (FPLC) a diferentes concentraciones de pH y sal. Todos los pasos deben ser controlados por Western Blot. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
Todos los experimentos se realizaron de conformidad con las directrices institucionales. El riñón porcino se obtuvo fresco del supermercado local. Los tejidos hepáticos se cosecharon de ratones de tipo salvaje C57BL6 mantenidos en el Instituto de Investigación del Envejecimiento Biomédico de la Universidad de Innsbruck, Rennweg 10, 6020 Innsbruck, Austria bajo la supervisión de Univ.-Doz. Dr. Pidder Jansen-Dürr, cubierto por un permiso ético como líder de proyecto emitido en 2013 (BMWF-66.008/0007-II/3b/2013). El mantenimiento y el uso de los ratones para el proyecto están cubiertos por el permiso ético No. 2020-0.242.978 del 5 de mayo de 2020, emitido por el Ministerio de Educación, Ciencia e Investigación de Austria (BMBWF).
1. Preparativos
NOTA: Antes de que comience el protocolo, se deben preparar varias cosas, es decir, el tampón de lisis de proteínas, la muestra de tejido crudo y un anticuerpo específico, además de productos químicos generales y materiales.
- Preparar 250 mL de tampón de lisis proteica por cada 100 g de peso neto de tejido: 250 mL de 1x PBS con 50 mM NaF, 1 mM PMSF, 2 μg/mL de aprotinina y 1 mM de ortovanadato activado (ver Tabla 1). Filtre la solución con una unidad de filtro de jeringa de 0,22 μm.
NOTA: Se requiere la activación del ortovanadato antes de su uso para convertirlo en un inhibidor más potente de la proteína tirosina fosfatasas18. El ortovanadato activado se puede obtener de proveedores comerciales, pero también se puede preparar de la siguiente manera.- Preparar una solución madre de 200 mM de ortovanadato (de sodio) en ddH2O. Para preparar 10 ml de solución, agregue 368 mg de Na3VO4 a 9 ml de agua y disuelva agitando. Una vez disuelto, enrasar el volumen hasta 10 mL con ddH2O.
NOTA: El pH inicial de la solución de ortovanadato de sodio puede variar con la fuente del material, y el pH debe ajustarse a 10 en un enfoque repetitivo de la siguiente manera. - Dependiendo del pH inicial de la solución, ajuste el pH a 10 con NaOH o HCl. A pH > 10, la solución tendrá un color amarillo. Hervir la solución hasta que se vuelva incolora, enfriarla a temperatura ambiente y verificar el pH. Si el pH es >10, agregue un pequeño volumen de HCl para ajustar el pH a 10. En este punto, la solución puede volverse amarilla nuevamente.
- Repita la ebullición y el enfriamiento hasta que la solución permanezca incolora y el pH se estabilice en 10 (aproximadamente 5-7 veces). En este punto, la adición de HCl da como resultado una apariencia débil de color amarillo en la solución. Conservar el ortovanadato activado en alícuotas de 1 ml a -20 °C.
- Preparar una solución madre de 200 mM de ortovanadato (de sodio) en ddH2O. Para preparar 10 ml de solución, agregue 368 mg de Na3VO4 a 9 ml de agua y disuelva agitando. Una vez disuelto, enrasar el volumen hasta 10 mL con ddH2O.
- Prepare tubos con 2 ml de tampón de lisis por gramo de tejido y colóquelos sobre hielo.
NOTA: Este protocolo utilizó ocho tubos de 50 ml, cada uno lleno de 30 ml de tampón de lisis en total para un riñón porcino (aproximadamente 100-150 g), y dos tubos cada uno lleno de 40 ml de tampón de lisis para 20 hígados de ratón (cada uno de 1-2 g) en total. - Prepare el tejido: diseccione el tejido en una placa de vidrio prelimpiada colocada sobre hielo en una caja de espuma de poliestireno. Corte trozos de tejido de aproximadamente 100 mg cada uno para ser transferidos fácilmente a los tubos respectivos para la lisis posterior. Transfiera las piezas de tejido a los tubos preparados (paso 1.2).
- Prepare una solución saturada de sulfato de amonio: caliente 500 ml de ddH2O a 70 °C y, mientras agita, agregue gradualmente polvo de sulfato de amonio (consulte la Tabla de materiales) hasta que no se disuelva más sulfato de amonio. Enfríe esta solución (sobre)saturada a temperatura ambiente y guárdela a 4 °C durante la noche.
2. Extracción total de proteínas
NOTA Después de preparar la muestra en tampón de lisis de proteínas frías (consulte el paso 1.3), homogeneice el tejido lo mejor posible mediante sonicación mediante una sonda ultrasónica o utilizando un homogeneizador eléctrico de la siguiente manera.
- Homogeneización de tejidos
- En el caso de un riñón porcino, sonicar la suspensión preferiblemente mediante una sonda ultrasónica manteniendo la muestra en hielo (10 ciclos de pulso de 15 s, con intervalos de 30 s entre los pulsos para enfriar la muestra sobre hielo, a amplitud media con ciclo de trabajo del 50%).
- En el caso de los órganos de ratón, homogeneizar la suspensión utilizando un homogeneizador eléctrico (comenzando con una fuerza baja y acelerando lentamente a fuerza media) manteniendo la muestra en hielo. Lave regularmente el homogeneizador eléctrico en PBS para eliminar cualquier material orgánico que obstruya el dispositivo.
- Saque 20 μL de las muestras y compruebe bajo el microscopio si las células del tejido homogeneizado se destruyen adecuadamente; de lo contrario, repita la homogeneización.
- Centrifugar los tubos en una centrífuga de mesa a 10.000 x g durante 30 min a 4 °C.
NOTA: Opcionalmente, centrifugar el sobrenadante por segunda vez a 20.000 x g durante 30 min a 4 °C para eliminar pequeñas fracciones del pellet inicial que puedan haber sido transferidas. Esto simplificará la filtración posterior en el paso 2.3. - Recoge el sobrenadante en un tubo fresco y colócalo sobre hielo. Filtre secuencialmente el sobrenadante utilizando unidades de filtro de jeringa de 0,45 μm y 0,22 μm. Alícuota el sobrenadante en lotes de 10 ml y congélelos a -20 °C para un almacenamiento a corto plazo o a -80 °C para un almacenamiento más largo.
NOTA: El filtrado previo con 0,45 μm elimina la mayoría de las partículas antes de que un segundo paso de filtrado con 0,22 μm elimine las partículas más finas. El uso del filtro de 0,22 μm directamente puede causar el riesgo de obstrucción de los filtros. - Prepare una muestra de 50 μL para el análisis SDS-PAGE/western blot agregando 10 μL de tampón de muestra SDS 5x (consulte la Tabla 1) a 40 μL del sobrenadante, y luego hirviendo a 95 °C durante 10 min.
- Opcionalmente, resuspenda aproximadamente 100 μL de pellet obtenido en el paso 2.2 en 900 μL de ddH2O, y prepare una muestra para el análisis SDS-PAGE/western blot como se describió anteriormente.
NOTA: La inclusión de muestras derivadas de pellets en el análisis de western blot, además del control positivo, indicará si la expresión de la proteína es baja o si el anticuerpo es problemático.
- Opcionalmente, resuspenda aproximadamente 100 μL de pellet obtenido en el paso 2.2 en 900 μL de ddH2O, y prepare una muestra para el análisis SDS-PAGE/western blot como se describió anteriormente.
3. SDS-PAGE y análisis de western blot
NOTA: Se requiere el análisis de Western blot para verificar la solubilidad de la proteína. A continuación se describe un protocolo para la electroblotting, utilizando un sistema de borrado húmedo/tanque (consulte la Tabla de materiales). Un protocolo alternativo para SDS-PAGE se puede encontrar en otra parte19.
- Prepare un gel discontinuo de poliacrilamida SDS-PAGE al 12,5% de acuerdo con las instrucciones del fabricante (es decir, un gel de apilamiento encima de un gel de resolución; consulte la Tabla 1). Ejecute los ejemplos preparados previamente durante el paso 2 (similar a los pasos 4, 5 y 6; consulte a continuación).
- Cargue una escalera de marcadores de proteínas en el primer pozo (ver Tabla de Materiales). Carga 5 ng de proteína recombinante hFAHD1 (obtenida de la bacteria12; ver Tabla 1) como control positivo en el segundo pozo.
- Posteriormente, cargue 20 μL de la muestra a analizar y llene todos los pozos restantes con 20 μL de tampón de muestra SDS-PAGE 1x preparado (es decir, 5x tampón de muestra diluido con ddH2O). Ejecute los geles SDS-PAGE a 125 V utilizando el búfer de ejecución SDS (consulte la Tabla 1).
- Una vez que se complete SDS-PAGE, realice un análisis de western blot y sondee las membranas utilizando el anticuerpo disponible contra FAHD1 (ver Tabla 1).
NOTA: Como las muestras se toman de homogeneizado de tejido crudo, generalmente la calidad del SDS-PAGE y el análisis de western blot en este punto se ve comprometida; sin embargo, es importante comprobar si la proteína a extraer es soluble en el sobrenadante. El siguiente protocolo se probó para el riñón porcino y diferentes órganos de ratón, incluidos el hígado, el corazón, el cerebro y el riñón.- Prepare el búfer de transferencia de western blot 10x (consulte la Tabla 1). Prepare el búfer de transferencia de western blot 1x (consulte la Tabla 1) y enfríelo a 4 °C.
- Activar una membrana de PVDF durante 2 min en metanol. Lavar la membrana en ddH2O durante 2 min. Equilibre la membrana durante 15 min en 1x western blot transfer buffer.
- Lave el SDS-gel con 1x PBS durante 10 minutos mientras agita para eliminar el tampón de funcionamiento de SDS, y luego incube el gel en 1x búfer de transferencia de western blot durante 10 min para el equilibrio. Ensamble el cassette electroblotting (es decir, combinando la membrana de PVDF activada y los geles) de acuerdo con las instrucciones del fabricante.
- Ejecute la mancha a través de electroblotting a 300 mA durante 1 h en una caja de espuma de poliestireno llena de hielo o en la cámara frigorífica (4 °C). Transfiera la membrana de PVDF a un tubo de 50 ml con su lado expuesto mirando hacia el lado interno del tubo. Incubar la membrana en 20 ml de tampón de bloqueo de western blot (ver Tabla 1) durante la noche a 4 °C mientras roda sobre un rodillo de tubo (ver Tabla de Materiales).
- Al día siguiente, lave la membrana durante 5 min con 20 ml de tampón de lavado de western blot (PBS con 0.1% (v / v) Tween 20) en el mismo tubo mientras rueda. Incubar la membrana en el mismo tubo con el anticuerpo primario2 (dirigido a FAHD1; ver Tabla 1) diluido 1:500 en el tampón de bloqueo de western blot durante 1 h a temperatura ambiente mientras se enrolla.
- Lave la membrana en el mismo tubo tres veces durante 10 minutos cada una con 20 ml de tampón de lavado de western blot mientras se enrolla. Incubar la membrana durante 30 min a temperatura ambiente con anticuerpo secundario conjugado con HRP (ver Tabla de Materiales) diluido 1:3000 en 5 mL de tampón de bloqueo de western blot.
- Lave la membrana en el mismo tubo tres veces durante 10 minutos cada una con 20 ml de tampón de lavado de western blot y dos veces durante 5 minutos cada una con 1x PBS. Seque la membrana sosteniéndola cuidadosamente con pinzas en un borde y tocando un trozo de celulosa o un trozo de papel Whatman con el borde opuesto (inferior) de la membrana. Coloque la membrana (lado expuesto hacia arriba) en una placa de vidrio limpia.
- Cubra cuidadosamente toda la membrana con 1 ml de sustrato ECL western blot preparado con una pipeta, teniendo cuidado de no crear burbujas de aire. Deje que la solución de ECL se incube durante 3 minutos e inmediatamente desarrolle la membrana utilizando una película de rayos X o utilizando un sistema de imágenes.
NOTA: Si la proteína no se detectó en ninguna de las muestras, sino solo en el control positivo, esto puede indicar que la proteína es insoluble o no está presente en cantidades adecuadas para ser detectada por el anticuerpo. Si solo se cargaran nanogramos del control positivo, el primer escenario es más probable. Si no se detectó ninguna proteína, verifique la calidad del anticuerpo y tal vez cambie a un anticuerpo policlonal en lugar de un anticuerpo monoclonal. En raras ocasiones, es decir, para algunas proteínas hidrofóbicas, la proteína puede ser detectable después de la centrifugación, pero no después de la filtración. En tal caso, se recomienda utilizar unidades de filtro especiales para proteínas hidrofóbicas.
- Opcionalmente, tiñe las membranas de PVDF después de western blot para controlar la transferencia exitosa de la proteína del gel SDS-PAGE a la membrana de PVDF.
NOTA: La tinción de coomassie se recomienda para la solución de problemas, el desarrollo de métodos y la documentación, pero tenga en cuenta que después de aplicar este protocolo, las membranas se pierden para un análisis posterior de Western Blot. La tinción de Ponceau S da una tinción más débil, pero se puede usar si se van a volver a sondear las membranas.- Prepare pequeñas bandejas que contengan las soluciones de tinción (Coomassie o Ponceau S) y desmanchas.
- Usando pinzas, coloque la membrana en la solución de tinción y agite suavemente hasta que la membrana se tiñe bien (5-10 min).
- Transfiera la membrana a la solución de retención y agite hasta que la solución esté saturada (5-10 min). Repita el paso de detención hasta que se puedan observar las bandas de proteínas en la membrana; si no se observan bandas, repita la tinción con un tiempo de incubación más largo. Seque la membrana colocándola en una placa de vidrio con pinzas.
4. Pruebas: Precipitación de sulfato de amonio
NOTA: La precipitación de sulfato de amonio es un método de purificación de proteínas al alterar la solubilidad de la proteína. En un experimento preliminar, la concentración de sulfato de amonio se incrementa secuencialmente a un valor que precipita una cantidad máxima de contaminantes proteicos, mientras se deja FAHD1 en solución. La solubilidad de la proteína se investiga nuevamente a través del análisis de western blot.
- Proceda a partir de la etapa 2.3: descongele una alícuota de la muestra o proceda directamente después de la extracción de proteínas (es decir, sin congelar la muestra). Filtre la muestra utilizando una unidad de filtro de 0,22 μm para excluir posibles precipitados después de la descongelación. Prepare seis tubos de 1,5 ml sobre hielo y transfiera 250 μL de muestra a cada tubo.
- Prepare una serie de dilución de 5%, 10%, 15%, 20%, 25% y 30% de sulfato de amonio en los tubos preparados anteriormente, y componga el volumen final a 1000 μL con tampón de lisis de proteínas. Incubar las muestras a 4 °C durante la noche en un rotador de tubo (ver Tabla de Materiales).
- Usando una centrífuga de mesa, centrífuga a 10,000 x g durante 30 min a 4 ° C y transfiera cuidadosamente todos los sobrenadantes en tubos separados. Seque al aire los gránulos resultantes y vuelva a suspender cada uno de ellos en 1000 μL de ddH2O.
- Para cada par de pellets y sobrenadantes resuspendidos del paso anterior, mezcle 40 μL con 10 μL de tampón de muestra 5x SDS y hierva a 95 ° C con tapas abiertas hasta que la mayor parte del líquido se haya vaporizado. Luego, vuelva a suspender el pellet en una mezcla de 50% DMSO en ddH2O.
- Realice SDS-PAGE (paso 3) pero ejecute los geles a 80 V durante 3 h. Para cada concentración de sulfato de amonio, cargue las muestras derivadas del pellet resuspendido y el sobrenadante (paso 4.3) en pares. Realice un análisis de western blot (paso 3).
- Compruebe si la concentración más alta de sulfato de amonio, a la que la proteína a purificar (es decir, FAHD1) permanece en la muestra derivada del sobrenadante. En base a los resultados, definir un protocolo de precipitación de sulfato de amonio para la proteína de interés, para ser utilizado en futuros experimentos.
NOTA: El sulfato de amonio es bien conocido por distorsionar SDS-PAGE y western blot. A medida que aumenta la concentración de sulfato de amonio, la calidad del análisis de western blot se verá comprometida. Sin embargo, al igual que con el paso 3 anterior, este análisis se utiliza para verificar la solubilidad de la proteína de interés a concentraciones dadas de sulfato de amonio. Este protocolo tiene como objetivo precipitar otras proteínas, mientras que la proteína a purificar debe permanecer soluble.
5. Pruebas: cromatografía de intercambio iónico con FPLC
NOTA: Las moléculas con grupos funcionales cargados se unen a una columna de partículas de sílice para FPLC, lo que permite la diferenciación de proteínas de acuerdo con su carga superficial. Realice este paso dos veces, utilizando la columna de intercambio catiónico y la columna de intercambio aniónico (consulte tabla de materiales). Los pasos del protocolo son los mismos para la cromatografía de intercambio catiónico o aniónico, pero los tampones que se utilizarán son diferentes (véase la Tabla 1); ambos con condiciones de "bajo contenido de sal" 15 mM NaCl y "alto contenido de sal" de 1 M NaCl. Para las columnas utilizadas, se recomienda un caudal de 1 ml/min.
- Configure el sistema FPLC con la columna de intercambio aniónico o catiónico. Lave la columna con 5 volúmenes de columna (CV) de 20% de EtOH (en H2O), seguido de 5 CV de ddH2O. Alternativamente, lave la columna con 1 CV de tampón de sal baja, tampón de sal alta y nuevamente tampón de sal baja en el orden hasta que no se observen más picos en el cromatograma, pero lave al menos una vez.
- Después de determinar el protocolo óptimo para la precipitación de sulfato de amonio a pequeña escala (paso 4), aplique el protocolo de precipitación a 10 ml de homogeneizado de tejido original (paso 2). Opcionalmente, dialice la muestra contra el tampón bajo en sal.
- Aplique la muestra sobre la columna (por ejemplo, mediante inyección o mediante el uso de una bomba de muestra) y recoja el flujo. Lave la columna con 1 CV del tampón bajo en sal.
- Configure una elución de gradiente lineal de 100% de tampón de sal baja / tampón de sal alta al 0% de tampón de sal baja / tampón de sal 100% alto dentro de 3 CV. Recolecte continuamente fracciones de 1 ml. Una vez finalizado el gradiente, continúe funcionando con el tampón de sal alta hasta que no se detecten más picos asociados a proteínas (absorción UV a 280/255 nm) en el cromatograma en el rango de 1 CV.
- Aplicar 1 mL de SDS al 25% disuelto en 0,5 M NaOH (en ddH2O) para limpiar la columna. Consecutivamente, lavar la columna con 3 CV de ddH2O y 3 CV de 20% EtOH (en ddH2O).
- Recolectar muestras SDS-PAGE de todas las fracciones pico y el flujo a través, y sondearlas a través de Western Blot para detectar la presencia de la proteína de interés (paso 3). Congele las fracciones recogidas en nitrógeno líquido y guárdelas a -80 °C.
- Una vez finalizado el análisis de western blot, descongele y agrupe las fracciones que contienen la proteína de interés y descarte las demás. Repita los pasos 5.1-5.5 con la columna alternativa (es decir, columna de intercambio catiónico o aniónico).
- Después de que ambas columnas hayan sido sondeadas, defina un protocolo FPLC para la proteína de interés, que se utilizará en futuros experimentos. Reduzca el volumen de la solución proteica utilizando unidades de filtro de ultracentrugación (10 kDa, consulte la Tabla de materiales) hasta 2 ml.
NOTA: Hay dos resultados esperados de esta serie de experimentos. O bien la proteína de interés se ha unido a una de las columnas, y la solución proteica ya es bastante pura después de la elución, o la proteína permanece en el flujo en ambos casos. En este último escenario, aunque la proteína está en el flujo, el efecto de limpieza de este paso aún podría ser significativo. En tal caso, como para FAHD1 en riñón porcino e hígado de ratón, este paso de intercambio iónico aún se realizará. Si ni la columna de intercambio catiónico o aniónico puede proporcionar un efecto de limpieza adecuado, se puede intentar modificar el pH del lisado y el tampón, y dializar la muestra contra el búfer en ejecución antes de la aplicación a FPLC.
6. Extracción de proteínas utilizando subprotocolos definidos para la precipitación de sulfato de amonio y FPLC
NOTA: Las partículas porosas en una columna de gel de sílice para FPLC (Ver Tabla de Materiales) permiten la diferenciación de proteínas según su radio hidrodinámico. Los pasos descritos deben realizarse con un sistema FPLC, utilizando cromatografía de exclusión de tamaño (SEC). Para la columna SEC utilizada (ver Tabla de Materiales), se recomienda un caudal de 0,3 mL/min.
- Prepare todos los materiales necesarios (consulte el paso 1) y extraiga la proteína total del tejido (consulte el paso 2). Realice una precipitación de sulfato de amonio con todo el homogeneizado tisular que no se utilizó para la prueba (ver paso 4). Para volúmenes más grandes, concentre el lisado utilizando unidades de filtro de ultracentrugación (10 kDa; consulte la Tabla de materiales) hasta un volumen más pequeño de 50 ml o menos.
- Realice un primer paso de purificación mediante cromatografía de intercambio iónico (ver paso 5).
- Prepare muestras para Western Blot, como se describe en los pasos anteriores. Realice análisis de western blot y agrupte todas las fracciones que contienen FAHD1 de la cromatografía de intercambio iónico.
- Reduzca el volumen de la solución proteica hasta 2 ml utilizando unidades de filtro de ultracentrugación (10 kDa). Filtre secuencialmente la solución con unidades de filtro de jeringa de 0,45 μm y 0,22 μm para eliminar cualquier microprecipitación.
- Equilibrar la columna SEC con 1 CV de búfer de ejecución SEC (ver Tabla 1), que contiene 1 mM TDT. Cargue la muestra en la columna y ejecute la cromatografía hasta que se eluyan todas las proteínas (1-2 CV).
- Recoger fracciones de 1 mL del flujo a través que corresponda a picos significativos en el cromatograma (absorción UV a 280/255 nm) y preparar muestras de 50 μL de cada fracción recogida para el análisis SDS-PAGE y western blot, como se describe en los pasos anteriores. Congele todas las fracciones con nitrógeno líquido y guárdelas a -80 °C.
- Lavar consecutivamente la columna SEC con 1 CV de ddH2O y 1 CV de 20% EtOH (en ddH2O). Realice análisis de western-Blot y agrupe todas las fracciones que contienen FAHD1. Reduzca el volumen de la solución proteica hasta 2 ml utilizando unidades de filtro de ultracentrugación (10 kDa, consulte la Tabla de materiales).
- Evalúe la concentración de proteínas utilizando un kit de ensayo comercial de BCA (consulte la Tabla de materiales).
NOTA: El pH y el contenido de sal de la fase móvil pueden influir en el perfil de elución de las proteínas globulares20. Las condiciones ácidas o básicas pueden dar lugar a picos menos definidos y un aumento de las interacciones proteína-matriz que conducen a la retención parcial de proteínas en la columna20. Este efecto puede ser explotado para una mayor purificación de proteínas. Una repetición del paso 6 con diferentes caudales, pH y concentraciones de sal puede mejorar la pureza de la proteína20.
7. Tinción de plata
NOTA: Se requiere un análisis de tinción de plata de los geles SDS-PAGE para verificar si hay contaminaciones de proteínas que pueden no verse con la tinción de Coomassie. El siguiente protocolo es una de las muchas versiones que se pueden encontrar en la literatura21. Realice todos los pasos de incubación agitando en una bandeja de vidrio limpia. Recoja todos los líquidos que contengan plata y formaldehído en un contenedor de desechos especial y deséchelos adecuadamente.
- Incubar los geles SDS-PAGE en solución fijadora de tinción de plata (ver Tabla 1) durante la noche en la cámara frigorífica. Incubar los geles en solución de incubación de tinción de plata (ver Tabla 1) durante 3 h a temperatura ambiente. Opcionalmente, agregue glutaraldehído (ver Tabla 1) para mejorar la detección de bandas débiles. Lave los geles cuatro veces en ddH2O durante 10 min cada uno.
- Incubar los geles en solución de plata tintineante de plata (ver Tabla 1) durante 1 h.
NOTA: Tenga en cuenta que a partir de ahora todos los líquidos y el gel en sí contienen plata y formaldehído que son tóxicos. - Incubar los geles en solución reveladora de tinción de plata (ver Tabla 1) con agitación vigorosa hasta que las bandas sean claramente visibles. Para detener la reacción, deseche la solución reveladora e incube inmediatamente los geles en solución de parada de tinción de plata (ver Tabla 1) durante un mínimo de 10 min.
NOTA: Las bandas teñidas en los pasos 7.2 y 7.3 se desarrollarán constantemente. Agregar más formaldehído a la solución de lo indicado puede ser necesario si la tinción es débil.
Resultados
La proteína FAHD1 se extrajo del riñón porcino y del hígado de ratón utilizando el protocolo presentado. Para el tejido de ratón, se requieren múltiples órganos para obtener varios μg después de la etapa final de purificación. Por esta razón, este artículo se centra en la extracción de FAHD1 de los riñones porcinos, que es un experimento mucho más ejemplar. La extracción de FAHD1 del hígado de ratón se realiza para presentar las dificultades y posibles escollos de este protocolo. En general, se recomie...
Discusión
Pasos críticos en el protocolo
Seguir pautas comunes para el manejo de proteínas es esencial, como trabajar en hielo y en condiciones moderadas de pH y sal. El uso de inhibidores de la proteasa es beneficioso para el método, mientras que el uso de inhibidores del proteasoma es muy recomendable. La congelación y descongelación de la muestra siempre puede dar lugar a la precipitación de proteínas (al menos parcialmente), por lo que cualquier alícuota descongelada de lisado proteico inicial (paso...
Divulgaciones
Los autores no tienen intereses financieros contrapuestos.
Agradecimientos
Los autores están muy agradecidos por la asistencia técnica de Ayse Öztürk y Eva Albertini. Los ratones utilizados para la generación de tejido hepático se mantuvieron bajo la supervisión de la Univ.-Doz. Dr. Pidder Jansen-Dürr (Instituto de Investigación Biomédica sobre el Envejecimiento de la Universidad de Innsbruck, Rennweg 10, 6020 Innsbruck, Austria).
Materiales
| Name | Company | Catalog Number | Comments |
| 0.22 µm filter units | MERCK | SLGP033RS | Millex-HP, 0.22 µm, PES 33 mm, not steril |
| 0.45 µm filter units | MERCK | SLHP033NS | Millex-HP, 0.45 µm, PES 33 mm, not steril |
| 15 mL Falcon tubes | VWR | 734-0451 | centrifugal tubes |
| 50 mL Falcon tubes | VWR | 734-0448 | centrifugal tubes |
| 96-Well UV Microplate | Thermo-Fischer | 8404 | UV/VIS transparent flat-bottom 96 well plates |
| Acrylamide/Bis Solution (40%, 29:1 ratio) | BIO-RAD | #1610147 | 40% acrylamide/bis-acrylamide, 29:1 (3.3% crosslinker) solution for casting polyacrylamide gels |
| ÄKTA FPLC system | GE Healthcare Life Sciences / Cytiva | - | using the FPLC system by GE Healthcare; different custom versions exist; this work used the "ÄKTA pure" system |
| Amicon Ultra-15, PLGC Ultracel-PL Membran, 10 kDa | MERCK | UFC901024 | centrifigal filters for protein enrichment; 10 kDa molecular mass filter; 15 mL |
| Amicon Ultra-4, PLGC Ultracel-PL Membran, 10 kDa | MERCK | UFC801024 | centrifigal filters for protein enrichment; 10 kDa molecular mass filter; 4 mL |
| Ammonium sulfate powder | MERCK | A4418 | ammonium sulphate for molecular biology, ≥99.0% |
| Ammoniumpersulfat reagent grade, 98% | MERCK | 215589 | Catalyst for acrylamide gel polymerization. |
| Coomassie Brilliant blue R 250 | MERCK | 1125530025 | Coomassie Brilliant blue R 250 (C.I. 42660) for electrophoresis Trademark of Imperial Chemical Industries PLC. CAS 6104-59-2, pH 6.2 (10 g/l, H2O, 25 °C) |
| Dialysis tubing cellulose membrane | MERCK | D9277 | Cellulose membranes for the exchange of buffers via dialysis. |
| Eppendof tubes 1.5 mL | VWR | 525-1042 | microcentrifugal tubes; autoclaved |
| HiLoad 26/600 Superdex 75 pg | GE Healthcare Life Sciences / Cytiva | 28989334 | HiLoad Superdex 75 pg prepacked columns are for high-resolution size exclusion chromatography of recombinant proteins |
| Immun-Blot PVDF Membrane | BIO-RAD | #1620177 | PVDF membranes are protein blotting membranes optimized for fluorescent and multiplex fluorescent applications. |
| Mini Trans-Blot Electrophoretic Transfer Cell | BIO-RAD | #1703930 | Use the Mini Trans-Blot Cell for rapid blotting of Mini-PROTEAN precast and handcast gels. |
| Mini-PROTEAN Tetra Vertical Electrophoresis Cell for Mini Precast Gels | BIO-RAD | #1658004 | 4-gel vertical electrophoresis system, includes electrode assembly, companion running module, tank, lid with power cables, mini cell buffer dam. |
| Mono Q 10/100 GL | GE Healthcare Life Sciences / Cytiva | 17516701 | Mono Q columns are strong anion exchange chromatography columns for protein analysis or small scale, high resolution polishing of proteins. |
| Mono S 10/100 GL | GE Healthcare Life Sciences / Cytiva | 17516901 | Mono S columns are strong cation exchange chromatography columns for protein analysis or small scale high resolution polishing of proteins. |
| PageRuler Prestained Protein Ladder, 10 to 180 kDa | Thermo-Fischer | 26616 | A mixture of 10 blue-, orange-, and green-stained proteins (10 to 180 kDa) for use as size standards in protein electrophoresis (SDS-PAGE) and western blotting. |
| Pierce BCA Protein Assay Kit | Thermo-Fischer | 23225 | A two-component, high-precision, detergent-compatible protein assay for determination of protein concentration. |
| Sonifier 250; Ultrasonic Cell Disruptor w/ Converter | Branson | - | New models at https://www.emerson.com/documents/automation/brochure-sonifier-sfx250-sfx550-cell-disruptors-homogenizers-branson-en-us-168180.pdf |
| Swine Anti-Rabbit Immunoglobulins/HRP (affinity isolated) | Agilent Dako | P0399 | The antibody used for horseradish peroxidase conjugation reacts with rabbit immunoglobulins of all classes. |
| TEMED, 1,2-Bis(dimethylamino)ethane, TMEDA | MERCK | T9281 | TEMED (N,N,N′,N′-Tetramethylethylenediamine) is molecule which allows rapid polymerization of polyacrylamide gels. |
| Tube Roller | - | - | A general tube rotator roller; e.g. a new model at https://labstac.com/de/Mixer/Roller/c/71 |
| Tube Rotator | - | - | A general tube rotator wheel; e.g. a new model at https://labstac.com/de/Tube-Roller/p/MT123 |
| ULTRA-TURRAX; T 25 digital | IKA | 0003725000 | New models at https://www.ika.com/de/Produkte-Lab-Eq/Dispergierer-Dipergiergeraet-Homogenisierer-Homogenisator-csp-177/T-25-digital-ULTRA-TURRAX-cpdt-3725000/ |
Referencias
- Pircher, H., et al. Identification of FAH domain-containing protein 1 (FAHD1) as oxaloacetate decarboxylase. Journal of Biological Chemistry. 290 (11), 6755-6762 (2015).
- Pircher, H., et al. Identification of human Fumarylacetoacetate Hydrolase Domain-containing Protein 1 (FAHD1) as a novel mitochondrial acylpyruvase. Journal of Biological Chemistry. 286 (42), 36500-36508 (2011).
- Kang, T. -. W., et al. Senescence surveillance of pre-malignant hepatocytes limits liver cancer development. Nature. 479 (7374), 547-551 (2011).
- Hong, H., Seo, H., Park, W., Kim, K. K. -. J. Sequence, structure and function-based classification of the broadly conserved FAH superfamily reveals two distinct fumarylpyruvate hydrolase subfamilies. Environmental Microbiology. 22 (1), 270-285 (2020).
- Timm, D. E., Mueller, H. A., Bhanumoorthy, P., Harp, J. M., Bunick, G. J. Crystal structure and mechanism of a carbon-carbon bond hydrolase. Structure. 7 (9), 1023-1033 (1999).
- Bateman, R. L., et al. Mechanistic inferences from the crystal structure of Fumarylacetoacetate Hydrolase with a bound phosphorus-based inhibitor. Journal of Biological Chemistry. 276 (18), 15284-15291 (2001).
- Weiss, A. K. H., et al. Structural basis for the bi-functionality of human oxaloacetate decarboxylase FAHD1. Biochemical Journal. 475 (22), 3561-3576 (2018).
- Etemad, S., et al. Oxaloacetate decarboxylase FAHD1 - a new regulator of mitochondrial function and senescence. Mechanisms of Ageing and Development. 177, 22-29 (2019).
- Weiss, A. K. H., et al. Regulation of cellular senescence by eukaryotic members of the FAH superfamily - A role in calcium homeostasis. Mechanisms of Ageing and Development. 190, 111284 (2020).
- Petit, M., Koziel, R., Etemad, S., Pircher, H., Jansen-Dürr, P. Depletion of oxaloacetate decarboxylase FAHD1 inhibits mitochondrial electron transport and induces cellular senescence in human endothelial cells. Experimental Gerontology. 92, 7-12 (2017).
- Wiley, C. D., et al. Mitochondrial dysfunction induces senescence with a distinct secretory phenotype. Cell Metabolism. 23 (2), 303-314 (2016).
- Weiss, A. K. H., et al. Expression, purification, crystallization, and enzyme assays of Fumarylacetoacetate Hydrolase Domain-containing proteins. Journal of Visualized Experiments: JoVE. (148), e59729 (2019).
- Weiss, A. K. H., et al. Inhibitors of Fumarylacetoacetate Hydrolase Domain Containing Protein 1 (FAHD1). Molcules. 26 (16), 5009 (2021).
- Mizutani, H., Kunishima, N. Purification, crystallization and preliminary X-ray analysis of the fumarylacetoacetase family member TTHA0809 from Thermus thermophilus HB8. Acta Crystallographica Section F Structural Biology and Crystallization Communications. 63 (9), 792-794 (2007).
- Lee, C. H. A simple outline of methods for protein isolation and purification. Endocrinology and Metabolism. 32 (1), 18-22 (2017).
- Amer, H. E. A. Purification of proteins: Between meaning and different methods). Proteomics Technologies and Applications. , (2019).
- Niu, L., Yuan, H., Gong, F., Wu, X., Wang, W. Protein extraction methods shape much of the extracted proteomes. Frontiers in Plant Science. 9, 802 (2018).
- Gordon, J. A. Use of vanadate as protein-phosphotyrosine phosphatase inhibitor. Methods in Enzymology. 201, 477-482 (1991).
- Gallagher, S. R. SDS-polyacrylamide gel electrophoresis (SDS-PAGE). Current Protocols in Essential Laboratory Techniques. , (2012).
- . Effect of pH on Protein Size Exclusion Chromatography Available from: https://www.agilent.com/cs/library/applications/5990-8138EN.pdf (2011)
- Sørensen, B. K., et al. Silver staining of proteins on electroblotting membranes and intensification of silver staining of proteins separated by polyacrylamide gel electrophoresis. Analytical Biochemistry. 304 (1), 33-41 (2002).
- Fagerberg, L., et al. Analysis of the human tissue-specific expression by genome-wide integration of transcriptomics and antibody-based proteomics. Molecular & Cellular Proteomics. 13 (2), 397-406 (2014).
- . Cytiva Life Fundamentals of size exclusion chromatography Available from: https://www.cytivalifesciences.com/en/us/solutions/protein-research/knowledge-center/protein-purification-methods/size-exclusion-chromatography (2022)
- Rosano, G. L., Ceccarelli, E. A. Recombinant protein expression in Escherichia coli: advances and challenges. Frontiers in Microbiology. 5, 172 (2014).
- Rosano, G. L., Morales, E. S., Ceccarelli, E. A. New tools for recombinant protein production in Escherichia coli: A 5-year update. Protein Science: A Publication of the Protein Society. 28 (8), 1412-1422 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados