Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Extraction et purification de la protéine FAHD1 du rein de porc et du foie de souris
Dans cet article
Résumé
Ce protocole décrit comment extraire la protéine 1 contenant le domaine de l’hydrolase fumarylacétoacétate (FAHD1) du rein et du foie de souris. Les méthodes énumérées peuvent être adaptées à d’autres protéines d’intérêt et modifiées pour d’autres tissus.
Résumé
La protéine 1 contenant le domaine de l’hydrolase fumarylacétoacétate (FAHD1) est le premier membre identifié de la superfamille FAH chez les eucaryotes, agissant comme oxaloacétate décarboxylase dans les mitochondries. Cet article présente une série de méthodes pour l’extraction et la purification de FAHD1 à partir de reins de porcs et de foies de souris. Les méthodes couvertes sont la chromatographie par échange ionique avec chromatographie liquide à protéines rapides (FPLC), la filtration sur gel préparatif et analytique avec FPLC et les approches protéomiques. Après l’extraction des protéines totales, la précipitation du sulfate d’ammonium et la chromatographie par échange ionique ont été explorées, et FAHD1 a été extrait via une stratégie séquentielle utilisant la chromatographie par échange ionique et par exclusion de taille. Cette approche représentative peut être adaptée à d’autres protéines d’intérêt (exprimées à des niveaux significatifs) et modifiée pour d’autres tissus. Les protéines purifiées des tissus peuvent favoriser le développement d’anticorps de haute qualité et/ou d’inhibiteurs pharmacologiques puissants et spécifiques.
Introduction
La protéine 1 eucaryote contenant le domaine FAH (FAHD1) agit sous forme d’oxaloacétate bifonctionnel (OAA), decarboxylase (ODx)1 et d’hydrolase acylpyruvate (ApH)2. Il est localisé dans les mitochondries2 et appartient à la vaste superfamille faH des enzymes 1,2,3,4,5,6. Bien que son activité ApH ne soit que d’une pertinence mineure, l’activité ODx de FAHD1 est impliquée dans la régulation du flux de cycle TCA 1,7,8,9. L’OAA est non seulement nécessaire pour la réaction centrale de la citrate synthase dans le cycle de l’acide tricarboxylique, mais agit également comme un inhibiteur compétitif de la succinate déshydrogénase dans le cadre du système de transport d’électrons et comme métabolite cataplérotique. La régulation négative de l’expression du gène FAHD1 dans les cellules endothéliales de la veine ombilicale humaine (HUVEC) a entraîné une réduction significative du taux de prolifération cellulaire10 et une inhibition significative du potentiel de la membrane mitochondriale, associée à un passage concomitant à la glycolyse. Le modèle de travail fait référence à la sénescence associée au dysfonctionnement mitochondrial (MiDAS)phénotype 8 de type MiDAS,11, où les niveaux d’OAA mitochondrial sont étroitement régulés par l’activité FAHD1 1,8,9.
La protéine recombinante est plus facile à obtenir par l’expression et la purification à partir de bactéries12 plutôt que de tissus. Cependant, une protéine exprimée dans les bactéries peut être biaisée par l’absence possible de modifications post-traductionnelles, ou peut simplement être problématique (c.-à-d. en raison de la perte de plasmides, des réponses au stress bactérien, des liaisons disulfures déformées / non formées, de l’absence ou d’une mauvaise sécrétion, de l’agrégation des protéines, du clivage protéolytique, etc.). Pour certaines applications, la protéine doit être obtenue à partir de lysat cellulaire ou de tissu, afin d’inclure de telles modifications et/ou d’exclure d’éventuels artefacts. Les protéines purifiées des tissus favorisent le développement d’anticorps de haute qualité et/ou d’inhibiteurs pharmacologiques puissants et spécifiques pour certaines enzymes, comme pour FAHD113.
Ce manuscrit présente une série de méthodes pour l’extraction et la purification de FAHD1 à partir de reins de porcs et de foies de souris. Les méthodes décrites nécessitent une chromatographie liquide à protéines rapides (FPLC), mais utilisent autrement un équipement de laboratoire commun. D’autres méthodes peuvent être trouvées ailleurs 14,15,16,17. Après l’extraction des protéines totales, le protocole proposé comprend une phase d’essai, au cours de laquelle les sous-protocoles de précipitation du sulfate d’ammonium et de chromatographie par échange ionique sont discutés (figure 1). Après avoir défini ces sous-protocoles, la protéine d’intérêt est extraite via une stratégie séquentielle utilisant l’échange ionique et la chromatographie d’exclusion de taille avec FPLC. Sur la base de ces lignes directrices, le protocole final peut être adapté individuellement à d’autres protéines d’intérêt.
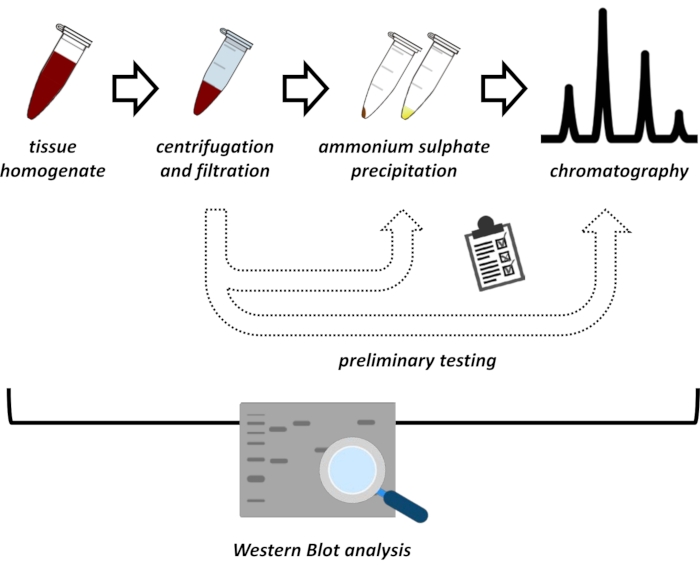
Figure 1 : La stratégie globale de ce protocole. De haut en bas : Les protéines sont extraites des tissus. L’homogénat tissulaire est préparé, centrifugé et filtré. Pour chaque paire d’échantillons surnageants et d’échantillons dérivés de granulés, des tests de précipitation de sulfate d’ammonium et de chromatographie par échange ionique (FPLC) doivent être effectués pour sonder les conditions optimales. Après avoir établi ces sous-protocoles, la protéine peut être extraite par une procédure séquentielle de précipitation du sulfate d’ammonium, de chromatographie par échange ionique et de chromatographie d’exclusion de taille répétitive (FPLC) à des concentrations variables de pH et de sel. Toutes les étapes doivent être contrôlées par transfert western. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Access restricted. Please log in or start a trial to view this content.
Protocole
Toutes les expériences ont été réalisées conformément aux directives institutionnelles. Le rein de porc a été obtenu frais du supermarché local. Des tissus hépatiques ont été prélevés sur des souris de type sauvage C57BL6 conservées à l’Institut de recherche biomédicale sur le vieillissement de l’Université d’Innsbruck, Rennweg 10, 6020 Innsbruck, Autriche, sous la supervision de l’Univ.-Doz. Dr. Pidder Jansen-Dürr, couvert par l’autorisation éthique en tant que chef de projet délivrée en 2013 (BMWF-66.008/0007-II/3b/2013). L’entretien et l’utilisation des souris pour le projet sont couverts par l’autorisation éthique n ° 2020-0.242.978 du 5 mai 2020, délivrée par le ministère autrichien de l’Éducation, des Sciences et de la Recherche (BMBWF).
1. Préparatifs
REMARQUE: Avant le début du protocole, plusieurs choses doivent être préparées, c’est-à-dire le tampon de lyse des protéines, l’échantillon de tissu brut et un anticorps spécifique, en plus des produits chimiques généraux et des matériaux.
- Préparer 250 mL de tampon de lyse des protéines par 100 g de poids net de tissu : 250 mL de 1x PBS avec 50 mM de NaF, 1 mM de PMSF, 2 μg/mL d’aprotinine et 1 mM d’orthovanadate activé (voir tableau 1). Filtrer la solution à l’aide d’une unité de filtration de seringue de 0,22 μm.
REMARQUE: L’activation de l’orthovanadate est nécessaire avant utilisation pour le convertir en un inhibiteur plus puissant de la protéine tyrosine phosphatases18. L’orthovanadate activé peut être obtenu auprès de fournisseurs commerciaux, mais également préparé comme suit.- Préparer une solution mère de 200 mM d’orthovanadate (sodium) dans ddH2O. Pour préparer 10 mL de solution, ajouter 368 mg de Na3VO4 à 9 mL d’eau et dissoudre par agitation. Une fois dissous, porter le volume à 10 mL avec ddH2O.
REMARQUE: Le pH de départ de la solution d’orthovanadate de sodium peut varier en fonction de la source du matériau, et le pH doit être ajusté à 10 dans une approche répétitive comme suit. - Selon le pH initial de la solution, ajuster le pH à 10 avec NaOH ou HCl. À un pH > 10, la solution aura une couleur jaune. Faites bouillir la solution jusqu’à ce qu’elle devienne incolore, refroidissez-la à la température ambiante et vérifiez le pH. Si le pH est >10, ajoutez un petit volume de HCl pour ajuster le pH à 10. À ce stade, la solution peut redevenir jaune.
- Répétez l’ébullition et le refroidissement jusqu’à ce que la solution reste incolore et que le pH se stabilise à 10 (environ 5-7 fois). À ce stade, l’ajout de HCl entraîne une légère apparence de couleur jaune dans la solution. Conserver l’orthovanadate activé dans des aliquotes de 1 mL à -20 °C.
- Préparer une solution mère de 200 mM d’orthovanadate (sodium) dans ddH2O. Pour préparer 10 mL de solution, ajouter 368 mg de Na3VO4 à 9 mL d’eau et dissoudre par agitation. Une fois dissous, porter le volume à 10 mL avec ddH2O.
- Préparez des tubes avec 2 mL de tampon de lyse par gramme de tissu et placez-les sur de la glace.
REMARQUE: Ce protocole utilisait huit tubes de 50 mL, chacun rempli de 30 mL de tampon de lyse au total pour un rein de porc (environ 100-150 g), et deux tubes remplis chacun de 40 mL de tampon de lyse pour 20 foies de souris (chacun 1-2 g) au total. - Préparez le tissu: disséquez le tissu sur une plaque de verre pré-nettoyée placée sur de la glace dans une boîte en mousse de polystyrène. Couper des morceaux de tissu d’environ 100 mg chacun pour être facilement transférés dans les tubes respectifs pour une lyse ultérieure. Transférer les morceaux de tissu dans les tubes préparés (étape 1.2).
- Préparer une solution saturée de sulfate d’ammonium : chauffer 500 mL de ddH2O à 70 °C et, en remuant, ajouter progressivement la poudre de sulfate d’ammonium (voir tableau des matières) jusqu’à ce qu’il n’y ait plus de sulfate d’ammonium dissous. Refroidir cette solution (sur)saturée à température ambiante et la conserver à 4 °C pendant la nuit.
2. Extraction des protéines totales
NOTE Après avoir préparé l’échantillon dans un tampon de lyse des protéines froides (voir étape 1.3), homogénéiser le tissu le mieux possible par sonication par une sonde à ultrasons, ou en utilisant un homogénéisateur électrique comme suit.
- Homogénéisation des tissus
- Dans le cas d’un rein de porc, soniquer la suspension de préférence par une sonde à ultrasons tout en gardant l’échantillon sur de la glace (10 cycles de 15 s d’impulsion, avec des intervalles de 30 s entre les impulsions pour refroidir l’échantillon sur de la glace, à amplitude moyenne avec un rapport cyclique de 50%).
- Dans le cas des organes de souris, homogénéiser la suspension à l’aide d’un homogénéisateur électrique (en commençant par une faible force et en accélérant lentement jusqu’à une force moyenne) tout en gardant l’échantillon sur la glace. Lavez régulièrement l’homogénéisateur électrique dans PBS pour éliminer toute matière organique obstruant l’appareil.
- Prélever 20 μL des échantillons et vérifier au microscope si les cellules du tissu homogénéisé sont correctement détruites; sinon, répétez l’homogénéisation.
- Centrifuger les tubes dans une centrifugeuse de table à 10 000 x g pendant 30 min à 4 °C.
REMARQUE: En option, centrifugez le surnageant une deuxième fois à 20 000 x g pendant 30 min à 4 °C pour éliminer les petites fractions de la pastille initiale qui peuvent avoir été transférées. Cela simplifiera la filtration ultérieure à l’étape 2.3. - Recueillez le surnageant dans un tube frais et placez-le sur de la glace. Filtrer séquentiellement le surnageant à l’aide d’unités de filtration de seringue de 0,45 μm et 0,22 μm. Aliquoter le surnageant en lots de 10 mL et les congeler à -20 °C pour un stockage à court terme ou à -80 °C pour un stockage plus long.
REMARQUE: Le préfiltrage à 0,45 μm élimine la majorité des particules avant qu’une deuxième étape de filtrage de 0,22 μm n’élimine les particules les plus fines. L’utilisation directe du filtre de 0,22 μm peut entraîner le risque d’encrassement des filtres. - Préparer un échantillon de 50 μL pour l’analyse SDS-PAGE/Western blot en ajoutant 10 μL de tampon d’échantillon SDS 5x (voir tableau 1) à 40 μL du surnageant, puis en les faisant bouillir à 95 °C pendant 10 min.
- Éventuellement, remettez en suspension environ 100 μL de pastille obtenue à l’étape 2.2 dans 900 μL de ddH2O et préparez un échantillon pour l’analyse SDS-PAGE/Western blot comme décrit ci-dessus.
REMARQUE : L’inclusion d’échantillons dérivés de granulés dans l’analyse par transfert western, en plus du témoin positif, indiquera si l’expression de la protéine est faible ou si l’anticorps est problématique.
- Éventuellement, remettez en suspension environ 100 μL de pastille obtenue à l’étape 2.2 dans 900 μL de ddH2O et préparez un échantillon pour l’analyse SDS-PAGE/Western blot comme décrit ci-dessus.
3. Analyse SDS-PAGE et Western Blot
REMARQUE: Une analyse par transfert western est nécessaire pour vérifier la solubilité des protéines. Ce qui suit décrit un protocole d’électrobuvardage, à l’aide d’un système de buvard humide/réservoir (voir tableau des matériaux). Un autre protocole pour SDS-PAGE peut être trouvé ailleurs19.
- Préparer un gel SDS-PAGE de polyacrylamide discontinu à 12,5 % conformément aux instructions du fabricant (c.-à-d. un gel empilable sur un gel résolvant; voir le tableau 1). Exécutez les exemples précédemment préparés à l’étape 2 (similaire aux étapes 4, 5 et 6 ; voir ci-dessous).
- Chargez une échelle de marqueurs protéiques dans le premier puits (voir tableau des matériaux). Charge 5 ng de protéine recombinante hFAHD1 (obtenue à partir de bactéries12; voir tableau 1) comme témoin positif dans le deuxième puits.
- Par la suite, chargez 20 μL de l’échantillon à analyser et remplissez tous les puits restants avec 20 μL de tampon d’échantillon SDS-PAGE 1x préparé (c.-à-d. 5x tampon d’échantillon dilué avec ddH2O). Exécutez les gels SDS-PAGE à 125 V à l’aide du tampon SDS (voir tableau 1).
- Une fois SDS-PAGE terminé, effectuez une analyse par transfert western et sondez les membranes à l’aide de l’anticorps disponible soulevé contre FAHD1 (voir le tableau 1).
REMARQUE: Comme les échantillons sont prélevés dans un homogénat de tissu brut, la qualité de la FDS-PAGE et de l’analyse par transfert western à ce stade est généralement compromise; cependant, il est important de vérifier si la protéine à extraire est soluble dans le surnageant. Le protocole suivant a été testé pour le rein porcin et différents organes de souris, y compris le foie, le cœur, le cerveau et les reins.- Préparez le tampon de transfert western blot 10x (voir tableau 1). Préparez le tampon de transfert western blot 1x (voir tableau 1) et refroidissez-le à 4 °C.
- Activer une membrane PVDF pendant 2 min dans du méthanol. Laver la membrane en ddH2O pendant 2 min. Équilibrez la membrane pendant 15 min dans 1x tampon de transfert western blot.
- Lavez le gel SDS avec 1x PBS pendant 10 min en agitant pour retirer le tampon SDS, puis incubez le gel dans 1x tampon de transfert western blot pendant 10 min pour l’équilibrage. Assemblez la cassette d’électrobuvardage (c’est-à-dire en combinant la membrane PVDF activée et les gels) conformément aux instructions du fabricant.
- Faire passer le blot par électroblotting à 300 mA pendant 1 h dans une boîte en mousse de polystyrène remplie de glace ou dans la chambre froide (4 °C). Transférez la membrane PVDF dans un tube de 50 mL avec son côté exposé face à la face interne du tube. Incuber la membrane dans 20 mL de tampon bloquant le transfert western (voir tableau 1) pendant la nuit à 4 °C tout en roulant sur un rouleau tubulaire (voir tableau des matériaux).
- Le lendemain, lavez la membrane pendant 5 min avec 20 mL de tampon de lavage western blot (PBS avec 0,1% (v/v) Tween 20) dans le même tube pendant le laminage. Incuber la membrane dans le même tube avec l’anticorps primaire2 (ciblant FAHD1; voir tableau 1) dilué à 1:500 dans un tampon bloquant le transfert western pendant 1 h à température ambiante pendant le laminage.
- Lavez la membrane dans le même tube trois fois pendant 10 minutes chacune avec 20 mL de tampon de lavage western pendant le laminage. Incuber la membrane pendant 30 min à température ambiante avec un anticorps secondaire conjugué HRP (voir Tableau des matériaux) dilué à 1:3000 dans 5 mL de tampon bloquant le transfert western.
- Lavez la membrane dans le même tube trois fois pendant 10 minutes chacune avec 20 mL de tampon de lavage western blot et deux fois pendant 5 minutes chacune avec 1x PBS. Séchez la membrane en la tenant soigneusement avec une pince à épiler sur un bord et en touchant un morceau de cellulose ou un morceau de papier Whatman avec le bord opposé (inférieur) de la membrane. Placez la membrane (côté exposé vers le haut) sur une plaque de verre nettoyée.
- Couvrez soigneusement toute la membrane avec 1 mL de substrat de western blot ECL préparé à l’aide d’une pipette, en prenant soin de ne pas créer de bulles d’air. Laissez la solution ECL incuber pendant 3 min et développez immédiatement la membrane à l’aide d’un film radiographique ou d’un système d’imagerie.
REMARQUE: Si la protéine n’a été détectée dans aucun des échantillons, mais seulement dans le témoin positif, cela peut indiquer que la protéine est insoluble ou qu’elle n’est pas présente en quantités suffisantes pour être détectée par l’anticorps. Si seulement des nanogrammes du témoin positif étaient chargés, le premier scénario est plus probable. Si aucune protéine n’a été détectée, vérifiez la qualité de l’anticorps et passez peut-être à un anticorps polyclonal plutôt qu’à un anticorps monoclonal. En de rares occasions, c’est-à-dire pour certaines protéines hydrophobes, la protéine peut être détectable après centrifugation, mais pas après filtration. Dans un tel cas, il est recommandé d’utiliser des unités de filtration spéciales pour les protéines hydrophobes.
- En option, colorez les membranes PVDF après le transfert western pour contrôler le transfert réussi de la protéine du gel SDS-PAGE vers la membrane PVDF.
REMARQUE: La coloration de Coomassie est recommandée pour le dépannage, le développement de méthodes et la documentation, mais gardez à l’esprit qu’après l’application de ce protocole, les membranes sont perdues pour une analyse ultérieure du transfert Western. La coloration Ponceau S donne une coloration plus faible, mais peut être utilisée si les membranes doivent être re-sondées.- Préparez de petits plateaux contenant les solutions de coloration (Coomassie ou Ponceau S) et de décoloration.
- À l’aide d’une pince à épiler, mettez la membrane dans la solution de coloration et secouez doucement jusqu’à ce que la membrane soit bien colorée (5-10 min).
- Transférer la membrane dans la solution de conservation et agiter jusqu’à ce que la solution soit saturée (5-10 min). Répétez l’étape de conservation jusqu’à ce que les bandes protéiques puissent être observées sur la membrane; si aucune bande n’est observée, répétez la coloration avec un temps d’incubation plus long. Séchez la membrane en la plaçant sur une plaque de verre à l’aide d’une pince à épiler.
4. Essais : Précipitation du sulfate d’ammonium
REMARQUE: La précipitation du sulfate d’ammonium est une méthode de purification des protéines en modifiant la solubilité de la protéine. Dans une expérience préliminaire, la concentration de sulfate d’ammonium est augmentée séquentiellement à une valeur qui précipite une quantité maximale de contaminants protéiques, tout en laissant FAHD1 en solution. La solubilité de la protéine est à nouveau sondée par l’analyse par transfert de Western.
- Procéder à l’étape 2.3 : soit décongeler une aliquote de l’échantillon, soit procéder directement après l’extraction des protéines (c.-à-d. sans congeler l’échantillon). Filtrer l’échantillon à l’aide d’une unité de filtration de 0,22 μm pour exclure les précipités possibles après décongélation. Préparer six tubes de 1,5 mL sur de la glace et transférer 250 μL d’échantillon dans chaque tube.
- Préparer une série de dilution de 5%, 10%, 15%, 20%, 25% et 30% de sulfate d’ammonium dans les tubes préparés ci-dessus, et porter le volume final à 1000 μL avec un tampon de lyse des protéines. Incuber les échantillons à 4 °C pendant la nuit sur un rotateur tubulaire (voir tableau des matériaux).
- À l’aide d’une centrifugeuse de table, centrifuger à 10 000 x g pendant 30 min à 4 °C et transférer soigneusement tous les surnageants dans des tubes séparés. Sécher à l’air libre les granulés résultants et remettre en suspension chacune d’elles dans 1000 μL de ddH2O.
- Pour chaque paire de granulés et de surnageants remis en suspension de l’étape précédente, mélanger 40 μL avec 10 μL de tampon d’échantillon 5x SDS et faire bouillir à 95 °C avec les couvercles ouverts jusqu’à ce que la majeure partie du liquide se soit vaporisée. Ensuite, remettez en suspension la pastille dans un mélange de 50% de DMSO dans ddH2O.
- Effectuez SDS-PAGE (étape 3) mais exécutez les gels à 80 V pendant 3 h. Pour chaque concentration de sulfate d’ammonium, charger les échantillons dérivés de la pastille remise en suspension et du surnageant (étape 4.3) par paires. Effectuez une analyse western blot (étape 3).
- Vérifiez la concentration la plus élevée de sulfate d’ammonium, à laquelle la protéine à purifier (c.-à-d. FAHD1) reste dans l’échantillon dérivé du surnageant. Sur la base des résultats, définir un protocole de précipitation de sulfate d’ammonium pour la protéine d’intérêt, à utiliser dans de futures expériences.
REMARQUE: Le sulfate d’ammonium est bien connu pour déformer SDS-PAGE et Western Blot. À mesure que la concentration de sulfate d’ammonium augmente, la qualité de l’analyse par transfert western sera compromise. Cependant, comme à l’étape 3 précédente, cette analyse est utilisée pour vérifier la solubilité de la protéine d’intérêt à des concentrations données de sulfate d’ammonium. Ce protocole vise à précipiter d’autres protéines, tandis que la protéine à purifier doit rester soluble.
5. Essais : chromatographie par échange ionique avec FPLC
REMARQUE: Les molécules avec des groupes fonctionnels chargés sont liées à une colonne de particules de silice pour FPLC, permettant la différenciation des protéines en fonction de leur charge de surface. Effectuez cette étape deux fois, à l’aide de la colonne d’échange cationique et de la colonne d’échange anionique (voir Tableau des matériaux). Les étapes du protocole sont les mêmes pour la chromatographie cationique ou anionique, mais les tampons à utiliser sont différents (voir tableau 1); les deux avec des conditions de NaCl « faible teneur en sel » de 15 mM et de « sel élevé » de 1 M NaCl. Pour les colonnes utilisées, un débit de 1 mL/min est recommandé.
- Configurez le système FPLC avec la colonne d’échange anionique ou cationique. Laver la colonne avec 5 volumes de colonne (CV) de 20% EtOH (enH2O), suivi de 5 CV de ddH2O. Alternativement, laver la colonne avec 1 CV de tampon à faible teneur en sel, un tampon à haute teneur en sel et à nouveau un tampon à faible teneur en sel dans l’ordre jusqu’à ce qu’aucun autre pic ne soit observé dans le chromatogramme, mais lavez au moins une fois.
- Après avoir déterminé le protocole optimal pour la précipitation du sulfate d’ammonium à petite échelle (étape 4), appliquez le protocole de précipitation à 10 mL d’homogénat tissulaire d’origine (étape 2). En option, dialysez l’échantillon par rapport au tampon à faible teneur en sel.
- Appliquez l’échantillon sur la colonne (p. ex., par injection ou à l’aide d’une pompe à échantillon) et prélevez le débit. Lavez la colonne avec 1 CV du tampon à faible teneur en sel.
- Mettez en place une élution à gradient linéaire de 100% de tampon à faible teneur en sel / 0% de tampon à sel élevé à 0% de tampon à faible teneur en sel / 100% de tampon à sel élevé dans 3 CV. Collectez en continu des fractions de 1 mL. Une fois le gradient terminé, continuez à fonctionner avec le tampon de sel élevé jusqu’à ce qu’aucun autre pic associé aux protéines (absorption UV à 280/255 nm) ne soit détecté dans le chromatogramme sur la plage de 1 CV.
- Appliquer 1 mL de FDS à 25 % dissous dans 0,5 M de NaOH (en ddH2O) pour nettoyer la colonne. Lavez consécutivement la colonne avec 3 CV de ddH2O et 3 CV de 20% EtOH (en ddH2O).
- Prélever des échantillons SDS-PAGE de toutes les fractions de crête et de l’écoulement, et les sonder via le transfert Western pour détecter la présence de la protéine d’intérêt (étape 3). Congeler les fractions collectées dans de l’azote liquide et les stocker à -80 °C.
- Une fois l’analyse par transfert western terminée, décongeler et mettre en commun les fractions contenant la protéine d’intérêt et jeter les autres. Répétez les étapes 5.1 à 5.5 avec la colonne alternative (c.-à-d. colonne d’échange cationique ou anionique).
- Une fois les deux colonnes sondées, définissez un protocole FPLC pour la protéine d’intérêt, à utiliser dans de futures expériences. Réduire le volume de la solution protéique à l’aide d’unités de filtration ultra-centrifugation (10 kDa, voir Tableau des matériaux) jusqu’à 2 mL.
NOTE: Il y a deux résultats attendus de cette série d’expériences. Soit la protéine d’intérêt s’est attachée à l’une des colonnes, et la solution protéique est déjà assez pure après l’élution, soit la protéine est restée dans le flux dans les deux cas. Dans ce dernier scénario, bien que la protéine soit dans le flux, l’effet nettoyant de cette étape peut encore être significatif. Dans un tel cas, comme pour FAHD1 dans le rein porcin et le foie de souris, cette étape d’échange ionique sera toujours effectuée. Si ni la colonne d’échange cationique ni la colonne d’échange anionique ne peuvent fournir un effet de nettoyage approprié, on peut essayer de modifier le pH du lysat et du tampon, et de dialyser l’échantillon par rapport au tampon en cours d’exécution avant l’application sur FPLC.
6. Extraction des protéines à l’aide de sous-protocoles définis pour la précipitation du sulfate d’ammonium et la FPLC
NOTE: Les particules poreuses dans une colonne de gel de silice pour FPLC (Voir Tableau des matériaux) permettent la différenciation des protéines en fonction de leur rayon hydrodynamique. Les étapes décrites doivent être effectuées avec un système FPLC, en utilisant la chromatographie d’exclusion de taille (SEC). Pour la colonne SEC utilisée (voir Tableau des matériaux), un débit de 0,3 mL/min est recommandé.
- Préparez tous les matériaux nécessaires (voir l’étape 1) et extrayez la protéine totale du tissu (voir l’étape 2). Effectuer une précipitation de sulfate d’ammonium avec tout l’homogénat tissulaire qui n’a pas été utilisé pour les tests (voir étape 4). Pour les plus grands volumes, concentrez le lysat à l’aide d’unités de filtration ultracentrifugation (10 kDa; voir Tableau des matériaux) jusqu’à un volume plus petit de 50 mL ou moins.
- Effectuer une première étape de purification à l’aide de la chromatographie par échange ionique (voir étape 5).
- Préparez des échantillons pour le transfert Western, comme décrit dans les étapes précédentes. Effectuer une analyse par transfert western et regrouper toutes les fractions contenant faHD1 provenant de la chromatographie par échange ionique.
- Réduire le volume de la solution protéique jusqu’à 2 mL à l’aide d’unités de filtration ultracentrifugation (10 kDa). Filtrer séquentiellement la solution avec des unités de filtration de seringue de 0,45 μm et 0,22 μm pour éliminer toute microprécipline.
- Équilibrez la colonne SEC avec 1 CV de tampon d’exécution SEC (voir Tableau 1), contenant 1 mM TNT. Chargez l’échantillon sur la colonne et exécutez la chromatographie jusqu’à ce que toutes les protéines soient éluées (1-2 CV).
- Prélever des fractions de 1 mL de l’écoulement qui correspondent à des pics significatifs dans le chromatogramme (absorption UV à 280/255 nm) et préparer des échantillons de 50 μL de chaque fraction collectée pour l’analyse SDS-PAGE et Western blot, comme décrit dans les étapes précédentes. Congelez toutes les fractions à l’aide d’azote liquide et stockez-les à -80 °C.
- Laver consécutivement la colonne SEC avec 1 CV de ddH2O et 1 CV de 20% EtOH (en ddH2O). Effectuez une analyse Western-Blot et regroupez toutes les fractions contenant FAHD1. Réduire le volume de la solution protéique jusqu’à 2 mL à l’aide d’unités de filtration à ultracentrifugation (10 kDa, voir tableau des matériaux).
- Évaluer la concentration en protéines à l’aide d’une trousse commerciale d’analyse de l’ACB (voir tableau des matériaux).
REMARQUE: Le pH et la teneur en sel de la phase mobile peuvent influencer le profil d’élution des protéines globulaires20. Les conditions acides ou basiques peuvent entraîner une diminution de la définition des pics et une augmentation des interactions protéine-matrice conduisant à une rétention partielle des protéines sur la colonne20. Cet effet peut être exploité pour une purification ultérieure des protéines. Une répétition de l’étape 6 avec des débits, des pH et des concentrations de sel différents peut améliorer la pureté de la protéine20.
7. Coloration à l’argent
REMARQUE: L’analyse de la coloration à l’argent des gels SDS-PAGE est nécessaire pour vérifier les contaminations protéiques qui peuvent ne pas être observées avec la coloration de Coomassie. Le protocole suivant est l’une des nombreuses versions que l’on peut trouver dans la littérature21. Effectuez toutes les étapes d’incubation en secouant dans un plateau en verre propre. Recueillir tous les liquides contenant de l’argent et du formaldéhyde dans un conteneur à déchets spéciaux et les jeter correctement.
- Incuber les gels SDS-PAGE dans une solution de fixation de coloration à l’argent (voir tableau 1) pendant la nuit dans la chambre froide. Incuber les gels dans une solution d’incubation de coloration à l’argent (voir tableau 1) pendant 3 h à température ambiante. Éventuellement, ajouter du glutaraldéhyde (voir tableau 1) pour améliorer la détection des bandes faibles. Laver les gels quatre fois dans ddH2O pendant 10 min chacun.
- Incuber les gels dans une solution d’argent colorant à l’argent (voir tableau 1) pendant 1 h.
REMARQUE: Gardez à l’esprit qu’à partir de maintenant, tous les liquides et le gel lui-même contiennent de l’argent et du formaldéhyde qui sont toxiques. - Incuber les gels dans une solution de révélateur de coloration à l’argent (voir tableau 1) en agitant vigoureusement jusqu’à ce que les bandes soient clairement visibles. Pour arrêter la réaction, jeter la solution de développement et incuber immédiatement les gels dans une solution d’arrêt de coloration à l’argent (voir tableau 1) pendant au moins 10 min.
REMARQUE: Les bandes colorées dans les étapes 7.2 et 7.3 deviendront constamment plus développées. L’ajout de plus de formaldéhyde à la solution que prévu peut être nécessaire si la coloration est faible.
Access restricted. Please log in or start a trial to view this content.
Résultats
La protéine FAHD1 a été extraite du rein du porc et du foie de souris à l’aide du protocole présenté. Pour les tissus de souris, plusieurs organes sont nécessaires pour obtenir plusieurs μg après l’étape finale de purification. Pour cette raison, cet article se concentre sur l’extraction de FAHD1 à partir de reins de porcs, ce qui est une expérience beaucoup plus exemplaire. L’extraction de FAHD1 du foie de souris est effectuée pour présenter les difficultés et les pièges possibles de ce protocole...
Access restricted. Please log in or start a trial to view this content.
Discussion
Étapes critiques du protocole
Il est essentiel de suivre les directives communes pour la manipulation des protéines, comme le travail sur la glace et à des conditions de pH et de sel modérées. L’utilisation d’inhibiteurs de la protéase est bénéfique pour la méthode, tandis que l’utilisation d’inhibiteurs du protéasome est fortement recommandée. La congélation et la décongélation de l’échantillon peuvent toujours entraîner une précipitation protéique (au moins partielle), de...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont pas d’intérêts financiers concurrents.
Remerciements
Les auteurs sont très reconnaissants de l’assistance technique d’Ayse Öztürk et Eva Albertini. Les souris utilisées pour la génération de tissu hépatique ont été maintenues sous la supervision de Univ.-Doz. Dr Pidder Jansen-Dürr (Institut de recherche biomédicale sur le vieillissement de l’Université d’Innsbruck, Rennweg 10, 6020 Innsbruck, Autriche).
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 0.22 µm filter units | MERCK | SLGP033RS | Millex-HP, 0.22 µm, PES 33 mm, not steril |
| 0.45 µm filter units | MERCK | SLHP033NS | Millex-HP, 0.45 µm, PES 33 mm, not steril |
| 15 mL Falcon tubes | VWR | 734-0451 | centrifugal tubes |
| 50 mL Falcon tubes | VWR | 734-0448 | centrifugal tubes |
| 96-Well UV Microplate | Thermo-Fischer | 8404 | UV/VIS transparent flat-bottom 96 well plates |
| Acrylamide/Bis Solution (40%, 29:1 ratio) | BIO-RAD | #1610147 | 40% acrylamide/bis-acrylamide, 29:1 (3.3% crosslinker) solution for casting polyacrylamide gels |
| ÄKTA FPLC system | GE Healthcare Life Sciences / Cytiva | - | using the FPLC system by GE Healthcare; different custom versions exist; this work used the "ÄKTA pure" system |
| Amicon Ultra-15, PLGC Ultracel-PL Membran, 10 kDa | MERCK | UFC901024 | centrifigal filters for protein enrichment; 10 kDa molecular mass filter; 15 mL |
| Amicon Ultra-4, PLGC Ultracel-PL Membran, 10 kDa | MERCK | UFC801024 | centrifigal filters for protein enrichment; 10 kDa molecular mass filter; 4 mL |
| Ammonium sulfate powder | MERCK | A4418 | ammonium sulphate for molecular biology, ≥99.0% |
| Ammoniumpersulfat reagent grade, 98% | MERCK | 215589 | Catalyst for acrylamide gel polymerization. |
| Coomassie Brilliant blue R 250 | MERCK | 1125530025 | Coomassie Brilliant blue R 250 (C.I. 42660) for electrophoresis Trademark of Imperial Chemical Industries PLC. CAS 6104-59-2, pH 6.2 (10 g/l, H2O, 25 °C) |
| Dialysis tubing cellulose membrane | MERCK | D9277 | Cellulose membranes for the exchange of buffers via dialysis. |
| Eppendof tubes 1.5 mL | VWR | 525-1042 | microcentrifugal tubes; autoclaved |
| HiLoad 26/600 Superdex 75 pg | GE Healthcare Life Sciences / Cytiva | 28989334 | HiLoad Superdex 75 pg prepacked columns are for high-resolution size exclusion chromatography of recombinant proteins |
| Immun-Blot PVDF Membrane | BIO-RAD | #1620177 | PVDF membranes are protein blotting membranes optimized for fluorescent and multiplex fluorescent applications. |
| Mini Trans-Blot Electrophoretic Transfer Cell | BIO-RAD | #1703930 | Use the Mini Trans-Blot Cell for rapid blotting of Mini-PROTEAN precast and handcast gels. |
| Mini-PROTEAN Tetra Vertical Electrophoresis Cell for Mini Precast Gels | BIO-RAD | #1658004 | 4-gel vertical electrophoresis system, includes electrode assembly, companion running module, tank, lid with power cables, mini cell buffer dam. |
| Mono Q 10/100 GL | GE Healthcare Life Sciences / Cytiva | 17516701 | Mono Q columns are strong anion exchange chromatography columns for protein analysis or small scale, high resolution polishing of proteins. |
| Mono S 10/100 GL | GE Healthcare Life Sciences / Cytiva | 17516901 | Mono S columns are strong cation exchange chromatography columns for protein analysis or small scale high resolution polishing of proteins. |
| PageRuler Prestained Protein Ladder, 10 to 180 kDa | Thermo-Fischer | 26616 | A mixture of 10 blue-, orange-, and green-stained proteins (10 to 180 kDa) for use as size standards in protein electrophoresis (SDS-PAGE) and western blotting. |
| Pierce BCA Protein Assay Kit | Thermo-Fischer | 23225 | A two-component, high-precision, detergent-compatible protein assay for determination of protein concentration. |
| Sonifier 250; Ultrasonic Cell Disruptor w/ Converter | Branson | - | New models at https://www.emerson.com/documents/automation/brochure-sonifier-sfx250-sfx550-cell-disruptors-homogenizers-branson-en-us-168180.pdf |
| Swine Anti-Rabbit Immunoglobulins/HRP (affinity isolated) | Agilent Dako | P0399 | The antibody used for horseradish peroxidase conjugation reacts with rabbit immunoglobulins of all classes. |
| TEMED, 1,2-Bis(dimethylamino)ethane, TMEDA | MERCK | T9281 | TEMED (N,N,N′,N′-Tetramethylethylenediamine) is molecule which allows rapid polymerization of polyacrylamide gels. |
| Tube Roller | - | - | A general tube rotator roller; e.g. a new model at https://labstac.com/de/Mixer/Roller/c/71 |
| Tube Rotator | - | - | A general tube rotator wheel; e.g. a new model at https://labstac.com/de/Tube-Roller/p/MT123 |
| ULTRA-TURRAX; T 25 digital | IKA | 0003725000 | New models at https://www.ika.com/de/Produkte-Lab-Eq/Dispergierer-Dipergiergeraet-Homogenisierer-Homogenisator-csp-177/T-25-digital-ULTRA-TURRAX-cpdt-3725000/ |
Références
- Pircher, H., et al. Identification of FAH domain-containing protein 1 (FAHD1) as oxaloacetate decarboxylase. Journal of Biological Chemistry. 290 (11), 6755-6762 (2015).
- Pircher, H., et al. Identification of human Fumarylacetoacetate Hydrolase Domain-containing Protein 1 (FAHD1) as a novel mitochondrial acylpyruvase. Journal of Biological Chemistry. 286 (42), 36500-36508 (2011).
- Kang, T. -W., et al. Senescence surveillance of pre-malignant hepatocytes limits liver cancer development. Nature. 479 (7374), 547-551 (2011).
- Hong, H., Seo, H., Park, W., Kim, K. K. -J. Sequence, structure and function-based classification of the broadly conserved FAH superfamily reveals two distinct fumarylpyruvate hydrolase subfamilies. Environmental Microbiology. 22 (1), 270-285 (2020).
- Timm, D. E., Mueller, H. A., Bhanumoorthy, P., Harp, J. M., Bunick, G. J. Crystal structure and mechanism of a carbon-carbon bond hydrolase. Structure. 7 (9), London, England. 1023-1033 (1999).
- Bateman, R. L., et al. Mechanistic inferences from the crystal structure of Fumarylacetoacetate Hydrolase with a bound phosphorus-based inhibitor. Journal of Biological Chemistry. 276 (18), 15284-15291 (2001).
- Weiss, A. K. H., et al. Structural basis for the bi-functionality of human oxaloacetate decarboxylase FAHD1. Biochemical Journal. 475 (22), 3561-3576 (2018).
- Etemad, S., et al. Oxaloacetate decarboxylase FAHD1 - a new regulator of mitochondrial function and senescence. Mechanisms of Ageing and Development. 177, 22-29 (2019).
- Weiss, A. K. H., et al. Regulation of cellular senescence by eukaryotic members of the FAH superfamily - A role in calcium homeostasis. Mechanisms of Ageing and Development. 190, 111284(2020).
- Petit, M., Koziel, R., Etemad, S., Pircher, H., Jansen-Dürr, P. Depletion of oxaloacetate decarboxylase FAHD1 inhibits mitochondrial electron transport and induces cellular senescence in human endothelial cells. Experimental Gerontology. 92, 7-12 (2017).
- Wiley, C. D., et al. Mitochondrial dysfunction induces senescence with a distinct secretory phenotype. Cell Metabolism. 23 (2), 303-314 (2016).
- Weiss, A. K. H., et al. Expression, purification, crystallization, and enzyme assays of Fumarylacetoacetate Hydrolase Domain-containing proteins. Journal of Visualized Experiments: JoVE. (148), e59729(2019).
- Weiss, A. K. H., et al. Inhibitors of Fumarylacetoacetate Hydrolase Domain Containing Protein 1 (FAHD1). Molcules. 26 (16), 5009(2021).
- Mizutani, H., Kunishima, N. Purification, crystallization and preliminary X-ray analysis of the fumarylacetoacetase family member TTHA0809 from Thermus thermophilus HB8. Acta Crystallographica Section F Structural Biology and Crystallization Communications. 63 (9), 792-794 (2007).
- Lee, C. H. A simple outline of methods for protein isolation and purification. Endocrinology and Metabolism. 32 (1), Seoul, Korea. 18-22 (2017).
- Amer, H. E. A. Purification of proteins: Between meaning and different methods). Proteomics Technologies and Applications. , (2019).
- Niu, L., Yuan, H., Gong, F., Wu, X., Wang, W. Protein extraction methods shape much of the extracted proteomes. Frontiers in Plant Science. 9, 802(2018).
- Gordon, J. A. Use of vanadate as protein-phosphotyrosine phosphatase inhibitor. Methods in Enzymology. 201, 477-482 (1991).
- Gallagher, S. R. SDS-polyacrylamide gel electrophoresis (SDS-PAGE). Current Protocols in Essential Laboratory Techniques. , (2012).
- Ahmed, U., Saunders, G. Effect of pH on Protein Size Exclusion Chromatography. , Available from: https://www.agilent.com/cs/library/applications/5990-8138EN.pdf (2011).
- Sørensen, B. K., et al. Silver staining of proteins on electroblotting membranes and intensification of silver staining of proteins separated by polyacrylamide gel electrophoresis. Analytical Biochemistry. 304 (1), 33-41 (2002).
- Fagerberg, L., et al. Analysis of the human tissue-specific expression by genome-wide integration of transcriptomics and antibody-based proteomics. Molecular & Cellular Proteomics. 13 (2), 397-406 (2014).
- Cytiva Life Fundamentals of size exclusion chromatography. , Available from: https://www.cytivalifesciences.com/en/us/solutions/protein-research/knowledge-center/protein-purification-methods/size-exclusion-chromatography (2022).
- Rosano, G. L., Ceccarelli, E. A. Recombinant protein expression in Escherichia coli: advances and challenges. Frontiers in Microbiology. 5, 172(2014).
- Rosano, G. L., Morales, E. S., Ceccarelli, E. A. New tools for recombinant protein production in Escherichia coli: A 5-year update. Protein Science: A Publication of the Protein Society. 28 (8), 1412-1422 (2019).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon