É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Extração e Purificação da Proteína FAHD1 do fígado de rim suíno e rato
Neste Artigo
Resumo
Este protocolo descreve como extrair a proteína de domínio de hidrolase 1 (FAHD1) de rim suíno e fígado de camundongo. Os métodos listados podem ser adaptados a outras proteínas de interesse e modificados para outros tecidos.
Resumo
Fumarylacetoacetate hydrolase proteína contendo domínio 1 (FAHD1) é o primeiro membro identificado da superfamília FAH em eucariotes, agindo como oxaloacetate decarboxylase em mitocôndrias. Este artigo apresenta uma série de métodos para a extração e purificação do FAHD1 a partir de fígado de rim suíno e rato. Os métodos cobertos são cromatografia de troca iônica com cromatografia líquida de proteína rápida (FPLC), filtragem de gel preparatório e analítico com FPLC e abordagens proteômicas. Após a extração total da proteína, a precipitação de sulfato de amônio e a cromatografia de troca iônica foram exploradas, e o FAHD1 foi extraído através de uma estratégia sequencial usando troca iônica e cromatografia de exclusão de tamanho. Esta abordagem representativa pode ser adaptada a outras proteínas de interesse (expressas em níveis significativos) e modificada para outros tecidos. A proteína purificada do tecido pode apoiar o desenvolvimento de anticorpos de alta qualidade e/ou potentes e específicos inibidores farmacológicos.
Introdução
A proteína de domínio fah eucariótico 1 (FAHD1) atua como oxaloacetato bifuncional (OAA) decarboxylase (ODx)1 e hidrolase acylpyruvate (ApH)2. Está localizada nas mitocôndrias2 e pertence à ampla superfamília FAH de enzimas 1,2,3,4,5,6. Embora sua atividade de ApH seja apenas de menor relevância, a atividade ODx do FAHD1 está envolvida na regulação do fluxo de ciclo TCA 1,7,8,9. OAA não é apenas necessário para a reação de sinthase citrato central no ciclo do ácido tricarboxílico, mas também age como um inibidor competitivo de succinato desidrogenase como parte do sistema de transporte de elétrons e como metabólito catapleótico. A redução da expressão genética FAHD1 nas células endoteliais da veia umbilical humana (HUVEC) resultou em uma redução significativa na taxa de proliferação celular10, e inibição significativa do potencial da membrana mitocondrial, associada a uma mudança concomitante para a glicólise. O modelo de trabalho refere-se à senescência associada à disfunção mitocondrial (MiDAS)11-like fenótipo8, onde os níveis mitocondrial de OAA são fortemente regulados pela atividade FAHD1 1,8,9.
A proteína recombinante é mais fácil de obter através da expressão e purificação de bactérias12 em vez de tecido. No entanto, uma proteína expressa em bactérias pode ser tendenciosa por possível falta de modificações pós-translacionais, ou pode ser simplesmente problemática (ou seja, devido à perda de plasmídeos, respostas de estresse bacteriano, ligações de dissulfeto distorcidas/não formadas, nenhuma ou má secreção, agregação de proteínas, decote protelítico, etc.). Para determinadas aplicações, a proteína precisa ser obtida a partir de lisesto celular ou tecido, a fim de incluir tais modificações e/ou excluir possíveis artefatos. A proteína purificada do tecido suporta o desenvolvimento de anticorpos de alta qualidade e/ou inibidores farmacológicos potentes e específicos para enzimas selecionadas, como para FAHD113.
Este manuscrito apresenta uma série de métodos para a extração e purificação do FAHD1 a partir de fígado de rim suíno e rato. Os métodos descritos requerem cromatografia líquida de proteína rápida (FPLC), mas de outra forma usam equipamentos de laboratório comuns. Métodos alternativos podem ser encontrados em outros lugares 14,15,16,17. Após a extração total da proteína, o protocolo proposto envolve uma fase de teste, na qual são discutidos sub-protocolos para precipitação de sulfato de amônio e cromatografia de troca iônica (Figura 1). Após a definição desses subtítnicos, a proteína de interesse é extraída através de uma estratégia sequencial usando troca iônica e cromatografia de exclusão de tamanho com FPLC. Com base nessas diretrizes, o protocolo final pode ser adaptado individualmente para outras proteínas de interesse.
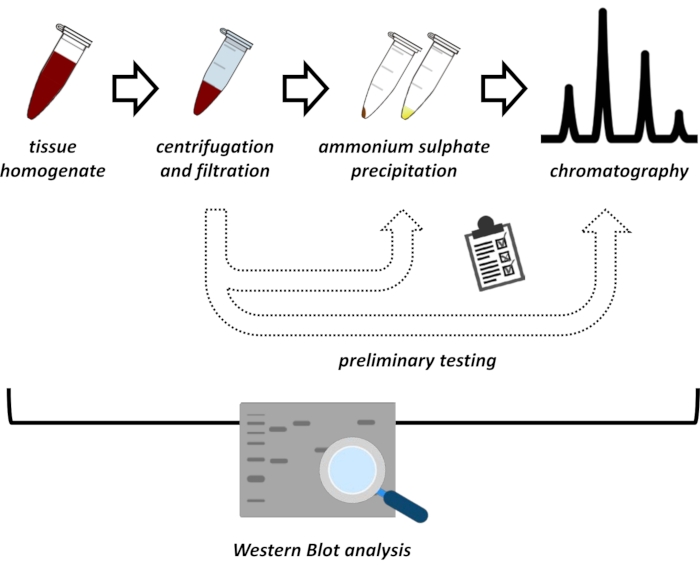
Figura 1: A estratégia global deste protocolo. De cima para baixo: A proteína é extraída de tecidos. A homogeneidade do tecido é preparada, centrifugada e filtrada. Para cada par de amostras supernascidas e derivadas de pelotas, os testes para precipitação de sulfato de amônio e cromatografia de troca iônica (FPLC) precisam ser realizados para sondar para condições ideais. Após a criação desses sub-protocolos, a proteína pode ser extraída através de um procedimento sequencial de precipitação de sulfato de amônio, cromatografia de troca iônica e cromatografia de exclusão de tamanho repetitivo (FPLC) em diferentes concentrações de pH e sal. Todos os passos precisam ser controlados por mancha ocidental. Clique aqui para ver uma versão maior desta figura.
Protocolo
Todos os experimentos foram realizados em conformidade com as diretrizes institucionais. O rim suíno foi obtido fresco do supermercado local. Os tecidos hepáticos foram colhidos de camundongos do tipo selvagem C57BL6 mantidos no Instituto de Pesquisa de Envelhecimento Biomédico da Universidade de Innsbruck, Rennweg 10, 6020 Innsbruck, Áustria sob a supervisão da Univ.-Doz. Dr. Pidder Jansen-Dürr, coberto pela permissão ética como líder do projeto emitido em 2013 (BMWF-66.008/0007-II/3b/2013). A manutenção e o uso dos camundongos para o projeto estão cobertos pela permissão ética nº 2020-0.242.978 a partir de 5 de maio de 2020, emitido pelo Ministério da Educação, Ciência e Pesquisa da Áustria (BMBWF).
1. Preparativos
NOTA: Antes do início do protocolo, várias coisas precisam ser preparadas, ou seja, o tampão de proteína, a amostra de tecido bruto e um anticorpo específico, além de produtos químicos gerais e materiais.
- Prepare 250 mL de tampão de proteína por 100 g de peso líquido do tecido: 250 mL de 1x PBS com 50 mM NaF, 1 mM PMSF, 2 μg/mL aprotinína e 1 mM de ortovanadate ativado (ver Tabela 1). Filtre a solução usando uma unidade de filtro de seringa de 0,22 μm.
NOTA: A ativação da ortovanadate é necessária antes do uso para convertê-lo em um inibidor mais potente de fosfates de proteína tyrosina18. A ortopedia ativada pode ser obtida a partir de fornecedores comerciais, mas também preparada da seguinte forma.- Prepare uma solução de estoque de 200 mM de ortovanadate (sódio) em ddH2O. Para preparar 10 mL de solução, adicione 368 mg de Na3VO4 a 9 mL de água e dissolva mexendo. Uma vez dissolvido, coma o volume até 10 mL com ddH2O.
NOTA: O pH inicial da solução de ortovanadato de sódio pode variar de acordo com a fonte do material, e o pH precisa ser ajustado para 10 em uma abordagem repetitiva da seguinte forma. - Dependendo do pH inicial da solução, ajuste o pH para 10 com NaOH ou HCl. No pH > 10, a solução terá uma cor amarela. Ferva a solução até ficar incolor, esfrie-a até a temperatura ambiente e verifique o pH. Se o pH estiver >10, adicione um pequeno volume de HCl para ajustar o pH a 10. Neste ponto, a solução pode ficar amarela novamente.
- Repita a ebulição e o resfriamento até que a solução permaneça incolor e o pH se estabilize em 10 (aproximadamente 5-7 vezes). Neste ponto, adicionar HCl resulta em uma leve aparência de cor amarela na solução. Armazene ortovanadate ativado em alíquotas de 1 mL a -20 °C.
- Prepare uma solução de estoque de 200 mM de ortovanadate (sódio) em ddH2O. Para preparar 10 mL de solução, adicione 368 mg de Na3VO4 a 9 mL de água e dissolva mexendo. Uma vez dissolvido, coma o volume até 10 mL com ddH2O.
- Prepare tubos com 2 mL de tampão de lise por grama de tecido e coloque-os no gelo.
NOTA: Este protocolo utilizou oito tubos de 50 mL, cada um preenchido com 30 mL de tampão de lise no total para um rim suíno (cerca de 100-150 g), e dois tubos cada um preenchidos com 40 mL de tampão de lise para 20 fígados de camundongos (cada 1-2 g) no total. - Prepare o tecido: disseque o tecido em uma placa de vidro pré-limpa colocada no gelo em uma caixa de espuma de poliestireno. Corte pedaços de tecido de cerca de 100 mg cada para ser facilmente transferido para os respectivos tubos para lise subsequente. Transfira os pedaços de tecido para os tubos preparados (passo 1.2).
- Prepare uma solução de sulfato de amônio saturado: aqueça 500 mL de ddH2O a 70 °C e, ao mexer, adicione gradualmente pó de sulfato de amônio (ver Tabela de Materiais) até que não haja mais sulfato de amônio. Esfrie esta solução (sobre)saturada à temperatura ambiente e armazene-a a 4 °C durante a noite.
2. Extração total de proteínas
NOTA Após o preparo da amostra em tampão de proteína fria (ver passo 1.3), homogeneize o tecido da melhor forma possível através da sônica por uma sonda ultrassônica, ou usando um homogeneizador elétrico da seguinte forma.
- Homogeneização dos tecidos
- No caso de um rim suíno, sonicar a suspensão preferencialmente por uma sonda ultrassônica, mantendo a amostra no gelo (10 ciclos de pulso de 15 s, com intervalos de 30 s entre os pulsos para resfriar a amostra no gelo, em amplitude média com ciclo de 50% de dever).
- No caso dos órgãos do camundongo, homogeneize a suspensão usando um homogeneizador elétrico (começando com baixa força, e acelerando lentamente para a força média) mantendo a amostra no gelo. Lave regularmente o homogeneizador elétrico em PBS para remover qualquer material orgânico que entupi o dispositivo.
- Tire 20 μL das amostras e verifique sob o microscópio se as células do tecido homogeneizado estão devidamente destruídas; caso contrário, repita a homogeneização.
- Centrifugar os tubos em uma centrífuga de mesa a 10.000 x g por 30 min a 4 °C.
NOTA: Opcionalmente, centrifugar o supernante uma segunda vez a 20.000 x g por 30 min a 4 °C para eliminar pequenas frações da pelota inicial que podem ter sido transferidas. Isso simplificará a filtragem subsequente na etapa 2.3. - Colete o supernatante em um tubo fresco e coloque-o no gelo. Filtrar sequencialmente o supernasário usando unidades de filtro de seringa de 0,45 μm e 0,22 μm. Alíquotar o supernatante em lotes de 10 mL e congelá-los a -20 °C para armazenamento a curto prazo ou a -80 °C para armazenamento mais longo.
NOTA: A pré-filtragem com 0,45 μm remove a maioria das partículas antes que uma segunda etapa de filtragem com 0,22 μm remova as partículas mais finas. Usar diretamente o filtro de 0,22 μm pode causar o risco de entupir os filtros. - Prepare uma amostra de 50 μL para análise de SDS-PAGE/western blot adicionando 10 μL de buffer de amostra SDS de 5x (ver Tabela 1) a 40 μL do sobrenante e, em seguida, fervendo a 95 °C por 10 min.
- Opcionalmente, resuspense cerca de 100 μL de pelota obtida na etapa 2.2 em 900 μL de ddH2O, e prepare uma amostra para análise de SDS-PAGE/western blot conforme descrito acima.
NOTA: A inclusão de amostras derivadas de pelotas na análise da mancha ocidental, além do controle positivo, indicará se a expressão da proteína é baixa ou se o anticorpo é problemático.
- Opcionalmente, resuspense cerca de 100 μL de pelota obtida na etapa 2.2 em 900 μL de ddH2O, e prepare uma amostra para análise de SDS-PAGE/western blot conforme descrito acima.
3. Análise SDS-PAGE e western blot
NOTA: A análise da mancha ocidental é necessária para verificar se há solubilidade de proteínas. Descreve um protocolo para eletroblotting, usando um sistema de manchas de tanques úmidos (ver Tabela de Materiais). Um protocolo alternativo para SDS-PAGE pode ser encontrado em outro lugar19.
- Prepare um gel SDS-PAGE de poliacrilamida de 12,5% de poliacrilamida de acordo com as instruções do fabricante (ou seja, um gel de empilhamento em cima de um gel de resolução; consulte a Tabela 1). Execute as amostras previamente preparadas durante a etapa 2 (semelhante às etapas 4, 5 e 6; veja abaixo).
- Coloque uma escada marcador de proteína no primeiro poço (ver Tabela de Materiais). Carregar 5 ng de proteína recombinante hFAHD1 (obtida a partir de bactérias12; ver Tabela 1) como um controle positivo para o segundo poço.
- Posteriormente, carregue 20 μL da amostra a ser analisada e preencha todos os poços restantes com 20 μL de buffer de amostra SDS-PAGE 1x preparado (ou seja, tampão de amostra de 5x diluído com ddH2O). Execute os géis SDS-PAGE a 125 V usando o buffer de execução do SDS (ver Tabela 1).
- Após a conclusão do SDS-PAGE, realize uma análise de manchas ocidentais e teste as membranas usando o anticorpo disponível levantado contra o FAHD1 (ver Tabela 1).
NOTA: Como as amostras são retiradas da homogeneidade do tecido bruto, geralmente a qualidade do SDS-PAGE e a análise da mancha ocidental neste momento estão comprometidas; no entanto, é importante verificar se a proteína a extrair é solúvel no sobrenante. O protocolo a seguir foi testado para rins suínos, e diferentes órgãos do camundongo, incluindo fígado, coração, cérebro e rim.- Prepare o buffer de transferência de manchas ocidentais de 10x (ver Tabela 1). Prepare o tampão de transferência de manchas ocidentais 1x (ver Tabela 1) e esfrie-o a 4 °C.
- Ative uma membrana PVDF por 2 minutos em metanol. Lave a membrana em ddH2O por 2 min. Equilibre a membrana por 15 min em 1x tampão de transferência de manchas ocidentais.
- Lave o gel SDS com 1x PBS por 10 minutos enquanto treme para remover o buffer de execução do SDS e, em seguida, incubar o gel em 1x tampão de transferência de mancha ocidental por 10 minutos para equilíbrio. Monte o de eletroblotting (ou seja, combinando a membrana PVDF ativada e os géis) de acordo com as instruções do fabricante.
- Execute a mancha através de eletroblotting a 300 mA por 1 h em uma caixa de espuma de poliestireno cheia de gelo ou na sala fria (4 °C). Transfira a membrana PVDF para um tubo de 50 mL com seu lado exposto voltado para o lado interno do tubo. Incubar a membrana em 20 mL de tampão de bloqueio de manchas ocidentais (ver Tabela 1) durante a noite a 4 °C enquanto rola em um rolo de tubo (ver Tabela de Materiais).
- No dia seguinte, lave a membrana por 5 min com 20 mL de tampão de lavagem de manchas ocidentais (PBS com 0,1% (v/v) Interpol 20) no mesmo tubo enquanto estiver rolando. Incubar a membrana no mesmo tubo com o anticorpo primário2 (visando FAHD1; ver Tabela 1) diluído 1:500 no tampão de bloqueio de manchas ocidentais por 1h à temperatura ambiente enquanto rola.
- Lave a membrana no mesmo tubo três vezes por 10 min cada com 20 mL de tampão de lavagem de manchas ocidentais enquanto rola. Incubar a membrana por 30 minutos à temperatura ambiente com anticorpo secundário conjugado pelo HRP (ver Tabela de Materiais) diluído 1:3000 em 5 mL de tampão de bloqueio de manchas ocidentais.
- Lave a membrana no mesmo tubo três vezes por 10 minutos cada com 20 mL de tampão de lavagem de manchas ocidentais e duas vezes por 5 min cada com 1x PBS. Seque a membrana segurando-a cuidadosamente com pinças em uma borda, e tocando um pedaço de celulose ou um pedaço de papel Whatman com a borda oposta (inferior) da membrana. Coloque a membrana (lado exposto para cima) em uma placa de vidro limpa.
- Cubra cuidadosamente toda a membrana com 1 mL de substrato de mancha ocidental ECL preparado usando uma pipeta, tomando cuidado para não criar bolhas de ar. Deixe a solução ECL incubar por 3 minutos, e desenvolva imediatamente a membrana usando filme de raio-X ou usando um sistema de imagem.
NOTA: Se a proteína foi detectada em nenhuma das amostras, mas apenas no controle positivo, isso pode indicar que a proteína é insolúvel, ou não está presente em quantidades adequadas a serem detectadas pelo anticorpo. Se apenas nanogramas do controle positivo foram carregados, o primeiro cenário é mais provável. Se nenhuma proteína foi detectada, verifique a qualidade do anticorpo, e talvez mude para um anticorpo policlonal em vez de um anticorpo monoclonal. Em raras ocasiões, ou seja, para algumas proteínas hidrofóbicas, a proteína pode ser detectável após centrifugação, mas não após a filtragem. Nesse caso, recomenda-se o uso de unidades de filtros especiais para proteínas hidrofóbicas.
- Opcionalmente, colorir as membranas PVDF após a mancha ocidental para controlar a transferência bem sucedida da proteína do gel SDS-PAGE para a membrana PVDF.
NOTA: A coloração coomassie é recomendada para solução de problemas, desenvolvimento de métodos e documentação, mas lembre-se que após a aplicação deste protocolo, as membranas são perdidas para uma análise mais aprofundada da mancha ocidental. A coloração de Ponceau S dá manchas mais fracas, mas pode ser usada se as membranas forem ressar sondadas.- Prepare pequenas bandejas contendo as soluções de coloração (Coomassie ou Ponceau S) e desoxidagem.
- Usando pinças, coloque a membrana na solução de coloração e agite suavemente até que a membrana esteja bem manchada (5-10 min).
- Transfira a membrana para a solução de desestabilização e agite até que a solução esteja saturada (5-10 min). Repita o passo de desestabilização até que as bandas de proteína possam ser observadas na membrana; se nenhuma banda for observada, repita a mancha com um tempo de incubação mais longo. Seque a membrana colocando-a em uma placa de vidro usando pinças.
4. Teste: Precipitação de sulfato de amônio
NOTA: A precipitação de sulfato de amônio é um método de purificação de proteínas alterando a solubilidade da proteína. Em um experimento preliminar, a concentração de sulfato de amônio é sequencialmente aumentada para um valor que precipita uma quantidade máxima de contaminantes proteicos, deixando o FAHD1 em solução. A solubilidade da proteína é novamente sondada através da análise de manchas ocidentais.
- Proceder a partir da etapa 2.3: descongelar uma alíquota da amostra ou proceder diretamente após a extração de proteínas (ou seja, sem congelar a amostra). Filtre a amostra usando uma unidade de filtro de 0,22 μm para excluir possíveis precipitados após o descongelamento. Prepare seis tubos de 1,5 mL no gelo e transfira 250 μL de amostra para cada tubo.
- Prepare uma série de diluição de 5%, 10%, 15%, 20%, 25% e 30% de sulfato de amônio nos tubos preparados acima, e compor o volume final até 1000 μL com tampão de proteína. Incubar as amostras a 4 °C durante a noite em uma rotadora de tubos (ver Tabela de Materiais).
- Usando uma centrífuga de mesa, centrífuga a 10.000 x g por 30 min a 4 °C e transfira cuidadosamente todos os supernantes em tubos separados. Seque as pelotas resultantes e resuspenque cada uma delas em 1000 μL de ddH2O.
- Para cada par de pelotas resuspended e sobrenante da etapa anterior, misture 40 μL com 10 μL de tampão de amostra SDS de 5x e ferva a 95 °C com tampas abertas até que a maior parte do líquido tenha vaporizado. Em seguida, resuspense a pelota em uma mistura de 50% DMSO em ddH2O.
- Execute O SDS-PAGE (etapa 3), mas execute os géis a 80 V por 3 h. Para cada concentração de sulfato de amônio, carregue as amostras derivadas da pelota resuspended e sobrenante (passo 4.3) em pares. Realize uma análise de manchas ocidentais (etapa 3).
- Verifique se há a maior concentração de sulfato de amônio, no qual a proteína a ser purificada (ou seja, FAHD1) permanece na amostra derivada do sobrenante. Com base nos resultados, defina um protocolo de precipitação de sulfato de amônio para a proteína de interesse, a ser usado em experimentos futuros.
NOTA: O sulfato de amônio é bem conhecido por distorcer SDS-PAGE e mancha ocidental. À medida que a concentração de sulfato de amônio aumenta, a qualidade da análise da mancha ocidental será comprometida. No entanto, como na etapa 3 anterior, esta análise é utilizada para verificar a solubilidade da proteína de interesse em determinadas concentrações de sulfato de amônio. Este protocolo visa precipitar outras proteínas, enquanto a proteína a ser purificada deve permanecer solúvel.
5. Teste: cromatografia de troca iônica com FPLC
NOTA: Moléculas com grupos funcionais carregados estão vinculadas a uma coluna de partículas de sílica para FPLC, permitindo a diferenciação de proteínas de acordo com sua carga superficial. Execute esta etapa duas vezes, utilizando a coluna de troca cationic e a coluna de troca aniônica (ver Tabela de Materiais). As etapas de protocolo são as mesmas para cromatografia de troca cônica ou aniônica, mas os buffers a serem utilizados são diferentes (ver Tabela 1); ambos com "sal baixo" 15 mM NaCl e "sal alto" 1 M Condições naCl. Para as colunas utilizadas, recomenda-se uma taxa de fluxo de 1 mL/min.
- Configure o sistema FPLC com a coluna de troca aniônica ou cônica. Lave a coluna com 5 volumes de coluna (CVs) de 20% EtOH (em H2O), seguida por 5 CVs de ddH2O. Alternadamente, lave a coluna com 1 CV de tampão de sal baixo, tampão de sal alto e novamente tampão de sal baixo na ordem até que não sejam observados mais picos no cromatógrafo, mas lave pelo menos uma vez.
- Após determinar o protocolo ideal para precipitação de sulfato de amônio em pequena escala (etapa 4), aplique o protocolo de precipitação a 10 mL de homogeneato de tecido original (passo 2). Opcionalmente, dilam a amostra contra o tampão de sal baixo.
- Aplique a amostra na coluna (por exemplo, por injeção ou usando uma bomba de amostra) e colete o fluxo-through. Lave a coluna com 1 CV do tampão de sal baixo.
- Configure uma elução linear de gradiente de 100% baixo tampão de sal/ tampão de sal de 0% alto para 0% baixo tampão de sal/100% tampão de sal 100% alto dentro de 3 CVs. Colete continuamente frações de 1 mL. Após o gradiente ter terminado, continue a funcionar com o tampão de sal alto até que não sejam detectados picos associados à proteína (absorção UV a 280/255 nm) no cromatograma ao longo da faixa de 1 CV.
- Aplique 1 mL de 25% SDS dissolvido em 0,5 M NaOH (em ddH2O) para limpar a coluna. Consecutivamente, lave a coluna com 3 CVs de ddH2O e 3 CVs de 20% EtOH (em ddH2O).
- Colete amostras de SDS-PAGE de todas as frações de pico e o fluxo-through, e teste-as via mancha ocidental para a presença da proteína de interesse (passo 3). Congele as frações coletadas em nitrogênio líquido e armazene-as a -80 °C.
- Após a análise da mancha ocidental ser concluída, descongele e acumule as frações contendo a proteína de interesse e descarte as outras. Repita as etapas 5.1-5.5 com a coluna alternativa (ou seja, coluna de troca cationic ou aniônica).
- Após ambas as colunas terem sido sondadas, defina um protocolo FPLC para a proteína de interesse, a ser usado em experimentos futuros. Reduza o volume da solução proteica usando unidades de filtro de ultra-centrifugação (10 kDa, ver Tabela de Materiais) até 2 mL.
NOTA: Há dois resultados esperados desta série de experimentos. Ou a proteína de interesse se apegou a uma das colunas, e a solução proteica já é bastante pura após a eluição, ou a proteína permaneceu no fluxo em ambos os casos. Neste último cenário, embora a proteína esteja no fluxo, o efeito de limpeza desta etapa ainda pode ser significativo. Nesse caso, quanto ao FAHD1 no fígado de suíno e rato, esta etapa de troca iônica ainda será realizada. Se nem a coluna de troca cationic ou aniônica pode fornecer um efeito de limpeza adequado, pode-se tentar modificar o pH do lysate e tampão, e dilisar a amostra contra o buffer em execução antes da aplicação para FPLC.
6. Extração de proteínas utilizando sub-protocolos definidos para sulfato-precipitação de amônio e FPLC
NOTA: Partículas porosas em uma coluna de gel de sílica para FPLC (Ver Tabela de Materiais) permitem a diferenciação de proteínas de acordo com seu raio hidrodinâmico. As etapas descritas devem ser realizadas com um sistema FPLC, utilizando cromatografia de exclusão de tamanho (SEC). Para a coluna SEC utilizada (ver Tabela de Materiais), recomenda-se uma taxa de fluxo de 0,3 mL/min.
- Prepare todos os materiais necessários (ver passo 1) e extraia a proteína total do tecido (ver passo 2). Realize uma precipitação de sulfato de amônio com toda a homogeneagem tecidual que não foi utilizada para testes (ver passo 4). Para maiores volumes, concentre o lysate usando unidades de filtro de ultra-centrifugação (10 kDa; ver Tabela de Materiais) até um volume menor de 50 mL ou menos.
- Realize um primeiro passo de purificação usando cromatografia de troca iônica (ver passo 5).
- Prepare amostras para a mancha ocidental, como descrito nas etapas anteriores. Realize a análise da mancha ocidental e acumule todas as FAHD1 contendo frações da cromatografia de troca iônica.
- Reduza o volume da solução proteica para 2 mL usando unidades de filtro de ultra-centrifugação (10 kDa). Filtrar sequencialmente a solução com unidades de filtro de seringa de 0,45 μm e 0,22 μm para remover qualquer micro-precipitação.
- Equilibre a coluna SEC com 1 CV de buffer de execução SEC (ver Tabela 1), contendo 1 mM DTT. Coloque a amostra na coluna e execute a cromatografia até que todas as proteínas sejam eluvadas (1-2 CV).
- Colete frações de 1 mL do fluxo-through que corresponde a picos significativos no cromatógrama (absorção UV a 280/255 nm) e prepare 50 amostras de μL de cada fração coletada para análise de SDS-PAGE e análise de manchas ocidentais, conforme descrito nas etapas anteriores. Congele todas as frações usando nitrogênio líquido e armazene-as a -80 °C.
- Lavar consecutivamente a coluna SEC com 1 CV de ddH2O e 1 CV de 20% EtOH (em ddH2O). Realize a análise western-Blot e acumule todas as FRAções FAHD1 contendo frações. Reduza o volume da solução proteica até 2 mL usando unidades de filtro de ultra-centrifugação (10 kDa, ver Tabela de Materiais).
- Avalie a concentração de proteínas utilizando um kit comercial de ensaio BCA (ver Tabela de Materiais).
NOTA: O teor de pH e sal da fase móvel pode influenciar o perfil de eluição das proteínas globulares20. Condições ácidas ou básicas podem resultar em picos sendo menos definidos e aumento das interações proteína-matriz levando à retenção parcial de proteína na coluna20. Este efeito pode ser explorado para uma purificação adicional de proteínas. Uma repetição da etapa 6 com diferentes taxas de fluxo, pH e concentrações de sal pode aumentar a pureza da proteína20.
7. Coloração de prata
NOTA: A análise de coloração de prata dos géis SDS-PAGE é necessária para verificar se há contaminações de proteínas que podem não ser vistas com a coloração de Coomassie. O protocolo a seguir é uma entre muitas versões que podem ser encontradas na literatura21. Realize todas as etapas de incubação tremendo em uma bandeja de vidro limpa. Colete todos os líquidos contendo prata e formaldeído em um recipiente de resíduos especiais e descarte-os adequadamente.
- Incubar os géis SDS-PAGE na solução de fixação de manchas de prata (ver Tabela 1) durante a noite na sala fria. Incubar os géis na solução de incubação de manchas de prata (ver Tabela 1) por 3 h em temperatura ambiente. Opcionalmente, adicione glutaraldeído (ver Tabela 1) para melhorar a detecção de bandas fracas. Lave os géis quatro vezes em ddH2O por 10 min cada.
- Incubar os géis na solução prata de coloração prata (ver Tabela 1) por 1h.
NOTA: Lembre-se que a partir de agora todos os líquidos e o gel em si contêm prata e formaldeído que são tóxicos. - Incubar os géis na solução de desenvolvedor de coloração de prata (ver Tabela 1) com agitação vigorosa até que as bandas estejam claramente visíveis. Para impedir a reação, descarte a solução do desenvolvedor e incuba imediatamente os géis na solução de parada de coloração prateada (ver Tabela 1) por um mínimo de 10 minutos.
NOTA: As bandas manchadas nas etapas 7.2 e 7.3 se tornarão constantemente mais desenvolvidas. Adicionar mais formaldeído à solução do que o indicado pode ser necessário se a coloração for fraca.
Resultados
A proteína FAHD1 foi extraída do fígado de rim e camundongo suíno usando o protocolo apresentado. Para o tecido do rato, vários órgãos são necessários para obter vários μg após a etapa final de purificação. Por essa razão, este artigo se concentra na extração do FAHD1 dos rins suínos, que é um experimento muito mais exemplar. A extração de FAHD1 do fígado do camundongo é realizada para apresentar as dificuldades e possíveis armadilhas deste protocolo. É geralmente recomendado o uso de órgãos qu...
Discussão
Passos críticos no protocolo
Seguir diretrizes comuns para o manuseio de proteínas é essencial, como trabalhar no gelo e em condições moderadas de pH e sal. O uso de inibidores de protease é benéfico para o método, enquanto o uso de inibidores proteasso é altamente recomendado. Congelar e descongelar a amostra pode sempre resultar em precipitação proteica (pelo menos parcialmente), por isso qualquer alíquota descongelada de lisato de proteína inicial (etapa 2) deve ser processada continua...
Divulgações
Os autores não têm interesses financeiros concorrentes.
Agradecimentos
Os autores agradecem muito a assistência técnica de Ayse Öztürk e Eva Albertini. Os camundongos utilizados para a geração de tecido hepático foram mantidos sob a supervisão da Univ.-Doz. Dr. Pidder Jansen-Dürr (Instituto de Pesquisa de Envelhecimento Biomédico da Universidade de Innsbruck, Rennweg 10, 6020 Innsbruck, Áustria).
Materiais
| Name | Company | Catalog Number | Comments |
| 0.22 µm filter units | MERCK | SLGP033RS | Millex-HP, 0.22 µm, PES 33 mm, not steril |
| 0.45 µm filter units | MERCK | SLHP033NS | Millex-HP, 0.45 µm, PES 33 mm, not steril |
| 15 mL Falcon tubes | VWR | 734-0451 | centrifugal tubes |
| 50 mL Falcon tubes | VWR | 734-0448 | centrifugal tubes |
| 96-Well UV Microplate | Thermo-Fischer | 8404 | UV/VIS transparent flat-bottom 96 well plates |
| Acrylamide/Bis Solution (40%, 29:1 ratio) | BIO-RAD | #1610147 | 40% acrylamide/bis-acrylamide, 29:1 (3.3% crosslinker) solution for casting polyacrylamide gels |
| ÄKTA FPLC system | GE Healthcare Life Sciences / Cytiva | - | using the FPLC system by GE Healthcare; different custom versions exist; this work used the "ÄKTA pure" system |
| Amicon Ultra-15, PLGC Ultracel-PL Membran, 10 kDa | MERCK | UFC901024 | centrifigal filters for protein enrichment; 10 kDa molecular mass filter; 15 mL |
| Amicon Ultra-4, PLGC Ultracel-PL Membran, 10 kDa | MERCK | UFC801024 | centrifigal filters for protein enrichment; 10 kDa molecular mass filter; 4 mL |
| Ammonium sulfate powder | MERCK | A4418 | ammonium sulphate for molecular biology, ≥99.0% |
| Ammoniumpersulfat reagent grade, 98% | MERCK | 215589 | Catalyst for acrylamide gel polymerization. |
| Coomassie Brilliant blue R 250 | MERCK | 1125530025 | Coomassie Brilliant blue R 250 (C.I. 42660) for electrophoresis Trademark of Imperial Chemical Industries PLC. CAS 6104-59-2, pH 6.2 (10 g/l, H2O, 25 °C) |
| Dialysis tubing cellulose membrane | MERCK | D9277 | Cellulose membranes for the exchange of buffers via dialysis. |
| Eppendof tubes 1.5 mL | VWR | 525-1042 | microcentrifugal tubes; autoclaved |
| HiLoad 26/600 Superdex 75 pg | GE Healthcare Life Sciences / Cytiva | 28989334 | HiLoad Superdex 75 pg prepacked columns are for high-resolution size exclusion chromatography of recombinant proteins |
| Immun-Blot PVDF Membrane | BIO-RAD | #1620177 | PVDF membranes are protein blotting membranes optimized for fluorescent and multiplex fluorescent applications. |
| Mini Trans-Blot Electrophoretic Transfer Cell | BIO-RAD | #1703930 | Use the Mini Trans-Blot Cell for rapid blotting of Mini-PROTEAN precast and handcast gels. |
| Mini-PROTEAN Tetra Vertical Electrophoresis Cell for Mini Precast Gels | BIO-RAD | #1658004 | 4-gel vertical electrophoresis system, includes electrode assembly, companion running module, tank, lid with power cables, mini cell buffer dam. |
| Mono Q 10/100 GL | GE Healthcare Life Sciences / Cytiva | 17516701 | Mono Q columns are strong anion exchange chromatography columns for protein analysis or small scale, high resolution polishing of proteins. |
| Mono S 10/100 GL | GE Healthcare Life Sciences / Cytiva | 17516901 | Mono S columns are strong cation exchange chromatography columns for protein analysis or small scale high resolution polishing of proteins. |
| PageRuler Prestained Protein Ladder, 10 to 180 kDa | Thermo-Fischer | 26616 | A mixture of 10 blue-, orange-, and green-stained proteins (10 to 180 kDa) for use as size standards in protein electrophoresis (SDS-PAGE) and western blotting. |
| Pierce BCA Protein Assay Kit | Thermo-Fischer | 23225 | A two-component, high-precision, detergent-compatible protein assay for determination of protein concentration. |
| Sonifier 250; Ultrasonic Cell Disruptor w/ Converter | Branson | - | New models at https://www.emerson.com/documents/automation/brochure-sonifier-sfx250-sfx550-cell-disruptors-homogenizers-branson-en-us-168180.pdf |
| Swine Anti-Rabbit Immunoglobulins/HRP (affinity isolated) | Agilent Dako | P0399 | The antibody used for horseradish peroxidase conjugation reacts with rabbit immunoglobulins of all classes. |
| TEMED, 1,2-Bis(dimethylamino)ethane, TMEDA | MERCK | T9281 | TEMED (N,N,N′,N′-Tetramethylethylenediamine) is molecule which allows rapid polymerization of polyacrylamide gels. |
| Tube Roller | - | - | A general tube rotator roller; e.g. a new model at https://labstac.com/de/Mixer/Roller/c/71 |
| Tube Rotator | - | - | A general tube rotator wheel; e.g. a new model at https://labstac.com/de/Tube-Roller/p/MT123 |
| ULTRA-TURRAX; T 25 digital | IKA | 0003725000 | New models at https://www.ika.com/de/Produkte-Lab-Eq/Dispergierer-Dipergiergeraet-Homogenisierer-Homogenisator-csp-177/T-25-digital-ULTRA-TURRAX-cpdt-3725000/ |
Referências
- Pircher, H., et al. Identification of FAH domain-containing protein 1 (FAHD1) as oxaloacetate decarboxylase. Journal of Biological Chemistry. 290 (11), 6755-6762 (2015).
- Pircher, H., et al. Identification of human Fumarylacetoacetate Hydrolase Domain-containing Protein 1 (FAHD1) as a novel mitochondrial acylpyruvase. Journal of Biological Chemistry. 286 (42), 36500-36508 (2011).
- Kang, T. -. W., et al. Senescence surveillance of pre-malignant hepatocytes limits liver cancer development. Nature. 479 (7374), 547-551 (2011).
- Hong, H., Seo, H., Park, W., Kim, K. K. -. J. Sequence, structure and function-based classification of the broadly conserved FAH superfamily reveals two distinct fumarylpyruvate hydrolase subfamilies. Environmental Microbiology. 22 (1), 270-285 (2020).
- Timm, D. E., Mueller, H. A., Bhanumoorthy, P., Harp, J. M., Bunick, G. J. Crystal structure and mechanism of a carbon-carbon bond hydrolase. Structure. 7 (9), 1023-1033 (1999).
- Bateman, R. L., et al. Mechanistic inferences from the crystal structure of Fumarylacetoacetate Hydrolase with a bound phosphorus-based inhibitor. Journal of Biological Chemistry. 276 (18), 15284-15291 (2001).
- Weiss, A. K. H., et al. Structural basis for the bi-functionality of human oxaloacetate decarboxylase FAHD1. Biochemical Journal. 475 (22), 3561-3576 (2018).
- Etemad, S., et al. Oxaloacetate decarboxylase FAHD1 - a new regulator of mitochondrial function and senescence. Mechanisms of Ageing and Development. 177, 22-29 (2019).
- Weiss, A. K. H., et al. Regulation of cellular senescence by eukaryotic members of the FAH superfamily - A role in calcium homeostasis. Mechanisms of Ageing and Development. 190, 111284 (2020).
- Petit, M., Koziel, R., Etemad, S., Pircher, H., Jansen-Dürr, P. Depletion of oxaloacetate decarboxylase FAHD1 inhibits mitochondrial electron transport and induces cellular senescence in human endothelial cells. Experimental Gerontology. 92, 7-12 (2017).
- Wiley, C. D., et al. Mitochondrial dysfunction induces senescence with a distinct secretory phenotype. Cell Metabolism. 23 (2), 303-314 (2016).
- Weiss, A. K. H., et al. Expression, purification, crystallization, and enzyme assays of Fumarylacetoacetate Hydrolase Domain-containing proteins. Journal of Visualized Experiments: JoVE. (148), e59729 (2019).
- Weiss, A. K. H., et al. Inhibitors of Fumarylacetoacetate Hydrolase Domain Containing Protein 1 (FAHD1). Molcules. 26 (16), 5009 (2021).
- Mizutani, H., Kunishima, N. Purification, crystallization and preliminary X-ray analysis of the fumarylacetoacetase family member TTHA0809 from Thermus thermophilus HB8. Acta Crystallographica Section F Structural Biology and Crystallization Communications. 63 (9), 792-794 (2007).
- Lee, C. H. A simple outline of methods for protein isolation and purification. Endocrinology and Metabolism. 32 (1), 18-22 (2017).
- Amer, H. E. A. Purification of proteins: Between meaning and different methods). Proteomics Technologies and Applications. , (2019).
- Niu, L., Yuan, H., Gong, F., Wu, X., Wang, W. Protein extraction methods shape much of the extracted proteomes. Frontiers in Plant Science. 9, 802 (2018).
- Gordon, J. A. Use of vanadate as protein-phosphotyrosine phosphatase inhibitor. Methods in Enzymology. 201, 477-482 (1991).
- Gallagher, S. R. SDS-polyacrylamide gel electrophoresis (SDS-PAGE). Current Protocols in Essential Laboratory Techniques. , (2012).
- . Effect of pH on Protein Size Exclusion Chromatography Available from: https://www.agilent.com/cs/library/applications/5990-8138EN.pdf (2011)
- Sørensen, B. K., et al. Silver staining of proteins on electroblotting membranes and intensification of silver staining of proteins separated by polyacrylamide gel electrophoresis. Analytical Biochemistry. 304 (1), 33-41 (2002).
- Fagerberg, L., et al. Analysis of the human tissue-specific expression by genome-wide integration of transcriptomics and antibody-based proteomics. Molecular & Cellular Proteomics. 13 (2), 397-406 (2014).
- . Cytiva Life Fundamentals of size exclusion chromatography Available from: https://www.cytivalifesciences.com/en/us/solutions/protein-research/knowledge-center/protein-purification-methods/size-exclusion-chromatography (2022)
- Rosano, G. L., Ceccarelli, E. A. Recombinant protein expression in Escherichia coli: advances and challenges. Frontiers in Microbiology. 5, 172 (2014).
- Rosano, G. L., Morales, E. S., Ceccarelli, E. A. New tools for recombinant protein production in Escherichia coli: A 5-year update. Protein Science: A Publication of the Protein Society. 28 (8), 1412-1422 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados