Method Article
Monitoreo de la cinética de agregación de proteínas in vivo mediante el conteo automatizado de inclusión en Caenorhabditis elegans
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí, se presenta un método para el análisis de la cinética de agregación de proteínas en el nematodo Caenorhabditis elegans. Los animales de una población sincronizada por edad se visualizan en diferentes puntos de tiempo, seguidos de un recuento de inclusión semiautomatizado en CellProfiler y se ajustan a un modelo matemático en AmyloFit.
Resumen
La agregación de proteínas en inclusiones insolubles es un sello distintivo de una variedad de enfermedades humanas, muchas de las cuales están relacionadas con la edad. El nematodo Caenorhabditis elegans es un organismo modelo bien establecido que ha sido ampliamente utilizado en el campo para estudiar la agregación de proteínas y la toxicidad. Su transparencia óptica permite la visualización directa de la agregación de proteínas mediante microscopía de fluorescencia. Además, el rápido ciclo reproductivo y la corta vida útil hacen del nematodo un modelo adecuado para detectar genes y moléculas que modulan este proceso.
Sin embargo, la cuantificación de la carga agregada en animales vivos está mal estandarizada, generalmente se realiza mediante el conteo manual de inclusión bajo un microscopio de disección de fluorescencia en un solo punto de tiempo. Este enfoque puede resultar en una alta variabilidad entre los observadores y limita la comprensión del proceso de agregación. Por el contrario, la agregación de proteínas similares a los amiloides in vitro se controla rutinariamente mediante fluorescencia de tioflavina T de una manera altamente cuantitativa y resuelta en el tiempo.
Aquí, se presenta un método análogo para el análisis imparcial de la cinética de agregación en C. elegans vivos, utilizando un microscopio confocal de alto rendimiento combinado con análisis de imágenes personalizados y ajuste de datos. La aplicabilidad de este método se demuestra mediante el monitoreo de la formación de inclusión de una proteína de poliglutamina (polyQ) marcada fluorescentemente en las células musculares de la pared corporal. El flujo de trabajo de análisis de imágenes permite la determinación del número de inclusiones en diferentes puntos de tiempo, que se ajustan a un modelo matemático basado en eventos de nucleación independientes en células musculares individuales. El método descrito aquí puede resultar útil para evaluar los efectos de los factores de proteostasis y las posibles terapias para las enfermedades de agregación de proteínas en un animal vivo de una manera robusta y cuantitativa.
Introducción
La acumulación de proteínas mal plegadas en depósitos insolubles ocurre en una amplia gama de enfermedades. Ejemplos bien conocidos son la agregación de β amiloide y tau en la enfermedad de Alzheimer, α-sinucleína en la enfermedad de Parkinson y huntingtina con poliQ expandido en la enfermedad de Huntington 1,2. El plegamiento incorrecto de estos polipéptidos en fibrillas amiloides se asocia con toxicidad y muerte celular por mecanismos que aún no están claros. Dilucidar los mecanismos de formación de amiloide será crucial para desarrollar terapias efectivas, que actualmente no están disponibles.
Se han realizado investigaciones detalladas de la formación de amiloide in vitro basadas en mediciones de fluorescencia de tioflavina T, lo que lleva a una comprensión mecanicista del proceso de agregación y el efecto de las moléculas inhibitorias 3,4,5. Sin embargo, no está claro si los mismos mecanismos de agregación son válidos en el complejo entorno de las células y organismos vivos. El gusano nematodo Caenorhabditis elegans es un organismo modelo adecuado para estudiar la agregación de proteínas in vivo. Tiene una anatomía relativamente simple, pero consiste en múltiples tejidos, incluidos el músculo, el intestino y un sistema nervioso. Está genéticamente bien caracterizado, y las herramientas para la modificación genética están fácilmente disponibles. Además, tiene un corto tiempo de generación de ~ 3 días y una vida útil total de 2-3 semanas. Como tal, la agregación de proteínas se puede examinar a lo largo de la vida útil del animal en una escala de tiempo experimentalmente conveniente. Finalmente, el nematodo es ópticamente transparente, lo que permite el seguimiento de la agregación de proteínas marcadas fluorescentemente en animales vivos.
Estas características de C. elegans han sido explotadas previamente para investigar la agregación de proteínas polyQ como modelo para la expansión de Huntington y otras enfermedades de expansión polyQ. Por encima del umbral patogénico de 35-40 residuos de glutamina, se puede observar que las proteínas polyQ marcadas con proteína fluorescente amarilla (YFP) forman inclusiones insolubles en el tejido muscular 6,7, las neuronas8 y el intestino 9,10. Estas características se han utilizado ampliamente para detectar los genes 11,12,13 y los modificadores de moléculas pequeñas 14 de la agregación de proteínas y la toxicidad.
C. elegans tiene el potencial de desempeñar un papel importante en la reducción de la brecha entre los estudios in vitro de agregación de proteínas y los modelos de enfermedades más complejos como los ratones15. C. elegans es susceptible de cribadofarmacológico 16 , pero también puede ser explotado para obtener una comprensión fundamental de los mecanismos moleculares de la agregación de proteínas in vivo, como se demostró recientemente17. Sin embargo, para ambas aplicaciones, es de suma importancia extraer una medida cuantitativa y reproducible de la agregación de proteínas. Aquí, esto se logra con el uso de un microscopio confocal de alto rendimiento combinado con una tubería de análisis de imágenes dedicada (Figura 1).
Protocolo
1. Crecimiento de una población sincronizada por edad de C. elegans
- Mantener las cepas de C. elegans en placas de medio de crecimiento de nematodos (NGM) sembradas con Escherichia coli OP50 a 20 °C según los procedimientos estándar18.
- Realice una puesta de huevo sincronizada colocando 10 nematodos adultos en una placa NGM sembrada de 6 cm con una púa de gusano de platino. Deje que los adultos pongan huevos durante ~ 2 h a 20 ° C antes de retirarlos. Preparar 1-4 placas por cepa, dependiendo de la fertilidad de la cepa y el número de puntos de tiempo a tomar.
- Coloque las placas con huevos en la incubadora a 20 °C. Monitorear el desarrollo de los animales hasta que alcancen la edad adulta.
NOTA: El día en que los animales han alcanzado la edad adulta se define aquí como el día 1. Por lo general, esto es tres días después de la puesta del huevo. - A partir del día 1, transfiera a los animales a nuevas placas NGM sembradas diariamente para separarlos de sus crías. Para compensar a los animales que mueren o se pierden durante la transferencia, transfiera ~ 40 animales por cepa multiplicado por el número de puntos a fotografiar (consulte el paso 2). Proceda hasta que los animales hayan dejado de poner huevos fertilizados (~ día 6 de la edad adulta).
NOTA: Excluya a los animales con embolsado u otros fenotipos del desarrollo. El embolsado se observa comúnmente en cepas que expresan proteínas propensas a la agregación.
2. Preparación de muestras de C. elegans en una placa multipocillo
NOTA: Como el procedimiento de imágenes requiere anestésicos que eventualmente matarán a los animales, los mismos animales no se pueden reutilizar para puntos de tiempo posteriores. En cambio, se toman imágenes de diferentes animales del mismo lote sincronizado por edad en diferentes días. Aunque la mayoría de las cepas tendrán pocas inclusiones en el día 1, se recomienda incluir este punto de tiempo como línea de base.
- Prepare la placa de 384 pocillos llenando el número requerido de pocillos con 100 μL de tampón M9 complementado con 25 mM de NaN3 como anestésico. Llene un pozo por cepa para ser fotografiado.
NOTA: La azida de sodio (NaN3) es tóxica y debe manipularse con cuidado. - Para cada cepa, transfiera 20 animales a un pozo usando una púa de gusano de platino.
NOTA: Los gusanos deben colocarse fuera del césped bacteriano antes de colocarlos en el pozo. Las bacterias hacen que los animales se adhieran a la selección del gusano, lo que puede evitar su liberación y nublará el contenido del pozo. En general, 20 es el número óptimo de animales por pozo para evitar la superposición entre los gusanos al tiempo que limita las imágenes innecesarias del espacio vacío del pozo. - Cubra la placa con la tapa para evitar la evaporación y tome una imagen de la placa dentro de 1 h después de la preparación.
- Repita los pasos 2.1-2.3 diariamente hasta que se alcance una meseta constante en los números de inclusión o hasta que la mayoría de los animales hayan muerto. Realice la preparación de la muestra y la obtención de imágenes a la misma hora todos los días para garantizar intervalos de 24 h.
3. Adquisición de imágenes en el microscopio confocal de alto rendimiento
NOTA: Este experimento también se puede configurar en un microscopio confocal de disco giratorio regular con un soporte de placa multipozo. Una cámara con un gran campo de visión es beneficiosa para limitar el número de mosaicos necesarios para ser fotografiados para abarcar todo el pozo. Consulte la Tabla de materiales para obtener detalles sobre el microscopio y el software utilizados en este protocolo.
- Encienda el instrumento y abra el software.
- Inicie un nuevo protocolo yendo a Configuración de medición | Nuevo. Seleccione el tipo de placa multipozo correcto y haga clic en Crear una nueva configuración de medición.
- Configure el canal para la fluorescencia yendo al Ch 1. Establezca el objetivo en 10x. Seleccione 488 nm como fuente de luz y BP525/25 como filtro de emisión para obtener imágenes de YFP. Establezca el binning en 2x2 para reducir el tamaño del archivo.
- Haga clic en Agregar canal y seleccione Brightfield como método.
- Para agregar una imagen de fluorescencia confocal de pila z a la medición, elija Adquisición de fluorescencia 3D en Lista de acciones. Vaya a Seleccionar y elija Ch 1. Para minimizar el tamaño de los archivos, establezca Procesamiento de imágenes en Máximo para que se guarde la imagen de proyección máxima en lugar de la pila z completa.
- Haga clic en | de adquisición de BF/Ph Seleccionar | Ch 2 para el canal brightfield.
- Haga clic en el botón de reproducción (busque el símbolo del triángulo que apunta a la derecha) junto a Descargar placas de pozo y coloque la placa de 384 pocillos en el microscopio.
- En Adquisición de fluorescencia 3D, haga clic en Probar y seleccione un pozo que contenga gusanos para determinar la distancia de desplazamiento óptima a la que los gusanos están centrados correctamente. Establezca la distancia ascendente en 50 μm, la distancia descendente en -50 μm y el intervalo de corte en 2 μm para capturar todo el grosor de los animales en la pila z.
- Optimice el tiempo de exposición para obtener una buena intensidad de señal para las cuatro cepas y evite la saturación. Use el mismo tiempo de exposición para todas las cepas y puntos de tiempo.
- Seleccione los pozos que se van a visualizar en Configuración de escaneo de placas de pozos. Seleccione Mosaico y adquiera todo bien.
- Guarde la configuración de medición e inicie el experimento haciendo clic en Iniciar medición. Para los puntos de tiempo posteriores, abra el mismo ajuste de medición y ajuste la distancia de desplazamiento y los pozos que se van a visualizar.
4. Costura de imágenes en mosaico en ImageJ
NOTA: Este paso solo es necesario cuando se utiliza un objetivo mayor de 4x, para el cual la imagen de cada pozo se adquiere como múltiples baldosas. En este flujo de trabajo de análisis, la costura de las baldosas se realiza utilizando el software libre FIJI/ImageJ19 (Figura 2). Dependiendo del instrumento utilizado en el paso 3, también puede ser posible realizar costuras directamente en el software que lo acompaña.
- Descargue FIJI20 y ábralo.
- Ir a la | Plugins | de costura Costuras de rejilla/colección21.
- En la ventana emergente, Costura de cuadrícula/colección, seleccione el tipo y el orden en que se recopilaron los mosaicos. Elija Cuadrícula: fila por fila y Derecha y abajo.
- En la siguiente ventana, Costura de cuadrícula: Cuadrícula: fila por fila, Derecha y abajo, inserte el número de mosaicos en las direcciones x e y. Para el objetivo 10x utilizado aquí, elija 2 como el tamaño de cuadrícula x, 3 como el tamaño de cuadrícula y y 0 como la superposición de mosaico.
- Haga clic en Examinar y seleccione la carpeta que contiene las imágenes TIFF que se van a coser.
- Inserte el nombre de archivo común en Nombres de archivo para iconos, utilizando {i} en la posición del número de icono en cada nombre de archivo.
- Desmarque todas las casillas a continuación.
- Ejecute el complemento.
- Guarde las imágenes resultantes como archivos TIFF para su análisis en el siguiente paso.
5. Conteo automatizado de la inclusión usando CellProfiler22
- Descargue e instale el software de análisis de imágenes de código abierto, CellProfiler23. Descargue la canalización InclusionCounting.cpproj desde github.com/sinnigelab/aggregate-quantification.
- Abra CellProfiler y arrastre la canalización a la ventana Colocar un archivo de canalización aquí. Haga clic en Sí para cargar el proyecto.
- Haga clic en el módulo de entrada Imágenes y arrastre las imágenes cosidas a la ventana Colocar archivos y carpeta aquí.
- Haga clic en el módulo de entrada de metadatos . Ajuste la expresión regular para extraer del nombre del archivo de acuerdo con los nombres de las imágenes cosidas.
- Haga clic en el módulo de entrada NamesandTypes y ajuste Seleccione los criterios de la regla para que coincidan con los canales en los nombres de archivo.
NOTA: En la configuración predeterminada de la canalización, los nombres de archivo que contienen BF se reconocen como imágenes de campo brillante y se denominan gusanos. Los nombres de archivo que contienen YFP se reconocen como imágenes de fluorescencia y se denominan fluorescencia. - Haga clic en Ver configuración de salida para seleccionar una carpeta predeterminada para guardar la salida de CellProfiler.
- Haga clic en Iniciar modo de prueba para comprobar la configuración de la canalización utilizando el primer conjunto de datos de imágenes. Haga clic en Ejecutar para ejecutar todos los módulos de la canalización o paso para ejecutar a través de la canalización un módulo a la vez. Para ajustar los contornos del gusano en el módulo EditObjectsManually , haga clic en Ayuda para ver las instrucciones y haga clic en Listo para continuar ejecutando la canalización.
NOTA: Las mediciones extraídas no se exportarán mientras estén en modo de prueba. Es posible que sea necesario ajustar los parámetros de umbral para detectar gusanos e inclusiones en función de las cepas y la ampliación utilizadas. - Haga clic en Salir del modo de prueba y analizar imágenes.
- Abra la carpeta de salida para ver los archivos de salida. Abra las imágenes con el nombre de archivo original seguido de contornos para comprobar si los gusanos y las inclusiones se superpusieron correctamente.
Nota : el número de inclusiones por gusano se puede encontrar en el archivo denominado ExpandedWormObjects. Puede encontrar más información sobre las imágenes de entrada en el archivo denominado Imagen. Se puede seleccionar un resultado adicional en el módulo ExportToSpreadsheet en la canalización.
6. Ajuste global de los datos de recuento de inclusión utilizando AmyloFit5
NOTA: Este paso solo se puede realizar cuando se dispone de datos para múltiples concentraciones de proteínas. Para Q40-YFP, se ha creado previamente un conjunto de cuatro cepas con diferentes niveles de sobreexpresión en las células musculares de la pared corporal17. En otros casos, se deben generar nuevas cepas utilizando microinyección plásmida e integración genómica24.
- Vaya a la plataforma gratuita de ajuste en línea para la cinética de agregación AmyloFit25. Regístrese o inicie sesión con una cuenta existente.
NOTA: Se puede acceder a un extenso manual sobre cómo usar AmyloFit para obtener ayuda adicional. Consulte el enlace en la parte superior izquierda de la página web (después de iniciar sesión) para obtener más información. - Para comenzar a usar AmyloFit, asigne un nombre al proyecto y haga clic en Crear proyecto. Abra el proyecto haciendo clic en Abrir y cree una sesión dándole un nombre y haciendo clic en Crear y cargar sesión.
- Haga clic en Agregar datos y cargue el archivo que contiene el número promedio de inclusiones por animal, siguiendo los requisitos de formato de datos que se muestran en el panel izquierdo. Haga clic en Cargar nuevos datos.
- Omita los pasos de preprocesamiento, que no son necesarios para los datos de recuento de inclusión, estableciendo el número de puntos para promediar sobre el desplazamiento de punto cero y el número de puntos para promediar para la meseta a 0. Haga clic en Enviar. Repita este paso para cada concentración de proteína (es decir, cada columna en el archivo cargado).
- Seleccione Personalizado en el panel del modelo, escriba Ncells*(1-exp(-Kcell*m**(n)*(t-1))) en el cuadro de ecuación y haga clic en Cargar modelo.
NOTA: Como AmyloFit fue diseñado originalmente para el análisis de datos cinéticos de ensayos in vitro, se debe cargar un modelo personalizado para analizar los datos de recuento de inclusión de C. elegans. En la ecuación utilizada aquí, Ncélulas es el número de células en las que tiene lugar la formación de inclusión, la célula K es la constante de la tasa de nucleación, m la concentración de proteína intracelular y n el orden de reacción de nucleación. - Establezca los tipos de parámetros en Global Const para Nceldas, Global fit para Kcell y n, y Const para m. Establezca el valor de Ncélulas en 95 para las células musculares de la pared corporal y la suposición inicial para las células K y n a 1. Introduzca los valores de m para las diferentes cepas en el panel izquierdo.
NOTA: Las conjeturas iniciales no son relevantes para el modelo relativamente simple utilizado aquí. Para modelos más complejos, es beneficioso introducir una estimación de los valores esperados para acortar el tiempo de cálculo. - Deje el número de saltos de lavabo sin cambios y haga clic en Ajustar en el panel de ajuste.
- Extraiga el ajuste haciendo clic en Descargar datos y Ajustar.
NOTA: Los parámetros extraídos por el ajuste global del modelo se enumerarán en la esquina inferior derecha. Una gráfica de los datos y el ajuste se generará automáticamente en el panel superior derecho. Esta gráfica se puede extraer haciendo clic en Descargar pdf y personalizarla yendo a Mostrar opciones de parcela. Lacélula K tiene unidades de concentración de moléculas-n tiempo-1 célula-1. Para comparar valores con diferentes n, la célula K se puede convertir a la tasa de nucleación a una concentración de proteína dada multiplicándola por mn.
Resultados
El método aquí descrito (Figura 1) se utilizó para analizar la cinética de agregación de un constructo compuesto por 40 glutaminas fusionadas a YFP (Q40-YFP). La proteína se expresa bajo el control del promotor unc-54 , impulsando la expresión en las células musculares de la pared corporal. Como estos son relativamente grandes y fáciles de visualizar, el uso de un objetivo 10x es suficiente para resolver las inclusiones formadas por Q40-YFP en este tejido. Previamente se desarrollaron cuatro cepas (líneas A-D) que expresan la proteína en diferentes grados para evaluar la concentración-dependencia de la agregación poliQ in vivo17.
Las poblaciones sincronizadas por edad de las líneas A-D se generaron mediante una puesta de huevos de 2 h, seguida de una transferencia diaria una vez que la descendencia alcanzó la edad adulta. Desde el día 1 hasta el día 10 de la edad adulta, se tomaron imágenes de 20 animales de cada una de las cuatro cepas en una placa de 384 pocillos, utilizando un microscopio confocal de alto rendimiento. Las imágenes de los pozos se adquirieron como 6 azulejos, que se unieron utilizando un complemento en ImageJ21 (Figura 2). Las imágenes cosidas se analizaron posteriormente utilizando una tubería CellProfiler22 hecha a medida (Figura 3) para cuantificar el número promedio de inclusión por animal para cada cepa y punto de tiempo.
Los datos se ajustaron a un modelo matemático en AmyloFit5 (Figura 4). El modelo se basa en la suposición de que cada una de las 95 células musculares de la pared corporal adquiere independientemente una inclusión por un evento de nucleación limitante de la velocidad, seguido de un rápido crecimiento agregado17. El ajuste produjo una constante de tasa de nucleación de 9,9 × 105 moléculas M-2.1 d-1 celular-1 y un orden de reacción de 2,1, correspondiente a una tasa de nucleación de 0,38 moléculas d-1 célula-1 a una concentración de proteína intracelular de 1 mM. Dos réplicas biológicas independientes condujeron a valores estrechamente correspondientes para la velocidad de nucleación y el orden de reacción, que están de acuerdo con un estudio previo que utiliza un protocolo similar17 (Tabla 1).
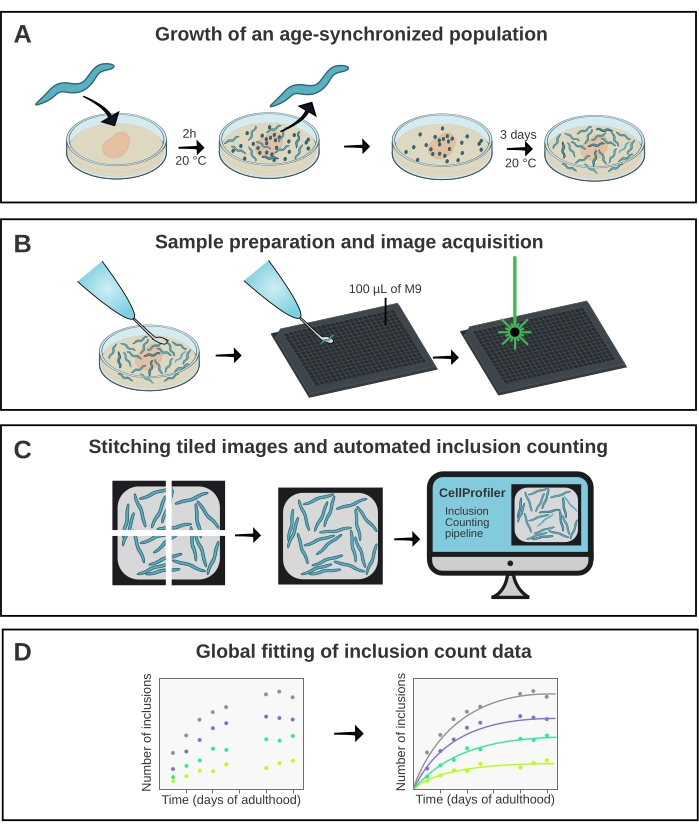
Figura 1: Descripción general esquemática del método. (A) Las poblaciones de C. elegans sincronizadas por edad se generan mediante una puesta de huevos cronometrada. (B) Los animales de la misma población son fotografiados en una placa de 384 pocillos en diferentes puntos de tiempo. (C) Las baldosas se unen para formar imágenes de pozos completos, que se analizan en CellProfiler para cuantificar los números de inclusión por animal. (D) Los datos se ajustan a un modelo matemático utilizando AmyloFit. Haga clic aquí para ver una versión más grande de esta figura.
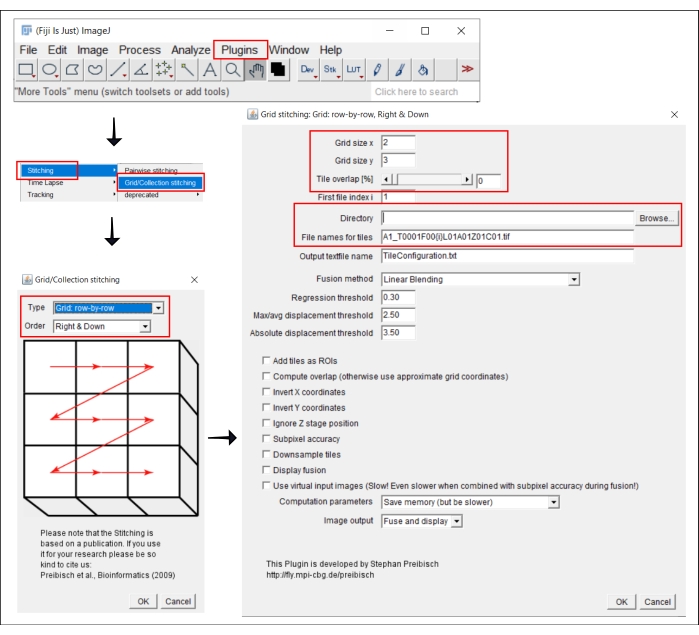
Figura 2: Capturas de pantalla del procedimiento de costura en ImageJ utilizando el plugin Grid/Collection stitching21. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Esquema de la canalización de CellProfiler para cuantificar los números de inclusiones. (A-C) La imagen de campo brillante (A) se utiliza para identificar los gusanos (B, primer plano en C). (D-G) La imagen de fluorescencia (D, primer plano en E) se utiliza para identificar las inclusiones (F). Los gusanos e inclusiones están relacionados para proporcionar el número de inclusiones para cada gusano en el pozo (G). Las imágenes mostradas son de animales de la línea A Q40 en el día 3 de la edad adulta. Haga clic aquí para ver una versión más grande de esta figura.
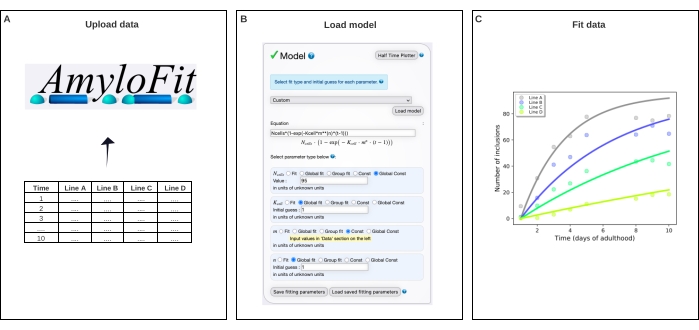
Figura 4: Ajuste de los datos a un modelo matemático en AmyloFit. (A) Los datos se cargan en AmyloFit. (B) Se introduce una ecuación personalizada para modelar la formación de inclusión, asumiendo eventos de nucleación independientes en cada célula. (C) Ajuste de la cinética de agregación para las líneas A-D de C. elegans que expresan diferentes niveles de Q40-YFP. Los datos son representativos de dos réplicas biológicas independientes. Haga clic aquí para ver una versión más grande de esta figura.
| Conjunto de datos 1 | Conjunto de datos 2 | Sinnige et al.17 | |
| n | 2.1 | 1.9 | 1.6 |
| Célula K (moléculas M-n d-1 célula-1) | 9,9 x 105 | 1,4 x 105 | 3,1 x 104 |
| Velocidad de nucleación a 1 mM (moléculas d-1 célula-1) | 0.38 | 0.21 | 0.35 |
Tabla 1: Valores de la velocidad de nucleación y orden de reacción de la agregación Q40-YFP. Datos para dos réplicas biológicas independientes del protocolo y comparación con datos publicados previamente17.
Discusión
El método presentado aquí facilita un análisis imparcial y cuantitativo de la cinética de agregación de proteínas en el organismo modelo C. elegans. Depende de cuatro elementos clave (Figura 1): 1) mantener una población de nematodos sincronizada por edad; 2) microscopía de fluorescencia en placas multipozo; 3) conteo automatizado de inclusión en CellProfiler; 4) ajuste de datos en AmyloFit. En comparación con el conteo manual de inclusiones en animales que se mueven libremente o de imágenes guardadas26, la cuantificación en CellProfiler es más rápida e imparcial. El otro avance clave del protocolo es la adquisición de datos cinéticos, en lugar de puntos de tiempo únicos, lo que proporciona información cuantitativa sobre el mecanismo de agregación al ajustar los datos a un modelo matemático.
Los cuatro elementos del protocolo se pueden utilizar como módulos independientes que se pueden modificar dependiendo de la aplicación. Las poblaciones sincronizadas por edad también se pueden mantener utilizando 5-fluoro-2′-desoxiuridina (FUDR) para esterilizar a los animales. Este compuesto afecta la vida útil y la proteostasis24,25 y es altamente cancerígeno para el experimentador; sin embargo, impide la transferencia manual de los gusanos, que puede ser intensiva en mano de obra cuando se manejan grandes cantidades. Otras alternativas son el uso de mutantes estériles29 o dispositivos de filtración para separar a la descendencia30.
El paso de microscopía de fluorescencia también se puede ajustar, por ejemplo, utilizando aumentos más altos para monitorear la agregación de proteínas en las neuronas. La microscopía de campo ancho puede ser suficiente para monitorear la agregación de polyQ en las células musculares cuando la diferencia relativa entre las condiciones es más importante que el número absoluto de inclusiones. La canalización de CellProfiler todavía se puede utilizar en estos casos, aunque el usuario deberá ajustar la configuración para reconocer gusanos e inclusiones. El rendimiento de la técnica está actualmente limitado por la necesidad de recoger manualmente los animales en la placa de 384 pocillos. Esto puede remediarse potencialmente mediante el uso de dispositivos microfluídicos16. La azida sódica es un anestésico relativamente duro, que podría ser sustituido por la inmovilización física con hidrogeles o perlas28,29.
El análisis en AmyloFit presentado aquí se basa en un mecanismo de agregación que consiste en eventos de nucleación independientes en células individuales. En los casos en que este modelo no encaje, el usuario deberá plantearse una alternativa como el modelo de agregación cooperativa desarrollado anteriormente17. Una limitación de este enfoque es que las cepas que expresan la proteína de interés deben estar disponibles a diferentes concentraciones, aunque estas pueden generarse utilizando métodos rutinarios de C. elegans 24.
En conjunto, este protocolo proporciona los medios para obtener datos de alta calidad para la cinética de agregación de proteínas en un sistema modelo in vivo , lo que permite un análisis detallado de los mecanismos de agregación17. Aunque el método se demostró para la agregación de polyQ en el tejido muscular de C. elegans , las aplicaciones futuras del protocolo pueden incluir otras proteínas y tejidos y los efectos de los factores de proteostasis y moléculas pequeñas.
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Agradecemos al laboratorio morimoto por las cepas de C. elegans y a Esmeralda Bosman por su asistencia en el microscopio confocal de alto rendimiento. Este trabajo fue financiado por una subvención inicial de la Universidad de Utrecht a T.S.
Materiales
| Name | Company | Catalog Number | Comments |
| 384-well plate | Greiner | 781091 | Black with flat clear bottom |
| AmyloFit | Knowles lab | v2.0 | Access at www.amylofit.ch.cam.ac.uk |
| C. elegans Q40 line A | Morimoto lab | AM1228 | Genotype rmIs404[unc-54p::Q40::YFP] |
| C. elegans Q40 line B | Morimoto lab | AM1229 | Genotype rmIs404[unc-54p::Q40::YFP] |
| C. elegans Q40 line C | Morimoto lab | AM1230 | Genotype rmIs404[unc-54p::Q40::YFP] |
| C. elegans Q40 line D | Morimoto lab | AM1231 | Genotype rmIs404[unc-54p::Q40::YFP] |
| CellProfiler | Broad Institute | 4.1.3 | Downloaded from https://cellprofiler.org |
| E. coli OP50 | Caenorhabditis Genetics Center (CGC) | OP50 | |
| FIJI | Open-source | (Fiji Is Just) ImageJ v2.1/1.5.3j | Downloaded from https://imagej.net/software/fiji/ |
| High-throughput confocal microscope | Yokogawa | CellVoyager CV7000S | |
| M9 buffer | Home-made | 3 g/L KH2PO4, 6 g/L Na2HPO4, 0.5 g/L NaCl, 1 mM MgSO4 | |
| NGM plates | Home-made | 17 g/L agar, 2.5 g/L bacto-peptone, 3 g/L NaCl, 25 mM KPO4 buffer pH 6.0, 1 mM MgSO4, 1 mM CaCl2, 5 mg/L cholesterol | |
| Pasteur pipette | WU Mainz | 250 | To make worm pick, 150 mm length |
| Platinum iridium wire | Alfa Aesar | 39383 | To make worm pick, 0.25 mm diameter |
| Sodium azide | Sigma-Aldrich | S2002 | |
| Stereomicroscope | Leica | S9 |
Referencias
- Knowles, T. P. J., Vendruscolo, M., Dobson, C. M. The amyloid state and its association with protein misfolding diseases. Nature Reviews. Molecular Cell Biology. 15 (6), 384-396 (2014).
- Chiti, F., Dobson, C. M. Protein misfolding, amyloid formation, and human disease: a summary of progress over the last decade. Annual Review of Biochemistry. 86 (1), 27-68 (2017).
- Knowles, T. P. J., et al. An analytical solution to the kinetics of breakable filament assembly. Science. 326 (5959), 1533-1537 (2009).
- Cohen, S. I. A., et al. Proliferation of amyloid-β42 aggregates occurs through a secondary nucleation mechanism. Proceedings of the National Academy of Sciences of the United States of America. 110 (24), 9758-9763 (2013).
- Meisl, G., et al. Molecular mechanisms of protein aggregation from global fitting of kinetic models. Nature Protocols. 11 (2), 252-272 (2016).
- Satyal, S. H., et al. Polyglutamine aggregates alter protein folding homeostasis in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 97 (11), 5750-5755 (2000).
- Morley, J. F., Brignull, H. R., Weyers, J. J., Morimoto, R. I. The threshold for polyglutamine-expansion protein aggregation and cellular toxicity is dynamic and influenced by aging in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 99 (16), 10417-10422 (2002).
- Brignull, H. R., Moore, F. E., Tang, S. J., Morimoto, R. I. Polyglutamine proteins at the pathogenic threshold display neuron-specific aggregation in a pan-neuronal Caenorhabditis elegans model. Journal of Neuroscience. 26 (29), 7597-7606 (2006).
- Moronetti Mazzeo, L. E., Dersh, D., Boccitto, M., Kalb, R. G., Lamitina, T. Stress and aging induce distinct polyQ protein aggregation states. Proceedings of the National Academy of Sciences of the United States of America. 109 (26), 10587-10592 (2012).
- Prahlad, V., Morimoto, R. I. Neuronal circuitry regulates the response of Caenorhabditis elegans to misfolded proteins. Proceedings of the National Academy of Sciences of the United States of America. 108 (34), 14204-14209 (2011).
- Nollen, E. A. A. Genome-wide RNA interference screen identifies previously undescribed regulators of polyglutamine aggregation. Proceedings of the National Academy of Sciences of the United States of America. 101 (17), 6403-6408 (2004).
- Silva, M. C., et al. A genetic screening strategy identifies novel regulators of the proteostasis network. PLoS Genetics. 7 (12), 1002438(2011).
- Brehme, M., et al. A chaperome subnetwork safeguards proteostasis in aging and neurodegenerative disease. Cell Reports. 9, 1-16 (2014).
- Calamini, B., et al. Small-molecule proteostasis regulators for protein conformational diseases. Nature Chemical Biology. 8 (2), 185-196 (2012).
- Sinnige, T., Stroobants, K., Dobson, C. M., Vendruscolo, M. Biophysical studies of protein misfolding and aggregation in in vivo models of Alzheimer's and Parkinson's diseases. Quarterly Reviews of Biophysics. 49, 22(2020).
- Mondal, S., et al. Large-scale microfluidics providing high-resolution and high-throughput screening of Caenorhabditis elegans poly-glutamine aggregation model. Nature Communications. 7, 13023(2016).
- Sinnige, T., et al. Kinetic analysis reveals that independent nucleation events determine the progression of polyglutamine aggregation in C. elegans. Proceedings of the National Academy of Sciences of the United States of America. 118 (11), 202188118(2021).
- Brenner, S. Caenorhabditis elegans. Methods. 77 (1), 71-94 (1974).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- FIJI/ImageJ. , Available from: https://imagej.net/downloads (2012).
- Preibisch, S., Saalfeld, S., Tomancak, P. Globally optimal stitching of tiled 3D microscopic image acquisitions. Bioinformatics. 25 (11), 1463-1465 (2009).
- Lamprecht, M. R., Sabatini, D. M., Carpenter, A. E. CellProfiler: free, versatile software for automated biological image analysis. BioTechniques. 42 (1), 71-75 (2007).
- CellProfiler. Broad Institute. , Available from: https://cellprofiler.org/releases (2021).
- Mello, C. C., Kramer, J. M., Stinchcomb, D., Ambros, V. Efficient gene transfer in C. elegans: extrachromosomal maintenance and integration of transforming sequences. EMBO Journal. 10 (12), 3959-3970 (1991).
- Knowles group, University of Cambridge. , Available from: https://amylofit.com/amylofitmain/login/ (2021).
- Lazaro-Pena, M. I., Cornwell, A. B., Samuelson, A. V. Quantifying tissue-specific proteostatic decline in Caenorhabditis elegans. Journal of Visualized Experiments: JoVE. (175), (2021).
- Feldman, N., Kosolapov, L., Ben-Zvi, A. Fluorodeoxyuridine improves Caenorhabditis elegans proteostasis independent of reproduction onset. PLoS One. 9 (1), 85964(2014).
- Brunquell, J., Bowers, P., Westerheide, S. D. Fluorodeoxyuridine enhances the heat shock response and decreases polyglutamine aggregation in an HSF-1-dependent manner in Caenorhabditis elegans. Mechanisms of Ageing and Development. 141-142, 1-4 (2014).
- David, D. C., et al. Widespread protein aggregation as an inherent part of aging in C. elegans. PLoS Biology. 8 (8), 47-48 (2010).
- Hunter, S., Maulik, M., Scerbak, C., Vayndorf, E., Taylor, B. E. Caenorhabditis sieve: A low-tech instrument and methodology for sorting small multicellular organisms. Journal of Visualized Experiments: JoVE. (137), (2018).
- Burnett, K., Edsinger, E., Albrecht, D. R. Rapid and gentle hydrogel encapsulation of living organisms enables long-term microscopy over multiple hours. Communications Biology. 1, 73(2018).
- Dong, L., et al. Reversible and long-term immobilization in a hydrogel-microbead matrix for high-resolution imaging of Caenorhabditis elegans and other small organisms. PLoS One. 13 (3), 0193989(2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados