Method Article
Überwachung der Proteinaggregationskinetik in vivo mittels automatisierter Einschlusszählung bei Caenorhabditis elegans
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier wird eine Methode zur Analyse der Proteinaggregationskinetik im Fadenwurm Caenorhabditis elegans vorgestellt. Tiere aus einer alterssynchronisierten Population werden zu verschiedenen Zeitpunkten abgebildet, gefolgt von einer halbautomatischen Inklusionszählung in CellProfiler und Anpassung an ein mathematisches Modell in AmyloFit.
Zusammenfassung
Die Proteinaggregation in unlösliche Einschlüsse ist ein Kennzeichen einer Vielzahl von menschlichen Krankheiten, von denen viele altersbedingt sind. Der Fadenfaden Caenorhabditis elegans ist ein etablierter Modellorganismus, der in diesem Bereich häufig zur Untersuchung der Proteinaggregation und -toxizität eingesetzt wurde. Seine optische Transparenz ermöglicht die direkte Visualisierung der Proteinaggregation durch Fluoreszenzmikroskopie. Darüber hinaus machen der schnelle Fortpflanzungszyklus und die kurze Lebensdauer den Fadenwurm zu einem geeigneten Modell, um nach Genen und Molekülen zu suchen, die diesen Prozess modulieren.
Die Quantifizierung der Gesamtbelastung bei lebenden Tieren ist jedoch schlecht standardisiert, typischerweise durch manuelle Einschlusszählung unter einem Fluoreszenzdissektionsmikroskop zu einem einzigen Zeitpunkt. Dieser Ansatz kann zu einer hohen Variabilität zwischen den Beobachtern führen und das Verständnis des Aggregationsprozesses einschränken. Im Gegensatz dazu wird die Amyloid-ähnliche Proteinaggregation in vitro routinemäßig durch Thioflavin-T-Fluoreszenz in einer hochquantitativen und zeitaufgelösten Weise überwacht.
Hier wird eine analoge Methode zur unvoreingenommenen Analyse der Aggregationskinetik in lebenden C. elegans vorgestellt, wobei ein konfokales Hochdurchsatzmikroskop in Kombination mit maßgeschneiderter Bildanalyse und Datenanpassung verwendet wird. Die Anwendbarkeit dieser Methode wird durch die Überwachung der Einschlussbildung eines fluoreszierend markierten Polyglutamins (PolyQ) -Proteins in den Körperwandmuskelzellen nachgewiesen. Der Bildanalyse-Workflow ermöglicht die Bestimmung der Anzahl der Einschlüsse zu verschiedenen Zeitpunkten, die an ein mathematisches Modell angepasst werden, das auf unabhängigen Keimbildungsereignissen in einzelnen Muskelzellen basiert. Die hier beschriebene Methode kann sich als nützlich erweisen, um die Auswirkungen von Proteostasefaktoren und potenziellen Therapeutika für Proteinaggregationskrankheiten bei einem lebenden Tier auf robuste und quantitative Weise zu bewerten.
Einleitung
Die Anreicherung von fehlgefalteten Proteinen in unlösliche Ablagerungen tritt bei einer Vielzahl von Krankheiten auf. Bekannte Beispiele sind die Aggregation von Amyloid-β und Tau bei der Alzheimer-Krankheit, α-Synuclein bei der Parkinson-Krankheit und Huntingtin mit expandiertem PolyQ bei der Huntington-Krankheit 1,2. Die Fehlfaltung dieser Polypeptide in Amyloidfibrillen ist mit Toxizität und Zelltod durch Mechanismen verbunden, die noch weitgehend unklar sind. Die Aufklärung der Mechanismen der Amyloidbildung wird entscheidend für die Entwicklung wirksamer Therapien sein, die derzeit nicht verfügbar sind.
Detaillierte Untersuchungen der Amyloidbildung wurden in vitro auf der Grundlage von Thioflavin-T-Fluoreszenzmessungen durchgeführt, was zu einem mechanistischen Verständnis des Aggregationsprozesses und der Wirkung hemmender Moleküleführte 3,4,5. Es ist jedoch nicht klar, ob die gleichen Aggregationsmechanismen in der komplexen Umgebung lebender Zellen und Organismen gelten. Der Fadenwurm Caenorhabditis elegans ist ein geeigneter Modellorganismus, um die Proteinaggregation in vivo zu untersuchen. Es hat eine relativ einfache Anatomie, besteht aber aus mehreren Geweben, einschließlich Muskeln, Darm und einem Nervensystem. Es ist genetisch gut charakterisiert, und Werkzeuge zur genetischen Veränderung sind leicht verfügbar. Darüber hinaus hat es eine kurze Generationszeit von ~ 3 Tagen und eine Gesamtlebensdauer von 2-3 Wochen. So kann die Proteinaggregation über die gesamte Lebensdauer des Tieres auf einer experimentell günstigen Zeitskala untersucht werden. Schließlich ist der Fadenwurm optisch transparent, was die Verfolgung der Aggregation fluoreszierend markierter Proteine in lebenden Tieren ermöglicht.
Diese Eigenschaften von C. elegans wurden zuvor ausgenutzt, um die Aggregation von PolyQ-Proteinen als Modell für Huntington- und andere PolyQ-Expansionskrankheiten zu untersuchen. Oberhalb der pathogenen Schwelle von 35-40 Glutaminresten kann beobachtet werden, dass die mit gelb fluoreszierendem Protein (YFP) markierten PolyQ-Proteine unlösliche Einschlüsse im Muskelgewebe6,7, Neuronen8 und im Darm 9,10 bilden. Diese Merkmale wurden häufig verwendet, um nach Genen 11,12,13 und niedermolekularen Modifikatoren 14 der Proteinaggregation und -toxizität zu suchen.
C. elegans hat das Potenzial, eine wichtige Rolle bei der Überbrückung der Lücke zwischen In-vitro-Studien zur Proteinaggregation und komplexeren Krankheitsmodellen wie Mäusenzu spielen 15. C. elegans ist für das Wirkstoff-Screening16 zugänglich, kann aber auch genutzt werden, um ein grundlegendes Verständnis der molekularen Mechanismen der Proteinaggregation in vivo zu erlangen, wie kürzlichgezeigt wurde 17. Für beide Anwendungen ist es jedoch von größter Bedeutung, ein quantitatives und reproduzierbares Maß für die Proteinaggregation zu extrahieren. Dies wird hier durch den Einsatz eines konfokalen Hochdurchsatzmikroskops in Kombination mit einer dedizierten Bildanalyse-Pipeline erreicht (Abbildung 1).
Protokoll
1. Wachstum einer alterssynchronen Population von C. elegans
- Pflegen Sie die C. elegans-Stämme auf NGM-Platten (Nematoden-Wachstumsmedium), die mit Escherichia coli OP50 ausgesät sind, bei 20 °C gemäß Standardverfahren18.
- Führen Sie eine synchronisierte Eiablage durch, indem Sie 10 erwachsene Nematoden mit einem Platinwurmpickel auf eine 6 cm große NGM-Platte legen. Lassen Sie die Erwachsenen ~ 2 h bei 20 ° C Eier legen, bevor Sie sie entfernen. Bereiten Sie 1-4 Platten pro Stamm vor, abhängig von der Fruchtbarkeit der Sorte und der Anzahl der zu nehmenden Zeitpunkte.
- Legen Sie die Platten mit Eiern in den Inkubator bei 20 °C. Überwachen Sie die Entwicklung der Tiere bis zum Erwachsenenalter.
HINWEIS: Der Tag, an dem die Tiere das Erwachsenenalter erreicht haben, wird hier als Tag 1 definiert. Typischerweise ist dies drei Tage nach der Eiablage. - Bringen Sie die Tiere ab Tag 1 täglich auf neu ausgesäte NGM-Platten, um sie von ihren Nachkommen zu trennen. Um Tiere zu kompensieren, die während des Transfers sterben oder verloren gehen, übertragen Sie ~ 40 Tiere pro Stamm multipliziert mit der Anzahl der abzubildenden Punkte (siehe Schritt 2). Fahren Sie fort, bis die Tiere aufgehört haben, befruchtete Eier zu legen (~ Tag 6 des Erwachsenenalters).
HINWEIS: Tiere mit Sack oder anderen Entwicklungsphänotypen ausschließen. Bagging wird häufig in Stämmen beobachtet, die aggregationsanfällige Proteine exprimieren.
2. Probenvorbereitung von C. elegans in einer Multiwell-Platte
HINWEIS: Da das bildgebende Verfahren Anästhetika erfordert, die die Tiere schließlich töten, können dieselben Tiere nicht für nachfolgende Zeitpunkte wiederverwendet werden. Stattdessen werden verschiedene Tiere aus derselben alterssynchronisierten Charge an verschiedenen Tagen abgebildet. Obwohl die meisten Stämme an Tag 1 nur wenige Einschlüsse haben, wird empfohlen, diesen Zeitpunkt als Basislinie anzugeben.
- Bereiten Sie die 384-Well-Platte vor, indem Sie die erforderliche Anzahl von Wells mit 100 μL M9-Puffer füllen, ergänzt durch 25 mMNaN 3 als Anästhetikum. Füllen Sie eine Vertiefung pro Sorte, die abgebildet werden soll.
HINWEIS: Natriumazid (NaN3) ist giftig und sollte mit Vorsicht behandelt werden. - Übertragen Sie für jeden Stamm 20 Tiere mit einem Platin-Wurmpickel in einen Brunnen.
HINWEIS: Die Würmer müssen außerhalb des Bakterienrasens platziert werden, bevor sie in den Brunnen gelegt werden. Bakterien bringen die Tiere dazu, sich an den Wurmpickel zu halten, was ihre Freisetzung verhindern kann und den Brunneninhalt trübt. Im Allgemeinen ist 20 die optimale Anzahl von Tieren pro Bohrloch, um Überlappungen zwischen den Würmern zu vermeiden und gleichzeitig unnötige Bildgebung von leerem Brunnenraum zu begrenzen. - Decken Sie die Platte mit dem Deckel ab, um eine Verdunstung zu verhindern, und stellen Sie die Platte innerhalb von 1 Stunde nach der Vorbereitung ab.
- Wiederholen Sie die Schritte 2.1-2.3 täglich, bis ein stetiges Plateau in den Einschlusszahlen erreicht ist oder bis die meisten Tiere gestorben sind. Führen Sie die Probenvorbereitung und Bildgebung jeden Tag zur gleichen Zeit durch, um Intervalle von 24 Stunden zu gewährleisten.
3. Bildaufnahme auf dem Hochdurchsatz-Konfokalmikroskop
HINWEIS: Dieses Experiment kann auch auf einem normalen konfokalen Scheibenmikroskop mit einem Multiwell-Plattenhalter eingerichtet werden. Eine Kamera mit einem großen Sichtfeld ist vorteilhaft, um die Anzahl der Kacheln zu begrenzen, die abgebildet werden müssen, um den gesamten Brunnen zu überspannen. Siehe die Tabelle der Materialien für Details über das Mikroskop und die Software, die in diesem Protokoll verwendet werden.
- Schalten Sie das Gerät ein und öffnen Sie die Software.
- Starten Sie ein neues Protokoll, indem Sie zu Measurement Settings | gehen Neu. Wählen Sie den richtigen Multiwell-Plattentyp aus und klicken Sie auf Neue Messeinstellung erstellen.
- Richten Sie den Kanal für die Fluoreszenz ein, indem Sie zu Ch 1 gehen. Setzen Sie das Ziel auf 10x. Wählen Sie 488 nm als Lichtquelle und BP525/25 als Emissionsfilter, um YFP abzubilden. Legen Sie Binning auf 2x2 fest, um die Dateigröße zu reduzieren.
- Klicken Sie auf Kanal hinzufügen und wählen Sie Brightfield als Methode aus.
- Um der Messung ein konfokales Z-Stack-Fluoreszenzbild hinzuzufügen, wählen Sie unter Aktionsliste die Option 3D-Fluoreszenzerfassung aus. Gehen Sie zu Auswählen und wählen Sie Ch 1. Um Dateigrößen zu minimieren, legen Sie Bildverarbeitung auf Maximum fest, damit das maximale Projektionsbild und nicht der vollständige Z-Stack gespeichert wird.
- Klicken Sie auf BF/Ph-Erfassung | | auswählen Ch 2 für den Hellfeldkanal.
- Klicken Sie auf den Play-Button (achten Sie auf das nach rechts zeigende Dreieckssymbol) neben Unload Well Plates und legen Sie die 384-Well-Platte in das Mikroskop.
- Klicken Sie unter 3D-Fluoreszenzerfassung auf Test und wählen Sie einen Brunnen aus, der Würmer enthält, um den optimalen Schaltabstand zu bestimmen, in dem die Würmer richtig zentriert sind. Stellen Sie den aufsteigenden Abstand auf 50 μm, den absteigenden Abstand auf -50 μm und das Schnittintervall auf 2 μm ein, um die gesamte Dicke der Tiere im Z-Stapel zu erfassen.
- Optimieren Sie die Belichtungszeit, um eine gute Signalintensität für alle vier Stämme zu erhalten und gleichzeitig eine Sättigung zu vermeiden. Verwenden Sie die gleiche Belichtungszeit für alle Dehnungen und Zeitpunkte.
- Wählen Sie unter Well Plate Scan Setting (Bohrlochplatten-Scaneinstellung) die zu bebildernden Vertiefungen aus. Wählen Sie Kachel und Ganzes gut erfassen.
- Speichern Sie die Messeinstellung, und starten Sie das Experiment, indem Sie auf Messung starten klicken. Öffnen Sie für nachfolgende Zeitpunkte dieselbe Messeinstellung und passen Sie den Schaltabstand und die abzubildenden Vertiefungen an.
4. Zusammenfügen von gekachelten Bildern in ImageJ
HINWEIS: Dieser Schritt ist nur erforderlich, wenn ein Objektiv größer als 4x verwendet wird, für das das Bild jedes Bohrlochs als mehrere Kacheln erfasst wird. In diesem Analyse-Workflow erfolgt das Stitching der Kacheln mit der kostenlosen Software FIJI/ImageJ19 (Abbildung 2). Je nachdem, welches Instrument in Schritt 3 verwendet wird, kann es auch möglich sein, das Nähen direkt in der begleitenden Software durchzuführen.
- Laden Sie FIJI20 herunter und öffnen Sie es.
- Gehe zu Plugins | Nähen | Raster-/Sammlungsnähte21.
- Wählen Sie im Popup-Fenster Raster/Sammelheft den Typ und die Reihenfolge aus, in der die Kacheln gesammelt wurden. Wählen Sie Raster: Zeile für Zeile und rechts und unten.
- Fügen Sie im nächsten Fenster, Rasterzusammenfügung: Raster: Zeile für Zeile, Nach rechts und unten, die Anzahl der Kacheln in x- und y-Richtung ein. Wählen Sie für das hier verwendete 10-fache Ziel 2 als Rastergröße x, 3 als Rastergröße y und 0 als Kachelüberlappung aus.
- Klicken Sie auf Durchsuchen und wählen Sie den Ordner aus, der die zu stitchenden TIFF-Bilder enthält.
- Fügen Sie den allgemeinen Dateinamen unter Dateinamen für Kacheln ein, indem Sie {i} an der Position der Kachelnummer in jedem Dateinamen verwenden.
- Deaktivieren Sie alle Kontrollkästchen unten.
- Führen Sie das Plugin aus.
- Speichern Sie die resultierenden Bilder als TIFF-Dateien zur Analyse im nächsten Schritt.
5. Automatisierte Einschlusszählung mit CellProfiler22
- Laden Sie die Open-Source-Bildanalysesoftware CellProfiler23 herunter und installieren Sie sie. Laden Sie die Pipeline InclusionCounting.cpproj von github.com/sinnigelab/aggregate-quantification herunter.
- Öffnen Sie CellProfiler, und ziehen Sie die Pipeline in das Fenster Pipeline-Datei hier ablegen . Klicken Sie auf Ja, um das Projekt zu laden.
- Klicken Sie auf das Eingabemodul Bilder und ziehen Sie die zusammengefügten Bilder in das Fenster Dateien und Ordner hier ablegen.
- Klicken Sie auf das Metadaten-Eingabemodul. Passen Sie den regulären Ausdruck an, um aus dem Dateinamen entsprechend den Namen der zusammengefügten Bilder zu extrahieren.
- Klicken Sie auf das Eingabemodul NamesandTypes und passen Sie Wählen Sie die Regelkriterien aus, um sie an die Kanäle in den Dateinamen anzupassen.
HINWEIS: In den Standardeinstellungen der Pipeline werden Dateinamen, die BF enthalten, als Hellfeldbilder erkannt und als Würmer bezeichnet. Dateinamen, die YFP enthalten, werden als Fluoreszenzbilder erkannt und als Fluoreszenz bezeichnet. - Klicken Sie auf Ausgabeeinstellungen anzeigen , um einen Standardordner auszuwählen, in dem die Ausgabe von CellProfiler gespeichert werden soll.
- Klicken Sie auf Testmodus starten , um die Einstellungen der Pipeline anhand des ersten Imaging-Datensatzes zu überprüfen. Klicken Sie auf Ausführen , um alle Module in der Pipeline auszuführen, oder auf Schritt , um die Pipeline Modul für Modul zu durchlaufen. Um die Wurmkonturen im Modul EditObjectsManually anzupassen, klicken Sie auf Hilfe , um die Anweisungen anzuzeigen, und klicken Sie auf Fertig , um die Pipeline weiter auszuführen.
HINWEIS: Die extrahierten Messungen werden im Testmodus nicht exportiert. Die Schwellenwertparameter zur Erkennung von Würmern und Einschlüssen müssen möglicherweise basierend auf den verwendeten Dehnungen und Vergrößerungen angepasst werden. - Klicken Sie auf Testmodus beenden und analysieren Sie Bilder.
- Öffnen Sie den Ausgabeordner, um die Ausgabedateien anzuzeigen. Öffnen Sie die Bilder mit dem ursprünglichen Dateinamen, gefolgt von Umrissen , um zu überprüfen, ob die Würmer und Einschlüsse korrekt überlagert wurden.
HINWEIS: Die Anzahl der Einschlüsse pro Wurm finden Sie in der Datei ExpandedWormObjects. Weitere Informationen zu den Eingabebildern finden Sie in der Datei Image. Zusätzliche Ausgabe kann im ExportToSpreadsheet-Modul in der Pipeline ausgewählt werden.
6. Globale Anpassung der Inklusionszähldaten mit AmyloFit5
HINWEIS: Dieser Schritt kann nur durchgeführt werden, wenn Daten für mehrere Proteinkonzentrationen verfügbar sind. Für Q40-YFP wurde zuvorein Satz von vier Stämmen mit unterschiedlichen Überexpressionsniveaus in den Körperwandmuskelzellen erstellt. In anderen Fällen sollten neuartige Stämme mittels Plasmid-Mikroinjektion und genomischer Integrationerzeugt werden 24.
- Besuchen Sie die kostenlose Online-Anpassungsplattform für AggregationskinetikAmyloFit 25. Registrieren Sie sich entweder oder melden Sie sich mit einem bestehenden Konto an.
HINWEIS: Auf ein umfangreiches Handbuch zur Verwendung von AmyloFit kann zugegriffen werden, um weitere Hilfe zu erhalten. Weitere Informationen finden Sie unter dem Link oben links auf der Webseite (nach dem Login). - Um AmyloFit zu verwenden, benennen Sie das Projekt und klicken Sie auf Projekt erstellen. Öffnen Sie das Projekt, indem Sie auf Öffnen klicken und eine Sitzung erstellen, indem Sie ihm einen Namen geben und auf Sitzung erstellen und laden klicken.
- Klicken Sie auf Daten hinzufügen und laden Sie die Datei mit der durchschnittlichen Anzahl von Einschlüssen pro Tier gemäß den im linken Bereich angezeigten Datenformatanforderungen hoch. Klicken Sie auf Neue Daten laden.
- Überspringen Sie die Vorverarbeitungsschritte, die für die Einschlusszählungsdaten nicht erforderlich sind, indem Sie die Anzahl der Punkte auf Mittelwert über für den Nullpunkt-Offset und die Anzahl der Punkte auf den Mittelwert für das Plateau auf 0 festlegen. Klicken Sie auf Senden. Wiederholen Sie diesen Schritt für jede Proteinkonzentration (d. h. für jede Spalte in der hochgeladenen Datei).
- Wählen Sie im Modellbedienfeld die Option Benutzerdefiniert aus, geben Sie Ncells*(1-exp(- Kcell*m**(n)*(t-1))) in das Gleichungsfeld ein und klicken Sie auf Modell laden.
HINWEIS: Da AmyloFit ursprünglich für die Analyse kinetischer Daten aus In-vitro-Assays entwickelt wurde, muss ein maßgeschneidertes Modell geladen werden, um die Einschlusszähldaten von C. elegans zu analysieren. In der hier verwendeten Gleichung ist N Zellen die Anzahl der Zellen, in denen die Einschlussbildung stattfindet, K-Zelle ist die Keimbildungsrate konstant, m die intrazelluläre Proteinkonzentration und n die Reaktionsreihenfolge der Keimbildung. - Legen Sie die Parametertypen für n Zellen auf Global Const, für Kcell und n global fit und für m auf Const fest. Setzen Sie den Wert von N Zellen auf 95 für Körperwandmuskelzellen unddie Anfangsschätzung für K-Zellen und n auf 1. Geben Sie die Werte von m für die verschiedenen Dehnungen im linken Bereich ein.
HINWEIS: Erste Vermutungen sind für das hier verwendete relativ einfache Modell nicht relevant. Bei komplexeren Modellen ist es sinnvoll, eine Schätzung der Erwartungswerte einzugeben, um die Berechnungszeit zu verkürzen. - Lassen Sie die Anzahl der Beckenhüpfer unverändert, und klicken Sie im Fitting-Panel auf Anpassen .
- Extrahieren Sie die Passform, indem Sie auf Daten herunterladen und anpassen klicken.
HINWEIS: Die Parameter, die durch die globale Anpassung des Modells extrahiert werden, werden in der unteren rechten Ecke aufgelistet. Ein Diagramm der Daten und der Anpassung wird automatisch im oberen rechten Bereich generiert. Dieses Diagramm kann extrahiert werden, indem Sie auf PDF herunterladen klicken, und angepasst werden, indem Sie zu Plotoptionen anzeigen gehen. K-Zelle hat Einheiten von Molekülkonzentration-n Zeit-1 Zelle-1. Um Werte mit verschiedenen n zu vergleichen, kann die K-Zelle in die Keimbildungsrate bei einer gegebenen Proteinkonzentration umgewandelt werden, indem sie mit mn multipliziert wird.
Ergebnisse
Das hier beschriebene Verfahren (Abbildung 1) wurde verwendet, um die Aggregationskinetik eines Konstrukts zu analysieren, das 40 zu YFP (Q40-YFP) verschmolzene Glutamine umfasst. Das Protein wird unter der Kontrolle des unc-54-Promotors exprimiert, der die Expression in den Muskelzellen der Körperwand antreibt. Da diese relativ groß und leicht zu visualisieren sind, reicht die Verwendung eines 10-fachen Objektivs aus, um die von Q40-YFP in diesem Gewebe gebildeten Einschlüsse aufzulösen. Vier Stämme (Linien A-D) wurden zuvor entwickelt, die das Protein in unterschiedlichem Ausmaß exprimieren, um die Konzentrationsabhängigkeit der PolyQ-Aggregation in vivo17 zu beurteilen.
Alterssynchronisierte Populationen der Linien A-D wurden durch eine 2-stündige Eiablage erzeugt, gefolgt von einem täglichen Transfer, sobald der Nachwuchs das Erwachsenenalter erreicht hatte. Von Tag 1 bis Tag 10 des Erwachsenenalters wurden 20 Tiere aus jedem der vier Stämme in einer 384-Well-Platte mit einem konfokalen Hochdurchsatzmikroskop abgebildet. Die Bilder der Bohrlöcher wurden als 6 Kacheln aufgenommen, die mit einem Plugin in ImageJ21 zusammengefügt wurden (Abbildung 2). Die zusammengefügten Bilder wurden anschließend mit einer maßgeschneiderten CellProfiler22-Pipeline (Abbildung 3) analysiert, um die durchschnittliche Einschlusszahl pro Tier für jeden Stamm und Zeitpunkt zu quantifizieren.
Die Daten wurden dann in AmyloFit 5 an ein mathematisches Modell angepasst (Abbildung 4). Das Modell basiert auf der Annahme, dass jede der 95 Körperwandmuskelzellen unabhängig voneinander einen Einschluss durch ein geschwindigkeitsbegrenzendes Keimbildungsereignis erhält, gefolgt von einem schnellen aggregierten Wachstum17. Die Anpassung ergab eine Keimbildungsratenkonstante von 9,9 × 105 Molekülen M-2,1 d-1 Zell-1 und eine Reaktionsordnung von 2,1, was einer Keimbildungsrate von 0,38 Molekülen d-1 Zell-1 bei einer intrazellulären Proteinkonzentration von 1 mM entspricht. Zwei unabhängige biologische Replikate führten zu sehr ähnlichen Werten für die Keimbildungsrate und die Reaktionsreihenfolge, die mit einer früheren Studie mit einem ähnlichen Protokoll17 übereinstimmen (Tabelle 1).
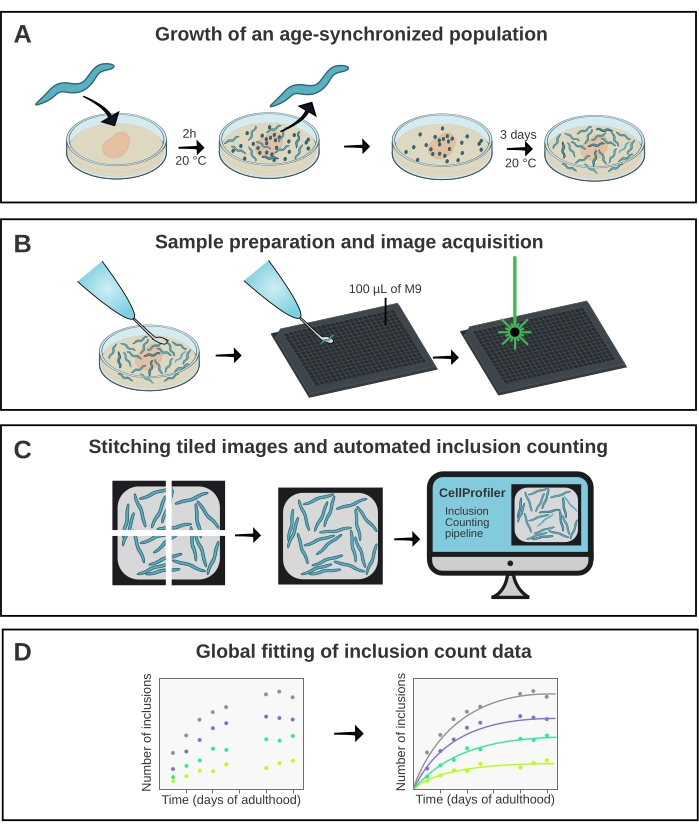
Abbildung 1: Schematische Übersicht über die Methode. (A) Alterssynchronisierte C. elegans-Populationen werden durch eine zeitgesteuerte Eiablage erzeugt. (B) Tiere aus derselben Population werden zu unterschiedlichen Zeitpunkten in einer 384-Well-Platte abgebildet. (C) Die Kacheln werden zusammengefügt, um Bilder der gesamten Vertiefungen zu bilden, die in CellProfiler analysiert werden, um die Einschlusszahlen pro Tier zu quantifizieren. (D) Die Daten werden mit AmyloFit an ein mathematisches Modell angepasst. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
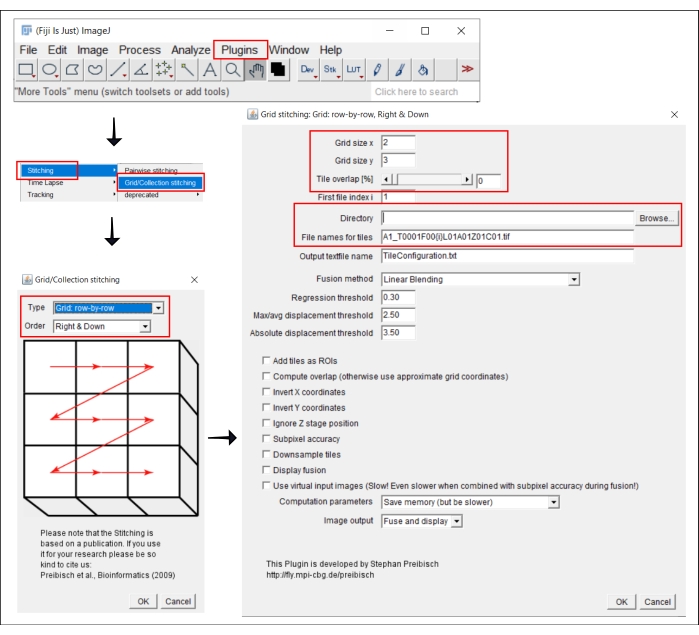
Abbildung 2: Screenshots des Stitching-Verfahrens in ImageJ mit dem Plugin Grid/Collection stitching21. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 3: Schematische Darstellung der CellProfiler-Pipeline zur Quantifizierung der Einschlusszahlen. (A-C) Das Hellfeldbild (A) wird verwendet, um die Würmer zu identifizieren (B, Nahaufnahme in C). (D-G) Das Fluoreszenzbild (D, Nahaufnahme in E) wird verwendet, um die Einschlüsse (F) zu identifizieren. Die Würmer und Einschlüsse beziehen sich auf die Anzahl der Einschlüsse für jeden Wurm im Brunnen (G). Die gezeigten Bilder zeigen Q40 Linie A Tiere am 3. Tag des Erwachsenenalters. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
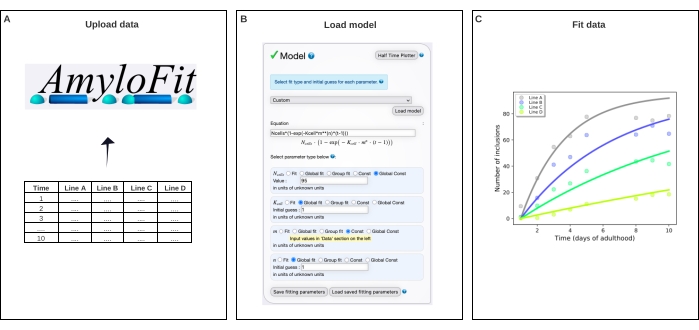
Abbildung 4: Anpassen der Daten an ein mathematisches Modell in AmyloFit. (A) Die Daten werden in AmyloFit hochgeladen. (B) Eine benutzerdefinierte Gleichung wird eingegeben, um die Einschlussbildung zu modellieren, wobei unabhängige Keimbildungsereignisse in jeder Zelle angenommen werden. (C) Anpassung der Aggregationskinetik für C. elegans Linien A-D, die unterschiedliche Konzentrationen von Q40-YFP ausdrücken. Die Daten sind repräsentativ für zwei unabhängige biologische Replikate. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
| Datensatz 1 | Datensatz 2 | Sinnige et al.17 | |
| n | 2.1 | 1.9 | 1.6 |
| K-Zelle (Moleküle M-n d-1 Zelle-1) | 9,9 x 105 | 1,4 x 105 | 3,1 x 104 |
| Keimbildungsrate bei 1 mM (Moleküle d-1 Zelle-1) | 0.38 | 0.21 | 0.35 |
Tabelle 1: Werte der Keimbildungsrate und Reaktionsreihenfolge der Q40-YFP-Aggregation. Daten für zwei unabhängige biologische Replikate des Protokolls und Vergleich mit zuvor veröffentlichten Daten17.
Diskussion
Die hierin vorgestellte Methode ermöglicht eine unvoreingenommene und quantitative Analyse der Proteinaggregationskinetik im Modellorganismus C. elegans. Es hängt von vier Schlüsselelementen ab (Abbildung 1): 1) Aufrechterhaltung einer alterssynchronisierten Population von Nematoden; 2) Fluoreszenzmikroskopie in Multiwell-Platten; 3) automatisierte Einschlusszählung in CellProfiler; 4) Datenanpassung in AmyloFit. Im Vergleich zur manuellen Zählung von Einschlüssen in frei beweglichen Tieren oder aus gespeicherten Bildern26 ist die Quantifizierung in CellProfiler sowohl schneller als auch unvoreingenommener. Die andere wichtige Weiterentwicklung des Protokolls ist die Erfassung kinetischer Daten anstelle von einzelnen Zeitpunkten, die quantitative Einblicke in den Aggregationsmechanismus bei der Anpassung der Daten an ein mathematisches Modell liefert.
Die vier Elemente des Protokolls können als unabhängige Module verwendet werden, die je nach Anwendung modifiziert werden können. Alterssynchronisierte Populationen können auch mit 5-Fluor-2'-Desoxyuridin (FUDR) aufrechterhalten werden, um die Tiere zu sterilisieren. Diese Verbindung beeinflusst die Lebensdauer und Proteostase24,25 und ist für den Experimentator stark krebserregend; Es schließt jedoch eine manuelle Übertragung der Würmer aus, die arbeitsintensiv sein kann, wenn große Mengen gehandhabt werden. Weitere Alternativen sind der Einsatz von sterilen Mutanten29 oder Filtergeräten zur Trennung von Nachkommen30.
Der Fluoreszenzmikroskopie-Schritt kann auch eingestellt werden, indem beispielsweise höhere Vergrößerungen verwendet werden, um die Proteinaggregation in Neuronen zu überwachen. Die Weitfeldmikroskopie kann ausreichen, um die PolyQ-Aggregation in Muskelzellen zu überwachen, wenn der relative Unterschied zwischen den Bedingungen wichtiger ist als die absolute Anzahl der Einschlüsse. Die CellProfiler-Pipeline kann in diesen Fällen weiterhin verwendet werden, obwohl die Einstellungen zur Erkennung von Würmern und Einschlüssen vom Benutzer angepasst werden müssen. Der Durchsatz der Technik ist derzeit durch die Notwendigkeit der manuellen Kommissionierung der Tiere in die 384-Well-Platte begrenzt. Dies kann möglicherweise durch den Einsatz von mikrofluidischen Gerätenbehoben werden 16. Natriumazid ist ein relativ hartes Anästhetikum, das durch physikalische Immobilisierung mit Hydrogelen oder Perlen ersetzt werden könnte28,29.
Die hier vorgestellte Analyse in AmyloFit basiert auf einem Aggregationsmechanismus, der aus unabhängigen Keimbildungsereignissen in einzelnen Zellen besteht. In Fällen, in denen dieses Modell nicht passt, sollte der Benutzer eine Alternative in Betracht ziehen, z. B. das zuvor entwickelte kooperativeAggregationsmodell 17. Eine Einschränkung dieses Ansatzes besteht darin, dass Stämme, die das interessierende Protein in verschiedenen Konzentrationen ausdrücken, verfügbar sein müssen, obwohl diese mit routinemäßigen C. elegans-Methoden erzeugt werden können24.
Insgesamt bietet dieses Protokoll die Möglichkeit, qualitativ hochwertige Daten für die Proteinaggregationskinetik in einem In-vivo-Modellsystem zu erhalten, das eine detaillierte Analyse der Aggregationsmechanismen ermöglicht17. Obwohl die Methode für die PolyQ-Aggregation im Muskelgewebe von C. elegans demonstriert wurde, können zukünftige Anwendungen des Protokolls andere Proteine und Gewebe sowie die Auswirkungen von Proteostasefaktoren und kleinen Molekülen umfassen.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Wir danken dem Morimoto-Labor für C. elegans-Stämme und Esmeralda Bosman für die Unterstützung beim konfokalen Hochdurchsatzmikroskop. Diese Arbeit wurde durch ein Start-up-Stipendium der Universität Utrecht an T.S.
Materialien
| Name | Company | Catalog Number | Comments |
| 384-well plate | Greiner | 781091 | Black with flat clear bottom |
| AmyloFit | Knowles lab | v2.0 | Access at www.amylofit.ch.cam.ac.uk |
| C. elegans Q40 line A | Morimoto lab | AM1228 | Genotype rmIs404[unc-54p::Q40::YFP] |
| C. elegans Q40 line B | Morimoto lab | AM1229 | Genotype rmIs404[unc-54p::Q40::YFP] |
| C. elegans Q40 line C | Morimoto lab | AM1230 | Genotype rmIs404[unc-54p::Q40::YFP] |
| C. elegans Q40 line D | Morimoto lab | AM1231 | Genotype rmIs404[unc-54p::Q40::YFP] |
| CellProfiler | Broad Institute | 4.1.3 | Downloaded from https://cellprofiler.org |
| E. coli OP50 | Caenorhabditis Genetics Center (CGC) | OP50 | |
| FIJI | Open-source | (Fiji Is Just) ImageJ v2.1/1.5.3j | Downloaded from https://imagej.net/software/fiji/ |
| High-throughput confocal microscope | Yokogawa | CellVoyager CV7000S | |
| M9 buffer | Home-made | 3 g/L KH2PO4, 6 g/L Na2HPO4, 0.5 g/L NaCl, 1 mM MgSO4 | |
| NGM plates | Home-made | 17 g/L agar, 2.5 g/L bacto-peptone, 3 g/L NaCl, 25 mM KPO4 buffer pH 6.0, 1 mM MgSO4, 1 mM CaCl2, 5 mg/L cholesterol | |
| Pasteur pipette | WU Mainz | 250 | To make worm pick, 150 mm length |
| Platinum iridium wire | Alfa Aesar | 39383 | To make worm pick, 0.25 mm diameter |
| Sodium azide | Sigma-Aldrich | S2002 | |
| Stereomicroscope | Leica | S9 |
Referenzen
- Knowles, T. P. J., Vendruscolo, M., Dobson, C. M. The amyloid state and its association with protein misfolding diseases. Nature Reviews. Molecular Cell Biology. 15 (6), 384-396 (2014).
- Chiti, F., Dobson, C. M. Protein misfolding, amyloid formation, and human disease: a summary of progress over the last decade. Annual Review of Biochemistry. 86 (1), 27-68 (2017).
- Knowles, T. P. J., et al. An analytical solution to the kinetics of breakable filament assembly. Science. 326 (5959), 1533-1537 (2009).
- Cohen, S. I. A., et al. Proliferation of amyloid-β42 aggregates occurs through a secondary nucleation mechanism. Proceedings of the National Academy of Sciences of the United States of America. 110 (24), 9758-9763 (2013).
- Meisl, G., et al. Molecular mechanisms of protein aggregation from global fitting of kinetic models. Nature Protocols. 11 (2), 252-272 (2016).
- Satyal, S. H., et al. Polyglutamine aggregates alter protein folding homeostasis in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 97 (11), 5750-5755 (2000).
- Morley, J. F., Brignull, H. R., Weyers, J. J., Morimoto, R. I. The threshold for polyglutamine-expansion protein aggregation and cellular toxicity is dynamic and influenced by aging in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 99 (16), 10417-10422 (2002).
- Brignull, H. R., Moore, F. E., Tang, S. J., Morimoto, R. I. Polyglutamine proteins at the pathogenic threshold display neuron-specific aggregation in a pan-neuronal Caenorhabditis elegans model. Journal of Neuroscience. 26 (29), 7597-7606 (2006).
- Moronetti Mazzeo, L. E., Dersh, D., Boccitto, M., Kalb, R. G., Lamitina, T. Stress and aging induce distinct polyQ protein aggregation states. Proceedings of the National Academy of Sciences of the United States of America. 109 (26), 10587-10592 (2012).
- Prahlad, V., Morimoto, R. I. Neuronal circuitry regulates the response of Caenorhabditis elegans to misfolded proteins. Proceedings of the National Academy of Sciences of the United States of America. 108 (34), 14204-14209 (2011).
- Nollen, E. A. A. Genome-wide RNA interference screen identifies previously undescribed regulators of polyglutamine aggregation. Proceedings of the National Academy of Sciences of the United States of America. 101 (17), 6403-6408 (2004).
- Silva, M. C., et al. A genetic screening strategy identifies novel regulators of the proteostasis network. PLoS Genetics. 7 (12), 1002438(2011).
- Brehme, M., et al. A chaperome subnetwork safeguards proteostasis in aging and neurodegenerative disease. Cell Reports. 9, 1-16 (2014).
- Calamini, B., et al. Small-molecule proteostasis regulators for protein conformational diseases. Nature Chemical Biology. 8 (2), 185-196 (2012).
- Sinnige, T., Stroobants, K., Dobson, C. M., Vendruscolo, M. Biophysical studies of protein misfolding and aggregation in in vivo models of Alzheimer's and Parkinson's diseases. Quarterly Reviews of Biophysics. 49, 22(2020).
- Mondal, S., et al. Large-scale microfluidics providing high-resolution and high-throughput screening of Caenorhabditis elegans poly-glutamine aggregation model. Nature Communications. 7, 13023(2016).
- Sinnige, T., et al. Kinetic analysis reveals that independent nucleation events determine the progression of polyglutamine aggregation in C. elegans. Proceedings of the National Academy of Sciences of the United States of America. 118 (11), 202188118(2021).
- Brenner, S. Caenorhabditis elegans. Methods. 77 (1), 71-94 (1974).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- FIJI/ImageJ. , Available from: https://imagej.net/downloads (2012).
- Preibisch, S., Saalfeld, S., Tomancak, P. Globally optimal stitching of tiled 3D microscopic image acquisitions. Bioinformatics. 25 (11), 1463-1465 (2009).
- Lamprecht, M. R., Sabatini, D. M., Carpenter, A. E. CellProfiler: free, versatile software for automated biological image analysis. BioTechniques. 42 (1), 71-75 (2007).
- CellProfiler. Broad Institute. , Available from: https://cellprofiler.org/releases (2021).
- Mello, C. C., Kramer, J. M., Stinchcomb, D., Ambros, V. Efficient gene transfer in C. elegans: extrachromosomal maintenance and integration of transforming sequences. EMBO Journal. 10 (12), 3959-3970 (1991).
- Knowles group, University of Cambridge. , Available from: https://amylofit.com/amylofitmain/login/ (2021).
- Lazaro-Pena, M. I., Cornwell, A. B., Samuelson, A. V. Quantifying tissue-specific proteostatic decline in Caenorhabditis elegans. Journal of Visualized Experiments: JoVE. (175), (2021).
- Feldman, N., Kosolapov, L., Ben-Zvi, A. Fluorodeoxyuridine improves Caenorhabditis elegans proteostasis independent of reproduction onset. PLoS One. 9 (1), 85964(2014).
- Brunquell, J., Bowers, P., Westerheide, S. D. Fluorodeoxyuridine enhances the heat shock response and decreases polyglutamine aggregation in an HSF-1-dependent manner in Caenorhabditis elegans. Mechanisms of Ageing and Development. 141-142, 1-4 (2014).
- David, D. C., et al. Widespread protein aggregation as an inherent part of aging in C. elegans. PLoS Biology. 8 (8), 47-48 (2010).
- Hunter, S., Maulik, M., Scerbak, C., Vayndorf, E., Taylor, B. E. Caenorhabditis sieve: A low-tech instrument and methodology for sorting small multicellular organisms. Journal of Visualized Experiments: JoVE. (137), (2018).
- Burnett, K., Edsinger, E., Albrecht, D. R. Rapid and gentle hydrogel encapsulation of living organisms enables long-term microscopy over multiple hours. Communications Biology. 1, 73(2018).
- Dong, L., et al. Reversible and long-term immobilization in a hydrogel-microbead matrix for high-resolution imaging of Caenorhabditis elegans and other small organisms. PLoS One. 13 (3), 0193989(2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten