Method Article
Caenorhabditisエレガンスにおける自動包接計数を用いた生体内のタンパク質凝集動態のモニタリング
要約
ここでは、線虫 Caenorhabditis elegansにおけるタンパク質凝集動態の解析のための方法を提示する。年齢同期された集団の動物は、異なる時点で画像化され、続いてCellProfilerで半自動包接カウントが行われ、AmyloFitの数学的モデルに適合します。
要約
不溶性封入体へのタンパク質凝集は、様々なヒト疾患の特徴であり、その多くは加齢に関連している。線虫 Caenorhabditis elegans は、タンパク質凝集および毒性を研究するために現場で広く使用されている十分に確立されたモデル生物である。その光透過性は、蛍光顕微鏡によるタンパク質凝集の直接可視化を可能にする。さらに、速い生殖サイクルと短い寿命により、線虫はこのプロセスを調節する遺伝子や分子をスクリーニングするのに適したモデルになります。
しかしながら、生きている動物における凝集体負荷の定量は、あまり標準化されておらず、典型的には、単一の時点で蛍光解剖顕微鏡下で手動包接計数によって行われる。このアプローチでは、オブザーバー間のばらつきが大きくなり、集計プロセスの理解が制限される可能性があります。対照的に、 インビトロでの アミロイド様タンパク質凝集は、チオフラビンT蛍光によって高度に定量的かつ時間分解された様式で日常的にモニターされる。
ここでは、カスタムメイドの画像解析とデータフィッティングを組み合わせたハイスループット共焦点顕微鏡を使用して、生きている C. elegansにおける凝集動力学の偏りのない解析のために同様の方法を提示する。この方法の適用可能性は、体壁筋細胞における蛍光標識ポリグルタミン(polyQ)タンパク質の包接形成をモニタリングすることによって実証される。画像解析ワークフローは、個々の筋肉細胞における独立した核生成事象に基づく数学的モデルに適合される異なる時点での封入物の数の決定を可能にする。ここに記載される方法は、生きている動物におけるタンパク質凝集疾患に対するタンパク質分解因子および潜在的な治療法の効果を堅牢かつ定量的な方法で評価するのに有用であることが証明され得る。
概要
ミスフォールディングされたタンパク質の不溶性沈着物への蓄積は、広範囲の疾患で起こる。よく知られている例は、アルツハイマー病におけるアミロイドβおよびタウの凝集、パーキンソン病におけるα-シヌクレイン、およびハンチントン病における拡張polyQを有するハンチンチン1,2である。これらのポリペプチドのアミロイド線維へのミスフォールディングは、依然としてほとんど不明であるメカニズムによる毒性および細胞死と関連している。アミロイド形成のメカニズムを解明することは、現在利用できない効果的な治療法を開発する上で極めて重要です。
チオフラビンT蛍光測定に基づいてアミロイド形成の詳細な調査がin vitroで行われており、凝集プロセスおよび阻害分子の効果の機構的理解につながっている3、4、5。しかし、同じ凝集機構が生細胞や生物の複雑な環境にも当てはまるかどうかは明らかではない。線虫Caenorhabditis elegansは、インビボでのタンパク質凝集を研究するのに好適なモデル生物である。それは比較的単純な解剖学を持っていますが、筋肉、腸、神経系を含む複数の組織で構成されています。それは遺伝的によく特徴付けられており、遺伝子組み換えのためのツールは容易に入手可能である。さらに、それは〜3日の短い生成時間と2〜3週間の総寿命を有する。そのため、タンパク質凝集は、実験的に都合のよい時間スケールで動物の寿命にわたって調べることができる。最後に、線虫は光学的に透明であり、生きている動物における蛍光標識タンパク質の凝集の追跡を可能にする。
C. elegansのこれらの特徴は、ハンチントンおよび他のpolyQ拡張疾患のモデルとしてのpolyQタンパク質の凝集を調べるために以前に利用されてきた。35〜40グルタミン残基の病原性閾値を超えると、黄色蛍光タンパク質(YFP)で標識されたpolyQタンパク質が、筋肉組織6、7、ニューロン8、および腸9、10において不溶性封入体を形成することが観察され得る。これらの特徴は、タンパク質凝集および毒性の遺伝子11、12、13および小分子修飾剤14についてスクリーニングするために広く使用されている。
C. elegansは 、タンパク質凝集の インビトロ 研究とマウスなどのより複雑な疾患モデルとの間のギャップを埋める上で重要な役割を果たす可能性を秘めている15。 C. elegans は薬物スクリーニング16 に順応可能であるが、最近実証されたように、 インビボでのタンパク質凝集の分子機構の基本的な理解を得るために利用することもできる17。しかし、どちらの用途でも、タンパク質凝集の定量的かつ再現性のある測定値を抽出することが最も重要です。ここでは、ハイスループットの共焦点顕微鏡と専用の画像解析パイプラインを組み合わせることでこれを実現します(図1)。
プロトコル
1. C. elegansの年齢同期人口の増加
- C . elegans 株を線虫増殖培地(NGM)プレート上に 、大腸菌 OP50を播種し、標準的な手順18に従って20°Cに維持する。
- プラチナワームピックで6cmの播種NGMプレート上に10匹の成虫線虫を配置して、同期産卵を行います。卵を取り除く前に、成虫を20°Cで約2時間産卵させます。株の繁殖力と採取する時間点の数に応じて、株ごとに1〜4枚のプレートを準備します。
- 卵を入れたプレートを20°Cのインキュベーターに入れます。 彼らが成人に達するまで動物の発達を監視します。
注:動物が成人に達した日は、ここでは1日目と定義されています。通常、これは産卵の3日後です。 - 1日目から始めて、動物を毎日新しい播種NGMプレートに移して、子孫から引き離します。移送中に死亡または失われた動物を補償するには、1株あたり約40匹の動物に、画像化する点数を掛けて転写します(ステップ2を参照)。動物が受精卵を産むのをやめるまで(成人期の〜6日目)進んでください。
注:袋詰めやその他の発達表現型を持つ動物は除外してください。バギングは、凝集を起こしやすいタンパク質を発現する株において一般的に観察される。
2. マルチウェルプレートにおける C.エレガンスの サンプル調製
注:イメージング手順には、最終的に動物を殺す麻酔薬が必要なため、同じ動物を後続の時点で再利用することはできません。代わりに、同じ年齢同期バッチの異なる動物が異なる日に画像化されます。ほとんどの株は1日目にインクルージョンをほとんど持たないが、この時点をベースラインとして含めることが推奨される。
- 麻酔薬として25 mM NaN 3を添加した100 μLのM9バッファーで必要な数のウェルを充填して、384 ウェルプレートを調製する。画像化する株ごとに1つのウェルを充填します。
注:アジ化ナトリウム(NaN3)は有毒であり、注意して取り扱う必要があります。 - 各株について、プラチナワームピックを使用して20匹の動物を1つの井戸に移します。
注:ワームは、井戸に入れる前に細菌の芝生の外に置く必要があります。バクテリアは動物をワームピックに付着させ、それは彼らの放出を妨げることができ、そして井戸の内容物を曇らせるでしょう。一般に、20匹は、ワーム間の重複を防ぎながら、空のウェルスペースの不必要なイメージングを制限するために、ウェルあたりの最適な動物数です。 - 蒸発を防ぐためにプレートを蓋で覆い、準備後1時間以内にプレートを画像化します。
- 包含数の安定したプラトーに達するまで、またはほとんどの動物が死ぬまで、ステップ2.1-2.3を毎日繰り返します。サンプル調製とイメージングを毎日同時に行い、24時間の間隔を確保します。
3. ハイスループット共焦点顕微鏡での画像取得
注:この実験は、マルチウェルプレートホルダーを備えた通常の回転ディスク共焦点顕微鏡で設定することもできます。広い視野を持つカメラは、井戸全体にまたがるために画像化する必要があるタイルの数を制限するのに有益です。このプロトコルで使用される顕微鏡とソフトウェアの詳細については、 材料表 を参照してください。
- 計測器の電源を入れ、ソフトウェアを開きます。
- 新しいプロトコルを開始するには 、測定設定|新規。正しいマルチウェルプレートタイプを選択し、「 新しい測定設定を作成」をクリックします。
- Ch 1 に移動して蛍光チャンネルを設定します。目標を 10 倍に設定します。光源として488nm、発光フィルタとしてBP525/25を選択し、YFPを画像化します。ビン分割を 2x2 に設定して、ファイル サイズを縮小します。
- [ チャンネルの追加 ] をクリックし、メソッドとして [ ブライトフィールド ] を選択します。
- zスタック共焦点蛍光画像を測定に追加するには、「アクションリスト」で「3D 蛍光取得」を選択します。[選択] に移動し、[Ch 1] を選択します。ファイルサイズを最小限に抑えるには、「画像処理」を「最大」に設定して、Z スタック全体ではなく最大投影イメージが保存されるようにします。
- BF/Ph取得|をクリックします。|を選択Ch 2 は明視野チャンネルを表します。
- 「ウェルプレートをアンロード」の横にある 再生 ボタン(右向きの三角形の記号を探します)をクリックし、384 ウェルプレート を顕微鏡に置きます。
- [ 3D 蛍光取得] で、[ テスト ] をクリックし、ワームを含むウェルを選択して、ワームが正しく中心に配置される最適なシフト距離を決定します。「上昇距離」を 「上昇 距離」を「下 降距離」 を「-50 μm」に、「 スライス間隔 」を「2 μm」に設定して、Z スタック内の動物の厚さ全体をキャプチャします。
- 露出時間を最適化して、飽和を避けながら4つの株すべてに対して良好な信号強度を得る。すべてのひずみとタイムポイントに同じ露出時間を使用します。
- 「ウェル プレートスキャン設定」で画像化するウェルを選択します。 タイル を選択し、 全体をうまく取得します。
- 測定設定を保存し、[測定の開始] をクリックして実験を開始します。それ以降のタイムポイントについては、同じ測定設定を開き、シフト距離と画像化するウェルを調整します。
4. ImageJ でタイル画像をステッチする
注: この手順は、各ウェルの画像が複数のタイルとして取得される 4 倍を超える対物レンズを使用する場合にのみ必要です。この分析ワークフローでは、タイルのステッチングは、フリーソフトウェアのFIJI/ImageJ19 を使用して実行されます(図2)。ステップ3で使用する器具によっては、付属のソフトウェアで直接ステッチングを行うこともできる。
- FIJI20をダウンロードして 開きます。
- プラグイン|に移動しますステッチング|グリッド/コレクションステッチ21.
- ポップアップ ウィンドウ の [グリッド/コレクションのステッチング] で、タイルが収集された 種類 と 順序 を選択します。 [グリッド: 行単位 ] と [右 & 下] を選択します。
- 次のウィンドウの [グリッド ステッチング: グリッド: 行単位]、[右 & 下]で、x 方向と y 方向にタイルの数を挿入します。ここで使用する 10x の目的では、グリッド サイズ x として 2、グリッド サイズ y として 3、タイルのオーバーラップとして 0 を選択します。
- [ 参照] をクリックし、ステッチする TIFF 画像を含むフォルダーを選択します。
- タイルの [ファイル名] の下に、各ファイル名のタイル番号の位置に {i} を使用して、共通ファイル名を挿入し ます。
- 下のすべてのボックスのチェックを外します。
- プラグインを実行します。
- 結果の画像をTIFFファイルとして保存し、次のステップで分析します。
5. セルプロファイラ22を使用した自動包接カウント
- オープンソースの画像解析ソフトウェア、CellProfiler23をダウンロードしてインストールします。github.com/sinnigelab/aggregate-quantification から InclusionCounting.cpproj パイプラインをダウンロードし てください。
- CellProfiler を開き、パイプラインを [パイプライン ファイルをここにドロップ] ウィンドウにドラッグします。「 はい 」をクリックしてプロジェクトをロードします。
- 画像入力モジュールをクリックし、ステッチされた画像をウィンドウにドラッグします ここにファイルとフォルダをドロップします。
- メタデータ入力モジュールをクリックします。正規表現を調整して、ステッチされた画像の名前に従ってファイル名から抽出します。
- NamesandTypes 入力モジュールをクリックし、ファイル名のチャンネルに一致するようにルール条件の選択を調整します。
メモ: パイプラインのデフォルト設定では、 BF を含むファイル名は明視野イメージとして認識され、 Worms という名前が付けられます。 YFP を含むファイル名は蛍光画像として認識され、 蛍光と名付けられます。 - [ 出力設定の表示 ]をクリックして、CellProfilerからの出力を保存するデフォルトのフォルダを選択します。
- [ テストモードの開始] をクリックして、最初のイメージングデータセットを使用してパイプラインの設定を確認します。[ 実行 ] をクリックしてパイプライン内のすべてのモジュールを実行するか、[ ステップ ] をクリックしてパイプラインを一度に 1 つのモジュールで実行します。 EditObjectsManual モジュールでワームのアウトラインを調整するには、[ ヘルプ ] をクリックして手順を表示し、[ 完了 ] をクリックしてパイプラインの実行を続行します。
メモ: 抽出された測定値は、テストモードの間はエクスポートされません。ワームおよび介在物を検出するための閾値化パラメータは、使用される株および倍率に基づいて調整する必要があるかもしれない。 - [テストモードの終了]をクリックし、画像を分析します。
- 出力フォルダを開いて、出力ファイルを表示します。元のファイル名の後に アウトライン が続く画像を開き、ワームとインクルージョンが正しくオーバーレイされているかどうかを確認します。
メモ: ワームごとのインクルージョンの数は、 ExpandedWormObjects という名前のファイルにあります。入力イメージの詳細については、 Image という名前のファイルを参照してください。追加の出力は、パイプラインの ExportToSpreadsheet モジュールで選択できます。
6. AmyloFit5を使用した包含カウントデータのグローバルフィッティング
注: このステップは、複数のタンパク質濃度のデータがある場合にのみ実行できます。Q40-YFPについて、体壁筋細胞における過剰発現の異なるレベルを有する4つの株のセットが以前に17個作成されている。他の場合には、新規株はプラスミドマイクロインジェクションおよびゲノム組込みを用いて作製されるべきである24。
- 集約動力学AmyloFit25のための無料のオンラインフィッティングプラットフォームにアクセスしてください。 既存のアカウントで登録またはログインします。
注:AmyloFitの使用方法に関する広範なマニュアルにアクセスして、追加のヘルプを得ることができます。詳細については、ウェブページの左上(ログイン後)のリンクを参照してください。 - AmyloFit の使用を開始するには、プロジェクトに 名前を付け て [ プロジェクトの作成] をクリックします。「開く」をクリックしてプロジェクト を開き 、名前を付けて「セッションの作成とロード」をクリックして セッションを作成します。
- [ データの追加 ] をクリックし、左側のパネルに示されているデータ形式の要件に従って、動物あたりの平均包含数を含むファイルをアップロードします。 「新規データのロード」をクリックします。
- 包含カウント データに必要でない前処理ステップをスキップするには、0 ポイント オフセットの場合は平均 するポイント数 を、 プラトーの場合は平均化するポイント数を 0 に設定します。「 送信」をクリックします。各タンパク質濃度(すなわち、アップロードされたファイルの各カラム)について、この手順を繰り返します。
- モデル パネルで「カスタム」 を選択し、方程式ボックスに 「Ncells*(1-exp(-Kcell*m**(n)*(t-1)) 」と入力して、「 モデルのロード」をクリックします。
注:AmyloFitはもともとin vitroアッセイからの動力学的データの分析のために設計されているため、C. elegansの包含数データを解析するには、カスタムメイドのモデルをロードする必要があります。ここで用いる式において、N細胞は包接形成が起こる細胞数、K細胞は核生成速度定数、mは 細胞内タンパク質濃度、nは核生成の反応順序である。 - パラメーターの種類を、N 個のセルの場合はグローバル定数、K 個のセルと n の場合はグローバル適合値、m の場合は定数に設定します。 体壁筋細胞の場合は N個の細胞の値を 95 に設定し、K個の細胞 の場合は初期推測、 n を 1 に設定します。左パネルに異なるひずみのmの値を入力します。
注: 最初の推測は、ここで使用する比較的単純なモデルには関係ありません。より複雑なモデルでは、期待値の推定値を入力して計算時間を短縮すると効果的です。 - 流域ホップの数を変更しないで、継ぎ手パネルで 「適合」(Fit ) をクリックします。
- 適合を抽出するには、[ データと適合のダウンロード]をクリックします。
メモ: モデルのグローバル適合によって抽出されたパラメータが右下隅に一覧表示されます。データと適合度のプロットが右上のパネルに自動的に生成されます。このプロットは、[PDFをダウンロード]をクリックして抽出し、[プロットオプションの表示]に移動してカスタマイズできます。K細胞は、細胞-1の分子濃度-n時間-1の単位を有する。異なるnの値を比較するために、K細胞は、mnと掛けることによって所与のタンパク質濃度における核生成速度に変換することができる。
結果
ここに記載した方法(図1)は、YFPに融合した40個のグルタミン(Q40−YFP)を含む構築物の凝集動態を分析するために使用された。このタンパク質はunc-54プロモーターの制御下で発現され、体壁筋細胞における発現を駆動する。これらは比較的大きく、視覚化が容易であるため、10倍の対物レンズの使用は、この組織においてQ40-YFPによって形成される封入体を解決するのに十分である。4つの株(系統A−D)が、インビボでのpolyQ凝集の濃度依存性を評価するために、異なる程度にタンパク質を発現させることが以前に開発された17。
系統A-Dの年齢同期集団は、2時間の産卵によって生成され、その後、子孫が成人期に達すると毎日移動した。成虫の1日目から10日目まで、4つの株のそれぞれから20匹の動物を、ハイスループット共焦点顕微鏡を用いて、384ウェルプレートに画像化した。井戸の画像は6つのタイルとして取得され、ImageJ21 のプラグインを使用して縫い合わされました(図2)。ステッチングされた画像は、その後、カスタムメイドのCellProfiler22 パイプライン(図3)を使用して分析され、各系統および時点の動物あたりの平均包接数を定量化した。
その後、データをAmyloFit5の数学的モデルに当てはめました(図4)。このモデルは、95個の体壁筋細胞の各々が、律速核生成事象によって1つの包接を独立して獲得し、続いて速い凝集体成長17を得るという仮定に基づいている。適合により、9.9×105分子M-2.1 d-1細胞-1の核生成速度定数および2.1の反応次数が得られ、細胞内タンパク質濃度1mMで0.38分子d-1細胞-1の核生成速度に相当する。2つの独立した生物学的反復は、核生成速度および反応順序について密接に対応する値をもたらし、これらは同様のプロトコル17を用いた以前の研究と一致している(表1)。
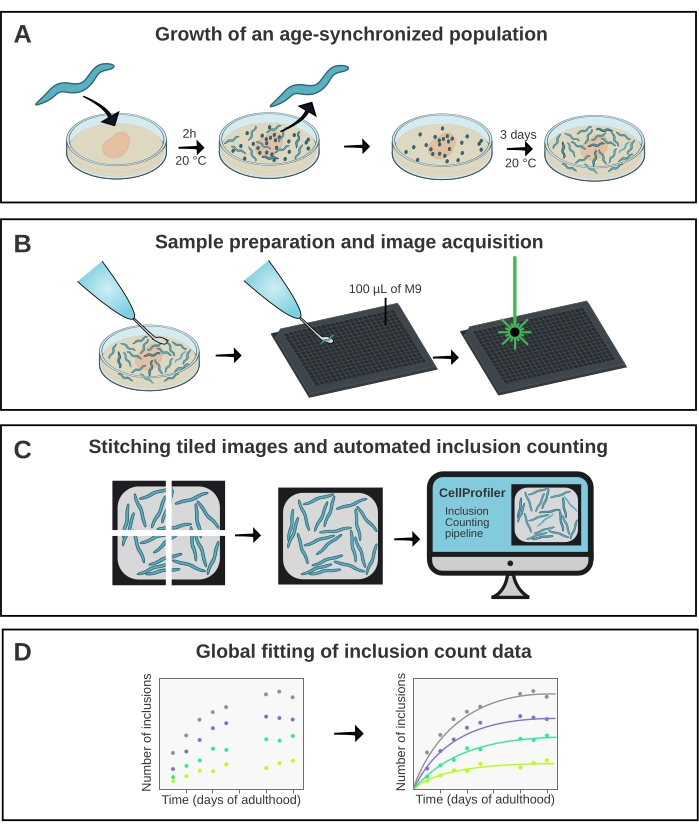
図1:方法の概略概要。 (A)年齢同期C .エレガンス 集団は、時間指定産卵によって生成される。(B)同じ集団の動物を、異なる時点で384ウェルプレートに画像化する。(C)タイルを縫い合わせてウェル全体の画像を形成し、これをCellProfilerで分析して動物ごとの包接数を定量化します。(D) データは、AmyloFitを使用して数学的モデルに適合させる。 この図の拡大版を表示するには、ここをクリックしてください。
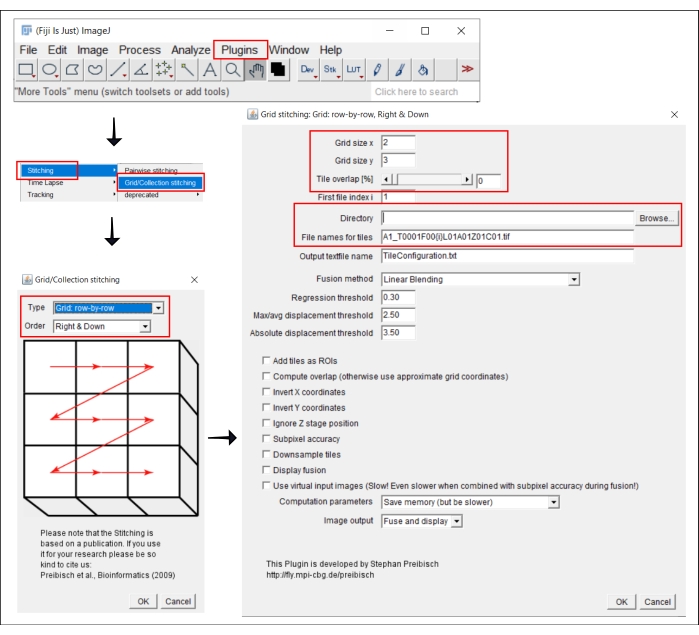
図 2: プラグイン Grid/Collection ステッチング21 を使用した ImageJ でのステッチング手順のスクリーンショット。 この図の拡大版を表示するには、ここをクリックしてください。

図 3: 封入体数を定量化するための CellProfiler パイプラインの概略図。(A-C) 明視野画像 (A) を使用してワームを識別します (B、C のクローズアップ)。(D-G)蛍光画像(D、Eにおけるクローズアップ)は、介在物(F)を識別するために使用される。ワームおよび介在物は、ウェル内の各ワーム(G)の介在物の数を提供することに関係している。示されている画像は、成人期3日目のQ40ラインA動物のものです。この図の拡大版を表示するには、ここをクリックしてください。
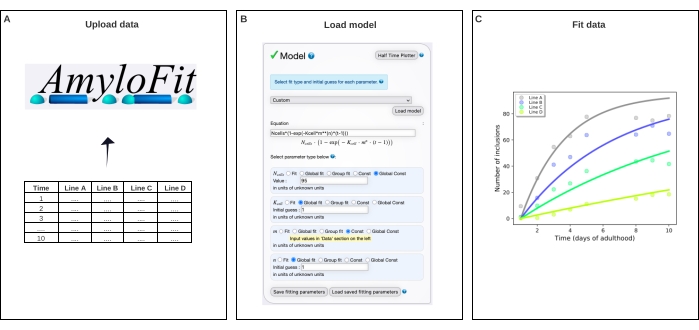
図 4: AmyloFit の数理モデルにデータを当てはめる。(A) データは AmyloFit にアップロードされます。(B)カスタム方程式は、各細胞における独立した核生成事象を仮定して、包接形成をモデル化するために入力される。(C)異なるレベルのQ40−YFPを発現するC.エレガンス線A−Dに対する凝集動態のフィッティング。データは、2つの独立した生物学的反復を表すものである。この図の拡大版を表示するには、ここをクリックしてください。
| データセット 1 | データセット 2 | Sinnige et al.17 | |
| n | 2.1 | 1.9 | 1.6 |
| K細胞(分子M-nd-1セル-1) | 9.9×105 | 1.4 × 105 | 3.1 × 104 |
| 1mMにおける核生成速度(分子d-1セル-1) | 0.38 | 0.21 | 0.35 |
表1:Q40-YFP凝集の核生成速度と反応順序の値。 プロトコールの2つの独立した生物学的複製についてのデータおよび以前に公開されたデータとの比較17。
ディスカッション
本明細書に提示される方法は、モデル生物 C. elegansにおけるタンパク質凝集動態の偏りのない定量的分析を容易にする。それは4つの重要な要素(図1)に依存する:1)線虫の年齢同期集団を維持する。2)マルチウェルプレートにおける蛍光顕微鏡;3)セルプロファイラにおける自動包接計数;4)AmyloFitのデータフィッティング。自由に動く動物や保存された画像26からの封入物の手動計数と比較して、CellProfilerでの定量化はより速く、より偏りがありません。プロトコルのもう1つの重要な進歩は、単一のタイムポイントではなく、動力学的データの取得であり、データを数学的モデルに適合させる際に集約メカニズムに定量的な洞察を提供します。
プロトコルの4つの要素は、アプリケーションに応じて変更できる独立したモジュールとして使用できます。年齢同期集団は、動物を滅菌するために5-フルオロ-2'-デオキシウリジン(FUDR)を使用して維持することもできる。この化合物は、寿命およびプロテオスタシス24,25に影響を及ぼし、実験者にとって非常に発癌性である。ただし、ワームを手動で転送することはできず、大量のワームが処理されると労働集約的になる可能性があります。他の代替手段は、子孫30を分離するための滅菌変異体29または濾過装置の使用である。
蛍光顕微鏡観察ステップは、例えば、ニューロンにおけるタンパク質凝集をモニターするためにより高い倍率を使用して調整することもできる。広視野顕微鏡は、条件間の相対的な差が封入物の絶対数よりも重要である場合に、筋細胞におけるpolyQ凝集をモニターするのに十分であり得る。このような場合でも CellProfiler パイプラインを使用できますが、ワームやインクルージョンを認識するための設定はユーザーが調整する必要があります。この技術のスループットは現在、384ウェルプレートへの動物の手動ピッキングの必要性によって制限されている。これは、マイクロ流体デバイス16の使用によって潜在的に改善され得る。アジ化ナトリウムは比較的過酷な麻酔薬であり、ヒドロゲルまたはビーズによる物理的固定化によって置き換えることができる28,29。
ここで紹介するAmyloFitでの解析は、個々の細胞における独立した核生成イベントからなる凝集メカニズムに基づいています。このモデルが適合しない場合、ユーザは、以前に開発された協調集約モデルなどの代替手段を検討するべきである17.このアプローチの限界は、異なる濃度で目的のタンパク質を発現する株が利用可能である必要があることであるが、これらは日常的な C. elegans 法24を用いて生成することができる。
全体として、このプロトコルは、 in vivo モデルシステムにおけるタンパク質凝集動態に関する高品質のデータを得る手段を提供し、凝集機構の詳細な分析を可能にする17。この方法は C.elegans 筋肉組織におけるpolyQ凝集について実証されたが、プロトコルの将来の適用は、他のタンパク質および組織ならびにプロテオスタシス因子および小分子の効果を含み得る。
開示事項
著者らは、開示する利益相反はありません。
謝辞
我々は、C. elegans株の森本研究室と、ハイスループット共焦点顕微鏡の支援に対するEsmeralda Bosmanに感謝する。この研究は、ユトレヒト大学からT.S.へのスタートアップ助成金によって資金提供されました。
資料
| Name | Company | Catalog Number | Comments |
| 384-well plate | Greiner | 781091 | Black with flat clear bottom |
| AmyloFit | Knowles lab | v2.0 | Access at www.amylofit.ch.cam.ac.uk |
| C. elegans Q40 line A | Morimoto lab | AM1228 | Genotype rmIs404[unc-54p::Q40::YFP] |
| C. elegans Q40 line B | Morimoto lab | AM1229 | Genotype rmIs404[unc-54p::Q40::YFP] |
| C. elegans Q40 line C | Morimoto lab | AM1230 | Genotype rmIs404[unc-54p::Q40::YFP] |
| C. elegans Q40 line D | Morimoto lab | AM1231 | Genotype rmIs404[unc-54p::Q40::YFP] |
| CellProfiler | Broad Institute | 4.1.3 | Downloaded from https://cellprofiler.org |
| E. coli OP50 | Caenorhabditis Genetics Center (CGC) | OP50 | |
| FIJI | Open-source | (Fiji Is Just) ImageJ v2.1/1.5.3j | Downloaded from https://imagej.net/software/fiji/ |
| High-throughput confocal microscope | Yokogawa | CellVoyager CV7000S | |
| M9 buffer | Home-made | 3 g/L KH2PO4, 6 g/L Na2HPO4, 0.5 g/L NaCl, 1 mM MgSO4 | |
| NGM plates | Home-made | 17 g/L agar, 2.5 g/L bacto-peptone, 3 g/L NaCl, 25 mM KPO4 buffer pH 6.0, 1 mM MgSO4, 1 mM CaCl2, 5 mg/L cholesterol | |
| Pasteur pipette | WU Mainz | 250 | To make worm pick, 150 mm length |
| Platinum iridium wire | Alfa Aesar | 39383 | To make worm pick, 0.25 mm diameter |
| Sodium azide | Sigma-Aldrich | S2002 | |
| Stereomicroscope | Leica | S9 |
参考文献
- Knowles, T. P. J., Vendruscolo, M., Dobson, C. M. The amyloid state and its association with protein misfolding diseases. Nature Reviews. Molecular Cell Biology. 15 (6), 384-396 (2014).
- Chiti, F., Dobson, C. M. Protein misfolding, amyloid formation, and human disease: a summary of progress over the last decade. Annual Review of Biochemistry. 86 (1), 27-68 (2017).
- Knowles, T. P. J., et al. An analytical solution to the kinetics of breakable filament assembly. Science. 326 (5959), 1533-1537 (2009).
- Cohen, S. I. A., et al. Proliferation of amyloid-β42 aggregates occurs through a secondary nucleation mechanism. Proceedings of the National Academy of Sciences of the United States of America. 110 (24), 9758-9763 (2013).
- Meisl, G., et al. Molecular mechanisms of protein aggregation from global fitting of kinetic models. Nature Protocols. 11 (2), 252-272 (2016).
- Satyal, S. H., et al. Polyglutamine aggregates alter protein folding homeostasis in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 97 (11), 5750-5755 (2000).
- Morley, J. F., Brignull, H. R., Weyers, J. J., Morimoto, R. I. The threshold for polyglutamine-expansion protein aggregation and cellular toxicity is dynamic and influenced by aging in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 99 (16), 10417-10422 (2002).
- Brignull, H. R., Moore, F. E., Tang, S. J., Morimoto, R. I. Polyglutamine proteins at the pathogenic threshold display neuron-specific aggregation in a pan-neuronal Caenorhabditis elegans model. Journal of Neuroscience. 26 (29), 7597-7606 (2006).
- Moronetti Mazzeo, L. E., Dersh, D., Boccitto, M., Kalb, R. G., Lamitina, T. Stress and aging induce distinct polyQ protein aggregation states. Proceedings of the National Academy of Sciences of the United States of America. 109 (26), 10587-10592 (2012).
- Prahlad, V., Morimoto, R. I. Neuronal circuitry regulates the response of Caenorhabditis elegans to misfolded proteins. Proceedings of the National Academy of Sciences of the United States of America. 108 (34), 14204-14209 (2011).
- Nollen, E. A. A. Genome-wide RNA interference screen identifies previously undescribed regulators of polyglutamine aggregation. Proceedings of the National Academy of Sciences of the United States of America. 101 (17), 6403-6408 (2004).
- Silva, M. C., et al. A genetic screening strategy identifies novel regulators of the proteostasis network. PLoS Genetics. 7 (12), 1002438(2011).
- Brehme, M., et al. A chaperome subnetwork safeguards proteostasis in aging and neurodegenerative disease. Cell Reports. 9, 1-16 (2014).
- Calamini, B., et al. Small-molecule proteostasis regulators for protein conformational diseases. Nature Chemical Biology. 8 (2), 185-196 (2012).
- Sinnige, T., Stroobants, K., Dobson, C. M., Vendruscolo, M. Biophysical studies of protein misfolding and aggregation in in vivo models of Alzheimer's and Parkinson's diseases. Quarterly Reviews of Biophysics. 49, 22(2020).
- Mondal, S., et al. Large-scale microfluidics providing high-resolution and high-throughput screening of Caenorhabditis elegans poly-glutamine aggregation model. Nature Communications. 7, 13023(2016).
- Sinnige, T., et al. Kinetic analysis reveals that independent nucleation events determine the progression of polyglutamine aggregation in C. elegans. Proceedings of the National Academy of Sciences of the United States of America. 118 (11), 202188118(2021).
- Brenner, S. Caenorhabditis elegans. Methods. 77 (1), 71-94 (1974).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- FIJI/ImageJ. , Available from: https://imagej.net/downloads (2012).
- Preibisch, S., Saalfeld, S., Tomancak, P. Globally optimal stitching of tiled 3D microscopic image acquisitions. Bioinformatics. 25 (11), 1463-1465 (2009).
- Lamprecht, M. R., Sabatini, D. M., Carpenter, A. E. CellProfiler: free, versatile software for automated biological image analysis. BioTechniques. 42 (1), 71-75 (2007).
- CellProfiler. Broad Institute. , Available from: https://cellprofiler.org/releases (2021).
- Mello, C. C., Kramer, J. M., Stinchcomb, D., Ambros, V. Efficient gene transfer in C. elegans: extrachromosomal maintenance and integration of transforming sequences. EMBO Journal. 10 (12), 3959-3970 (1991).
- Knowles group, University of Cambridge. , Available from: https://amylofit.com/amylofitmain/login/ (2021).
- Lazaro-Pena, M. I., Cornwell, A. B., Samuelson, A. V. Quantifying tissue-specific proteostatic decline in Caenorhabditis elegans. Journal of Visualized Experiments: JoVE. (175), (2021).
- Feldman, N., Kosolapov, L., Ben-Zvi, A. Fluorodeoxyuridine improves Caenorhabditis elegans proteostasis independent of reproduction onset. PLoS One. 9 (1), 85964(2014).
- Brunquell, J., Bowers, P., Westerheide, S. D. Fluorodeoxyuridine enhances the heat shock response and decreases polyglutamine aggregation in an HSF-1-dependent manner in Caenorhabditis elegans. Mechanisms of Ageing and Development. 141-142, 1-4 (2014).
- David, D. C., et al. Widespread protein aggregation as an inherent part of aging in C. elegans. PLoS Biology. 8 (8), 47-48 (2010).
- Hunter, S., Maulik, M., Scerbak, C., Vayndorf, E., Taylor, B. E. Caenorhabditis sieve: A low-tech instrument and methodology for sorting small multicellular organisms. Journal of Visualized Experiments: JoVE. (137), (2018).
- Burnett, K., Edsinger, E., Albrecht, D. R. Rapid and gentle hydrogel encapsulation of living organisms enables long-term microscopy over multiple hours. Communications Biology. 1, 73(2018).
- Dong, L., et al. Reversible and long-term immobilization in a hydrogel-microbead matrix for high-resolution imaging of Caenorhabditis elegans and other small organisms. PLoS One. 13 (3), 0193989(2018).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved