Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Demostración de la alineación de secuencias para predecir la susceptibilidad entre especies herramienta para una evaluación rápida de la conservación de proteínas
En este artículo
Resumen
Aquí, presentamos un protocolo para utilizar la última versión de la herramienta de alineación de secuencias de la Agencia de Protección Ambiental de los Estados Unidos para predecir la susceptibilidad a través de especies (SeqAPASS). Este protocolo demuestra la aplicación de la herramienta en línea para analizar rápidamente la conservación de proteínas y proporcionar predicciones personalizables y fácilmente interpretables de susceptibilidad química en todas las especies.
Resumen
La herramienta Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS) de la Agencia de Protección Ambiental de los Estados Unidos es una aplicación de detección en línea rápida y disponible gratuitamente que permite a los investigadores y reguladores extrapolar información de toxicidad entre especies. Para objetivos biológicos en sistemas modelo como células humanas, ratones, ratas y peces cebra, los datos de toxicidad están disponibles para una variedad de productos químicos. A través de la evaluación de la conservación de objetivos proteicos, esta herramienta se puede utilizar para extrapolar los datos generados a partir de dichos sistemas modelo a miles de otras especies que carecen de datos de toxicidad, lo que produce predicciones de susceptibilidad química intrínseca relativa. Las últimas versiones de la herramienta (versiones 2.0-6.1) han incorporado nuevas características que permiten la rápida síntesis, interpretación y uso de los datos para la publicación más gráficos de calidad de presentación.
Entre estas características se encuentran visualizaciones de datos personalizables y un informe de resumen completo diseñado para resumir los datos de SeqAPASS para facilitar la interpretación. Este documento describe el protocolo para guiar a los usuarios a través del envío de trabajos, la navegación por los diversos niveles de comparaciones de secuencias de proteínas y la interpretación y visualización de los datos resultantes. Se destacan las nuevas características de SeqAPASS v2.0-6.0. Además, se describen dos casos de uso centrados en la conservación de proteínas transtiretinas y receptoras opioides utilizando esta herramienta. Finalmente, se discuten las fortalezas y limitaciones de SeqAPASS para definir el dominio de aplicabilidad de la herramienta y resaltar diferentes aplicaciones para la extrapolación entre especies.
Introducción
Tradicionalmente, el campo de la toxicología se ha basado en gran medida en el uso de pruebas con animales enteros para proporcionar los datos necesarios para las evaluaciones de seguridad química. Tales métodos suelen ser costosos y requieren muchos recursos. Sin embargo, debido al gran número de productos químicos utilizados actualmente y al rápido ritmo al que se están desarrollando nuevos productos químicos, a nivel mundial existe una necesidad reconocida de métodos más eficientes de detección química 1,2. Esta necesidad y el cambio de paradigma resultante de las pruebas con animales ha llevado al desarrollo de muchos métodos de enfoque nuevos, incluidos los ensayos de detección de alto rendimiento, la transcriptómica de alto rendimiento, la secuenciación de próxima generación y el modelado computacional, que son estrategias de prueba alternativas prometedoras 3,4.
La evaluación de la seguridad química en toda la diversidad de especies potencialmente afectadas por las exposiciones químicas ha sido un desafío permanente, no solo con las pruebas de toxicidad tradicionales, sino también con los nuevos métodos de enfoque. Los avances en toxicología comparativa y predictiva han proporcionado marcos para comprender la sensibilidad relativa de diferentes especies, y los avances tecnológicos en métodos computacionales continúan aumentando la aplicabilidad de estos métodos. En la última década se han discutido varias estrategias que aprovechan las bases de datos existentes de secuencias de genes y proteínas, junto con el conocimiento de objetivos moleculares químicos específicos, para apoyar los enfoques predictivos para la extrapolación entre especies y mejorar las evaluaciones de seguridad química más allá de los organismos modelo típicos 5,6,7,8.
Para avanzar en la ciencia hacia la acción, construir sobre estos estudios fundamentales en toxicología predictiva, priorizar los esfuerzos de pruebas químicas y apoyar la toma de decisiones, se creó la herramienta de alineación de secuencias de la Agencia de Protección Ambiental de los Estados Unidos para predecir la susceptibilidad entre especies (SeqAPASS). Esta herramienta es una aplicación basada en la web pública y de libre acceso que utiliza repositorios públicos de información de secuencias de proteínas en constante expansión para predecir la susceptibilidad química en la diversidad de especies9. Basándose en el principio de que la susceptibilidad intrínseca relativa de una especie a una sustancia química en particular puede determinarse evaluando la conservación de las dianas proteicas conocidas de esa sustancia química, esta herramienta compara rápidamente las secuencias de aminoácidos proteicos de una especie con sensibilidad conocida a todas las especies con los datos de secuencias de proteínas existentes. Esta evaluación se completa a través de tres niveles de análisis, que incluyen (1) secuencia primaria de aminoácidos, (2) dominio funcional y (3) comparaciones de residuos críticos de aminoácidos, cada uno de los cuales requiere un conocimiento más profundo de la interacción químico-proteína y proporciona una mayor resolución taxonómica en la predicción de susceptibilidad. Una fortaleza importante de SeqAPASS es que los usuarios pueden personalizar y refinar su evaluación agregando líneas adicionales de evidencia hacia la conservación del objetivo en función de la cantidad de información disponible sobre la interacción químico-proteína o proteína-proteína de interés.
La primera versión se lanzó en 2016, que permitió a los usuarios evaluar secuencias de aminoácidos primarios y dominios funcionales de manera simplificada para predecir la susceptibilidad química y contenía capacidades mínimas de visualización de datos (Tabla 1). Se ha demostrado que las diferencias individuales de aminoácidos son determinantes importantes de las diferencias entre especies en las interacciones químico-proteicas, que pueden afectar la susceptibilidad química de las especies10,11,12. Por lo tanto, las versiones posteriores fueron desarrolladas para considerar los aminoácidos críticos que son importantes para la interacción química directa13. En respuesta a los comentarios de las partes interesadas y los usuarios, esta herramienta ha sido lanzada anualmente con nuevas características adicionales diseñadas para satisfacer las necesidades tanto de los investigadores como de las comunidades reguladoras para abordar los desafíos en la extrapolación entre especies (Tabla 1). El lanzamiento de SeqAPASS versión 5.0 en 2020 trajo características centradas en el usuario que incorporan opciones de visualización y síntesis de datos, enlaces externos, opciones de tabla de resumen e informe y características gráficas. En general, los nuevos atributos y capacidades de esta versión mejoraron la síntesis de datos, la interoperabilidad entre bases de datos externas y la facilidad de interpretación de datos para las predicciones de susceptibilidad entre especies.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Primeros pasos
NOTA: El protocolo presentado aquí se centra en la utilidad de la herramienta y las características clave. Las descripciones detalladas de los métodos, características y componentes se pueden encontrar en el sitio web en una Guía del usuario completa (Tabla 1).
Tabla 1: Evolución de la herramienta SeqAPASS. Una lista de características y actualizaciones agregadas a la herramienta SeqAPASS desde su implementación inicial. Abreviaturas: SeqAPASS = alineación de secuencias para predecir la susceptibilidad entre especies; ECOTOX = Base de conocimiento de ECOTOXicology. Haga clic aquí para descargar esta tabla.
- Ve a https://seqapass.epa.gov/seqapass usando Chrome. Seleccione Iniciar sesión para usar una cuenta existente o siga las instrucciones para crear una cuenta SeqAPASS, que permitirá a los usuarios ejecutar, almacenar, acceder y personalizar sus trabajos completados.
- Antes de realizar un análisis, primero identifique una proteína de interés y una especie objetivo o sensible revisando la literatura existente o los datos preexistentes (Figura 1). Como SeqAPASS contiene enlaces a recursos externos para ayudar a identificar la proteína de consulta, haga clic en los botones desplegables en Identificar un objetivo de proteína para acceder a los recursos relevantes.
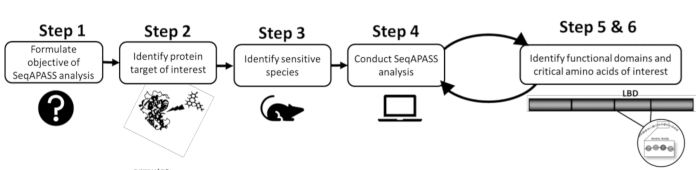
Figura 1: Formulación del problema SeqAPASS: diagrama esquemático de la información preliminar necesaria para un análisis exitoso. Abreviaturas: SeqAPASS = alineación de secuencias para predecir la susceptibilidad entre especies; LBD = dominio de unión al ligando. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Interoperabilidad de SeqAPASS entre bases de datos. Diagrama esquemático de herramientas externas, bases de datos y recursos integrados en SeqAPASS. Abreviaturas: SeqAPASS = alineación de secuencias para predecir la susceptibilidad entre especies; AOP = vía de resultado adverso; NCBI = Centro Nacional de Información Biotecnológica; ECOTOX = Base de conocimiento de ECOTOXicology. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 2: Enlaces, recursos y herramientas integradas en la herramienta SeqAPASS. Una lista de las diversas fuentes de datos, enlaces y recursos aprovechados en la herramienta SeqAPASS. Abreviatura: SeqAPASS = alineación de secuencias para predecir la susceptibilidad entre especies. Haga clic aquí para descargar esta tabla.
2. Desarrollo y ejecución de una consulta SeqAPASS: Level 1
NOTA: En un análisis de Nivel 1, toda la secuencia de aminoácidos primarios de una proteína de consulta se compara con las secuencias de aminoácidos primarios de todas las especies con información de secuencia disponible. Esta herramienta utiliza algoritmos para extraer, recopilar y compilar datos disponibles públicamente para alinear y comparar rápidamente las secuencias de aminoácidos entre especies. El backend almacena información de las bases de datos del Centro Nacional de Información Biotecnológica (NCBI) y utiliza estratégicamente las versiones independientes de la Herramienta de búsqueda de alineación local básica de proteínas (BLASTp)54 y la Herramienta de alineación múltiple basada en restricciones (COBALT)55.
- En Comparar secuencias de aminoácidos primarios, haga clic en Por especie o Por adhesión. Utilice la selección Por especie para escribir o seleccionar de una lista de especies para elegir el objetivo de proteína de interés.
- Envíe las accesiones de proteínas (es decir, identificación de proteína NCBI) directamente ingresando la(s) accesibilidad(es) en el cuadro de texto Por accesión .
- Seleccione Solicitar ejecución para enviar la consulta. Una vez enviado, espere a que aparezca una notificación en la esquina superior derecha de la ventana del navegador que indique un envío exitoso.
- Seleccione la pestaña Estado de ejecución de SeqAPASS en la parte superior de la página para mostrar una lista de todas las ejecuciones de SeqAPASS realizadas en esa cuenta de usuario y verificar el porcentaje de finalización.
- Haga clic en Actualizar datos mientras se selecciona el botón de opción apropiado para comprobar el estado de las ejecuciones de nivel 2 y nivel 3.
- Seleccione la pestaña Ver informes de SeqAPASS en la parte superior de la página para acceder a una lista de todos los informes completados con esa cuenta.
- En la pestaña Ver informes de SeqPASS , seleccione la proteína de consulta que le interese. Haga clic en Solicitar informe seleccionado para abrir la página Información de proteína de consulta de nivel 1 y ver resultados, opciones de personalización de datos, visualizaciones e informes de resumen.
- De forma predeterminada, seleccione Ver informe para ver los datos en el explorador web. Como alternativa, seleccione Guardar informe para descargar datos sin procesar como un archivo .zip.
NOTA: El tiempo requerido para un análisis de Nivel 1 variará (promedio de 23 minutos para la versión 5.1) dependiendo de la demanda global del usuario en ese momento, el número de trabajos enviados a la cola y la cantidad de información de proteínas que existe para un trabajo enviado. Si un objetivo de proteína se ha completado previamente, los datos estarán disponibles en segundos después de la presentación.
3. Desarrollo y ejecución de una consulta SeqAPASS: Level 2
NOTA: Como toda la secuencia de proteínas no está directamente involucrada en una interacción química, un análisis de Nivel 2 compara solo la secuencia de aminoácidos del dominio funcional para hacer predicciones de susceptibilidad en rangos taxonómicos más bajos (por ejemplo, clase, orden, familia).
- En la página Información de proteína de consulta de nivel 1, haga clic en el signo más + situado junto al encabezado de nivel 2 para rellenar el menú Consulta de nivel 2.
- Identificar el(los) dominio(s) apropiado(s) en la proteína de interés (proteína de consulta).
- Si no se ha identificado un dominio, haga clic en el enlace integrado a la Base de datos de dominios conservados (CDD) del NCBI (Tabla 1), que puede ayudar en la identificación de la selección de dominio adecuada.
NOTA: Normalmente, solo se seleccionan dominios de visitas específicos como consultas en el nivel 2.
- Si no se ha identificado un dominio, haga clic en el enlace integrado a la Base de datos de dominios conservados (CDD) del NCBI (Tabla 1), que puede ayudar en la identificación de la selección de dominio adecuada.
- Haga clic en el cuadro Seleccionar dominio para rellenar automáticamente una lista de dominios funcionales para la proteína de consulta.
- Seleccione las adhesiones al dominio de la lista desplegable e inicie la consulta de nivel 2 haciendo clic en el botón Solicitar ejecución de dominio . Una vez enviado, espere a que aparezca una notificación que indique un envío exitoso.
- Haga clic en Actualizar ejecuciones de nivel 2 y 3 para rellenar los datos de nivel 2 , que estarán disponibles en cuestión de segundos después del envío.
- En Ver datos de nivel 2, seleccione las accesiones de dominio completadas de la lista desplegable y haga clic en el botón Ver datos de nivel 2 para abrir los resultados en una página nueva.
4. Acceso y comprensión de los datos: SeqAPASS Nivel 1 y Nivel 2
- Desplácese hasta la parte inferior de la página Consultar información de proteínas para ver un informe de los resultados: se proporciona un informe primario con análisis de nivel 1 y 2 de forma predeterminada. Seleccione el botón de opción Informe completo para ver un informe más detallado que proporciona todos los aciertos de secuencia y métricas de alineación. Haga clic en la accesición/ID/nombre apropiado en ambos informes para acceder a la alineación transparente de proteínas y la información de taxonomía en la base de datos NCBI.
- Desplácese hasta el lado derecho de la tabla de resultados para ver la columna ECOTOX . Haga clic en los enlaces a la base de conocimiento de ECOTOXicology (ECOTOX) para recopilar rápidamente los datos de toxicidad correspondientes para especies con predicciones de susceptibilidad.
NOTA: ECOTOX es una base de conocimientos completa y disponible públicamente que proporciona datos únicos de toxicidad química para plantas acuáticas y terrestres y vida silvestre. SeqAPASS v6.0 incluye un widget ECOTOX para conectarse con datos relevantes de ECOTOX más rápidamente por producto químico y especies de interés. - Haga clic en Descargar tabla para guardar la tabla como un archivo de hoja de cálculo. Haga clic en el botón Ver informe de resumen para ver y descargar una tabla de informe de resumen que presenta datos ordenados por grupo taxonómico.
NOTA: Las tablas de resumen de datos están disponibles para los informes primarios y completos y proporcionan una descripción general de las predicciones para un objetivo determinado.
5. Manipulación de la configuración de datos: SeqAPASS Nivel 1 y Nivel 2
NOTA: En los análisis de Nivel 1 y Nivel 2, se supone que cuanto mayor sea la similitud de la proteína, mayor será la probabilidad de que una sustancia química interactúe con la proteína de manera similar a la especie/proteína consulta, haciéndola susceptible a los posibles impactos de las sustancias químicas con este objetivo molecular. Debido a la similitud de estos datos, los pasos para comprender los datos de Nivel 1 y 2 se describen juntos en un solo protocolo.
- Consulte los submenús en la parte superior de Consultar información de proteínas para acceder y manipular la configuración del informe y usar la configuración predeterminada para todas las opciones de informe para la mayoría de los análisis. Si hay una justificación científica para cambiar la configuración predeterminada, siga estos pasos opcionales:
- (OPCIONAL) Haga clic en el signo más + junto a Corte de susceptibilidad para ver y ajustar la configuración de corte de susceptibilidad en una pestaña nueva. Seleccione un nuevo valor de corte de una lista desplegable o introduzca un valor de corte definido por el usuario.
- (OPCIONAL) Cambie el número en el campo E-Value (el número de alineaciones diferentes que se espera que ocurran por casualidad) si se desea algo distinto al predeterminado.
NOTA: Cualquier proteína con un valor E mayor que el número en el cuadro se eliminará del informe primario. - (OPCIONAL) Utilice la opción Ordenar por grupo taxonómico para elegir el nivel de jerarquía taxonómica que desea mostrar en la columna Grupo taxonómico filtrado de la tabla de resultados.
NOTA: Cambiar la jerarquía taxonómica también alterará la predicción de susceptibilidad basada en las especies de cada grupo filtrado que se encuentran por encima del límite. - (OPCIONAL) Cambie el campo Dominio común (cuántos dominios comunes debe compartir una proteína con la proteína de consulta para incluirla en los resultados) si se desea algo distinto del predeterminado.
NOTA: Como el valor predeterminado es 1, se excluirá cualquier secuencia que no comparta al menos un dominio común con la proteína de consulta. - (OPCIONAL) Seleccione No en Lectura de especies para recuperar las predicciones de susceptibilidad de Y solo si la similitud porcentual es mayor o igual que el límite o si el resultado se identifica como un candidato ortológico.
NOTA: Esta configuración predeterminada es Sí, lo que significa que se informará una predicción de susceptibilidad de Y para todos los candidatos ortológicos, todas las especies enumeradas por encima del límite de susceptibilidad y todas las especies por debajo del límite del mismo grupo taxonómico con una o más especies por encima del límite.
- Haga clic en el botón Descargar configuración de informe actual para descargar un archivo que capture la configuración actual aplicada.
NOTA: El nivel de evaluación específico (1, 2 o 3) seleccionado dictará la configuración que se presenta en el informe.
6. Visualización de los datos: SeqAPASS Nivel 1 y Nivel 2
- Haga clic en el signo más + junto a Visualización y haga clic en el botón Visualizar datos para abrir una pestaña separada que muestra la información definida por el usuario y la opción de seleccionar un gráfico interactivo de resultados.
- Haga clic en Diagrama de cuadro para abrir el diagrama de cuadro interactivo y los controles de trazado y permitir que la visualización del diagrama de cuadro se actualice activamente para reflejar los cambios en la tabla de datos y proporcionar gráficos con calidad de publicación y presentación.
NOTA: El diagrama de caja predeterminado muestra los grupos de especies en el eje x y la similitud porcentual en el eje y. Los diagramas de caja muestran el límite de susceptibilidad (línea discontinua), el porcentaje de similitud entre especies en comparación con la especie de consulta, y los valores medios y medianos para cada grupo taxonómico junto con los percentiles 25 y 75 y el rango intercuartílico. Dependiendo del objetivo del análisis y las necesidades del usuario, muchas características de diagrama de caja se pueden modificar a través de los siguientes pasos opcionales.- (OPCIONAL) Para personalizar los grupos taxonómicos mostrados, consulte el cuadro Grupos taxonómicos en la sección Controles . Elimine grupos desplazándose sobre los nombres y seleccionando x o usando el menú desplegable Grupos taxonómicos .
- (OPCIONAL) Para agregar una leyenda que señale una especie de interés o grupos específicos predefinidos (por ejemplo, especies en peligro de extinción o amenazadas), coloque el cursor sobre el nombre de un grupo taxonómico en el eje x para activar un cuadro emergente que enumera las tres especies principales ordenadas por mayor similitud porcentual. Desplácese sobre las especies de la leyenda para generar un cuadro emergente con la información de especies correspondiente. Haga clic en el cuadro de un grupo taxonómico específico para generar una tabla de resumen descargable que enumere especies y predicciones.
- Haga clic en Descargar diagrama de caja para elegir un tipo de archivo, personalizar la resolución de ancho / alto y guardar la visualización.
7. Desarrollo y ejecución de un análisis SeqAPASS: Level 3
NOTA: Un análisis de Nivel 3 evalúa los residuos de aminoácidos identificados por el usuario dentro de la proteína de consulta y compara rápidamente la conservación de estos residuos en todas las especies. Se supone que las especies en las que se conservan estos residuos tienen más probabilidades de interactuar con una sustancia química de manera similar a la especie/proteína modelo. Como el Nivel 3 se centra en aminoácidos individuales, solo se puede realizar un análisis cuando se dispone de un conocimiento detallado de los residuos de aminoácidos críticos para la interacción químico-proteína o proteína-proteína.
- Haga clic en el signo más + situado junto al encabezado de nivel 3 en la página Información de proteína de consulta de nivel 1 para rellenar el menú Consulta de nivel 3.
- Haga clic en el signo más + junto al Explorador de referencias para abrir la herramienta del explorador de referencias, que genera una cadena booleana predefinida para consultar la literatura disponible y ayuda a los usuarios a identificar la literatura apropiada para apoyar la identificación de aminoácidos críticos que se utilizarán en la evaluación de Nivel 3 (Tabla 2 y Figura 2).
- (OPCIONAL) Una vez que la proteína de consulta se rellena automáticamente, use la función Agregar nombre de proteína para agregar proteínas adicionales.
- Haga clic en Generar enlace de Google Académico para abrir una ventana emergente que contenga una cadena de búsqueda generada automáticamente que incluya términos de búsqueda relevantes.
- Haga clic en Buscar en Google Académico para consultar la base de datos de literatura utilizando la cadena de búsqueda.
- Como alternativa, haga clic en Copiar al Portapapeles y personalice la cadena de búsqueda agregando o quitando términos mediante las funciones del Explorador de referencias.
8. Identificar residuos críticos de aminoácidos utilizando literatura identificada
- Seleccione la secuencia de plantilla con la que se alinearán las especies seleccionadas por el usuario en el menú Consulta de nivel 3.
NOTA: Esta secuencia de plantilla se elige comúnmente en base a la literatura para la cual se identificaron los aminoácidos críticos y puede ser la misma especie o una especie diferente a las consultadas en el Nivel 1 y Nivel 2.- (OPCIONAL) Utilice el cuadro Comparaciones adicionales para comparar las accesiones o secuencias que no aparecen en las tablas Principal/Informe completo .
- Introduzca un nombre definido por el usuario para la ejecución de nivel 3 en el cuadro de texto Introducir nombre de ejecución de nivel 3 para identificar la ejecución de nivel 3 completada. Elija un nombre único para cada evaluación.
- Seleccione el grupo taxonómico de interés en el campo Elegir grupo(s) taxonómico(s). Seleccione un grupo taxonómico para filtrar automáticamente la tabla por ese grupo taxonómico.
- En la tabla de resultados, haga clic manualmente en la casilla de verificación situada junto a cualquier especie que desee alinearse con la secuencia de plantilla.
NOTA: Para garantizar una alineación adecuada, se debe comparar un grupo taxonómico a la vez con la plantilla. Solo seleccione proteínas con anotaciones similares para la especie de interés. Al seleccionar secuencias para la comparación, es importante prestar atención a ciertas secuencias (por ejemplo , hipotéticas, de BAJA CALIDAD o parciales). A menos que haya una justificación transparente para la inclusión, es mejor excluir estas secuencias, ya que pueden distorsionar las predicciones debido a información de secuencia incompleta o inadecuada. - Repita los pasos para alinear todos los grupos taxonómicos de interés.
- Haga clic en Actualizar ejecuciones de nivel 2 y 3 una vez que todas las especies se hayan alineado para rellenar el menú Seleccionar nombre de ejecución de nivel 3 con los trabajos de nivel 3 completados y obtener los datos de una alineación de nivel 3 inmediatamente.
- Haga clic en Combinar datos de nivel 3 para combinar alineaciones de varios grupos taxonómicos.
- Como alternativa, para ver un solo informe, seleccione el nombre definido por el usuario en Elegir consulta para ver y haga clic en Ver datos de nivel 3.
- Seleccione la plantilla de nivel 3 que se utilizará como base para la comparación de residuos de aminoácidos en el menú Combinar informes de nivel 3 y haga clic en Siguiente.
- En Trabajos de nivel 3, seleccione trabajos completados para compararlos y haga clic en Siguiente. Utilice la función Trabajos de nivel de orden 3 para reordenar los grupos taxonómicos si lo desea. Haga clic en Ver datos de nivel 3 para generar una página de informe de nivel 3 con los grupos taxonómicos combinados alineados.
- Seleccione las posiciones de aminoácidos previamente identificadas para las especies de plantilla escribiendo las posiciones de aminoácidos, separadas por comas, en el cuadro Introducir posiciones de residuos de aminoácidos y, a continuación, seleccionando Copiar a la lista de residuos. Seleccione directamente los residuos en la secuencia de plantillas de la caja lanzadera.
- Haga clic en Actualizar informe para actualizar la página y mostrar las predicciones de susceptibilidad de nivel 3.
NOTA: El nivel 3 utiliza un conjunto simple de reglas derivadas de descriptores básicos de propiedades funcionales de cadena lateral (por ejemplo, alifáticas, aromáticas) y dimensiones moleculares (diferencias de peso molecular >30 g/mol) para determinar si es probable que las diferencias en posiciones clave afecten las interacciones de proteínas13.
9. Visualización de datos SeqAPASS de nivel 3
NOTA: Al igual que en los niveles anteriores, los informes primario y completo están disponibles. Además de los datos idénticos a los datos de los Niveles 1 y 2, el Informe primario muestra posiciones de aminoácidos, abreviaturas y una susceptibilidad sí/no (Y/N) similar a la predicción de la plantilla. Del mismo modo, el informe completo contiene información sobre la clasificación de la cadena lateral de aminoácidos y el peso molecular.
- En la página Informe de nivel 3, desplácese hasta la parte inferior para ver un informe de los resultados. Haga clic en Descargar tabla en la parte inferior del informe para guardar la tabla .
- Haga clic en Ver informe de resumen de nivel 3 para ver y descargar una tabla de informe de resumen que presenta datos ordenados por grupo taxonómico. Haga clic en el signo más + junto a Visualización en la página Informe de nivel 3 para abrir una pestaña separada del navegador que muestra la información definida por el usuario y la opción de ver los resultados en forma de un mapa de calor interactivo.
- Haga clic en Mapa de calor en la página Información de visualización para abrir el gráfico interactivo y los controles y permitir que la visualización del mapa de calor se actualice activamente para reflejar los cambios en la tabla de datos. Realice los siguientes pasos opcionales para personalizar el mapa de calor.
- (OPCIONAL) Seleccione Opciones de informe para cambiar entre un informe simple, que muestra la posición del aminoácido, la abreviatura de una letra y la similitud de aminoácidos, o un informe completo, que muestra información detallada sobre cada aminoácido seleccionado.
- (OPCIONAL) Seleccione Opciones de informe para cambiar la forma en que se muestran las especies, ya sea por Nombre común o Nombre científico.
NOTA: Dentro del Informe simple, los aminoácidos se clasifican como una coincidencia total (azul oscuro), coincidencia parcial (azul claro, sustituciones que cumplen solo un criterio) o no una coincidencia (amarillo, sustituciones que no cumplen ninguno de los criterios) al aminoácido plantilla. El informe completo muestra las comparaciones como una coincidencia total (azul oscuro) o no una coincidencia (amarillo). - (OPCIONAL) Seleccione Selecciones opcionales para resaltar información útil como candidatos ortológicos, especies amenazadas, especies en peligro de extinción u organismos modelo comunes.
- (OPCIONAL) Seleccione Configuración del mapa de calor para seleccionar opciones de personalización adicionales, incluida la adición o eliminación de columnas, leyendas y texto.
- Haga clic en Descargar diagrama de caja para elegir un tipo de archivo y guardar la visualización.
10. Interpretación de los resultados de SeqAPASS: Líneas de evidencia para la conservación de proteínas
NOTA: Para facilitar la interpretación, esta herramienta incluye un Informe de resumen de decisiones (Informe DS) diseñado para integrar datos entre niveles. El Informe DS contiene los resultados (es decir, tablas de datos y/o visualizaciones) que el usuario ha seleccionado y permite la evaluación rápida de las predicciones de susceptibilidad en múltiples niveles para múltiples especies simultáneamente.
- Haga clic en Push Level # to DS Report en las páginas de visualización de resultados o datos y espere a que los datos se "inserten" y la pestaña DS Report se active.
NOTA: Si los resultados o cualquier cambio no se han insertado en el Informe de DS, el nivel de inserción # al Informe de DS permanecerá activo hasta que se seleccione. Si se ha cambiado una configuración, aparecerá el texto Haga clic para insertar nuevos cambios hasta que los cambios se inserten en el informe. Las visualizaciones se pueden enviar al Informe de DS en cualquier momento durante la evaluación. - Seleccione la ficha Informe de DS en cualquier momento para acceder a la página de DS .
NOTA: Para todas las especies alineadas en el Nivel 1, la tabla del Informe Resumido de Decisión Final contiene los datos importantes y las predicciones de susceptibilidad para cada análisis. Si una especie en la tabla DS no se incluyó en el informe de Nivel 3 pero se encontró en trabajos de Nivel 1 y / o Nivel 2, la celda de la tabla recibirá una designación no aplicable (NA) para la predicción de susceptibilidad de Nivel 3.
Access restricted. Please log in or start a trial to view this content.
Resultados
Para demostrar la aplicación de la herramienta SeqAPASS y resaltar nuevas características, se describen dos estudios de caso que representan casos en los que la conservación de proteínas predice que existen diferencias en la susceptibilidad química entre especies (transtiretina humana) y que no hay diferencias (μ receptor opioide [MOR]). El primero de estos ejemplos aborda las comparaciones estructurales / secuencia de proteínas para predecir el dominio de aplicabilidad para las vías de resultados adversos (AOP, ...
Access restricted. Please log in or start a trial to view this content.
Discusión
Existe un reconocimiento generalizado de que no es factible probar empíricamente suficientes especies para capturar la diversidad genómica, fenotípica, fisiológica y de comportamiento de los organismos vivos que pueden estar expuestos a productos químicos de interés toxicológico. El objetivo de SeqAPASS es maximizar el uso de la secuencia de proteínas existente y en continua expansión y los datos estructurales para ayudar e informar la extrapolación de datos / conocimientos de toxicidad química de organismos p...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Los autores agradecen al Dr. Daniel L. Villeneuve (U.S. EPA, Center for Computational Toxicology and Exposure) y al Dr. Jon A. Doering (Departamento de Ciencias Ambientales, Louisiana State University) por proporcionar comentarios sobre un borrador anterior del manuscrito. Este trabajo fue apoyado por la Agencia de Protección Ambiental de los Estados Unidos. Las opiniones expresadas en este documento son las de los autores y no reflejan necesariamente las opiniones o políticas de la Agencia de Protección Ambiental de los Estados Unidos, ni la mención de nombres comerciales o productos comerciales indica respaldo por parte del gobierno federal.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Spreadsheet program | N/A | N/A | Any program that can be used to view and work with csv files (e.g. Microsoft Excel, OpenOffice Calc, Google Docs) can be used to access data export files. |
| Basic computing setup and internet access | N/A | N/A | SeqAPASS is a free, online tool that can be easily used via an internet connection. No software downloads are required. |
Referencias
- Krewski, D., et al. Toxicity testing in the 21st century: a vision and a strategy. Journal of Toxicology and Environmental Health, Part B. 13 (2-4), 51-138 (2010).
- Wang, Z., Walker, G. W., Muir, D. C. G., Nagatani-Yoshida, K. Toward a global understanding of chemical pollution: A first comprehensive analysis of national and regional chemical inventories. Environmental Science & Technology. 54 (5), 2575-2584 (2020).
- Brooks, B. W., et al. Toxicology advances for 21st century chemical pollution. One Earth. 2 (4), 312-316 (2020).
- Kostal, J., Voutchkova-Kostal, A. Going all in: A strategic investment in in silico toxicology. Chemical Research in Toxicology. 33 (4), 880-888 (2020).
- Cheng, W., Doering, J. A., LaLone, C., Ng, C. Integrative computational approaches to inform relative bioaccumulation potential of per- and polyfluoroalkyl substances (PFAS) across species. Toxicology Sciences. 180 (2), 212-223 (2021).
- Kostich, M. S., Lazorchak, J. M. Risks to aquatic organisms posed by human pharmaceutical use. Science of the Total Environment. 389 (2-3), 329-339 (2008).
- Gunnarsson, L., Jauhiainen, A., Kristiansson, E., Nerman, O., Larsson, D. G. Evolutionary conservation of human drug targets in organisms used for environmental risk assessments. Environmental Science & Technology. 42 (15), 5807-5813 (2008).
- LaLone, C. A., et al. Evidence for cross species extrapolation of mammalian-based high-throughput screening assay results. Environmental Science & Technology. 52 (23), 13960-13971 (2018).
- LaLone, C. A., et al. Editor's highlight: Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS): A web-based tool for addressing the challenges of cross-species extrapolation of chemical toxicity. Toxicology Sciences. 153 (2), 228-245 (2016).
- Head, J. A., Hahn, M. E., Kennedy, S. W. Key amino acids in the aryl hydrocarbon receptor predict dioxin sensitivity in avian species. Environmental Science & Technology. 42 (19), 7535-7541 (2008).
- Bass, C., et al. Mutation of a nicotinic acetylcholine receptor β subunit is associated with resistance to neonicotinoid insecticides in the aphid Myzus persicae. BMC Neuroscience. 12, 51-51 (2011).
- Erdmanis, L., et al. Association of neonicotinoid insensitivity with a conserved residue in the loop d binding region of the tick nicotinic acetylcholine receptor. Biochemistry. 51 (23), 4627-4629 (2012).
- Doering, J. A., et al. et al. In silico site-directed mutagenesis informs species-specific predictions of chemical susceptibility derived from the Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS) tool. Toxicology Sciences. 166 (1), 131-145 (2018).
- Noyes, P. D., et al. Evaluating chemicals for thyroid disruption: Opportunities and challenges with in vitro testing and adverse outcome pathway approaches. Environmental Health Perspectives. 127 (9), 95001(2019).
- Park, G. Y., Jamerlan, A., Shim, K. H., An, S. S. A. Diagnostic and treatment approaches involving transthyretin in amyloidogenic diseases. Int J Mol Sci. 20 (12), 2982(2019).
- Rabah, S. A., Gowan, I. L., Pagnin, M., Osman, N., Richardson, S. J. Thyroid hormone distributor proteins during development in vertebrates. Front Endocrinol (Lausane). 10, 506(2019).
- Richardson, S. J. Cell and molecular biology of transthyretin and thyroid hormones. International Review of Cytology. 258, 137-193 (2007).
- Yamauchi, K., Ishihara, A. Transthyretin and Endocrine Disruptors. Recent Advances in Transthyretin Evolution, Structure and Biological Functions. Richardson, S. J., Cody, V. , Springer. Berlin Heidelberg, Germany. 159-171 (2009).
- Iakovleva, I., et al. Tetrabromobisphenol A is an efficient stabilizer of the transthyretin tetramer. PLoS One. 11 (4), 0153529(2016).
- Ishihara, A., Sawatsubashi, S., Yamauchi, K. Endocrine disrupting chemicals: Interference of thyroid hormone binding to transthyretins and to thyroid hormone receptors. Molecular and Cellular Endocrinology. 199 (1), 105-117 (2003).
- Kar, S., Sepúlveda, M. S., Roy, K., Leszczynski, J. Endocrine-disrupting activity of per- and polyfluoroalkyl substances: Exploring combined approaches of ligand and structure based modeling. Chemosphere. 184, 514-523 (2017).
- Morais-de-Sa, E., Pereira, P. J., Saraiva, M. J., Damas, A. M. The crystal structure of transthyretin in complex with diethylstilbestrol: A promising template for the design of amyloid inhibitors. Journal of Biological Chemistry. 279 (51), 53483-53490 (2004).
- Morgado, I., Campinho, M. A., Costa, R., Jacinto, R., Power, D. M. Disruption of the thyroid system by diethylstilbestrol and ioxynil in the sea bream (Sparus aurata). Aquatic Toxicology. 92 (4), 271-280 (2009).
- Yamauchi, K., Prapunpoj, P., Richardson, S. J. Effect of diethylstilbestrol on thyroid hormone binding to amphibian transthyretins. General and Comparative Endocrinology. 119 (3), 329-339 (2000).
- Zhang, J., et al. Structure-based virtual screening protocol for in silico identification of potential thyroid disrupting chemicals targeting transthyretin. Environmental Science & Technology. 50 (21), 11984-11993 (2016).
- Ren, X. M., et al. Binding interactions of perfluoroalkyl substances with thyroid hormone transport proteins and potential toxicological implications. Toxicology. 366-367, 32-42 (2016).
- Wilson, N., Mbabazi, K., Seth, P., Smith, H., Davis, N. L. Drug and opioid-involved overdose deaths - United States, 2017-2018. Morbidity and Mortality Weekly Report. 69 (11), 290-297 (2020).
- EPA. National Pollutant Discharge Elimination System (NPDES). United States Environmental Protection Agency. , Available from: https://www.epa.gov/npdes/npdes-resources (2018).
- Duvallet, C., Hayes, B. D., Erickson, T. B., Chai, P. R., Matus, M. Mapping community opioid exposure through wastewater-based epidemiology as a means to engage pharmacies in harm reduction efforts. Preventing Chronic Disease. 17, 200053(2020).
- Gushgari, A. J., Venkatesan, A. K., Chen, J., Steele, J. C., Halden, R. U. Long-term tracking of opioid consumption in two United States cities using wastewater-based epidemiology approach. Water Research. 161, 171-180 (2019).
- Lau, B., Bretaud, S., Huang, Y., Lin, E., Guo, S. Dissociation of food and opiate preference by a genetic mutation in zebrafish. Genes Brain Behave. 5 (7), 497-505 (2006).
- Bossé, G. D., Peterson, R. T. Development of an opioid self-administration assay to study drug seeking in zebrafish. Behavioural Brain Research. 335, 158-166 (2017).
- Mottaz, H., et al. Dose-dependent effects of morphine on lipopolysaccharide (LPS)-induced inflammation, and involvement of multixenobiotic resistance (MXR) transporters in LPS efflux in teleost fish. Environmental Pollution. 221, 105-115 (2017).
- Manglik, A., et al. Crystal structure of the µ-opioid receptor bound to a morphinan antagonist. Nature. 485 (7398), 321-326 (2012).
- Comer, S. D., Cahill, C. M. Fentanyl: Receptor pharmacology, abuse potential, and implications for treatment. Neuroscience & Biobehavioral Reviews. 106, 49-57 (2019).
- Podlewska, S., Bugno, R., Kudla, L., Bojarski, A. J., Przewlocki, R. Molecular modeling of µ opioid receptor ligands with various functional properties: PZM21, SR-17018, morphine, and fentanyl-simulated interaction patterns confronted with experimental data. Molecules. 25 (20), 4636(2020).
- Huang, W., et al. Structural insights into µ-opioid receptor activation. Nature. 524 (7565), 315-321 (2015).
- Lipiński, P. F. J., et al. Fentanyl family at the mu-opioid receptor: Uniform assessment of binding and computational analysis. Molecules. 24 (4), 740(2019).
- Boland, L. A., Angles, J. M. Feline permethrin toxicity: Retrospective study of 42 cases. Journal of Feline Medicine and Surgery. 12 (2), 61-71 (2010).
- Stevenson, B. J., Pignatelli, P., Nikou, D., Paine, M. J. Pinpointing P450s associated with pyrethroid metabolism in the dengue vector, Aedes aegypti: developing new tools to combat insecticide resistance. PLoS Neglected Tropical Diseases. 6 (3), 1595(2012).
- Ankley, G. T., Gray, L. E. Cross-species conservation of endocrine pathways: A critical analysis of tier 1 fish and rat screening assays with 12 model chemicals. Environmental Toxicology and Chemistry. 32 (5), 1084-1087 (2013).
- Meteyer, C. U., Rideout, B. A., Gilbert, M., Shivaprasad, H. L., Oaks, J. L. Pathology and proposed pathophysiology of diclofenac poisoning in free-living and experimentally exposed oriental white-backed vultures (Gyps bengalensis). Journal of Wildlife Diseases. 41 (4), 707-716 (2005).
- EPA. ECOTOX User Guide: ECOTOXicology Knowledgebase System. EPA, United States Environmental Protection Agency. , Available from: https://cfpub.epa.gov/ecotox/index.cfm (2021).
- ECOS Environmental Conservation Online System. U.S. Fish & Wildlife Service. , Available from: https://ecos.fws.gov/ecp/ (2021).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados