このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
タンパク質保存の迅速な評価のための種間の感受性を予測するための配列アラインメントツールのデモンストレーション
要約
ここでは、米国環境保護庁の配列アライメントを利用して種感受性を予測する(SeqAPASS)ツールの最新バージョンを利用するためのプロトコルを紹介します。このプロトコルは、タンパク質の保存を迅速に分析し、種全体の化学物質感受性のカスタマイズ可能で簡単に解釈可能な予測を提供するためのオンラインツールの適用を示しています。
要約
米国環境保護庁の種感受性を予測するための配列調整(SeqAPASS)ツールは、研究者や規制当局が種全体の毒性情報を推定できるようにする、高速で無料で入手できるオンラインスクリーニングアプリケーションです。ヒト細胞、マウス、ラット、ゼブラフィッシュなどのモデル系の生物学的標的については、さまざまな化学物質の毒性データを利用できます。タンパク質ターゲット保全の評価を通じて、このツールは、そのようなモデルシステムから生成されたデータを、毒性データが不足している他の何千もの種に外挿し、相対的な固有の化学物質感受性の予測をもたらすために使用できます。ツールの最新リリース(バージョン2.0〜6.1)には、公開用のデータの迅速な合成、解釈、および使用とプレゼンテーション品質のグラフィックスを可能にする新機能が組み込まれています。
これらの機能の中には、カスタマイズ可能なデータ視覚化と、解釈を容易にするためにSeqAPASSデータを要約するように設計された包括的な要約レポートがあります。このホワイトペーパーでは、ジョブの送信、さまざまなレベルのタンパク質配列比較のナビゲート、および結果のデータの解釈と表示を通じてユーザーをガイドするプロトコルについて説明します。SeqAPASS v2.0-6.0 の新機能が強調表示されています。さらに、このツールを使用してトランスサイレチンとオピオイド受容体タンパク質の保存に焦点を当てた2つのユースケースについて説明します。最後に、SeqAPASSの長所と限界について説明し、ツールの適用性の範囲を定義し、種間外挿のさまざまなアプリケーションを強調します。
概要
従来、毒物学の分野は、化学物質の安全性評価に必要なデータを提供するために、全動物試験の使用に大きく依存してきました。このような方法は、通常、コストとリソースを大量に消費します。しかし、現在使用されている化学物質の数が多く、新しい化学物質が急速に開発されているため、世界的には、より効率的な化学物質スクリーニング方法の必要性が認識されています1,2。この必要性とその結果としての動物実験からのパラダイムシフトは、ハイスループットスクリーニングアッセイ、ハイスループットトランスクリプトミクス、次世代シーケンシング、計算モデリングなど、多くの新しいアプローチ方法の開発につながり、これらは有望な代替試験戦略です3,4。
化学物質曝露によって潜在的に影響を受ける多様な種全体で化学物質の安全性を評価することは、従来の毒性試験だけでなく、新しいアプローチ方法でも永続的な課題でした。比較および予測毒物学の進歩は、異なる種の相対的な感受性を理解するためのフレームワークを提供し、計算方法の技術的進歩はこれらの方法の適用可能性を高め続けています。過去10年間、既存の遺伝子およびタンパク質配列データベースと特定の化学分子標的の知識を活用して、種間外挿の予測アプローチをサポートし、典型的なモデル生物を超えて化学物質の安全性評価を強化するいくつかの戦略が議論されてきました5,6,7,8。
科学を行動に移し、予測毒物学におけるこれらの基礎研究に基づいて構築し、化学試験の取り組みに優先順位を付け、意思決定をサポートするために、米国環境保護庁の種感受性を予測するための配列調整(SeqAPASS)ツールが作成されました。このツールは、絶えず拡大するタンパク質配列情報のパブリックリポジトリを使用して、種の多様性にわたる化学的感受性を予測する、公開された無料で利用可能なWebベースのアプリケーションです9。このツールは、特定の化学物質に対する種の相対的な固有感受性は、その化学物質の既知のタンパク質標的の保存を評価することによって決定できるという原則に基づいて、すべての種に対する既知の感受性を持つ種のタンパク質アミノ酸配列を既存のタンパク質配列データと迅速に比較します。この評価は、(1)一次アミノ酸配列、(2)機能ドメイン、(3)重要なアミノ酸残基の比較を含む3つのレベルの分析によって完了し、それぞれが化学タンパク質相互作用に関するより深い知識を必要とし、感受性予測における分類学的分解能を高めます。SeqAPASSの大きな強みは、目的の化学-タンパク質またはタンパク質-タンパク質相互作用に関して利用可能な情報の量に基づいて、ターゲットの保存に向けた証拠の行を追加することで、ユーザーが評価をカスタマイズして洗練できることです。
最初のバージョンは2016年にリリースされ、ユーザーは一次アミノ酸配列と機能ドメインを合理化して化学物質感受性を予測でき、最小限のデータ視覚化機能しか含まれていませんでした(表1)。個々のアミノ酸の違いは、化学-タンパク質相互作用における種間の違いの重要な決定要因であることが示されており、これは種の化学的感受性に影響を与える可能性があります10,11,12。したがって、直接的な化学的相互作用に重要な重要なアミノ酸を考慮するために、後続のバージョンが開発されました13。利害関係者とユーザーのフィードバックに応えて、このツールは毎年バージョンリリースされ、種間外挿の課題に対処するための研究者と規制コミュニティの両方のニーズを満たすように設計された追加の新機能が追加されています(表1)。2020年のSeqAPASSバージョン5.0のリリースにより、データの視覚化とデータ合成のオプション、外部リンク、要約テーブルとレポートのオプション、およびグラフィック機能を組み込んだユーザー中心の機能が導入されました。全体として、このバージョンの新しい属性と機能により、データ合成、外部データベース間の相互運用性、および種間感受性の予測のためのデータ解釈の容易さが向上しました。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1. はじめに
メモ: ここで紹介するプロトコルは、ツールの有用性と主要な機能に重点を置いています。方法、機能、およびコンポーネントの詳細な説明は、包括的なユーザーガイド(表1)のWebサイトにあります。
表 1: SeqAPASS ツールの進化 初期展開から SeqAPASS ツールに追加された機能と更新プログラムの一覧。略語:SeqAPASS =種全体の感受性を予測するための配列アラインメント。ECOTOX = ECOTOXicologyナレッジベース。 この表をダウンロードするには、ここをクリックしてください。
- Chrome を使用した https://seqapass.epa.gov/seqapass に移動します。[ログイン] を選択して既存のアカウントを使用するか、指示に従って SeqAPASS アカウントを作成し、ユーザーが完了したジョブを実行、保存、アクセス、およびカスタマイズできるようにします。
- 分析を行う前に、まず既存の文献または既存のデータを確認して、目的のタンパク質と標的種または感受性種を特定します(図1)。SeqAPASSには、クエリタンパク質の識別に役立つ外部リソースへのリンクが含まれているため、タンパク質ターゲットの特定の下にあるドロップダウンボタンをクリックして、関連するリソースにアクセスします。
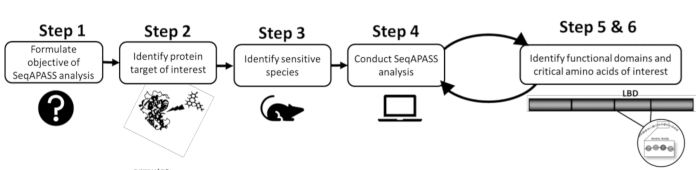
図1:SeqAPASS問題の定式化:分析を成功させるために必要な予備情報の概略図。略語:SeqAPASS =種全体の感受性を予測するための配列アラインメント。LBD = リガンド結合ドメイン。この図の拡大版を表示するには、ここをクリックしてください。

図 2: データベース間の SeqAPASS 相互運用性。 SeqAPASSに統合された外部ツール、データベース、およびリソースの概略図。略語:SeqAPASS =種全体の感受性を予測するための配列アラインメント。AOP =有害転帰経路;NCBI = 国立バイオテクノロジー情報センター;ECOTOX = ECOTOXicologyナレッジベース。 この図の拡大版を表示するには、ここをクリックしてください。
表 2: SeqAPASS ツールに統合されたリンク、リソース、およびツール。 SeqAPASSツールに活用されるさまざまなデータソース、リンク、およびリソースのリスト。略語: SeqAPASS = 種全体の感受性を予測するための配列アラインメント。 この表をダウンロードするには、ここをクリックしてください。
2. SeqAPASS クエリの開発と実行: レベル 1
注:レベル1解析では、クエリタンパク質の一次アミノ酸配列全体が、利用可能な配列情報を持つすべての種の一次アミノ酸配列と比較されます。このツールは、アルゴリズムを使用して公開されているデータをマイニング、収集、コンパイルし、種間でアミノ酸配列を迅速に調整および比較します。バックエンドは、国立バイオテクノロジー情報センター(NCBI)データベースからの情報を保存し、タンパク質基本ローカルアライメント検索ツール(BLASTp)54 および制約ベースのマルチプルアライメントツール(COBALT)55のスタンドアロンバージョンを戦略的に利用します。
- [一次アミノ酸配列の比較] で、[種別] または [アクセッション別] をクリックします。「種別」選択を使用して、種を入力するか、種のリストから選択して目的のタンパク質ターゲットを選択します。
- タンパク質アクセッション(NCBIタンパク質ID)をアクセッション別テキストボックスにアクセッションを入力して直接提出します。
- [ 実行の要求 ] を選択してクエリを送信します。送信が完了したら、送信が成功したことを示す通知がブラウザウィンドウの右上隅に表示されるのを待ちます。
- ページの上部にある [SeqAPASS 実行ステータス ] タブを選択して、そのユーザー アカウントで実行されたすべての SeqAPASS 実行の一覧を表示し、完了率を確認します。
- 適切なラジオボタンが選択されているときに[ データの更新 ]をクリックして、レベル2およびレベル3の実行のステータスを確認します。
- ページの上部にある [ SeqAPASS レポートの表示 ] タブを選択して、そのアカウントで完了したすべてのレポートのリストにアクセスします。
- SeqAPASSレポートの表示タブで、目的のクエリタンパク質を選択します。「選択レポートの要求」をクリックして「レベル 1 照会タンパク質情報」ページを開き、結果、データ・カスタマイズ・オプション、視覚化、および要約レポートを表示します。
- 既定では、[ レポートの表示 ] を選択して、Web ブラウザーでデータを表示します。または、[ レポートの保存 ] を選択して、生データを.zipファイルとしてダウンロードします。
注:レベル1分析に必要な時間は、その時点でのグローバルユーザーの要求、キューにサブミットされたジョブの数、およびサブミットされたジョブに存在するタンパク質情報の量によって異なります(バージョン5.1の平均23分)。タンパク質ターゲットが以前に完成している場合は、提出後数秒でデータが利用可能になります。
3. SeqAPASS クエリの開発と実行: レベル 2
注:タンパク質配列全体が化学的相互作用に直接関与しているわけではないため、レベル2分析では、機能ドメインのアミノ酸配列のみを比較して、より低い分類ランク(クラス、オーダー、ファミリーなど)での感受性予測を行います。
- 「レベル 1 クエリタンパク質情報」ページで、レベル 2 ヘッダーの横にあるプラス記号 + をクリックして、「レベル 2 クエリー」メニューに入力します。
- 目的のタンパク質(クエリタンパク質)の適切なドメインを特定します。
- ドメインが識別されていない場合は、NCBI保存ドメインデータベース(CDD)(表1)への統合リンクをクリックして、適切なドメイン選択の特定に役立てることができます。
注: 通常、レベル 2 では、特定のヒット ドメインのみがクエリとして選択されます。
- ドメインが識別されていない場合は、NCBI保存ドメインデータベース(CDD)(表1)への統合リンクをクリックして、適切なドメイン選択の特定に役立てることができます。
- [ ドメインの選択 ] ボックスをクリックして、クエリタンパク質の機能ドメインのリストを自動入力します。
- ドロップダウン リストからドメイン アクセッション を選択し、[ ドメインの実行の要求 ] ボタンをクリックしてレベル 2 クエリを開始します。送信が完了したら、送信が成功したことを示す通知が表示されるのを待ちます。
- [ レベル 2 と 3 の実行 を更新] をクリックしてレベル 2 のデータを入力すると、送信から数秒以内に使用できるようになります。
- [ レベル 2 データの表示] で、 ドロップダウン リストから完了したドメイン アクセッション を選択し、[ レベル 2 データの表示 ] ボタンをクリックして結果を新しいページで開きます。
4.データへのアクセスと理解:SeqAPASSレベル1およびレベル2
- タンパク質 情報のクエリ ページの一番下までスクロールして、結果のレポートを表示します - プライマリレポート はデフォルトでレベル1と2の分析で提供されます。[ 完全なレポート ] ラジオ ボタンを選択すると、すべてのシーケンス ヒットとアライメント メトリックを提供するより詳細なレポートが表示されます。両方のレポートで適切な アクセッション/ID/名前 をクリックすると、NCBIデータベース内の透過的なタンパク質アラインメントおよび分類情報にアクセスできます。
- 結果テーブルの右側までスクロールして、 ECOTOX 列を表示します。ECOTOXicologyナレッジベース(ECOTOX) へのリンク をクリックして、感受性予測のある種に対応する毒性データを迅速に収集します。
注:ECOTOXは、水生および陸生植物および野生生物の単一の化学毒性データを提供する、包括的で公開されているナレッジベースです。SeqAPASS v6.0には、化学物質や関心のある種ごとに関連するECOTOXデータにすばやく接続するためのECOTOXウィジェットが含まれています。 - [ 表のダウンロード ] をクリックして、表をスプレッドシート ファイルとして保存します。[ 概要レポートの表示 ] ボタンをクリックして、分類グループ別に並べ替えられたデータを表示する概要レポート テーブルを表示およびダウンロードします。
注: データサマリーテーブルは、プライマリレポートとフルレポートの両方で使用でき、特定のターゲットの予測の概要を提供します。
5. データ設定の操作: SeqAPASS レベル 1 およびレベル 2
注:レベル1とレベル2の両方の分析では、タンパク質の類似性が高いほど、化学物質がクエリ種/タンパク質と同様の方法でタンパク質と相互作用する可能性が高くなり、この分子標的を持つ化学物質の潜在的な影響を受けやすくなると想定されています。これらのデータは類似しているため、レベル1と2のデータを理解するための手順は、単一のプロトコルでまとめられています。
- タンパク質情報の照会の上部にあるサブメニューを参照して、レポート設定にアクセスして操作し、ほとんどの分析ですべてのレポートオプションにデフォルト設定を使用します。既定の設定を変更することが科学的な正当性がある場合は、次の省略可能な手順に従います。
- (オプション)[感受性カットオフ] の横にあるプラス記号 + をクリックして、新しいタブで感受性カットオフ設定を表示および調整します。ドロップダウン リストから新しいカットオフ値を選択するか、ユーザー定義のカットオフ値を入力します。
- (オプション)デフォルト以外のものが必要な場合は、 E-Value (偶然に発生すると予想される異なるアライメントの数)フィールドの数値を変更します。
注:箱の中の数字よりも大きいE値を持つタンパク質は、一次レポートから削除されます。 - (オプション) [分類グループで並べ替え] オプションを使用して、結果テーブルの [ フィルターされた 分類グループ] 列に表示する分類階層のレベルを選択します。
注:分類階層を変更すると、カットオフの上に見つかった各フィルタリングされたグループの種に基づく感受性の予測も変更されます。 - (オプション)デフォルト以外のものが必要な場合は、共通ドメイン(タンパク質がクエリタンパク質と共有する必要がある共通 ドメイン の数)フィールドを変更します。
注: デフォルト設定は 1 であるため、クエリタンパク質と少なくとも 1 つの共通ドメインを共有しない配列は除外されます。 - (オプション)類似度がカットオフ以上の場合、またはヒットがオルソログ候補として識別された場合にのみ、Y の感受性予測を返すには、[種の読み取り] で [いいえ] を選択します。
注: この設定の既定値は [はい] で、すべてのオルソログ候補、感受性カットオフより上にリストされているすべての種、およびカットオフより上の 1 つ以上の種を持つ同じ分類グループからのカットオフより下のすべての種について、Y の感受性予測が報告されることを意味します。
- [ 現在のレポート設定のダウンロード ] ボタンをクリックして、現在適用されている設定をキャプチャしたファイルをダウンロードします。
注: 選択した特定の評価レベル (1、2、または 3) によって、レポートに表示される設定が決まります。
6. データの視覚化: シークアパス レベル 1 およびレベル 2
- [ビジュアライゼーション] の横にあるプラス記号 + をクリックし、[データの視覚化] ボタンをクリックすると、別のタブが開き、ユーザー定義の情報と結果の対話型グラフを選択するオプションが表示されます。
- [ 箱ひげ図 ] をクリックして、インタラクティブな箱ひげ図コントロールと印刷コントロールを開き、箱ひげ図ビジュアリゼーションをアクティブに更新してデータ テーブルへの変更を反映し、出版品質およびプレゼンテーション品質のグラフィックスを提供します。
注: デフォルトの箱ひげ図では、X 軸に種グループ、Y 軸に類似度が表示されます。箱ひげ図には、感受性カットオフ(破線)、クエリ種と比較した種間の類似率、各分類群の平均値と中央値、25パーセンタイルと75パーセンタイル、四分位数の範囲が表示されます。分析の目的とユーザーのニーズに応じて、多くの箱ひげ図機能は、次のオプションのステップで変更できます。- (オプション)表示される分類グループをカスタマイズするには、[コントロール]セクションの[分類グループ]ボックスを参照してください。グループを削除するには、名前をスクロールして [x] を選択するか、[分類グループ] ドロップダウン メニューを使用します。
- (オプション)関心のある種または特定の事前定義されたグループ(絶滅危惧種や絶滅危惧種など)を特定する凡例を追加するには、X軸の 分類グループ名 にカーソルを合わせると、類似度が最も高い順に上位3種を一覧表示するポップアップボックスがアクティブになります。凡例の種にカーソルを合わせると、対応する 種 情報を含むポップアップ ボックスが生成されます。特定の分類グループの ボックス をクリックして、種と予測をリストしたダウンロード可能な要約表を生成します。
- [ 箱ひげ図をダウンロード ]をクリックして、ファイルの種類を選択し、幅/高さの解像度をカスタマイズして、ビジュアリゼーションを保存します。
7. シークアパス解析の開発と実行: レベル 3
注:レベル3分析では、クエリタンパク質内のユーザーが同定したアミノ酸残基を評価し、種間でこれらの残基の保存を迅速に比較します。これらの残基が保存されている種は、テンプレート種/タンパク質と同様の方法で化学物質と相互作用する可能性が高いと考えられています。レベル3は個々のアミノ酸に焦点を当てているため、化学-タンパク質またはタンパク質-タンパク質相互作用に重要なアミノ酸残基の詳細な知識が利用可能な場合にのみ分析を実行できます。
- 「レベル 1 クエリタンパク質情報」ページの「レベル 3」ヘッダーの横にあるプラス記号 + をクリックして、「レベル 3 クエリー」メニューに入力します。
- 参照エクスプローラーの横にあるプラス記号 + をクリックして参照エクスプローラー ツールを開きます。これは、使用可能な文献を照会するための定義済みのブール文字列を生成し、レベル 3 評価で使用される重要なアミノ酸の同定をサポートする適切な文献を特定するのに役立ちます (表 2 および図 2)。
- (オプション)クエリタンパク質が自動入力されたら、タンパク質 名の追加機能を使用してタンパク質 を追加します。
- [ Google Scholar リンクの生成 ] をクリックすると、関連する検索語句を含む自動生成された検索文字列を含むポップアップが開きます。
- [ Google 学者を 検索] をクリックして、検索文字列を使用して文献データベースを検索します。
- または、[ クリップボードにコピー ] をクリックし、 参照エクスプローラーの関数を使用して用語を追加または削除して検索文字列をカスタマイズします。
8.同定された文献を使用して重要なアミノ酸残基を特定する
- ユーザーが選択した種を整列させる テンプレートシーケンス を、レベル3クエリメニューで選択します。
注:このテンプレート配列は、通常、重要なアミノ酸が同定された文献に基づいて選択され、レベル1およびレベル2で照会されたものと同じ種または異なる種である可能性があります。- (オプション) 「追加 比較」ボックスを使用して、「 プライマリ/フル・レポート 」表に表示されないアクセッション/シーケンスを比較します。
- 完了したレベル 3 実行を識別するために、[ レベル 3 実行名を入力 ] テキスト ボックスにレベル 3 実行のユーザー定義名を入力します。評価ごとに一意の名前を選択します。
- [ 分類グループの選択] フィールドで目的の分類グループを選択します。分類グループを選択して、その分類グループでテーブルを自動フィルター処理します。
- 結果テーブルで、テンプレートシーケンスに整列させる 種の横にあるチェックボックス を手動でクリックします。
注: 適切な配置を行うには、一度に 1 つの分類グループをテンプレートと比較する必要があります。目的の種に対して同様にアノテーションされたタンパク質のみを選択します。比較するシーケンスを選択するときは、特定のシーケンス(仮説 、低品質、部分的など)に注意を払うことが重要です。含めることの明確な正当性がない限り、これらの配列は不完全または不適切な配列情報のために予測を歪める可能性があるため、除外するのが最善です。 - 手順を繰り返して、関心のあるすべての分類グループを揃えます。
- すべての種が整列されたら、[ レベル 2 と 3 の実行 を更新] をクリックして、[レベル 3 の実行名の選択 ] メニューに完了したレベル 3 のジョブを入力し、レベル 3 の配置からデータをすぐに取得します。
- [ レベル 3 データの結合 ] をクリックして、複数の分類グループの線形を結合します。
- または、1 つのレポートを表示するには、[ 表示するクエリの選択 ] でユーザー定義名を選択し、[ レベル 3 データの表示] をクリックします。
- 「レベル 3 レポートの結合」メニューでアミノ酸残基比較の基礎として使用するレベル 3 テンプレートを選択し、「次へ」をクリックします。
- [レベル 3 ジョブ] で、 比較する完了したジョブ を選択し、[ 次へ] をクリックします。 注文レベル 3 ジョブ 機能を使用して、必要に応じて分類グループを並べ替えます。[ レベル 3 データの表示 ] をクリックして、結合された分類グループを揃えたレベル 3 レポート ページを生成します。
- [アミノ酸残基位置の入力] ボックスにアミノ酸位置をコンマで区切って入力し、[残基リストにコピー] を選択して、テンプレート種に対して以前に特定されたアミノ酸位置を選択します。シャトルボックスからテンプレート配列の残基を直接選択します。
- [ レポートの更新 ] をクリックしてページを更新し、レベル 3 の感受性予測を表示します。
注:レベル3は、側鎖の機能特性(脂肪族、芳香族など)および分子寸法(分子量差>30 g / mol)の基本的な記述子から派生した単純な一連の規則を使用して、重要な位置の違いがタンパク質相互作用に影響を与える可能性があるかどうかを判断します13。
9. レベル 3 シーク APASS データの視覚化
注: 以前のレベルと同様に、プライマリレポートとフルレポートを使用できます。レベル1および2のデータと同じデータに加えて、プライマリレポートには、アミノ酸の位置、略語、およびテンプレート予測と同様のはい/いいえ(Y/N)の感受性が表示されます。同様に、完全なレポートには、アミノ酸側鎖の分類と分子量に関する情報が含まれています。
- レベル 3 のレポート ページで、一番下までスクロールして結果のレポートを表示します。レポートの下部にある [表のダウンロード] をクリックして、表を保存します。
- [レベル 3 サマリー レポートの表示] をクリックして、分類グループ別に並べ替えられたデータを表示するサマリー レポート テーブルを表示およびダウンロードします。[レベル 3 レポート] ページの [ビジュアライゼーション] の横にあるプラス記号 + をクリックすると、別のブラウザー タブが開き、ユーザー定義の情報と、対話型のヒート マップの形式で結果を表示するオプションが表示されます。
- [ビジュアリゼーション情報] ページの [ヒート マップ] をクリックして、対話型のグラフィックスとコントロールを開き、ヒート マップ ビジュアリゼーションをアクティブに更新してデータ テーブルへの変更を反映します。次のオプションの手順を実行して、ヒート マップをカスタマイズします。
- (オプション)[レポート オプション ] を選択して、アミノ酸の位置、1 文字の省略形、およびアミノ酸の類似性を表示する 簡易レポートと、選択した各アミノ酸に関する詳細情報を表示する 完全なレポートを切り替えます。
- (オプション)[ レポート オプション ] を選択して、種の表示方法を [ 共通 名] または [学名] で変更します。
注: シンプルレポート内では、アミノ酸はテンプレートアミノ酸に対して完全一致(濃い青)、部分一致(水色、1つの基準のみを満たす置換)、または一致しない(黄色、どちらの基準も満たさない置換)に分類されます。 完全なレポート には、合計一致(濃い青)または一致しない(黄色)のいずれかで比較が表示されます。 - (オプション) [オプションの選択 ] を選択して、オルソログ候補、絶滅危惧種、絶滅危惧種、共通モデル生物などの有用な情報をハイライト表示します。
- (オプション)[ ヒート マップ設定 ] を選択して、列、凡例、テキストの追加または削除など、追加のカスタマイズ オプションを選択します。
- [ 箱ひげ図をダウンロード ]をクリックしてファイル タイプを選択し、ビジュアリゼーションを保存します。
10. SeqAPASS結果の解釈:タンパク質保存のエビデンスライン
注: 解釈を容易にするために、このツールには、レベル間でデータを統合するように設計された 決定サマリー レポート (DS レポート) が含まれています。DSレポートには、ユーザーが選択した結果(データテーブルやビジュアライゼーションなど)が含まれており、複数の種の複数のレベルにわたる感受性予測を同時に迅速に評価できます。
- 結果ページまたはデータ視覚化ページから [レベル # を DS レポートにプッシュ] をクリックし、データが「プッシュ」されて [DS レポート ] タブがアクティブになるまで待ちます。
注: 結果または変更が DS レポートにプッシュされていない場合、 プッシュ レベル # to DS レポート は選択されるまでアクティブのままになります。設定が変更されている場合は、変更がレポートにプッシュされるまで、[ クリックして新しい変更をプッシュ ] というテキストが表示されます。ビジュアライゼーションは、評価中いつでも DS レポートにプッシュできます。 - [ DS レポート ] タブをいつでも選択して、DS ページにアクセスできます。
注:レベル1に分類されたすべての種について、 最終決定要約レポートの 表には、各分析の重要なデータと感受性予測が含まれています。DS 表の種がレベル 3 レポートに含まれていないが、レベル 1 および/またはレベル 2 のジョブで見つかった場合、表のセルはレベル 3 感受性予測に該当なし(NA)の指定を受けます。
Access restricted. Please log in or start a trial to view this content.
結果
SeqAPASSツールの適用を実証し、新機能を強調するために、タンパク質保存が種間で化学的感受性に違いがあること(ヒトトランスサイレチン)と違いがないこと(オピオイド受容体[MOR])μ予測する事例を表す2つのケーススタディについて説明します。これらの例の最初の例は、有害な結果経路(AOP、定義については表2を参照)の適用性のドメインを予測するためのタンパク質配列/構造の比較に対処?...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
毒物学的に関心のある化学物質にさらされる可能性のある生物のゲノム的、表現型的、生理学的、および行動的多様性を捉えるのに十分な種を経験的にテストすることは現実的ではないという認識が広まっています。SeqAPASSの目標は、既存および継続的に拡大しているタンパク質配列および構造データを最大限に活用して、分子レベルの比較を通じて、試験された生物から数百または数千の?...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者は開示する利益相反を持っていません。
謝辞
著者らは、Daniel L. Villeneuve博士(米国EPA、計算毒性学および曝露センター)とJon A. Doering博士(ルイジアナ州立大学環境科学部)が原稿の以前の草稿にコメントを提供してくれたことに感謝する。この作業は、米国環境保護庁の支援を受けました。このホワイトペーパーで表明された見解は著者のものであり、必ずしも米国環境保護庁の見解や政策を反映しているわけではなく、商号や商用製品の言及は連邦政府による承認を示すものでもありません。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| Spreadsheet program | N/A | N/A | Any program that can be used to view and work with csv files (e.g. Microsoft Excel, OpenOffice Calc, Google Docs) can be used to access data export files. |
| Basic computing setup and internet access | N/A | N/A | SeqAPASS is a free, online tool that can be easily used via an internet connection. No software downloads are required. |
参考文献
- Krewski, D., et al. Toxicity testing in the 21st century: a vision and a strategy. Journal of Toxicology and Environmental Health, Part B. 13 (2-4), 51-138 (2010).
- Wang, Z., Walker, G. W., Muir, D. C. G., Nagatani-Yoshida, K. Toward a global understanding of chemical pollution: A first comprehensive analysis of national and regional chemical inventories. Environmental Science & Technology. 54 (5), 2575-2584 (2020).
- Brooks, B. W., et al. Toxicology advances for 21st century chemical pollution. One Earth. 2 (4), 312-316 (2020).
- Kostal, J., Voutchkova-Kostal, A. Going all in: A strategic investment in in silico toxicology. Chemical Research in Toxicology. 33 (4), 880-888 (2020).
- Cheng, W., Doering, J. A., LaLone, C., Ng, C. Integrative computational approaches to inform relative bioaccumulation potential of per- and polyfluoroalkyl substances (PFAS) across species. Toxicology Sciences. 180 (2), 212-223 (2021).
- Kostich, M. S., Lazorchak, J. M. Risks to aquatic organisms posed by human pharmaceutical use. Science of the Total Environment. 389 (2-3), 329-339 (2008).
- Gunnarsson, L., Jauhiainen, A., Kristiansson, E., Nerman, O., Larsson, D. G. Evolutionary conservation of human drug targets in organisms used for environmental risk assessments. Environmental Science & Technology. 42 (15), 5807-5813 (2008).
- LaLone, C. A., et al. Evidence for cross species extrapolation of mammalian-based high-throughput screening assay results. Environmental Science & Technology. 52 (23), 13960-13971 (2018).
- LaLone, C. A., et al. Editor's highlight: Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS): A web-based tool for addressing the challenges of cross-species extrapolation of chemical toxicity. Toxicology Sciences. 153 (2), 228-245 (2016).
- Head, J. A., Hahn, M. E., Kennedy, S. W. Key amino acids in the aryl hydrocarbon receptor predict dioxin sensitivity in avian species. Environmental Science & Technology. 42 (19), 7535-7541 (2008).
- Bass, C., et al. Mutation of a nicotinic acetylcholine receptor β subunit is associated with resistance to neonicotinoid insecticides in the aphid Myzus persicae. BMC Neuroscience. 12, 51-51 (2011).
- Erdmanis, L., et al. Association of neonicotinoid insensitivity with a conserved residue in the loop d binding region of the tick nicotinic acetylcholine receptor. Biochemistry. 51 (23), 4627-4629 (2012).
- Doering, J. A., et al. et al. In silico site-directed mutagenesis informs species-specific predictions of chemical susceptibility derived from the Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS) tool. Toxicology Sciences. 166 (1), 131-145 (2018).
- Noyes, P. D., et al. Evaluating chemicals for thyroid disruption: Opportunities and challenges with in vitro testing and adverse outcome pathway approaches. Environmental Health Perspectives. 127 (9), 95001(2019).
- Park, G. Y., Jamerlan, A., Shim, K. H., An, S. S. A. Diagnostic and treatment approaches involving transthyretin in amyloidogenic diseases. Int J Mol Sci. 20 (12), 2982(2019).
- Rabah, S. A., Gowan, I. L., Pagnin, M., Osman, N., Richardson, S. J. Thyroid hormone distributor proteins during development in vertebrates. Front Endocrinol (Lausane). 10, 506(2019).
- Richardson, S. J. Cell and molecular biology of transthyretin and thyroid hormones. International Review of Cytology. 258, 137-193 (2007).
- Yamauchi, K., Ishihara, A. Transthyretin and Endocrine Disruptors. Recent Advances in Transthyretin Evolution, Structure and Biological Functions. Richardson, S. J., Cody, V. , Springer. Berlin Heidelberg, Germany. 159-171 (2009).
- Iakovleva, I., et al. Tetrabromobisphenol A is an efficient stabilizer of the transthyretin tetramer. PLoS One. 11 (4), 0153529(2016).
- Ishihara, A., Sawatsubashi, S., Yamauchi, K. Endocrine disrupting chemicals: Interference of thyroid hormone binding to transthyretins and to thyroid hormone receptors. Molecular and Cellular Endocrinology. 199 (1), 105-117 (2003).
- Kar, S., Sepúlveda, M. S., Roy, K., Leszczynski, J. Endocrine-disrupting activity of per- and polyfluoroalkyl substances: Exploring combined approaches of ligand and structure based modeling. Chemosphere. 184, 514-523 (2017).
- Morais-de-Sa, E., Pereira, P. J., Saraiva, M. J., Damas, A. M. The crystal structure of transthyretin in complex with diethylstilbestrol: A promising template for the design of amyloid inhibitors. Journal of Biological Chemistry. 279 (51), 53483-53490 (2004).
- Morgado, I., Campinho, M. A., Costa, R., Jacinto, R., Power, D. M. Disruption of the thyroid system by diethylstilbestrol and ioxynil in the sea bream (Sparus aurata). Aquatic Toxicology. 92 (4), 271-280 (2009).
- Yamauchi, K., Prapunpoj, P., Richardson, S. J. Effect of diethylstilbestrol on thyroid hormone binding to amphibian transthyretins. General and Comparative Endocrinology. 119 (3), 329-339 (2000).
- Zhang, J., et al. Structure-based virtual screening protocol for in silico identification of potential thyroid disrupting chemicals targeting transthyretin. Environmental Science & Technology. 50 (21), 11984-11993 (2016).
- Ren, X. M., et al. Binding interactions of perfluoroalkyl substances with thyroid hormone transport proteins and potential toxicological implications. Toxicology. 366-367, 32-42 (2016).
- Wilson, N., Mbabazi, K., Seth, P., Smith, H., Davis, N. L. Drug and opioid-involved overdose deaths - United States, 2017-2018. Morbidity and Mortality Weekly Report. 69 (11), 290-297 (2020).
- EPA. National Pollutant Discharge Elimination System (NPDES). United States Environmental Protection Agency. , Available from: https://www.epa.gov/npdes/npdes-resources (2018).
- Duvallet, C., Hayes, B. D., Erickson, T. B., Chai, P. R., Matus, M. Mapping community opioid exposure through wastewater-based epidemiology as a means to engage pharmacies in harm reduction efforts. Preventing Chronic Disease. 17, 200053(2020).
- Gushgari, A. J., Venkatesan, A. K., Chen, J., Steele, J. C., Halden, R. U. Long-term tracking of opioid consumption in two United States cities using wastewater-based epidemiology approach. Water Research. 161, 171-180 (2019).
- Lau, B., Bretaud, S., Huang, Y., Lin, E., Guo, S. Dissociation of food and opiate preference by a genetic mutation in zebrafish. Genes Brain Behave. 5 (7), 497-505 (2006).
- Bossé, G. D., Peterson, R. T. Development of an opioid self-administration assay to study drug seeking in zebrafish. Behavioural Brain Research. 335, 158-166 (2017).
- Mottaz, H., et al. Dose-dependent effects of morphine on lipopolysaccharide (LPS)-induced inflammation, and involvement of multixenobiotic resistance (MXR) transporters in LPS efflux in teleost fish. Environmental Pollution. 221, 105-115 (2017).
- Manglik, A., et al. Crystal structure of the µ-opioid receptor bound to a morphinan antagonist. Nature. 485 (7398), 321-326 (2012).
- Comer, S. D., Cahill, C. M. Fentanyl: Receptor pharmacology, abuse potential, and implications for treatment. Neuroscience & Biobehavioral Reviews. 106, 49-57 (2019).
- Podlewska, S., Bugno, R., Kudla, L., Bojarski, A. J., Przewlocki, R. Molecular modeling of µ opioid receptor ligands with various functional properties: PZM21, SR-17018, morphine, and fentanyl-simulated interaction patterns confronted with experimental data. Molecules. 25 (20), 4636(2020).
- Huang, W., et al. Structural insights into µ-opioid receptor activation. Nature. 524 (7565), 315-321 (2015).
- Lipiński, P. F. J., et al. Fentanyl family at the mu-opioid receptor: Uniform assessment of binding and computational analysis. Molecules. 24 (4), 740(2019).
- Boland, L. A., Angles, J. M. Feline permethrin toxicity: Retrospective study of 42 cases. Journal of Feline Medicine and Surgery. 12 (2), 61-71 (2010).
- Stevenson, B. J., Pignatelli, P., Nikou, D., Paine, M. J. Pinpointing P450s associated with pyrethroid metabolism in the dengue vector, Aedes aegypti: developing new tools to combat insecticide resistance. PLoS Neglected Tropical Diseases. 6 (3), 1595(2012).
- Ankley, G. T., Gray, L. E. Cross-species conservation of endocrine pathways: A critical analysis of tier 1 fish and rat screening assays with 12 model chemicals. Environmental Toxicology and Chemistry. 32 (5), 1084-1087 (2013).
- Meteyer, C. U., Rideout, B. A., Gilbert, M., Shivaprasad, H. L., Oaks, J. L. Pathology and proposed pathophysiology of diclofenac poisoning in free-living and experimentally exposed oriental white-backed vultures (Gyps bengalensis). Journal of Wildlife Diseases. 41 (4), 707-716 (2005).
- EPA. ECOTOX User Guide: ECOTOXicology Knowledgebase System. EPA, United States Environmental Protection Agency. , Available from: https://cfpub.epa.gov/ecotox/index.cfm (2021).
- ECOS Environmental Conservation Online System. U.S. Fish & Wildlife Service. , Available from: https://ecos.fws.gov/ecp/ (2021).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved