Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Демонстрация выравнивания последовательностей для прогнозирования восприимчивости различных видов для быстрой оценки сохранения белка
В этой статье
Резюме
Здесь мы представляем протокол для использования последней версии инструмента Агентства по охране окружающей среды США По согласованию последовательностей для прогнозирования восприимчивости к видам (SeqAPASS). Этот протокол демонстрирует применение онлайн-инструмента для быстрого анализа сохранения белка и предоставления настраиваемых и легко интерпретируемых прогнозов химической восприимчивости у разных видов.
Аннотация
Инструмент Агентства по охране окружающей среды США по согласованию последовательностей для прогнозирования восприимчивости видов (SeqAPASS) - это быстрое, свободно доступное онлайн-приложение для скрининга, которое позволяет исследователям и регулирующим органам экстраполировать информацию о токсичности между видами. Для биологических мишеней в модельных системах, таких как клетки человека, мыши, крысы и рыбки данио, данные о токсичности доступны для различных химических веществ. Благодаря оценке сохранения белковых мишеней этот инструмент может быть использован для экстраполяции данных, полученных из таких модельных систем, на тысячи других видов, не имеющих данных о токсичности, что дает прогнозы относительной внутренней химической восприимчивости. Последние выпуски инструмента (версии 2.0-6.1) включали новые функции, которые позволяют быстро синтезировать, интерпретировать и использовать данные для публикации, а также графику качества презентации.
Среди этих функций - настраиваемые визуализации данных и всеобъемлющий сводный отчет, предназначенный для обобщения данных SeqAPASS для удобства интерпретации. В этом документе описывается протокол, который поможет пользователям при отправке заданий, навигации по различным уровням сравнения последовательностей белков, а также интерпретации и отображении полученных данных. Выделены новые возможности SeqAPASS v2.0-6.0. Кроме того, описаны два варианта использования, ориентированные на сохранение транстиретина и белка опиоидных рецепторов с использованием этого инструмента. Наконец, обсуждаются сильные и слабые стороны SeqAPASS, чтобы определить область применимости инструмента и выделить различные приложения для межвидовой экстраполяции.
Введение
Традиционно область токсикологии в значительной степени полагалась на использование испытаний на животных для предоставления данных, необходимых для оценки химической безопасности. Такие методы, как правило, являются дорогостоящими и ресурсоемкими. Однако в связи с большим количеством используемых в настоящее время химических веществ и быстрыми темпами разработки новых химических веществ во всем мире существует признанная потребность в более эффективных методах химического скрининга 1,2. Эта потребность и вытекающий из этого сдвиг парадигмы от испытаний на животных привели к разработке многих новых методов подхода, включая высокопроизводительные скрининговые анализы, высокопроизводительную транскриптомику, секвенирование следующего поколения и вычислительное моделирование, которые являются многообещающими альтернативными стратегиями тестирования 3,4.
Оценка химической безопасности во всем разнообразии видов, потенциально подверженных воздействию химических веществ, является постоянной проблемой не только с традиционными испытаниями на токсичность, но и с новыми методами подхода. Достижения в сравнительной и прогностической токсикологии обеспечили основу для понимания относительной чувствительности различных видов, а технологические достижения в вычислительных методах продолжают повышать применимость этих методов. За последнее десятилетие обсуждалось несколько стратегий, которые используют существующие базы данных генов и белковых последовательностей, а также знания конкретных химических молекулярных мишеней для поддержки прогностических подходов к межвидовой экстраполяции и улучшения оценок химической безопасности за пределами типичных модельных организмов 5,6,7,8.
Чтобы продвинуть науку в действие, опираясь на эти фундаментальные исследования в области прогностической токсикологии, приоритизировать усилия по химическому тестированию и поддерживать принятие решений, был создан инструмент Агентства по охране окружающей среды США по согласованию последовательностей для прогнозирования восприимчивости к видам (SeqAPASS). Этот инструмент представляет собой общедоступное и свободно доступное веб-приложение, которое использует общедоступные репозитории постоянно расширяющейся информации о последовательностях белков для прогнозирования химической восприимчивости в разнообразии видов9. Основываясь на принципе, что относительная внутренняя восприимчивость вида к конкретному химическому веществу может быть определена путем оценки сохранения известных белковых мишеней этого химического вещества, этот инструмент быстро сравнивает последовательности белковых аминокислот от вида с известной чувствительностью ко всем видам с существующими данными о последовательностях белков. Эта оценка завершается с помощью трех уровней анализа, включая (1) последовательность первичных аминокислот, (2) функциональную область и (3) сравнение критических аминокислотных остатков, каждое из которых требует более глубоких знаний о химическо-белковом взаимодействии и обеспечивает большее таксономическое разрешение в прогнозировании восприимчивости. Основной сильной стороной SeqAPASS является то, что пользователи могут настраивать и уточнять свою оценку, добавляя дополнительные строки доказательств для сохранения цели на основе того, сколько информации доступно относительно интересующего взаимодействия химического белка или белка-белка.
Первая версия была выпущена в 2016 году, которая позволила пользователям оценивать последовательности первичных аминокислот и функциональные домены оптимизированным образом для прогнозирования химической восприимчивости и содержала минимальные возможности визуализации данных (таблица 1). Было показано, что индивидуальные аминокислотные различия являются важными детерминантами межвидовых различий в химических и белковых взаимодействиях, которые могут влиять на химическую восприимчивость видов 10,11,12. Поэтому последующие версии были разработаны для рассмотрения критических аминокислот, которые важны для прямого химического взаимодействия13. Реагируя на отзывы заинтересованных сторон и пользователей, этот инструмент претерпел ежегодные выпуски версий с дополнительными новыми функциями, предназначенными для удовлетворения потребностей как исследователей, так и регулирующих сообществ для решения проблем межвидовой экстраполяции (таблица 1). Запуск SeqAPASS версии 5.0 в 2020 году принес ориентированные на пользователя функции, которые включают в себя параметры визуализации и синтеза данных, внешние ссылки, сводные таблицы и параметры отчетов, а также графические функции. В целом, новые атрибуты и возможности этой версии улучшили синтез данных, функциональную совместимость между внешними базами данных и простоту интерпретации данных для прогнозирования межвидовой восприимчивости.
Access restricted. Please log in or start a trial to view this content.
протокол
1. Начало работы
ПРИМЕЧАНИЕ: Протокол, представленный здесь, ориентирован на полезность инструмента и ключевые функции. Подробное описание методов, функций и компонентов можно найти на веб-сайте во всеобъемлющем руководстве пользователя (таблица 1).
Таблица 1: Эволюция инструмента SeqAPASS. Список функций и обновлений, добавленных в инструмент SeqAPASS с момента его первоначального развертывания. Сокращения: SeqAPASS = выравнивание последовательностей для прогнозирования восприимчивости различных видов; ECOTOX = база знаний ECOTOXicology. Пожалуйста, нажмите здесь, чтобы загрузить эту таблицу.
- Перейдите в раздел https://seqapass.epa.gov/seqapass с помощью Chrome. Выберите Войти, чтобы использовать существующую учетную запись, или следуйте инструкциям по созданию учетной записи SeqAPASS, которая позволит пользователям запускать, хранить, получать доступ и настраивать свои завершенные задания.
- Перед проведением анализа сначала определите интересующий белок и целевой или чувствительный вид, просмотрев существующую литературу или ранее существовавшие данные (рисунок 1). Поскольку SeqAPASS содержит ссылки на внешние ресурсы, помогающие идентифицировать белок запроса, нажмите кнопки раскрывающегося списка в разделе Определение целевого объекта белка для доступа к соответствующим ресурсам.
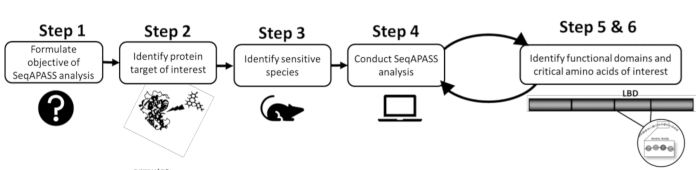
Рисунок 1: Постановка задачи SeqAPASS: принципиальная схема предварительной информации, необходимой для успешного анализа. Сокращения: SeqAPASS = выравнивание последовательностей для прогнозирования восприимчивости различных видов; LBD = лигандсвязывающий домен. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.

Рисунок 2: Взаимодействие SeqAPASS между базами данных. Принципиальная схема внешних инструментов, баз данных и ресурсов, интегрированных в SeqAPASS. Сокращения: SeqAPASS = выравнивание последовательностей для прогнозирования восприимчивости различных видов; AOP = путь неблагоприятных исходов; NCBI = Национальный центр биотехнологической информации; ECOTOX = база знаний ECOTOXicology. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Таблица 2: Ссылки, ресурсы и инструменты, интегрированные в инструмент SeqAPASS. Список различных источников данных, ссылок и ресурсов, используемых в инструменте SeqAPASS. Аббревиатура: SeqAPASS = выравнивание последовательностей для прогнозирования восприимчивости различных видов. Пожалуйста, нажмите здесь, чтобы загрузить эту таблицу.
2. Разработка и выполнение запроса SeqAPASS: Уровень 1
ПРИМЕЧАНИЕ: В анализе уровня 1 вся первичная аминокислотная последовательность белка запроса сравнивается с последовательностями первичных аминокислот всех видов с доступной информацией о последовательностях. Этот инструмент использует алгоритмы для добычи, сбора и компиляции общедоступных данных для быстрого выравнивания и сравнения аминокислотных последовательностей между видами. Серверная часть хранит информацию из баз данных Национального центра биотехнологической информации (NCBI) и стратегически использует автономные версии инструмента поиска базового локального выравнивания белка (BLASTp)54 и инструмента множественного выравнивания на основе ограничений (COBALT)55.
- В разделе Сравнение последовательностей первичных аминокислот щелкните По видам или По присоединению. Используйте выбор по видам для ввода или выбора из списка видов, чтобы выбрать интересующую белковую мишень.
- Представьте присоединение к белку (т.е. идентификатор белка NCBI) непосредственно путем ввода присоединения (присоединения) в текстовом поле «При присоединении ».
- Выберите Запуск запроса , чтобы отправить запрос. После отправки дождитесь появления уведомления в правом верхнем углу окна браузера, указывающего на успешную отправку.
- Выберите вкладку Состояние выполнения SeqAPASS в верхней части страницы, чтобы отобразить список всех запусков SeqAPASS, выполненных под этой учетной записью пользователя, и проверить процент завершения.
- Нажмите кнопку Обновить данные , когда будет выбран соответствующий переключатель, чтобы проверить состояние запусков уровня 2 и уровня 3.
- Выберите вкладку Просмотр отчетов SeqAPASS в верхней части страницы, чтобы получить доступ к списку всех отчетов, завершенных под этой учетной записью.
- На вкладке Просмотр отчетов SeqAPASS выберите интересующий белок запроса. Щелкните Запросить выбранный отчет, чтобы открыть страницу Сведения о белке запроса уровня 1 и просмотреть результаты, параметры настройки данных, визуализации и сводные отчеты.
- По умолчанию выберите Просмотреть отчет , чтобы просмотреть данные в веб-браузере. Кроме того, можно выбрать Сохранить отчет , чтобы загрузить необработанные данные в виде файла .zip.
ПРИМЕЧАНИЕ: Время, необходимое для анализа уровня 1, будет варьироваться (в среднем 23 минуты для версии 5.1) в зависимости от глобального спроса пользователей на данный момент времени, количества заданий, отправленных в очередь, и количества информации о белке, которая существует для отправленного задания. Если белковая мишень была ранее завершена, данные будут доступны через несколько секунд после отправки.
3. Разработка и выполнение запроса SeqAPASS: уровень 2
ПРИМЕЧАНИЕ: Поскольку вся последовательность белка непосредственно не участвует в химическом взаимодействии, анализ уровня 2 сравнивает только аминокислотную последовательность функционального домена, чтобы сделать прогнозы восприимчивости при более низких таксономических рангах (например, класс, порядок, семейство).
- На странице Сведения о белке запроса уровня 1 щелкните знак плюс + рядом с заголовком уровня 2 , чтобы заполнить меню Запрос уровня 2.
- Определите соответствующий домен (домены) в интересующем белке (протеин запроса).
- Если домен не был идентифицирован, щелкните интегрированную ссылку на базу данных NCBI Conserved Domains Database (CDD) (таблица 1), которая может помочь в определении соответствующего выбора домена.
ПРИМЕЧАНИЕ: Как правило, в качестве запросов на уровне 2 выбираются только определенные домены попадания.
- Если домен не был идентифицирован, щелкните интегрированную ссылку на базу данных NCBI Conserved Domains Database (CDD) (таблица 1), которая может помочь в определении соответствующего выбора домена.
- Щелкните поле Выбрать домен , чтобы автоматически заполнить список функциональных доменов для белка запроса.
- Выберите присоединение к домену из раскрывающегося списка и инициируйте запрос уровня 2, нажав кнопку Запросить запуск домена . После отправки дождитесь появления уведомления об успешной отправке.
- Щелкните Обновить уровень 2, и запустится 3 , чтобы заполнить данные уровня 2, которые будут доступны в течение нескольких секунд после отправки.
- В разделе Просмотр данных уровня 2 выберите завершенное присоединение к домену из раскрывающегося списка и нажмите кнопку Просмотреть данные уровня 2 , чтобы открыть результаты на новой странице.
4. Доступ и понимание данных: SeqAPASS Уровень 1 и Уровень 2
- Прокрутите страницу Запрос информации о белке вниз, чтобы просмотреть отчет о результатах - первичный отчет по умолчанию предоставляется с анализами уровня 1 и 2. Установите переключатель Полный отчет , чтобы просмотреть более подробный отчет, содержащий все метрики совпадений последовательностей и выравнивания. Нажмите на соответствующее присоединение / идентификатор / имя в обоих отчетах, чтобы получить доступ к прозрачной информации о выравнивании белков и таксономии в базе данных NCBI.
- Прокрутите таблицу результатов вправо, чтобы просмотреть столбец ECOTOX . Нажмите на ссылки на базу знаний ECOTOXicology (ECOTOX), чтобы быстро собрать соответствующие данные о токсичности для видов с прогнозами восприимчивости.
ПРИМЕЧАНИЕ: ECOTOX представляет собой всеобъемлющую, общедоступную базу знаний, содержащую единые данные о химической токсичности для водных и наземных растений и дикой природы. SeqAPASS v6.0 включает в себя виджет ECOTOX для более быстрого подключения к соответствующим данным ECOTOX по химическим веществам и видам, представляющим интерес. - Нажмите кнопку Загрузить таблицу , чтобы сохранить таблицу в виде файла электронной таблицы. Нажмите кнопку Просмотреть сводный отчет , чтобы просмотреть и загрузить сводную таблицу отчета, содержащую данные, отсортированные по таксономическим группам.
ПРИМЕЧАНИЕ: Сводные таблицы данных доступны как для первичных, так и для полных отчетов и содержат обзор прогнозов для данного целевого объекта.
5. Управление настройками данных: SeqAPASS Level 1 и Level 2
ПРИМЕЧАНИЕ: В анализах уровня 1 и уровня 2 предполагается, что чем больше сходство белков, тем больше вероятность того, что химическое вещество будет взаимодействовать с белком аналогично запрашиваемому виду / белку, что делает их восприимчивыми к потенциальному воздействию химических веществ с этой молекулярной мишенью. Из-за сходства этих данных шаги для понимания данных уровня 1 и 2 изложены вместе в одном протоколе.
- Обратитесь к подменю в верхней части Query Protein Information для доступа к параметрам отчета и управления ими, а также для использования параметров по умолчанию для всех параметров отчета для большинства анализов. Если изменение параметра по умолчанию имеется научное обоснование, выполните следующие необязательные действия.
- (НЕОБЯЗАТЕЛЬНО) Щелкните знак плюс + рядом с пунктом Отсечение восприимчивости , чтобы просмотреть и настроить параметры отключения восприимчивости в новой вкладке. Выберите новое значение отсечки из раскрывающегося списка или введите определяемое пользователем значение отсечки.
- (НЕОБЯЗАТЕЛЬНО) Измените число в поле E-Value (количество различных выравниваний, которые, как ожидается, произойдут случайно), если требуется что-то отличное от значения по умолчанию.
ПРИМЕЧАНИЕ: Любой белок со значением Е, превышающим число в коробке, будет исключен из первичного отчета. - (НЕОБЯЗАТЕЛЬНО) Используйте параметр Сортировать по таксономической группе , чтобы выбрать уровень таксономической иерархии для отображения в столбце Отфильтрованная таксономическая группа в таблице результатов.
ПРИМЕЧАНИЕ: Изменение таксономической иерархии также изменит прогноз восприимчивости на основе видов из каждой отфильтрованной группы, которые находятся выше отсечения. - (НЕОБЯЗАТЕЛЬНО) Измените поле Общий домен (сколько общих доменов белок должен разделять с белком запроса для включения в результаты), если требуется что-то иное, чем по умолчанию.
ПРИМЕЧАНИЕ: Поскольку по умолчанию установлено значение 1, любая последовательность, которая не разделяет хотя бы один общий домен с белком запроса, будет исключена. - (НЕОБЯЗАТЕЛЬНО) Выберите Нет в разделе Виды, прочитанные поперек , чтобы вернуть прогнозы восприимчивости Y только в том случае, если процент сходства больше или равен отсечке или если попадание идентифицировано как кандидат в ортологи.
ПРИМЕЧАНИЕ: Этот параметр по умолчанию имеет значение Да, что означает, что прогноз восприимчивости Y будет сообщен для всех кандидатов в ортологи, всех видов, перечисленных выше отсечки восприимчивости, и всех видов ниже отсечки из одной и той же таксономической группы с одним или несколькими видами выше отсечки.
- Нажмите кнопку Загрузить параметры текущего отчета, чтобы загрузить файл, фиксирующий примененные текущие параметры.
ПРИМЕЧАНИЕ: Выбранный конкретный уровень оценки (1, 2 или 3) будет диктовать параметры, представленные в отчете.
6. Визуализация данных: SeqAPASS Уровень 1 и Уровень 2
- Щелкните знак плюс + рядом с пунктом Визуализация и нажмите кнопку Визуализировать данные , чтобы открыть отдельную вкладку, отображающую пользовательскую информацию и возможность выбора интерактивного графика результатов.
- Щелкните Boxplot , чтобы открыть интерактивные элементы управления boxplot и plot и разрешить активное обновление визуализации boxplot для отражения изменений в таблице данных и предоставления графики качества публикации и представления.
ПРИМЕЧАНИЕ: Boxplot по умолчанию отображает группы видов по оси X и процентное сходство по оси Y. Boxplots отображают отсечение восприимчивости (пунктирная линия), процентное сходство между видами по сравнению с запрашиваемыми видами, а также средние и медианные значения для каждой таксономической группы, а также 25-й и 75-й процентиля и межквартильный диапазон. В зависимости от цели анализа и потребностей пользователя многие функции boxplot могут быть изменены с помощью следующих дополнительных шагов.- (НЕОБЯЗАТЕЛЬНО) Чтобы настроить отображаемые таксономические группы, обратитесь к полю Таксономические группы в разделе Элементы управления . Удалите группы, прокрутив имена и выбрав x или с помощью раскрывающегося меню Таксономические группы .
- (НЕОБЯЗАТЕЛЬНО) Чтобы добавить легенду, которая точно определит интересующий вид или конкретные предопределенные группы (например, исчезающие или находящиеся под угрозой исчезновения виды), наведите курсор на название таксономической группы на оси X, чтобы активировать всплывающее окно, в котором перечислены три лучших вида, упорядоченных по наибольшему процентному подобию. Наведите указатель мыши на виды в легенде, чтобы создать всплывающее окно с соответствующей информацией о видах. Нажмите на поле для конкретной таксономической группы, чтобы создать загружаемую сводную таблицу со списком видов и прогнозов.
- Нажмите кнопку Загрузить Boxplot , чтобы выбрать тип файла, настроить разрешение ширины/высоты и сохранить визуализацию.
7. Разработка и проведение анализа SeqAPASS: Уровень 3
ПРИМЕЧАНИЕ: Анализ уровня 3 оценивает идентифицированные пользователем аминокислотные остатки в исследуемом белке и быстро сравнивает сохранение этих остатков по видам. Предполагается, что виды, у которых эти остатки сохраняются, с большей вероятностью взаимодействуют с химическим веществом аналогично типовому/белку шаблона. Поскольку уровень 3 фокусируется на отдельных аминокислотах, анализ может быть выполнен только тогда, когда доступны подробные знания аминокислотных остатков, критически важных для взаимодействия химико-белка или белка-белка.
- Щелкните знак плюс + рядом с заголовком уровня 3 на странице Сведения о белке запроса уровня 1 , чтобы заполнить меню Запрос уровня 3.
- Щелкните знак плюс + рядом с обозревателем ссылок , чтобы открыть инструмент обозревателя ссылок, который генерирует предопределенную логическую строку для запроса доступной литературы и помогает пользователям определить соответствующую литературу для поддержки идентификации критических аминокислот, которые будут использоваться в оценке уровня 3 (таблица 2 и рисунок 2).
- (НЕОБЯЗАТЕЛЬНО) После автоматического заполнения белка запроса используйте функцию Add Protein Name (Добавить имя белка ) для добавления дополнительных белков.
- Нажмите создать ссылку Академии Google , чтобы открыть всплывающее окно, содержащее автоматически сгенерированную строку поиска, содержащую соответствующие условия поиска.
- Нажмите Поиск в Академии Google , чтобы запросить базу данных литературы с помощью строки поиска.
- Также можно нажать кнопку Копировать в буфер обмена и настроить строку поиска, добавив или удалив термины с помощью функций обозревателя ссылок.
8. Идентификация критических аминокислотных остатков с использованием идентифицированной литературы
- Выберите шаблонную последовательность , по которой выбранные пользователем виды будут выровнены, в меню Запрос уровня 3.
ПРИМЕЧАНИЕ: Эта шаблонная последовательность обычно выбирается на основе литературы, для которой были идентифицированы критические аминокислоты, и может быть тем же или другим видом, чем те, которые запрашиваются на уровне 1 и уровне 2.- (НЕОБЯЗАТЕЛЬНО) Поле Дополнительные сравнения используется для сравнения всех присоединений/последовательностей, которые не отображаются в таблицах Основной/Полный отчет .
- Введите определяемое пользователем имя запуска уровня 3 в текстовом поле Введите имя выполнения уровня 3 , чтобы определить завершенный запуск уровня 3. Выберите уникальное имя для каждой оценки.
- Выберите интересующую таксономическую группу в поле Выбрать таксономическую группу (группы ). Выберите таксономическую группу для автоматической фильтрации таблицы по этой таксономической группе.
- В таблице результатов вручную установите флажок рядом с любыми видами , которые необходимо выровнять по последовательности шаблона.
ПРИМЕЧАНИЕ: Для обеспечения надлежащего выравнивания следует сравнивать одну таксономическую группу за раз с шаблоном. Выбирайте только аналогично аннотированные белки для интересующих видов. При выборе последовательностей для сравнения важно обращать внимание на определенные последовательности (например, гипотетические, НИЗКОКАЧЕСТВЕННЫЕ или частичные). Если нет прозрачного обоснования для включения, лучше всего исключить эти последовательности, поскольку они могут искажать прогнозы из-за неполной или ненадлежащей информации о последовательностях. - Повторите шаги, чтобы выровнять все интересующие таксономические группы.
- Щелкните Обновить уровни 2 и 3 после того, как все виды будут выровнены, чтобы заполнить меню «Выбрать имя запуска уровня 3 » завершенными заданиями уровня 3 и немедленно получить данные из выравнивания уровня 3.
- Щелкните Объединить данные уровня 3 , чтобы объединить выравнивания из нескольких таксономических групп.
- Кроме того, чтобы просмотреть один отчет, выберите определяемое пользователем имя в разделе Выбрать запрос для просмотра и щелкните Просмотр данных уровня 3.
- Выберите шаблон уровня 3, который будет использоваться в качестве основы для сравнения аминокислотных остатков, в меню «Объединить отчеты уровня 3» и нажмите кнопку «Далее».
- В разделе Задания уровня 3 выберите завершенные задания для сравнения и нажмите кнопку Далее. При необходимости используйте функцию Задания уровня 3 для изменения порядка таксономических групп. Щелкните Просмотр данных уровня 3 , чтобы создать страницу отчета уровня 3 с объединенными таксономическими группами.
- Выберите ранее определенные позиции аминокислот для типов шаблонов , введя позиции аминокислот, разделенные запятыми, в поле Введите позиции аминокислотных остатков , а затем выбрав Копировать в список остатков . Непосредственно выберите остатки в последовательности шаблона из коробки шаттла.
- Щелкните Обновить отчет , чтобы обновить страницу и отобразить прогнозы восприимчивости уровня 3.
ПРИМЕЧАНИЕ: Уровень 3 использует простой набор правил, полученных из основных дескрипторов функциональных свойств боковой цепи (например, алифатических, ароматических) и молекулярных измерений (различия молекулярной массы >30 г/моль), чтобы определить, могут ли различия в ключевых позициях влиять на белковые взаимодействия13.
9. Визуализация данных SeqAPASS уровня 3
ПРИМЕЧАНИЕ: Как и на предыдущих уровнях, доступны первичные и полные отчеты. В дополнение к данным, идентичным данным уровня 1 и 2, первичный отчет отображает аминокислотные позиции, аббревиатуры и «да/нет» (Y/N), аналогичную восприимчивости к шаблону прогноза. Аналогичным образом, полный отчет содержит информацию о классификации боковых цепей аминокислот и молекулярной массе.
- На странице отчета уровня 3 прокрутите вниз, чтобы просмотреть отчет о результатах. Нажмите кнопку Загрузить таблицу в нижней части отчета, чтобы сохранить таблицу.
- Щелкните Просмотр сводного отчета уровня 3 , чтобы просмотреть и загрузить сводную таблицу отчета, содержащую данные, отсортированные по таксономическим группам. Щелкните знак плюс + рядом с пунктом Визуализация на странице Отчета уровня 3, чтобы открыть отдельную вкладку браузера, отображающую пользовательскую информацию и возможность просмотра результатов в виде интерактивной тепловой карты.
- Щелкните Тепловая карта на странице Сведения о визуализации , чтобы открыть интерактивный рисунок и элементы управления и разрешить активное обновление визуализации тепловой карты для отражения изменений в таблице данных. Выполните следующие дополнительные действия для настройки тепловой карты.
- (НЕОБЯЗАТЕЛЬНО) Выберите Параметры отчета, чтобы перейти между простым отчетом, в котором отображается положение аминокислот, однобуквенная аббревиатура и сходство аминокислот, или полным отчетом, в котором отображаются подробные сведения о каждой выбранной аминокислоте.
- (НЕОБЯЗАТЕЛЬНО) Выберите Параметры отчета, чтобы изменить способ отображения видов по общему имени или научному названию.
ПРИМЕЧАНИЕ: В простом отчете аминокислоты классифицируются как Полное соответствие (темно-синий), Частичное совпадение (светло-синий, замены, отвечающие только одному критерию) или Не соответствует (желтый, замены, не отвечающие ни одному из критериев) аминокислоте шаблона. В полном отчете отображаются сравнения как «Итоговое совпадение» (темно-синий) или «Не совпадение» (желтый). - (НЕОБЯЗАТЕЛЬНО) Выберите «Дополнительные выборки», чтобы выделить полезную информацию, такую как кандидаты в ортологи, виды, находящиеся под угрозой исчезновения, виды, находящиеся под угрозой исчезновения, или общие модельные организмы.
- (НЕОБЯЗАТЕЛЬНО) Выберите Параметры тепловой карты , чтобы выбрать дополнительные параметры настройки, включая добавление или удаление столбцов, легенд и текста.
- Нажмите кнопку Загрузить Boxplot , чтобы выбрать тип файла и сохранить визуализацию.
10. Интерпретация результатов SeqAPASS: строки доказательств сохранения белка
ПРИМЕЧАНИЕ: Для удобства интерпретации этот инструмент включает в себя отчет о принятии резюме решений (DS Report), предназначенный для интеграции данных между уровнями. Отчет DS содержит результаты (т.е. таблицы данных и/или визуализации), выбранные пользователем, и позволяет быстро оценить прогнозы восприимчивости на нескольких уровнях для нескольких видов одновременно.
- Щелкните Push Level # to DS Report на страницах результатов или визуализации данных и подождите, пока данные будут «отправлены» и вкладка DS Report станет активной.
ПРИМЕЧАНИЕ: Если результаты или какие-либо изменения не были отправлены в отчет DS, Push Level # to DS Report будет оставаться активным до тех пор, пока не будет выбран. Если параметр был изменен, текст Щелкните, чтобы отправить новые изменения , будет отображаться до тех пор, пока изменения не будут отправлены в отчет. Визуализации могут быть отправлены в отчет DS в любое время во время оценки. - Выберите вкладку Отчет DS в любое время, чтобы получить доступ к странице DS.
ПРИМЕЧАНИЕ: Для всех видов, выровненных на уровне 1, таблица сводного отчета об окончательном решении содержит важные данные и прогнозы восприимчивости для каждого анализа. Если вид в таблице DS не был включен в отчет уровня 3, но был обнаружен в заданиях уровня 1 и/или уровня 2, ячейка в таблице получит неприменимое обозначение (NA) для прогноза восприимчивости уровня 3.
Access restricted. Please log in or start a trial to view this content.
Результаты
Чтобы продемонстрировать применение инструмента SeqAPASS и подчеркнуть новые особенности, описаны два тематических исследования, представляющих случаи, в которых сохранение белка предсказывает, что существуют различия в химической восприимчивости между видами (транстиретин человека) и...
Access restricted. Please log in or start a trial to view this content.
Обсуждение
Широко признано, что невозможно эмпирически проверить достаточное количество видов, чтобы охватить геномное, фенотипическое, физиологическое и поведенческое разнообразие живых организмов, которые могут подвергаться воздействию химических веществ, представляющих токсикологический...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
У авторов нет конфликта интересов для раскрытия.
Благодарности
Авторы благодарят д-ра Даниэля Л. Вильнёва (Агентство по охране окружающей среды США, Центр вычислительной токсикологии и воздействия) и доктора Джона А. Дёринга (Департамент наук об окружающей среде, Университет штата Луизиана) за комментарии к более раннему проекту рукописи. Эта работа была поддержана Агентством по охране окружающей среды США. Мнения, выраженные в настоящем документе, являются мнениями авторов и не обязательно отражают взгляды или политику Агентства по охране окружающей среды США, а упоминание торговых наименований или коммерческих продуктов не указывает на одобрение со стороны федерального правительства.
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| Spreadsheet program | N/A | N/A | Any program that can be used to view and work with csv files (e.g. Microsoft Excel, OpenOffice Calc, Google Docs) can be used to access data export files. |
| Basic computing setup and internet access | N/A | N/A | SeqAPASS is a free, online tool that can be easily used via an internet connection. No software downloads are required. |
Ссылки
- Krewski, D., et al. Toxicity testing in the 21st century: a vision and a strategy. Journal of Toxicology and Environmental Health, Part B. 13 (2-4), 51-138 (2010).
- Wang, Z., Walker, G. W., Muir, D. C. G., Nagatani-Yoshida, K. Toward a global understanding of chemical pollution: A first comprehensive analysis of national and regional chemical inventories. Environmental Science & Technology. 54 (5), 2575-2584 (2020).
- Brooks, B. W., et al. Toxicology advances for 21st century chemical pollution. One Earth. 2 (4), 312-316 (2020).
- Kostal, J., Voutchkova-Kostal, A. Going all in: A strategic investment in in silico toxicology. Chemical Research in Toxicology. 33 (4), 880-888 (2020).
- Cheng, W., Doering, J. A., LaLone, C., Ng, C. Integrative computational approaches to inform relative bioaccumulation potential of per- and polyfluoroalkyl substances (PFAS) across species. Toxicology Sciences. 180 (2), 212-223 (2021).
- Kostich, M. S., Lazorchak, J. M. Risks to aquatic organisms posed by human pharmaceutical use. Science of the Total Environment. 389 (2-3), 329-339 (2008).
- Gunnarsson, L., Jauhiainen, A., Kristiansson, E., Nerman, O., Larsson, D. G. Evolutionary conservation of human drug targets in organisms used for environmental risk assessments. Environmental Science & Technology. 42 (15), 5807-5813 (2008).
- LaLone, C. A., et al. Evidence for cross species extrapolation of mammalian-based high-throughput screening assay results. Environmental Science & Technology. 52 (23), 13960-13971 (2018).
- LaLone, C. A., et al. Editor's highlight: Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS): A web-based tool for addressing the challenges of cross-species extrapolation of chemical toxicity. Toxicology Sciences. 153 (2), 228-245 (2016).
- Head, J. A., Hahn, M. E., Kennedy, S. W. Key amino acids in the aryl hydrocarbon receptor predict dioxin sensitivity in avian species. Environmental Science & Technology. 42 (19), 7535-7541 (2008).
- Bass, C., et al. Mutation of a nicotinic acetylcholine receptor β subunit is associated with resistance to neonicotinoid insecticides in the aphid Myzus persicae. BMC Neuroscience. 12, 51-51 (2011).
- Erdmanis, L., et al. Association of neonicotinoid insensitivity with a conserved residue in the loop d binding region of the tick nicotinic acetylcholine receptor. Biochemistry. 51 (23), 4627-4629 (2012).
- Doering, J. A., et al. et al. In silico site-directed mutagenesis informs species-specific predictions of chemical susceptibility derived from the Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS) tool. Toxicology Sciences. 166 (1), 131-145 (2018).
- Noyes, P. D., et al. Evaluating chemicals for thyroid disruption: Opportunities and challenges with in vitro testing and adverse outcome pathway approaches. Environmental Health Perspectives. 127 (9), 95001(2019).
- Park, G. Y., Jamerlan, A., Shim, K. H., An, S. S. A. Diagnostic and treatment approaches involving transthyretin in amyloidogenic diseases. Int J Mol Sci. 20 (12), 2982(2019).
- Rabah, S. A., Gowan, I. L., Pagnin, M., Osman, N., Richardson, S. J. Thyroid hormone distributor proteins during development in vertebrates. Front Endocrinol (Lausane). 10, 506(2019).
- Richardson, S. J. Cell and molecular biology of transthyretin and thyroid hormones. International Review of Cytology. 258, 137-193 (2007).
- Yamauchi, K., Ishihara, A. Transthyretin and Endocrine Disruptors. Recent Advances in Transthyretin Evolution, Structure and Biological Functions. Richardson, S. J., Cody, V. , Springer. Berlin Heidelberg, Germany. 159-171 (2009).
- Iakovleva, I., et al. Tetrabromobisphenol A is an efficient stabilizer of the transthyretin tetramer. PLoS One. 11 (4), 0153529(2016).
- Ishihara, A., Sawatsubashi, S., Yamauchi, K. Endocrine disrupting chemicals: Interference of thyroid hormone binding to transthyretins and to thyroid hormone receptors. Molecular and Cellular Endocrinology. 199 (1), 105-117 (2003).
- Kar, S., Sepúlveda, M. S., Roy, K., Leszczynski, J. Endocrine-disrupting activity of per- and polyfluoroalkyl substances: Exploring combined approaches of ligand and structure based modeling. Chemosphere. 184, 514-523 (2017).
- Morais-de-Sa, E., Pereira, P. J., Saraiva, M. J., Damas, A. M. The crystal structure of transthyretin in complex with diethylstilbestrol: A promising template for the design of amyloid inhibitors. Journal of Biological Chemistry. 279 (51), 53483-53490 (2004).
- Morgado, I., Campinho, M. A., Costa, R., Jacinto, R., Power, D. M. Disruption of the thyroid system by diethylstilbestrol and ioxynil in the sea bream (Sparus aurata). Aquatic Toxicology. 92 (4), 271-280 (2009).
- Yamauchi, K., Prapunpoj, P., Richardson, S. J. Effect of diethylstilbestrol on thyroid hormone binding to amphibian transthyretins. General and Comparative Endocrinology. 119 (3), 329-339 (2000).
- Zhang, J., et al. Structure-based virtual screening protocol for in silico identification of potential thyroid disrupting chemicals targeting transthyretin. Environmental Science & Technology. 50 (21), 11984-11993 (2016).
- Ren, X. M., et al. Binding interactions of perfluoroalkyl substances with thyroid hormone transport proteins and potential toxicological implications. Toxicology. 366-367, 32-42 (2016).
- Wilson, N., Mbabazi, K., Seth, P., Smith, H., Davis, N. L. Drug and opioid-involved overdose deaths - United States, 2017-2018. Morbidity and Mortality Weekly Report. 69 (11), 290-297 (2020).
- EPA. National Pollutant Discharge Elimination System (NPDES). United States Environmental Protection Agency. , Available from: https://www.epa.gov/npdes/npdes-resources (2018).
- Duvallet, C., Hayes, B. D., Erickson, T. B., Chai, P. R., Matus, M. Mapping community opioid exposure through wastewater-based epidemiology as a means to engage pharmacies in harm reduction efforts. Preventing Chronic Disease. 17, 200053(2020).
- Gushgari, A. J., Venkatesan, A. K., Chen, J., Steele, J. C., Halden, R. U. Long-term tracking of opioid consumption in two United States cities using wastewater-based epidemiology approach. Water Research. 161, 171-180 (2019).
- Lau, B., Bretaud, S., Huang, Y., Lin, E., Guo, S. Dissociation of food and opiate preference by a genetic mutation in zebrafish. Genes Brain Behave. 5 (7), 497-505 (2006).
- Bossé, G. D., Peterson, R. T. Development of an opioid self-administration assay to study drug seeking in zebrafish. Behavioural Brain Research. 335, 158-166 (2017).
- Mottaz, H., et al. Dose-dependent effects of morphine on lipopolysaccharide (LPS)-induced inflammation, and involvement of multixenobiotic resistance (MXR) transporters in LPS efflux in teleost fish. Environmental Pollution. 221, 105-115 (2017).
- Manglik, A., et al. Crystal structure of the µ-opioid receptor bound to a morphinan antagonist. Nature. 485 (7398), 321-326 (2012).
- Comer, S. D., Cahill, C. M. Fentanyl: Receptor pharmacology, abuse potential, and implications for treatment. Neuroscience & Biobehavioral Reviews. 106, 49-57 (2019).
- Podlewska, S., Bugno, R., Kudla, L., Bojarski, A. J., Przewlocki, R. Molecular modeling of µ opioid receptor ligands with various functional properties: PZM21, SR-17018, morphine, and fentanyl-simulated interaction patterns confronted with experimental data. Molecules. 25 (20), 4636(2020).
- Huang, W., et al. Structural insights into µ-opioid receptor activation. Nature. 524 (7565), 315-321 (2015).
- Lipiński, P. F. J., et al. Fentanyl family at the mu-opioid receptor: Uniform assessment of binding and computational analysis. Molecules. 24 (4), 740(2019).
- Boland, L. A., Angles, J. M. Feline permethrin toxicity: Retrospective study of 42 cases. Journal of Feline Medicine and Surgery. 12 (2), 61-71 (2010).
- Stevenson, B. J., Pignatelli, P., Nikou, D., Paine, M. J. Pinpointing P450s associated with pyrethroid metabolism in the dengue vector, Aedes aegypti: developing new tools to combat insecticide resistance. PLoS Neglected Tropical Diseases. 6 (3), 1595(2012).
- Ankley, G. T., Gray, L. E. Cross-species conservation of endocrine pathways: A critical analysis of tier 1 fish and rat screening assays with 12 model chemicals. Environmental Toxicology and Chemistry. 32 (5), 1084-1087 (2013).
- Meteyer, C. U., Rideout, B. A., Gilbert, M., Shivaprasad, H. L., Oaks, J. L. Pathology and proposed pathophysiology of diclofenac poisoning in free-living and experimentally exposed oriental white-backed vultures (Gyps bengalensis). Journal of Wildlife Diseases. 41 (4), 707-716 (2005).
- EPA. ECOTOX User Guide: ECOTOXicology Knowledgebase System. EPA, United States Environmental Protection Agency. , Available from: https://cfpub.epa.gov/ecotox/index.cfm (2021).
- ECOS Environmental Conservation Online System. U.S. Fish & Wildlife Service. , Available from: https://ecos.fws.gov/ecp/ (2021).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены