Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Demonstration der Sequenzausrichtung zur Vorhersage über Spezies hinweg Suszeptibilitätstool zur schnellen Bewertung der Proteinkonservierung
In diesem Artikel
Zusammenfassung
Hier stellen wir ein Protokoll vor, um die neueste Version des SeqAPASS-Tools (Sequence Alignment to Predict Across Species Susceptibility) der US Environmental Protection Agency zu verwenden. Dieses Protokoll demonstriert die Anwendung des Online-Tools zur schnellen Analyse der Proteinkonservierung und zur Bereitstellung anpassbarer und leicht interpretierbarer Vorhersagen der chemischen Empfindlichkeit über Spezies hinweg.
Zusammenfassung
Das SeqAPASS-Tool (Sequence Alignment to Predict Across Species Susceptibility) der US-Umweltschutzbehörde ist eine schnelle, frei verfügbare Online-Screening-Anwendung, mit der Forscher und Regulierungsbehörden Toxizitätsinformationen über Spezies hinweg extrapolieren können. Für biologische Ziele in Modellsystemen wie menschlichen Zellen, Mäusen, Ratten und Zebrafischen liegen Toxizitätsdaten für eine Vielzahl von Chemikalien vor. Durch die Bewertung der Proteinzielkonservierung kann dieses Werkzeug verwendet werden, um Daten, die aus solchen Modellsystemen generiert werden, auf Tausende anderer Arten ohne Toxizitätsdaten zu extrapolieren, was Vorhersagen der relativen intrinsischen chemischen Suszeptibilität liefert. Die neuesten Versionen des Tools (Versionen 2.0-6.1) enthalten neue Funktionen, die eine schnelle Synthese, Interpretation und Verwendung der Daten für die Veröffentlichung sowie Grafiken in Präsentationsqualität ermöglichen.
Zu diesen Funktionen gehören anpassbare Datenvisualisierungen und ein umfassender zusammenfassender Bericht, der SeqAPASS-Daten zur einfachen Interpretation zusammenfasst. In diesem Dokument wird das Protokoll beschrieben, mit dem Benutzer durch das Einreichen von Aufträgen, das Navigieren durch die verschiedenen Ebenen von Proteinsequenzvergleichen sowie die Interpretation und Anzeige der resultierenden Daten geführt werden. Neue Funktionen von SeqAPASS v2.0-6.0 werden hervorgehoben. Darüber hinaus werden zwei Anwendungsfälle beschrieben, die sich auf die Konservierung von Transthyretin- und Opioidrezeptorproteinen mit diesem Werkzeug konzentrieren. Schließlich werden die Stärken und Grenzen von SeqAPASS diskutiert, um den Anwendungsbereich für das Tool zu definieren und verschiedene Anwendungen für die artenübergreifende Extrapolation hervorzuheben.
Einleitung
Traditionell stützt sich die Toxikologie stark auf die Verwendung von Ganztierversuchen, um die für die Stoffsicherheitsbeurteilung erforderlichen Daten zu liefern. Solche Methoden sind typischerweise kostspielig und ressourcenintensiv. Aufgrund der großen Anzahl der derzeit verwendeten Chemikalien und des rasanten Tempos, mit dem neue Chemikalien entwickelt werden, besteht jedoch weltweit ein anerkannter Bedarf an effizienteren Methoden des chemischen Screenings 1,2. Dieser Bedarf und der daraus resultierende Paradigmenwechsel weg von Tierversuchen hat zur Entwicklung vieler neuer Ansatzmethoden geführt, darunter Hochdurchsatz-Screening-Assays, Hochdurchsatz-Transkriptomik, Next-Generation-Sequenzierung und Computermodellierung, die vielversprechende alternative Teststrategien sind 3,4.
Die Bewertung der Stoffsicherheit über die Vielfalt der Arten hinweg, die potenziell von chemischen Expositionen betroffen sind, war eine anhaltende Herausforderung, nicht nur mit traditionellen Toxizitätstests, sondern auch mit neuen Ansatzmethoden. Fortschritte in der vergleichenden und prädiktiven Toxikologie haben Rahmenbedingungen für das Verständnis der relativen Sensitivität verschiedener Arten geschaffen, und technologische Fortschritte bei Computermethoden erhöhen weiterhin die Anwendbarkeit dieser Methoden. In den letzten zehn Jahren wurden mehrere Strategien diskutiert, die bestehende Gen- und Proteinsequenzdatenbanken zusammen mit dem Wissen über spezifische chemisch-molekulare Ziele nutzen, um prädiktive Ansätze für die artenübergreifende Extrapolation zu unterstützen und die Bewertung der chemischen Sicherheit über die typischen Modellorganismen hinaus zu verbessern 5,6,7,8.
Um die Wissenschaft in die Tat umzusetzen, auf diesen grundlegenden Studien in der prädiktiven Toxikologie aufzubauen, chemische Testbemühungen zu priorisieren und die Entscheidungsfindung zu unterstützen, wurde das SeqAPASS-Tool (Sequence Alignment to Predict Across Species Susceptibility) der US-Umweltschutzbehörde (Sequence Alignment to Predict Across Species Susceptibility) entwickelt. Dieses Tool ist eine öffentliche und frei verfügbare webbasierte Anwendung, die öffentliche Repositorien mit ständig wachsenden Proteinsequenzinformationen verwendet, um die chemische Empfindlichkeit über die Vielfalt der Spezies vorherzusagen9. Basierend auf dem Prinzip, dass die relative intrinsische Suszeptibilität einer Spezies für eine bestimmte Chemikalie durch die Bewertung der Erhaltung der bekannten Proteinziele dieser Chemikalie bestimmt werden kann, vergleicht dieses Tool schnell Proteinaminosäuresequenzen einer Spezies mit bekannter Empfindlichkeit gegenüber allen Spezies mit vorhandenen Proteinsequenzdaten. Diese Bewertung wird durch drei Analyseebenen abgeschlossen, darunter (1) primäre Aminosäuresequenz, (2) funktionelle Domäne und (3) kritische Aminosäurerestvergleiche, die jeweils tiefere Kenntnisse der chemischen Protein-Interaktion erfordern und eine größere taxonomische Auflösung in der Suszeptibilitätsvorhersage bieten. Eine große Stärke von SeqAPASS besteht darin, dass Benutzer ihre Bewertung anpassen und verfeinern können, indem sie zusätzliche Evidenzlinien für die Zielkonservierung hinzufügen, basierend darauf, wie viele Informationen über die interessierende Chemie-Protein- oder Protein-Protein-Interaktion verfügbar sind.
Die erste Version wurde 2016 veröffentlicht, die es Benutzern ermöglichte, primäre Aminosäuresequenzen und funktionelle Domänen auf optimierte Weise zu bewerten, um die chemische Anfälligkeit vorherzusagen, und enthielt minimale Datenvisualisierungsfunktionen (Tabelle 1). Es hat sich gezeigt, dass individuelle Aminosäureunterschiede wichtige Determinanten von artenübergreifenden Unterschieden in chemischen Proteininteraktionen sind, die die chemische Suszeptibilität von Arten beeinflussen können10,11,12. Daher wurden nachfolgende Versionen entwickelt, um die kritischen Aminosäuren zu berücksichtigen, die für die direkte chemische Wechselwirkung wichtig sind13. Als Reaktion auf das Feedback von Interessengruppen und Benutzern wurde dieses Tool jährlich mit zusätzlichen neuen Funktionen veröffentlicht, die sowohl den Bedürfnissen von Forschern als auch Regulierungsbehörden bei der Bewältigung von Herausforderungen bei der artenübergreifenden Extrapolation gerecht werden (Tabelle 1). Die Einführung von SeqAPASS Version 5.0 im Jahr 2020 brachte benutzerzentrierte Funktionen hervor, die Datenvisualisierungs- und Datensyntheseoptionen, externe Links, Übersichtstabellen- und Berichtsoptionen sowie grafische Funktionen umfassen. Insgesamt verbesserten die neuen Attribute und Funktionen dieser Version die Datensynthese, die Interoperabilität zwischen externen Datenbanken und die einfache Dateninterpretation für Vorhersagen der artenübergreifenden Suszeptibilität.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Erste Schritte
HINWEIS: Das hier vorgestellte Protokoll konzentriert sich auf das Dienstprogramm und die wichtigsten Funktionen des Tools. Detaillierte Beschreibungen von Methoden, Funktionen und Komponenten finden Sie auf der Website in einem umfassenden Benutzerhandbuch (Tabelle 1).
Tabelle 1: Entwicklung des SeqAPASS-Tools. Eine Liste der Funktionen und Updates, die dem SeqASPASS-Tool seit der ersten Bereitstellung hinzugefügt wurden. Abkürzungen: SeqAPASS = Sequence Alignment to Predict Across Species Susceptibility; ECOTOX = ECOTOXicology Wissensdatenbank. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
- Wechseln Sie mit Chrome zu https://seqapass.epa.gov/seqapass. Wählen Sie entweder Anmelden, um ein vorhandenes Konto zu verwenden, oder folgen Sie den Anweisungen, um ein SeqAPASS-Konto zu erstellen, mit dem Benutzer ihre abgeschlossenen Jobs ausführen, speichern, darauf zugreifen und anpassen können.
- Bevor Sie eine Analyse durchführen, identifizieren Sie zunächst ein Protein von Interesse und eine gezielte oder empfindliche Spezies, indem Sie die vorhandene Literatur oder bereits vorhandene Daten überprüfen (Abbildung 1). Da SeqAPASS Links zu externen Ressourcen enthält, um das Abfrageprotein zu identifizieren, klicken Sie auf die Dropdown-Schaltflächen unter Proteinziel identifizieren , um auf relevante Ressourcen zuzugreifen.
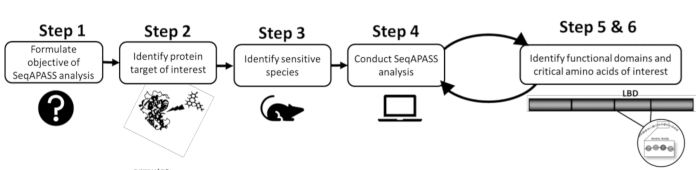
Abbildung 1: SeqAPASS-Problemformulierung: schematische Darstellung der für eine erfolgreiche Analyse notwendigen Vorabinformationen. Abkürzungen: SeqAPASS = Sequence Alignment to Predict Across Species Susceptibility; LBD = ligandenbindende Domäne. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 2: SeqAPASS-Interoperabilität zwischen Datenbanken. Schematische Darstellung externer Tools, Datenbanken und Ressourcen, die in SeqAPASS integriert sind. Abkürzungen: SeqAPASS = Sequence Alignment to Predict Across Species Susceptibility; AOP = adverse outcome pathway; NCBI = National Center for Biotechnology Information; ECOTOX = ECOTOXicology Wissensdatenbank. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Tabelle 2: Links, Ressourcen und Tools, die in das SeqAPASS-Tool integriert sind. Eine Liste der verschiedenen Datenquellen, Links und Ressourcen, die im SeqAPASS-Tool genutzt werden. Abkürzung: SeqAPASS = Sequence Alignment to Predict Across Species Susceptibility. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
2. Entwicklung und Ausführung einer SeqAPASS-Abfrage: Ebene 1
HINWEIS: In einer Level-1-Analyse wird die gesamte primäre Aminosäuresequenz eines Abfrageproteins mit den primären Aminosäuresequenzen aller Spezies mit verfügbaren Sequenzinformationen verglichen. Dieses Tool verwendet Algorithmen, um öffentlich verfügbare Daten zu sammeln, zu sammeln und zusammenzustellen, um Aminosäuresequenzen über Arten hinweg schnell auszurichten und zu vergleichen. Das Backend speichert Informationen aus den Datenbanken des National Center for Biotechnology Information (NCBI) und nutzt strategisch die eigenständigen Versionen des Protein Basic Local Alignment Search Tool (BLASTp)54 und des Constraint-based Multiple Alignment Tool (COBALT)55.
- Klicken Sie unter Primäre Aminosäuresequenzen vergleichen entweder auf Nach Art oder Nach Akzession. Verwenden Sie die Auswahl Nach Spezies , um das gewünschte Proteinziel auszuwählen, oder wählen Sie aus einer Liste von Arten aus.
- Reichen Sie Proteinakzessionen (d. h. NCBI-Protein-ID) direkt ein, indem Sie die Beitritt(e) in das Textfeld Nach Beitritt eingeben.
- Wählen Sie Anforderungsausführung aus, um die Abfrage zu senden. Warten Sie nach dem Absenden, bis in der oberen rechten Ecke des Browserfensters eine Benachrichtigung angezeigt wird, die eine erfolgreiche Übermittlung anzeigt.
- Wählen Sie die Registerkarte SeqAPASS Run Status oben auf der Seite, um eine Liste aller SeqAPASS-Läufe anzuzeigen, die unter diesem Benutzerkonto durchgeführt wurden, und überprüfen Sie den Prozentsatz des Abschlusses.
- Klicken Sie auf Daten aktualisieren , während das entsprechende Optionsfeld ausgewählt ist, um den Status von Läufen der Ebenen 2 und 3 zu überprüfen.
- Wählen Sie oben auf der Seite die Registerkarte SeqASPASS-Berichte anzeigen aus, um auf eine Liste aller unter diesem Konto erstellten Berichte zuzugreifen.
- Wählen Sie auf der Registerkarte SeqAPASS-Berichte anzeigen das gewünschte Abfrageprotein aus. Klicken Sie auf Ausgewählten Bericht anfordern , um die Seite Level 1 Query Protein Information zu öffnen und Ergebnisse, Datenanpassungsoptionen, Visualisierungen und Zusammenfassungsberichte anzuzeigen.
- Wählen Sie standardmäßig Bericht anzeigen aus, um Daten im Webbrowser anzuzeigen. Alternativ können Sie Bericht speichern auswählen, um Rohdaten als .zip Datei herunterzuladen.
HINWEIS: Die für eine Level-1-Analyse erforderliche Zeit variiert (durchschnittlich 23 Minuten für Version 5.1), abhängig von der globalen Benutzeranforderung zu diesem Zeitpunkt, der Anzahl der an die Warteschlange übermittelten Aufträge und der Menge an Proteininformationen, die für einen übermittelten Auftrag vorhanden sind. Wenn ein Proteinziel zuvor erreicht wurde, sind die Daten innerhalb von Sekunden nach der Einreichung verfügbar.
3. Entwickeln und Ausführen einer SeqAPASS-Abfrage: Ebene 2
HINWEIS: Da die gesamte Proteinsequenz nicht direkt an einer chemischen Wechselwirkung beteiligt ist, vergleicht eine Level-2-Analyse nur die Aminosäuresequenz der funktionellen Domäne, um Empfindlichkeitsvorhersagen auf niedrigeren taxonomischen Rängen (z. B. Klasse, Ordnung, Familie) zu treffen.
- Klicken Sie auf der Seite Level 1 Query Protein Information (Proteininformationen der Ebene 1) auf das Pluszeichen + neben der Kopfzeile der Ebene 2, um das Menü "Level 2-Abfrage" aufzufüllen.
- Identifizieren Sie die geeignete(n) Domäne(n) im interessierenden Protein (Abfrageprotein).
- Wenn eine Domäne nicht identifiziert wurde, klicken Sie auf den integrierten Link zur NCBI Conserved Domains Database (CDD) (Tabelle 1), die bei der Identifizierung der entsprechenden Domänenauswahl helfen kann.
HINWEIS: In der Regel werden nur bestimmte Trefferdomänen als Abfragen in Ebene 2 ausgewählt.
- Wenn eine Domäne nicht identifiziert wurde, klicken Sie auf den integrierten Link zur NCBI Conserved Domains Database (CDD) (Tabelle 1), die bei der Identifizierung der entsprechenden Domänenauswahl helfen kann.
- Klicken Sie auf das Feld Domäne auswählen , um automatisch eine Liste funktionaler Domänen für das Abfrageprotein aufzufüllen.
- Wählen Sie die Domain-Mitgliedschaft(en) aus der Dropdown-Liste aus und initiieren Sie die Level 2-Abfrage, indem Sie auf die Schaltfläche Domain Run anfordern klicken. Warten Sie nach dem Senden, bis eine Benachrichtigung angezeigt wird, die auf eine erfolgreiche Übermittlung hinweist.
- Klicken Sie auf Refresh Level 2 and 3 runs, um Level 2-Daten aufzufüllen, die innerhalb von Sekunden nach der Übermittlung verfügbar sind.
- Wählen Sie unter Daten der Ebene 2 anzeigen die abgeschlossenen Domänenbeitritte aus der Dropdown-Liste aus und klicken Sie auf die Schaltfläche Daten der Ebene 2 anzeigen, um die Ergebnisse auf einer neuen Seite zu öffnen.
4. Zugriff und Verständnis der Daten: SeqAPASS Level 1 und Level 2
- Scrollen Sie auf der Seite Proteininformationen abfragen nach unten, um einen Bericht über die Ergebnisse anzuzeigen - standardmäßig wird ein Primärbericht mit Analysen der Ebenen 1 und 2 bereitgestellt. Aktivieren Sie das Optionsfeld Vollständiger Bericht, um einen detaillierteren Bericht anzuzeigen, der alle Sequenztreffer und Ausrichtungsmetriken enthält. Klicken Sie in beiden Berichten auf den entsprechenden Beitritt/ID/Namen, um auf die transparenten Proteinausrichtungs- und Taxonomieinformationen in der NCBI-Datenbank zuzugreifen.
- Scrollen Sie zur rechten Seite der Ergebnistabelle, um die ECOTOX-Spalte anzuzeigen. Klicken Sie auf Links zur ECOTOXicology Knowledgebase (ECOTOX), um schnell entsprechende Toxizitätsdaten für Arten mit Suszeptibilitätsvorhersagen zu sammeln.
HINWEIS: ECOTOX ist eine umfassende, öffentlich zugängliche Wissensdatenbank, die einzelne chemische Toxizitätsdaten für aquatische und terrestrische Pflanzen und Wildtiere bereitstellt. SeqAPASS v6.0 enthält ein ECOTOX-Widget, um sich schneller mit relevanten ECOTOX-Daten nach Chemikalien und Spezies von Interesse zu verbinden. - Klicken Sie auf Tabelle herunterladen, um die Tabelle als Tabellenkalkulationsdatei zu speichern. Klicken Sie auf die Schaltfläche Zusammenfassungsbericht anzeigen, um eine Zusammenfassungsberichtstabelle anzuzeigen und herunterzuladen, die Daten nach taxonomischer Gruppe sortiert enthält.
HINWEIS: Datenzusammenfassungstabellen sind sowohl für primäre als auch für vollständige Berichte verfügbar und bieten einen Überblick über Vorhersagen für ein bestimmtes Ziel.
5. Bearbeiten von Dateneinstellungen: SeqAPASS Level 1 und Level 2
HINWEIS: Sowohl in Level-1- als auch in Level-2-Analysen wird davon ausgegangen, dass je größer die Proteinähnlichkeit ist, desto größer ist die Wahrscheinlichkeit, dass eine Chemikalie mit dem Protein in ähnlicher Weise wie die Abfragespezies / -protein interagiert, wodurch sie anfällig für mögliche Auswirkungen von Chemikalien mit diesem molekularen Ziel wird. Aufgrund der Ähnlichkeit dieser Daten werden Schritte zum Verständnis von Level-1- und Level-2-Daten in einem einzigen Protokoll zusammengefasst.
- In den Untermenüs oben unter Proteininformationen abfragen können Sie auf die Berichtseinstellungen zugreifen und diese bearbeiten und die Standardeinstellungen für alle Berichtsoptionen für die meisten Analysen verwenden. Wenn es eine wissenschaftliche Begründung für die Änderung der Standardeinstellung gibt, führen Sie die folgenden optionalen Schritte aus:
- (OPTIONAL) Klicken Sie auf das Pluszeichen + neben Empfindlichkeitsgrenzwert, um die Einstellungen für den Empfindlichkeitsgrenzwert auf einer neuen Registerkarte anzuzeigen und anzupassen. Wählen Sie entweder einen neuen Cutoff-Wert aus einer Dropdown-Liste aus oder geben Sie einen benutzerdefinierten Cutoff-Wert ein.
- (OPTIONAL) Ändern Sie die Zahl im Feld E-Wert (die Anzahl der verschiedenen Ausrichtungen, die zufällig auftreten können), wenn etwas anderes als der Standardwert gewünscht wird.
HINWEIS: Jedes Protein mit einem E-Wert, der größer als die Zahl in der Box ist, wird aus dem primären Bericht entfernt. - (OPTIONAL) Verwenden Sie die Option Nach taxonomischer Gruppe sortieren, um die Ebene der taxonomischen Hierarchie auszuwählen, die in der Spalte Gefilterte taxonomische Gruppe in der Ergebnistabelle angezeigt werden soll.
HINWEIS: Wenn Sie die taxonomische Hierarchie ändern, ändert sich auch die Vorhersage der Anfälligkeit basierend auf den Arten aus jeder gefilterten Gruppe, die oberhalb des Grenzwerts gefunden werden. - (OPTIONAL) Ändern Sie das Feld Common Domain (wie viele gemeinsame Domänen ein Protein mit dem Abfrageprotein teilen muss, um in die Ergebnisse aufgenommen zu werden), wenn etwas anderes als der Standardwert gewünscht wird.
HINWEIS: Da die Standardeinstellung 1 ist, wird jede Sequenz, die nicht mindestens eine gemeinsame Domäne mit dem Abfrageprotein teilt, ausgeschlossen. - (OPTIONAL) Wählen Sie Nein unter Spezies lesen aus, um die Suszeptibilitätsvorhersagen von Y nur dann zurückzuerhalten, wenn die prozentuale Ähnlichkeit größer oder gleich dem Cutoff ist oder wenn der Treffer als orthologischer Kandidat identifiziert wird.
HINWEIS: Diese Einstellung ist standardmäßig auf Ja gesetzt, was bedeutet, dass eine Suszeptibilitätsvorhersage von Y für alle orthologischen Kandidaten, alle Arten, die oberhalb des Grenzwerts für die Anfälligkeit aufgeführt sind, und alle Arten unterhalb des Grenzwerts aus derselben taxonomischen Gruppe mit einer oder mehreren Arten über dem Grenzwert gemeldet wird.
- Klicken Sie auf die Schaltfläche Aktuelle Berichtseinstellungen herunterladen , um eine Datei herunterzuladen, in der die aktuell angewendeten Einstellungen erfasst werden.
HINWEIS: Die ausgewählte spezifische Bewertungsstufe (1, 2 oder 3) bestimmt die Einstellungen, die im Bericht angezeigt werden.
6. Visualisierung der Daten: SeqAPASS Level 1 und Level 2
- Klicken Sie auf das Pluszeichen + neben Visualisierung und klicken Sie auf die Schaltfläche Daten visualisieren , um eine separate Registerkarte mit den benutzerdefinierten Informationen und der Option zur Auswahl eines interaktiven Ergebnisdiagramms zu öffnen.
- Klicken Sie auf Boxplot, um den interaktiven Boxplot und die Plotsteuerelemente zu öffnen und die Boxplot-Visualisierung aktiv zu aktualisieren, um Änderungen an der Datentabelle widerzuspiegeln und Grafiken in Publikations- und Präsentationsqualität bereitzustellen.
HINWEIS: Das Standard-Boxplot zeigt Artengruppen auf der x-Achse und prozentuale Ähnlichkeit auf der y-Achse an. Boxplots zeigen den Suszeptibilitätsgrenzwert (gestrichelte Linie), die prozentuale Ähnlichkeit zwischen den Arten im Vergleich zu den Abfragearten sowie Mittel- und Medianwerte für jede taxonomische Gruppe zusammen mit dem 25. und 75. Perzentil und dem Interquartilbereich an. Abhängig vom Ziel der Analyse und den Bedürfnissen des Benutzers können viele Boxplot-Funktionen durch die folgenden optionalen Schritte geändert werden.- (OPTIONAL) Informationen zum Anpassen der angezeigten taxonomischen Gruppen finden Sie im Feld Taxonomische Gruppen im Abschnitt Steuerelemente . Entfernen Sie Gruppen, indem Sie über die Namen scrollen und x auswählen oder das Dropdown-Menü Taxonomische Gruppen verwenden.
- (OPTIONAL) Um eine Legende hinzuzufügen, die eine Art von Interesse oder bestimmte vordefinierte Gruppen (z. B. gefährdete oder bedrohte Arten) identifiziert, bewegen Sie den Mauszeiger über einen taxonomischen Gruppennamen auf der x-Achse, um ein Popup-Fenster zu aktivieren, in dem die drei wichtigsten Arten nach höchster prozentualer Ähnlichkeit sortiert sind. Bewegen Sie den Mauszeiger über die Arten in der Legende, um ein Popup-Fenster mit den entsprechenden Arteninformationen zu generieren. Klicken Sie auf das Kästchen für eine bestimmte taxonomische Gruppe, um eine herunterladbare Übersichtstabelle mit Arten und Vorhersagen zu erstellen.
- Klicken Sie auf Boxplot herunterladen , um einen Dateityp auszuwählen, die Auflösung in Breite/Höhe anzupassen und die Visualisierung zu speichern.
7. Entwicklung und Durchführung einer SeqAPASS-Analyse: Ebene 3
HINWEIS: Eine Level-3-Analyse bewertet vom Benutzer identifizierte Aminosäurereste innerhalb des Abfrageproteins und vergleicht schnell die Erhaltung dieser Rückstände über Arten hinweg. Es wird angenommen, dass Spezies, in denen diese Rückstände konserviert sind, eher mit einer Chemikalie in ähnlicher Weise interagieren wie die Template-Spezies/Protein. Da sich Level 3 auf einzelne Aminosäuren konzentriert, kann eine Analyse nur durchgeführt werden, wenn detaillierte Kenntnisse über die Aminosäurereste verfügbar sind, die für die chemische Protein- oder Protein-Protein-Wechselwirkung entscheidend sind.
- Klicken Sie auf das Pluszeichen + neben der Kopfzeile Ebene 3 auf der Seite Level 1 Query Protein Information, um das Menü Level 3 Query aufzufüllen.
- Klicken Sie auf das Pluszeichen + neben dem Referenz-Explorer, um das Referenz-Explorer-Tool zu öffnen, das eine vordefinierte boolesche Zeichenfolge generiert, um die verfügbare Literatur abzufragen, und Benutzer bei der Identifizierung geeigneter Literatur unterstützt, um die Identifizierung kritischer Aminosäuren zu unterstützen, die in der Bewertung der Stufe 3 verwendet werden sollen (Tabelle 2 und Abbildung 2).
- (OPTIONAL) Sobald das Abfrageprotein automatisch ausgefüllt wurde, verwenden Sie die Funktion Proteinnamen hinzufügen , um weitere Proteine hinzuzufügen.
- Klicken Sie auf Google Scholar-Link generieren , um ein Popup-Fenster mit einer automatisch generierten Suchzeichenfolge mit relevanten Suchbegriffen zu öffnen.
- Klicken Sie auf Google Scholar durchsuchen , um die Literaturdatenbank anhand der Suchzeichenfolge abzufragen.
- Alternativ können Sie auf In Zwischenablage kopieren klicken und die Suchzeichenfolge anpassen, indem Sie Begriffe mithilfe der Funktionen im Verweis-Explorer hinzufügen oder entfernen.
8. Identifizierung kritischer Aminosäurereste anhand identifizierter Literatur
- Wählen Sie im Menü Abfrage der Ebene 3 die Vorlagensequenz aus, an der die vom Benutzer ausgewählten Arten ausgerichtet werden sollen.
HINWEIS: Diese Vorlagensequenz wird üblicherweise auf der Grundlage der Literatur ausgewählt, für die die kritischen Aminosäuren identifiziert wurden, und kann die gleiche oder eine andere Art sein als die in Level 1 und Level 2 abgefragten.- (OPTIONAL) Verwenden Sie das Feld Zusätzliche Vergleiche , um alle Zugänge/Sequenzen zu vergleichen, die nicht in den Tabellen Primärer/Vollständiger Bericht angezeigt werden.
- Geben Sie einen benutzerdefinierten Namen für den Level 3-Lauf in das Textfeld Level 3-Ausführungsnamen eingeben ein, um den abgeschlossenen Level 3-Lauf zu identifizieren. Wählen Sie für jede Bewertung einen eindeutigen Namen.
- Wählen Sie im Feld Taxonomische Gruppe(n) auswählen die gewünschte taxonomische Gruppe aus. Wählen Sie eine taxonomische Gruppe aus, um die Tabelle automatisch nach dieser taxonomischen Gruppe zu filtern.
- Klicken Sie in der Ergebnistabelle manuell auf das Kontrollkästchen neben allen Arten , die an der Vorlagensequenz ausgerichtet werden sollen.
HINWEIS: Um eine angemessene Ausrichtung zu gewährleisten, sollte jeweils eine taxonomische Gruppe mit der Vorlage verglichen werden. Wählen Sie nur ähnlich annotierte Proteine für die interessierende Spezies aus. Bei der Auswahl von Sequenzen zum Vergleich ist es wichtig, auf bestimmte Sequenzen zu achten (z. B. hypothetisch, NIEDRIGE QUALITÄT oder Teilsequenzen). Sofern es keine transparente Begründung für die Aufnahme gibt, ist es am besten, diese Sequenzen auszuschließen, da sie Vorhersagen aufgrund unvollständiger oder unangemessener Sequenzinformationen verzerren können. - Wiederholen Sie die Schritte, um alle taxonomischen Interessengruppen auszurichten.
- Klicken Sie auf Level-2- und -3-Läufe aktualisieren, sobald alle Arten ausgerichtet wurden, um das Menü Run Name der Ebene 3 auswählen mit den abgeschlossenen Level-3-Aufträgen zu füllen und die Daten sofort von einer Level-3-Ausrichtung abzurufen.
- Klicken Sie auf Daten der Ebene 3 kombinieren, um Ausrichtungen aus mehreren taxonomischen Gruppen zu kombinieren.
- Um einen einzelnen Bericht anzuzeigen, wählen Sie alternativ den benutzerdefinierten Namen unter Zu anzeigende Abfrage auswählen aus, und klicken Sie auf Daten der Ebene 3 anzeigen.
- Wählen Sie im Menü "Level 3-Berichte kombinieren" die Vorlage Level 3 aus, die als Grundlage für den Vergleich der Aminosäurerückstände verwendet werden soll, und klicken Sie auf Next (Weiter).
- Wählen Sie unter Aufträge der Ebene 3 abgeschlossene Aufträge zum Vergleich aus und klicken Sie auf Weiter. Verwenden Sie die Funktion Aufträge der Bestellebene 3 , um die taxonomischen Gruppen bei Bedarf neu anzuordnen. Klicken Sie auf Level-3-Daten anzeigen , um eine Level-3-Berichtsseite mit den kombinierten taxonomischen Gruppen zu erstellen.
- Wählen Sie zuvor identifizierte Aminosäurepositionen für die Vorlagenspezies aus, indem Sie die Aminosäureposition(en), getrennt durch Kommas, in das Feld Enter Amino Acid Residue Positions (Positionen für Aminosäurereste eingeben ) eingeben und dann In Rückstandsliste kopieren auswählen. Selektieren Sie Rückstände in der Schablonensequenz direkt aus der Shuttlebox.
- Klicken Sie auf Bericht aktualisieren, um die Seite zu aktualisieren und die Vorhersagen zur Anfälligkeit der Stufe 3 anzuzeigen.
HINWEIS: Level 3 verwendet ein einfaches Regelwerk, das aus grundlegenden Deskriptoren der funktionellen Eigenschaften der Seitenkette (z. B. aliphatisch, aromatisch) und der molekularen Dimensionen (Molekulargewichtsunterschiede >30 g / mol) abgeleitet wird, um zu bestimmen, ob Unterschiede an Schlüsselpositionen wahrscheinlich Proteininteraktionen beeinflussen13.
9. Visualisierung von Level 3 SeqAPASS-Daten
HINWEIS: Wie in den vorherigen Stufen sind die Berichte "Primär" und "Vollständig" verfügbar. Zusätzlich zu den Daten, die mit den Daten in Level 1 und 2 identisch sind, zeigt der primäre Bericht Aminosäurepositionen, Abkürzungen und eine Ja/Nein-ähnliche Anfälligkeit (Y/N) wie die Vorlagenvorhersage an. In ähnlicher Weise enthält der vollständige Bericht Informationen über die Klassifizierung der Aminosäureseitenkette und das Molekulargewicht.
- Führen Sie auf der Berichtsseite der Ebene 3 einen Bildlauf nach unten durch, um einen Bericht mit den Ergebnissen anzuzeigen. Klicken Sie unten im Bericht auf Tabelle herunterladen, um die Tabelle zu speichern.
- Klicken Sie auf Zusammenfassungsbericht der Ebene 3 anzeigen , um eine Zusammenfassungsberichtstabelle mit Daten nach taxonomischer Gruppe sortiert anzuzeigen und herunterzuladen. Klicken Sie auf das Pluszeichen + neben Visualisierung auf der Seite Level 3 Report, um eine separate Browserregisterkarte mit den benutzerdefinierten Informationen und der Option zum Anzeigen der Ergebnisse in Form einer interaktiven Heatmap zu öffnen.
- Klicken Sie auf der Seite Visualisierungsinformationen auf Heatmap, um die interaktive Grafik und die Steuerelemente zu öffnen und die Heatmap-Visualisierung aktiv zu aktualisieren, um Änderungen an der Datentabelle widerzuspiegeln. Führen Sie die folgenden optionalen Schritte aus, um die Heatmap anzupassen.
- (OPTIONAL) Wählen Sie Berichtsoptionen , um zwischen einem einfachen Bericht, der die Aminosäureposition, die Abkürzung aus einem Buchstaben und die Aminosäureähnlichkeit anzeigt, oder einem vollständigen Bericht, der detaillierte Informationen zu jeder ausgewählten Aminosäure anzeigt, zu wechseln.
- (OPTIONAL) Wählen Sie Berichtsoptionen , um die Anzeige von Arten zu ändern, entweder nach Allgemeiner Name oder Wissenschaftlicher Name.
HINWEIS: Innerhalb des einfachen Berichts werden Aminosäuren als Gesamtübereinstimmung (dunkelblau), Teilübereinstimmung (hellblau, Substitutionen, die nur ein Kriterium erfüllen) oder Keine Übereinstimmung (gelb, Substitutionen, die keine der Kriterien erfüllen) zur Vorlagenaminosäure kategorisiert. Im vollständigen Bericht werden Vergleiche entweder als Gesamtübereinstimmung (dunkelblau) oder "Keine Übereinstimmung" (gelb) angezeigt. - (OPTIONAL) Wählen Sie Optionale Auswahl , um nützliche Informationen wie Ortholog-Kandidaten, Bedrohte Arten, gefährdete Arten oder häufige Modellorganismen hervorzuheben.
- (OPTIONAL) Wählen Sie Heatmap-Einstellungen , um zusätzliche Anpassungsoptionen auszuwählen, einschließlich Hinzufügen oder Entfernen von Spalten, Legenden und Text.
- Klicken Sie auf Boxplot herunterladen , um einen Dateityp auszuwählen und die Visualisierung zu speichern.
10. Interpretation der SeqAPASS-Ergebnisse: Evidenzlinien für Proteinkonservierung
HINWEIS: Zur leichteren Interpretation enthält dieses Tool einen zusammenfassenden Entscheidungsbericht (DS-Bericht), der für die Integration von Daten über Ebenen hinweg entwickelt wurde. Der DS-Bericht enthält die Ergebnisse (d.h. Datentabellen und/oder Visualisierungen), die der Benutzer ausgewählt hat, und ermöglicht die schnelle Auswertung von Suszeptibilitätsvorhersagen über mehrere Ebenen für mehrere Arten gleichzeitig.
- Klicken Sie auf den Ergebnis- oder Datenvisualisierungsseiten auf Stufe # an DS-Bericht senden, und warten Sie, bis die Daten "gepusht" und die Registerkarte DS-Bericht aktiv wird.
HINWEIS: Wenn Ergebnisse oder Änderungen nicht per Push Level # to DS Report übertragen wurden, bleibt Push Level # to DS Report aktiv, bis es ausgewählt ist. Wenn eine Einstellung geändert wurde, wird der Text Zum Pushen neuer Änderungen klicken angezeigt, bis die Änderungen per Push in den Bericht übertragen werden. Visualisierungen können jederzeit während der Auswertung in den DS Report übertragen werden. - Wählen Sie jederzeit die Registerkarte DS-Bericht aus, um auf die DS-Seite zuzugreifen.
HINWEIS: Für alle Arten, die in Stufe 1 ausgerichtet sind, enthält die Tabelle des zusammenfassenden Abschlussberichts die wichtigen Daten und Vorhersagen zur Anfälligkeit für jede Analyse. Wenn eine Art in der DS-Tabelle nicht im Level-3-Bericht enthalten war, aber in Level 1- und/oder Level 2-Jobs gefunden wurde, erhält die Zelle in der Tabelle eine nicht zutreffende (NA)-Bezeichnung für die Level-3-Suszeptibilitätsvorhersage.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Um die Anwendung des SeqAPASS-Tools zu demonstrieren und neue Funktionen hervorzuheben, werden zwei Fallstudien beschrieben, die Fälle darstellen, in denen die Proteinkonservierung vorhersagt, dass es Unterschiede in der chemischen Anfälligkeit zwischen den Arten gibt (humanes Transthyretin) und dass es keine Unterschiede gibt (μ Opioidrezeptor [MOR]). Das erste dieser Beispiele befasst sich mit Proteinsequenz-/Strukturvergleichen zur Vorhersage des Anwendungsbereichs für unerwünschte Ergebnispfade (AOPs, siehe
Access restricted. Please log in or start a trial to view this content.
Diskussion
Es ist allgemein anerkannt, dass es nicht möglich ist, genügend Arten empirisch zu testen, um die genomische, phänotypische, physiologische und Verhaltensvielfalt lebender Organismen zu erfassen, die Chemikalien von toxikologischem Interesse ausgesetzt sein können. Das Ziel von SeqAPASS ist es, die Nutzung bestehender und kontinuierlich expandierender Proteinsequenz- und Strukturdaten zu maximieren, um die Extrapolation von chemischen Toxizitätsdaten / Wissen von getesteten Organismen auf Hunderte oder Tausende ande...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Die Autoren danken Dr. Daniel L. Villeneuve (U.S. EPA, Center for Computational Toxicology and Exposure) und Dr. Jon A. Doering (Department of Environmental Sciences, Louisiana State University) für die Kommentare zu einem früheren Entwurf des Manuskripts. Diese Arbeit wurde von der U.S. Environmental Protection Agency unterstützt. Die in diesem Papier geäußerten Ansichten sind die der Autoren und spiegeln nicht unbedingt die Ansichten oder Richtlinien der US-Umweltschutzbehörde wider, noch deutet die Erwähnung von Handelsnamen oder kommerziellen Produkten auf eine Billigung durch die Bundesregierung hin.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Spreadsheet program | N/A | N/A | Any program that can be used to view and work with csv files (e.g. Microsoft Excel, OpenOffice Calc, Google Docs) can be used to access data export files. |
| Basic computing setup and internet access | N/A | N/A | SeqAPASS is a free, online tool that can be easily used via an internet connection. No software downloads are required. |
Referenzen
- Krewski, D., et al. Toxicity testing in the 21st century: a vision and a strategy. Journal of Toxicology and Environmental Health, Part B. 13 (2-4), 51-138 (2010).
- Wang, Z., Walker, G. W., Muir, D. C. G., Nagatani-Yoshida, K. Toward a global understanding of chemical pollution: A first comprehensive analysis of national and regional chemical inventories. Environmental Science & Technology. 54 (5), 2575-2584 (2020).
- Brooks, B. W., et al. Toxicology advances for 21st century chemical pollution. One Earth. 2 (4), 312-316 (2020).
- Kostal, J., Voutchkova-Kostal, A. Going all in: A strategic investment in in silico toxicology. Chemical Research in Toxicology. 33 (4), 880-888 (2020).
- Cheng, W., Doering, J. A., LaLone, C., Ng, C. Integrative computational approaches to inform relative bioaccumulation potential of per- and polyfluoroalkyl substances (PFAS) across species. Toxicology Sciences. 180 (2), 212-223 (2021).
- Kostich, M. S., Lazorchak, J. M. Risks to aquatic organisms posed by human pharmaceutical use. Science of the Total Environment. 389 (2-3), 329-339 (2008).
- Gunnarsson, L., Jauhiainen, A., Kristiansson, E., Nerman, O., Larsson, D. G. Evolutionary conservation of human drug targets in organisms used for environmental risk assessments. Environmental Science & Technology. 42 (15), 5807-5813 (2008).
- LaLone, C. A., et al. Evidence for cross species extrapolation of mammalian-based high-throughput screening assay results. Environmental Science & Technology. 52 (23), 13960-13971 (2018).
- LaLone, C. A., et al. Editor's highlight: Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS): A web-based tool for addressing the challenges of cross-species extrapolation of chemical toxicity. Toxicology Sciences. 153 (2), 228-245 (2016).
- Head, J. A., Hahn, M. E., Kennedy, S. W. Key amino acids in the aryl hydrocarbon receptor predict dioxin sensitivity in avian species. Environmental Science & Technology. 42 (19), 7535-7541 (2008).
- Bass, C., et al. Mutation of a nicotinic acetylcholine receptor β subunit is associated with resistance to neonicotinoid insecticides in the aphid Myzus persicae. BMC Neuroscience. 12, 51-51 (2011).
- Erdmanis, L., et al. Association of neonicotinoid insensitivity with a conserved residue in the loop d binding region of the tick nicotinic acetylcholine receptor. Biochemistry. 51 (23), 4627-4629 (2012).
- Doering, J. A., et al. et al. In silico site-directed mutagenesis informs species-specific predictions of chemical susceptibility derived from the Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS) tool. Toxicology Sciences. 166 (1), 131-145 (2018).
- Noyes, P. D., et al. Evaluating chemicals for thyroid disruption: Opportunities and challenges with in vitro testing and adverse outcome pathway approaches. Environmental Health Perspectives. 127 (9), 95001(2019).
- Park, G. Y., Jamerlan, A., Shim, K. H., An, S. S. A. Diagnostic and treatment approaches involving transthyretin in amyloidogenic diseases. Int J Mol Sci. 20 (12), 2982(2019).
- Rabah, S. A., Gowan, I. L., Pagnin, M., Osman, N., Richardson, S. J. Thyroid hormone distributor proteins during development in vertebrates. Front Endocrinol (Lausane). 10, 506(2019).
- Richardson, S. J. Cell and molecular biology of transthyretin and thyroid hormones. International Review of Cytology. 258, 137-193 (2007).
- Yamauchi, K., Ishihara, A. Transthyretin and Endocrine Disruptors. Recent Advances in Transthyretin Evolution, Structure and Biological Functions. Richardson, S. J., Cody, V. , Springer. Berlin Heidelberg, Germany. 159-171 (2009).
- Iakovleva, I., et al. Tetrabromobisphenol A is an efficient stabilizer of the transthyretin tetramer. PLoS One. 11 (4), 0153529(2016).
- Ishihara, A., Sawatsubashi, S., Yamauchi, K. Endocrine disrupting chemicals: Interference of thyroid hormone binding to transthyretins and to thyroid hormone receptors. Molecular and Cellular Endocrinology. 199 (1), 105-117 (2003).
- Kar, S., Sepúlveda, M. S., Roy, K., Leszczynski, J. Endocrine-disrupting activity of per- and polyfluoroalkyl substances: Exploring combined approaches of ligand and structure based modeling. Chemosphere. 184, 514-523 (2017).
- Morais-de-Sa, E., Pereira, P. J., Saraiva, M. J., Damas, A. M. The crystal structure of transthyretin in complex with diethylstilbestrol: A promising template for the design of amyloid inhibitors. Journal of Biological Chemistry. 279 (51), 53483-53490 (2004).
- Morgado, I., Campinho, M. A., Costa, R., Jacinto, R., Power, D. M. Disruption of the thyroid system by diethylstilbestrol and ioxynil in the sea bream (Sparus aurata). Aquatic Toxicology. 92 (4), 271-280 (2009).
- Yamauchi, K., Prapunpoj, P., Richardson, S. J. Effect of diethylstilbestrol on thyroid hormone binding to amphibian transthyretins. General and Comparative Endocrinology. 119 (3), 329-339 (2000).
- Zhang, J., et al. Structure-based virtual screening protocol for in silico identification of potential thyroid disrupting chemicals targeting transthyretin. Environmental Science & Technology. 50 (21), 11984-11993 (2016).
- Ren, X. M., et al. Binding interactions of perfluoroalkyl substances with thyroid hormone transport proteins and potential toxicological implications. Toxicology. 366-367, 32-42 (2016).
- Wilson, N., Mbabazi, K., Seth, P., Smith, H., Davis, N. L. Drug and opioid-involved overdose deaths - United States, 2017-2018. Morbidity and Mortality Weekly Report. 69 (11), 290-297 (2020).
- EPA. National Pollutant Discharge Elimination System (NPDES). United States Environmental Protection Agency. , Available from: https://www.epa.gov/npdes/npdes-resources (2018).
- Duvallet, C., Hayes, B. D., Erickson, T. B., Chai, P. R., Matus, M. Mapping community opioid exposure through wastewater-based epidemiology as a means to engage pharmacies in harm reduction efforts. Preventing Chronic Disease. 17, 200053(2020).
- Gushgari, A. J., Venkatesan, A. K., Chen, J., Steele, J. C., Halden, R. U. Long-term tracking of opioid consumption in two United States cities using wastewater-based epidemiology approach. Water Research. 161, 171-180 (2019).
- Lau, B., Bretaud, S., Huang, Y., Lin, E., Guo, S. Dissociation of food and opiate preference by a genetic mutation in zebrafish. Genes Brain Behave. 5 (7), 497-505 (2006).
- Bossé, G. D., Peterson, R. T. Development of an opioid self-administration assay to study drug seeking in zebrafish. Behavioural Brain Research. 335, 158-166 (2017).
- Mottaz, H., et al. Dose-dependent effects of morphine on lipopolysaccharide (LPS)-induced inflammation, and involvement of multixenobiotic resistance (MXR) transporters in LPS efflux in teleost fish. Environmental Pollution. 221, 105-115 (2017).
- Manglik, A., et al. Crystal structure of the µ-opioid receptor bound to a morphinan antagonist. Nature. 485 (7398), 321-326 (2012).
- Comer, S. D., Cahill, C. M. Fentanyl: Receptor pharmacology, abuse potential, and implications for treatment. Neuroscience & Biobehavioral Reviews. 106, 49-57 (2019).
- Podlewska, S., Bugno, R., Kudla, L., Bojarski, A. J., Przewlocki, R. Molecular modeling of µ opioid receptor ligands with various functional properties: PZM21, SR-17018, morphine, and fentanyl-simulated interaction patterns confronted with experimental data. Molecules. 25 (20), 4636(2020).
- Huang, W., et al. Structural insights into µ-opioid receptor activation. Nature. 524 (7565), 315-321 (2015).
- Lipiński, P. F. J., et al. Fentanyl family at the mu-opioid receptor: Uniform assessment of binding and computational analysis. Molecules. 24 (4), 740(2019).
- Boland, L. A., Angles, J. M. Feline permethrin toxicity: Retrospective study of 42 cases. Journal of Feline Medicine and Surgery. 12 (2), 61-71 (2010).
- Stevenson, B. J., Pignatelli, P., Nikou, D., Paine, M. J. Pinpointing P450s associated with pyrethroid metabolism in the dengue vector, Aedes aegypti: developing new tools to combat insecticide resistance. PLoS Neglected Tropical Diseases. 6 (3), 1595(2012).
- Ankley, G. T., Gray, L. E. Cross-species conservation of endocrine pathways: A critical analysis of tier 1 fish and rat screening assays with 12 model chemicals. Environmental Toxicology and Chemistry. 32 (5), 1084-1087 (2013).
- Meteyer, C. U., Rideout, B. A., Gilbert, M., Shivaprasad, H. L., Oaks, J. L. Pathology and proposed pathophysiology of diclofenac poisoning in free-living and experimentally exposed oriental white-backed vultures (Gyps bengalensis). Journal of Wildlife Diseases. 41 (4), 707-716 (2005).
- EPA. ECOTOX User Guide: ECOTOXicology Knowledgebase System. EPA, United States Environmental Protection Agency. , Available from: https://cfpub.epa.gov/ecotox/index.cfm (2021).
- ECOS Environmental Conservation Online System. U.S. Fish & Wildlife Service. , Available from: https://ecos.fws.gov/ecp/ (2021).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten