JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
단백질 보존의 신속한 평가를 위한 종 간 감수성 도구를 예측하기 위한 서열 정렬 입증
요약
여기에서는 미국 환경 보호국 서열 정렬을 활용하여 종 감수성 (SeqAPASS) 도구를 예측하는 프로토콜을 제시합니다. 이 프로토콜은 단백질 보존을 신속하게 분석하고 종 전반에 걸쳐 화학적 감수성에 대한 사용자 정의 가능하고 쉽게 해석 가능한 예측을 제공하는 온라인 도구의 적용을 보여줍니다.
초록
미국 환경 보호국 서열 정렬을 통해 종 감수성 예측(SeqAPASS) 도구는 연구자와 규제 기관이 종 전반에 걸쳐 독성 정보를 외삽할 수 있는 빠르고 무료로 제공되는 온라인 스크리닝 애플리케이션입니다. 인간 세포, 생쥐, 쥐 및 제브라 피쉬와 같은 모델 시스템의 생물학적 표적의 경우 다양한 화학 물질에 대한 독성 데이터를 사용할 수 있습니다. 단백질 표적 보존의 평가를 통해이 도구는 이러한 모델 시스템에서 생성 된 데이터를 독성 데이터가없는 수천 종의 다른 종으로 외삽하여 상대적 고유 화학적 감수성을 예측하는 데 사용할 수 있습니다. 도구의 최신 릴리스(버전 2.0-6.1)에는 출판을 위한 데이터의 빠른 합성, 해석 및 사용과 프레젠테이션 품질의 그래픽이 가능한 새로운 기능이 통합되어 있습니다.
이러한 기능 중에는 사용자 정의 가능한 데이터 시각화와 해석의 용이성을 위해 SeqAPASS 데이터를 요약하도록 설계된 포괄적인 요약 보고서가 있습니다. 이 백서에서는 사용자가 작업을 제출하고, 다양한 수준의 단백질 서열 비교를 탐색하고, 결과 데이터를 해석 및 표시하는 과정을 안내하는 프로토콜에 대해 설명합니다. SeqAPASS v2.0-6.0의 새로운 기능이 강조됩니다. 또한이 도구를 사용하여 트랜스 티레틴 및 오피오이드 수용체 단백질 보존에 초점을 맞춘 두 가지 사용 사례가 설명됩니다. 마지막으로 SeqAPASS의 강점과 한계에 대해 논의하여 도구에 대한 적용 가능성 영역을 정의하고 종 간 외삽을 위한 다양한 응용 분야를 강조합니다.
서문
전통적으로 독성학 분야는 화학 안전성 평가에 필요한 데이터를 제공하기 위해 전체 동물 실험의 사용에 크게 의존해 왔습니다. 이러한 방법은 일반적으로 비용이 많이 들고 리소스 집약적입니다. 그러나 현재 사용되는 화학 물질의 수가 많고 새로운 화학 물질이 개발되는 속도가 빠르기 때문에 전 세계적으로 보다 효율적인화학 스크리닝 방법에 대한 필요성이 인식되고 있습니다1,2. 이러한 필요성과 그에 따른 동물 실험으로부터의 패러다임 전환은 유망한 대체 검사 전략3,4인 고처리량 스크리닝 분석, 고처리량 전사체학, 차세대 시퀀싱 및 컴퓨터 모델링을 포함한 많은 새로운 접근 방법의 개발로 이어졌습니다.
화학 물질 노출의 영향을 받을 수 있는 다양한 종에 대한 화학적 안전성을 평가하는 것은 기존의 독성 테스트뿐만 아니라 새로운 접근 방법으로도 지속적인 과제였습니다. 비교 및 예측 독성학의 발전은 다양한 종의 상대적 민감도를 이해하기 위한 프레임워크를 제공했으며 계산 방법의 기술 발전은 이러한 방법의 적용 가능성을 계속 높이고 있습니다. 지난 10년 동안 특정 화학 분자 표적에 대한 지식과 함께 기존 유전자 및 단백질 서열 데이터베이스를 활용하여 종 간 외삽에 대한 예측 접근 방식을 지원하고 일반적인 모델 유기체 5,6,7,8을 넘어 화학적 안전성 평가를 향상시키는 몇 가지 전략이 논의되었습니다.
과학을 행동으로 옮기고, 예측 독성학에 대한 이러한 기초 연구를 기반으로 화학 테스트 노력의 우선 순위를 지정하고, 의사 결정을 지원하기 위해 미국 환경 보호국 서열 정렬을 통해 종 감수성을 예측하는 서열 정렬(SeqAPASS) 도구가 만들어졌습니다. 이 도구는 지속적으로 확장되는 단백질 서열 정보의 공개 저장소를 사용하여 종의 다양성에 걸쳐 화학적 감수성을 예측하는 공개 및 무료 웹 기반 응용 프로그램입니다9. 특정 화학 물질에 대한 종의 상대적 고유 감수성은 해당 화학 물질의 알려진 단백질 표적의 보존을 평가하여 결정할 수 있다는 원칙에 따라 이 도구는 모든 종에 대해 알려진 민감성을 가진 종의 단백질 아미노산 서열을 기존 단백질 서열 데이터와 신속하게 비교합니다. 이 평가는 (1) 1차 아미노산 서열, (2) 기능 도메인 및 (3) 중요한 아미노산 잔기 비교를 포함한 세 가지 수준의 분석을 통해 완료되며, 각각은 화학-단백질 상호 작용에 대한 보다 심층적인 지식이 필요하고 감수성 예측에서 더 큰 분류학적 분해능을 제공합니다. SeqAPASS의 주요 강점은 사용자가 관심 있는 화학-단백질 또는 단백질-단백질 상호 작용과 관련하여 사용 가능한 정보의 양을 기반으로 표적 보존에 대한 추가 증거를 추가하여 평가를 맞춤화하고 개선할 수 있다는 것입니다.
첫 번째 버전은 2016년에 출시되었으며, 이를 통해 사용자는 1차 아미노산 서열과 기능 도메인을 간소화된 방식으로 평가하여 화학적 감수성을 예측할 수 있었고 최소한의 데이터 시각화 기능을 포함했습니다(표 1). 개별 아미노산 차이는 종의 화학적 감수성에 영향을 미칠 수 있는 화학-단백질 상호 작용에서 종 간 차이의 중요한 결정 요인으로 나타났습니다10,11,12. 따라서, 후속 버전은 직접적인 화학적 상호작용에 중요한 중요한 아미노산을 고려하기 위해 개발되었다13. 이해 관계자 및 사용자 피드백에 응답하여이 도구는 종 간 외삽의 문제를 해결하기 위해 연구자와 규제 커뮤니티 모두의 요구를 충족하도록 설계된 추가 새로운 기능과 함께 연간 버전 릴리스를 거쳤습니다 (표 1). 2020년 SeqAPASS 버전 5.0의 출시는 데이터 시각화 및 데이터 합성 옵션, 외부 링크, 요약 테이블 및 보고서 옵션, 그래픽 기능을 통합하는 사용자 중심 기능을 선보였습니다. 전반적으로 이 버전의 새로운 속성과 기능은 데이터 합성, 외부 데이터베이스 간의 상호 운용성 및 종 간 감수성 예측을 위한 데이터 해석의 용이성을 개선했습니다.
Access restricted. Please log in or start a trial to view this content.
프로토콜
1. 시작하기
참고: 여기에 제시된 프로토콜은 도구 유틸리티 및 주요 기능에 중점을 둡니다. 방법, 기능 및 구성 요소에 대한 자세한 설명은 웹 사이트의 포괄적인 사용자 가이드(표 1)에서 찾을 수 있습니다.
표 1: SeqAPASS 도구의 진화. 초기 배포에서 SeqAPASS 도구에 추가된 기능 및 업데이트 목록입니다. 약어: SeqAPASS = 종 감수성을 예측하기 위한 서열 정렬; 에코톡스 = 에코톡시콜로지 지식 기반. 이 표를 다운로드하려면 여기를 클릭하십시오.
- 크롬을 사용하여 https://seqapass.epa.gov/seqapass 로 이동합니다. 로그인을 선택하여 기존 계정을 사용하거나 지침에 따라 SeqAPASS 계정을 만들면 사용자가 완료된 작업을 실행, 저장, 액세스 및 사용자 지정할 수 있습니다.
- 분석을 수행하기 전에 먼저 기존 문헌 또는 기존 데이터를 검토하여 관심 단백질과 표적 또는 민감한 종을 식별합니다(그림 1). SeqAPASS에는 쿼리 단백질을 식별하는 데 도움이 되는 외부 리소스에 대한 링크가 포함되어 있으므로 단백질 표적 식별 아래의 드롭다운 버튼을 클릭하여 관련 리소스에 액세스합니다.
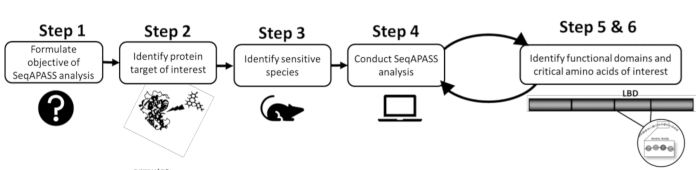
그림 1: SeqAPASS 문제 공식화: 성공적인 분석에 필요한 예비 정보의 개략도. 약어: SeqAPASS = 종 감수성을 예측하기 위한 서열 정렬; LBD = 리간드-결합 도메인. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2: 데이터베이스 간 SeqAPASS 상호 운용성. SeqAPASS에 통합된 외부 도구, 데이터베이스 및 리소스의 회로도입니다. 약어: SeqAPASS = 종 감수성을 예측하기 위한 서열 정렬; AOP = 불리한 결과 경로; NCBI = 국립 생명 공학 정보 센터; 에코톡스 = 에코톡시콜로지 지식 기반. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
표 2: SeqAPASS 도구에 통합된 링크, 리소스 및 도구. SeqAPASS 도구에 활용되는 다양한 데이터 소스, 링크 및 리소스 목록입니다. 약어: SeqAPASS = 종 감수성을 예측하기 위한 서열 정렬. 이 표를 다운로드하려면 여기를 클릭하십시오.
2. SeqAPASS 쿼리 개발 및 실행: 수준 1
참고: 레벨 1 분석에서, 질의 단백질의 전체 1차 아미노산 서열은 사용 가능한 서열 정보를 사용하여 모든 종의 1차 아미노산 서열과 비교됩니다. 이 도구는 알고리즘을 사용하여 공개적으로 사용 가능한 데이터를 마이닝, 수집 및 컴파일하여 종 간의 아미노산 서열을 신속하게 정렬하고 비교합니다. 백엔드는 국립 생명 공학 정보 센터 (NCBI) 데이터베이스의 정보를 저장하고 단백질 기본 로컬 정렬 검색 도구 (BLASTp) 54 및 제약 조건 기반 다중 정렬 도구 (COBALT) 55의 독립형 버전을 전략적으로 사용합니다.
- 1차 아미노산 서열 비교에서 종별 또는 수탁별을 클릭합니다. 종별 선택을 사용하여 종 목록을 입력하거나 선택하여 관심 있는 단백질 표적을 선택합니다.
- 수탁 텍스트 상자에 수탁을 입력하여 단백질 수탁(즉, NCBI 단백질 ID)을 직접 제출합니다.
- 요청 실행을 선택하여 쿼리를 제출합니다. 제출되면 브라우저 창의 오른쪽 상단 모서리에 성공적인 제출을 나타내는 알림이 나타날 때까지 기다립니다.
- 페이지 맨 위에 있는 SeqAPASS 실행 상태 탭을 선택하여 해당 사용자 계정으로 수행된 모든 SeqAPASS 실행 목록을 표시하고 완료율을 확인합니다.
- 적절한 라디오 단추가 선택된 상태에서 데이터 새로 고침을 클릭하여 수준 2 및 수준 3 실행 상태를 확인합니다.
- 페이지 맨 위에 있는 SeqAPASS 보고서 보기 탭을 선택하여 해당 계정에서 완료된 모든 보고서 목록에 액세스합니다.
- SeqAPASS 보고서 보기 탭에서 관심 있는 쿼리 단백질을 선택합니다. 선택한 보고서 요청을 클릭하여 수준 1 쿼리 단백질 정보 페이지를 열고 결과, 데이터 사용자 지정 옵션, 시각화 및 요약 보고서를 봅니다.
- 기본적으로 보고서 보기를 선택하여 웹 브라우저에서 데이터를 봅니다. 또는 보고서 저장을 선택하여 원시 데이터를 .zip 파일로 다운로드합니다.
참고: 레벨 1 분석에 필요한 시간은 해당 시점의 글로벌 사용자 요구, 대기열에 제출된 작업 수 및 제출된 작업에 대해 존재하는 단백질 정보의 양에 따라 달라집니다(버전 5.1의 경우 평균 23분). 단백질 표적이 이전에 완료된 경우 제출 즉시 몇 초 안에 데이터를 사용할 수 있습니다.
3. SeqAPASS 쿼리 개발 및 실행: 레벨 2
참고: 전체 단백질 서열이 화학적 상호 작용에 직접 관여하지 않기 때문에 레벨 2 분석은 기능 도메인의 아미노산 서열만 비교하여 더 낮은 분류학적 순위(예: 클래스, 순서, 패밀리)에서 감수성 예측을 수행합니다.
- 수준 1 쿼리 단백질 정보 페이지에서 수준 2 머리글 옆에 있는 더하기 기호 + 를 클릭하여 수준 2 쿼리 메뉴를 채웁니다.
- 관심 단백질(쿼리 단백질)에서 적절한 도메인을 식별합니다.
- 도메인이 식별되지 않은 경우 NCBI CDD(보존된 도메인 데이터베이스)(표 1)에 대한 통합 링크를 클릭하면 적절한 도메인 선택을 식별하는 데 도움이 될 수 있습니다.
참고: 일반적으로 특정 히트 도메인만 수준 2에서 쿼리로 선택됩니다.
- 도메인이 식별되지 않은 경우 NCBI CDD(보존된 도메인 데이터베이스)(표 1)에 대한 통합 링크를 클릭하면 적절한 도메인 선택을 식별하는 데 도움이 될 수 있습니다.
- 도메인 선택 상자를 클릭하여 쿼리 단백질에 대한 기능 도메인 목록을 자동으로 채웁니다.
- 드롭다운 목록에서 도메인 가입 을 선택하고 도메인 실행 요청 버튼을 클릭하여 수준 2 쿼리를 시작합니다. 제출이 완료되면 제출 성공을 나타내는 알림이 나타날 때까지 기다립니다.
- 수준 2 및 3 실행 새로 고침 을 클릭하여 제출 후 몇 초 이내에 사용할 수 있는 수준 2 데이터를 채웁니다.
- 수준 2 데이터 보기의 드롭다운 목록에서 완료된 도메인 가입을 선택하고 수준 2 데이터 보기 단추를 클릭하여 결과를 새 페이지에서 엽니다.
4. 데이터 접근 및 이해: SeqAPASS 레벨 1 및 레벨 2
- 쿼리 단백질 정보 페이지의 맨 아래로 스크롤하여 결과 보고서를 봅니다 - 기본 보고서는 기본적으로 레벨 1 및 2 분석과 함께 제공됩니다. 전체 보고서 라디오 단추를 선택하여 모든 시퀀스 적중 및 정렬 메트릭을 제공하는 보다 자세한 보고서를 봅니다. 두 보고서에서 적절한 수탁/ID/이름을 클릭하여 NCBI 데이터베이스의 투명한 단백질 정렬 및 분류 정보에 액세스합니다.
- 결과 표의 오른쪽으로 스크롤하여 ECOTOX 열을 봅니다. ECOTOXicology 지식 베이스(ECOTOX)에 대한 링크를 클릭하여 감수성 예측이 있는 종에 대한 해당 독성 데이터를 신속하게 수집합니다.
참고: ECOTOX는 수생 및 육상 식물과 야생 동물에 대한 단일 화학 독성 데이터를 제공하는 포괄적이고 공개적으로 사용 가능한 지식 기반입니다. SeqAPASS v6.0에는 화학 물질 및 관심 종별로 관련 ECOTOX 데이터와 보다 신속하게 연결할 수 있는 ECOTOX 위젯이 포함되어 있습니다. - 테이블 다운로드를 클릭하여 테이블을 스프레드시트 파일로 저장합니다. 요약 보고서 보기 버튼을 클릭하여 분류 그룹별로 정렬된 데이터를 표시하는 요약 보고서 테이블을 보고 다운로드합니다.
참고: 데이터 요약 테이블은 기본 보고서와 전체 보고서 모두에 사용할 수 있으며 지정된 대상에 대한 예측에 대한 개요를 제공합니다.
5. 데이터 설정 조작: SeqAPASS 레벨 1 및 레벨 2
참고: 레벨 1 및 레벨 2 분석 모두에서 단백질 유사성이 클수록 화학 물질이 쿼리 종/단백질과 유사한 방식으로 단백질과 상호 작용할 가능성이 커져 이 분자 표적을 가진 화학 물질의 잠재적 영향에 취약하다고 가정합니다. 이러한 데이터의 유사성으로 인해 레벨 1 및 2 데이터를 이해하기 위한 단계가 단일 프로토콜에 함께 요약되어 있습니다.
- 단백질 정보 쿼리 상단의 하위 메뉴를 참조하여 보고서 설정에 액세스하고 조작하며 대부분의 분석에 대한 모든 보고서 옵션에 대해 기본 설정을 사용합니다. 기본 설정을 변경하는 과학적 근거가 있는 경우 다음 선택적 단계를 수행합니다.
- (선택 사항) 감수성 컷오프 옆에 있는 더하기 기호 + 를 클릭하여 새 탭에서 감수 성 컷오프 설정을 보고 조정합니다. 드롭다운 리스트에서 새 컷오프 값을 선택하거나 사용자정의 컷오프 값을 입력합니다.
- (선택 사항) 기본값 이외의 다른 것이 필요한 경우 E-Value (우연히 발생할 것으로 예상되는 다른 선형의 수) 필드의 숫자를 변경합니다.
참고: 상자 안의 숫자보다 큰 E 값을 가진 단백질은 기본 보고서에서 제거됩니다. - (선택 사항) 분류 그룹 별 정렬 옵션을 사용하여 결과 테이블의 필터링된 분류 그룹 열에 표시할 분류 계층 수준을 선택합니다.
참고: 분류학적 계층 구조를 변경하면 컷오프 위에서 발견된 각 필터링된 그룹의 종을 기반으로 한 감수성 예측도 변경됩니다. - (선택 사항) Common Domain (단백질이 결과에 포함되기 위해 쿼리 단백질과 공유해야 하는 공통 도메인 수) 필드를 기본값 이외의 다른 것이 필요한 경우 변경합니다.
참고: 기본 설정은 1이므로 쿼리 단백질과 하나 이상의 공통 도메인을 공유하지 않는 시퀀스는 제외됩니다. - (선택 사항) Species Read Across 아래에서 아니요를 선택하여 유사성 백분율이 컷오프보다 크거나 같거나 히트가 ortholog 후보로 식별되는 경우에만 Y의 감수성 예측을 다시 가져옵니다.
참고: 이 설정은 기본적으로 예로 설정되며, 이는 모든 ortholog 후보, 감수성 컷오프 위에 나열된 모든 종 및 컷오프 위에 하나 이상의 종이 있는 동일한 분류학적 그룹의 컷오프 아래의 모든 종에 대해 Y의 감수성 예측이 보고됨을 의미합니다.
- 현재 보고서 설정 다운로드 버튼을 클릭하여 적용된 현재 설정을 캡처하는 파일을 다운로드합니다.
참고: 선택한 특정 평가 수준(1, 2 또는 3)에 따라 보고서에 표시되는 설정이 결정됩니다.
6. 데이터 시각화: SeqAPASS 레벨 1 및 레벨 2
- 시각화 옆에 있는 더하기 기호 +를 클릭하고 데이터 시각화 버튼을 클릭하여 사용자 정의 정보와 대화형 결과 그래프를 선택하는 옵션을 표시하는 별도의 탭을 엽니다.
- Boxplot을 클릭하여 대화형 상자 그림 및 그림 컨트롤을 열고 상자 그림 시각화가 능동적으로 업데이트되어 데이터 테이블의 변경 사항을 반영하고 출판 및 프레젠테이션 품질의 그래픽을 제공할 수 있도록 합니다.
참고: 기본 상자 그림은 x-축에 종 그룹을 표시하고 y-축에 백분율 유사성을 표시합니다. 상자 그림은 민감도 컷오프(점선), 쿼리 종과 비교한 종 간 유사성 백분율, 각 분류 그룹의 평균 및 중앙값과 함께 25번째 및 75번째 백분위수 및 사분위수 범위를 표시합니다. 분석의 목적과 사용자의 요구에 따라 다음과 같은 선택적 단계를 통해 많은 상자 그림 특징을 수정할 수 있습니다.- (선택 사항) 표시된 분류 그룹을 사용자 지정하려면 컨트롤 섹션 아래의 분류 그룹 상자를 참조하십시오. 이름 위로 스크롤하고 x를 선택하거나 분류 그룹 드롭다운 메뉴를 사용하여 그룹을 제거합니다.
- (선택 사항) 관심 종 또는 미리 정의된 특정 그룹(예: 멸종 위기에 처하거나 멸종 위기에 처한 종)을 정확히 찾아내는 범례를 추가하려면 x축의 분류 그룹 이름 위로 마우스를 가져가면 가장 높은 유사성 백분율로 정렬된 상위 3개 종을 나열하는 팝업 상자가 활성화됩니다. 범례에서 종 위로 마우스를 가져가면 해당 종 정보가 포함된 팝업 상자가 생성됩니다. 특정 분류 그룹에 대한 상자를 클릭하여 종과 예측을 나열하는 다운로드 가능한 요약 테이블을 생성합니다.
- Boxplot 다운로드를 클릭하여 파일 유형을 선택하고, 너비/높이 해상도를 사용자 지정하고, 시각화를 저장합니다.
7. SeqAPASS 분석 개발 및 실행: 레벨 3
참고: 레벨 3 분석은 쿼리 단백질 내에서 사용자가 식별한 아미노산 잔기를 평가하고 종 간에 이러한 잔기의 보존을 신속하게 비교합니다. 이러한 잔기가 보존되는 종은 주형 종/단백질과 유사한 방식으로 화학 물질과 상호 작용할 가능성이 더 높은 것으로 가정합니다. 레벨 3은 개별 아미노산에 초점을 맞추기 때문에 화학-단백질 또는 단백질-단백질 상호 작용에 중요한 아미노산 잔기에 대한 자세한 지식이 있는 경우에만 분석을 수행할 수 있습니다.
- 수준 1 쿼리 단백질 정보 페이지에서 수준 3 머리글 옆에 있는 더하기 기호 +를 클릭하여 수준 3 쿼리 메뉴를 채웁니다.
- 참조 탐색기 옆에 있는 더하기 기호 + 를 클릭하여 참조 탐색기 도구를 열면 사전 정의된 부울 문자열을 생성하여 사용 가능한 문헌을 쿼리하고 사용자가 레벨 3 평가에 사용할 중요한 아미노산 식별을 지원하는 적절한 문헌을 식별하는 데 도움이 됩니다(표 2 및 그림 2).
- (선택 사항) 쿼리 단백질이 자동으로 채워지면 단백질 이름 추가 기능을 사용하여 단백질을 추가합니다.
- Google 학술 검색 링크 생성을 클릭하여 관련 검색어가 포함된 자동 생성된 검색어가 포함된 팝업을 엽니다.
- Google 학술검색 검색을 클릭하여 검색 문자열을 사용하여 문헌 데이터베이스를 쿼리합니다.
- 또는 클립보드에 복사 를 클릭하고 참조 탐색기의 함수를 사용하여 용어를 추가하거나 제거하여 검색 문자열을 사용자 지정합니다.
8. 확인된 문헌을 사용하여 중요한 아미노산 잔기 식별
- 사용자가 선택한 종을 Level 3 쿼리 메뉴에서 정렬할 템플릿 시퀀스 를 선택합니다.
참고: 이 주형 서열은 일반적으로 중요 아미노산이 확인된 문헌을 기반으로 선택되며 수준 1 및 수준 2에서 쿼리된 것과 동일하거나 다른 종일 수 있습니다.- (선택 사항) 추가 비교 상자를 사용하여 기본/전체 보고서 테이블에 나타나지 않는 가입/시퀀스를 비교할 수 있습니다.
- 수준 3 실행 이름 입력 텍스트 상자에 수준 3 실행에 대한 사용자 정의 이름을 입력하여 완료된 수준 3 실행을 식별합니다. 각 평가에 대해 고유한 이름을 선택합니다.
- 분류 그룹 선택 필드에서 관심 있는 분류 그룹을 선택합니다. 분류 그룹을 선택하여 해당 분류 그룹을 기준으로 테이블을 자동 필터링합니다.
- 결과 테이블에서 템플릿 시퀀스에 정렬할 종 옆에 있는 확인란을 수동으로 클릭합니다.
참고: 적절한 정렬을 보장하려면 한 번에 하나의 분류 그룹을 템플릿과 비교해야 합니다. 관심 종에 대해 유사하게 주석이 달린 단백질만 선택하십시오. 비교를 위해 시퀀스를 선택할 때 특정 시퀀스(예: 가설, LOW QUALITY 또는 부분)에 주의를 기울이는 것이 중요합니다. 포함에 대한 투명한 근거가 없는 한, 불완전하거나 부적절한 시퀀스 정보로 인해 예측을 왜곡할 수 있으므로 이러한 시퀀스를 제외하는 것이 가장 좋습니다. - 단계를 반복하여 관심 있는 모든 분류 그룹을 정렬합니다.
- 모든 종이 정렬되면 수준 2 및 3 실행 새로 고침 을 클릭하여 수준 3 실행 이름 선택 메뉴를 완료된 수준 3 작업으로 채우고 수준 3 맞춤에서 데이터를 즉시 가져옵니다.
- 레벨 3 데이터 결합을 클릭하여 여러 분류 그룹의 정렬을 결합합니다.
- 또는 단일 보고서를 보려면 조회할 쿼리 선택 에서 사용자 정의 이름을 선택하고 수준 3 데이터 보기를 클릭합니다.
- 레벨 3 보고서 결합 메뉴에서 아미노산 잔기 비교의 기초로 사용할 레벨 3 템플릿을 선택하고 다음을 클릭합니다.
- 레벨 3 작업에서 비교를 위해 완료된 작업을 선택하고 다음을 클릭합니다. 원하는 경우 주문 레벨 3 작업 함수를 사용하여 분류 그룹을 재정렬합니다. 수준 3 데이터 보기를 클릭하여 결합된 분류 그룹이 정렬된 수준 3 보고서 페이지를 생성합니다.
- Entry Amino Residue Positions 입력 상자에 쉼표로 구분된 아미노산 위치를 입력한 다음 잔차 목록에 복사를 선택하여 템플릿 종에 대해 이전에 식별된 아미노산 위치를 선택합니다. 셔틀 박스에서 템플릿 시퀀스의 잔류물을 직접 선택합니다.
- 보고서 업데이트를 클릭하여 페이지를 새로 고치고 수준 3 민감도 예측을 표시합니다.
참고: 레벨 3은 측쇄 기능적 특성(예: 지방족, 방향족) 및 분자 치수(분자량 차이>30g/mol)의 기본 설명자에서 파생된 간단한 규칙 세트를 사용하여 주요 위치의 차이가 단백질 상호 작용에 영향을 미칠 가능성이 있는지 여부를 결정합니다13.
9. 레벨 3 시퀀스 데이터 시각화
참고: 이전 레벨과 마찬가지로 기본 및 전체 보고서를 사용할 수 있습니다. 수준 1 및 2의 데이터와 동일한 데이터 외에도 기본 보고서에는 아미노산 위치, 약어 및 예/아니요(Y/N) 유사한 감수성이 템플릿 예측과 표시됩니다. 유사하게, 전체 보고서에는 아미노산 측쇄 분류 및 분자량에 대한 정보가 포함되어 있습니다.
- 수준 3 보고서 페이지에서 아래쪽으로 스크롤하여 결과 보고서를 봅니다. 보고서 아래쪽에 있는 테이블 다운로드를 클릭하여 테이블을 저장합니다.
- 수준 3 요약 보고서 보기를 클릭하여 분류 그룹별로 정렬된 데이터를 표시하는 요약 보고서 테이블을 보고 다운로드합니다. 수준 3 보고서 페이지에서 시각화 옆에 있는 더하기 기호 +를 클릭하여 사용자 정의 정보와 대화형 히트 맵 형식으로 결과를 볼 수 있는 옵션을 표시하는 별도의 브라우저 탭을 엽니다.
- 시각화 정보 페이지에서 히트 맵을 클릭하여 대화형 그래픽 및 컨트롤을 열고 히트 맵 시각화가 데이터 테이블의 변경 내용을 반영하도록 적극적으로 업데이트되도록 합니다. 다음 선택적 단계를 수행하여 히트 맵을 사용자 지정합니다.
- (선택 사항) 보고서 옵션을 선택하여 아미노산 위치, 한 글자 약어 및 아미노산 유사성을 표시하는 단순 보고서 또는 선택한 각 아미노산에 대한 자세한 정보를 표시하는 전체 보고서 간에 변경합니다.
- (선택 사항) 보고서 옵션을 선택하여 종이 표시되는 방식( 일반 이름 또는 학명)을 변경합니다.
참고: 단순 보고서 내에서 아미노산은 템플릿 아미노산에 대한 총 일치(진한 파란색), 부분 일치(연한 파란색, 한 가지 기준만 충족하는 대체) 또는 일치하지 않음(노란색, 두 기준을 모두 충족하는 대체)으로 분류됩니다. 전체 보고서 에는 비교가 총 일치(진한 파란색) 또는 일치하지 않음(노란색)으로 표시됩니다. - (선택 사항) 선택적 선택을 선택하여 Ortholog 후보, 멸종 위기 종, 멸종 위기 종 또는 공통 모델 유기체와 같은 유용한 정보를 강조 표시합니다.
- (선택 사항) 히트 맵 설정을 선택하여 열, 범례 및 텍스트 추가 또는 제거를 포함한 추가 사용자 지정 옵션을 선택합니다.
- Boxplot 다운로드를 클릭하여 파일 유형을 선택하고 시각화를 저장합니다.
10. SeqAPASS 결과의 해석: 단백질 보존을 위한 증거 라인
참고: 해석의 편의를 위해 이 도구에는 레벨 간 데이터를 통합하도록 설계된 결정 요약 보고서(DS 보고서)가 포함되어 있습니다. DS 보고서에는 사용자가 선택한 결과(즉, 데이터 테이블 및/또는 시각화)가 포함되어 있으며 여러 종에 대한 여러 레벨에서 동시에 감수성 예측을 빠르게 평가할 수 있습니다.
- 결과 또는 데이터 시각화 페이지에서 수준 #을 DS Report로 푸시를 클릭하고 데이터가 "푸시"되고 DS 보고서 탭이 활성화될 때까지 기다립니다.
참고: 결과 또는 변경 사항이 DS 보고서에 푸시되지 않은 경우 DS 보고서로 수준 #을 밀어넣기는 선택할 때까지 활성 상태로 유지됩니다. 설정이 변경된 경우 변경 내용이 보고서에 푸시될 때까지 클릭하여 새 변경 내용 푸시 텍스트가 표시됩니다. 시각화는 평가 중에 언제든지 DS Report에 푸시할 수 있습니다. - 언제든지 DS 보고서 탭을 선택하여 DS 페이지에 액세스합니다.
참고: 레벨 1에 정렬된 모든 종의 경우 최종 결정 요약 보고서 테이블에는 각 분석에 대한 중요한 데이터와 감수성 예측이 포함되어 있습니다. DS 테이블의 종이 수준 3 보고서에 포함되지 않았지만 수준 1 및/또는 수준 2 작업에서 발견된 경우 표의 셀은 수준 3 감수성 예측에 대해 해당 없음(NA) 지정을 받게 됩니다.
Access restricted. Please log in or start a trial to view this content.
결과
SeqAPASS 도구의 적용을 시연하고 새로운 기능을 강조하기 위해 단백질 보존이 종(인간 트랜스티레틴) 간에 화학적 감수성에 차이가 있고 차이가 없다고 예측하는 사례(μ 오피오이드 수용체[MOR])를 나타내는 두 가지 사례 연구가 설명됩니다. 이 중 첫 번째 예는 부작용 경로(AOP, 정의는 표 2 참조)에 대한 적용 가능성 영역을 예측하기 위한 단백질 서열/구조 비교를 다루는 반면, 두 번째?...
Access restricted. Please log in or start a trial to view this content.
토론
독성 학적 관심의 화학 물질에 노출 될 수있는 살아있는 유기체의 게놈, 표현형, 생리 학적 및 행동 적 다양성을 포착하기에 충분한 종을 경험적으로 테스트하는 것이 불가능하다는 인식이 널리 퍼져 있습니다. SeqAPASS의 목표는 분자 수준 비교를 통해 테스트된 유기체에서 수백 또는 수천 종의 다른 종으로 화학 독성 데이터/지식을 외삽하는 데 도움이 되고 정보를 제공하기 위해 기존의 지속적으?...
Access restricted. Please log in or start a trial to view this content.
공개
저자는 공개 할 이해 상충이 없습니다.
감사의 말
저자는 Daniel L. Villeneuve 박사 (미국 EPA, 전산 독성학 및 노출 센터)와 Jon A. Doering 박사 (루이지애나 주립 대학 환경 과학과)에게 원고의 초기 초안에 대한 의견을 제공 한 것에 대해 감사드립니다. 이 작업은 미국 환경 보호국의 지원을 받았습니다. 이 논문에 표현 된 견해는 저자의 견해이며 반드시 미국 환경 보호국의 견해 나 정책을 반영하는 것은 아니며 상표명 또는 상업용 제품에 대한 언급이 연방 정부의 보증을 나타내는 것도 아닙니다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| Spreadsheet program | N/A | N/A | Any program that can be used to view and work with csv files (e.g. Microsoft Excel, OpenOffice Calc, Google Docs) can be used to access data export files. |
| Basic computing setup and internet access | N/A | N/A | SeqAPASS is a free, online tool that can be easily used via an internet connection. No software downloads are required. |
참고문헌
- Krewski, D., et al. Toxicity testing in the 21st century: a vision and a strategy. Journal of Toxicology and Environmental Health, Part B. 13 (2-4), 51-138 (2010).
- Wang, Z., Walker, G. W., Muir, D. C. G., Nagatani-Yoshida, K. Toward a global understanding of chemical pollution: A first comprehensive analysis of national and regional chemical inventories. Environmental Science & Technology. 54 (5), 2575-2584 (2020).
- Brooks, B. W., et al. Toxicology advances for 21st century chemical pollution. One Earth. 2 (4), 312-316 (2020).
- Kostal, J., Voutchkova-Kostal, A. Going all in: A strategic investment in in silico toxicology. Chemical Research in Toxicology. 33 (4), 880-888 (2020).
- Cheng, W., Doering, J. A., LaLone, C., Ng, C. Integrative computational approaches to inform relative bioaccumulation potential of per- and polyfluoroalkyl substances (PFAS) across species. Toxicology Sciences. 180 (2), 212-223 (2021).
- Kostich, M. S., Lazorchak, J. M. Risks to aquatic organisms posed by human pharmaceutical use. Science of the Total Environment. 389 (2-3), 329-339 (2008).
- Gunnarsson, L., Jauhiainen, A., Kristiansson, E., Nerman, O., Larsson, D. G. Evolutionary conservation of human drug targets in organisms used for environmental risk assessments. Environmental Science & Technology. 42 (15), 5807-5813 (2008).
- LaLone, C. A., et al. Evidence for cross species extrapolation of mammalian-based high-throughput screening assay results. Environmental Science & Technology. 52 (23), 13960-13971 (2018).
- LaLone, C. A., et al. Editor's highlight: Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS): A web-based tool for addressing the challenges of cross-species extrapolation of chemical toxicity. Toxicology Sciences. 153 (2), 228-245 (2016).
- Head, J. A., Hahn, M. E., Kennedy, S. W. Key amino acids in the aryl hydrocarbon receptor predict dioxin sensitivity in avian species. Environmental Science & Technology. 42 (19), 7535-7541 (2008).
- Bass, C., et al. Mutation of a nicotinic acetylcholine receptor β subunit is associated with resistance to neonicotinoid insecticides in the aphid Myzus persicae. BMC Neuroscience. 12, 51-51 (2011).
- Erdmanis, L., et al. Association of neonicotinoid insensitivity with a conserved residue in the loop d binding region of the tick nicotinic acetylcholine receptor. Biochemistry. 51 (23), 4627-4629 (2012).
- Doering, J. A., et al. et al. In silico site-directed mutagenesis informs species-specific predictions of chemical susceptibility derived from the Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS) tool. Toxicology Sciences. 166 (1), 131-145 (2018).
- Noyes, P. D., et al. Evaluating chemicals for thyroid disruption: Opportunities and challenges with in vitro testing and adverse outcome pathway approaches. Environmental Health Perspectives. 127 (9), 95001(2019).
- Park, G. Y., Jamerlan, A., Shim, K. H., An, S. S. A. Diagnostic and treatment approaches involving transthyretin in amyloidogenic diseases. Int J Mol Sci. 20 (12), 2982(2019).
- Rabah, S. A., Gowan, I. L., Pagnin, M., Osman, N., Richardson, S. J. Thyroid hormone distributor proteins during development in vertebrates. Front Endocrinol (Lausane). 10, 506(2019).
- Richardson, S. J. Cell and molecular biology of transthyretin and thyroid hormones. International Review of Cytology. 258, 137-193 (2007).
- Yamauchi, K., Ishihara, A. Transthyretin and Endocrine Disruptors. Recent Advances in Transthyretin Evolution, Structure and Biological Functions. Richardson, S. J., Cody, V. , Springer. Berlin Heidelberg, Germany. 159-171 (2009).
- Iakovleva, I., et al. Tetrabromobisphenol A is an efficient stabilizer of the transthyretin tetramer. PLoS One. 11 (4), 0153529(2016).
- Ishihara, A., Sawatsubashi, S., Yamauchi, K. Endocrine disrupting chemicals: Interference of thyroid hormone binding to transthyretins and to thyroid hormone receptors. Molecular and Cellular Endocrinology. 199 (1), 105-117 (2003).
- Kar, S., Sepúlveda, M. S., Roy, K., Leszczynski, J. Endocrine-disrupting activity of per- and polyfluoroalkyl substances: Exploring combined approaches of ligand and structure based modeling. Chemosphere. 184, 514-523 (2017).
- Morais-de-Sa, E., Pereira, P. J., Saraiva, M. J., Damas, A. M. The crystal structure of transthyretin in complex with diethylstilbestrol: A promising template for the design of amyloid inhibitors. Journal of Biological Chemistry. 279 (51), 53483-53490 (2004).
- Morgado, I., Campinho, M. A., Costa, R., Jacinto, R., Power, D. M. Disruption of the thyroid system by diethylstilbestrol and ioxynil in the sea bream (Sparus aurata). Aquatic Toxicology. 92 (4), 271-280 (2009).
- Yamauchi, K., Prapunpoj, P., Richardson, S. J. Effect of diethylstilbestrol on thyroid hormone binding to amphibian transthyretins. General and Comparative Endocrinology. 119 (3), 329-339 (2000).
- Zhang, J., et al. Structure-based virtual screening protocol for in silico identification of potential thyroid disrupting chemicals targeting transthyretin. Environmental Science & Technology. 50 (21), 11984-11993 (2016).
- Ren, X. M., et al. Binding interactions of perfluoroalkyl substances with thyroid hormone transport proteins and potential toxicological implications. Toxicology. 366-367, 32-42 (2016).
- Wilson, N., Mbabazi, K., Seth, P., Smith, H., Davis, N. L. Drug and opioid-involved overdose deaths - United States, 2017-2018. Morbidity and Mortality Weekly Report. 69 (11), 290-297 (2020).
- EPA. National Pollutant Discharge Elimination System (NPDES). United States Environmental Protection Agency. , Available from: https://www.epa.gov/npdes/npdes-resources (2018).
- Duvallet, C., Hayes, B. D., Erickson, T. B., Chai, P. R., Matus, M. Mapping community opioid exposure through wastewater-based epidemiology as a means to engage pharmacies in harm reduction efforts. Preventing Chronic Disease. 17, 200053(2020).
- Gushgari, A. J., Venkatesan, A. K., Chen, J., Steele, J. C., Halden, R. U. Long-term tracking of opioid consumption in two United States cities using wastewater-based epidemiology approach. Water Research. 161, 171-180 (2019).
- Lau, B., Bretaud, S., Huang, Y., Lin, E., Guo, S. Dissociation of food and opiate preference by a genetic mutation in zebrafish. Genes Brain Behave. 5 (7), 497-505 (2006).
- Bossé, G. D., Peterson, R. T. Development of an opioid self-administration assay to study drug seeking in zebrafish. Behavioural Brain Research. 335, 158-166 (2017).
- Mottaz, H., et al. Dose-dependent effects of morphine on lipopolysaccharide (LPS)-induced inflammation, and involvement of multixenobiotic resistance (MXR) transporters in LPS efflux in teleost fish. Environmental Pollution. 221, 105-115 (2017).
- Manglik, A., et al. Crystal structure of the µ-opioid receptor bound to a morphinan antagonist. Nature. 485 (7398), 321-326 (2012).
- Comer, S. D., Cahill, C. M. Fentanyl: Receptor pharmacology, abuse potential, and implications for treatment. Neuroscience & Biobehavioral Reviews. 106, 49-57 (2019).
- Podlewska, S., Bugno, R., Kudla, L., Bojarski, A. J., Przewlocki, R. Molecular modeling of µ opioid receptor ligands with various functional properties: PZM21, SR-17018, morphine, and fentanyl-simulated interaction patterns confronted with experimental data. Molecules. 25 (20), 4636(2020).
- Huang, W., et al. Structural insights into µ-opioid receptor activation. Nature. 524 (7565), 315-321 (2015).
- Lipiński, P. F. J., et al. Fentanyl family at the mu-opioid receptor: Uniform assessment of binding and computational analysis. Molecules. 24 (4), 740(2019).
- Boland, L. A., Angles, J. M. Feline permethrin toxicity: Retrospective study of 42 cases. Journal of Feline Medicine and Surgery. 12 (2), 61-71 (2010).
- Stevenson, B. J., Pignatelli, P., Nikou, D., Paine, M. J. Pinpointing P450s associated with pyrethroid metabolism in the dengue vector, Aedes aegypti: developing new tools to combat insecticide resistance. PLoS Neglected Tropical Diseases. 6 (3), 1595(2012).
- Ankley, G. T., Gray, L. E. Cross-species conservation of endocrine pathways: A critical analysis of tier 1 fish and rat screening assays with 12 model chemicals. Environmental Toxicology and Chemistry. 32 (5), 1084-1087 (2013).
- Meteyer, C. U., Rideout, B. A., Gilbert, M., Shivaprasad, H. L., Oaks, J. L. Pathology and proposed pathophysiology of diclofenac poisoning in free-living and experimentally exposed oriental white-backed vultures (Gyps bengalensis). Journal of Wildlife Diseases. 41 (4), 707-716 (2005).
- EPA. ECOTOX User Guide: ECOTOXicology Knowledgebase System. EPA, United States Environmental Protection Agency. , Available from: https://cfpub.epa.gov/ecotox/index.cfm (2021).
- ECOS Environmental Conservation Online System. U.S. Fish & Wildlife Service. , Available from: https://ecos.fws.gov/ecp/ (2021).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유