Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Démonstration de l’alignement des séquences pour prédire l’outil de sensibilité entre espèces pour une évaluation rapide de la conservation des protéines
Dans cet article
Résumé
Ici, nous présentons un protocole pour utiliser la dernière version de l’outil SeqAPASS (Sequence Alignment to Predict Across Species Susceptibility) de l’Environmental Protection Agency des États-Unis. Ce protocole démontre l’application de l’outil en ligne pour analyser rapidement la conservation des protéines et fournir des prédictions personnalisables et facilement interprétables de la sensibilité chimique entre les espèces.
Résumé
L’outil SeqAPASS (Sequence Alignment to Predict Across Species Susceptibility) de l’Environmental Protection Agency des États-Unis est une application de dépistage en ligne rapide et gratuite qui permet aux chercheurs et aux organismes de réglementation d’extrapoler les informations sur la toxicité entre les espèces. Pour les cibles biologiques dans des systèmes modèles tels que les cellules humaines, les souris, les rats et les poissons-zèbres, des données sur la toxicité sont disponibles pour une variété de produits chimiques. Grâce à l’évaluation de la conservation des cibles protéiques, cet outil peut être utilisé pour extrapoler les données générées par ces systèmes modèles à des milliers d’autres espèces dépourvues de données de toxicité, ce qui donne des prédictions de la susceptibilité chimique intrinsèque relative. Les dernières versions de l’outil (versions 2.0-6.1) ont incorporé de nouvelles fonctionnalités qui permettent la synthèse, l’interprétation et l’utilisation rapides des données pour la publication et des graphiques de qualité présentation.
Parmi ces fonctionnalités figurent des visualisations de données personnalisables et un rapport de synthèse complet conçu pour résumer les données SeqAPASS pour faciliter l’interprétation. Cet article décrit le protocole pour guider les utilisateurs dans la soumission des tâches, la navigation dans les différents niveaux de comparaison des séquences de protéines, et l’interprétation et l’affichage des données résultantes. Les nouvelles fonctionnalités de SeqAPASS v2.0-6.0 sont mises en évidence. En outre, deux cas d’utilisation axés sur la conservation de la transthyrétine et des protéines des récepteurs opioïdes à l’aide de cet outil sont décrits. Enfin, les forces et les limites de SeqAPASS sont discutées pour définir le domaine d’applicabilité de l’outil et mettre en évidence différentes applications pour l’extrapolation inter-espèces.
Introduction
Traditionnellement, le domaine de la toxicologie s’est fortement appuyé sur l’utilisation d’essais sur des animaux entiers pour fournir les données nécessaires aux évaluations de la sécurité chimique. Ces méthodes sont généralement coûteuses et gourmandes en ressources. Toutefois, en raison du grand nombre de produits chimiques actuellement utilisés et du rythme rapide auquel de nouveaux produits chimiques sont mis au point, il est reconnu à l’échelle mondiale qu’il existe un besoin de méthodes plus efficaces de dépistage des produits chimiques 1,2. Ce besoin et le changement de paradigme qui en a résulté pour s’éloigner de l’expérimentation animale ont conduit au développement de nombreuses nouvelles méthodes d’approche, y compris les tests de criblage à haut débit, la transcriptomique à haut débit, le séquençage de nouvelle génération et la modélisation informatique, qui sont des stratégies de test alternativesprometteuses 3,4.
L’évaluation de la sécurité chimique dans la diversité des espèces potentiellement touchées par les expositions chimiques a été un défi permanent, non seulement avec les tests de toxicité traditionnels, mais aussi avec les nouvelles méthodes d’approche. Les progrès de la toxicologie comparative et prédictive ont fourni des cadres pour comprendre la sensibilité relative de différentes espèces, et les progrès technologiques dans les méthodes de calcul continuent d’accroître l’applicabilité de ces méthodes. Plusieurs stratégies ont été discutées au cours de la dernière décennie qui tirent parti des bases de données existantes sur les séquences de gènes et de protéines, ainsi que de la connaissance de cibles moléculaires chimiques spécifiques, pour soutenir les approches prédictives pour l’extrapolation interspécifique et améliorer les évaluations de l’innocuité chimique au-delà des organismes modèles typiques 5,6,7,8.
Pour faire progresser la science dans l’action, s’appuyer sur ces études fondamentales en toxicologie prédictive, prioriser les efforts d’essais chimiques et soutenir la prise de décision, l’outil SeqAPASS (Sequence Alignment to Predict Across Species Susceptibility) de l’Environmental Protection Agency des États-Unis a été créé. Cet outil est une application Web publique et gratuite qui utilise des dépôts publics d’informations sur les séquences de protéines en constante expansion pour prédire la susceptibilité chimique à travers la diversité des espèces9. Basé sur le principe selon lequel la susceptibilité intrinsèque relative d’une espèce à un produit chimique particulier peut être déterminée en évaluant la conservation des cibles protéiques connues de ce produit chimique, cet outil compare rapidement les séquences d’acides aminés protéiques d’une espèce ayant une sensibilité connue à toutes les espèces avec des données de séquence protéique existantes. Cette évaluation est complétée par trois niveaux d’analyse, y compris (1) la séquence d’acides aminés primaires, (2) le domaine fonctionnel et (3) les comparaisons de résidus d’acides aminés critiques, chacune nécessitant une connaissance plus approfondie de l’interaction chimique-protéine et fournissant une plus grande résolution taxonomique dans la prédiction de la susceptibilité. L’une des principales forces de SeqAPASS est que les utilisateurs peuvent personnaliser et affiner leur évaluation en ajoutant des sources de données supplémentaires pour la conservation des cibles en fonction de la quantité d’informations disponibles concernant l’interaction chimique-protéine ou protéine-protéine d’intérêt.
La première version a été publiée en 2016, ce qui a permis aux utilisateurs d’évaluer les séquences d’acides aminés primaires et les domaines fonctionnels de manière rationalisée pour prédire la sensibilité chimique et contenait des capacités minimales de visualisation des données (tableau 1). Il a été démontré que les différences individuelles d’acides aminés sont des déterminants importants des différences entre les espèces dans les interactions chimique-protéine, ce qui peut affecter la susceptibilité chimiquedes espèces 10,11,12. Par conséquent, des versions ultérieures ont été développées pour prendre en compte les acides aminés critiques qui sont importants pour l’interaction chimique directe13. En réponse aux commentaires des intervenants et des utilisateurs, cet outil a fait l’objet d’une version annuelle avec de nouvelles fonctionnalités supplémentaires conçues pour répondre aux besoins des chercheurs et des organismes de réglementation en matière de résolution des problèmes liés à l’extrapolation interspécifique (tableau 1). Le lancement de la version 5.0 de SeqAPASS en 2020 a apporté des fonctionnalités centrées sur l’utilisateur qui intègrent des options de visualisation et de synthèse des données, des liens externes, des options de tableau récapitulatif et de rapport, ainsi que des fonctionnalités graphiques. Dans l’ensemble, les nouveaux attributs et capacités de cette version ont amélioré la synthèse des données, l’interopérabilité entre les bases de données externes et la facilité d’interprétation des données pour les prédictions de la sensibilité interspécifique.
Protocole
1. Mise en route
REMARQUE: Le protocole présenté ici est axé sur l’utilitaire de l’outil et les fonctionnalités clés. Des descriptions détaillées des méthodes, des caractéristiques et des composants sont disponibles sur le site Web dans un guide de l’utilisateur complet (tableau 1).
Tableau 1 : Évolution de l’outil SeqAPASS. Liste des fonctionnalités et mises à jour ajoutées à l’outil SeqAPASS depuis son déploiement initial. Abréviations : SeqAPASS = alignement des séquences pour prédire la sensibilité de l’espèce à l’ensemble des espèces; ECOTOX = base de connaissances ECOTOXicology. Veuillez cliquer ici pour télécharger ce tableau.
- Accédez à https://seqapass.epa.gov/seqapass à l’aide de Chrome. Sélectionnez Connexion pour utiliser un compte existant ou suivez les instructions pour créer un compte SeqAPASS, ce qui permettra aux utilisateurs d’exécuter, de stocker, d’accéder et de personnaliser leurs tâches terminées.
- Avant d’effectuer une analyse, identifiez d’abord une protéine d’intérêt et une espèce ciblée ou sensible en examinant la littérature existante ou les données préexistantes (figure 1). Comme SeqAPASS contient des liens vers des ressources externes pour aider à identifier la protéine de requête, cliquez sur les boutons déroulants sous Identifier une cible protéique pour accéder aux ressources pertinentes.
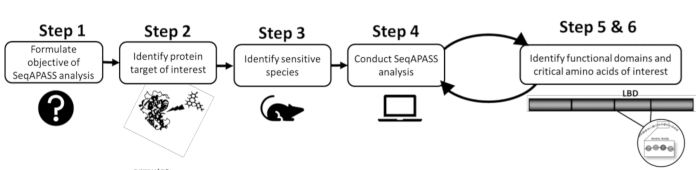
Figure 1: Formulation du problème SeqAPASS: schéma des informations préliminaires nécessaires à une analyse réussie. Abréviations : SeqAPASS = alignement des séquences pour prédire la sensibilité de l’espèce à l’ensemble des espèces; LBD = domaine de liaison au ligand. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Interopérabilité SeqAPASS entre les bases de données. Diagramme schématique des outils, bases de données et ressources externes intégrés dans SeqAPASS. Abréviations : SeqAPASS = alignement des séquences pour prédire la sensibilité de l’espèce à l’ensemble des espèces; POA = voie de résultat indésirable; NCBI = Centre national d’information sur la biotechnologie; ECOTOX = base de connaissances ECOTOXicology. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 2 : Liens, ressources et outils intégrés dans l’outil SeqAPASS. Une liste des différentes sources de données, liens et ressources exploités dans l’outil SeqAPASS. Abréviation : SeqAPASS = alignement de séquence pour prédire la sensibilité de l’espèce. Veuillez cliquer ici pour télécharger ce tableau.
2. Développement et exécution d’une requête SeqAPASS : Niveau 1
REMARQUE: Dans une analyse de niveau 1, toute la séquence d’acides aminés primaires d’une protéine de requête est comparée aux séquences d’acides aminés primaires de toutes les espèces avec des informations de séquence disponibles. Cet outil utilise des algorithmes pour exploiter, collecter et compiler des données accessibles au public afin d’aligner et de comparer rapidement les séquences d’acides aminés entre les espèces. Le backend stocke des informations provenant des bases de données du National Center for Biotechnology Information (NCBI) et utilise stratégiquement les versions autonomes de l’outil de recherche d’alignement local de base des protéines (BLASTp)54 et de l’outil d’alignement multiple basé sur les contraintes (COBALT)55.
- Sous Comparer les séquences d’acides aminés primaires, cliquez sur Par espèce ou Par accession. Utilisez la sélection Par espèce pour taper ou sélectionner dans une liste d’espèces pour choisir la protéine cible d’intérêt.
- Soumettez les accessions de protéines (c.-à-d. ID de protéine NCBI) directement en entrant l'(les ) accession(s) dans la zone de texte Par accession .
- Sélectionnez Exécuter la demande pour envoyer la requête. Une fois soumis, attendez qu’une notification apparaisse dans le coin supérieur droit de la fenêtre du navigateur indiquant une soumission réussie.
- Sélectionnez l’onglet État d’exécution SeqAPASS en haut de la page pour afficher une liste de toutes les exécutions SeqAPASS effectuées sous ce compte d’utilisateur et vérifier le pourcentage d’achèvement.
- Cliquez sur Actualiser les données lorsque la case d’option appropriée est sélectionnée pour vérifier l’état des exécutions de niveau 2 et de niveau 3.
- Sélectionnez l’onglet Afficher les rapports SeqAPASS en haut de la page pour accéder à la liste de tous les rapports complétés sous ce compte.
- Dans l’onglet Afficher les rapports SeqAPASS , sélectionnez la protéine de requête qui vous intéresse. Cliquez sur Demander un rapport sélectionné pour ouvrir la page Informations sur les protéines de requête de niveau 1 et afficher les résultats, les options de personnalisation des données, les visualisations et les rapports récapitulatifs.
- Par défaut, sélectionnez Afficher le rapport pour afficher les données dans le navigateur Web. Vous pouvez également sélectionner Enregistrer le rapport pour télécharger les données brutes sous forme de fichier .zip.
REMARQUE : Le temps requis pour une analyse de niveau 1 varie (moyenne de 23 minutes pour la version 5.1) en fonction de la demande globale de l’utilisateur à ce moment-là, du nombre de tâches soumises à la file d’attente et de la quantité d’informations de protéines qui existent pour une tâche soumise. Si une cible protéique a déjà été complétée, les données seront disponibles en quelques secondes après la soumission.
3. Développement et exécution d’une requête SeqAPASS : Niveau 2
REMARQUE : Comme la séquence protéique entière n’est pas directement impliquée dans une interaction chimique, une analyse de niveau 2 compare uniquement la séquence d’acides aminés du domaine fonctionnel pour faire des prédictions de sensibilité aux rangs taxonomiques inférieurs (p. ex., classe, ordre, famille).
- Dans la page Informations sur la protéine de requête de niveau 1, cliquez sur le signe plus + en regard de l’en-tête de niveau 2 pour remplir le menu Requête de niveau 2 .
- Identifiez le(s) domaine(s) approprié(s) dans la protéine d’intérêt (protéine de requête).
- Si aucun domaine n’a été identifié, cliquez sur le lien intégré vers la base de données des domaines conservés (CDD) du NCBI (Tableau 1), ce qui peut faciliter l’identification de la sélection de domaine appropriée.
REMARQUE : En règle générale, seuls des domaines d’accès spécifiques sont sélectionnés comme requêtes au niveau 2.
- Si aucun domaine n’a été identifié, cliquez sur le lien intégré vers la base de données des domaines conservés (CDD) du NCBI (Tableau 1), ce qui peut faciliter l’identification de la sélection de domaine appropriée.
- Cliquez sur la zone Sélectionner un domaine pour remplir automatiquement une liste de domaines fonctionnels pour la protéine de requête.
- Sélectionnez la ou les accessions de domaine dans la liste déroulante et lancez la requête de niveau 2 en cliquant sur le bouton Demander l’exécution du domaine . Une fois soumis, attendez qu’une notification apparaisse indiquant une soumission réussie.
- Cliquez sur Actualiser les exécutions de niveau 2 et 3 pour remplir les données de niveau 2, qui seront disponibles dans les secondes suivant la soumission.
- Sous Afficher les données de niveau 2, sélectionnez l’acquisition de domaine terminée dans la liste déroulante et cliquez sur le bouton Afficher les données de niveau 2 pour ouvrir les résultats dans une nouvelle page.
4. Accès et compréhension des données : SeqAPASS Niveau 1 et Niveau 2
- Faites défiler jusqu’au bas de la page Interroger les informations sur les protéines pour afficher un rapport des résultats - un rapport principal est fourni avec des analyses de niveaux 1 et 2 par défaut. Sélectionnez la case d’option Rapport complet pour afficher un rapport plus détaillé qui fournit tous les accès de séquence et les mesures d’alignement. Cliquez sur l’accession/ID /nom approprié dans les deux rapports pour accéder aux informations transparentes sur l’alignement des protéines et la taxonomie dans la base de données NCBI.
- Faites défiler vers la droite du tableau des résultats pour afficher la colonne ECOTOX . Cliquez sur les liens vers la base de connaissances ECOTOXicology (ECOTOX) pour recueillir rapidement les données de toxicité correspondantes pour les espèces dont la sensibilité est prédictée.
NOTE: ECOTOX est une base de connaissances complète et accessible au public fournissant des données uniques sur la toxicité chimique pour les plantes aquatiques et terrestres et la faune. SeqAPASS v6.0 inclut un widget ECOTOX pour se connecter plus rapidement aux données ECOTOX pertinentes par produit chimique et espèce d’intérêt. - Cliquez sur Télécharger le tableau pour enregistrer le tableau en tant que fichier de feuille de calcul. Cliquez sur le bouton Afficher le rapport récapitulatif pour afficher et télécharger un tableau de rapport récapitulatif présentant les données triées par groupe taxonomique.
REMARQUE : Des tableaux sommaires des données sont disponibles pour les rapports principal et complet et donnent un aperçu des prévisions pour une cible donnée.
5. Manipulation des paramètres de données : SeqAPASS Niveau 1 et Niveau 2
REMARQUE : Dans les analyses de niveau 1 et de niveau 2, on suppose que plus la similitude protéique est grande, plus il est probable qu’un produit chimique interagisse avec la protéine de la même manière que l’espèce ou la protéine interrogée, ce qui les rend vulnérables aux impacts potentiels des produits chimiques avec cette cible moléculaire. En raison de la similitude de ces données, les étapes de compréhension des données de niveaux 1 et 2 sont décrites ensemble dans un seul protocole.
- Reportez-vous aux sous-menus situés en haut de la section Interroger les informations sur les protéines pour accéder aux paramètres du rapport et les manipuler, et utilisez les paramètres par défaut pour toutes les options de rapport pour la plupart des analyses. S’il existe une justification scientifique pour modifier le paramètre par défaut, procédez comme suit :
- (FACULTATIF) Cliquez sur le signe plus + en regard de Seuil de sensibilité pour afficher et ajuster les paramètres de seuil de susceptibilité dans un nouvel onglet. Sélectionnez une nouvelle valeur seuil dans une liste déroulante ou entrez une valeur seuil définie par l’utilisateur.
- (FACULTATIF) Modifiez le nombre dans le champ Valeur E (le nombre d’alignements différents attendus par hasard) si quelque chose d’autre que la valeur par défaut est souhaité.
REMARQUE: Toute protéine avec une valeur E supérieure au nombre dans la boîte sera éliminée du rapport primaire. - (FACULTATIF) Utilisez l’option Trier par groupe taxonomique pour choisir le niveau de hiérarchie taxonomique à afficher dans la colonne Groupe taxonomique filtré du tableau des résultats.
REMARQUE : La modification de la hiérarchie taxonomique modifiera également la prédiction de la sensibilité en fonction des espèces de chaque groupe filtré qui se trouvent au-dessus du seuil. - (FACULTATIF) Modifiez le champ Domaine commun (combien de domaines communs une protéine doit partager avec la protéine de requête à inclure dans les résultats) si quelque chose d’autre que la valeur par défaut est souhaité.
Remarque : Comme le paramètre par défaut est 1, toute séquence qui ne partage pas au moins un domaine commun avec la protéine de requête sera exclue. - (FACULTATIF) Sélectionnez Non sous Lecture croisée des espèces pour récupérer les prédictions de sensibilité de Y uniquement si le pourcentage de similarité est supérieur ou égal au seuil ou si le résultat positif est identifié comme un candidat orthologue.
REMARQUE : Ce paramètre par défaut est Oui, ce qui signifie qu’une prédiction de sensibilité de Y sera signalée pour tous les candidats orthologues, toutes les espèces énumérées au-dessus du seuil de sensibilité et toutes les espèces en dessous du seuil du même groupe taxonomique avec une ou plusieurs espèces au-dessus du seuil.
- Cliquez sur le bouton Télécharger les paramètres actuels du rapport pour télécharger un fichier capturant les paramètres actuels appliqués.
REMARQUE : Le niveau d’évaluation spécifique (1, 2 ou 3) sélectionné dictera les paramètres présentés dans le rapport.
6. Visualisation des données : SeqAPASS Niveau 1 et Niveau 2
- Cliquez sur le signe plus + en regard de Visualisation et cliquez sur le bouton Visualiser les données pour ouvrir un onglet séparé affichant les informations définies par l’utilisateur et la possibilité de sélectionner un graphique interactif des résultats.
- Cliquez sur Boxplot pour ouvrir les contrôles interactifs boxplot et plot et permettre à la visualisation boxplot de se mettre à jour activement pour refléter les modifications apportées au tableau de données et fournir des graphiques de qualité publication et présentation.
Remarque : Le boxplot par défaut affiche les groupes d’espèces sur l’axe des x et le pourcentage de similarité sur l’axe y. Les diagrammes en boîte affichent le seuil de sensibilité (ligne pointillée), le pourcentage de similitude entre les espèces par rapport aux espèces interrogées, et les valeurs moyennes et médianes pour chaque groupe taxonomique ainsi que les 25e et 75e percentiles et l’intervalle interquartile. En fonction de l’objectif de l’analyse et des besoins de l’utilisateur, de nombreuses fonctionnalités du boxplot peuvent être modifiées par les étapes facultatives suivantes.- (FACULTATIF) Pour personnaliser les groupes taxonomiques affichés, reportez-vous à la zone Groupes taxonomiques de la section Contrôles . Supprimez des groupes en faisant défiler les noms et en sélectionnant x ou en utilisant le menu déroulant Groupes taxonomiques .
- (FACULTATIF) Pour ajouter une légende qui identifiera une espèce d’intérêt ou des groupes prédéfinis spécifiques (par exemple, des espèces en voie de disparition ou menacées), passez la souris sur un nom de groupe taxonomique sur l’axe des x pour activer une boîte contextuelle répertoriant les trois principales espèces classées par pourcentage de similitude le plus élevé. Survolez les espèces dans la légende pour générer une boîte contextuelle contenant les informations sur les espèces correspondantes. Cliquez sur la case correspondant à un groupe taxonomique spécifique pour générer un tableau récapitulatif téléchargeable répertoriant les espèces et les prévisions.
- Cliquez sur Télécharger Boxplot pour choisir un type de fichier, personnaliser la résolution largeur/hauteur et enregistrer la visualisation.
7. Élaboration et exécution d’une analyse SeqAPASS : Niveau 3
REMARQUE : Une analyse de niveau 3 évalue les résidus d’acides aminés identifiés par l’utilisateur dans la protéine de requête et compare rapidement la conservation de ces résidus entre les espèces. On présume que les espèces dans lesquelles ces résidus sont conservés sont plus susceptibles d’interagir avec un produit chimique d’une manière similaire à l’espèce/protéine modèle. Comme le niveau 3 se concentre sur les acides aminés individuels, une analyse ne peut être effectuée que lorsque des connaissances détaillées sur les résidus d’acides aminés essentiels à l’interaction chimique-protéine ou protéine-protéine sont disponibles.
- Cliquez sur le signe plus + en regard de l’en-tête de niveau 3 de la page Informations sur les protéines de requête de niveau 1 pour remplir le menu Requête de niveau 3.
- Cliquez sur le signe plus + en regard de l’Explorateur de références pour ouvrir l’outil Explorateur de références, qui génère une chaîne booléenne prédéfinie pour interroger la documentation disponible et aide les utilisateurs à identifier la littérature appropriée pour appuyer l’identification des acides aminés critiques à utiliser dans l’évaluation de niveau 3 (tableau 2 et figure 2).
- (FACULTATIF) Une fois la protéine de requête remplie automatiquement, utilisez la fonction Ajouter un nom de protéine pour ajouter des protéines supplémentaires.
- Cliquez sur le lien Générer Google Scholar pour ouvrir une fenêtre contextuelle contenant une chaîne de recherche générée automatiquement qui inclut les termes de recherche pertinents.
- Cliquez sur Rechercher dans Google Scholar pour interroger la base de données de littérature à l’aide de la chaîne de recherche.
- Vous pouvez également cliquer sur Copier dans le Presse-papiers et personnaliser la chaîne de recherche en ajoutant ou en supprimant des termes à l’aide des fonctions de l’Explorateur de références.
8. Identifier les résidus d’acides aminés critiques à l’aide de la littérature identifiée
- Sélectionnez la séquence de modèles à laquelle les espèces sélectionnées par l’utilisateur seront alignées dans le menu Requête de niveau 3.
REMARQUE : Cette séquence modèle est généralement choisie en fonction de la littérature pour laquelle les acides aminés critiques ont été identifiés et peut être la même espèce ou une espèce différente de celles interrogées aux niveaux 1 et 2.- (FACULTATIF) Utilisez la zone Comparaisons supplémentaires pour comparer les accessions/séquences qui n’apparaissent pas dans les tableaux du rapport principal/complet .
- Entrez un nom défini par l’utilisateur pour l’exécution de niveau 3 dans la zone de texte Entrer le nom de l’exécution de niveau 3 pour identifier l’exécution de niveau 3 terminée. Choisissez un nom unique pour chaque évaluation.
- Sélectionnez le groupe taxonomique qui vous intéresse dans le champ Choisir le(s) groupe(s ) taxonomique(s). Sélectionnez un groupe taxonomique pour filtrer automatiquement la table en fonction de ce groupe taxonomique.
- Dans le tableau des résultats, cochez manuellement la case en regard de n’importe quelle espèce à aligner sur la séquence de modèles.
REMARQUE : Pour assurer un alignement approprié, un groupe taxonomique à la fois doit être comparé au modèle. Ne sélectionnez que des protéines annotées de manière similaire pour l’espèce d’intérêt. Lors de la sélection des séquences à comparer, il est important de prêter attention à certaines séquences (par exemple, hypothétiques, de MAUVAISE QUALITÉ ou partielles). À moins qu’il n’y ait une justification transparente de l’inclusion, il est préférable d’exclure ces séquences car elles peuvent fausser les prédictions en raison d’informations de séquence incomplètes ou inappropriées. - Répétez les étapes pour aligner tous les groupes taxonomiques d’intérêt.
- Cliquez sur Actualiser les exécutions de niveau 2 et 3 une fois que toutes les espèces ont été alignées pour remplir le menu Sélectionner le nom de la course de niveau 3 avec les tâches de niveau 3 terminées et obtenir immédiatement les données d’un alignement de niveau 3.
- Cliquez sur Combiner les données de niveau 3 pour combiner les alignements de plusieurs groupes taxonomiques.
- Pour afficher un seul rapport, vous pouvez également sélectionner le nom défini par l’utilisateur sous Choisir la requête à afficher et cliquer sur Afficher les données de niveau 3.
- Sélectionnez le modèle de niveau 3 à utiliser comme base pour la comparaison des résidus d’acides aminés dans le menu Combiner les rapports de niveau 3 et cliquez sur Suivant.
- Dans les tâches de niveau 3, sélectionnez les tâches terminées pour la comparaison et cliquez sur Suivant. Utilisez la fonction Order Level 3 Jobs pour réorganiser les groupes taxonomiques si vous le souhaitez. Cliquez sur Afficher les données de niveau 3 pour produire une page de rapport de niveau 3 avec les groupes taxonomiques combinés alignés.
- Sélectionnez les positions des acides aminés précédemment identifiées pour l’espèce modèle en tapant la ou les positions des acides aminés, séparées par des virgules, dans la zone Entrer les positions des résidus d’acides aminés , puis en sélectionnant Copier dans la liste des résidus. Sélectionnez directement les résidus dans la séquence du modèle à partir de la boîte de navette.
- Cliquez sur Mettre à jour le rapport pour actualiser la page et afficher les prévisions de susceptibilité de niveau 3.
REMARQUE : Le niveau 3 utilise un ensemble simple de règles dérivées des descripteurs de base des propriétés fonctionnelles de la chaîne latérale (p. ex., aliphatique, aromatique) et des dimensions moléculaires (différences de poids moléculaire >30 g/mol) pour déterminer si les différences à des positions clés sont susceptibles d’affecter les interactions protéiques13.
9. Visualisation des données SeqAPASS de niveau 3
REMARQUE : Comme dans les niveaux précédents, les rapports principal et complet sont disponibles. En plus des données identiques aux données des niveaux 1 et 2, le rapport principal affiche les positions des acides aminés, les abréviations et une susceptibilité oui/non (O/N) similaire à celle du modèle de prédiction. De même, le rapport complet contient des informations sur la classification de la chaîne latérale des acides aminés et le poids moléculaire.
- Sur la page Rapport de niveau 3, faites défiler vers le bas pour afficher un rapport des résultats. Cliquez sur Télécharger le tableau en bas du rapport pour enregistrer le tableau .
- Cliquez sur Afficher le rapport récapitulatif de niveau 3 pour afficher et télécharger un tableau de rapport récapitulatif présentant les données triées par groupe taxonomique. Cliquez sur le signe plus + en regard de Visualisation sur la page Rapport de niveau 3 pour ouvrir un onglet de navigateur distinct affichant les informations définies par l’utilisateur et la possibilité d’afficher les résultats sous la forme d’une carte thermique interactive.
- Cliquez sur Carte thermique sur la page Informations sur la visualisation pour ouvrir le graphique interactif et les contrôles et permettre à la visualisation de la carte thermique de se mettre à jour activement pour refléter les modifications apportées au tableau de données. Effectuez les étapes facultatives suivantes pour personnaliser la carte thermique.
- (FACULTATIF) Sélectionnez Options de rapport pour basculer entre un rapport simple, qui affiche la position des acides aminés, l’abréviation d’une lettre et la similitude des acides aminés, ou un rapport complet, qui affiche des informations détaillées sur chaque acide aminé sélectionné.
- (FACULTATIF) Sélectionnez Options de rapport pour modifier la façon dont les espèces sont affichées, soit par nom commun , soit par nom scientifique.
REMARQUE : Dans le rapport simple, les acides aminés sont classés comme une correspondance totale (bleu foncé), une correspondance partielle (bleu clair, substitutions ne répondant qu’à un seul critère) ou une correspondance non (jaune, substitutions ne répondant à aucun des critères) à l’acide aminé modèle. Le rapport complet affiche les comparaisons sous la forme d’une correspondance totale (bleu foncé) ou d’une correspondance non conforme (jaune). - (FACULTATIF) Sélectionnez Sélections facultatives pour mettre en évidence des informations utiles telles que les candidats orthologues, les espèces menacées, les espèces en voie de disparition ou les organismes modèles communs.
- (FACULTATIF) Sélectionnez Paramètres de carte thermique pour sélectionner des options de personnalisation supplémentaires, notamment l’ajout ou la suppression de colonnes, de légendes et de texte.
- Cliquez sur Télécharger Boxplot pour choisir un type de fichier et enregistrer la visualisation.
10. Interprétation des résultats de SeqAPASS : Sources de données pour la conservation des protéines
REMARQUE : Pour faciliter l’interprétation, cet outil comprend un rapport sommaire de décision (rapport DS) conçu pour intégrer les données entre les niveaux. Le rapport DS contient les résultats (c.-à-d. tableaux de données et/ou visualisations) que l’utilisateur a sélectionnés et permet d’évaluer rapidement les prévisions de sensibilité à plusieurs niveaux pour plusieurs espèces simultanément.
- Cliquez sur Push Level # to DS Report dans les pages de résultats ou de visualisation des données et attendez que les données soient « poussées » et que l’onglet DS Report devienne actif.
REMARQUE : Si les résultats ou les modifications n’ont pas été transmis au rapport DS, l’option Niveau de diffusion # vers le rapport DS restera active jusqu’à ce qu’elle soit sélectionnée. Si un paramètre a été modifié, le texte Cliquez pour pousser les nouvelles modifications s’affiche jusqu’à ce que les modifications soient transmises au rapport. Les visualisations peuvent être envoyées au rapport DS à tout moment au cours de l’évaluation. - Sélectionnez l’onglet Rapport DS à tout moment pour accéder à la page DS .
REMARQUE : Pour toutes les espèces alignées au niveau 1, le tableau du Rapport sommaire de décision finale contient les données importantes et les prévisions de sensibilité pour chaque analyse. Si une espèce figurant dans le tableau DS n’a pas été incluse dans le rapport de niveau 3, mais qu’elle a occupé des emplois de niveau 1 et/ou de niveau 2, la cellule du tableau recevra une désignation sans objet (NA) pour la prédiction de sensibilité de niveau 3.
Résultats
Pour démontrer l’application de l’outil SeqAPASS et mettre en évidence de nouvelles fonctionnalités, deux études de cas sont décrites représentant des cas dans lesquels la conservation des protéines prédit qu’il existe des différences de sensibilité chimique entre les espèces (transthyrétine humaine) et qu’il n’y a pas de différences (μ récepteur opioïde [MOR]). Le premier de ces exemples porte sur les comparaisons de séquences protéiques et de structures pour prédire le domaine d’applicabi...
Discussion
Il est largement reconnu qu’il n’est pas possible de tester empiriquement suffisamment d’espèces pour saisir la diversité génomique, phénotypique, physiologique et comportementale des organismes vivants susceptibles d’être exposés à des produits chimiques d’intérêt toxicologique. L’objectif de SeqAPASS est de maximiser l’utilisation des données existantes et en expansion continue de la séquence protéique et des données structurelles pour aider et informer l’extrapolation des données / connai...
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Les auteurs remercient le Dr Daniel L. Villeneuve (U.S. EPA, Center for Computational Toxicology and Exposure) et le Dr Jon A. Doering (Department of Environmental Sciences, Louisiana State University) d’avoir fourni des commentaires sur une version antérieure du manuscrit. Ce travail a été soutenu par l’Environmental Protection Agency des États-Unis. Les opinions exprimées dans le présent document sont celles des auteurs et ne reflètent pas nécessairement les opinions ou les politiques de l’Environmental Protection Agency des États-Unis, et la mention de noms commerciaux ou de produits commerciaux n’indique pas non plus l’approbation du gouvernement fédéral.
matériels
| Name | Company | Catalog Number | Comments |
| Spreadsheet program | N/A | N/A | Any program that can be used to view and work with csv files (e.g. Microsoft Excel, OpenOffice Calc, Google Docs) can be used to access data export files. |
| Basic computing setup and internet access | N/A | N/A | SeqAPASS is a free, online tool that can be easily used via an internet connection. No software downloads are required. |
Références
- Krewski, D., et al. Toxicity testing in the 21st century: a vision and a strategy. Journal of Toxicology and Environmental Health, Part B. 13 (2-4), 51-138 (2010).
- Wang, Z., Walker, G. W., Muir, D. C. G., Nagatani-Yoshida, K. Toward a global understanding of chemical pollution: A first comprehensive analysis of national and regional chemical inventories. Environmental Science & Technology. 54 (5), 2575-2584 (2020).
- Brooks, B. W., et al. Toxicology advances for 21st century chemical pollution. One Earth. 2 (4), 312-316 (2020).
- Kostal, J., Voutchkova-Kostal, A. Going all in: A strategic investment in in silico toxicology. Chemical Research in Toxicology. 33 (4), 880-888 (2020).
- Cheng, W., Doering, J. A., LaLone, C., Ng, C. Integrative computational approaches to inform relative bioaccumulation potential of per- and polyfluoroalkyl substances (PFAS) across species. Toxicology Sciences. 180 (2), 212-223 (2021).
- Kostich, M. S., Lazorchak, J. M. Risks to aquatic organisms posed by human pharmaceutical use. Science of the Total Environment. 389 (2-3), 329-339 (2008).
- Gunnarsson, L., Jauhiainen, A., Kristiansson, E., Nerman, O., Larsson, D. G. Evolutionary conservation of human drug targets in organisms used for environmental risk assessments. Environmental Science & Technology. 42 (15), 5807-5813 (2008).
- LaLone, C. A., et al. Evidence for cross species extrapolation of mammalian-based high-throughput screening assay results. Environmental Science & Technology. 52 (23), 13960-13971 (2018).
- LaLone, C. A., et al. Editor's highlight: Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS): A web-based tool for addressing the challenges of cross-species extrapolation of chemical toxicity. Toxicology Sciences. 153 (2), 228-245 (2016).
- Head, J. A., Hahn, M. E., Kennedy, S. W. Key amino acids in the aryl hydrocarbon receptor predict dioxin sensitivity in avian species. Environmental Science & Technology. 42 (19), 7535-7541 (2008).
- Bass, C., et al. Mutation of a nicotinic acetylcholine receptor β subunit is associated with resistance to neonicotinoid insecticides in the aphid Myzus persicae. BMC Neuroscience. 12, 51-51 (2011).
- Erdmanis, L., et al. Association of neonicotinoid insensitivity with a conserved residue in the loop d binding region of the tick nicotinic acetylcholine receptor. Biochemistry. 51 (23), 4627-4629 (2012).
- Doering, J. A., et al. et al. In silico site-directed mutagenesis informs species-specific predictions of chemical susceptibility derived from the Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS) tool. Toxicology Sciences. 166 (1), 131-145 (2018).
- Noyes, P. D., et al. Evaluating chemicals for thyroid disruption: Opportunities and challenges with in vitro testing and adverse outcome pathway approaches. Environmental Health Perspectives. 127 (9), 95001 (2019).
- Park, G. Y., Jamerlan, A., Shim, K. H., An, S. S. A. Diagnostic and treatment approaches involving transthyretin in amyloidogenic diseases. Int J Mol Sci. 20 (12), 2982 (2019).
- Rabah, S. A., Gowan, I. L., Pagnin, M., Osman, N., Richardson, S. J. Thyroid hormone distributor proteins during development in vertebrates. Front Endocrinol (Lausane). 10, 506 (2019).
- Richardson, S. J. Cell and molecular biology of transthyretin and thyroid hormones. International Review of Cytology. 258, 137-193 (2007).
- Yamauchi, K., Ishihara, A., Richardson, S. J., Cody, V. Transthyretin and Endocrine Disruptors. Recent Advances in Transthyretin Evolution, Structure and Biological Functions. , 159-171 (2009).
- Iakovleva, I., et al. Tetrabromobisphenol A is an efficient stabilizer of the transthyretin tetramer. PLoS One. 11 (4), 0153529 (2016).
- Ishihara, A., Sawatsubashi, S., Yamauchi, K. Endocrine disrupting chemicals: Interference of thyroid hormone binding to transthyretins and to thyroid hormone receptors. Molecular and Cellular Endocrinology. 199 (1), 105-117 (2003).
- Kar, S., Sepúlveda, M. S., Roy, K., Leszczynski, J. Endocrine-disrupting activity of per- and polyfluoroalkyl substances: Exploring combined approaches of ligand and structure based modeling. Chemosphere. 184, 514-523 (2017).
- Morais-de-Sa, E., Pereira, P. J., Saraiva, M. J., Damas, A. M. The crystal structure of transthyretin in complex with diethylstilbestrol: A promising template for the design of amyloid inhibitors. Journal of Biological Chemistry. 279 (51), 53483-53490 (2004).
- Morgado, I., Campinho, M. A., Costa, R., Jacinto, R., Power, D. M. Disruption of the thyroid system by diethylstilbestrol and ioxynil in the sea bream (Sparus aurata). Aquatic Toxicology. 92 (4), 271-280 (2009).
- Yamauchi, K., Prapunpoj, P., Richardson, S. J. Effect of diethylstilbestrol on thyroid hormone binding to amphibian transthyretins. General and Comparative Endocrinology. 119 (3), 329-339 (2000).
- Zhang, J., et al. Structure-based virtual screening protocol for in silico identification of potential thyroid disrupting chemicals targeting transthyretin. Environmental Science & Technology. 50 (21), 11984-11993 (2016).
- Ren, X. M., et al. Binding interactions of perfluoroalkyl substances with thyroid hormone transport proteins and potential toxicological implications. Toxicology. 366-367, 32-42 (2016).
- Wilson, N., Mbabazi, K., Seth, P., Smith, H., Davis, N. L. Drug and opioid-involved overdose deaths - United States, 2017-2018. Morbidity and Mortality Weekly Report. 69 (11), 290-297 (2020).
- National Pollutant Discharge Elimination System (NPDES). United States Environmental Protection Agency Available from: https://www.epa.gov/npdes/npdes-resources (2018)
- Duvallet, C., Hayes, B. D., Erickson, T. B., Chai, P. R., Matus, M. Mapping community opioid exposure through wastewater-based epidemiology as a means to engage pharmacies in harm reduction efforts. Preventing Chronic Disease. 17, 200053 (2020).
- Gushgari, A. J., Venkatesan, A. K., Chen, J., Steele, J. C., Halden, R. U. Long-term tracking of opioid consumption in two United States cities using wastewater-based epidemiology approach. Water Research. 161, 171-180 (2019).
- Lau, B., Bretaud, S., Huang, Y., Lin, E., Guo, S. Dissociation of food and opiate preference by a genetic mutation in zebrafish. Genes Brain Behave. 5 (7), 497-505 (2006).
- Bossé, G. D., Peterson, R. T. Development of an opioid self-administration assay to study drug seeking in zebrafish. Behavioural Brain Research. 335, 158-166 (2017).
- Mottaz, H., et al. Dose-dependent effects of morphine on lipopolysaccharide (LPS)-induced inflammation, and involvement of multixenobiotic resistance (MXR) transporters in LPS efflux in teleost fish. Environmental Pollution. 221, 105-115 (2017).
- Manglik, A., et al. Crystal structure of the µ-opioid receptor bound to a morphinan antagonist. Nature. 485 (7398), 321-326 (2012).
- Comer, S. D., Cahill, C. M. Fentanyl: Receptor pharmacology, abuse potential, and implications for treatment. Neuroscience & Biobehavioral Reviews. 106, 49-57 (2019).
- Podlewska, S., Bugno, R., Kudla, L., Bojarski, A. J., Przewlocki, R. Molecular modeling of µ opioid receptor ligands with various functional properties: PZM21, SR-17018, morphine, and fentanyl-simulated interaction patterns confronted with experimental data. Molecules. 25 (20), 4636 (2020).
- Huang, W., et al. Structural insights into µ-opioid receptor activation. Nature. 524 (7565), 315-321 (2015).
- Lipiński, P. F. J., et al. Fentanyl family at the mu-opioid receptor: Uniform assessment of binding and computational analysis. Molecules. 24 (4), 740 (2019).
- Boland, L. A., Angles, J. M. Feline permethrin toxicity: Retrospective study of 42 cases. Journal of Feline Medicine and Surgery. 12 (2), 61-71 (2010).
- Stevenson, B. J., Pignatelli, P., Nikou, D., Paine, M. J. Pinpointing P450s associated with pyrethroid metabolism in the dengue vector, Aedes aegypti: developing new tools to combat insecticide resistance. PLoS Neglected Tropical Diseases. 6 (3), 1595 (2012).
- Ankley, G. T., Gray, L. E. Cross-species conservation of endocrine pathways: A critical analysis of tier 1 fish and rat screening assays with 12 model chemicals. Environmental Toxicology and Chemistry. 32 (5), 1084-1087 (2013).
- Meteyer, C. U., Rideout, B. A., Gilbert, M., Shivaprasad, H. L., Oaks, J. L. Pathology and proposed pathophysiology of diclofenac poisoning in free-living and experimentally exposed oriental white-backed vultures (Gyps bengalensis). Journal of Wildlife Diseases. 41 (4), 707-716 (2005).
- ECOTOX User Guide: ECOTOXicology Knowledgebase System. EPA, United States Environmental Protection Agency Available from: https://cfpub.epa.gov/ecotox/index.cfm (2021)
- ECOS Environmental Conservation Online System. U.S. Fish & Wildlife Service Available from: https://ecos.fws.gov/ecp/ (2021)
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon