Method Article
Visualización de la localización subcelular de una proteína en el corazón utilizando inmunoetiquetado mediado por puntos cuánticos seguido de microscopía electrónica de transmisión
En este artículo
Resumen
El presente protocolo describe un método de inmunomarcaje de una proteína en las secciones del tejido cardíaco utilizando puntos cuánticos. Esta técnica proporciona una herramienta útil para visualizar la localización y expresión subcelular de cualquier proteína a nivel ultraestructural.
Resumen
La localización subcelular es crítica para delinear la función adecuada y determinar los mecanismos moleculares de una proteína en particular. Se utilizan varias técnicas cualitativas y cuantitativas para determinar la localización subcelular de las proteínas. Una de las técnicas emergentes para determinar la localización subcelular de una proteína es el inmunomarcaje mediado por puntos cuánticos (QD) de una proteína, seguido de imágenes de ellas con microscopía electrónica de transmisión (TEM). QD es un nanocristal semiconductor con una doble propiedad de estructura cristalina y alta densidad de electrones, lo que los hace aplicables a la microscopía electrónica. Este método actual visualizó la localización subcelular de la proteína del receptor Sigma 1 (Sigmar1) utilizando QD-TEM en el tejido cardíaco a nivel ultraestructural. Pequeños cubos de las secciones de tejido cardíaco de un ratón de tipo salvaje se fijaron en glutaraldehído al 3%, posteriormente osmicados, teñidos con acetato de uranilo, seguidos de deshidratación secuencial con etanol y acetona. Estas secciones de tejido cardíaco deshidratado se incrustaron en resinas epoxi de baja viscosidad, se cortaron en secciones delgadas de 500 nm de espesor, se colocaron en la rejilla y posteriormente se sometieron a desenmascaramiento de antígenos con metaperiodato de sodio al 5%, seguido de enfriamiento de los aldehídos residuales con glicina. Los tejidos fueron bloqueados, seguidos de incubación secuencial en anticuerpo primario, anticuerpo secundario biotinilado y QD conjugado con estreptavidina. Estas secciones teñidas se secaron con manchas y se obtuvieron imágenes a gran aumento utilizando TEM. La técnica QD-TEM permitió la visualización de la localización subcelular de la proteína Sigmar1 a nivel ultraestructural en el corazón. Estas técnicas se pueden utilizar para visualizar la presencia de cualquier proteína y localización subcelular en cualquier sistema de órganos.
Introducción
El cuerpo humano está compuesto de muchas proteínas responsables de numerosas funciones corporales. La función de las proteínas depende en gran medida de su localización en el órgano y los orgánulos celulares. Varias técnicas, incluyendo el fraccionamiento subcelular, la inmunofluorescencia y la extracción de proteínas mediada por detergente, se utilizan comúnmente para determinar la localización subcelular de la proteína 1,2. La microscopía con colorante inmunofluorescente es el método más utilizado entre estas técnicas. Sin embargo, los colorantes fluorescentes utilizados en esta técnica son menos estables y propensos al fotoblanqueo3. Otras técnicas involucraron microscopía de alta resolución para visualizar la proteína a nivel ultraestructural mediante inmunomarcaje de proteínas con nanocristales densos en electrones, metales pesados (oro, ferritina) o puntos cuánticos y seguidos visualizándolos mediante microscopía electrónica de transmisión (TEM)4,5.
QD es un nanocristal semiconductor compuesto de compuestos metálicos semiconductores con propiedades de fotoluminiscencia controlables que tienen una gran importancia en los sistemas biológicos3. Los nanocristales QD se fabrican en un formato núcleo-cáscara donde un nanocristal se encapsula en la formación de nanocristales para asegurar su correcta estabilidad y funcionamiento. La combinación de nanocristales núcleo-cáscara comúnmente utilizada es CdSe/ZnS, CdSe/CdS CdSe/ZnSe, CdTe/CdS, CdTe/ZnS y CdTe/CdS/ZnS (núcleo/capa/cáscara)3. Entre estas combinaciones de nanocristales, CdSe/ZnS y CdSe/CdS son estudiadas más vigorosamente y utilizadas con frecuencia como conjugados secundariosde anticuerpos 3,6. Estas nanopartículas QD también poseen propiedades fluorescentes con diferentes espectros de excitación y emisión que los fluoróforos tradicionales. QD emplea la excitación de electrones de la banda de valencia a granel para exhibir mayores rendimientos cuánticos de fluorescencia en comparación con los fluoróforos tradicionales. La disposición de nanocristales de los metales semiconductores hace que el etiquetado mediado por QD sea más estable y resistente al fotoblanqueo6. Además, el núcleo nanocristalino en QD y su estructura cristalina permite que QD de diferentes tamaños tenga una amplia gama de espectros de absorción y picos de emisión muy estrechos7. Además, estas partículas QD son lo suficientemente grandes como para producir una alta densidad de electrones, lo que las hace útiles en técnicas de microscopía de alta resolución, incluida la microscopía electrónica de transmisión 5,8,9. Estos nanocristales QD también están disponibles comercialmente en múltiples tamaños con diferentes espectros y formas de emisión de fluorescencia, lo que los convierte en un gran candidato para el etiquetado con múltiples anticuerpos10,11.
La tecnología QD atrajo gran importancia en la investigación biológica debido a múltiples propiedades funcionales, incluido el uso en imágenes de células vivas, el estudio de los mecanismos de transporte en la célula, el transporte de membrana del movimiento de difusión de proteínas, la heterogeneidad funcional de las células y el marcado del orgánulo intracelular 3,12,13,14,15,16 . QD también es útil en el diagnóstico molecular para atacar y detectar tejidos cancerosos, caracterizar el perfil molecular y el estado inmune del tumor, y visualizar el cuerpo vítreo y las membranas epirretinianas 3,17,18. Además, QD también se puede utilizar en terapias médicas para tratar tumores malignos a través de terapia fotodinámica y anomalías oftálmicas mediante la administración de medicamentos a los ojos 3,17,18,19.
Utilizando estos nanocristales QD altamente útiles, el presente estudio determinó la localización subcelular de una proteína llamada receptor Sigma 1 (Sigmar1). Sigmar1 es una proteína chaperona molecular multitarea expresada ubicuamente. Extensos estudios centrados en la localización subcelular de Sigmar1 en diferentes tejidos y órganos reportaron una localización subcelular específica de células y tejidos que provoca una función molecular20. En diferentes células (neuronales, fotorreceptoras y células inmunes) y tejidos (hígado y cerebro) utilizando diferentes enfoques bioquímicos, los estudios informaron la localización de Sigmar1 en retículo endoplásmico (RE), membrana ER asociada a mitocondrias (MAM), envoltura nuclear, membrana plasmática, retículo nucleoplásmico, núcleo y mitocondrias. A pesar de todos estos estudios, la localización subcelular de Sigmar1 en el corazón permaneció desconocida20. Por lo tanto, la localización subcelular de Sigmar1 en el tejido cardíaco se determinó utilizando el inmunomarcaje mediado por QD seguido de imágenes TEM.
Protocolo
Los procedimientos de manejo de animales en este protocolo cumplieron con la Guía para el Cuidado y Uso de Animales de Laboratorio (Octava Edición, Institutos Nacionales de Salud, Bethesda, MD) y fueron aprobados por el Comité de Cuidado y Uso de Animales del Centro de Ciencias de la Salud de la Universidad Estatal de Louisiana-Shreveport. Para el presente estudio se utilizaron ratones machos de seis meses de edad con antecedentes de FVB / N. Los ratones se obtuvieron de fuentes comerciales (ver Tabla de materiales). Los ratones utilizados fueron alojados en un ambiente bien regulado en jaulas que permiten un ciclo de luz-oscuridad de 12 h, provistos de agua y una dieta regular de comida ad libitum, y fueron atendidos de acuerdo con la Guía de los NIH para el Cuidado y Uso de los Animales de Laboratorio. El proceso general se ilustra en la Figura 1.
1. Preparación de animales
- Anestesiar a los ratones usando isoflurano al 3%. Abra el pecho haciendo una incisión horizontal en la región por encima del abdomen medio y tirando de la piel. Con cuidado, haga otra incisión para abrir la cavidad torácica sin perforar ningún otro órgano21,22,23.
- Perfundir24 el corazón a través del ápice primero, y luego el ventrículo derecho usando glutaraldehído frío al 3% en solución cardiopléjica (50 mM KCl, dextrosa al 5% en PBS, ver Archivo Suplementario 1) durante 2 min para asegurar una relajación miocárdica completa.
- Perfundir el corazón usando glutaraldehído al 3% helado en 0.1 M de tampón de cacodilato de sodio (pH 7.4, paso 2.1.1) durante otros 2 minutos. Usando la presión de gravedad, use una aguja de 25 G 5/8" para introducir los fijadores en el corazón. Inmediatamente después de que el corazón comience a llenarse con el fijador, levante el ápice del corazón y corte los vasos debajo a 1-2 mm del corazón para aliviar la presión y permitir que los líquidos drenen.
- Diseccionar el corazón, eliminar las aurículas24 y dejar caer los ventrículos en glutaraldehído helado al 3% / cacodilato de sodio 0.1 M en una placa de Petri. Haga un corte de mariposa después de 30-60 minutos de fijación y colóquelo en una placa de Petri que contenga 3% de glutaraldehído/cacodilato (ver Tabla de materiales). Picar el corazón con una cuchilla quirúrgica en cubos pequeños de 1 mm3 mientras está en solución de glutaraldehído/cacodilato.
- Fijar/sumergir el tejido cardíaco disecado en solución de glutaraldehído/cacodilato durante 24 h a 4 °C.
2. Procesamiento del tejido cardíaco
- Después de 24 h de fijación, lavar los tejidos en 0,1 M de tampón de cacodilato de sodio durante 20 min.
- Prepare el tampón de cacodilato 0,1 M siguiendo los pasos a continuación.
- Para preparar tampón de cacodilato de sodio 0,1 M, prepare un stock de tampón de cacodilato de sodio 1 M disolviendo cacodilato de sodio (21,4 g) y de solución de cloruro de calcio al 1% (3 ml) en 90 ml de agua destilada. Suba el volumen de la solución a 100 ml agregando el agua destilada. Revuelva bien la solución y déjela toda la noche para disolver el soluto.
- A continuación, tome 10 ml de solución madre de cacodilato de sodio 1 M y agréguela a 80 ml de agua destilada. Ajuste el pH a 7.4 usando HCl y aumente el volumen hasta 100 ml agregando agua destilada.
- Prepare el tampón de cacodilato 0,1 M siguiendo los pasos a continuación.
- Repita el lavado en tampón de cacodilato de sodio 0,1 M durante otros 20 minutos. Retire los tejidos del tampón de cacodilato de sodio y sumérjalos en una solución de tetróxido de osmio al 2% durante 4 h a temperatura ambiente. Este proceso se llama osmicación.
- Prepare una solución de tetróxido de osmio al 2% siguiendo los pasos a continuación.
- Para preparar una solución de tetróxido de osmio al 2%, tome una solución de tetróxido de osmio al 4%, 4 ml (consulte la tabla de materiales), una solución madre de cacodilato sódico al 1 m (0,8 ml) y agua destilada (3,2 ml) para hacer un total de 8 ml de solución.
NOTA: Todo este proceso y los pasos a partir de aquí deben llevarse a cabo en la campana extractora.
- Para preparar una solución de tetróxido de osmio al 2%, tome una solución de tetróxido de osmio al 4%, 4 ml (consulte la tabla de materiales), una solución madre de cacodilato sódico al 1 m (0,8 ml) y agua destilada (3,2 ml) para hacer un total de 8 ml de solución.
- Prepare una solución de tetróxido de osmio al 2% siguiendo los pasos a continuación.
- Después de la osmicación, sumerja los tejidos en una solución de acetato de sodio al 2% durante 10 minutos a temperatura ambiente.
- Prepare una solución de acetato de sodio al 2% disolviendo 4 g de acetato de sodio en 20 ml de agua destilada.
- A continuación, sumerja los tejidos en una solución de acetato de uranilo al 2% durante 1 h a temperatura ambiente.
- Prepare una solución de acetato de uranilo al 2% disolviendo 4 g de acetato de uranilo en 20 ml de agua destilada.
- Después de la tinción de acetato de uranilo, deshidratar los tejidos secuencialmente a través de los alcoholes clasificados y la acetona en el orden mencionado en el Archivo Suplementario 1.
3. Incrustación del tejido cardíaco
- Incruste los tejidos deshidratados en resina epoxi de baja viscosidad siguiendo los pasos a continuación.
- Para hacer resina epoxi de baja viscosidad, mezcle 10.24 ml de monómero epoxi de dióxido de ciclohexeno de vinilo (ERL 4221), 6.74 ml de éter diglicílico de polipropilenglicol (DER 732) y 30.05 ml de anhídrido succínico de nonenil (NSA) (consulte la Tabla de materiales) en un tubo de 50 ml. Revuelva bien la suspensión a mano durante 2 min.
- Agregue 18 gotas de acelerador epoxi de 2-dimetilaminoetanol (DMAE, consulte la Tabla de materiales) y revuelva la suspensión para mezclar bien los componentes.
- Impregnar los tejidos en resina epoxi en la siguiente secuencia de mezcla.
- Sustituir la acetona 100% por resina 1:1: suspensión de acetona y sumergir los tejidos en ella durante 1 h a temperatura ambiente.
- Reemplace la resina 1:1: suspensión de acetona por resina 6:1: suspensión de acetona y sumerja los tejidos en ella durante 3 h.
- Finalmente, reemplaza la resina 6:1: suspensión de acetona por suspensión 100% resina y deja los tejidos sumergidos en ella durante toda la noche a temperatura ambiente.
- Poner los tejidos en resina fresca en micromoldes de 8 mm (ver Tabla de materiales) y curar los tejidos incrustados a 70 °C durante la noche.
NOTA: Asegúrese de que la resina sea dura pero no quebradiza después del curado.
4. Sección de tejido con un ultramicrotomo
- Recorte los bloques de resina con el tejido a no más de 1 mm antes de montarlos en el ultramicrótomo (consulte la Tabla de materiales). Coloque el molde con la mayor precisión posible en el brazo segmentado del ultramicrotomo y avance manualmente el molde de muestra hacia el cuchillo de diamante.
- Corte las secciones a un grosor de 500 nm (media micra) con un cuchillo Histo (consulte la Tabla de materiales) y, utilizando la herramienta de bucle EM, recójalas y colóquelas en un portaobjetos de vidrio.
- Coloque el portaobjetos de vidrio en una placa caliente para la tinción azul de toluidina25 para encontrar el área de interés.
- Después de encontrar el área de interés, use un cuchillo Ultra 45 ° (consulte la Tabla de materiales) para producir secciones ultrafinas de oro pálido (100 nm). Coloque estas secciones ultrafinas en el lado opaco de una rejilla de cobre de malla 200.
5. Tinción de la sección ultrafina del corazón
- Comience la tinción desenmascarando el antígeno con una solución de grabado, es decir, una solución de metaperiodato de sodio al 5%.
- Prepare 500 μL de solución de metaperiodato de sodio al 5% (consulte la Tabla de materiales) en agua destilada.
NOTA: Prepare la solución fresca antes de usar. - Poner 20 μL de solución de metaperiodato de sodio en una película de parafina limpia. Coloque las rejillas completamente secas con secciones de tejido en la gota de la solución de grabado.
NOTA: El lado opaco de la rejilla con la sección de tejido debe mirar hacia la solución de grabado. - Deje la rejilla de sección en la solución durante 30 minutos a temperatura ambiente.
- Prepare 500 μL de solución de metaperiodato de sodio al 5% (consulte la Tabla de materiales) en agua destilada.
- Lave la sección de tejido grabado colocándola en una gota de agua destilada durante 60 s.
- Bloquear los aldehídos residuales colocando las rejillas de sección en una gota de solución de glicina 0,05 M durante 10 min a temperatura ambiente. Seque los bordes de la rejilla en papel de filtro para eliminar la solución de glicina residual.
- Preparar solución de glicina 0,05 M (ver Tabla de materiales) disolviendo 3,75 mg de glicina en 1 ml de 1x PBS (pH 7,4).
- Coloque las rejillas de sección en 10-20 μL de solución de bloqueo durante 25 min a temperatura ambiente.
NOTA: Composición de la solución de bloqueo: 2 μL de suero normal de cabra al 1% (NGS) + 20 μL de BSA al 1% (concentración final: 10%) + 178 μL de 1x PBS (pH 7.4) para hacer un volumen final de 200 μL. - Seque los bordes de la rejilla en papel de filtro y coloque la sección de la rejilla en el diluyente de anticuerpos para su acondicionamiento a temperatura ambiente durante 10 minutos.
- Incubar las secciones de rejilla con anticuerpo primario (Sigmar1 en este caso, ver Tabla de materiales; diluido 1:10 en diluyente de anticuerpos) durante 1 h 30 min en una cámara humidificada.
- Seque la rejilla y lave las secciones de la rejilla con diluyente de anticuerpos dos veces durante 5 minutos cada una.
- Incubar las secciones de la rejilla con anticuerpo secundario biotinilado (en este caso, anticuerpo secundario policlonal anti-conejo de cabra biotinilada, ver Tabla de materiales; diluido 1:10 en diluyente de anticuerpos) durante 1 h en una cámara humidificada.
- Seque la rejilla y lave las secciones de la rejilla con diluyente de anticuerpos dos veces durante 5 minutos cada una.
- Incubar las secciones de rejilla en QD conjugado con estreptavidina disponible comercialmente (QD655nm, ver Tabla de materiales; diluido 1:10 en diluyente de anticuerpos) durante 1 h en una cámara humidificada a temperatura ambiente. Evite la exposición a la luz cubriendo la cámara con papel de aluminio.
- Seque los bordes de la rejilla con papel de filtro y lave las secciones de la rejilla colocándolas en gotas de agua durante 2 minutos.
- Seque los bordes de la rejilla para que se sequen.
6. Imágenes de microscopía electrónica de transmisión (TEM)
- Coloque las secciones ultrafinas en rejillas de cobre en un soporte de cuarteto de muestras y sujetarlas en una posición que permita la selección de rejillas en el haz de electrones.
- Inserte el portamuestras en la columna del microscopio y active el interruptor de la bomba para evacuar el goniómetro, seguido de la inserción completa del portamuestras en la columna del microscopio (consulte la Tabla de materiales).
- Ajuste el voltaje a 80 kV antes de generar el haz para la observación de imágenes.
- Enfoque bien en el área deseada, capture la imagen con una cámara digital de alta velocidad y guarde el archivo en .tif formato.
NOTA: El ajuste del microscopio para capturar la imagen en este estudio fue voltaje de aceleración o voltaje de alta tensión de 80 kV y un aumento de 20,000x.
Resultados
La presente metodología QD-TEM visualizó la presencia de Sigmar1 y su localización en los compartimentos cardíacos subcelulares mediante la realización de un etiquetado QD dirigido anti-Sigmar1 en secciones cardíacas ultradelgadas de ratones adultos. Las imágenes QD marcadas con anti-Sigmar1 denso en electrones en membranas mitocondriales (internas y externas), lisosomas y la interfaz membrana-mitocondrial del retículo endoplásmico / retículo sarcoplásmico (ER / SR) (Figura 2A, B) se ilustraron con imágenes QD-TEM. Además, las secciones del corazón también se marcaron con IgG y QD de conejo como un control de isotipo para el anticuerpo primario de conejo anti-Sigmar1 (Figura 2C, D). Por lo tanto, la imagen QD-TEM destacó la localización de Sigmar1 endógeno y su enriquecimiento principalmente en los lisosomas y las membranas mitocondriales.
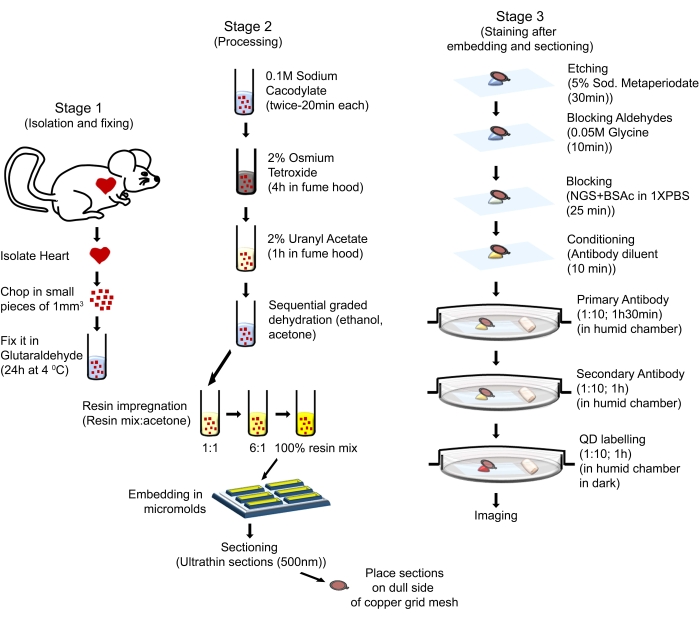
Figura 1: Ilustración esquemática que muestra los pasos secuenciales y los procedimientos utilizados para realizar QD-TEM. Haga clic aquí para ver una versión más grande de esta figura.
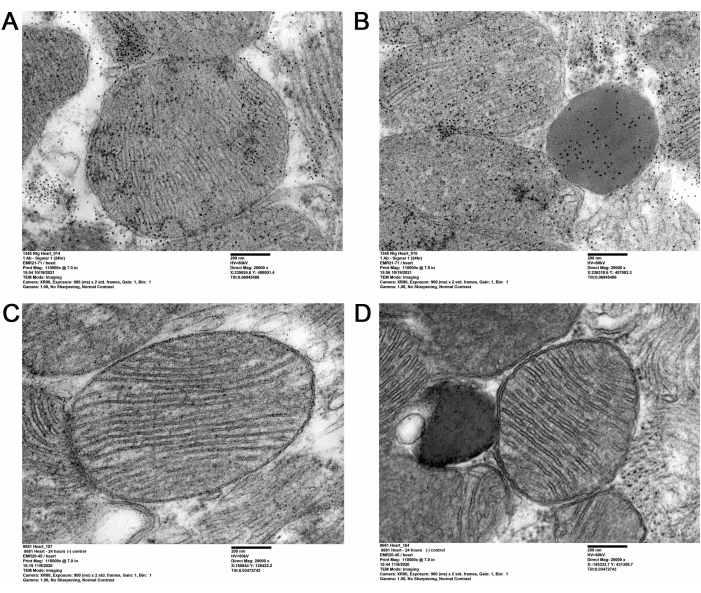
Figura 2: Sigmar1 inmuno-marcado QD en tejidos cardíacos de ratones adultos . (A,B) Imágenes representativas de TEM que muestran Sigmar1 marcado QD en mitocondrias (membrana externa e interna), retículo endoplásmico asociado a mitocondrias (RE) / retículo sarcoplásmico (SR) membranas y lisosomas. (C,D) Imagen TEM de las secciones cardíacas del control del isotipo marcadas con QDs e IgG de conejo. Barra de escala: 200 nm. Haga clic aquí para ver una versión más grande de esta figura.
Archivo complementario 1: Composición de PBS y otras soluciones utilizadas para la deshidratación tisular. Haga clic aquí para descargar este archivo.
Discusión
En el presente estudio, se utilizó inmunomarcaje mediado por QD para mostrar claramente la localización subcelular de Sigmar1. Usando QD, la localización de Sigmar1 en la membrana mitocondrial, especialmente la membrana mitocondrial interna, se representó en el tejido cardíaco. Además, también se encontró que Sigmar1 estaba localizado en retículo sarcoplásmico/endoplásmico (S/ER) y lisosomas a nivel ultraestructural, como se muestra en la Figura 2A-D.
Un paso crítico en este protocolo es el paso de grabado o desenmascaramiento de antígenos utilizando una solución de metaperiodato de sodio altamente concentrada para desenmascarar el antígeno después de la fijación y osmicación de glutaraldehído. Este protocolo utilizó un tratamiento único con una alta concentración (5%) de metaperiodato de sodio durante 30 min a temperatura ambiente. Se necesita un cuidado adicional en este paso, ya que una mayor duración o mayor concentración para la incubación de metaperiodato de sodio dará como resultado la agregación de estructuras, la pérdida de la definición de membrana para los orgánulos y causará perforaciones en la sección, lo que dificultará la visualización de la proteína o la estructura. Alternativamente, también se puede usar una concentración más baja de solución de metaperiodato (3%) en dos pasos durante 30 minutos en lugar de metaperiodato al 5%. Los estudios han demostrado que esta opción exhibe resultados similares a los de la solución de metaperiodato al 5% para la incubación de un paso y 30 minutos. Sin embargo, una solución de metaperiodato al 3% durante 30 min de incubación durante dos veces proporciona un mejor control sobre el proceso26,27,28. Inicialmente, este protocolo utilizó la incubación de las secciones con solución de metaperiodato al 10% durante 30 min. Sin embargo, debido a demasiadas perforaciones creadas en la sección de tejido por esta concentración, la concentración final y la duración de la incubación de la solución de metaperiodato se disminuyeron y se optimizaron al 5% durante 30 min.
Otro paso requirió la optimización del tiempo de fijación con glutaraldehído. La fijación subóptima de los tejidos da como resultado un etiquetado QD inadecuado, mientras que la fijación excesiva de los tejidos da como resultado un etiquetado no específico más alto. Por lo tanto, se debe considerar cuidadosamente la determinación y titulación de un nivel óptimo de fijación tisular para el etiquetado adecuado y específico de las proteínas. En este método utilizando tejidos cardíacos, el tiempo de fijación con glutaraldehído se tituló utilizando 24 h y 48 h como puntos de tiempo. A partir de las imágenes de tinción de las secciones fijadas para ambos puntos temporales, se encontró que las secciones fijadas durante 24 h mostraron mejores resultados. Hasta la fecha, los nanocristales QD están disponibles en múltiples tamaños, incluidos 525, 565, 585, 605, 655 y 705 nm11,29. Cada uno de estos QD tiene sus propios espectros de emisión y emite fluorescencia a diferentes longitudes de onda. Además, estos QD disponibles comercialmente de diferentes tamaños muestran diferentes formas; por ejemplo, QD 525, 565 y 585 son virtualmente esféricos con diferentes tamaños, mientras que QD 605, 655 y 705 son irregulares de forma oblonga. De estos diferentes nanocristales QD, QD 525, 565 y 655 son fácilmente distinguibles entre sí11,29. Estas diferencias en los espectros y formas de emisión hacen de QD un gran candidato para el etiquetado múltiple de proteínas y la visualización por fluorescencia y microscopía electrónica. En este estudio, se utilizó un QD disponible comercialmente, QD 655, para etiquetar la proteína Sigmar1 para distinguirla de cualquier fondo no específico en las secciones teñidas.
Otra contraparte de QD para el etiquetado de proteínas en microscopía de alta resolución es la partícula de inmunooro. Las partículas de inmunooro se utilizan tradicionalmente para etiquetar proteínas para microscopía de alta resolución. Estas partículas de oro son altamente densas en electrones y fácilmente identificables en comparación con los nanocristales QD. Sin embargo, QD exhibe una mejor eficiencia con mejor penetración en los tejidos, mayor estabilidad y vida útil, y mejor retención de componentes ultraestructurales, lo que los convierte en un mejor candidato para el etiquetado de proteínas 4,5. QD también tiene una capacidad única para ser detectado por microscopía óptica y electrónica, lo que aumenta su valor sobre el etiquetado de inmunooro10.
Una limitación de este inmunomarcaje mediado por QD es el uso de tetróxido de osmio durante el procesamiento. El tetróxido de osmio se utiliza para aumentar la densidad electrónica, la conductividad y el contraste de estructuras de membrana biológicas menos densas en electrones y menos contrastantes 5,30. Sin embargo, el uso de tetróxido de osmio destruye instantánea e irreversiblemente la propiedad del espécimen para crear fluorescencia cuando se marca con QD6. Esto limita el uso de QD en microscopía de fluorescencia. Un enfoque alternativo que omita el uso de tetróxido de osmio será ventajoso para conservar las propiedades fluorescentes y, por lo tanto, la aplicación dual del inmunomarcado mediado por QD. Algunos de los modelos más nuevos de TEM tienen la opción de conectar el sistema de análisis de rayos X por dispersión de energía (EDX) que permite la identificación de la composición elemental de los materiales. Otra limitación del estudio es la falta de mapeo elemental de una muestra y análisis de imágenes utilizando EDX. Por lo tanto, los estudios futuros deben centrarse en el análisis EDX de los espectros QD para analizar la composición elemental.
El etiquetado QD de proteínas ha ganado mucha atención en los últimos tiempos. QD ofrece varias aplicaciones y ventajas tanto en la investigación biológica como en la terapéutica médica. QD al estar envuelto adicionalmente con ligando polidentado exhibe una mayor estabilidad manteniendo el rendimiento cuántico. Además, la encapsulación de QD con estos agentes biofavorables también aumenta su biodisponibilidad en los tejidos, lo que lo convierte en un buen candidato para su posible aplicación en la detección de tumores, imágenes de células vivas, administración de fármacos e imágenes de tejidos 3,31,32.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por las subvenciones de los Institutos Nacionales de Salud: R01HL145753, R01HL145753-01S1, R01HL145753-03S1 y R01HL152723; Premio LSUHSC-S CCDS Finish Line, Premio de Investigación COVID-19 y Premio de Investigación LARC a MSB; LSUHSC-S Malcolm Feist Cardiovascular and AHA Postdoctoral Fellowship to CSA (20POST35210789); y LSUHSC-S Malcolm Feist Pre-doctoral Fellowship to RA.
Materiales
| Name | Company | Catalog Number | Comments |
| 200 Mesh copper grid | Ted Pella | G200HH | |
| 6-month-old male mice with FVB/N background | Jackson Laboratory, Bar Harbor, ME | ||
| Acetone 100% | Fisher Chemicals | A949 | |
| Antibody diluent | Dako | S3022 | |
| anti-Sigmar1 antibody | Cell Signaling | 61994S | |
| Biotinylated goat anti-rabbit IgG antibody | Sigma Aldrich | B7389 | |
| BSAc (10%) | Electron Microscopy Sciences | 25557 | |
| Calcium chloride | Sigma Aldrich | C7902 | |
| Cytoseal Xyl | Thermo fisher | 8312-4 | |
| DER 732C36AA10:C33 | Electron Microscopy Sciences | 13010 | |
| Dextrose | Sigma Aldrich | G7528 | |
| Diamond Knife | Diatome | Histo; Ultra 450 | |
| DMAE | Electron Microscopy Sciences | 13300 | |
| Electron microscope | JEOL | JEOL-1400 Flash | |
| ERL 4221 | Electron Microscopy Sciences | 15004 | |
| Ethanol 100% | Fisher Chemicals | A405P | |
| Glutaraldehyde 3% | Electron Microscopy Sciences | 16538-15 | |
| Glycine | Alfa Aesar | A13816 | |
| Hydrochloric acid | Fisher Scientific | SA56 | |
| Micromolds | Ted Pella | 10505 | |
| Microtome | Leica Microsystem | EM UC7 | |
| Normal goat serum | Invitrogen | PCN5000 | |
| NSA | Electron Microscopy Sciences | 19050 | |
| Osmium tetroxide | Electron Microscopy Sciences | 19150 | |
| Parafilm | Genesse Scientific | 16-101 | |
| Potassium Chloride | Sigma Aldrich | P5655 | |
| Potassium Phosphate monobasic | Sigma Aldrich | 71640 | |
| Qdot 655 Streptavidin Conjugate | Invitrogen | Q10121MP | |
| Sodium Acetate | Fisher Scientific | BP334 | |
| Sodium Cacodylate | Electron Microscopy Sciences | 12300 | |
| Sodium Chloride | Fisher Scientific | BP358 | |
| Sodium metaperiodate | Sigma Aldrich | 71859 | |
| Sodium Phosphate dibasic | Sigma Aldrich | P9541 | |
| Surgical blade (size 10) | Aspen surgical | 371110 | |
| TEM image software | AMT-V700 | AMT TEM imaging systems | |
| TEM imaging camera | XR80 TEM series | AMT TEM imaging systems | |
| Toluidine Blue O solution (0.5%) | Fisher Scientic | S25612 | |
| Uranyl acetate | Polysciences | 21447 |
Referencias
- Lidke, D. S., Lidke, K. A. Advances in high-resolution imaging - techniques for three-dimensional imaging of cellular structures. Journal of Cell Science. 125 (11), 2571-2580 (2012).
- de Duve, C. Tissue fraction-past and present. The Journal of Cell Biology. 50 (1), 20 (1971).
- Pleskova, S., Mikheeva, E., Gornostaeva, E., Saquib, Q., Faisal, M., Al-Khedhairy, A. A., Alatar, A. A. . Cellular and Molecular Toxicology of Nanoparticles. , 323-334 (2018).
- Mayhew, T. M., Mühlfeld, C., Vanhecke, D., Ochs, M. A review of recent methods for efficiently quantifying immunogold and other nanoparticles using TEM sections through cells, tissues and organs. Annals of Anatomy - Anatomischer Anzeiger. 191 (2), 153-170 (2009).
- Kuipers, J., de Boer, P., Giepmans, B. N. G. Scanning EM of non-heavy metal stained biosamples: Large-field of view, high contrast and highly efficient immunolabeling. Experimental Cell Research. 337 (2), 202-207 (2015).
- Deerinck, T. J. The application of fluorescent quantum dots to confocal, multiphoton, and electron microscopic imaging. Toxicologic Pathology. 36 (1), 112-116 (2008).
- Nisman, R., Dellaire, G., Ren, Y., Li, R., Bazett-Jones, D. P. Application of quantum dots as probes for correlative fluorescence, conventional, and energy-filtered transmission electron microscopy. Journal of Histochemistry & Cytochemistry. 52 (1), 13-18 (2004).
- Michalet, X., et al. Quantum dots for live cells, in vivo imaging, and diagnostics. Science. 307 (5709), 538-544 (2005).
- Killingsworth, M. C., Bobryshev, Y. V. Correlative light- and electron microscopy using quantum dot nanoparticles. Journal of Visualized Experiments. (114), e54307 (2016).
- Deerinck, T. J., Giepmans, B. N. G., Smarr, B. L., Martone, M. E., Ellisman, M. H., Bruchez, M. P., Hotz, C. Z., Ellisman, M. H. . Quantum Dots: Applications in Biology. , 43-53 (2007).
- Giepmans, B. N. G., Deerinck, T. J., Smarr, B. L., Jones, Y. Z., Ellisman, M. H. Correlated light and electron microscopic imaging of multiple endogenous proteins using Quantum dots. Nature Methods. 2 (10), 743-749 (2005).
- Lidke, D. S., et al. Quantum dot ligands provide new insights into erbB/HER receptor-mediated signal transduction. Nature Biotechnology. 22 (2), 198-203 (2004).
- Pleskova, S. N., et al. Differences in the functional activity of human neutrophilic granulocytes in their interactions with semiconductor quantum dots. Morfologiia. 135 (3), 47-49 (2009).
- Crane, J., Haggie, P., Verkman, A. Quantum dot single molecule tracking reveals a wide range of diffusive motions of membrane transport proteins. Proceedings of SPIE. 7189, (2009).
- Hanaki, K. -. i., et al. Semiconductor quantum dot/albumin complex is a long-life and highly photostable endosome marker. Biochemical and Biophysical Research Communications. 302 (3), 496-501 (2003).
- Lim, Y. T., et al. . Selection of quantum dot wavelengths for biomedical assays and imaging. 2, 50-64 (2003).
- Gao, X., Cui, Y., Levenson, R. M., Chung, L. W. K., Nie, S. In vivo cancer targeting and imaging with semiconductor quantum dots. Nature Biotechnology. 22 (8), 969-976 (2004).
- Åkerman, M. E., Chan, W. C. W., Laakkonen, P., Bhatia, S. N., Ruoslahti, E. Nanocrystal targeting in vivo. Proceedings of the National Academy of Sciences. 99 (20), 12617-12621 (2002).
- Sarwat, S. A. -. O. X., Stapleton, F., Willcox, M., Roy, M. A. -. O. Quantum dots in ophthalmology: A literature review. Current Eye Research. 44 (10), 1037-1046 (2019).
- Aishwarya, R., Abdullah, C. S., Morshed, M., Remex, N. S., Bhuiyan, M. S. Sigmar1's molecular, cellular, and biological functions in regulating cellular pathophysiology. Frontiers in Physiology. 12, 705575 (2021).
- Bohne, B., Harding, G. . Processing the mouse temporal bone for gross, cellular and subcellular evaluation poster. , (2004).
- Roszkowski, M. . The effects of acute stress on Apold1 gene expression and blood-brain barrier permeability. , (2014).
- Wu, J., et al. Transcardiac perfusion of the mouse for brain tissue dissection and fixation. Bio-protocol. 11 (5), 3988 (2021).
- Jones, W. K., et al. Ablation of the murine alpha myosin heavy chain gene leads to dosage effects and functional deficits in the heart. Journal of Clinical Investigation. 98 (8), 1906-1917 (1996).
- Hunter, E. E. . Practical Electron Microscopy: A Beginner's Illustrated Guide. , (1993).
- Morris, R. E., Ciraolo, G. M. A universal post-embedding protocol for immunogold labelling of osmium-fixed, epoxy resin-embedded tissue. Journal of Electron Microscopy. 46 (4), 315-319 (1997).
- Brorson, S. H. Deplasticizing or etching of epoxy sections with different concentrations of sodium ethoxide to enhance the immunogold labeling. Micron. 32 (2), 101-105 (2001).
- Lobo, M. V. T., et al. Ultrastructural Staining with Sodium Metaperiodate and Sodium Borohydride. Journal of Histochemistry & Cytochemistry. 50 (1), 11-19 (2002).
- Bruchez, M., Moronne, M., Gin, P., Weiss, S., Alivisatos, A. P. Semiconductor nanocrystals as fluorescent biological labels. Science. 281 (5385), 2013-2016 (1998).
- Tapia, J. C., et al. High-contrast en bloc staining of neuronal tissue for field emission scanning electron microscopy. Nature Protocols. 7 (2), 193-206 (2012).
- Smith, A. M., Duan, H., Mohs, A. M., Nie, S. Bioconjugated quantum dots for in vivo molecular and cellular imaging. Advanced Drug Delivery Reviews. 60 (11), 1226-1240 (2008).
- Gour, A., Ramteke, S., Jain, N. K. Pharmaceutical applications of quantum dots. AAPS PharmSciTech. 22 (7), 233 (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados