Method Article
Visualizzazione della localizzazione subcellulare di una proteina nel cuore utilizzando l'immuno-marcatura mediata da punti quantici seguita da microscopia elettronica a trasmissione
In questo articolo
Riepilogo
Il presente protocollo descrive un metodo di immuno-marcatura di una proteina nelle sezioni del tessuto cardiaco utilizzando punti quantici. Questa tecnica fornisce uno strumento utile per visualizzare la localizzazione e l'espressione subcellulare di qualsiasi proteina a livello ultrastrutturale.
Abstract
La localizzazione subcellulare è fondamentale per delineare il corretto funzionamento e determinare i meccanismi molecolari di una particolare proteina. Diverse tecniche qualitative e quantitative sono utilizzate per determinare la localizzazione subcellulare delle proteine. Una delle tecniche emergenti nel determinare la localizzazione subcellulare di una proteina è l'immunomarcatura mediata da punti quantici (QD) di una proteina seguita dall'imaging con microscopia elettronica a trasmissione (TEM). QD è un nanocristallo semiconduttore con una duplice proprietà di struttura cristallina e alta densità elettronica, che li rende applicabili alla microscopia elettronica. Questo metodo attuale ha visualizzato la localizzazione subcellulare della proteina del recettore Sigma 1 (Sigmar1) utilizzando QD-TEM nel tessuto cardiaco a livello ultrastrutturale. Piccoli cubi delle sezioni del tessuto cardiaco di un topo selvatico sono stati fissati in glutaraldeide al 3%, successivamente osmicati, colorati con acetato di uranile, seguiti da disidratazione sequenziale con etanolo e acetone. Queste sezioni di tessuto cardiaco disidratato sono state incorporate in resine epossidiche a bassa viscosità, tagliate in sezioni sottili di 500 nm di spessore, messe sulla griglia e successivamente sottoposte a smascheramento dell'antigene con metaperiodato di sodio al 5%, seguito da tempra delle aldeidi residue con glicina. I tessuti sono stati bloccati, seguiti dall'incubazione sequenziale nell'anticorpo primario, nell'anticorpo secondario biotinilato e nel QD coniugato con streptavidina. Queste sezioni colorate sono state asciugate a macchia e riprodotte ad alto ingrandimento utilizzando TEM. La tecnica QD-TEM ha permesso la visualizzazione della localizzazione subcellulare della proteina Sigmar1 a livello ultrastrutturale nel cuore. Queste tecniche possono essere utilizzate per visualizzare la presenza di qualsiasi proteina e localizzazione subcellulare in qualsiasi sistema di organi.
Introduzione
Il corpo umano è composto da molte proteine responsabili di numerose funzioni corporee. La funzione delle proteine dipende in gran parte dalla loro localizzazione nell'organo e negli organelli cellulari. Diverse tecniche, tra cui il frazionamento subcellulare, l'immunofluorescenza e l'estrazione proteica mediata da detergenti, sono comunemente usate per determinare la localizzazione subcellulare della proteina 1,2. La microscopia con colorante immunofluorescente è il metodo più utilizzato tra queste tecniche. Tuttavia, i coloranti fluorescenti utilizzati in questa tecnica sono meno stabili e inclini al fotosbiancamento3. Altre tecniche hanno coinvolto la microscopia ad alta risoluzione per visualizzare la proteina a livello ultrastrutturale mediante immunomarcatura di proteine con nanocristalli di metalli pesanti densi di elettroni (oro, ferritina) o punti quantici e seguita dalla visualizzazione utilizzando la microscopia elettronica a trasmissione (TEM)4,5.
QD è un nanocristallo semiconduttore composto da composti metallici semiconduttori con proprietà di fotoluminescenza controllabili che hanno un grande significato nei sistemi biologici3. I nanocristalli QD sono realizzati in un formato core-shell in cui un nanocristallo è incapsulato nella formazione di nanocristalli per garantire la loro corretta stabilità e funzionamento. Le combinazioni di nanocristalli core-shell comunemente usate sono CdSe/ZnS, CdSe/CdS CdSe/ZnSe, CdTe/CdS, CdTe/ZnS e CdTe/CdS/ZnS (core/shell/shell)3. Tra queste combinazioni di nanocristalli, CdSe/ZnS e CdSe/CdS sono le più vigorosamente studiate e frequentemente usate come coniugati anticorpali secondari 3,6. Queste nanoparticelle QD possiedono anche proprietà fluorescenti con spettri di eccitazione ed emissione diversi rispetto ai fluorofori tradizionali. La QD impiega l'eccitazione di elettroni dalla banda di valenza di massa per mostrare rese quantiche di fluorescenza più elevate rispetto ai fluorofori tradizionali. La disposizione dei nanocristalli dei metalli semiconduttori rende l'etichettatura mediata da QD più stabile e resistente al fotosbiancamento6. Inoltre, il nucleo del nanocristallo in QD e la sua struttura cristallina consentono a QD di diverse dimensioni di avere una vasta gamma di spettri di assorbimento e picchi di emissione molto stretti7. Inoltre, queste particelle QD sono abbastanza grandi da produrre un'alta densità di elettroni, rendendole utili nelle tecniche di microscopia ad alta risoluzione, inclusa la microscopia elettronica a trasmissione 5,8,9. Questi nanocristalli QD sono anche disponibili in commercio in più dimensioni con diversi spettri e forme di emissione di fluorescenza, rendendoli un ottimo candidato per l'etichettatura con anticorpi multipli10,11.
La tecnologia QD ha attratto grande importanza nella ricerca biologica a causa di molteplici proprietà funzionali, tra cui l'uso nell'imaging di cellule vive, lo studio dei meccanismi di trasporto nella cellula, il trasporto di membrana del movimento di diffusione della proteina, l'eterogeneità funzionale delle cellule e la marcatura degli organelli intracellulari 3,12,13,14,15,16 . La QD è utile anche nella diagnostica molecolare per il targeting e la rilevazione dei tessuti tumorali, caratterizzando il profilo molecolare del tumore e lo stato immunitario e visualizzando il corpo vitreo e le membrane epiretiniche 3,17,18. Inoltre, il QD può essere utilizzato anche nelle terapie mediche per trattare i tumori maligni attraverso la terapia fotodinamica e le anomalie oftalmiche somministrando farmaci agli occhi 3,17,18,19.
Utilizzando questi nanocristalli QD altamente utili, il presente studio ha determinato la localizzazione subcellulare di una proteina chiamata recettore Sigma 1 (Sigmar1). Sigmar1 è una proteina chaperone molecolare multi-tasking ubiquitariamente espressa. Studi approfonditi incentrati sulla localizzazione subcellulare di Sigmar1 in diversi tessuti e organi hanno riportato una localizzazione subcellulare specifica per cellula e tipo di tessuto che induce la funzione molecolare20. In diverse cellule (neuronali, fotorecettori e cellule immunitarie) e tessuti (fegato e cervello) utilizzando diversi approcci biochimici, gli studi hanno riportato la localizzazione di Sigmar1 sul reticolo endoplasmatico (ER), sulla membrana ER associata ai mitocondri (MAM), sull'involucro nucleare, sulla membrana plasmatica, sul reticolo nucleoplasmatico, sul nucleo e sui mitocondri. Nonostante tutti questi studi, la localizzazione subcellulare di Sigmar1 nel cuore è rimasta sconosciuta20. Pertanto, la localizzazione subcellulare di Sigmar1 nel tessuto cardiaco è stata determinata utilizzando l'immunomarcatura mediata da QD seguita dall'imaging TEM.
Protocollo
Le procedure di gestione degli animali in questo protocollo sono conformi alla Guida per la cura e l'uso degli animali da laboratorio (ottava edizione, National Institutes of Health, Bethesda, MD) e sono state approvate dal Comitato per la cura e l'uso degli animali del Centro di scienze della salute della Louisiana State University-Shreveport. Per il presente studio sono stati utilizzati topi maschi di sei mesi con background FVB/N. I topi sono stati ottenuti da fonti commerciali (vedi Tabella dei materiali). I topi utilizzati sono stati alloggiati in un ambiente ben regolato in gabbie che consentono un ciclo luce-buio di 12 ore, fornito di acqua e una dieta regolare di chow ad libitum, e sono stati curati secondo la Guida NIH per la cura e l'uso degli animali da laboratorio. Il processo complessivo è illustrato nella Figura 1.
1. Preparazione degli animali
- Anestetizzare i topi usando isoflurano al 3%. Aprire il torace praticando un'incisione orizzontale nella regione sopra l'addome medio e tirando la pelle. Con attenzione, fare un'altra incisione per aprire la cavità toracica senza perforare altri organi21,22,23.
- Perfondere24 il cuore attraverso l'apice prima, e poi il ventricolo destro usando glutaraldeide fredda al 3% in soluzione cardioplegica (50 mM KCl, 5% destrosio in PBS, vedi file supplementare 1) per 2 minuti per garantire un completo rilassamento miocardico.
- Perfondere il cuore usando glutaraldeide ghiacciata al 3% in 0,1 M di tampone di cacodilato di sodio (pH 7,4, passo 2.1.1) per altri 2 minuti. Utilizzando la pressione di gravità, utilizzare un ago da 25 G 5/8" per introdurre i fissativi nel cuore. Immediatamente dopo che il cuore inizia a riempirsi con il fissativo, sollevare l'apice del cuore e tagliare i vasi sottostanti a 1-2 mm dal cuore per alleviare la pressione e consentire ai liquidi di drenare.
- Sezionare il cuore, rimuovere gli atri24 e far cadere i ventricoli in glutaraldeide ghiacciata al 3% / cacodilato di sodio 0,1 M in una capsula di Petri. Effettuare un taglio a farfalla dopo 30-60 minuti di fissazione e metterlo in una capsula di Petri contenente il 3% di glutaraldeide/cacodilato (vedi Tabella dei materiali). Tritare il cuore usando una lama chirurgica in piccoli cubetti di 1 mm3 mentre è in soluzione di glutaraldeide/cacodilato.
- Fissare/immergere il tessuto cardiaco sezionato in soluzione di glutaraldeide/cacodilato per 24 ore a 4 °C.
2. Elaborazione del tessuto cardiaco
- Dopo 24 ore di fissazione, lavare i tessuti in 0,1 M di tampone cacodilato di sodio per 20 minuti.
- Preparare un tampone cacodilato da 0,1 M seguendo i passaggi seguenti.
- Per preparare un tampone di cacodilato di sodio 0,1 M, preparare una scorta di tampone cacodilato di sodio 1 M sciogliendo il cacodilato di sodio (21,4 g) e la soluzione di cloruro di calcio all'1% (3 ml) in 90 ml di acqua distillata. Volume fino a 100 ml aggiungendo l'acqua distillata. Mescolare bene la soluzione e lasciarla per una notte per sciogliere il soluto.
- Quindi, prendere 10 ml di soluzione madre di cacodalilato di sodio 1 M e aggiungerla a 80 ml di acqua distillata. Regolare il pH a 7,4 utilizzando HCl e volumetrico fino a 100 ml aggiungendo acqua distillata.
- Preparare un tampone cacodilato da 0,1 M seguendo i passaggi seguenti.
- Ripetere il lavaggio in tampone di cacodilato di sodio 0,1 M per altri 20 minuti. Rimuovere i tessuti dal tampone di cacodilato di sodio e immergerli in una soluzione di tetrossido di osmio al 2% per 4 ore a temperatura ambiente. Questo processo è chiamato osmication.
- Preparare la soluzione di tetrossido di osmio al 2% seguendo i passaggi seguenti.
- Per preparare la soluzione di tetrossido di osmio al 2%, prendere una soluzione di tetrossido di osmio al 4%, 4 ml (vedere la tabella dei materiali), una soluzione madre di cacodilato di sodio 1 M (0,8 ml) e acqua distillata (3,2 ml) per ottenere un totale di 8 ml di soluzione.
NOTA: L'intero processo e le fasi successive devono essere eseguiti nella cappa aspirante.
- Per preparare la soluzione di tetrossido di osmio al 2%, prendere una soluzione di tetrossido di osmio al 4%, 4 ml (vedere la tabella dei materiali), una soluzione madre di cacodilato di sodio 1 M (0,8 ml) e acqua distillata (3,2 ml) per ottenere un totale di 8 ml di soluzione.
- Preparare la soluzione di tetrossido di osmio al 2% seguendo i passaggi seguenti.
- Dopo l'osmicazione, immergere i tessuti in una soluzione di acetato di sodio al 2% per 10 minuti a temperatura ambiente.
- Preparare una soluzione di acetato di sodio al 2% sciogliendo 4 g di acetato di sodio in 20 ml di acqua distillata.
- Quindi, immergere i tessuti in una soluzione di acetato di uranile al 2% per 1 ora a temperatura ambiente.
- Preparare una soluzione di acetato di uranile al 2% sciogliendo 4 g di acetato di uranile in 20 ml di acqua distillata.
- Dopo la colorazione con acetato di uranile, disidratare i tessuti in sequenza attraverso gli alcoli classificati e l'acetone nell'ordine indicato nella scheda supplementare 1.
3. Incorporazione del tessuto cardiaco
- Incorporare i tessuti disidratati in resina epossidica a bassa viscosità seguendo i passaggi seguenti.
- Per ottenere resina epossidica a bassa viscosità, mescolare 10,24 mL di biossido di vinile cicloesene (ERL 4221) monomero epossidico, 6,74 mL di etere diglicidil di glicole propilenico (DER 732) e 30,05 ml di anidride nonenilsuccinica (NSA) (vedere Tabella dei materiali) in un tubo da 50 ml. Mescolare bene la sospensione a mano per 2 minuti.
- Aggiungere 18 gocce di 2-dimetilamminoetanolo (DMAE, vedere Tabella dei materiali) acceleratore epossidico e mescolare la sospensione per mescolare accuratamente i componenti.
- Impregnare i tessuti in resina epossidica nella seguente sequenza di miscela.
- Sostituire l'acetone al 100% con resina 1:1: sospensione di acetone e immergere i tessuti in esso per 1 h a temperatura ambiente.
- Sostituire la resina 1:1: sospensione di acetone con resina 6:1: sospensione di acetone e immergere i tessuti in essa per 3 ore.
- Infine, sostituire la resina 6:1: sospensione di acetone con sospensione di resina al 100% e lasciare i tessuti immersi in essa per una notte a temperatura ambiente.
- Mettere i tessuti in resina fresca in micro stampi da 8 mm (vedi Tabella dei materiali) e polimerizzare i tessuti incorporati a 70 °C durante la notte.
NOTA: Assicurarsi che la resina sia dura ma non fragile dopo la polimerizzazione.
4. Sezionamento dei tessuti con un ultramicrotomo
- Tagliare i blocchi di resina con il tessuto a non più di 1 mm prima di montare sull'ultramicrotomo (vedere Tabella dei materiali). Posizionare lo stampo il più precisamente possibile nel braccio segmentato dell'ultramicrotomo e far avanzare manualmente lo stampo del campione verso il coltello diamantato.
- Tagliare le sezioni con uno spessore di 500 nm (mezzo micron) con un coltello Histo (vedi Tabella dei materiali) e, utilizzando lo strumento EM loop, raccoglierle e posizionarle su un vetrino.
- Posizionare il vetrino su una piastra riscaldante per toluidina blu colorazione25 per trovare l'area di interesse.
- Dopo aver trovato l'area di interesse, utilizzare un coltello Ultra 45° (vedi Tabella dei materiali) per produrre sezioni ultrasottili in oro pallido (100 nm). Posiziona queste sezioni ultrasottili sul lato opaco di una griglia di rame a 200 maglie.
5. Colorazione della sezione cardiaca ultrasottile
- Iniziare la colorazione smascherando l'antigene usando una soluzione di incisione, cioè una soluzione di metaperiodato di sodio al 5%.
- Preparare 500 μL di soluzione di metaperiodato di sodio al 5% (vedere Tabella dei materiali) in acqua distillata.
NOTA: Preparare una soluzione fresca prima dell'uso. - Mettere 20 μL di soluzione di metaperiodato di sodio su un film di paraffina pulito. Posizionare le griglie completamente asciutte con sezioni di tessuto sulla goccia della soluzione di incisione.
NOTA: Il lato opaco della griglia con la sezione di tessuto deve essere rivolto verso la soluzione di incisione. - Lasciare la griglia di sezione sulla soluzione per 30 minuti a temperatura ambiente.
- Preparare 500 μL di soluzione di metaperiodato di sodio al 5% (vedere Tabella dei materiali) in acqua distillata.
- Lavare la sezione di tessuto inciso posizionandola su una goccia di acqua distillata per 60 secondi.
- Bloccare le aldeidi residue posizionando le griglie di sezione su una goccia di soluzione di glicina 0,05 M per 10 minuti a temperatura ambiente. Tamponare i bordi della griglia su carta da filtro per rimuovere la soluzione di glicina residua.
- Preparare la soluzione di glicina 0,05 M (vedere Tabella dei materiali) sciogliendo 3,75 mg di glicina in 1 mL di 1x PBS (pH 7,4).
- Posizionare le griglie di sezione in 10-20 μL di soluzione bloccante per 25 minuti a temperatura ambiente.
NOTA: Composizione della soluzione bloccante: 2 μL di siero di capra normale all'1% (NGS) + 20 μL di BSA all'1% (concentrazione finale: 10%) + 178 μL di 1x PBS (pH 7,4) per ottenere un volume finale di 200 μL. - Asciugare i bordi della griglia su carta da filtro e posizionare la sezione della griglia sul diluente anticorpale per il condizionamento a temperatura ambiente per 10 minuti.
- Incubare le sezioni della griglia con anticorpo primario (Sigmar1 in questo caso, vedi Tabella dei materiali; diluito 1:10 in diluente anticorpale) per 1 h 30 min in camera umidificata.
- Asciugare la griglia e lavare le sezioni della griglia con diluente anticorpale due volte per 5 minuti ciascuna.
- Incubare le sezioni della griglia con anticorpo secondario biotinilato (in questo caso, anticorpo secondario policlonale anti-coniglio di capra biotinilato, vedi Tabella dei materiali; diluito 1:10 in diluente anticorpale) per 1 ora in camera umidificata.
- Asciugare la griglia e lavare le sezioni della griglia con diluente anticorpale due volte per 5 minuti ciascuna.
- Incubare le sezioni della griglia in QD coniugato con streptavidina disponibile in commercio (QD655nm, vedi Tabella dei materiali; diluito 1:10 in diluente anticorpale) per 1 ora in una camera umidificata a temperatura ambiente. Prevenire l'esposizione alla luce coprendo la camera con un foglio di alluminio.
- Asciugare i bordi della griglia con carta da filtro e lavare le sezioni della griglia posizionandole su goccioline d'acqua per 2 minuti.
- Asciugare i bordi della griglia.
6. Microscopia elettronica a trasmissione (TEM)
- Posizionare le sezioni ultrasottili su griglie di rame in un porta-quartetto di campioni e bloccarle in una posizione tale da consentire la selezione delle griglie nel fascio di elettroni.
- Inserire il supporto del campione nella colonna del microscopio e azionare l'interruttore della pompa per evacuare il goniometro, seguito dall'inserimento completo del supporto del campione nella colonna del microscopio (vedere Tabella dei materiali).
- Impostare la tensione su 80 kV prima di generare il fascio per l'osservazione delle immagini.
- Metti a fuoco bene l'area desiderata, cattura l'immagine utilizzando una fotocamera digitale ad alta velocità e salva il file in .tif formato.
NOTA: L'impostazione del microscopio per catturare l'immagine in questo studio era l'accelerazione della tensione o della tensione ad alta tensione di 80 kV e un ingrandimento di 20.000x.
Risultati
L'attuale metodologia QD-TEM ha visualizzato la presenza di Sigmar1 e la sua localizzazione sui compartimenti cardiaci subcellulari eseguendo la marcatura QD mirata anti-Sigmar1 su sezioni cardiache ultrasottili di topo adulto. La QD marcata anti-Sigmar1 densa di elettroni sulle membrane mitocondriali (interne ed esterne), sui lisosomi e sull'interfaccia membrana-mitocondriale reticolo/reticolo sarcoplasmatico (ER/SR) (Figura 2A,B) sono state illustrate dalle immagini QD-TEM. Inoltre, le sezioni cardiache sono state anche etichettate con IgG e QD di coniglio come controllo isotipo per l'anticorpo primario del coniglio anti-Sigmar1 (Figura 2C, D). Pertanto, l'imaging QD-TEM ha evidenziato la localizzazione di Sigmar1 endogeno e il suo arricchimento principalmente sui lisosomi e sulle membrane mitocondriali.
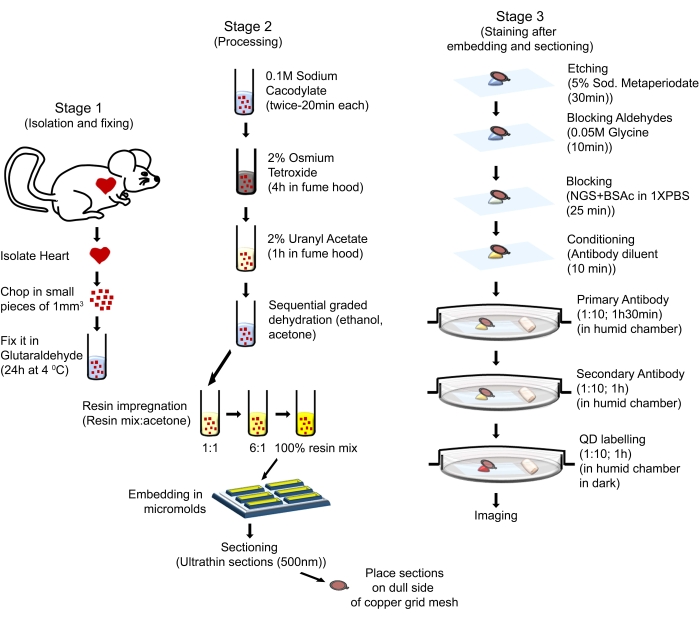
Figura 1: Illustrazione schematica che mostra i passaggi sequenziali e le procedure utilizzate per eseguire QD-TEM. Fare clic qui per visualizzare una versione ingrandita di questa figura.
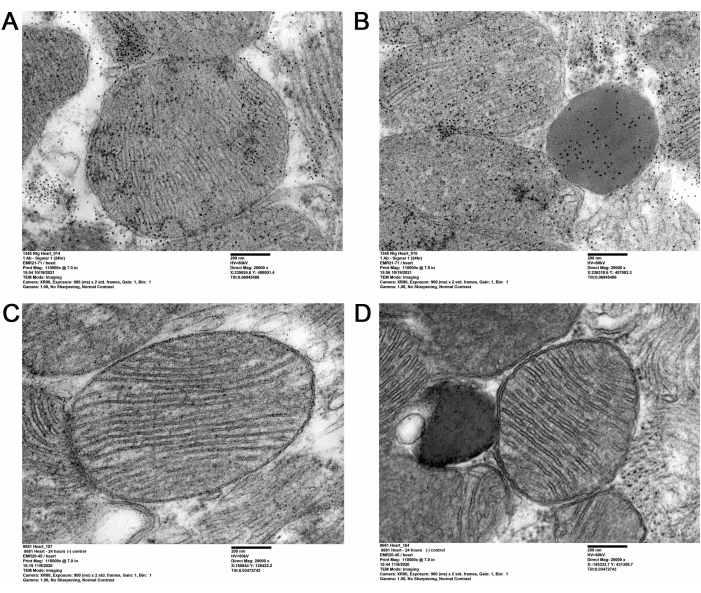
Figura 2: QD immuno-marcato con Sigmar1 nei tessuti cardiaci di topi adulti. (A,B) Immagini TEM rappresentative che mostrano Sigmar1 marcato QD su mitocondri (membrana esterna e interna), reticolo endoplasmatico associato ai mitocondri (ER) / reticolo sarcoplasmatico (SR) e lisosomi. (C,D) Immagine TEM delle sezioni cardiache del controllo isotipo marcate con QD e IgG di coniglio. Barra scala: 200 nm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
File supplementare 1: Composizione di PBS e altre soluzioni utilizzate per la disidratazione dei tessuti. Clicca qui per scaricare questo file.
Discussione
Nel presente studio, l'immunomarcatura mediata da QD è stata utilizzata per mostrare in modo distintivo la localizzazione subcellulare di Sigmar1. Utilizzando la QD, la localizzazione di Sigmar1 sulla membrana mitocondriale, in particolare sulla membrana mitocondriale interna, è stata rappresentata nel tessuto cardiaco. Inoltre, Sigmar1 è stato trovato localizzato anche sul reticolo sarcoplasmatico/endoplasmatico (S/ER) e sui lisosomi a livello ultrastrutturale, come mostrato nella Figura 2A-D.
Un passaggio critico in questo protocollo è la fase di incisione o smascheramento dell'antigene utilizzando una soluzione di metaperiodato di sodio altamente concentrata per smascherare l'antigene dopo la fissazione e l'osmizione della glutaraldeide. Questo protocollo ha utilizzato un singolo trattamento con un'alta concentrazione (5%) di metaperiodato di sodio per 30 minuti a temperatura ambiente. È necessaria particolare attenzione in questa fase poiché una durata più lunga o una concentrazione più elevata per l'incubazione del metaperiodato di sodio comporterà l'aggregazione delle strutture, la perdita della definizione della membrana per gli organelli e causerà perforazioni nella sezione, rendendo difficile visualizzare la proteina o la struttura. In alternativa, è possibile utilizzare anche una concentrazione inferiore di soluzione di metaperiodato (3%) in due fasi per 30 minuti invece del metaperiodato al 5%. Gli studi hanno dimostrato che questa opzione mostra risultati simili a quelli della soluzione metaperiodato al 5% per l'incubazione in una fase di 30 minuti. Tuttavia, una soluzione di metaperiodato al 3% per 30 minuti di incubazione per due volte fornisce un migliore controllo sul processo26,27,28. Inizialmente, questo protocollo prevedeva l'incubazione delle sezioni con soluzione di metaperiodato al 10% per 30 minuti. Tuttavia, a causa delle troppe perforazioni create nella sezione tissutale da questa concentrazione, la concentrazione finale e la durata di incubazione della soluzione di metaperiodato sono state ridotte e ottimizzate al 5% per 30 minuti.
Un altro passo ha richiesto l'ottimizzazione del tempo di fissazione con glutaraldeide. La fissazione non ottimale dei tessuti si traduce in un'etichettatura QD inadeguata, mentre la fissazione eccessiva dei tessuti si traduce in una maggiore etichettatura non specifica. Pertanto, è necessario prestare particolare attenzione nel determinare e titolare un livello ottimale di fissazione tissutale per una corretta e specifica etichettatura delle proteine. In questo metodo utilizzando tessuti cardiaci, il tempo di fissazione con glutaraldeide è stato titolato utilizzando 24 ore e 48 ore come timepoint. Sulla base delle immagini di colorazione delle sezioni fissate per entrambi i timepoint, è stato riscontrato che le sezioni fissate per 24 ore hanno mostrato risultati migliori. Ad oggi, i nanocristalli QD sono disponibili in più dimensioni, tra cui 525, 565, 585, 605, 655 e 705 nm11,29. Ognuno di questi QD ha i propri spettri di emissione ed emette fluorescenza a diverse lunghezze d'onda. Inoltre, questi QD disponibili in commercio di diverse dimensioni mostrano forme diverse; ad esempio, QD 525, 565 e 585 sono praticamente sferici con dimensioni diverse, mentre QD 605, 655 e 705 sono di forma oblunga irregolare. Di questi diversi nanocristalli QD, QD 525, 565 e 655 sono facilmente distinguibili l'uno dall'altro11,29. Queste differenze negli spettri di emissione e nelle forme rendono la QD un ottimo candidato per la marcatura multipla delle proteine e la visualizzazione mediante fluorescenza e microscopia elettronica. In questo studio, un QD disponibile in commercio, QD 655, è stato utilizzato per etichettare la proteina Sigmar1 per distinguerla da qualsiasi sfondo non specifico nelle sezioni colorate.
Un'altra controparte del QD per la marcatura delle proteine in microscopia ad alta risoluzione è la particella immunogold. Le particelle immunogold sono tradizionalmente utilizzate per marcare le proteine per la microscopia ad alta risoluzione. Queste particelle d'oro sono altamente dense di elettroni e facilmente identificabili rispetto ai nanocristalli QD. Tuttavia, QD mostra una migliore efficienza con una migliore penetrazione nei tessuti, maggiore stabilità e durata di conservazione e una migliore ritenzione dei componenti ultrastrutturali, rendendoli un candidato migliore per l'etichettatura delle proteine 4,5. QD ha anche una capacità unica di essere rilevata sia dalla microscopia ottica che elettronica, che aggiunge valore alla marcatura immunogold10.
Una limitazione di questa immunomarcatura mediata da QD è l'uso di tetrossido di osmio durante la lavorazione. Il tetrossido di osmio viene utilizzato per aumentare la densità elettronica, la conduttività e il contrasto di strutture biologiche a membrana altrimenti meno dense di elettroni e meno contrastanti 5,30. Tuttavia, l'uso del tetrossido di osmio distrugge istantaneamente e irreversibilmente la proprietà del campione di creare fluorescenza quando etichettato con QD6. Ciò limita l'uso della QD nella microscopia a fluorescenza. Un approccio alternativo che omette l'uso del tetrossido di osmio sarà vantaggioso nel mantenere le proprietà fluorescenti e quindi la doppia applicazione dell'immunomarcatura mediata da QD. Alcuni dei modelli più recenti di TEM hanno la possibilità di collegare il sistema di analisi a raggi X a dispersione di energia (EDX) che consente l'identificazione della composizione elementare dei materiali. Un'altra limitazione dello studio è la mancanza di mappatura elementare di un campione e l'analisi delle immagini utilizzando EDX. Pertanto, gli studi futuri dovrebbero concentrarsi sull'analisi EDX degli spettri QD per analizzare la composizione elementare.
La marcatura QD delle proteine ha guadagnato molta attenzione negli ultimi tempi. QD offre diverse applicazioni e vantaggi sia nella ricerca biologica che nelle terapie mediche. Il QD essendo inoltre avvolto con ligando polidentato mostra una maggiore stabilità mantenendo la resa quantica. Inoltre, l'incapsulamento di QD con questi agenti bio-favorevoli aumenta anche la sua biodisponibilità nei tessuti, rendendolo un buon candidato per potenziali applicazioni nella rilevazione di tumori, imaging di cellule vive, somministrazione di farmaci e imaging tissutale 3,31,32.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto dalle sovvenzioni del National Institutes of Health: R01HL145753, R01HL145753-01S1, R01HL145753-03S1 e R01HL152723; LSUHSC-S CCDS Finish Line Award, COVID-19 Research Award e LARC Research Award a MSB; LSUHSC-S Malcolm Feist Cardiovascular e AHA Postdoctoral Fellowship to CSA (20POST35210789); e LSUHSC-S Malcolm Feist Pre-doctoral Fellowship a RA.
Materiali
| Name | Company | Catalog Number | Comments |
| 200 Mesh copper grid | Ted Pella | G200HH | |
| 6-month-old male mice with FVB/N background | Jackson Laboratory, Bar Harbor, ME | ||
| Acetone 100% | Fisher Chemicals | A949 | |
| Antibody diluent | Dako | S3022 | |
| anti-Sigmar1 antibody | Cell Signaling | 61994S | |
| Biotinylated goat anti-rabbit IgG antibody | Sigma Aldrich | B7389 | |
| BSAc (10%) | Electron Microscopy Sciences | 25557 | |
| Calcium chloride | Sigma Aldrich | C7902 | |
| Cytoseal Xyl | Thermo fisher | 8312-4 | |
| DER 732C36AA10:C33 | Electron Microscopy Sciences | 13010 | |
| Dextrose | Sigma Aldrich | G7528 | |
| Diamond Knife | Diatome | Histo; Ultra 450 | |
| DMAE | Electron Microscopy Sciences | 13300 | |
| Electron microscope | JEOL | JEOL-1400 Flash | |
| ERL 4221 | Electron Microscopy Sciences | 15004 | |
| Ethanol 100% | Fisher Chemicals | A405P | |
| Glutaraldehyde 3% | Electron Microscopy Sciences | 16538-15 | |
| Glycine | Alfa Aesar | A13816 | |
| Hydrochloric acid | Fisher Scientific | SA56 | |
| Micromolds | Ted Pella | 10505 | |
| Microtome | Leica Microsystem | EM UC7 | |
| Normal goat serum | Invitrogen | PCN5000 | |
| NSA | Electron Microscopy Sciences | 19050 | |
| Osmium tetroxide | Electron Microscopy Sciences | 19150 | |
| Parafilm | Genesse Scientific | 16-101 | |
| Potassium Chloride | Sigma Aldrich | P5655 | |
| Potassium Phosphate monobasic | Sigma Aldrich | 71640 | |
| Qdot 655 Streptavidin Conjugate | Invitrogen | Q10121MP | |
| Sodium Acetate | Fisher Scientific | BP334 | |
| Sodium Cacodylate | Electron Microscopy Sciences | 12300 | |
| Sodium Chloride | Fisher Scientific | BP358 | |
| Sodium metaperiodate | Sigma Aldrich | 71859 | |
| Sodium Phosphate dibasic | Sigma Aldrich | P9541 | |
| Surgical blade (size 10) | Aspen surgical | 371110 | |
| TEM image software | AMT-V700 | AMT TEM imaging systems | |
| TEM imaging camera | XR80 TEM series | AMT TEM imaging systems | |
| Toluidine Blue O solution (0.5%) | Fisher Scientic | S25612 | |
| Uranyl acetate | Polysciences | 21447 |
Riferimenti
- Lidke, D. S., Lidke, K. A. Advances in high-resolution imaging - techniques for three-dimensional imaging of cellular structures. Journal of Cell Science. 125 (11), 2571-2580 (2012).
- de Duve, C. Tissue fraction-past and present. The Journal of Cell Biology. 50 (1), 20(1971).
- Pleskova, S., Mikheeva, E., Gornostaeva, E. Cellular and Molecular Toxicology of Nanoparticles. Saquib, Q., Faisal, M., Al-Khedhairy, A. A., Alatar, A. A. , Springer International Publishing. 323-334 (2018).
- Mayhew, T. M., Mühlfeld, C., Vanhecke, D., Ochs, M. A review of recent methods for efficiently quantifying immunogold and other nanoparticles using TEM sections through cells, tissues and organs. Annals of Anatomy - Anatomischer Anzeiger. 191 (2), 153-170 (2009).
- Kuipers, J., de Boer, P., Giepmans, B. N. G. Scanning EM of non-heavy metal stained biosamples: Large-field of view, high contrast and highly efficient immunolabeling. Experimental Cell Research. 337 (2), 202-207 (2015).
- Deerinck, T. J. The application of fluorescent quantum dots to confocal, multiphoton, and electron microscopic imaging. Toxicologic Pathology. 36 (1), 112-116 (2008).
- Nisman, R., Dellaire, G., Ren, Y., Li, R., Bazett-Jones, D. P. Application of quantum dots as probes for correlative fluorescence, conventional, and energy-filtered transmission electron microscopy. Journal of Histochemistry & Cytochemistry. 52 (1), 13-18 (2004).
- Michalet, X., et al. Quantum dots for live cells, in vivo imaging, and diagnostics. Science. 307 (5709), 538-544 (2005).
- Killingsworth, M. C., Bobryshev, Y. V. Correlative light- and electron microscopy using quantum dot nanoparticles. Journal of Visualized Experiments. (114), e54307(2016).
- Deerinck, T. J., Giepmans, B. N. G., Smarr, B. L., Martone, M. E., Ellisman, M. H. Quantum Dots: Applications in Biology. Bruchez, M. P., Hotz, C. Z., Ellisman, M. H. , Humana Press. 43-53 (2007).
- Giepmans, B. N. G., Deerinck, T. J., Smarr, B. L., Jones, Y. Z., Ellisman, M. H. Correlated light and electron microscopic imaging of multiple endogenous proteins using Quantum dots. Nature Methods. 2 (10), 743-749 (2005).
- Lidke, D. S., et al. Quantum dot ligands provide new insights into erbB/HER receptor-mediated signal transduction. Nature Biotechnology. 22 (2), 198-203 (2004).
- Pleskova, S. N., et al. Differences in the functional activity of human neutrophilic granulocytes in their interactions with semiconductor quantum dots. Morfologiia. 135 (3), Saint Petersburg, Russia. 47-49 (2009).
- Crane, J., Haggie, P., Verkman, A. Quantum dot single molecule tracking reveals a wide range of diffusive motions of membrane transport proteins. Proceedings of SPIE. 7189, (2009).
- Hanaki, K. -i, et al. Semiconductor quantum dot/albumin complex is a long-life and highly photostable endosome marker. Biochemical and Biophysical Research Communications. 302 (3), 496-501 (2003).
- Lim, Y. T., et al. Selection of quantum dot wavelengths for biomedical assays and imaging. 2, 50-64 (2003).
- Gao, X., Cui, Y., Levenson, R. M., Chung, L. W. K., Nie, S. In vivo cancer targeting and imaging with semiconductor quantum dots. Nature Biotechnology. 22 (8), 969-976 (2004).
- Åkerman, M. E., Chan, W. C. W., Laakkonen, P., Bhatia, S. N., Ruoslahti, E. Nanocrystal targeting in vivo. Proceedings of the National Academy of Sciences. 99 (20), 12617-12621 (2002).
- Sarwat, S. A. -O. X., Stapleton, F., Willcox, M., Roy, M. A. -O. Quantum dots in ophthalmology: A literature review. Current Eye Research. 44 (10), 1037-1046 (2019).
- Aishwarya, R., Abdullah, C. S., Morshed, M., Remex, N. S., Bhuiyan, M. S. Sigmar1's molecular, cellular, and biological functions in regulating cellular pathophysiology. Frontiers in Physiology. 12, 705575(2021).
- Bohne, B., Harding, G. Processing the mouse temporal bone for gross, cellular and subcellular evaluation poster. , (2004).
- Roszkowski, M. The effects of acute stress on Apold1 gene expression and blood-brain barrier permeability. , (2014).
- Wu, J., et al. Transcardiac perfusion of the mouse for brain tissue dissection and fixation. Bio-protocol. 11 (5), 3988(2021).
- Jones, W. K., et al. Ablation of the murine alpha myosin heavy chain gene leads to dosage effects and functional deficits in the heart. Journal of Clinical Investigation. 98 (8), 1906-1917 (1996).
- Hunter, E. E. Practical Electron Microscopy: A Beginner's Illustrated Guide. , Cambridge University Press. (1993).
- Morris, R. E., Ciraolo, G. M. A universal post-embedding protocol for immunogold labelling of osmium-fixed, epoxy resin-embedded tissue. Journal of Electron Microscopy. 46 (4), 315-319 (1997).
- Brorson, S. H. Deplasticizing or etching of epoxy sections with different concentrations of sodium ethoxide to enhance the immunogold labeling. Micron. 32 (2), 101-105 (2001).
- Lobo, M. V. T., et al. Ultrastructural Staining with Sodium Metaperiodate and Sodium Borohydride. Journal of Histochemistry & Cytochemistry. 50 (1), 11-19 (2002).
- Bruchez, M., Moronne, M., Gin, P., Weiss, S., Alivisatos, A. P. Semiconductor nanocrystals as fluorescent biological labels. Science. 281 (5385), 2013-2016 (1998).
- Tapia, J. C., et al. High-contrast en bloc staining of neuronal tissue for field emission scanning electron microscopy. Nature Protocols. 7 (2), 193-206 (2012).
- Smith, A. M., Duan, H., Mohs, A. M., Nie, S. Bioconjugated quantum dots for in vivo molecular and cellular imaging. Advanced Drug Delivery Reviews. 60 (11), 1226-1240 (2008).
- Gour, A., Ramteke, S., Jain, N. K. Pharmaceutical applications of quantum dots. AAPS PharmSciTech. 22 (7), 233(2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon