Method Article
Visualisation de la localisation subcellulaire d’une protéine dans le cœur à l’aide de l’immunomarquage médié par les points quantiques suivi de la microscopie électronique à transmission
Dans cet article
Résumé
Le présent protocole décrit une méthode d’immunomarquage d’une protéine dans les coupes de tissu cardiaque à l’aide de points quantiques. Cette technique fournit un outil utile pour visualiser la localisation et l’expression subcellulaire de toute protéine au niveau ultrastructurel.
Résumé
La localisation subcellulaire est essentielle pour délimiter le bon fonctionnement et déterminer les mécanismes moléculaires d’une protéine particulière. Plusieurs techniques qualitatives et quantitatives sont utilisées pour déterminer la localisation subcellulaire des protéines. L’une des techniques émergentes pour déterminer la localisation subcellulaire d’une protéine est l’immunomarquage médié par points quantiques (QD) d’une protéine, suivi de son imagerie par microscopie électronique à transmission (MET). QD est un nanocristal semi-conducteur avec une double propriété de structure cristalline et de densité électronique élevée, ce qui les rend applicables à la microscopie électronique. Cette méthode actuelle a visualisé la localisation subcellulaire de la protéine du récepteur Sigma 1 (Sigmar1) en utilisant QD-TEM dans le tissu cardiaque au niveau ultrastructurel. De petits cubes de sections de tissu cardiaque provenant d’une souris de type sauvage ont été fixés dans du glutaraldéhyde à 3%, puis osmicés, colorés avec de l’acétate d’uranyle, suivis d’une déshydratation séquentielle avec de l’éthanol et de l’acétone. Ces coupes de tissu cardiaque déshydratées ont été incorporées dans des résines époxy à faible viscosité, coupées en sections minces de 500 nm d’épaisseur, mises sur la grille, puis soumises à un démasquage de l’antigène avec du métaperiodate de sodium à 5%, suivi d’une trempe des aldéhydes résiduels avec de la glycine. Les tissus ont été bloqués, suivis d’une incubation séquentielle dans l’anticorps primaire, l’anticorps secondaire biotinylé et le QD conjugué à la streptavidine. Ces sections colorées ont été séchées par transfert et imagées à fort grossissement à l’aide de TEM. La technique QD-TEM a permis de visualiser la localisation subcellulaire de la protéine Sigmar1 au niveau ultrastructurel du cœur. Ces techniques peuvent être utilisées pour visualiser la présence de n’importe quelle protéine et localisation subcellulaire dans n’importe quel système organique.
Introduction
Le corps humain est composé de nombreuses protéines responsables de nombreuses fonctions corporelles. La fonction des protéines dépend en grande partie de leur localisation dans l’organe et les organites cellulaires. Plusieurs techniques, y compris le fractionnement subcellulaire, l’immunofluorescence et l’extraction de protéines médiée par un détergent, sont couramment utilisées pour déterminer la localisation subcellulaire de la protéine 1,2. La microscopie utilisant un colorant immunofluorescent est la méthode la plus largement utilisée parmi ces techniques. Cependant, les colorants fluorescents utilisés dans cette technique sont moins stables et sujets au photoblanchiment3. D’autres techniques impliquaient la microscopie à haute résolution pour visualiser la protéine à un niveau ultrastructurel en immunomarquant des protéines avec des nanocristaux denses en électrons et lourds (or, ferritine) ou à points quantiques, puis en les visualisant par microscopie électronique à transmission (MET)4,5.
QD est un nanocristal semi-conducteur composé de composés métalliques semi-conducteurs avec des propriétés de photoluminescence contrôlables ayant une grande importance dans les systèmes biologiques3. Les nanocristaux QD sont fabriqués dans un format noyau-coquille où un nanocristal est encapsulé dans la formation de nanocristaux pour assurer leur stabilité et leur bon fonctionnement. Les combinaisons de nanocristaux noyau-enveloppe couramment utilisées sont CdSe/ZnS, CdSe/CdS CdSe/ZnSe, CdTe/CdS, CdTe/ZnS et CdTe/CdS/ZnS (noyau/coque/coque)3. Parmi ces combinaisons de nanocristaux, CdSe/ZnS et CdSe/CdS sont les plus vigoureusement étudiés et fréquemment utilisés comme conjugués d’anticorps secondaires 3,6. Ces nanoparticules QD possèdent également des propriétés fluorescentes avec des spectres d’excitation et d’émission différents de ceux des fluorophores traditionnels. QD utilise l’excitation des électrons de la bande de valence en vrac pour présenter des rendements quantiques de fluorescence plus élevés que les fluorophores traditionnels. La disposition nanocristalline des métaux semi-conducteurs rend le marquage médié par QD plus stable et résistant au photoblanchiment6. De plus, le noyau nanocristallin dans QD et sa structure cristalline permet à QD de différentes tailles d’avoir une large gamme de spectres d’absorption et des pics d’émission très étroits7. De plus, ces particules QD sont suffisamment grosses pour produire une densité électronique élevée, ce qui les rend utiles dans les techniques de microscopie à haute résolution, y compris la microscopie électronique à transmission 5,8,9. Ces nanocristaux QD sont également disponibles dans le commerce en plusieurs tailles avec différents spectres et formes d’émission de fluorescence, ce qui en fait un excellent candidat pour le marquage avec plusieurs anticorps10,11.
La technologie QD a attiré une grande importance dans la recherche biologique en raison de multiples propriétés fonctionnelles, y compris l’utilisation dans l’imagerie de cellules vivantes, l’étude des mécanismes de transport dans la cellule, le transport membranaire du mouvement de diffusion des protéines, l’hétérogénéité fonctionnelle des cellules et le marquage des organites intracellulaires 3,12,13,14,15,16 . QD est également utile dans le diagnostic moléculaire pour cibler et détecter les tissus cancéreux, caractériser le profil moléculaire et l’état immunitaire de la tumeur, et visualiser le corps vitré et les membranes épirétiniennes 3,17,18. En outre, QD peut également être utilisé dans les thérapies médicales pour traiter les tumeurs malignes via la thérapie photodynamique et les anomalies ophtalmiques en administrant des médicaments aux yeux 3,17,18,19.
En utilisant ces nanocristaux QD très utiles, la présente étude a déterminé la localisation subcellulaire d’une protéine appelée récepteur Sigma 1 (Sigmar1). Sigmar1 est une protéine chaperonne moléculaire multitâche omniprésente. Des études approfondies portant sur la localisation subcellulaire de Sigmar1 dans différents tissus et organes ont rapporté une localisation subcellulaire spécifique à la cellule et au type tissulaire déclenchant une fonction moléculaire20. Dans différentes cellules (neuronales, photorécepteurs et cellules immunitaires) et tissus (foie et cerveau) utilisant différentes approches biochimiques, des études ont rapporté la localisation de Sigmar1 sur le réticulum endoplasmique (ER), la membrane ER associée aux mitochondries (MAM), l’enveloppe nucléaire, la membrane plasmique, le réticulum nucléoplasmique, le noyau et les mitochondries. Malgré toutes ces études, la localisation subcellulaire de Sigmar1 dans le cœur restaitinconnue20. Par conséquent, la localisation subcellulaire de Sigmar1 dans le tissu cardiaque a été déterminée à l’aide de l’immunomarquage médié par QD suivi de l’imagerie MET.
Protocole
Les procédures de manipulation des animaux dans ce protocole étaient conformes au Guide for the Care and Use of Laboratory Animals (huitième édition, National Institutes of Health, Bethesda, MD) et ont été approuvées par le Comité de soin et d’utilisation des animaux du Louisiana State University Health Sciences Center-Shreveport. Des souris mâles âgées de six mois avec un fond FVB / N ont été utilisées pour la présente étude. Les souris ont été obtenues de sources commerciales (voir le tableau des matériaux). Les souris utilisées ont été logées dans un environnement bien régulé dans des cages permettant un cycle lumière-obscurité de 12 heures, fournies avec de l’eau et un régime alimentaire régulier ad libitum, et ont été soignées conformément au Guide des NIH pour le soin et l’utilisation des animaux de laboratoire. Le processus global est illustré à la figure 1.
1. Préparation des animaux
- Anesthésier les souris à l’aide d’isoflurane à 3%. Ouvrez la poitrine en faisant une incision horizontale dans la région au-dessus du milieu de l’abdomen et en tirant la peau. Soigneusement, faites une autre incision pour ouvrir la cavité thoracique sans perforer d’autres organes21,22,23.
- Perfuser24 le cœur à travers l’apex d’abord, puis le ventricule droit en utilisant du glutaraldéhyde froid à 3% dans une solution cardioplégique (50 mM KCl, 5% de dextrose dans PBS, voir dossier supplémentaire 1) pendant 2 min pour assurer une relaxation complète du myocarde.
- Perfuser le cœur en utilisant du glutaraldéhyde glacé à 3% dans 0,1 M de tampon cacodylate de sodium (pH 7,4, étape 2.1.1) pendant encore 2 min. En utilisant la pression de gravité, utilisez une aiguille de 25 G 5/8 » pour introduire les fixateurs dans le cœur. Immédiatement après que le cœur commence à se remplir avec le fixateur, soulevez l’apex du cœur et coupez les vaisseaux en dessous à 1-2 mm du cœur pour soulager la pression et permettre aux liquides de s’écouler.
- Disséquez le cœur, retirez les oreillettes24 et laissez tomber les ventricules dans du glutaraldéhyde glacé à 3 % / cacodylate de sodium 0,1 M dans une boîte de Pétri. Faire une coupe papillon après 30-60 min de fixation et la placer dans une boîte de Petri contenant 3% de glutaraldéhyde/cacodylate (voir le tableau des matières). Hacher le cœur à l’aide d’une lame chirurgicale en petits cubes de 1mm3 pendant qu’il est dans une solution de glutaraldéhyde / cacodylate.
- Fixer/immerger le tissu cardiaque disséqué dans une solution de glutaraldéhyde/cacodylate pendant 24 h à 4 °C.
2. Traitement des tissus cardiaques
- Après 24 h de fixation, laver les tissus dans 0,1 M de tampon cacodylate de sodium pendant 20 min.
- Préparer un tampon cacodylate 0,1 M en suivant les étapes ci-dessous.
- Pour préparer un tampon de cacodylate de sodium 0,1 M, préparer un stock de tampon cacodylate de sodium 1 M en dissolvant du cacodylate de sodium (21,4 g) et d’une solution de chlorure de calcium à 1 % (3 mL) dans 90 mL d’eau distillée. Augmenter le volume de la solution à 100 mL en ajoutant l’eau distillée. Bien mélanger la solution et la laisser toute la nuit pour dissoudre le soluté.
- Ensuite, prenez 10 mL de solution mère de cacodylate de sodium 1 M et ajoutez-la à 80 mL d’eau distillée. Ajustez le pH à 7,4 à l’aide de HCl et augmentez le volume jusqu’à 100 mL en ajoutant de l’eau distillée.
- Préparer un tampon cacodylate 0,1 M en suivant les étapes ci-dessous.
- Répéter le lavage dans un tampon cacodylate de sodium 0,1 M pendant encore 20 min. Retirer les tissus du tampon cacodylate de sodium et les immerger dans une solution de tétroxyde d’osmium à 2% pendant 4 h à température ambiante. Ce processus est appelé osmication.
- Préparer une solution de tétroxyde d’osmium à 2 % en suivant les étapes ci-dessous.
- Pour préparer une solution de tétroxyde d’osmium à 2 %, prendre une solution de tétroxyde d’osmium à 4 %, 4 mL (voir le tableau des matières), une solution mère de cacodylate de sodium 1 M (0,8 mL) et de l’eau distillée (3,2 mL) pour obtenir une solution totale de 8 mL.
REMARQUE: Tout ce processus et les étapes à partir de là doivent être effectués dans la hotte.
- Pour préparer une solution de tétroxyde d’osmium à 2 %, prendre une solution de tétroxyde d’osmium à 4 %, 4 mL (voir le tableau des matières), une solution mère de cacodylate de sodium 1 M (0,8 mL) et de l’eau distillée (3,2 mL) pour obtenir une solution totale de 8 mL.
- Préparer une solution de tétroxyde d’osmium à 2 % en suivant les étapes ci-dessous.
- Après osmication, immerger les tissus dans une solution d’acétate de sodium à 2% pendant 10 min à température ambiante.
- Préparer une solution d’acétate de sodium à 2 % en dissolvant 4 g d’acétate de sodium dans 20 mL d’eau distillée.
- Ensuite, immerger les tissus dans une solution d’acétate d’uranyle à 2% pendant 1 h à température ambiante.
- Préparer une solution d’acétate d’uranyle à 2 % en dissolvant 4 g d’acétate d’uranyle dans 20 mL d’eau distillée.
- Après coloration à l’acétate d’uranyle, déshydrater les tissus séquentiellement à travers les alcools classés et l’acétone dans l’ordre mentionné dans le dossier supplémentaire 1.
3. Intégration du tissu cardiaque
- Intégrez les tissus déshydratés dans de la résine époxy à faible viscosité en suivant les étapes ci-dessous.
- Pour fabriquer une résine époxy à faible viscosité, mélanger 10,24 mL de dioxyde de vinylcyclohexène (ERL 4221) monomère époxy, 6,74 mL d’éther diglycidylique de polypropylène glycol (DER 732) et 30,05 mL d’anhydride nonénylsuccinique (ANE) (voir le tableau des matériaux) dans un tube de 50 mL. Bien mélanger la suspension à la main pendant 2 min.
- Ajouter 18 gouttes d’accélérateur époxy de 2-diméthylaminoéthanol (DMAE, voir le tableau des matières) et agiter la suspension pour bien mélanger les composants.
- Imprégner les tissus dans de la résine époxy dans la séquence suivante de mélange.
- Remplacez la 100% acétone par une suspension d’acétone 1:1 résine : acétone et immergez-y les tissus pendant 1 h à température ambiante.
- Remplacer la résine 1:1 : suspension d’acétone par la résine 6:1 : suspension d’acétone et y immerger les tissus pendant 3 h.
- Enfin, remplacez la suspension de résine 6:1 : acétone par une suspension 100% résine et laissez les mouchoirs immergés dedans pendant une nuit à température ambiante.
- Mettez les tissus dans de la résine fraîche dans des micromoules de 8 mm (voir Tableau des matériaux) et durcissez les tissus incrustés à 70 °C pendant la nuit.
REMARQUE: Assurez-vous que la résine est dure mais pas fragile après durcissement.
4. Sectionnement des tissus à l’aide d’un ultramicrotome
- Couper les blocs de résine avec le tissu à une taille maximale de 1 mm avant de les monter sur l’ultramicrotome (voir le tableau des matériaux). Placez le moule aussi précisément que possible dans le bras segmenté de l’ultramicrotome et avancez manuellement le moule d’échantillon vers le couteau diamanté.
- Coupez les sections à une épaisseur de 500 nm (un demi-micron) avec un couteau Histo (voir Tableau des matériaux) et, à l’aide de l’outil de boucle EM, ramassez-les et placez-les sur une lame de verre.
- Placez la lame de verre sur une plaque chauffante pour la tache bleue toluidine25 pour trouver la zone d’intérêt.
- Après avoir trouvé la zone d’intérêt, utilisez un couteau Ultra 45° (voir Tableau des matériaux) pour produire des sections ultraminces en or pâle (100 nm). Placez ces sections ultraminces sur le côté terne d’une grille de cuivre de 200 mailles.
5. Coloration ultramince de la section cardiaque
- Commencez la coloration en démasquant l’antigène à l’aide d’une solution de gravure, c’est-à-dire une solution de métaperiodate de sodium à 5%.
- Préparer 500 μL de solution de métaperiodate de sodium à 5 % (voir le tableau des matières) dans de l’eau distillée.
NOTE: Préparer la solution fraîche avant utilisation. - Mettre 20 μL de solution de métaperiodate de sodium sur un film de paraffine propre. Placez les grilles complètement séchées avec des sections de tissu sur la gouttelette de la solution de gravure.
REMARQUE: Le côté terne de la grille avec la section de tissu doit être orienté vers la solution de gravure. - Laisser la grille de section sur la solution pendant 30 min à température ambiante.
- Préparer 500 μL de solution de métaperiodate de sodium à 5 % (voir le tableau des matières) dans de l’eau distillée.
- Laver la section de tissu gravée en la plaçant sur une gouttelette d’eau distillée pendant 60 s.
- Bloquer les aldéhydes résiduels en plaçant les grilles de section sur une gouttelette de solution de glycine 0,05 M pendant 10 min à température ambiante. Éponger les bords de la grille sur du papier filtre pour éliminer la solution résiduelle de glycine.
- Préparer la solution de glycine 0,05 M (voir le tableau des matières) en dissolvant 3,75 mg de glycine dans 1 mL de 1x PBS (pH 7,4).
- Placer les grilles de section dans 10-20 μL de solution de blocage pendant 25 min à température ambiante.
NOTE: Composition de la solution bloquante: 2 μL de sérum de chèvre normal (NGS) à 1% + 20 μL de BSA à 1% (concentration finale: 10%) + 178 μL de 1x PBS (pH 7,4) pour obtenir un volume final de 200 μL. - Éponger les bords de la grille sur du papier filtre et placer la section de grille sur un diluant d’anticorps pour le conditionnement à température ambiante pendant 10 min.
- Incuber les sections de grille avec l’anticorps primaire (Sigmar1 dans ce cas, voir Tableau des matériaux; dilué 1:10 dans un diluant d’anticorps) pendant 1 h 30 min dans une chambre humidifiée.
- Sécher la grille et laver les sections de grille avec un diluant d’anticorps deux fois pendant 5 minutes chacune.
- Incuber les sections de grille avec un anticorps secondaire biotinylé (dans ce cas, anticorps secondaire polyclonal anti-lapin biotinylé de chèvre, voir le tableau des matériaux; dilué 1:10 dans un diluant d’anticorps) pendant 1 h dans une chambre humidifiée.
- Sécher la grille et laver les sections de grille avec un diluant d’anticorps deux fois pendant 5 minutes chacune.
- Incuber les sections de grille dans un QD conjugué à la streptavidine disponible dans le commerce (QD655 nm, voir le tableau des matériaux; dilué 1:10 dans un diluant d’anticorps) pendant 1 h dans une chambre humidifiée à température ambiante. Prévenez l’exposition à la lumière en recouvrant la chambre de papier d’aluminium.
- Sécher les bords de la grille avec du papier filtre et laver les sections de la grille en les plaçant sur des gouttelettes d’eau pendant 2 min.
- Épongez les bords de la grille pour les faire sécher.
6. Imagerie par microscopie électronique à transmission (MET)
- Placer les sections ultraminces sur des grilles de cuivre dans un porte-quatuor d’échantillons et les serrer de manière à permettre la sélection des grilles dans le faisceau d’électrons.
- Insérez le porte-échantillon dans la colonne du microscope et enclenchez l’interrupteur de la pompe pour évacuer le goniomètre, puis l’insertion complète du porte-échantillon dans la colonne du microscope (voir le tableau des matériaux).
- Réglez la tension à 80 kV avant de générer le faisceau pour l’observation de l’image.
- Concentrez-vous bien sur la zone souhaitée, capturez l’image à l’aide d’un appareil photo numérique haute vitesse et enregistrez le fichier dans .tif format.
REMARQUE: Le réglage du microscope pour capturer l’image dans cette étude était une tension d’accélération ou une tension haute tension de 80 kV et un grossissement de 20 000x.
Résultats
La méthodologie QD-TEM actuelle a visualisé la présence de Sigmar1 et sa localisation sur les compartiments cardiaques subcellulaires en effectuant un marquage QD ciblé anti-Sigmar1 sur des sections cardiaques ultra-minces de souris adultes. Les anticorps anti-Sigmar1 marqués QD à forte densité d’électrons sur les membranes mitochondriales (internes et externes), les lysosomes et l’interface membrane-mitochondrie du réticulum endoplasmique / réticulum sarcoplasmique (ER/SR) (Figure 2A, B) ont été illustrés par les images QD-TEM. De plus, des coupes cardiaques ont également été marquées avec des IgG et QD de lapin comme témoin d’isotype pour l’anticorps primaire anti-Sigmar1 du lapin (Figure 2C,D). Par conséquent, l’imagerie QD-TEM a mis en évidence la localisation de Sigmar1 endogène et son enrichissement principalement sur les lysosomes et les membranes mitochondriales.
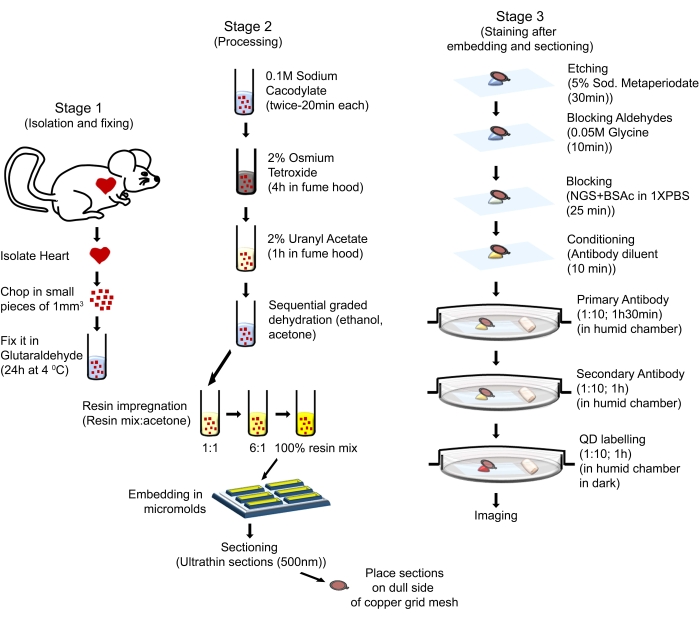
Figure 1 : Illustration schématique montrant les étapes séquentielles et les procédures utilisées pour effectuer QD-TEM. Veuillez cliquer ici pour voir une version agrandie de cette figure.
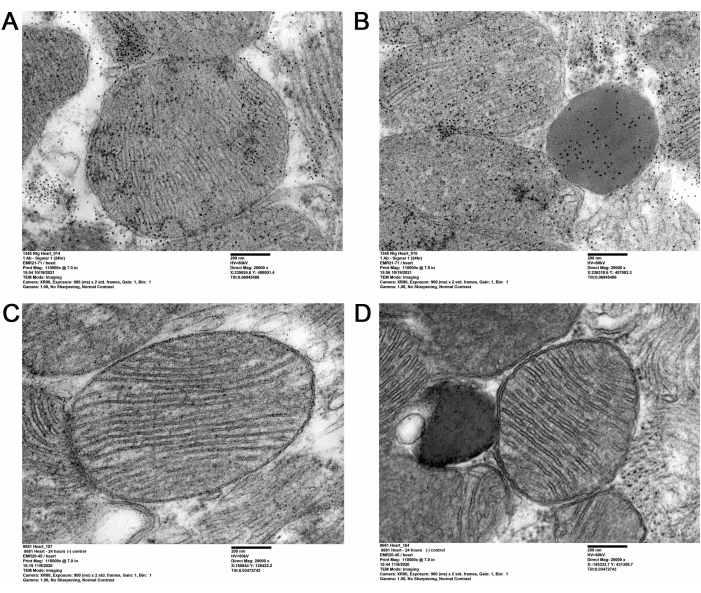
Figure 2 : QD immuno-marqué Sigmar1 dans les tissus cardiaques de souris adultes. (A,B) Images représentatives de TEM montrant Sigmar1 marqué QD sur les mitochondries (membrane externe et interne), les membranes du réticulum endoplasmique (RE) / réticulum sarcoplasmique (SR) associées aux mitochondries et les lysosomes. (C, D) Image TEM des sections cardiaques du contrôle d’isotype marquées avec QDs et IgG de lapin. Barre d’échelle: 200 nm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Fichier supplémentaire 1 : Composition du PBS et d’autres solutions utilisées pour la déshydratation des tissus. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Dans la présente étude, l’immunomarquage médié par QD a été utilisé pour montrer distinctement la localisation subcellulaire de Sigmar1. En utilisant QD, la localisation de Sigmar1 sur la membrane mitochondriale, en particulier la membrane mitochondriale interne, a été représentée dans le tissu cardiaque. De plus, Sigmar1 a également été trouvé sur le réticulum sarcoplasmique / endoplasmique (S / ER) et les lysosomes au niveau ultrastructurel, comme le montre la figure 2A-D.
Une étape critique de ce protocole est l’étape de gravure ou de démasquage de l’antigène à l’aide d’une solution hautement concentrée de métaperiodate de sodium pour démasquer l’antigène après fixation et osmication du glutaraldéhyde. Ce protocole utilisait un seul traitement avec une concentration élevée (5%) de métaperiodate de sodium pendant 30 minutes à température ambiante. Des précautions supplémentaires sont nécessaires à cette étape, car une durée plus longue ou une concentration plus élevée pour l’incubation du métaperiodate de sodium entraînera l’agrégation des structures, la perte de la définition de la membrane pour les organites et provoquera des perforations dans la section, ce qui rendra difficile la visualisation de la protéine ou de la structure. Alternativement, une concentration plus faible de (3%) solution de métaperiodate en deux étapes pendant 30 min peut également être utilisée à la place du métaperiodate à 5%. Des études ont montré que cette option présentait des résultats similaires à ceux d’une solution de métaperiodate à 5% pour une incubation en une étape de 30 minutes. Cependant, une solution de métaperiodate à 3% pendant 30 min d’incubation pendant deux fois permet un meilleur contrôle du processus26,27,28. Initialement, ce protocole utilisait l’incubation des sections avec une solution de métaperiodate à 10% pendant 30 min. Cependant, en raison d’un trop grand nombre de perforations créées dans la section tissulaire par cette concentration, la concentration finale et la durée d’incubation de la solution de métaperiodate ont été réduites et optimisées à 5% pendant 30 minutes.
Une autre étape nécessitait l’optimisation du temps de fixation avec le glutaraldéhyde. La fixation sous-optimale des tissus entraîne un étiquetage QD inadéquat, tandis qu’une fixation excessive des tissus entraîne un étiquetage non spécifique plus élevé. Par conséquent, une attention particulière doit être accordée à la détermination et au titrage d’un niveau optimal de fixation tissulaire pour un marquage approprié et spécifique des protéines. Dans cette méthode utilisant des tissus cardiaques, le temps de fixation avec le glutaraldéhyde a été titré en utilisant 24 h et 48 h comme points temporels. Sur la base des images de coloration des sections fixées pour les deux points temporels, il a été constaté que les sections fixées pour 24 h affichaient de meilleurs résultats. À ce jour, les nanocristaux QD sont disponibles en plusieurs tailles, y compris 525, 565, 585, 605, 655 et 705 nm 11,29. Chacun de ces QD a ses propres spectres d’émission et émet une fluorescence à différentes longueurs d’onde. De plus, ces QD disponibles dans le commerce de différentes tailles affichent des formes différentes; par exemple, QD 525, 565 et 585 sont pratiquement sphériques avec des tailles différentes, tandis que QD 605, 655 et 705 sont de forme oblongue irrégulière. Parmi ces différents nanocristaux QD, QD 525, 565 et 655 se distinguent facilement les uns des autres11,29. Ces différences dans les spectres et les formes d’émission font de QD un excellent candidat pour le multi-marquage des protéines et la visualisation par fluorescence et microscopie électronique. Dans cette étude, un QD disponible dans le commerce, QD 655, a été utilisé pour marquer la protéine Sigmar1 afin de la distinguer de tout fond non spécifique dans les sections colorées.
Une autre contrepartie de QD pour le marquage des protéines en microscopie à haute résolution est la particule immunogold. Les particules d’immunoor sont traditionnellement utilisées pour marquer les protéines pour la microscopie à haute résolution. Ces particules d’or sont très denses en électrons et facilement identifiables par rapport aux nanocristaux QD. Cependant, QD présente une meilleure efficacité avec une meilleure pénétration dans les tissus, une stabilité et une durée de conservation plus élevées, et une meilleure rétention des composants ultrastructuraux, ce qui en fait un meilleur candidat pour le marquage des protéines 4,5. QD a également une capacité unique à être détecté à la fois par microscopie optique et électronique, ce qui ajoute à sa valeur par rapport au marquage immunogold10.
L’une des limites de cet immunomarquage médié par QD est l’utilisation de tétroxyde d’osmium pendant le traitement. Le tétroxyde d’osmium est utilisé pour augmenter la densité électronique, la conductivité et le contraste de structures membranaires biologiques autrement moins denses en électrons et moins contrastées 5,30. Cependant, l’utilisation du tétroxyde d’osmium détruit instantanément et irréversiblement la propriété de l’échantillon de créer une fluorescence lorsqu’il est marqué avec QD6. Cela limite l’utilisation de QD en microscopie à fluorescence. Une autre approche omettant l’utilisation du tétroxyde d’osmium sera avantageuse pour conserver les propriétés fluorescentes et donc la double application de l’immunomarquage médié par QD. Certains des modèles les plus récents de TEM ont la possibilité de fixer le système d’analyse des rayons X à dispersion d’énergie (EDX) qui permet d’identifier la composition élémentaire des matériaux. Une autre limite de l’étude est l’absence de cartographie élémentaire d’un échantillon et d’analyse d’image à l’aide d’EDX. Par conséquent, les études futures devraient se concentrer sur l’analyse EDX des spectres QD pour analyser la composition élémentaire.
Le marquage QD des protéines a attiré beaucoup d’attention ces derniers temps. QD offre plusieurs applications et avantages dans la recherche biologique et la thérapeutique médicale. QD étant en outre enveloppé de ligand polydenté présente une stabilité accrue maintenant le rendement quantique. En outre, l’encapsulation de QD avec ces agents bio-favorables augmente également sa biodisponibilité dans les tissus, ce qui en fait un bon candidat pour une application potentielle dans la détection des tumeurs, l’imagerie de cellules vivantes, l’administration de médicaments et l’imagerie tissulaire 3,31,32.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été soutenu par les subventions des National Institutes of Health : R01HL145753, R01HL145753-01S1, R01HL145753-03S1 et R01HL152723; Prix de la ligne d’arrivée du CCDS LSUHSC-S, Prix de recherche sur la COVID-19 et Prix de recherche du LARC à MSB; Bourse postdoctorale Malcolm Feist de LSUHSC-S pour les maladies cardiovasculaires et l’AHA à l’ASC (20POST35210789); et LSUHSC-S Malcolm Feist Pre-doctoral Fellowship to RA.
matériels
| Name | Company | Catalog Number | Comments |
| 200 Mesh copper grid | Ted Pella | G200HH | |
| 6-month-old male mice with FVB/N background | Jackson Laboratory, Bar Harbor, ME | ||
| Acetone 100% | Fisher Chemicals | A949 | |
| Antibody diluent | Dako | S3022 | |
| anti-Sigmar1 antibody | Cell Signaling | 61994S | |
| Biotinylated goat anti-rabbit IgG antibody | Sigma Aldrich | B7389 | |
| BSAc (10%) | Electron Microscopy Sciences | 25557 | |
| Calcium chloride | Sigma Aldrich | C7902 | |
| Cytoseal Xyl | Thermo fisher | 8312-4 | |
| DER 732C36AA10:C33 | Electron Microscopy Sciences | 13010 | |
| Dextrose | Sigma Aldrich | G7528 | |
| Diamond Knife | Diatome | Histo; Ultra 450 | |
| DMAE | Electron Microscopy Sciences | 13300 | |
| Electron microscope | JEOL | JEOL-1400 Flash | |
| ERL 4221 | Electron Microscopy Sciences | 15004 | |
| Ethanol 100% | Fisher Chemicals | A405P | |
| Glutaraldehyde 3% | Electron Microscopy Sciences | 16538-15 | |
| Glycine | Alfa Aesar | A13816 | |
| Hydrochloric acid | Fisher Scientific | SA56 | |
| Micromolds | Ted Pella | 10505 | |
| Microtome | Leica Microsystem | EM UC7 | |
| Normal goat serum | Invitrogen | PCN5000 | |
| NSA | Electron Microscopy Sciences | 19050 | |
| Osmium tetroxide | Electron Microscopy Sciences | 19150 | |
| Parafilm | Genesse Scientific | 16-101 | |
| Potassium Chloride | Sigma Aldrich | P5655 | |
| Potassium Phosphate monobasic | Sigma Aldrich | 71640 | |
| Qdot 655 Streptavidin Conjugate | Invitrogen | Q10121MP | |
| Sodium Acetate | Fisher Scientific | BP334 | |
| Sodium Cacodylate | Electron Microscopy Sciences | 12300 | |
| Sodium Chloride | Fisher Scientific | BP358 | |
| Sodium metaperiodate | Sigma Aldrich | 71859 | |
| Sodium Phosphate dibasic | Sigma Aldrich | P9541 | |
| Surgical blade (size 10) | Aspen surgical | 371110 | |
| TEM image software | AMT-V700 | AMT TEM imaging systems | |
| TEM imaging camera | XR80 TEM series | AMT TEM imaging systems | |
| Toluidine Blue O solution (0.5%) | Fisher Scientic | S25612 | |
| Uranyl acetate | Polysciences | 21447 |
Références
- Lidke, D. S., Lidke, K. A. Advances in high-resolution imaging - techniques for three-dimensional imaging of cellular structures. Journal of Cell Science. 125 (11), 2571-2580 (2012).
- de Duve, C. Tissue fraction-past and present. The Journal of Cell Biology. 50 (1), 20 (1971).
- Pleskova, S., Mikheeva, E., Gornostaeva, E., Saquib, Q., Faisal, M., Al-Khedhairy, A. A., Alatar, A. A. . Cellular and Molecular Toxicology of Nanoparticles. , 323-334 (2018).
- Mayhew, T. M., Mühlfeld, C., Vanhecke, D., Ochs, M. A review of recent methods for efficiently quantifying immunogold and other nanoparticles using TEM sections through cells, tissues and organs. Annals of Anatomy - Anatomischer Anzeiger. 191 (2), 153-170 (2009).
- Kuipers, J., de Boer, P., Giepmans, B. N. G. Scanning EM of non-heavy metal stained biosamples: Large-field of view, high contrast and highly efficient immunolabeling. Experimental Cell Research. 337 (2), 202-207 (2015).
- Deerinck, T. J. The application of fluorescent quantum dots to confocal, multiphoton, and electron microscopic imaging. Toxicologic Pathology. 36 (1), 112-116 (2008).
- Nisman, R., Dellaire, G., Ren, Y., Li, R., Bazett-Jones, D. P. Application of quantum dots as probes for correlative fluorescence, conventional, and energy-filtered transmission electron microscopy. Journal of Histochemistry & Cytochemistry. 52 (1), 13-18 (2004).
- Michalet, X., et al. Quantum dots for live cells, in vivo imaging, and diagnostics. Science. 307 (5709), 538-544 (2005).
- Killingsworth, M. C., Bobryshev, Y. V. Correlative light- and electron microscopy using quantum dot nanoparticles. Journal of Visualized Experiments. (114), e54307 (2016).
- Deerinck, T. J., Giepmans, B. N. G., Smarr, B. L., Martone, M. E., Ellisman, M. H., Bruchez, M. P., Hotz, C. Z., Ellisman, M. H. . Quantum Dots: Applications in Biology. , 43-53 (2007).
- Giepmans, B. N. G., Deerinck, T. J., Smarr, B. L., Jones, Y. Z., Ellisman, M. H. Correlated light and electron microscopic imaging of multiple endogenous proteins using Quantum dots. Nature Methods. 2 (10), 743-749 (2005).
- Lidke, D. S., et al. Quantum dot ligands provide new insights into erbB/HER receptor-mediated signal transduction. Nature Biotechnology. 22 (2), 198-203 (2004).
- Pleskova, S. N., et al. Differences in the functional activity of human neutrophilic granulocytes in their interactions with semiconductor quantum dots. Morfologiia. 135 (3), 47-49 (2009).
- Crane, J., Haggie, P., Verkman, A. Quantum dot single molecule tracking reveals a wide range of diffusive motions of membrane transport proteins. Proceedings of SPIE. 7189, (2009).
- Hanaki, K. -. i., et al. Semiconductor quantum dot/albumin complex is a long-life and highly photostable endosome marker. Biochemical and Biophysical Research Communications. 302 (3), 496-501 (2003).
- Lim, Y. T., et al. . Selection of quantum dot wavelengths for biomedical assays and imaging. 2, 50-64 (2003).
- Gao, X., Cui, Y., Levenson, R. M., Chung, L. W. K., Nie, S. In vivo cancer targeting and imaging with semiconductor quantum dots. Nature Biotechnology. 22 (8), 969-976 (2004).
- Åkerman, M. E., Chan, W. C. W., Laakkonen, P., Bhatia, S. N., Ruoslahti, E. Nanocrystal targeting in vivo. Proceedings of the National Academy of Sciences. 99 (20), 12617-12621 (2002).
- Sarwat, S. A. -. O. X., Stapleton, F., Willcox, M., Roy, M. A. -. O. Quantum dots in ophthalmology: A literature review. Current Eye Research. 44 (10), 1037-1046 (2019).
- Aishwarya, R., Abdullah, C. S., Morshed, M., Remex, N. S., Bhuiyan, M. S. Sigmar1's molecular, cellular, and biological functions in regulating cellular pathophysiology. Frontiers in Physiology. 12, 705575 (2021).
- Bohne, B., Harding, G. . Processing the mouse temporal bone for gross, cellular and subcellular evaluation poster. , (2004).
- Roszkowski, M. . The effects of acute stress on Apold1 gene expression and blood-brain barrier permeability. , (2014).
- Wu, J., et al. Transcardiac perfusion of the mouse for brain tissue dissection and fixation. Bio-protocol. 11 (5), 3988 (2021).
- Jones, W. K., et al. Ablation of the murine alpha myosin heavy chain gene leads to dosage effects and functional deficits in the heart. Journal of Clinical Investigation. 98 (8), 1906-1917 (1996).
- Hunter, E. E. . Practical Electron Microscopy: A Beginner's Illustrated Guide. , (1993).
- Morris, R. E., Ciraolo, G. M. A universal post-embedding protocol for immunogold labelling of osmium-fixed, epoxy resin-embedded tissue. Journal of Electron Microscopy. 46 (4), 315-319 (1997).
- Brorson, S. H. Deplasticizing or etching of epoxy sections with different concentrations of sodium ethoxide to enhance the immunogold labeling. Micron. 32 (2), 101-105 (2001).
- Lobo, M. V. T., et al. Ultrastructural Staining with Sodium Metaperiodate and Sodium Borohydride. Journal of Histochemistry & Cytochemistry. 50 (1), 11-19 (2002).
- Bruchez, M., Moronne, M., Gin, P., Weiss, S., Alivisatos, A. P. Semiconductor nanocrystals as fluorescent biological labels. Science. 281 (5385), 2013-2016 (1998).
- Tapia, J. C., et al. High-contrast en bloc staining of neuronal tissue for field emission scanning electron microscopy. Nature Protocols. 7 (2), 193-206 (2012).
- Smith, A. M., Duan, H., Mohs, A. M., Nie, S. Bioconjugated quantum dots for in vivo molecular and cellular imaging. Advanced Drug Delivery Reviews. 60 (11), 1226-1240 (2008).
- Gour, A., Ramteke, S., Jain, N. K. Pharmaceutical applications of quantum dots. AAPS PharmSciTech. 22 (7), 233 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon