Method Article
Visualisierung der subzellulären Lokalisation eines Proteins im Herzen mittels Quantenpunkt-vermittelter Immunmarkierung gefolgt von Transmissionselektronenmikroskopie
In diesem Artikel
Zusammenfassung
Das vorliegende Protokoll beschreibt eine Methode zur Immunmarkierung eines Proteins in den Herzgewebeschnitten unter Verwendung von Quantenpunkten. Diese Technik bietet ein nützliches Werkzeug, um die subzelluläre Lokalisation und Expression eines Proteins auf ultrastruktureller Ebene zu visualisieren.
Zusammenfassung
Die subzelluläre Lokalisation ist entscheidend für die Beschreibung der richtigen Funktion und die Bestimmung der molekularen Mechanismen eines bestimmten Proteins. Mehrere qualitative und quantitative Techniken werden verwendet, um die subzelluläre Lokalisation von Proteinen zu bestimmen. Eine der aufkommenden Techniken zur Bestimmung der subzellulären Lokalisation eines Proteins ist die Quantenpunkt-(QD)-vermittelte Immunmarkierung eines Proteins, gefolgt von der Abbildung mit Transmissionselektronenmikroskopie (TEM). QD ist ein Halbleiter-Nanokristall mit einer doppelten Eigenschaft der kristallinen Struktur und der hohen Elektronendichte, was sie für die Elektronenmikroskopie anwendbar macht. Diese aktuelle Methode visualisierte die subzelluläre Lokalisation des Sigma-1-Rezeptors (Sigmar1) mittels QD-TEM im Herzgewebe auf ultrastruktureller Ebene. Kleine Würfel der Herzgewebeschnitte einer Wildtyp-Maus wurden in 3% Glutaraldehyd fixiert, anschließend osmiert, mit Uranylacetat gefärbt, gefolgt von sequentieller Dehydratisierung mit Ethanol und Aceton. Diese dehydrierten Herzgewebeschnitte wurden in niedrigviskose Epoxidharze eingebettet, in dünne Abschnitte von 500 nm Dicke geschnitten, auf das Gitter gelegt und anschließend einer Antigen-Demaskierung mit 5% Natriummetaperiodat unterzogen, gefolgt von der Abschreckung der Restaldehyde mit Glycin. Die Gewebe wurden blockiert, gefolgt von einer sequentiellen Inkubation in primären Antikörpern, biotinylierten sekundären Antikörpern und streptavidin-konjugierter QD. Diese gefärbten Abschnitte wurden getupft getrocknet und mit hoher Vergrößerung mit TEM abgebildet. Die QD-TEM-Technik ermöglichte die Visualisierung der subzellulären Lokalisation des Sigmar1-Proteins auf ultrastruktureller Ebene im Herzen. Diese Techniken können verwendet werden, um das Vorhandensein von Proteinen und subzellulären Lokalisationen in jedem Organsystem zu visualisieren.
Einleitung
Der menschliche Körper besteht aus vielen Proteinen, die für zahlreiche Körperfunktionen verantwortlich sind. Die Funktion von Proteinen hängt maßgeblich von ihrer Lokalisation im Organ und in den Zellorganellen ab. Verschiedene Techniken, einschließlich subzellulärer Fraktionierung, Immunfluoreszenz und detergensvermittelter Proteinextraktion, werden häufig verwendet, um die subzelluläre Lokalisation des Proteins zu bestimmen 1,2. Die Mikroskopie mit Immunfluoreszenzfarbstoff ist die am weitesten verbreitete Methode unter diesen Techniken. Die bei dieser Technik verwendeten Fluoreszenzfarbstoffe sind jedoch weniger stabil und anfällig für Photobleiche3. Andere Techniken beinhalteten hochauflösende Mikroskopie, um das Protein auf ultrastruktureller Ebene sichtbar zu machen, indem Proteine mit elektronendichten, Schwermetallen (Gold, Ferritin) oder Quantenpunkt-Nanokristallen immunisiert und anschließend mit Transmissionselektronenmikroskopie (TEM) sichtbar gemacht wurden4,5.
QD ist ein Halbleiter-Nanokristall, der aus Halbleitermetallverbindungen mit kontrollierbaren Photolumineszenzeigenschaften besteht, die in biologischen Systemen eine große Bedeutung haben3. QD-Nanokristalle werden in einem Kern-Schale-Format hergestellt, bei dem ein Nanokristall in die Bildung von Nanokristallen eingekapselt wird, um ihre ordnungsgemäße Stabilität und Funktion zu gewährleisten. Häufig verwendete Kern-Schale-Nanokristallkombinationen sind CdSe/ZnS, CdSe/CdS CdSe/ZnSe, CdTe/CdS, CdTe/ZnS und CdTe/CdS/ZnS (Kern/Schale/Schale)3. Unter diesen Nanokristallkombinationen werden CdSe/ZnS und CdSe/CdS am intensivsten untersucht und häufig als sekundäre Antikörperkonjugate 3,6 verwendet. Diese QD-Nanopartikel besitzen auch fluoreszierende Eigenschaften mit anderen Anregungs- und Emissionsspektren als herkömmliche Fluorophore. QD nutzt die Anregung von Elektronen aus dem Bulk-Valenzband, um im Vergleich zu herkömmlichen Fluorophoren höhere Fluoreszenzquantenausbeuten zu zeigen. Die Nanokristallanordnung von Halbleitermetallen macht QD-vermittelte Markierungen stabiler und resistenter gegen Photobleiche6. Darüber hinaus ermöglicht der Nanokristallkern in QD und seine kristalline Struktur QD unterschiedlicher Größe eine breite Palette von Absorptionsspektren und sehr enge Emissionspeaks7. Darüber hinaus sind diese QD-Partikel groß genug, um eine hohe Elektronendichte zu erzielen, was sie in hochauflösenden Mikroskopietechniken, einschließlich der Transmissionselektronenmikroskopie 5,8,9, nützlich macht. Diese QD-Nanokristalle sind auch kommerziell in mehreren Größen mit unterschiedlichen Fluoreszenzemissionsspektren und -formen erhältlich, was sie zu einem großartigen Kandidaten für die Markierung mit mehreren Antikörpernmacht 10,11.
Die QD-Technologie erlangte in der biologischen Forschung aufgrund ihrer vielfältigen funktionellen Eigenschaften große Bedeutung, einschließlich der Verwendung in der Bildgebung lebender Zellen, der Untersuchung von Transportmechanismen in der Zelle, des Membrantransports der Diffusionsbewegung des Proteins, der funktionellen Heterogenität von Zellen und der Markierung intrazellulärer Organellen 3,12,13,14,15,16 . QD ist auch nützlich in der molekularen Diagnostik für die gezielte und Erkennung von Krebsgewebe, die Charakterisierung des molekularen Profils und des Immunstatus des Tumors und die Visualisierung von Glaskörper- und Epiretinalmembranen 3,17,18. Darüber hinaus kann QD auch in medizinischen Therapien verwendet werden, um Tumormalignome durch photodynamische Therapie und ophthalmologische Anomalien zu behandeln, indem Medikamente an die Augen abgegebenwerden 3,17,18,19.
Unter Verwendung dieser hochnützlichen QD-Nanokristalle bestimmte die vorliegende Studie die subzelluläre Lokalisation eines Proteins namens Sigma-1-Rezeptor (Sigmar1). Sigmar1 ist ein ubiquitär exprimiertes multitasking-molekulares Chaperonprotein. Umfangreiche Studien, die sich auf die subzelluläre Lokalisation von Sigmar1 in verschiedenen Geweben und Organen konzentrierten, berichteten über eine zell- und gewebetypspezifische subzelluläre Lokalisation, die molekulare Funktionen auslöste20. In verschiedenen Zellen (neuronale, Photorezeptor- und Immunzellen) und Geweben (Leber und Gehirn) mit unterschiedlichen biochemischen Ansätzen berichteten Studien über die Lokalisation von Sigmar1 auf endoplasmatischem Retikulum (ER), Mitochondrien-assoziierter ER-Membran (MAM), Kernhülle, Plasmamembran, nukleoplasmatischem Retikulum, Kern und Mitochondrien. Trotz all dieser Studien blieb die subzelluläre Lokalisation von Sigmar1 im Herzen unbekannt20. Daher wurde die subzelluläre Lokalisation von Sigmar1 im Herzgewebe mittels QD-vermittelter Immunmarkierung und anschließender TEM-Bildgebung bestimmt.
Protokoll
Die Verfahren zur Handhabung von Tieren in diesem Protokoll entsprachen dem Leitfaden für die Pflege und Verwendung von Labortieren (achte Ausgabe, National Institutes of Health, Bethesda, MD) und wurden vom Animal Care and Use Committee des Louisiana State University Health Sciences Center-Shreveport genehmigt. Sechs Monate alte männliche Mäuse mit FVB / N-Hintergrund wurden für die vorliegende Studie verwendet. Die Mäuse wurden aus kommerziellen Quellen gewonnen (siehe Materialtabelle). Die verwendeten Mäuse wurden in einer gut regulierten Umgebung in Käfigen untergebracht, die einen 12-stündigen Hell-Dunkel-Zyklus ermöglichten, mit Wasser und einer regelmäßigen Chow-Diät ad libitum versorgt und gemäß dem NIH-Leitfaden für die Pflege und Verwendung der Labortiere gepflegt. Der Gesamtprozess ist in Abbildung 1 dargestellt.
1. Tierische Zubereitung
- Anästhesieren Sie die Mäuse mit 3% Isofluran. Öffnen Sie die Brust, indem Sie einen horizontalen Schnitt in der Region über dem Mittelbauch machen und die Haut ziehen. Machen Sie vorsichtig einen weiteren Schnitt, um die Brusthöhle zu öffnen, ohne andere Organe zu punktieren21,22,23.
- Perfusion24 zuerst das Herz durch die Spitze und dann den rechten Ventrikel mit kaltem 3% Glutaraldehyd in kardioplegischer Lösung (50 mM KCl, 5% Dextrose in PBS, siehe Zusatzdatei 1) für 2 min, um eine vollständige myokardiale Entspannung zu gewährleisten.
- Durchbluten Sie das Herz mit eiskaltem 3% Glutaraldehyd in 0,1 M Natriumcacodylatpuffer (pH 7,4, Schritt 2.1.1) für weitere 2 min. Verwenden Sie unter Schwerkraftdruck eine 25 G 5/8 "Nadel, um die Fixiermittel in das Herz einzuführen. Unmittelbar nachdem sich das Herz mit dem Fixiermittel gefüllt hat, heben Sie die Herzspitze an und schneiden Sie die darunter liegenden Gefäße 1-2 mm vom Herzen ab, um den Druck zu verringern und die Flüssigkeiten abfließen zu lassen.
- Sezieren Sie das Herz, entfernen Sie die Vorhöfe24 und lassen Sie die Ventrikel in eiskaltes 3% Glutaraldehyd / 0,1 M Natriumcacodylat in einer Petrischale fallen. Nach 30-60 min Fixierung einen Schmetterlingsschnitt machen und in eine Petrischale geben, die 3% Glutaraldehyd/Cacodylat enthält (siehe Materialtabelle). Hacken Sie das Herz mit einer chirurgischen Klinge in kleine Würfel von 1 mm3 , während es in Glutaraldehyd / Cacodylatlösung ist.
- Fixieren/tauchen Sie das zerlegte Herzgewebe in Glutaraldehyd/Cacodylat-Lösung für 24 h bei 4 °C.
2. Herzgewebeaufbereitung
- Nach 24 h Fixierung waschen Sie das Gewebe in 0,1 M Natriumcacodylatpuffer für 20 min.
- Bereiten Sie 0,1 M Cacodylatpuffer vor, indem Sie die folgenden Schritte ausführen.
- Zur Herstellung von 0,1 M Natriumcacodylatpuffer wird eine Brühe von 1 M Natriumcacodylatpuffer durch Auflösen von Natriumcacodylat (21,4 g) und von 1% iger Calciumchloridlösung (3 ml) in 90 ml destilliertem Wasser hergestellt. Erhöhen Sie die Lösung auf 100 ml, indem Sie das destillierte Wasser hinzufügen. Rühren Sie die Lösung gut um und lassen Sie sie über Nacht stehen, um den gelösten Stoff aufzulösen.
- Als nächstes nehmen Sie 10 ml 1 M Natriumcacodylat-Stammlösung und fügen Sie sie zu 80 ml destilliertem Wasser hinzu. Stellen Sie den pH-Wert mit HCl auf 7,4 ein und bemessen Sie ihn durch Zugabe von destilliertem Wasser auf bis zu 100 ml.
- Bereiten Sie 0,1 M Cacodylatpuffer vor, indem Sie die folgenden Schritte ausführen.
- Wiederholen Sie die Wäsche in 0,1 M Natriumcacodylatpuffer für weitere 20 Minuten. Entfernen Sie die Gewebe aus dem Natriumcacodylatpuffer und tauchen Sie sie für 4 h bei Raumtemperatur in 2% ige Osmiumtetroxidlösung. Dieser Prozess wird Osmierung genannt.
- Bereiten Sie 2% ige Osmiumtetroxidlösung wie folgt vor.
- Um 2% ige Osmiumtetroxidlösung herzustellen, nehmen Sie 4% Osmiumtetroxidlösung, 4 ml (siehe Materialtabelle), 1 M Natriumcacodylat-Stammlösung (0,8 ml) und destilliertes Wasser (3,2 ml), um insgesamt 8 ml Lösung herzustellen.
HINWEIS: Dieser gesamte Prozess und die Schritte von nun an müssen im Abzug durchgeführt werden.
- Um 2% ige Osmiumtetroxidlösung herzustellen, nehmen Sie 4% Osmiumtetroxidlösung, 4 ml (siehe Materialtabelle), 1 M Natriumcacodylat-Stammlösung (0,8 ml) und destilliertes Wasser (3,2 ml), um insgesamt 8 ml Lösung herzustellen.
- Bereiten Sie 2% ige Osmiumtetroxidlösung wie folgt vor.
- Nach der Osmulation tauchen Sie das Gewebe für 10 min bei Raumtemperatur in 2% ige Natriumacetatlösung.
- Bereiten Sie 2% ige Natriumacetatlösung vor, indem Sie 4 g Natriumacetat in 20 ml destilliertem Wasser auflösen.
- Als nächstes tauchen Sie das Gewebe für 1 h bei Raumtemperatur in 2% ige Uranylacetatlösung.
- 2%ige Uranylacetatlösung durch Auflösen von 4 g Uranylacetat in 20 ml destilliertem Wasser hergestellt.
- Nach der Uranylacetatfärbung dehydrieren Sie das Gewebe nacheinander durch die abgestuften Alkohole und das Aceton in der in der Zusatzakte 1 angegebenen Reihenfolge.
3. Einbettung von Herzgewebe
- Betten Sie das dehydrierte Gewebe in niedrigviskoses Epoxidharz ein, indem Sie die folgenden Schritte ausführen.
- Zur Herstellung von niedrigviskosem Epoxidharz mischen Sie 10,24 ml Vinylcyclohexendioxid (ERL 4221) Epoxidmonomer, 6,74 ml Diglycidylether Polypropylenglykol (DER 732) und 30,05 ml Nonenylbernsteinsäureanhydrid (NSA) (siehe Materialtabelle) in einem 50-ml-Röhrchen. Rühren Sie die Suspension 2 min lang gut von Hand um.
- Fügen Sie 18 Tropfen 2-Dimethylaminoethanol (DMAE, siehe Materialtabelle) Epoxidbeschleuniger hinzu und rühren Sie die Suspension um, um die Komponenten gründlich zu mischen.
- Imprägnant die Gewebe in Epoxidharz in der folgenden Mischungssequenz.
- Ersetzen Sie das 100% ige Aceton durch 1:1 Harz: Acetonsuspension und tauchen Sie das Gewebe für 1 h bei Raumtemperatur ein.
- Ersetzen Sie 1:1 Harz: Acetonsuspension durch 6:1 Harz: Acetonsuspension und tauchen Sie das Gewebe für 3 h hinein.
- Ersetzen Sie schließlich die 6:1-Harz-Aceton-Suspension durch eine 100% ige Harzsuspension und lassen Sie das Gewebe über Nacht bei Raumtemperatur darin eintauchen.
- Die Gewebe in 8 mm Mikroformen in frisches Harz geben (siehe Materialtabelle) und das eingebettete Gewebe bei 70 °C über Nacht aushärten.
HINWEIS: Stellen Sie sicher, dass das Harz hart, aber nach dem Aushärten nicht spröde ist.
4. Gewebeschnitt mit einem Ultramikrotom
- Schneiden Sie die Harzblöcke mit dem Gewebe auf nicht größer als 1 mm, bevor Sie sie auf das Ultramikrotom montieren (siehe Materialtabelle). Platzieren Sie die Form so präzise wie möglich im segmentierten Arm des Ultramikrotoms und schieben Sie die Probenform manuell in Richtung Diamantmesser.
- Schneiden Sie die Abschnitte mit einer Dicke von 500 nm (ein halbes Mikrometer) mit einem Histo-Messer (siehe Materialtabelle), heben Sie sie mit dem EM-Schlaufenwerkzeug auf und legen Sie sie auf einen Glasobjektträger.
- Legen Sie den Glasobjektträger auf eine Heizplatte für ToluidinblauFärbung 25 , um den interessierenden Bereich zu finden.
- Nachdem Sie den interessierenden Bereich gefunden haben, verwenden Sie ein Ultra 45°-Messer (siehe Materialtabelle), um ultradünne Schnitte aus hellem Gold (100 nm) herzustellen. Platzieren Sie diese ultradünnen Abschnitte auf der stumpfen Seite eines 200-mesh-Kupfergitters.
5. Ultradünne Herzschnittfärbung
- Beginnen Sie die Färbung, indem Sie das Antigen mit einer Ätzlösung, d. H. 5% Natriummetaperiodatlösung, demaskieren.
- 500 μL 5%ige Natriummetaperiodatlösung (siehe Materialtabelle) in destilliertem Wasser herstellen.
HINWEIS: Bereiten Sie vor Gebrauch eine frische Lösung vor. - Geben Sie 20 μL Natriummetaperiodatlösung auf einen sauberen Paraffinfilm. Legen Sie die vollständig getrockneten Gitter mit Gewebeschnitten auf den Tropfen der Ätzlösung.
HINWEIS: Die stumpfe Seite des Gitters mit dem Gewebeschnitt muss zur Ätzlösung zeigen. - Lassen Sie das Schnittgitter 30 min bei Raumtemperatur auf der Lösung.
- 500 μL 5%ige Natriummetaperiodatlösung (siehe Materialtabelle) in destilliertem Wasser herstellen.
- Waschen Sie den geätzten Gewebeabschnitt, indem Sie ihn 60 s lang auf einen Tropfen destilliertes Wasser legen.
- Blockieren Sie die Restaldehydehyde, indem Sie die Abschnittsgitter für 10 min bei Raumtemperatur auf einen Tröpfchen von 0,05 M Glycinlösung legen. Tupfen Sie die Ränder des Gitters auf Filterpapier ab, um die restliche Glycinlösung zu entfernen.
- Bereiten Sie 0,05 M Glycinlösung (siehe Materialtabelle) vor, indem Sie 3,75 mg Glycin in 1 ml 1x PBS (pH 7,4) auflösen.
- Legen Sie die Abschnittsgitter für 25 min bei Raumtemperatur in 10-20 μL Blockierlösung.
HINWEIS: Zusammensetzung der Blockierungslösung: 2 μL 1% normales Ziegenserum (NGS) + 20 μL 1% BSA (Endkonzentration: 10%) + 178 μL 1x PBS (pH 7,4), um ein Endvolumen von 200 μL zu erhalten. - Tupfen Sie die Gitterränder auf Filterpapier und legen Sie den Gitterabschnitt auf Antikörperverdünner zur Konditionierung bei Raumtemperatur für 10 min.
- Die Gitterschnitte mit primärem Antikörper (in diesem Fall Sigmar1, siehe Materialtabelle; verdünnt 1:10 in Antikörperverdünnungsmittel) für 1 h 30 min in einer befeuchteten Kammer inkubieren.
- Trocknen Sie das Rost und waschen Sie die Gitterabschnitte zweimal für jeweils 5 min mit Antikörperverdünnungsmittel.
- Die Gitterschnitte mit biotinyliertem sekundärem Antikörper (in diesem Fall biotinylierter polyklonaler Ziegenantikaninchen-Sekundärantikörper, siehe Materialtabelle; 1:10 in Antikörperverdünnungsmittel verdünnt) für 1 h in einer befeuchteten Kammer inkubieren.
- Trocknen Sie das Rost und waschen Sie die Gitterabschnitte zweimal für jeweils 5 min mit Antikörperverdünnungsmittel.
- Inkubation der Gitterschnitte in handelsüblicher Streptavidin-konjugierter QD (QD655nm, siehe Materialtabelle; verdünnt 1:10 in Antikörperverdünnungsmittel) für 1 h in einer befeuchteten Kammer bei Raumtemperatur. Verhindern Sie Lichteinwirkung, indem Sie die Kammer mit Aluminiumfolie abdecken.
- Die Gitterränder mit Filterpapier abtupfen und die Gitterabschnitte waschen, indem Sie sie 2 min auf Wassertröpfchen legen.
- Tupfen Sie die Ränder des Gitters zum Trocknen ab.
6. Bildgebung der Transmissionselektronenmikroskopie (TEM)
- Legen Sie die ultradünnen Schnitte auf Kupfergitter in einen Probenquartetthalter und klemmen Sie sie so fest, dass die Auswahl von Gittern im Elektronenstrahl möglich ist.
- Führen Sie den Probenhalter in die Mikroskopsäule ein und betätigen Sie den Pumpenschalter, um das Goniometer zu evakuieren, gefolgt von einem vollständigen Einsetzen des Probenhalters in die Mikroskopsäule (siehe Materialtabelle).
- Stellen Sie die Spannung auf 80 kV ein, bevor Sie den Strahl für die Bildbeobachtung erzeugen.
- Fokussieren Sie sich gut auf den gewünschten Bereich, nehmen Sie das Bild mit einer Hochgeschwindigkeits-Digitalkamera auf und speichern Sie die Datei in .tif Format.
HINWEIS: Die Mikroskopeinstellung zur Aufnahme des Bildes in dieser Studie war eine Beschleunigungsspannung oder Hochspannungsspannung von 80 kV und eine Vergrößerung von 20.000x.
Ergebnisse
Die vorliegende QD-TEM-Methodik visualisierte das Vorhandensein von Sigmar1 und seine Lokalisation in den subzellulären Herzkompartimenten, indem Anti-Sigmar1-gezielte QD-Markierungen an ultradünnen Herzabschnitten erwachsener Mäuse durchgeführt wurden. Elektronendichte Anti-Sigmar1-markierte QD auf mitochondrialen Membranen (innen und außen), Lysosomen und der endoplasmatischen Retikulum/sarkoplasmatischen Retikulum (ER/SR) Membran-Mitochondrien-Grenzfläche (Abbildung 2A,B) wurden durch die QD-TEM-Bilder veranschaulicht. Darüber hinaus wurden Herzabschnitte auch mit Kaninchen-IgG und QD als Isotyp-Kontrolle für Anti-Sigmar1-Kaninchen-Primärantikörper markiert (Abbildung 2C, D). Daher hob die QD-TEM-Bildgebung die Lokalisation von endogenem Sigmar1 und seine Anreicherung hauptsächlich an den Lysosomen und den Mitochondrienmembranen hervor.
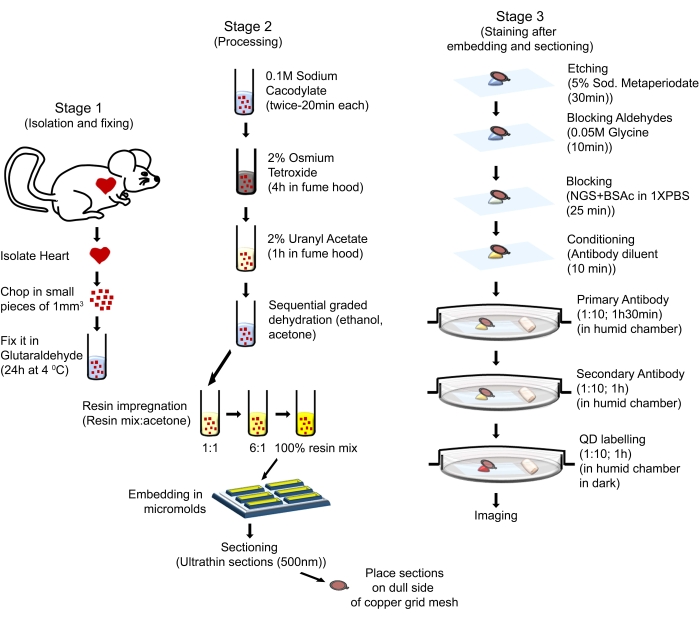
Abbildung 1: Schematische Darstellung der aufeinanderfolgenden Schritte und Verfahren zur Durchführung von QD-TEM. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
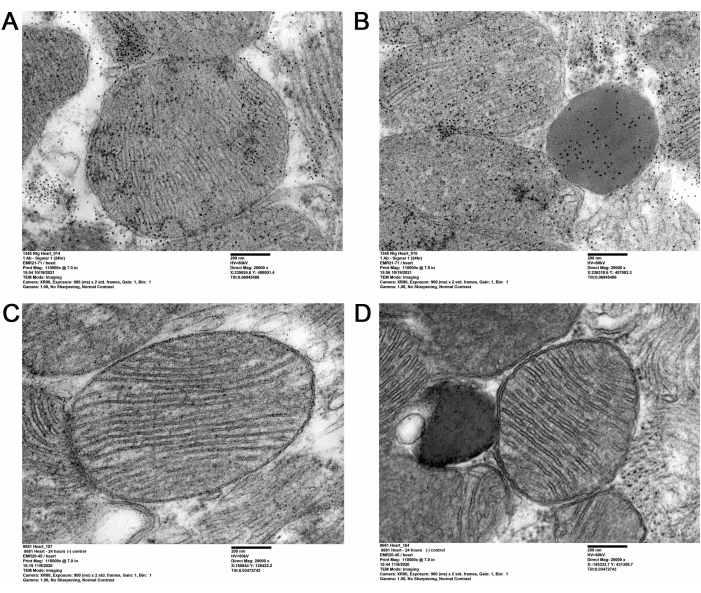
Abbildung 2: Sigmar1 immun-markierte QD in adulten Mäuseherzgeweben. (A,B) Repräsentative TEM-Bilder, die Sigmar1-markierte QD auf Mitochondrien (äußere und innere Membran), Mitochondrien-assoziierten endoplasmatischen Retikulum (ER) / sarkoplasmatischen Retikulummembranen (SR) und Lysosomen zeigen. (C,D) TEM-Aufnahme der Herzabschnitte der Isotypkontrolle, markiert mit QDs und Kaninchen-IgG. Maßstabsleiste: 200 nm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Ergänzende Datei 1: Zusammensetzung von PBS und anderen Lösungen zur Gewebetrocknung. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
In der vorliegenden Studie wurde QD-vermittelte Immunmarkierung verwendet, um die subzelluläre Lokalisation von Sigmar1 deutlich zu zeigen. Mit Hilfe von QD wurde die Lokalisation von Sigmar1 auf der mitochondrialen Membran, insbesondere der inneren Mitochondrienmembran, im Herzgewebe dargestellt. Darüber hinaus wurde festgestellt, dass Sigmar1 auch auf sarkoplasmatischen/endoplasmatischen Retikulum (S/ER) und Lysosomen auf ultrastruktureller Ebene lokalisiert ist, wie in Abbildung 2A-D gezeigt.
Ein kritischer Schritt in diesem Protokoll ist der Ätz- oder Antigen-Demaskierungsschritt unter Verwendung hochkonzentrierter Natriummetaperiodatlösung zur Demaskierung des Antigens nach Glutaraldehydfixierung und Osmierung. Dieses Protokoll verwendete eine einmalige Behandlung mit einer hohen Konzentration (5%) Natriummetaperiodat für 30 Minuten bei Raumtemperatur. In diesem Schritt ist besondere Vorsicht geboten, da eine längere Dauer oder höhere Konzentration für die Natriummetaperiodat-Inkubation zur Aggregation von Strukturen, zum Verlust der Membrandefinition für die Organellen führt und Perforationen im Abschnitt verursacht, was es schwierig macht, das Protein oder die Struktur zu visualisieren. Alternativ kann anstelle von 5% Metaperiodat auch eine niedrigere Konzentration von (3%) Metaperiodatlösung in zwei Schritten für 30 min verwendet werden. Studien haben gezeigt, dass diese Option ähnliche Ergebnisse wie bei einer 5% igen Metaperiodatlösung für eine einstufige 30-minütige Inkubation zeigt. Eine 3% ige Metaperiodatlösung für 30 Minuten Inkubation für zwei Mal bietet jedoch eine bessere Kontrolle über den Prozess26,27,28. Anfangs verwendete dieses Protokoll die Inkubation der Abschnitte mit 10% iger Metaperiodatlösung für 30 min. Aufgrund zu vieler Perforationen, die durch diese Konzentration im Gewebeschnitt entstehen, wurde jedoch die Endkonzentration und Inkubationsdauer der Metaperiodatlösung reduziert und für 30 min auf 5% optimiert.
Ein weiterer Schritt erforderte die Optimierung der Fixierungszeit mit Glutaraldehyd. Eine suboptimale Fixierung von Geweben führt zu einer unzureichenden QD-Markierung, während eine Überfixierung von Geweben zu einer höheren unspezifischen Markierung führt. Daher muss bei der Bestimmung und Titration eines optimalen Gewebefixierungsgrades für eine korrekte und spezifische Markierung von Proteinen sorgfältig abgewogen werden. Bei dieser Methode mit Herzgewebe wurde die Fixierungszeit mit Glutaraldehyd unter Verwendung von 24 h und 48 h als Zeitpunkte titriert. Basierend auf den Färbebildern der Abschnitte, die für beide Zeitpunkte festgelegt wurden, wurde festgestellt, dass Abschnitte, die für 24 Stunden fixiert waren, bessere Ergebnisse zeigten. Bis heute sind QD-Nanokristalle in verschiedenen Größen erhältlich, darunter 525, 565, 585, 605, 655 und 705 nm11,29. Jede dieser QD hat ihre eigenen Emissionsspektren und emittiert Fluoreszenz bei verschiedenen Wellenlängen. Darüber hinaus weisen diese kommerziell erhältlichen QDs unterschiedlicher Größe unterschiedliche Formen auf; Zum Beispiel sind QD 525, 565 und 585 praktisch kugelförmig mit unterschiedlichen Größen, während QD 605, 655 und 705 unregelmäßig länglich geformt sind. Von diesen verschiedenen QD-Nanokristallen sind QD 525, 565 und 655 leicht voneinander zu unterscheiden11,29. Diese Unterschiede in Emissionsspektren und -formen machen QD zu einem großartigen Kandidaten für die Multi-Markierung von Proteinen und die Visualisierung durch Fluoreszenz- und Elektronenmikroskopie. In dieser Studie wurde eine kommerziell erhältliche QD, QD 655, verwendet, um das Sigmar1-Protein zu markieren, um es von jedem unspezifischen Hintergrund in den gefärbten Abschnitten zu unterscheiden.
Ein weiteres Gegenstück zur QD für die Proteinmarkierung in der hochauflösenden Mikroskopie ist das Immunogoldpartikel. Die Immungoldpartikel werden traditionell verwendet, um Proteine für die hochauflösende Mikroskopie zu markieren. Diese Goldpartikel sind sehr elektronendicht und im Vergleich zu QD-Nanokristallen leicht identifizierbar. QD zeigt jedoch eine bessere Effizienz mit besserer Penetration in Gewebe, höherer Stabilität und Haltbarkeit und besserer Retention ultrastruktureller Komponenten, was sie zu einem besseren Kandidaten für die Proteinmarkierung macht 4,5. QD hat auch eine einzigartige Fähigkeit, sowohl durch Licht- als auch durch Elektronenmikroskopie nachgewiesen zu werden, was seinen Wert gegenüber der Immunogold-Markierung10 erhöht.
Eine Einschränkung dieser QD-vermittelten Immunmarkierung ist die Verwendung von Osmiumtetroxid während der Verarbeitung. Osmiumtetroxid wird verwendet, um die Elektronendichte, Leitfähigkeit und den Kontrast von ansonsten weniger elektronendichten und weniger kontrastierenden biologischen Membranstrukturen zu erhöhen 5,30. Die Verwendung von Osmiumtetroxid zerstört jedoch sofort und irreversibel die Eigenschaft der Probe, Fluoreszenz zu erzeugen, wenn sie mit QD6 markiert wird. Dies schränkt die Verwendung von QD in der Fluoreszenzmikroskopie ein. Ein alternativer Ansatz, der auf die Verwendung von Osmiumtetroxid verzichtet, wird vorteilhaft sein, um die fluoreszierenden Eigenschaften und damit die doppelte Anwendung der QD-vermittelten Immunmarkierung beizubehalten. Einige der neueren Modelle von TEM haben die Möglichkeit, das energiedispersive Röntgenanalysesystem (EDX) anzubringen, das die Identifizierung der Elementzusammensetzung von Materialien ermöglicht. Eine weitere Einschränkung der Studie ist das Fehlen einer Elementkartierung einer Probe und Bildanalyse mittels EDX. Daher sollten sich zukünftige Studien auf die EDX-Analyse der QD-Spektren konzentrieren, um die elementare Zusammensetzung zu analysieren.
Die QD-Markierung von Proteinen hat in letzter Zeit viel Aufmerksamkeit erregt. QD bietet mehrere Anwendungen und Vorteile sowohl in der biologischen Forschung als auch in der medizinischen Therapeutika. QD, das zusätzlich mit polyzähnigen Liganden umwickelt ist, zeigt eine erhöhte Stabilität, die die Quantenausbeute beibehält. Darüber hinaus erhöht die Verkapselung von QD mit diesen biogünstigen Wirkstoffen auch seine Bioverfügbarkeit in Geweben, was es zu einem guten Kandidaten für eine mögliche Anwendung bei der Erkennung von Tumoren, der Bildgebung lebender Zellen, der Wirkstoffabgabe und der Gewebebildgebung macht 3,31,32.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Diese Arbeit wurde durch die Zuschüsse der National Institutes of Health unterstützt: R01HL145753, R01HL145753-01S1, R01HL145753-03S1 und R01HL152723; LSUHSC-S CCDS Finish Line Award, COVID-19 Research Award und LARC Research Award an MSB; LSUHSC-S Malcolm Feist Herz-Kreislauf- und AHA-Postdoc-Stipendium für CSA (20POST35210789); und LSUHSC-S Malcolm Feist Pre-doctoral Fellowship zu RA.
Materialien
| Name | Company | Catalog Number | Comments |
| 200 Mesh copper grid | Ted Pella | G200HH | |
| 6-month-old male mice with FVB/N background | Jackson Laboratory, Bar Harbor, ME | ||
| Acetone 100% | Fisher Chemicals | A949 | |
| Antibody diluent | Dako | S3022 | |
| anti-Sigmar1 antibody | Cell Signaling | 61994S | |
| Biotinylated goat anti-rabbit IgG antibody | Sigma Aldrich | B7389 | |
| BSAc (10%) | Electron Microscopy Sciences | 25557 | |
| Calcium chloride | Sigma Aldrich | C7902 | |
| Cytoseal Xyl | Thermo fisher | 8312-4 | |
| DER 732C36AA10:C33 | Electron Microscopy Sciences | 13010 | |
| Dextrose | Sigma Aldrich | G7528 | |
| Diamond Knife | Diatome | Histo; Ultra 450 | |
| DMAE | Electron Microscopy Sciences | 13300 | |
| Electron microscope | JEOL | JEOL-1400 Flash | |
| ERL 4221 | Electron Microscopy Sciences | 15004 | |
| Ethanol 100% | Fisher Chemicals | A405P | |
| Glutaraldehyde 3% | Electron Microscopy Sciences | 16538-15 | |
| Glycine | Alfa Aesar | A13816 | |
| Hydrochloric acid | Fisher Scientific | SA56 | |
| Micromolds | Ted Pella | 10505 | |
| Microtome | Leica Microsystem | EM UC7 | |
| Normal goat serum | Invitrogen | PCN5000 | |
| NSA | Electron Microscopy Sciences | 19050 | |
| Osmium tetroxide | Electron Microscopy Sciences | 19150 | |
| Parafilm | Genesse Scientific | 16-101 | |
| Potassium Chloride | Sigma Aldrich | P5655 | |
| Potassium Phosphate monobasic | Sigma Aldrich | 71640 | |
| Qdot 655 Streptavidin Conjugate | Invitrogen | Q10121MP | |
| Sodium Acetate | Fisher Scientific | BP334 | |
| Sodium Cacodylate | Electron Microscopy Sciences | 12300 | |
| Sodium Chloride | Fisher Scientific | BP358 | |
| Sodium metaperiodate | Sigma Aldrich | 71859 | |
| Sodium Phosphate dibasic | Sigma Aldrich | P9541 | |
| Surgical blade (size 10) | Aspen surgical | 371110 | |
| TEM image software | AMT-V700 | AMT TEM imaging systems | |
| TEM imaging camera | XR80 TEM series | AMT TEM imaging systems | |
| Toluidine Blue O solution (0.5%) | Fisher Scientic | S25612 | |
| Uranyl acetate | Polysciences | 21447 |
Referenzen
- Lidke, D. S., Lidke, K. A. Advances in high-resolution imaging - techniques for three-dimensional imaging of cellular structures. Journal of Cell Science. 125 (11), 2571-2580 (2012).
- de Duve, C. Tissue fraction-past and present. The Journal of Cell Biology. 50 (1), 20 (1971).
- Pleskova, S., Mikheeva, E., Gornostaeva, E., Saquib, Q., Faisal, M., Al-Khedhairy, A. A., Alatar, A. A. . Cellular and Molecular Toxicology of Nanoparticles. , 323-334 (2018).
- Mayhew, T. M., Mühlfeld, C., Vanhecke, D., Ochs, M. A review of recent methods for efficiently quantifying immunogold and other nanoparticles using TEM sections through cells, tissues and organs. Annals of Anatomy - Anatomischer Anzeiger. 191 (2), 153-170 (2009).
- Kuipers, J., de Boer, P., Giepmans, B. N. G. Scanning EM of non-heavy metal stained biosamples: Large-field of view, high contrast and highly efficient immunolabeling. Experimental Cell Research. 337 (2), 202-207 (2015).
- Deerinck, T. J. The application of fluorescent quantum dots to confocal, multiphoton, and electron microscopic imaging. Toxicologic Pathology. 36 (1), 112-116 (2008).
- Nisman, R., Dellaire, G., Ren, Y., Li, R., Bazett-Jones, D. P. Application of quantum dots as probes for correlative fluorescence, conventional, and energy-filtered transmission electron microscopy. Journal of Histochemistry & Cytochemistry. 52 (1), 13-18 (2004).
- Michalet, X., et al. Quantum dots for live cells, in vivo imaging, and diagnostics. Science. 307 (5709), 538-544 (2005).
- Killingsworth, M. C., Bobryshev, Y. V. Correlative light- and electron microscopy using quantum dot nanoparticles. Journal of Visualized Experiments. (114), e54307 (2016).
- Deerinck, T. J., Giepmans, B. N. G., Smarr, B. L., Martone, M. E., Ellisman, M. H., Bruchez, M. P., Hotz, C. Z., Ellisman, M. H. . Quantum Dots: Applications in Biology. , 43-53 (2007).
- Giepmans, B. N. G., Deerinck, T. J., Smarr, B. L., Jones, Y. Z., Ellisman, M. H. Correlated light and electron microscopic imaging of multiple endogenous proteins using Quantum dots. Nature Methods. 2 (10), 743-749 (2005).
- Lidke, D. S., et al. Quantum dot ligands provide new insights into erbB/HER receptor-mediated signal transduction. Nature Biotechnology. 22 (2), 198-203 (2004).
- Pleskova, S. N., et al. Differences in the functional activity of human neutrophilic granulocytes in their interactions with semiconductor quantum dots. Morfologiia. 135 (3), 47-49 (2009).
- Crane, J., Haggie, P., Verkman, A. Quantum dot single molecule tracking reveals a wide range of diffusive motions of membrane transport proteins. Proceedings of SPIE. 7189, (2009).
- Hanaki, K. -. i., et al. Semiconductor quantum dot/albumin complex is a long-life and highly photostable endosome marker. Biochemical and Biophysical Research Communications. 302 (3), 496-501 (2003).
- Lim, Y. T., et al. . Selection of quantum dot wavelengths for biomedical assays and imaging. 2, 50-64 (2003).
- Gao, X., Cui, Y., Levenson, R. M., Chung, L. W. K., Nie, S. In vivo cancer targeting and imaging with semiconductor quantum dots. Nature Biotechnology. 22 (8), 969-976 (2004).
- Åkerman, M. E., Chan, W. C. W., Laakkonen, P., Bhatia, S. N., Ruoslahti, E. Nanocrystal targeting in vivo. Proceedings of the National Academy of Sciences. 99 (20), 12617-12621 (2002).
- Sarwat, S. A. -. O. X., Stapleton, F., Willcox, M., Roy, M. A. -. O. Quantum dots in ophthalmology: A literature review. Current Eye Research. 44 (10), 1037-1046 (2019).
- Aishwarya, R., Abdullah, C. S., Morshed, M., Remex, N. S., Bhuiyan, M. S. Sigmar1's molecular, cellular, and biological functions in regulating cellular pathophysiology. Frontiers in Physiology. 12, 705575 (2021).
- Bohne, B., Harding, G. . Processing the mouse temporal bone for gross, cellular and subcellular evaluation poster. , (2004).
- Roszkowski, M. . The effects of acute stress on Apold1 gene expression and blood-brain barrier permeability. , (2014).
- Wu, J., et al. Transcardiac perfusion of the mouse for brain tissue dissection and fixation. Bio-protocol. 11 (5), 3988 (2021).
- Jones, W. K., et al. Ablation of the murine alpha myosin heavy chain gene leads to dosage effects and functional deficits in the heart. Journal of Clinical Investigation. 98 (8), 1906-1917 (1996).
- Hunter, E. E. . Practical Electron Microscopy: A Beginner's Illustrated Guide. , (1993).
- Morris, R. E., Ciraolo, G. M. A universal post-embedding protocol for immunogold labelling of osmium-fixed, epoxy resin-embedded tissue. Journal of Electron Microscopy. 46 (4), 315-319 (1997).
- Brorson, S. H. Deplasticizing or etching of epoxy sections with different concentrations of sodium ethoxide to enhance the immunogold labeling. Micron. 32 (2), 101-105 (2001).
- Lobo, M. V. T., et al. Ultrastructural Staining with Sodium Metaperiodate and Sodium Borohydride. Journal of Histochemistry & Cytochemistry. 50 (1), 11-19 (2002).
- Bruchez, M., Moronne, M., Gin, P., Weiss, S., Alivisatos, A. P. Semiconductor nanocrystals as fluorescent biological labels. Science. 281 (5385), 2013-2016 (1998).
- Tapia, J. C., et al. High-contrast en bloc staining of neuronal tissue for field emission scanning electron microscopy. Nature Protocols. 7 (2), 193-206 (2012).
- Smith, A. M., Duan, H., Mohs, A. M., Nie, S. Bioconjugated quantum dots for in vivo molecular and cellular imaging. Advanced Drug Delivery Reviews. 60 (11), 1226-1240 (2008).
- Gour, A., Ramteke, S., Jain, N. K. Pharmaceutical applications of quantum dots. AAPS PharmSciTech. 22 (7), 233 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten