Method Article
Disección manual de los intestinos de Caenorhabditis elegans
En este artículo
Resumen
El presente protocolo describe un procedimiento para aislar intestinos de gusanos nematodos Caenorhabditis elegans adultos a mano para su aporte en genómica, proteómica, microbioma u otros ensayos.
Resumen
Compuesto por solo 20 células, el intestino de Caenorhabditis elegans es el nexo de muchas funciones de soporte vital, incluida la digestión, el metabolismo, el envejecimiento, la inmunidad y la respuesta ambiental. Las interacciones críticas entre el huésped de C. elegans y su entorno convergen dentro del intestino, donde se concentra la microbiota intestinal. Por lo tanto, la capacidad de aislar el tejido intestinal lejos del resto del gusano es necesaria para evaluar los procesos específicos del intestino. Este protocolo describe un método para diseccionar a mano los intestinos adultos de C. elegans. El procedimiento se puede realizar en cepas marcadas con fluorescencia para fines de facilidad o entrenamiento. Una vez que se perfecciona la técnica, los intestinos se pueden recolectar de gusanos no marcados de cualquier genotipo. Este enfoque de microdisección permite la captura simultánea del tejido intestinal del huésped y la microbiota intestinal, un beneficio para muchos estudios de microbiomas. Como tal, las aplicaciones posteriores para las preparaciones intestinales generadas por este protocolo pueden incluir, entre otras, el aislamiento de ARN de las células intestinales y el aislamiento del ADN de la microbiota capturada. En general, la disección manual de los intestinos de C. elegans ofrece un método simple y robusto para investigar aspectos críticos de la biología intestinal.
Introducción
El gusano nematodo Caenorhabditis elegans, con apenas 959 células y un ciclo de vida de 4 días de huevo a huevo, es un sistema modelo ideal para muchos estudios de genética, genómica y desarrollo 1,2. La facilidad de detección genética directa e inversa, la prevalencia de marcadores fluorescentes diseñados, la capacidad de realizar la edición del genoma específico de nucleótidos y los numerosos recursos de toda la comunidad han contribuido a los principales descubrimientos y conocimientos en el sistema de C. elegans. Sin embargo, un inconveniente importante es la dificultad de obtener poblaciones puras de células, tejidos u órganos, que son pequeños, frágiles y pueden estar interconectados. Como las poblaciones puras de células son importantes para los ensayos genómicos como RNA-seq, ChIP-seq y ATAC-seq, han surgido varios enfoques para obtener preparaciones puras de células, tejidos y órganos de C. elegans. Aquí, se describe un método para diseccionar intestinos manuales, en grandes secciones, a partir de gusanos adultos C. elegans. Las preparaciones resultantes son adecuadas para ensayos genómicos posteriores (Figura 1).
El método de disección de tejido a escala fina descrito aquí (Figura 2) es solo un enfoque. Otras técnicas alternativas, como el marcado molecular, la desagregación de gusanos y la purificación de tipos celulares de interés con clasificación celular activada por fluorescencia (FACS) y análisis post hoc, también se han utilizado con éxito para estudiar las características específicas del tejido de la biología molecular de C. elegans. Una ventaja de la disección manual sobre estos otros enfoques, sin embargo, es que puede ser utilizada para explorar simultáneamente las características del intestino de C. elegans y su contenido bacteriano 3,4,5. Esto permite la secuenciación del gen 16S rRNA y facilita los estudios de microbioma dentro del sistema de C. elegans. Una limitación importante, sin embargo, es que las células intestinales no están aisladas individualmente.
El marcado molecular imparte una etiqueta específica del tipo de célula a las moléculas solo dentro del tejido o células de interés especificados. Estas etiquetas se pueden aislar de las preparaciones de gusanos totales. De esta manera, los promotores específicos de tejido que conducen una proteína de unión a poliA marcada o un líder empalmado han permitido el perfil del transcriptoma específico del tejido 6,7,8,9,10 y el mapeo 3'UTR 11,12. Del mismo modo, se han realizado perfiles de factores de transcripción específicos de tejido utilizando ChIP-seq y DamID, en los que se agregaron variantes de factores de transcripción específicos del promotor con etiquetas o fusiones enzimáticas13,14.
FACS permite aislar los tipos celulares de interés de gusanos disociados en función de sus características celulares intrínsecas y propiedades fluorescentes. Este enfoque ha generado transcriptomas específicos de tejido de diversos órganos 8,15,16 y tipos de células neuronales individuales 8,9,15,16,17,18 y se ha utilizado para crear un mapa de expresión de todo el sistema nervioso de C. elegans 19,20 . El FACS, y su primo la clasificación de núcleos activados por fluorescencia (FANS), también se han utilizado para generar perfiles de cromatina específicos de células21,22.
Finalmente, el análisis post-hoc se puede realizar en ensayos de resolución de una sola célula. En este método, se examinan todas las células individuales, el tipo de célula de cada una se atribuye en la etapa de análisis y los tipos de células de interés se filtran selectivamente para su posterior estudio. El análisis post-hoc se ha utilizado con éxito para obtener transcriptomas de células en desarrollo con alta resolución espacial y temporal en embriones de C. elegans 23,24,25,26,27 y gusanos en etapa L1 28. La accesibilidad a la cromatina también ha sido caracterizada utilizando ATAC-seq en lugar de RNA-seq utilizando una estrategia similar29.
Cada enfoque tiene sus ventajas y limitaciones. Para el intestino de C. elegans, la desagregación de gusanos y el aislamiento de células intestinales por FACS se pueden lograr en las etapas embrionaria y larvaria30, pero es un desafío en adultos. Se cree que esto se debe a que las células grandes, endo-reduplicadas y fuertemente adherentes del intestino las hacen difíciles de disociar sin daños. El método de disección de manos descrito aquí evita estos desafíos, permitiendo el aislamiento de grandes secciones del intestino del gusano adulto. La práctica de diseccionar gónadas a mano de esta misma etapa es generalizada y directa. La disección intestinal es similar a la disección de gónadas, pero se realiza con menos frecuencia32. El protocolo presentado aquí está adaptado de un protocolo más largo e inédito desarrollado por el Dr. James McGhee y Barb Goszczynski. Este protocolo simplificado toma prestadas técnicas para aislar blastómeros de embriones en etapa temprana 23,33,34,35. Aunque la disección manual no es factible para aislar la mayoría de los tipos de células o tejidos en C. elegans, es ideal para aislar los intestinos de los gusanos adultos. Por lo tanto, la disección de la mano complementa otros medios para obtener preparaciones celulares específicas del intestino.
Protocolo
Para el presente estudio se utilizaron gusanos CL2122. Los gusanos se obtuvieron a través del Centro de Genética Caenorhabditis (CGC, ver Tabla de Materiales), financiado por la Oficina de Programas de Infraestructura de Investigación de los NIH (P40 OD010440).
1. Cultivo de gusanos para disección
- Cultivar una placa grande de gusanos CL2122 de etapa mixta para la sincronización siguiendo los procedimientos de cultivo estándar (es decir, placas NGM sembradas con E. coli OP50)36,37.
NOTA: Generalmente toma ~ 96 h para alcanzar la capacidad máxima de puesta de huevos.- El embrión prepara los gusanos dentro del período de 72-96 h. Después de la preparación del embrión, dejar que los embriones eclosionen en M9 (ver Tabla 1) durante 48 h. Esto producirá una población sincrónica de gusanos en etapa L1.
NOTA: Los gusanos CL2122 albergan un transgen-GFP integrado (proteína fluorescente verde) expulsado del promotor mtl-2 específico del intestino (MeTaLlothionein 2)38. Este promotor es específico del citoplasma de células intestinales y permite visualizar el intestino en un microscopio de disección fluorescente. Una vez entrenados, los usuarios pueden no encontrar necesaria la guía de fluorescencia.
- El embrión prepara los gusanos dentro del período de 72-96 h. Después de la preparación del embrión, dejar que los embriones eclosionen en M9 (ver Tabla 1) durante 48 h. Esto producirá una población sincrónica de gusanos en etapa L1.
- Gusanos L1 sincronizados en placa en un mínimo de dos placas pequeñas. Cultivar gusanos en placas NGM con suficiente alimento hasta que alcancen la etapa adulta (identificados por la presencia de embriones capaces de poner huevos). Esto tarda entre 38-46 h a 20 °C.
- Tenga cuidado de asegurarse de que la misma etapa de desarrollo se cosecha en las réplicas y en las cepas comparativas.
NOTA: El tiempo para cultivar los gusanos depende de la cepa, la temperatura, la fuente de alimento y la etapa de desarrollo objetivo39. También se pueden usar otras etapas cercanas a la etapa adulta, como las etapas L4 y las etapas adultas mayores. Para planificar experimentos de microbioma, se pueden utilizar diferentes fuentes de alimentos bacterianos más allá de la fuente de alimento tradicional (E. coli OP50) para cultivar gusanos, como las cepas4 de CeMbio o un patógeno de interés40.
- Tenga cuidado de asegurarse de que la misma etapa de desarrollo se cosecha en las réplicas y en las cepas comparativas.
2. Preparación de soluciones madre y pipetas microcapilares
NOTA: La Tabla 1 proporciona los detalles de todos los tampones y soluciones utilizados para el presente estudio.
- Prepare 50 ml de sales de huevo (también conocido como tampón de huevo) en un tubo cónico libre de nucleasa (consulte la Tabla de materiales). Una vez hecho, almacenar a temperatura ambiente.
- Preparar 10 ml de solución madre de levamisol de 100 mM en un tubo cónico libre de nucleasa. Una vez preparados, hacer alícuotas de 500 μL y almacenarlas a -20 °C. Las alícuotas pueden descongelarse y almacenarse a 4 °C durante 1 semana a la vez.
- Preparar 1 ml de solución de albúmina sérica bovina acetilada (ASC) de 20 mg/ml en un tubo de microcentrífuga libre de nucleasas. Una vez preparados, elaborar alícuotas de un solo uso de 50 μL y conservarlas a −20 °C.
NOTA: Es importante utilizar la versión acetilada de BSA porque esta es la única forma de BSA que está libre de nucleasa. La BSA no acetilada puede ser una fuente importante de nucleasas y, por lo tanto, conducir a la degradación de las muestras. - Preparar al menos cinco pipetas microcapilares de 50 μm y 100 μm, cada una siguiendo los pasos que se indican a continuación.
- Primero, tire de los capilares de vidrio estándar (4 pulgadas de largo y 1,2 mm de diámetro exterior) en forma de aguja de inyección usando un extractor de agujas (consulte la Tabla de materiales) y las condiciones para "Célula adherente, C. elegans y Drosophila" del libro de cocina de pipetas41 de Sutter Instruments (Condiciones: calor = rampa + 5; Pull = 100; Vel. = 75; Retraso = 90; Presión = 500; Caja de 2,5 mm x 2,5 mm).
- A continuación, forje las pipetas microcapilares a un tamaño de 50 μm (tres marcas de tick indicadas por la regla ocular de microforge bajo el objetivo M5/0.1) o de 100 μm (cinco marcas de tick en las mismas condiciones) utilizando una microforja (ver Tabla de materiales). Estos tamaños representan el diámetro de apertura estimado de la pipeta microcapilar (Figura 3).
- Luego, fije un tubo aspirador bucal a las pipetas microcapilares.
NOTA: Las pipetas aspiradoras se controlan tradicionalmente mediante pipeteo bucal, pero muchos protocolos de seguridad modernos no permiten este método. Como tal, los usuarios pueden controlar la aspiración con el tubo aspirador bucal pellizcando el tubo entre el dedo y el pulgar. Además, se puede instalar un filtro de jeringa dentro del sistema de tubos del aspirador bucal para mayor seguridad.
3. Preparación experimental
- Prepare 5 ml de tampón de disección en un tubo cónico libre de nucleasa. Conservar a temperatura ambiente.
- Preparar 1 ml de solución de BSA acetilada de trabajo en un tubo de microcentrífuga libre de nucleasa. Manténgase en hielo.
- Hacer 350 μL de solución de levamisol funcional en un tubo de microcentrífuga libre de nucleasa. Preparar por triplicado (un tubo para unos 20 gusanos). Manténgase en hielo.
- Preparar el tampón de quelación en un tubo de microcentrífuga libre de nucleasa (Tabla 1). Hacer en replicar (un tubo para unos 10 gusanos disecados). Manténgase en hielo. El uso de tampón de quelación durante las disecciones manuales mejora la calidad y cantidad de ARN (Figura 4).
- Prepare un tubo de microcentrífuga por grupo experimental que contenga 500 μL de reactivo de aislamiento de ácido nucleico o reactivo de aislamiento especificado por kit (consulte la Tabla de materiales) en un tubo libre de nucleasa. Manténgase en hielo. Este tubo se utilizará para recoger los intestinos finales aislados para su almacenamiento o uso posterior.
- Prepare un baño M9 y un baño amortiguador de disección. Para ello, obtenga dos placas de Petri estériles de 35 mm de diámetro. Luego, agregue 2 ml de M9 a un plato y 2 ml de tampón de disección al otro. Finalmente, agregue 100 μL de solución de BSA de trabajo a cada baño. Agitar para mezclar. Agregar BSA a los baños evitará que los gusanos se peguen al plástico.
- Prepare la matriz de disección. Obtenga un portaobjetos de concavidad de 2 pocillos (consulte la Tabla de materiales) y agregue 150 μL de solución de levamisol de trabajo al primer pocillo. Luego, agregue 150 μL de tampón de disección al segundo pocillo. Finalmente, agregue 20 μL de solución BSA de trabajo a cada pozo. Agregar BSA a cada pocillo evitará que los gusanos se peguen al portaobjetos.
4. Disección manual del intestino de C. elegans
- Con un recogecabezas de gusanos, mueva 20 gusanos adultos de la placa NGM al baño M9 (paso 3.6). Esto lavará las bacterias externas de los gusanos.
- Luego, mueva los 20 gusanos del baño M9 al baño tampón de disección (paso 3.6). Esto eliminará aún más las bacterias externas y equilibrará los gusanos en el tampón de disección.
- A continuación, mueva los lotes de gusanos (es decir, en conjuntos de 10) del baño tampón de disección al pocillo que contiene la solución de levamisol (paso 3.7).
NOTA: El levamisol paralizará temporalmente los gusanos.- Una vez que los movimientos del gusano disminuyan, muévalos rápidamente del pocillo de levamisol al pozo que contiene el tampón de disección (paso 3.7). Tenga cuidado de no paralizar demasiado los gusanos.
NOTA: El número de gusanos movidos en lotes a la solución de levamisol puede variar según la comodidad del usuario. Esto también es cierto para el número de gusanos recogidos inicialmente en el baño M9 y luego trasladados al baño amortiguador de disección. Es una buena práctica tener gusanos adicionales disponibles para la disección, especialmente durante el entrenamiento, a medida que el usuario aprende y se siente cómodo con el protocolo.
- Una vez que los movimientos del gusano disminuyan, muévalos rápidamente del pocillo de levamisol al pozo que contiene el tampón de disección (paso 3.7). Tenga cuidado de no paralizar demasiado los gusanos.
- Luego, permita que los gusanos comiencen a moverse un poco en el búfer de disección antes de comenzar las disecciones.
NOTA: Los intestinos no se extruyen bien de los gusanos demasiado paralizados (es decir, gusanos que no se mueven en absoluto). - Cuando esté listo, diseccione los gusanos bajo un endoscopio fluorescente con una aguja hipodérmica (es decir, 27 G x 1/2 pulg.) (consulte la Tabla de materiales) haciendo un corte justo detrás de la faringe (Figura 5Aa) o justo delante del recto (Figura 5Ab). Esto producirá dos grandes secciones del intestino, una mitad anterior-media y una mitad medio-posteriorI. Continuar este patrón para el resto de los gusanos, manteniendo el número de secciones intestinales obtenidas de las secciones anterior-media y medio-posterior equivalente hasta que se adquiera el número total deseado de intestinos.
NOTA: La colocación de la aguja hipodérmica en un cilindro vacío de jeringa de 1 ml puede ayudar en su manipulación durante las disecciones de manos. Dependiendo de la comodidad y experiencia del usuario, no es raro hacer un corte de disección que no produzca ningún fragmento intestinal. Sin embargo, con la práctica, esto se vuelve mucho menos común. - Espere aproximadamente 1 minuto para que los intestinos se extruyan al máximo del cuerpo. Por lo general, toman forma de bucle y pueden estar pegadas a una parte de las gónadas (Figura 5A). Mientras espera, agregue 50 μL de tampón de quelación al pocillo para ayudar a reducir la degradación del ARN (Figura 4).
- Mientras espera y para ayudar aún más a facilitar la extrusión intestinal, use la pipeta microcapilar de 100 μm conectada a un aspirador bucal y extraiga el intestino / gusano dentro y fuera de la pipeta (Figura 5B). Esto también ayudará a liberar el intestino de la gónada. Tenga cuidado de no perder el intestino succionándolo accidentalmente completamente en la pipeta microcapilar.
NOTA: El usuario puede alternar entre las pipetas microcapilares de 100 μm y 50 μm para liberar el intestino del resto del cuerpo y la gónada. La abertura de menor diámetro de la pipeta microcapilar de 50 μm puede proporcionar un beneficio para eliminar piezas pegajosas del intestino. Las pipetas aspiradoras se controlan tradicionalmente mediante pipeteo bucal, pero muchos protocolos de seguridad modernos no permiten este método. Como tal, los usuarios pueden controlar la aspiración con el tubo aspirador bucal pellizcando el tubo entre el dedo y el pulgar. Además, se puede instalar un filtro de jeringa dentro del sistema de tubos del aspirador bucal para mayor seguridad. - Una vez que un intestino esté suficientemente extruido, use la aguja hipodérmica de 27 G para cortarlo del resto del cuerpo y de cualquier gónada restante. El tamaño de la sección intestinal aislada puede variar mucho dependiendo de la experiencia del cosechador, la calidad del corte y el nivel de parálisis en el gusano.
NOTA: La integridad del intestino y los fragmentos intestinales se pueden monitorear fácilmente a lo largo del protocolo a través de su fluorescencia GFP. - Ahora, use la pipeta microcapilar para aspirar la sección intestinal y moverla fuera del pozo y dentro del tubo de microcentrífuga que contiene el reactivo de aislamiento de ácido nucleico o el reactivo de aislamiento especificado por el kit. Mantenga los intestinos aislados en reactivo sobre hielo y repita para los intestinos restantes.
NOTA: Se pueden agregar múltiples intestinos al tubo de microcentrífuga que contiene reactivo de aislamiento de ácido nucleico u otro reactivo de aislamiento especificado por el kit durante todo el día. Manténgase en hielo. Si no se pueden aislar todos los intestinos en 1 día, los que ya están aislados y almacenados en un reactivo de aislamiento de ácido nucleico (que preserva los ácidos nucleicos) se pueden mantener a -80 ° C hasta que se reanuden los aislamientos y / o se puedan completar. Aquí, las muestras son estables a largo plazo (es decir, de varios meses a 1 año) y pueden permanecer hasta que estén listas para aislar los ácidos nucleicos. Los intestinos se pueden cosechar en agua o en cualquier tampón de lisis específico del kit. Sin embargo, se debe tener en cuenta al determinar cuánto tiempo pueden permanecer los intestinos en un tampón dado sobre hielo (u otra temperatura deseada) antes de pasar a aplicaciones aguas abajo.
5. Aislamiento de ARN de intestinos disecados
- Homogeneizar los tejidos adquiridos almacenados en el reactivo de aislamiento de ácido nucleico realizando tres ciclos de congelación/descongelación/vórtice. Para ello, utilice un baño de perlas a 37 °C y nitrógeno líquido (u otro medio comparable).
- Luego, agregue 0.2 volúmenes de fenol: cloroformo: reactivo IAA (consulte la Tabla de materiales) a la muestra y vórtice brevemente. Por ejemplo, para un volumen de muestra inicial de 500 μL, 0,2 volúmenes de cloroformo serían 100 μL.
- Agitar el tubo a mano durante 20 s y, a continuación, incubar a temperatura ambiente durante 3 min.
- Separar las fases de la muestra por centrifugación (10.000 x g, 18 min, 4 °C).
- Retire la fase acuosa y transfiérala a un nuevo tubo de microcentrífuga libre de nucleasas.
NOTA: Tenga cuidado de no aspirar o perturbar la interfaz. - Luego, agregue un volumen igual de etanol al 100% a la fase acuosa y agite la muestra a mano durante 20 s.
- Transfiera 700 μL de la muestra a la columna de centrifugado (ver Tabla de materiales). Luego, adhiera el ARN a la columna con centrifugación (≥8,000 x g, 30 s, RT [temperatura ambiente]). Deseche el flujo continuo. Repita el procedimiento para cualquier muestra restante adicional.
- Agregue 350 μL de tampón RW1 (consulte la Tabla de materiales) a la columna para lavar la muestra. Luego, centrifugar (≥8,000 x g, 30 s, RT) y desechar el flujo continuo.
- Realizar la digestión del ADN en columna. Agregue 80 μL de DNasa I en el tampón RDD (consulte la Tabla de materiales) a la columna de muestra. Luego, incubar durante 15 minutos en RT.
- Luego, agregue 350 μL de tampón RW1 a la columna para lavar la muestra. Centrífuga (≥8,000 x g, 30 s, RT) y transferir la columna a un nuevo tubo de recolección. Deseche el tubo de recolección viejo y de flujo. Este lavado elimina la DNasa.
- Agregue 500 μL de EPR (consulte la Tabla de materiales) a la columna de muestra. A continuación, centrifugar (≥8.000 x g, 30 s, RT) y desechar el flujo. Repita para un segundo lavado.
- Luego, centrifugar durante 1 minuto adicional para secar aún más la membrana de la columna (≥8,000 x g, 1 min, RT). A continuación, transfiera la columna de muestra a un tubo de recolección de microcentrífuga sin nucleasa fresco con tapa. Deseche el tubo de recolección viejo y de flujo.
- Añadir 14 μL de agua libre de nucleasas directamente a la membrana de la columna de muestra. Luego, incubar la muestra durante 2 minutos en RT. A continuación, centrifugar (≥8.000 x g, 1 min, RT) para eluyir el ARN.
- Guarde la muestra en hielo. A continuación, evalúe la calidad y cantidad de ARN de la muestra utilizando kits de ensayo disponibles comercialmente (consulte la Tabla de materiales). Una vez hecho esto, guarde la muestra en un congelador de -80 °C.
NOTA: El ARN es generalmente estable a -80 °C durante un máximo de 1 año sin degradación.
Resultados
El presente protocolo se utilizó para aislar grandes secciones del intestino de C. elegans adultos a mano (Figura 2). La muestra final del intestino para cada grupo experimental que se muestra se compone de una colección igual de secciones del intestino anterior-medio y medio-posterior. Sin embargo, dependiendo de la pregunta experimental, también podría comprender una colección de secciones del intestino anterior, medio o posterior. En conjunto, se presentan tres resultados representativos para este protocolo. El primero representa la disección y el aislamiento exitosos de los intestinos (Figura 6). El segundo informa los resultados del aislamiento de ARN de intestinos aislados (Figura 7). El tercero muestra los resultados de la vigilancia microbiana de intestinos aislados (Figura 8).
Para el primer resultado, la Figura 6A muestra cómo se ve un intestino extruido después de hacer una incisión primaria en el sitio "a" en la Figura 5A dentro de gusanos CL2122 adultos. Como los gusanos CL2122 albergan el promotor mtl-2 específico del intestino fusionado con GFP (mtl-2 p::GFP), los intestinos aislados brillarán en verde bajo el telescopio de disección fluorescente. La disección exitosa de un segmento intestinal se muestra en la Figura 6B. Esta sección del intestino está libre de contaminantes visibles, como restos de la gónada o la carcasa. En contraste, la Figura 6C muestra intestinos que no se diseccionan con éxito, ya que la gónada y la carcasa todavía están visiblemente unidas al segmento intestinal.
Para el segundo resultado, los intestinos se cosecharon en un reactivo de aislamiento de ácido nucleico y se procesaron al día siguiente utilizando un protocolo de extracción de ARN total de baja entrada (ver Tabla de materiales). Una muestra intestinal final de 60 secciones intestinales totales de las secciones anterior-media y medio-posterior produce aproximadamente 15 ng de ARN total de alta calidad (Figura 7A). Esta cantidad de intestinos totales se puede obtener fácilmente en un solo día, pero también se puede dividir durante varios días si es necesario. Los rendimientos de ARN de la disección manual son más eficientes que la desagregación de gusanos y el aislamiento FACS de células intestinales, ya que se requieren cientos de miles de células intestinales para obtener cantidades proporcionales de ARN total (Figura 7B). Es importante destacar que los rendimientos de ARN generados por disección manual son más que suficientes como entrada para los kits comerciales de biblioteca RNA-seq (es decir, NEBNext Ultra II y NEBNext Single Cell / Low Input), que pueden tomar tan solo 2 pg de ARN.
Para el tercer resultado, los intestinos se cosecharon en ddH20 estéril y se procesaron el mismo día utilizando un kit comercial de aislamiento de ADN microbiano (ver Tabla de materiales). Una muestra intestinal final de 40 secciones intestinales totales de las secciones anterior-media y medio-posterior produce alrededor de 0,009 pg de ADN microbiano total utilizando un ensayo de detección panbacteriana (ver Tabla de materiales) (Figura 8). Estas cantidades son demasiado bajas para los métodos de cuantificación tradicionales y deben extrapolarse a partir de las curvas estándar de qPCR. Idealmente, los usuarios deben apuntar a una cantidad total final de intestinos >40, ya que esto aumenta el límite de detección y el límite de la señal a ruido con respecto a los niveles de contaminación del reactivo.
Al considerar el diseño experimental, siempre es necesario recolectar muestras de control apropiadas. Para los experimentos de transcriptómica, un control experimental adecuado puede incluir preparaciones de todo el gusano recolectado de la misma manera que los intestinos. Sin embargo, durante la disección y el aislamiento de los intestinos de C. elegans, es común ver trozos de cadáver y gónada aferrados a las secciones intestinales. Si bien idealmente estos tejidos contaminantes se eliminarán de los intestinos antes del almacenamiento para su uso posterior, los controles experimentales adicionales pueden incluir la recolección de los cadáveres de gusanos sobrantes después de la disección intestinal y / o la recolección de gónadas disecadas. Para los experimentos con microbiomas, los controles también pueden incluir preparaciones de todo el gusano recolectado de la misma manera que los intestinos, además de los controles convencionales positivos (cultivo bacteriano) y negativos (agua).
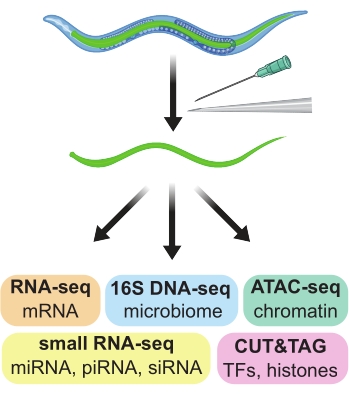
Figura 1: Disección manual de intestinos de C. elegans adultos utilizados para generar preparaciones específicas de tejido para su uso en una amplia variedad de ensayos ómicos posteriores. Haga clic aquí para ver una versión más grande de esta figura.
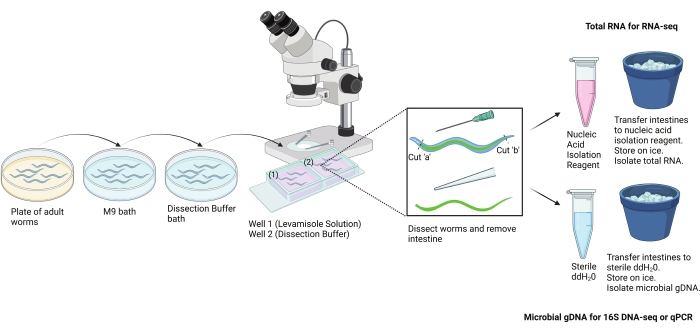
Figura 2: Esquema para el protocolo de disección manual. Este protocolo se utilizó para aislar grandes secciones del intestino de C. elegans adultos a mano. Los intestinos se pueden aislar para diferentes ensayos posteriores. Aquí se muestra el uso de intestinos para el aislamiento de ARN y el aislamiento de ADN microbiano. Imagen creada con BioRender.com. Haga clic aquí para ver una versión más grande de esta figura.
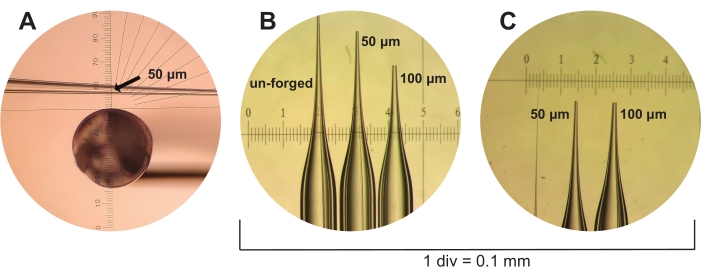
Figura 3: Fabricación de pipetas microcapilares. (A) Una pipeta microcapilar recién tirada pero sin forjar se muestra a través de la pieza ocular de una microforja. Una regla ocular se utiliza para medir pipetas microcapilares de 50 μm de tamaño a un diámetro interno estimado de tres marcas de garrapatas (mostradas, indicadas por flecha). (B) Las pipetas microcapilares no forjadas, de 50 μm y 100 μm se muestran bajo el alcance de disección junto con una regla de calibración con 1 div = 0,1 mm. (C) Las pipetas microcapilares de 50 μm y 100 μm de (B) se muestran de nuevo, pero desde un punto de vista diferente. Haga clic aquí para ver una versión más grande de esta figura.
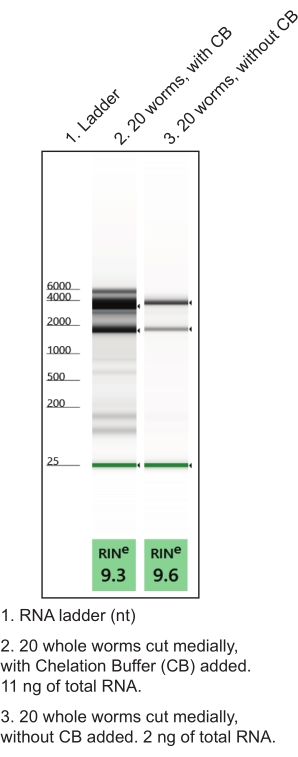
Figura 4: El uso de tampón de quelación (CB) durante la disección manual para mejorar el rendimiento de ARN. Un protocolo de extracción de ARN total de baja entrada generó preparaciones de ARN a partir de tejidos nombrados. Se muestran ejecuciones representativas de electroforesis en gel que caracterizan la calidad y cantidad de ARN total aislado (RIN, número de integridad del ARN). Haga clic aquí para ver una versión más grande de esta figura.
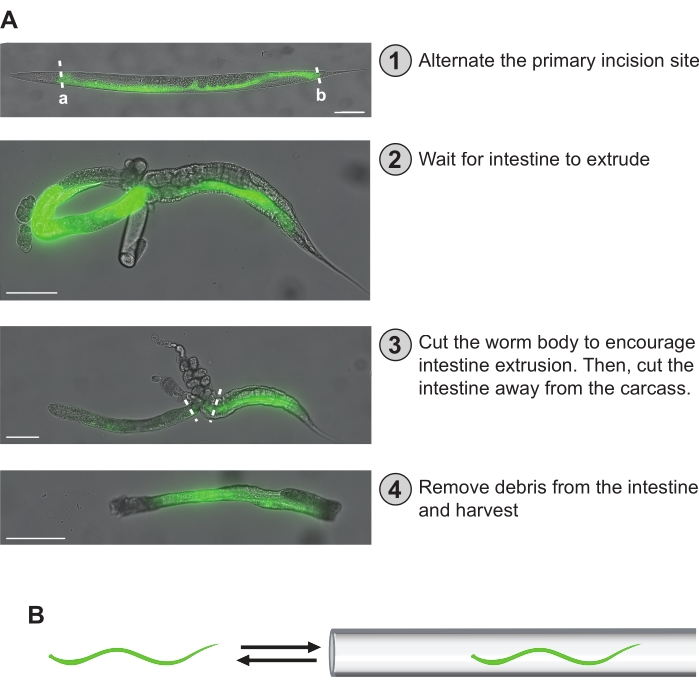
Figura 5: Pasos de disección manual. (A) Los pasos de disección descritos en el protocolo se describen aquí. En (1), el intestino del gusano se visualiza mediante fluorescencia GFP. El sitio de incisión primario se puede distribuir uniformemente entre los gusanos para garantizar una cobertura uniforme en toda la longitud del intestino. Alternativamente, se pueden seleccionar sitios de incisión primaria "a" o "b" para obtener una preparación específica de fragmento intestinal anterior-medio o medio posterior. En (2), el intestino se ha extruido, tomando forma de bucle. En (3), el intestino se corta primero del cuerpo del gusano mientras se intenta liberarlo de la carcasa y la gónada. Luego se extrae el intestino de cualquier cadáver o gónada restante que no se pueda extraer. En (4), una sección limpia del intestino está aislada y lista para su almacenamiento. (B) Un paso crítico es desalojar cualquier residuo adherente (es decir, gónada, cadáver) del intestino pasándolo dentro y fuera de la pipeta microcapilar formada al final del aspirador bucal. Barras de escala = 100 μm. Haga clic aquí para ver una versión más grande de esta figura.
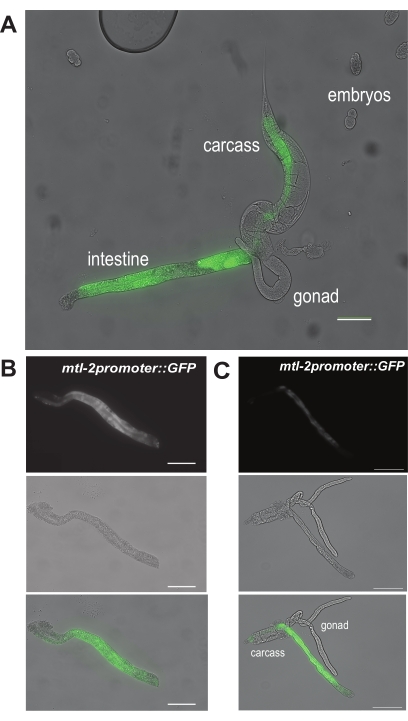
Figura 6: Resultados representativos de los pasos de disección manual. Una imagen representativa que muestra (A) la extrusión del intestino del gusano. Estos son gusanos del genotipo CL2122, que expresan GFP bajo el promotor mtl-2 específico de la célula intestinal. Dos imágenes representativas muestran (B) una sección limpia y completamente aislada del intestino y (C) un intestino aislado con gónadas contaminantes y tejidos de la canal. Barras de escala = 100 μm. Haga clic aquí para ver una versión más grande de esta figura.
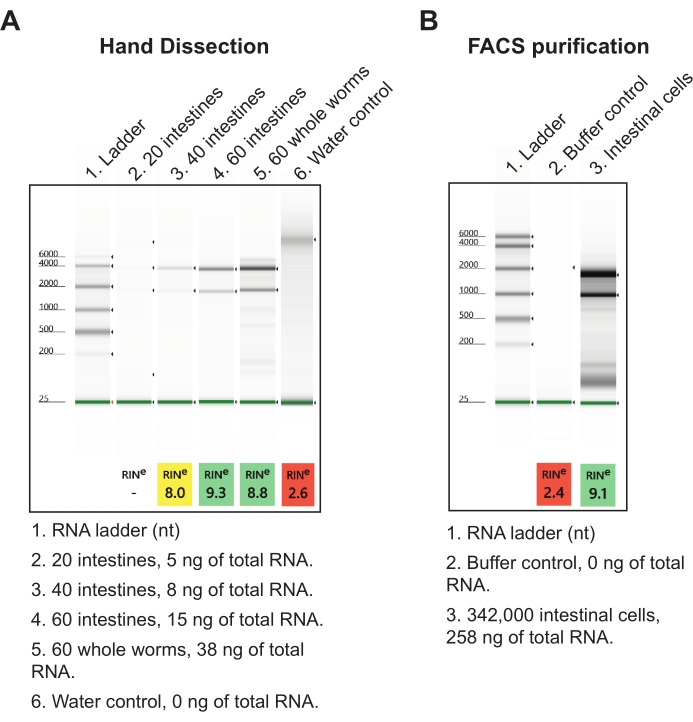
Figura 7: Resultados representativos de la extracción de ARN total de intestinos aislados. (A) Se muestra una imagen representativa de preparaciones de ARN resueltas por electroforesis en gel de agarosa, caracterizando la calidad (RINs, número de integridad de ARN) y cantidad de ARN totales aislados. (B) Se muestra para la comparación un gel representativo de ARN total aislado de células intestinales recolectadas de gusanos en etapa L1 mediante clasificación celular activada por fluorescencia (FACS). A 20 células por intestino, se puede inferir que el método de disección manual produce más ARN total por intestino que las células intestinales purificadas por FACS. Haga clic aquí para ver una versión más grande de esta figura.
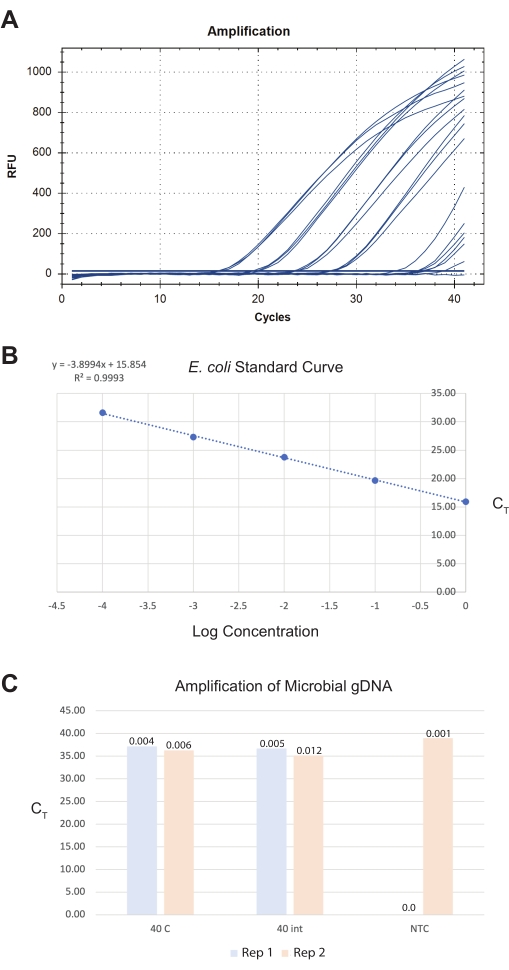
Figura 8: Resultados representativos para la extracción de ADN microbiano total de intestinos aislados. Un kit comercial de aislamiento de ADN microbiano generó preparaciones de ADN a partir de intestinos, gusanos enteros y controles. Se utilizó un ensayo genético panbacteriano para cuantificar el número de bacterias en las muestras. Una imagen representativa de (A) las curvas de amplificación de qPCR generadas, (B) la curva estándar OP50 de E. coli utilizada para cuantificar las muestras, y (C) las muestras. En (B), las concentraciones iniciales logarítmicas de los patrones de E. coli se grafican contra sus valores deC T . En (C), dos réplicas (repeticiones) de 40 gusanos enteros se cortaron medialmente, y 40 secciones intestinales aisladas se procesaron para el aislamiento microbiano de ADNg. También se muestra el No-Template-Control (NTC) de la ejecución de qPCR. El eje Y representa los valores CT de muestra. La cantidad de ADN cuantificada a partir de PCR se muestra como valores totales de picogramos (pg) por encima de las barras de muestra. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 1: Composiciones de los tampones y soluciones utilizadas en este estudio. Haga clic aquí para descargar esta tabla.
Discusión
Este artículo describe el protocolo paso a paso para la disección manual de intestinos de C. elegans adultos, generando preparaciones puras para ensayos aguas abajo. Los pasos críticos en este protocolo incluyen (1) asegurarse de no paralizar demasiado los gusanos, (2) hacer cortes de disección precisos, (3) forjar micropipetas del tamaño adecuado para la disección y (4) garantizar la rápida recuperación de intestinos sanos durante la cosecha final. Por estas razones, se debe tener cuidado al exponer los gusanos a la solución de levamisol, y las agujas hipodérmicas deben actualizarse con frecuencia para garantizar la máxima nitidez. Manipular el intestino con la pipeta microcapilar y el aspirador bucal es otro paso que requerirá práctica. Las micropipetas forjadas correctamente del tamaño adecuado también marcan una diferencia sustancial en el aislamiento de grandes secciones del intestino durante las disecciones, además de reducir el riesgo de perder intestinos dentro de la micropipeta. Los nuevos usuarios del protocolo comúnmente pierden intestinos en el borde interno de la pipeta microcapilar antes de que puedan ser expulsados al reactivo de aislamiento. Este problema se puede modificar con la práctica y pipetas microcapilares forjadas correctamente.
El protocolo descrito en este documento fue diseñado para su uso en gusanos adultos. Los ensayos preliminares apoyan que este protocolo también es efectivo para su uso en gusanos L4 y gusanos adultos mayores. Sin embargo, la eficacia de este protocolo aún no se ha evaluado en gusanos en etapa larvaria temprana. Una limitación de este enfoque es la pequeña cantidad de material que produce. Aunque las cantidades son suficientes para RNA-seq y PCR, pueden no ser adecuadas para otros ensayos. Como tal, los usuarios deben determinar si la entrada mínima requerida para un ensayo se puede recopilar de manera factible con este protocolo.
Nuestro laboratorio utiliza rutinariamente FACS para purificar las células intestinales después del aislamiento 30, métodos de análisis post-hoc para la identificación de células intestinales y este método de disección de manos30,42. La disección manual tiene la ventaja de ser susceptible de uso en gusanos adultos cuando la desagregación de gusanos y el aislamiento celular son menos exitosos. Además, la eficiencia y la calidad del ARN total extraído de las preparaciones de disección manual son altas, probablemente porque los tejidos se arrancan rápidamente de los gusanos y luego se depositan rápidamente en un reactivo de aislamiento de ácido nucleico, reduciendo la degradación del ARN. Otro beneficio del método de disección manual es que es de bajo costo, fácil de aprender y no requiere equipo especializado. Finalmente, este enfoque permite la recolección y el aislamiento de bacterias intestinales de intestinos de gusanos, lo que permite estudios de microbiomas aguas abajo.
El protocolo de disección de la mano descrito aquí para aislar los intestinos de C. elegans adultos representa una herramienta poderosa para estudiar varios aspectos de la biología de C. elegans. Por ejemplo, con una preparación pura de intestinos, los investigadores pueden investigar la intersección entre la inmunidad, el envejecimiento, el metabolismo y el microbioma.
Divulgaciones
Los autores declaran no tener conflictos de intereses.
Agradecimientos
Estamos en deuda con el trabajo pionero de James McGhee y Barb Goszczynski, quienes inicialmente desarrollaron el método de disección intestinal a partir del cual se adapta este protocolo. Nuestro trabajo está respaldado por un Premio MIRA (R35) supervisado por el Instituto Nacional de Ciencias Médicas Generales (Institutos Nacionales de Salud, R35GM124877 a EON) y un Premio NSF-CAREER supervisado por el NSF MCB Div Of Molecular and Cellular Bioscience (Premio # 2143849 a EON).
Materiales
| Name | Company | Catalog Number | Comments |
| Acetylated Bovine Serum Albumin (BSA) | VWR | 97061-420 | Nuclease free BSA |
| CL2122 worm strain | CGC (Caenorhabditis Genetics Center) | CL2122 | dvIs15 [(pPD30.38) unc-54(vector) + (pCL26) mtl-2::GFP]. Control strain for CL2120. Phenotype apparently WT. |
| Calcium Chloride Dihydrate | Fisher | C79 | needed for making Egg Salts |
| 50 mL Centrifuge Tubes, Bulk | Olympus Plastics | 28-108 | Nuclease free conical tube needed for solution making. |
| 15 mL Centrifuge Tubes, Bulk | Olympus Plastics | 28-103 | Nuclease free conical tube needed for solution making. |
| Concavity slide (2-well) | Electron Microscopy Sciences | 71878-08 | 12-pk of 2-well concavity slides |
| Ethylene glycol-bis(2-amino-ethylether)-N,N,N',N'-tetraacetic acid (EGTA) | Millipore Sigma | E3889 | needed for making chelation buffer |
| Fluorescent Dissection Microscope | Leica | M205 FCA | This is an optional piece of equipment that can be used with fluorescent C. elegans strains to help guid users during hand dissections |
| N-(2-Hydroxyethyl)piperazine-N′-(2-ethanesulfonic acid), 4-(2-Hydroxyethyl)piperazine-1-ethanesulfonic acid (HEPES) | Millipore Sigma | H4034 | needed for making dissection buffer |
| High Sensitivity RNA ScreenTape | Agilent | 5067-5579 | for assesment of total RNA quality and quantity |
| High Sensitivity RNA ScreenTape Ladder | Agilent | 5067-5581 | for assesment of total RNA quality and quantity |
| High Sensitivity RNA ScreenTape Sample Buffer | Agilent | 5067-5580 | for assesment of total RNA quality and quantity |
| HostZERO Microbial DNA Kit | Zymo Research | D4310 | Isolation of microbial DNA from worm intestines/worms |
| Hypodermic Needle (27G x 1/2") | BD Scientific | 305109 | needed for hand dissection of intestines |
| Levamisole (a.k.a. (-)-Tetramisole hydrochloride) | Millipore Sigma | L9756 | used to temporarily paralyze worms prior to hand dissection of intestines. |
| Luer-Lok General Use Disposable Syringe (1 mL) | BD Scientific | 309628 | Optional. Can be used to affix the hypodermic needle to, allowing easier manipulation of the needle during dissection. Remove the plunger. |
| Magnesium Chloride Hexahydrate | Fisher | M33 | needed for making Egg Salts |
| Magnesium Sulfate Hpetahydrate | Sigma-Aldrich | 230391-500G | needed for making M9 buffer |
| MF-900 Microforge | Narishige | MF-900 | Used to forge the microcapillary pipettes. Available through Tritech Research. |
| 1.7 mL Microtubes, Clear | Olympus Plastics | 22-282 | Nuclease free microfuge tube needed for solution making and sample storage. |
| Mouth Aspirator Tube | Millipore Sigma | A5177 | Mouth aspirator tube is needed in combination with the microcapillary pipette to allow aspiration of dissected intestines. |
| 16S Pan-Bacterial Control TaqMan Assay | Thermo Fisher | A50137 | Assay ID: Ba04930791_s1. Assay used for gut microbial detection via qPCR. |
| P-1000 Micropipette Puller | Sutter Instruments | Model P-1000 | Used to pull the microcapillary pippettes prior to forging. |
| Petri Dish (35 x 10 mm) | Genesee Scientific - Olympus Plastics | 32-103 | Used to make M9 bath and Disseciton Buffer bath for washing worms prior to dissection. |
| Phenol:Chloroform:IAA | Ambion | AM9730 | Used in the isolation of total RNA |
| Potassium Chloride | Millipore Sigma | 529552 | needed for making Egg Salts |
| Potassium Phosphate Monobasic | Sigma-Aldrich | P0662-500G | needed for making M9 buffer |
| Qubit 3 Fluorometer | Invitrogen | Q33216 | Accompanies the Qubit RNA HS Assay Kit. Can be used to quantify RNA prior to running sample on the Agilent ScreenTape. |
| Qubit RNA HS Assay Kit | Invitrogen | Q32852 | Can be used to quantify RNA prior to running sample on the Agilent ScreenTape. |
| RNasin Ribonuclease Inhibitor | Promega | N2111 | Broad spectrum inhibition of common eukaryotic Rnases |
| RNase-Free DNase Set | Qiagen | 79254 | used for on-column DNA digestion during RNA isolation protocol. |
| RNeasy Micro Kit | Qiagen | 74004 | Used for isolation of total RNA from worm intestines/worms |
| Standard Glass Capillaries | World Precision Instruments | 1B100F-4 | 4 in OD 1.2 mm standard borosilicate glass capillaries used to make microcapillary pipettes for dissection |
| Sodium Chloride | Fisher | S271 | needed for making Egg Salts |
| Sodium phosphate dibasic heptahydrate | Fisher Scientific | S373-500 | needed for making M9 buffer |
| Syringe filter (0.2 micrometer SCFA) | Thermo Fisher | 72302520 | Optional for use with the mouth aspirator tube when mouth pipetting. |
| 4150 TapeStation System | Agilent | G2992AA | Accompanies the RNA ScreenTape reagents for assessing RNA quality and quantity |
| TaqPath BactoPure Microbial Detection Master Mix | Applied Biosystems | A52699 | master mix used for qPCR |
| TRIzol Reagent | Thermo Fisher Scientific | 15596026 | Nucleic acid isolation and preservation. QIAzol (Qiagen; 79306) can be substituted if preferred. |
| Worm Pick | NA | NA | Made in house from a pasteur pipette and a platinum wire. See wormbook for details. |
Referencias
- Riddel, D. L., Blumenthal, T., Meyer, B. J., Priess, J. . C. elegans II. , (1997).
- Corsi, A. K., Wightman, B., Chalfie, M. A transparent window into biology: A primer on Caenorhabditis elegans. Genetics. 200 (2), 387-407 (2015).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 485 (2017).
- Roy, P. J., Stuart, J. M., Lund, J., Kim, S. K. Chromosomal clustering of muscle-expressed genes in Caenorhabditis elegans. Nature. 418 (6901), 975-979 (2002).
- Pauli, F., Liu, Y., Kim, Y. A., Chen, P. -. J., Kim, S. K. Chromosomal clustering and GATA transcriptional regulation of intestine-expressed genes in C. elegans. Development. 133 (2), 287-295 (2005).
- Spencer, W. C., et al. A spatial and temporal map of C. elegans gene expression. Genome Research. 21 (2), 325-341 (2011).
- Spencer, W. C., et al. Isolation of specific neurons from C. elegans larvae for gene expression profiling. PLoS One. 9 (11), 112102 (2014).
- Ma, X., et al. Analysis of C. elegans muscle transcriptome using trans-splicing-based RNA tagging (SRT). Nucleic Acids Research. 44 (21), 156 (2016).
- Blazie, S. M., et al. Comparative RNA-Seq analysis reveals pervasive tissue-specific alternative polyadenylation in Caenorhabditis elegans intestine and muscles. BMC Biology. 13 (1), 4 (2015).
- Blazie, S. M., et al. Alternative polyadenylation directs tissue-specific miRNA targeting in Caenorhabditis elegans somatic tissues. Genetics. 206 (2), 757-774 (2017).
- Gómez-Saldivar, G., et al. et al.Tissue-specific transcription footprinting using RNA PoI DamID (RAPID) in Caenorhabditis elegans. Genetics. 216 (4), 931-945 (2020).
- Katsanos, D., Barkoulas, M. Targeted DamID in C. elegans reveals a direct role for LIN-22 and NHR-25 in antagonizing the epidermal stem cell fate. Science Advances. 8 (5), 3141 (2022).
- Kaletsky, R., et al. The C. elegans adult neuronal IIS/FOXO transcriptome reveals adult phenotype regulators. Nature. 529 (7584), 92-96 (2015).
- Kaletsky, R., et al. Transcriptome analysis of adult Caenorhabditis elegans cells reveals tissue-specific gene and isoform expression. PLoS Genetics. 14 (8), 1007559 (2018).
- Mathies, L. D., et al. mRNA profiling reveals significant transcriptional differences between a multipotent progenitor and its differentiated sister. BMC Genomics. 20 (1), 427 (2019).
- Liang, X., Calovich-Benne, C., Norris, A. Sensory neuron transcriptomes reveal complex neuron-specific function and regulation of mec-2/ Stomatin splicing. Nucleic Acids Research. 50 (5), 2401-2416 (2021).
- Glenwinkel, L., et al. In silico analysis of the transcriptional regulatory logic of neuronal identity specification throughout the C. elegans nervous system. eLife. 10, 64906 (2021).
- Taylor, S. R., et al. Molecular topography of an entire nervous system. Cell. 184 (16), 4329-4347 (2021).
- Charest, J., et al. Combinatorial action of temporally segregated transcription factors. Developmental Cell. 55 (4), 483-499 (2020).
- Steiner, F. A., Talbert, P. B., Kasinathan, S., Deal, R. B., Henikoff, S. Cell-type-specific nuclei purification from whole animals for genome-wide expression and chromatin profiling. Genome Research. 22 (4), 766-777 (2012).
- Tintori, S. C., Nishimura, E. O., Golden, P., Lieb, J. D., Goldstein, B. A transcriptional lineage of the early C. embryo. Developmental Cell. 38 (4), 430-444 (2016).
- Hashimshony, T., Wagner, F., Sher, N., Yanai, I. CEL-Seq: Single-cell RNA-Seq by multiplexed linear amplification. Cell Reports. 2 (3), 666-673 (2012).
- Hashimshony, T., Feder, M., Levin, M., Hall, B. K., Yanai, I. Spatiotemporal transcriptomics reveals the evolutionary history of the endoderm germ layer. Nature. 519 (7542), 219-222 (2015).
- Packer, J. S., et al. A lineage-resolved molecular atlas of C. elegans embryogenesis at single-cell resolution. Science. 365 (6459), 1971 (2019).
- Warner, A. D., Gevirtzman, L., Hillier, L. W., Ewing, B., Waterston, R. H. The C. elegans embryonic transcriptome with tissue, time, and alternative splicing resolution. Genome Research. 29 (6), 1036-1045 (2019).
- Cao, J., et al. Comprehensive single-cell transcriptional profiling of a multicellular organism. Science. 357 (6352), 661-667 (2017).
- Durham, T. J., et al. Comprehensive characterization of tissue-specific chromatin accessibility in L2 Caenorhabditis elegans nematodes. Genome Research. 31 (10), 1952-1969 (2021).
- King, D. C., et al. The transcription factor ELT-2 positively and negatively impacts direct target genes to modulate the Caenorhabditis elegans intestinal transcriptome. bioRxiv. , (2021).
- Kocsisova, Z., Mohammad, A., Kornfeld, K., Schedl, T. Cell cycle analysis in the C. elegans germline with the thymidine analog EdU. Journal of Visualized Experiments. (140), e58339 (2018).
- Han, S., et al. Mono-unsaturated fatty acids link H3K4me3 modifiers to C. elegans lifespan. Nature. 544 (7649), 185-190 (2017).
- Edgar, L. G., Wolf, N., Wood, W. B. Early transcription in Caenorhabditis elegans embryos. Development. 120 (2), 443-451 (1994).
- Edgar, L. G., Goldstein, B. Culture and manipulation of embryonic cells. Methods in Cell Biology. 107, 151-175 (2012).
- Nishimura, E. O., Zhang, J. C., Werts, A. D., Goldstein, B., Lieb, J. D. Asymmetric transcript discovery by RNA-seq in C. elegans blastomeres identifies neg-1, a gene important for anterior morphogenesis. PLoS Genetics. 11 (4), 1005117 (2015).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Cerón, J. Basic Caenorhabditis elegans methods: Synchronization and observation. Journal of Visualized Experiments. (64), e4019 (2012).
- Fay, D. S., Fluet, A., Johnson, C. J., Link, C. D. In vivo aggregation of β-amyloid peptide variants. Journal of Neurochemistry. 71 (4), 1616-1625 (1998).
- Altun, Z. F., Hall, D. H. WormAtas Hermaphrodite Handbook – Introduction to C. elegans Anatomy. WormAtlas. , (2006).
- Tan, M. -. W., Mahajan-Miklos, S., Ausubel, F. M. Killing of Caenorhabditis elegans by Pseudomonas aeruginosa used to model mammalian bacterial pathogenesis. Proceedings of the National Academy of Sciences of the United States of America. 96 (2), 715-720 (1999).
- Oesterle, A. Pipette Cookbook 2018: P-97 & P-1000 Micropipette Pullers. Sutter Instrument Company. , (2018).
- Dineen, A., Nishimura, E. O., Goszczynski, B., Rothman, J. H., McGhee, J. D. Quantitating transcription factor redundancy: The relative roles of the ELT-2 and ELT-7 GATA factors in the C. elegans endoderm. Developmental Biology. 435 (2), 150-161 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados