Method Article
Caenorhabditis elegans 내장의 손 해부
요약
본 프로토콜은 유전체학, 단백질체학, 마이크로바이옴 또는 기타 분석에 입력하기 위해 손으로 성인 Caenorhabditis elegans 선충으로부터 장을 분리하는 절차를 설명합니다.
초록
단 20개의 세포로 구성된 Caenorhabditis elegans 장은 소화, 신진대사, 노화, 면역 및 환경 반응을 포함한 많은 생명 유지 기능의 연결체입니다. C. elegans 숙주와 그 환경 사이의 중요한 상호 작용은 장내 미생물이 집중되는 장 내에서 수렴됩니다. 따라서 장 조직을 나머지 웜에서 분리하는 능력은 장 특이적 과정을 평가하는 데 필요합니다. 이 프로토콜은 성인 C. elegans 내장을 손으로 해부하는 방법을 설명합니다. 절차는 용이함 또는 훈련 목적을 위해 형광 표지된 균주에서 수행할 수 있습니다. 기술이 완성되면 모든 유전자형의 표지되지 않은 벌레에서 장을 수집 할 수 있습니다. 이 미세 해부 접근법은 숙주 장 조직과 장내 미생물총을 동시에 포획할 수 있게 하며, 이는 많은 미생물군집 연구에 도움이 됩니다. 이와 같이, 이 프로토콜에 의해 생성된 장내 제제에 대한 다운스트림 적용은 장 세포로부터의 RNA 분리 및 포획된 미생물총으로부터의 DNA 분리를 포함할 수 있지만 이에 국한되지는 않는다. 전반적으로 C. elegans 장의 손 해부는 장 생물학의 중요한 측면을 조사하는 간단하고 강력한 방법을 제공합니다.
서문
Caenorhabditis elegans 선충류는 959개의 세포와 4일의 난자 대 계란 수명 주기를 가지며 많은 유전학, 유전체학 및 발달 연구 1,2에 이상적인 모델 시스템입니다. 정방향 및 역방향 유전자 스크리닝의 용이성, 조작된 형광 마커의 보급, 뉴클레오티드 특이적 게놈 편집을 수행할 수 있는 능력, 수많은 커뮤니티 전체 리소스는 모두 C. elegans 시스템의 주요 발견과 통찰력에 기여했습니다. 그러나 중요한 단점은 작고 깨지기 쉽고 상호 연결될 수있는 세포, 조직 또는 기관의 순수한 집단을 얻는 것이 어렵다는 것입니다. RNA-seq, ChIP-seq 및 ATAC-seq와 같은 유전체학 분석에는 순수한 세포 집단이 중요하기 때문에 C. elegans 세포, 조직 및 기관의 순수한 제제를 얻기 위한 몇 가지 접근 방식이 등장했습니다. 여기에서는 창자를 큰 부분으로 손으로 해부하는 방법을 성인 C. elegans 벌레에서 설명합니다. 결과 제제는 다운스트림 유전체학 분석에 적합합니다(그림 1).
여기에 설명된 미세 조직 해부 방법(그림 2)은 한 가지 접근 방식일 뿐입니다. 분자 태깅, 웜 분해, 형광 활성화 세포 분류(FACS) 및 사후 분석을 통한 관심 세포 유형 정제와 같은 다른 대체 기술도 C. elegans 분자 생물학의 조직 특이적 특징을 조사하는 데 성공적으로 사용되었습니다. 그러나 이러한 다른 접근법에 비해 손 해부의 장점은 C. elegans 장의 특징과 박테리아 내용물 3,4,5를 동시에 탐색하는 데 사용할 수 있다는 것입니다. 이를 통해 16S rRNA 유전자 시퀀싱이 가능하고 C. elegans 시스템 내에서 마이크로바이옴 연구를 용이하게 합니다. 그러나 중요한 한계는 장 세포가 개별적으로 분리되지 않는다는 것입니다.
분자 태깅은 지정된 조직 또는 관심 세포 내에서만 분자에 세포 유형별 태그를 부여합니다. 그런 다음 이러한 태그를 전체 웜 준비에서 분리할 수 있습니다. 이러한 방식으로, 태그된 폴리A-결합 단백질 또는 스플라이싱된 리더를 구동하는 조직-특이적 프로모터는 조직-특이적 전사체 프로파일링 6,7,8,9,10 및 3'UTR 맵핑(11,12)을 가능하게 하였다. 유사하게, 조직-특이적 전사 인자 프로파일은 ChIP-seq 및 DamID를 사용하여 수행되었으며, 여기서 프로모터-특이적 전사 인자 변이체는 태그 또는 효소 융합체13,14로 추가되었다.
FACS는 고유한 세포 특성 및 형광 특성에 기초하여 해리된 웜으로부터 관심 세포 유형을 분리할 수 있게 한다. 이 접근법은 다양한 기관 8,15,16 및 개별 신경 세포 유형 8,9,15,16,17,18에서 조직 특이 적 전사체를 생성했으며 전체 C. elegans 신경계의 발현지도를 만드는 데 사용되었습니다 19,20 . FACS 및 그의 사촌 형광-활성화 핵 분류(FANS)는 또한 세포-특이적 염색질 프로파일21,22를 생성하는데 사용되었다.
마지막으로, 사후 분석은 단일 세포 분해능 분석에서 수행할 수 있습니다. 이 방법에서는 모든 개별 세포를 조사하고 각각의 세포 유형을 분석 단계에 귀속하며 추가 연구를 위해 관심 세포 유형을 선택적으로 필터링합니다. 사후 분석은 C. elegans 배아23,24,25,26,27 및 L1 28 단계 웜에서 높은 공간 및 시간 해상도를 가진 발달 세포의 전사체를 얻는 데 성공적으로 사용되었습니다. 염색질 접근성은 또한 유사한 전략29를 사용하여 RNA-seq 대신 ATAC-seq를 사용하여 특성화되었습니다.
각 접근 방식에는 장점과 한계가 있습니다. C. elegans 장의 경우 장 세포의 웜 분해 및 FACS 분리는 배아 및 유충 단계30에서 달성 할 수 있지만 성인에서는 어렵습니다. 이것은 장의 크고 내로 복제되며 강하게 부착 된 세포로 인해 손상되지 않은 상태로 해리되기 어렵 기 때문인 것으로 생각됩니다. 여기에 설명 된 손 해부 방법은 이러한 문제를 우회하여 성인 벌레 장의 많은 부분을 분리 할 수 있습니다. 이 같은 단계에서 생식선을 손으로 해부하는 관행은 광범위하고 간단합니다. 장 박리는 생식선 박리와 유사하지만 덜 일반적으로 수행됩니다32. 여기에 제시된 프로토콜은 James McGhee 박사와 Barb Goszczynski가 개발 한 더 긴 미공개 프로토콜에서 채택되었습니다. 이 간소화 된 프로토콜은 초기 단계 배아 23,33,34,35에서 분열구를 분리하는 기술을 차용합니다. 손 해부는 C. elegans에서 대부분의 세포 또는 조직 유형을 분리하는 데 적합하지 않지만 성인 벌레로부터 장을 분리하는 데 이상적입니다. 따라서 손 해부는 장 특이 적 세포 제제를 얻기위한 다른 수단을 보완합니다.
프로토콜
CL2122 웜을 본 연구에 사용하였다. 벌레는 NIH 연구 인프라 프로그램 사무소 (P40 OD010440)에서 자금을 지원하는 Caenorhabditis Genetics Center (CGC, 재료 표 참조)를 통해 획득되었습니다.
1. 해부를위한 벌레의 성장
- 표준 배양 절차 (즉, 대장균 OP2122로 파종 된 NGM 플레이트)36,37에 따라 동기화를 위해 혼합 단계 CL50 웜의 큰 플레이트 하나를 성장시킵니다.
알림: 일반적으로 최대 알을 낳는 능력에 도달하는 데 ~ 96 시간이 걸립니다.- 배아는 72-96 시간 내에 벌레를 준비합니다. 배아 준비 후 배아가 M9 ( 표 1 참조)에서 48 시간 동안 부화하도록합니다. 이렇게 하면 L1 단계 웜의 동기 모집단이 생성됩니다.
참고: CL2122 웜은 장 특이적 mtl-2 (MeTaLlothionein 2) 프로모터38에서 구동되는 통합 전이유전자-GFP(녹색 형광 단백질)를 보유하고 있습니다. 이 프로모터는 장 세포질에 특이적이며 장을 형광 해부 현미경으로 시각화할 수 있습니다. 일단 교육을 받으면 사용자는 형광 지침이 필요하다고 생각하지 않을 수 있습니다.
- 배아는 72-96 시간 내에 벌레를 준비합니다. 배아 준비 후 배아가 M9 ( 표 1 참조)에서 48 시간 동안 부화하도록합니다. 이렇게 하면 L1 단계 웜의 동기 모집단이 생성됩니다.
- 최소 두 개의 작은 플레이트에 동기화된 L1 웜을 플레이트합니다. 성인기에 도달 할 때까지 충분한 음식으로 NGM 플레이트에서 벌레를 키울 수 있습니다 (알을 낳을 수있는 배아의 존재로 확인됨). 20 ° C에서 38-46 시간이 걸립니다.
- 동일한 발달 단계가 반복실험과 비교 균주 간에 수확되도록 주의하십시오.
참고: 벌레를 키우는 데 걸리는 시간은 균주, 온도, 먹이 공급원 및 대상 발달 단계에 따라 다릅니다39. L4 및 노인 단계와 같은 성인 단계 근처의 다른 단계도 사용할 수 있습니다. 마이크로바이옴 실험을 계획하기 위해 전통적인 식품 공급원(E. coli OP50)을 넘어 다양한 박테리아 식품 공급원을 사용하여 CeMbio 균주4 또는 관심 병원체40와 같은 벌레를 성장시킬 수 있습니다.
- 동일한 발달 단계가 반복실험과 비교 균주 간에 수확되도록 주의하십시오.
2. 원액 및 미세모세관 피펫의 준비
참고: 표 1 은 본 연구에 사용된 모든 완충액 및 용액의 세부 정보를 제공합니다.
- 뉴클레아제가 없는 원뿔형 튜브에 50mL의 계란 소금(일명 계란 완충액)을 준비합니다( 재료 표 참조). 일단 만들어지면 실온에서 보관하십시오.
- 뉴클레아제가 없는 원뿔형 튜브에 100mM 레바미솔 원액 10mL를 준비합니다. 일단 준비되면 500 μL 분취량을 만들어 -20 ° C에서 보관하십시오. 분취량은 해동되어 4°C에서 한 번에 1주일 동안 보관할 수 있습니다.
- 뉴클레아제가 없는 미세 원심분리 튜브에 20mg/mL 스톡 아세틸화 소 혈청 알부민(BSA) 용액 1mL를 준비합니다. 일단 준비되면 50 μL 일회용 분취량을 만들어 -20 °C에서 보관하십시오.
참고: BSA의 아세틸화 버전은 뉴클레아제가 없는 유일한 형태의 BSA이기 때문에 사용하는 것이 중요합니다. 아세틸화되지 않은 BSA는 뉴클레아제의 중요한 공급원이 될 수 있으며, 따라서 샘플의 분해로 이어질 수 있습니다. - 아래 단계에 따라 각각 최소 5개의 50μm 및 100μm 미세모세관 피펫을 준비합니다.
- 먼저, 표준 유리 모세관(길이 4인치, 외경 1.2mm)을 바늘 풀러(재료 표 참조)와 Sutter Instruments Pipette Cookbook41의 "Adherent Cell, C. elegans, & Drosophila"에 대한 조건(조건: 열 = 램프 + 5; 풀 = 100; 벨. = 75; 지연 = 90; 압력 = 500; 2.5 mm x 2.5 mm 상자).
- 다음으로, 마이크로포지를 사용하여 마이크로모세관 피펫을 50μm(M5/0.1 대물렌즈 아래의 마이크로포지 안구 눈금자에 표시된 3개의 눈금 표시) 또는 100μm(동일한 조건에서 5개의 눈금 표시) 크기로 단조합니다( 재료 표 참조). 이러한 크기는 미세모세관 피펫의 예상 개구부 직경을 나타냅니다(그림 3).
- 그런 다음 구강 흡인기 튜브를 미세 모세관 피펫에 부착합니다.
참고: 흡인기 피펫은 전통적으로 마우스 피펫팅으로 제어되지만 많은 최신 안전 프로토콜은 이 방법을 허용하지 않습니다. 따라서 사용자는 손가락과 엄지 손가락 사이에 튜브를 끼워 입 흡인기 튜브로 흡인을 제어 할 수 있습니다. 또한 안전을 강화하기 위해 구강 흡인기 튜브 시스템 내에 주사기 필터를 설치할 수 있습니다.
3. 실험 준비
- 뉴클레아제가 없는 원추형 튜브에 5mL의 해부 완충액을 준비합니다. 실온에서 보관하십시오.
- 뉴클레아제가 없는 미세 원심분리 튜브에 작동 아세틸화 BSA 용액 1mL를 준비합니다. 얼음 위에 계속하십시오.
- 뉴클레아제가 없는 미세 원심분리 튜브에 350μL의 작동 레바미솔 용액을 만듭니다. 3 배 (약 20 개의 웜을위한 하나의 튜브)로 준비하십시오. 얼음 위에 계속하십시오.
- 뉴클레아제가 없는 마이크로원심분리 튜브에서 킬레이트화 완충액을 준비합니다(표 1). 복제하십시오 (약 10 개의 해부 된 벌레를위한 하나의 튜브). 얼음 위에 계속하십시오. 손 해부 중에 킬레이트 처리 버퍼를 사용하면 RNA 품질과 양이 향상됩니다(그림 4).
- 뉴클레아제가 없는 튜브에 500μL의 핵산 분리 시약 또는 키트 지정 분리 시약( 재료 표 참조)이 포함된 실험 그룹당 하나의 마이크로 원심분리 튜브를 준비합니다. 얼음 위에 계속하십시오. 이 튜브는 저장 또는 나중에 사용하기 위해 최종적이고 격리 된 장을 수집하는 데 사용됩니다.
- M9 배스 1개와 해부 버퍼 배스 1개를 준비합니다. 그렇게 하려면 직경 35mm의 멸균 페트리 접시 2개를 구하십시오. 그런 다음 한 접시에 M9 2mL를 추가하고 다른 접시에 2mL의 해부 완충액을 추가합니다. 마지막으로, 각 수조에 100 μL의 작동 BSA 용액을 추가합니다. 소용돌이 섞습니다. 욕조에 BSA를 추가하면 벌레가 플라스틱에 달라붙는 것을 방지할 수 있습니다.
- 해부 배열을 준비합니다. 2웰 오목한 슬라이드( 재료 표 참조)를 얻고 150μL의 작동 레바미솔 용액을 첫 번째 웰에 추가합니다. 그런 다음 150μL의 해부 버퍼를 두 번째 웰에 추가합니다. 마지막으로, 각 웰에 20 μL의 작동 BSA 용액을 추가합니다. 각 웰에 BSA를 추가하면 웜이 슬라이드에 달라붙는 것을 방지할 수 있습니다.
4. C. 엘레 간스 장의 손 해부
- 웜 픽을 사용하여 NGM 플레이트에서 20 마리의 성인 벌레를 M9 욕조로 옮깁니다 (3.6 단계). 이것은 웜에서 외부 박테리아를 씻어냅니다.
- 그런 다음 M20 배스에서 9 개의 웜을 모두 해부 버퍼 배스로 옮깁니다 (3.6 단계). 이것은 외부 박테리아를 더 씻어 내고 해부 완충액에서 벌레를 평형화시킵니다.
- 다음으로, 웜 배치(즉, 10개 세트)를 해부 완충 수조에서 레바미솔 용액이 포함된 웰로 이동합니다(단계 3.7).
알림: 레바미솔은 일시적으로 웜을 마비시킵니다.- 웜의 움직임이 느려지면 레바 미솔 웰에서 해부 완충액이 들어있는 웰로 빠르게 이동합니다 (3.7 단계). 벌레를 과도하게 마비시키지 않도록주의하십시오.
알림: 레바미솔 용액으로 일괄적으로 이동하는 웜의 수는 사용자의 편안함에 따라 달라질 수 있습니다. 이것은 처음에 M9 수조로 채집된 다음 해부 완충 수조로 이동한 벌레의 수에 대해서도 마찬가지입니다. 특히 훈련 중에 사용자가 프로토콜을 배우고 익숙해지기 때문에 해부에 사용할 수 있는 추가 웜을 확보하는 것이 좋습니다.
- 웜의 움직임이 느려지면 레바 미솔 웰에서 해부 완충액이 들어있는 웰로 빠르게 이동합니다 (3.7 단계). 벌레를 과도하게 마비시키지 않도록주의하십시오.
- 그런 다음 해부를 시작하기 전에 벌레가 해부 버퍼에서 약간 움직이기 시작하도록합니다.
알림: 내장은 지나치게 마비 된 벌레 (즉, 전혀 움직이지 않는 벌레)에서 잘 돌출되지 않습니다. - 준비가 되면 인두 바로 뒤(그림 5Aa) 또는 직장 바로 앞(그림 5Ab)에서 한 번 절단하여 피하 주사 바늘(즉, 27G x 1/2인치)(재료 표 참조)을 사용하여 형광 해부 스코프 아래에서 벌레를 해부합니다. 이것은 장의 두 개의 큰 부분, 즉 전방 중간 절반과 중간 후방 절반I를 생성합니다. 나머지 웜에 대해이 패턴을 계속하여 원하는 총 장 수를 얻을 때까지 전방 - 중간 및 중간 후방 섹션에서 얻은 장 섹션의 수를 동일하게 유지하십시오.
알림: 피하 주사 바늘을 빈 1mL 주사기 통에 부착하면 손 해부 중에 조작하는 데 도움이 될 수 있습니다. 사용자의 편안함과 경험에 따라 장 조각이 나오지 않는 해부 절단을하는 것은 드문 일이 아닙니다. 그러나 연습을 통해 이것은 훨씬 덜 일반적입니다. - 내장이 몸에서 최대한 돌출 될 때까지 약 1 분 정도 기다리십시오. 그들은 일반적으로 루프 모양을 취하고 생식선의 일부에 붙어있을 수 있습니다 (그림 5A). 기다리는 동안 50μL의 킬레이트 처리 버퍼를 웰에 추가하여 RNA 분해를 줄입니다(그림 4).
- 기다리는 동안 장 압출을 더욱 촉진하려면 구강 흡인기에 부착된 100μm 미세모세관 피펫을 사용하여 장/벌레를 피펫 안팎으로 끌어들입니다(그림 5B). 이것은 또한 생식선에서 장을 해방시키는 데 도움이됩니다. 실수로 장을 미세 모세관 피펫으로 완전히 빨아 들여 장을 잃지 않도록주의하십시오.
알림: 사용자는 100μm와 50μm 미세모세관 피펫을 번갈아 가며 신체의 나머지 부분과 생식선에서 장을 유리시킬 수 있습니다. 50μm 미세모세관 피펫의 더 작은 직경의 개구부는 장에서 끈적끈적한 조각을 제거하는 데 도움이 될 수 있습니다. 흡인기 피펫은 전통적으로 마우스 피펫팅으로 제어되지만 많은 최신 안전 프로토콜은 이 방법을 허용하지 않습니다. 따라서 사용자는 손가락과 엄지 손가락 사이에 튜브를 끼워 입 흡인기 튜브로 흡인을 제어 할 수 있습니다. 또한 안전을 강화하기 위해 구강 흡인기 튜브 시스템 내에 주사기 필터를 설치할 수 있습니다. - 장이 충분히 압출되면 27G 피하 주사 바늘을 사용하여 신체의 나머지 부분과 나머지 생식선에서 잘라냅니다. 격리 된 장 섹션의 크기는 수확기의 경험, 절단 품질 및 웜의 마비 수준에 따라 크게 달라질 수 있습니다.
참고: 장 및 장 조각의 무결성은 GFP 형광을 통해 프로토콜 전체에서 쉽게 모니터링할 수 있습니다. - 이제 미세 모세관 피펫을 사용하여 장 섹션을 흡인하고 웰에서 핵산 분리 시약 또는 키트 지정 분리 시약이 들어 있는 미세 원심분리 튜브로 옮깁니다. 분리 된 장을 시약에 얼음 위에 보관하고 나머지 내장에 대해 반복하십시오.
참고: 핵산 분리 시약 또는 키트로 지정된 다른 분리 시약이 들어 있는 마이크로 원심분리 튜브에 하루 종일 여러 장을 추가할 수 있습니다. 얼음 위에 계속하십시오. 모든 장을 1일 안에 분리할 수 없는 경우, 이미 분리되어 핵산 분리 시약(핵산을 보존함)에 보관된 장은 분리가 재개되거나 완료될 때까지 -80°C에서 보관할 수 있습니다. 여기서, 샘플은 장기간 안정하고(즉, 수개월 내지 1년) 핵산을 단리할 준비가 될 때까지 남아있을 수 있다. 내장은 물 또는 키트 특이적 용해 완충액으로 수확할 수 있습니다. 그러나 다운스트림 응용 프로그램으로 이동하기 전에 장이 얼음(또는 다른 원하는 온도)의 주어진 완충액에 얼마나 오래 머무를 수 있는지 결정할 때 고려해야 합니다.
5. 해부된 장에서 RNA 분리
- 핵산 분리 시약에 저장된 획득한 조직을 3번의 동결/해동/와류 주기를 수행하여 균질화합니다. 그렇게 하려면 37°C 비드 배스 및 액체 질소(또는 다른 유사한 수단)를 사용하십시오.
- 그런 다음 0.2 부피의 페놀 : 클로로포름 : IAA 시약 ( 재료 표 참조)을 샘플에 추가하고 간단히 와동시킵니다. 예를 들어, 시작 시료 부피가 500μL인 경우 클로로포름 부피 0.2는 100μL입니다.
- 튜브를 손으로 20초 동안 흔든 다음 실온에서 3분 동안 배양합니다.
- 원심분리(10,000 x g, 18분, 4°C)로 시료상을 분리합니다.
- 수성 상을 제거하고 새로운 뉴클레아제가 없는 마이크로원심분리 튜브로 옮깁니다.
참고: 인터페이스를 흡입하거나 방해하지 않도록 주의하십시오. - 그런 다음 동일한 부피의 100 % 에탄올을 수성 상에 첨가하고 샘플을 손으로 20 초 동안 흔 듭니다.
- 700μL의 샘플을 스핀 컬럼으로 옮깁니다( 재료 표 참조). 그런 다음 원심분리(≥8,000 x g, 30초, RT[실온])로 컬럼에 RNA를 부착합니다. 플로우 스루를 폐기합니다. 남은 추가 샘플에 대해 반복합니다.
- 350μL의 RW1 버퍼( 재료 표 참조)를 컬럼에 추가하여 샘플을 세척합니다. 이어서, 원심분리기(≥8,000 x g, 30 s, RT)를 버리고 유동을 폐기한다.
- 컬럼 내 DNA 분해를 수행합니다. RDD 버퍼( 재료 표 참조)에 80μL의 DNase I을 샘플 컬럼에 추가합니다. 그런 다음 RT에서 15분 동안 배양합니다.
- 그런 다음 350μL의 RW1 버퍼를 컬럼에 추가하여 샘플을 세척합니다. 원심분리기(≥8,000 x g, 30초, RT)를 사용하여 컬럼을 새 수집 튜브로 옮깁니다. 플로우 스루 및 오래된 수집 튜브를 폐기하십시오. 이 세척은 DNase를 제거합니다.
- 500 μL의 RPE( 재료 표 참조)를 샘플 컬럼에 추가합니다. 이어서, 원심분리(≥8,000 x g, 30 s, RT)하고 유동을 폐기한다. 두 번째 세척을 반복합니다.
- 그런 다음 컬럼 멤브레인을 추가로 건조시키기 위해 추가 1분 동안 원심분리합니다(≥8,000 x g, 1분, RT). 다음으로, 샘플 컬럼을 뚜껑이 있는 새로운 뉴클레아제가 없는 마이크로 원심분리기 수집 튜브로 옮깁니다. 플로우 스루 및 오래된 수집 튜브를 폐기하십시오.
- 14 μL의 뉴클레아제가 없는 물을 샘플 컬럼의 멤브레인에 직접 추가합니다. 이어서, 샘플을 RT에서 2분 동안 인큐베이션한다. 다음으로, 원심분리(≥8,000 x g, 1분, RT)하여 RNA를 용리시킨다.
- 샘플을 얼음에 보관하십시오. 다음으로, 시중에서 판매하는 분석 키트를 사용하여 샘플의 RNA 품질과 양을 평가합니다( 재료 표 참조). 완료되면 샘플을 -80 ° C 냉동고에 보관하십시오.
참고: RNA는 일반적으로 분해 없이 최대 1년 동안 -80°C에서 안정적입니다.
결과
본 프로토콜은 장내 많은 부분을 성인 C. elegans 에서 손으로 분리하는 데 사용되었습니다 (그림 2). 표시된 각 실험 그룹에 대한 최종 장 샘플은 전방 중후 장 섹션의 동일한 모음으로 구성됩니다. 그러나 실험 질문에 따라 전방, 중간 또는 후방 장 섹션만 포함할 수도 있습니다. 총체적으로, 이 프로토콜에 대한 세 가지 대표적인 결과가 제시된다. 첫 번째는 장의 성공적인 해부와 격리를 보여줍니다 (그림 6). 두 번째는 분리 된 장에서 RNA 분리 결과를보고합니다 (그림 7). 세 번째는 격리 된 장에서 미생물 감시 결과를 보여줍니다 (그림 8).
첫 번째 결과의 경우, 그림 6A는 성인 CL2122 웜 내에서 그림 5A의 부위 "a"에서 1차 절개를 한 후 압출된 장의 모습을 보여줍니다. CL2122 웜은 GFP(mtl-2 p::GFP)에 융합된 장 특이적 mtl-2 프로모터를 보유하고 있기 때문에 분리된 장은 형광 해부 범위 아래에서 녹색으로 빛납니다. 장 세그먼트의 성공적인 해부는 그림 6B에 나와 있습니다. 이 장 섹션에는 생식선이나 시체의 파편과 같은 눈에 보이는 오염 물질이 없습니다. 대조적으로, 그림 6C는 생식선과 시체가 여전히 장 세그먼트에 눈에 띄게 부착되어 있기 때문에 성공적으로 해부되지 않은 장을 표시합니다.
두 번째 결과의 경우, 장을 핵산 분리 시약으로 수확하고 다음날 저 입력 총 RNA 추출 프로토콜을 사용하여 처리했습니다 ( 재료 표 참조). 전방 중후 및 후방 중간 절편에서 60 개의 총 장 절편의 최종 장 샘플은 약 15ng의 고품질 총 RNA를 산출합니다 (그림 7A). 이 양의 총 내장은 하루에 쉽게 얻을 수 있지만 필요한 경우 며칠에 걸쳐 분해 할 수도 있습니다. 손 해부로 인한 RNA 수율은 그에 상응하는 양의 총 RNA를 얻기 위해 수십만 개의 장 세포가 필요하다는 점에서 웜 분해 및 장 세포의 FACS 분리보다 더 효율적입니다(그림 7B). 중요하게도, 손 해부에 의해 생성된 RNA 수율은 2pg의 RNA를 적게 사용할 수 있는 상용 RNA-seq 라이브러리 키트(즉, NEBNext Ultra II 및 NEBNext 단일 세포/낮은 입력)에 대한 입력으로 충분합니다.
세 번째 결과를 위해, 장을 멸균ddH20으로 수확하고, 상용 미생물 DNA 분리 키트를 사용하여 당일 처리하였다( 재료 표 참조). 전방 중간 및 중간 후방 절편에서 40 개의 총 장 절편의 최종 장 샘플은 범 박테리아 검출 분석을 사용하여 약 0.009 pg의 총 미생물 DNA를 산출합니다 ( 재료 표 참조) (그림 8). 이러한 양은 기존의 정량화 방법에 비해 너무 적으며 qPCR 표준 곡선에서 외삽해야 합니다. 이상적으로, 사용자는 시약 오염 수준과 관련된 신호 대 노이즈의 검출 한계와 경계를 증가시키기 때문에 장의 최종 총량을 >40으로 목표로 삼아야 합니다.
실험 설계를 고려할 때 항상 적절한 대조 샘플을 수집해야 합니다. 전사체학 실험의 경우, 적합한 실험 대조군은 장과 동일한 방식으로 수집된 전체 웜의 제제를 포함할 수 있다. 그러나 C. elegans에서 장을 해부하고 분리하는 동안 시체와 생식선 조각이 장 섹션에 달라 붙는 것을 보는 것이 일반적입니다. 이상적으로 이러한 오염 조직은 다운스트림 사용을 위해 저장하기 전에 장에서 제거되지만 추가 실험 제어에는 장 해부 후 남은 벌레 사체 수집 및/또는 해부된 생식선 수집이 포함될 수 있습니다. 마이크로바이옴 실험의 경우, 대조군은 기존의 양성(박테리아 배양) 및 음성(물) 대조군 외에도 장과 동일한 방식으로 수집된 전체 벌레의 제제를 포함할 수도 있습니다.
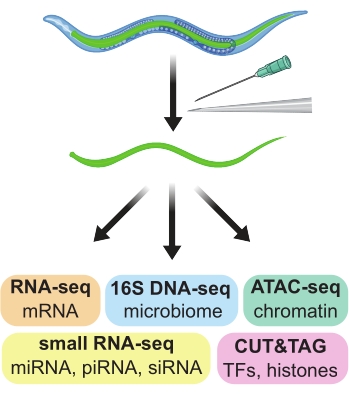
그림 1: 다양한 다운스트림 오믹스 분석에 사용하기 위한 조직 특이적 제제를 생성하는 데 사용되는 성인 C. elegans의 장 손 해부. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
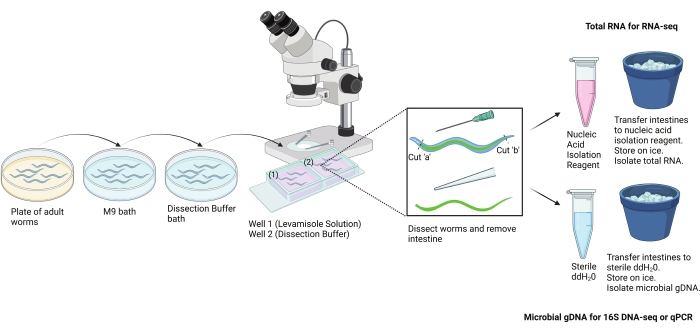
그림 2: 손 해부 프로토콜에 대한 구성표. 이 프로토콜은 장내 많은 부분을 성인 C. elegans 에서 손으로 분리하는 데 사용되었습니다. 장은 다양한 다운스트림 분석을 위해 분리될 수 있습니다. 여기에 표시된 것은 RNA 분리 및 미생물 DNA 분리를 위한 장의 사용입니다. BioRender.com 로 만든 이미지입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
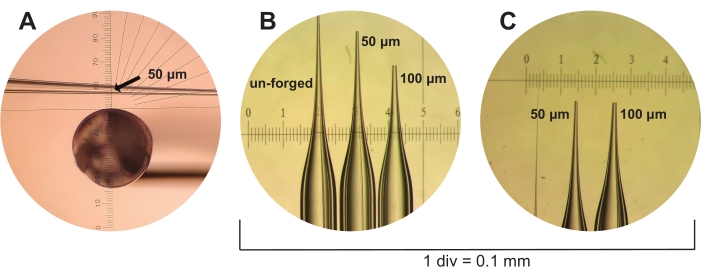
그림 3: 미세모세관 피펫 제조. (A) 새로 당겨졌지만 단조되지 않은 미세 모세관 피펫이 미세 단조의 안구 조각을 통해 표시됩니다. 안구 눈금자는 50μm 크기의 미세 모세관 피펫을 3 개의 눈금 표시 (화살표로 표시)의 추정 내경까지 측정하는 데 사용됩니다. (B) 단조되지 않은 50 μm 및 100 μm 미세 모세관 피펫은 1 div = 0.1 mm의 교정 눈금자와 함께 해부 범위 아래에 표시됩니다. (C) (B)의 50 μm 및 100 μm 미세 모세관 피펫은 다시 표시되지만 다른 유리한 지점에서 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
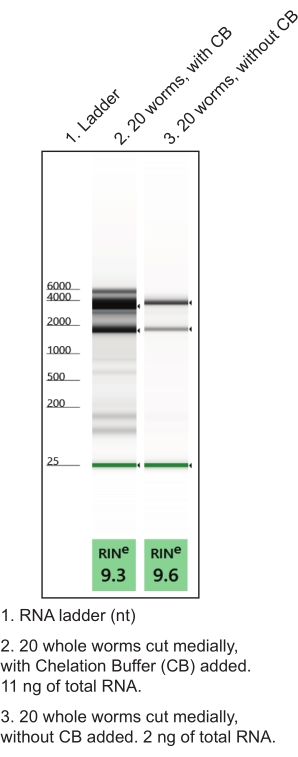
그림 4: RNA 수율을 개선하기 위해 손 해부 중 킬레이트화 완충액(CB) 사용. 낮은 입력 총 RNA 추출 프로토콜은 명명 된 조직으로부터 RNA 준비를 생성했다. 분리된 총 RNA 품질(RIN, RNA 무결성 번호) 및 양을 특성화하는 대표적인 겔 전기영동 실행이 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
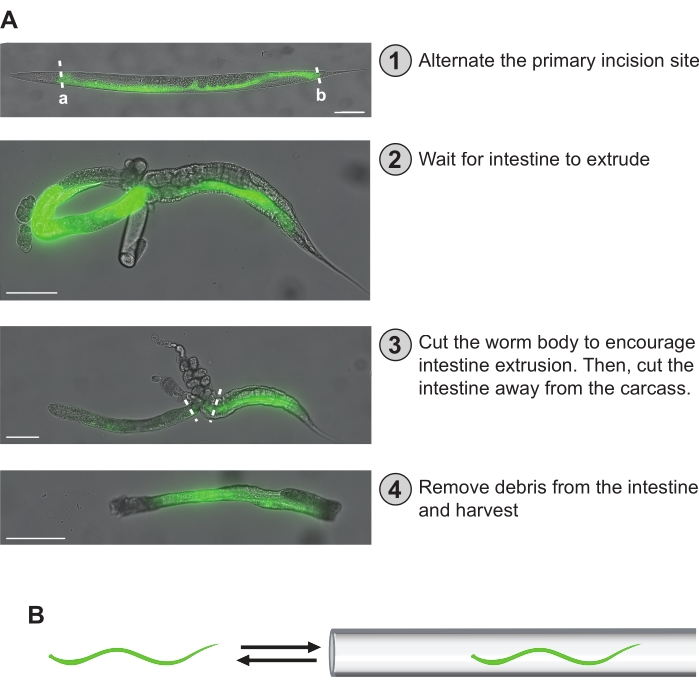
그림 5: 손 해부 단계 . (A) 프로토콜에 설명된 해부 단계는 여기에 요약되어 있습니다. (1)에서 웜의 장은 GFP 형광으로 시각화됩니다. 1차 절개 부위는 벌레 사이에 고르게 분포되어 장의 전체 길이에 걸쳐 균일한 커버리지를 보장할 수 있습니다. 대안적으로, "a" 또는 "b" 1차 절개 부위를 선택하여 전방 중후 또는 중후 장 단편 특이적 제제를 수득할 수 있다. (2)에서 장은 압출되어 루프 모양을 취합니다. (3)에서 장은 먼저 벌레 몸에서 잘라내어 시체와 생식선에서 해방을 시도합니다. 그런 다음 제거 할 수없는 나머지 시체 또는 생식선에서 장을 제거합니다. (4)에서, 장의 청소 된 부분이 격리되어 보관 준비가됩니다. (B) 중요한 단계는 입 흡인기 끝에 형성된 미세 모세관 피펫 안팎으로 통과시켜 장에서 달라붙는 파편(즉, 생식선, 시체)을 제거하는 것입니다. 스케일 바 = 100 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
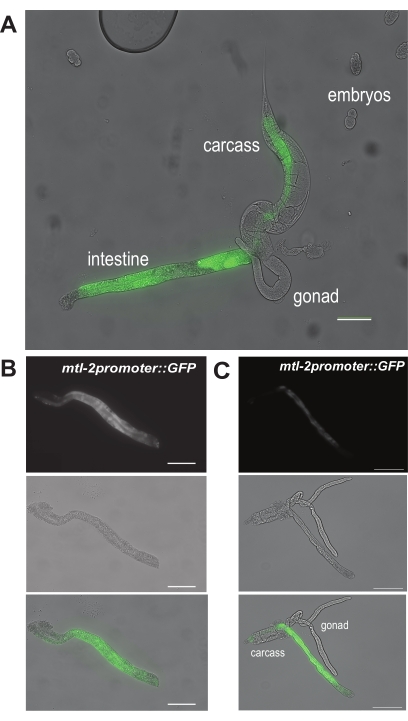
그림 6: 손 해부 단계의 대표적인 결과. (A) 웜으로부터 장의 압출을 보여주는 대표 이미지. 이들은 CL2122 유전자형의 웜으로, 장 세포 특이적 mtl-2 프로모터 하에서 GFP를 발현합니다. 두 개의 대표적인 이미지는 (B) 장의 깨끗하고 완전히 격리된 부분과 (C) 생식선 및 사체 조직을 오염시키는 격리된 장을 보여줍니다. 스케일 바 = 100 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
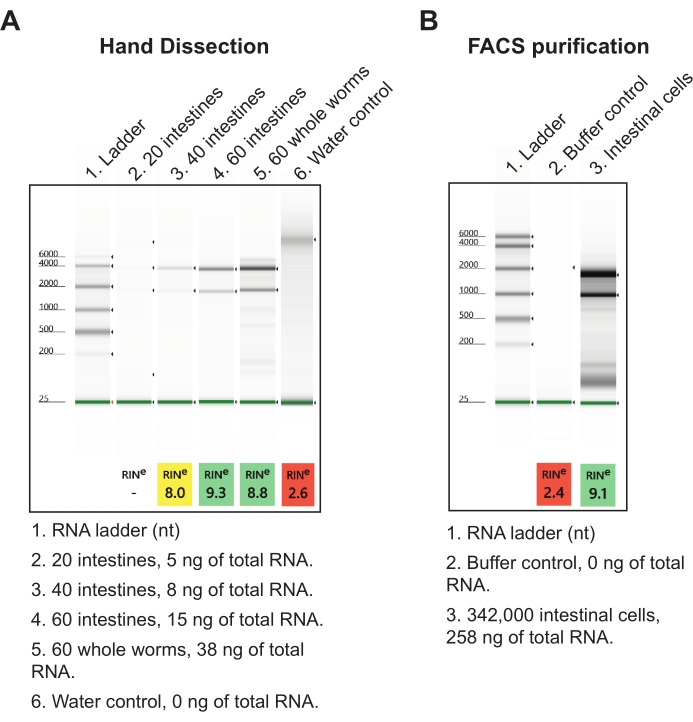
그림 7: 분리된 장에서 총 RNA 추출의 대표적인 결과. (A) 분리된 총 RNA의 품질(RIN, RNA 무결성 번호) 및 양을 특성화하여 아가로스 겔 전기영동에 의해 분해된 RNA 제제의 대표 이미지를 보여줍니다. (B) 비교를 위해 형광 활성화 세포 분류(FACS)를 통해 L1 단계 웜으로부터 수확된 장 세포로부터 분리된 총 RNA의 대표적인 겔을 나타낸다. 장당 20개의 세포에서, 손 해부 방법이 FACS로 정제된 장 세포보다 장당 더 많은 총 RNA를 산출한다고 추론할 수 있다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
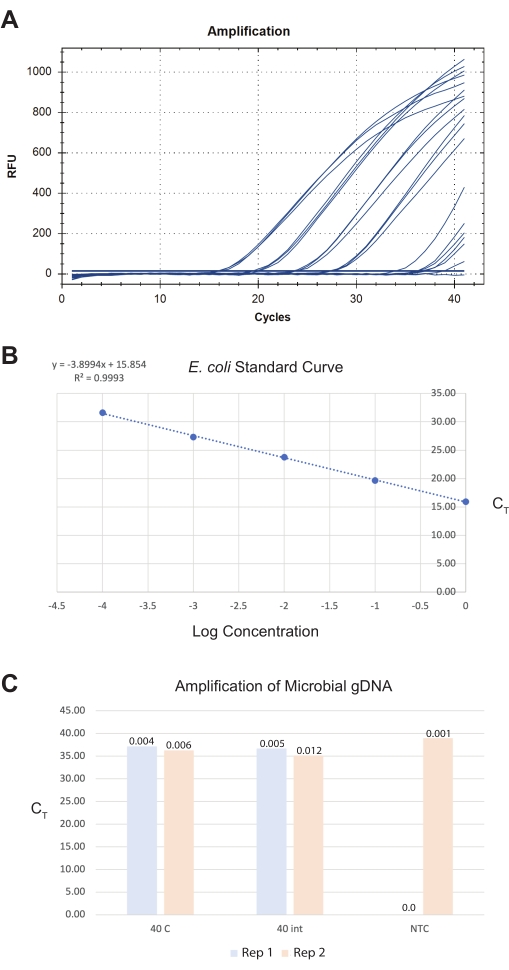
그림 8: 분리된 장에서 전체 미생물 DNA 추출에 대한 대표적인 결과. 상업용 미생물 DNA 분리 키트는 장, 전체 벌레 및 대조군에서 DNA 준비를 생성했습니다. 범박테리아 유전자 분석을 사용하여 샘플 내의 박테리아 수를 정량화했습니다. (A) 생성된 qPCR 증폭 곡선, (B) 샘플을 정량하기 위해 사용된 대장균 OP50 표준 곡선, 및 (C) 샘플의 대표 이미지가 도시되어 있다. (B)에서, 대장균 표준물질의 로그 시작 농도는 그들의CT 값에 대해 그래프로 표시된다. (C)에서, 40 개의 전체 벌레의 2 회 복제 (반복)를 내측으로 절단하고, 40 개의 분리 된 장 절편을 미생물 gDNA 분리를 위해 처리했다. qPCR 실행의 NTC(No-Template-Control)도 표시됩니다. Y축은 샘플CT 값을 나타낸다. PCR에서 정량화된 DNA의 양은 샘플 막대 위의 총 피코그램(pg) 값으로 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
표 1: 본 연구에 사용된 완충액 및 용액의 조성. 이 표를 다운로드하려면 여기를 클릭하십시오.
토론
이 기사에서는 성인 C. elegans에서 장을 손으로 해부하여 다운스트림 분석을 위한 순수한 준비를 생성하는 단계별 프로토콜을 설명합니다. 이 프로토콜의 중요한 단계에는 (1) 벌레가 과도하게 마비되지 않도록 하고, (2) 정확한 해부 절단을 하고, (3) 해부를 위해 적절한 크기의 마이크로 피펫을 단조하고, (4) 최종 수확 중에 건강한 장의 빠른 회복을 보장하는 것이 포함됩니다. 이러한 이유로 레바 미솔 용액에 웜을 노출 할 때는주의를 기울여야하며 최대 선명도를 보장하기 위해 피하 주사 바늘을 자주 새로 고쳐야합니다. 미세 모세관 피펫과 구강 흡인기를 사용하여 장을 다루는 것은 연습이 필요한 또 다른 단계입니다. 적절한 크기의 적절하게 단조된 마이크로 피펫은 마이크로 피펫 내에서 장을 잃을 위험을 줄이는 것 외에도 해부 중에 장의 큰 부분을 분리하는 데 상당한 차이를 만듭니다. 새로운 프로토콜 사용자는 일반적으로 분리 시약으로 배출되기 전에 미세 모세관 피펫의 내부 가장자리에서 장을 잃습니다. 이 문제는 연습과 적절하게 단조 된 미세 모세관 피펫으로 수정할 수 있습니다.
본원에 기술된 프로토콜은 성충에서 사용하기 위해 설계되었다. 예비 시험은 이 프로토콜이 L4 웜 및 노인 성인 웜에도 효과적임을 뒷받침합니다. 그러나이 프로토콜의 효능은 초기 애벌레 단계 웜에서 아직 평가되지 않았습니다. 이 접근법의 한계는 그것이 산출하는 재료의 양이 적다는 것입니다. 양은 RNA-seq 및 PCR에 충분하지만 다른 분석에는 적합하지 않을 수 있습니다. 따라서 사용자는 분석에 필요한 최소 입력을 이 프로토콜로 실현 가능하게 수집할 수 있는지 결정해야 합니다.
우리 연구실은 FACS를 일상적으로 활용하여 분리 후 장 세포를 정제하고30, 장 세포 식별을 위한 사후 분석 방법 및 이 손 해부 방법(30,42)을 사용합니다. 손 해부는 웜 분해 및 세포 분리가 덜 성공적일 때 성인 웜에서 사용할 수 있다는 장점이 있습니다. 또한, 손 해부 제제에서 추출한 총 RNA의 효율성과 품질이 높은데, 이는 조직이 벌레에서 빠르게 뽑힌 다음 핵산 분리 시약에 빠르게 침착되어 RNA 분해를 감소시키기 때문일 수 있습니다. 손 해부 방법의 또 다른 이점은 비용이 저렴하고 배우기 쉬우 며 특수 장비가 필요하지 않다는 것입니다. 마지막으로, 이 접근 방식은 벌레 장에서 장내 박테리아를 수확하고 분리하여 다운스트림 마이크로바이옴 연구를 가능하게 합니다.
성인 C. elegans에서 장을 분리하기 위해 여기에 설명 된 손 해부 프로토콜은 C. elegans 생물학의 다양한 측면을 연구하기위한 강력한 도구입니다 . 예를 들어, 장의 순수한 준비로 연구자들은 면역, 노화, 신진 대사 및 미생물 군집 사이의 교차점을 조사 할 수 있습니다.
공개
저자는 이해 상충이 없음을 선언합니다.
감사의 말
우리는 처음에 이 프로토콜이 적용된 장 해부 방법을 개발한 James McGhee와 Barb Goszczynski의 선구적인 작업에 빚을 지고 있습니다. 우리의 연구는 국립 일반 의학 연구소 (국립 보건원, R35GM124877에서 EON까지)가 감독하는 MIRA (R35) 상과 NSF MCB 분자 및 세포 생명 과학 부서가 감독하는 NSF-CAREER 상 (EON에 대한 상 #2143849)의 지원을 받습니다.
자료
| Name | Company | Catalog Number | Comments |
| Acetylated Bovine Serum Albumin (BSA) | VWR | 97061-420 | Nuclease free BSA |
| CL2122 worm strain | CGC (Caenorhabditis Genetics Center) | CL2122 | dvIs15 [(pPD30.38) unc-54(vector) + (pCL26) mtl-2::GFP]. Control strain for CL2120. Phenotype apparently WT. |
| Calcium Chloride Dihydrate | Fisher | C79 | needed for making Egg Salts |
| 50 mL Centrifuge Tubes, Bulk | Olympus Plastics | 28-108 | Nuclease free conical tube needed for solution making. |
| 15 mL Centrifuge Tubes, Bulk | Olympus Plastics | 28-103 | Nuclease free conical tube needed for solution making. |
| Concavity slide (2-well) | Electron Microscopy Sciences | 71878-08 | 12-pk of 2-well concavity slides |
| Ethylene glycol-bis(2-amino-ethylether)-N,N,N',N'-tetraacetic acid (EGTA) | Millipore Sigma | E3889 | needed for making chelation buffer |
| Fluorescent Dissection Microscope | Leica | M205 FCA | This is an optional piece of equipment that can be used with fluorescent C. elegans strains to help guid users during hand dissections |
| N-(2-Hydroxyethyl)piperazine-N′-(2-ethanesulfonic acid), 4-(2-Hydroxyethyl)piperazine-1-ethanesulfonic acid (HEPES) | Millipore Sigma | H4034 | needed for making dissection buffer |
| High Sensitivity RNA ScreenTape | Agilent | 5067-5579 | for assesment of total RNA quality and quantity |
| High Sensitivity RNA ScreenTape Ladder | Agilent | 5067-5581 | for assesment of total RNA quality and quantity |
| High Sensitivity RNA ScreenTape Sample Buffer | Agilent | 5067-5580 | for assesment of total RNA quality and quantity |
| HostZERO Microbial DNA Kit | Zymo Research | D4310 | Isolation of microbial DNA from worm intestines/worms |
| Hypodermic Needle (27G x 1/2") | BD Scientific | 305109 | needed for hand dissection of intestines |
| Levamisole (a.k.a. (-)-Tetramisole hydrochloride) | Millipore Sigma | L9756 | used to temporarily paralyze worms prior to hand dissection of intestines. |
| Luer-Lok General Use Disposable Syringe (1 mL) | BD Scientific | 309628 | Optional. Can be used to affix the hypodermic needle to, allowing easier manipulation of the needle during dissection. Remove the plunger. |
| Magnesium Chloride Hexahydrate | Fisher | M33 | needed for making Egg Salts |
| Magnesium Sulfate Hpetahydrate | Sigma-Aldrich | 230391-500G | needed for making M9 buffer |
| MF-900 Microforge | Narishige | MF-900 | Used to forge the microcapillary pipettes. Available through Tritech Research. |
| 1.7 mL Microtubes, Clear | Olympus Plastics | 22-282 | Nuclease free microfuge tube needed for solution making and sample storage. |
| Mouth Aspirator Tube | Millipore Sigma | A5177 | Mouth aspirator tube is needed in combination with the microcapillary pipette to allow aspiration of dissected intestines. |
| 16S Pan-Bacterial Control TaqMan Assay | Thermo Fisher | A50137 | Assay ID: Ba04930791_s1. Assay used for gut microbial detection via qPCR. |
| P-1000 Micropipette Puller | Sutter Instruments | Model P-1000 | Used to pull the microcapillary pippettes prior to forging. |
| Petri Dish (35 x 10 mm) | Genesee Scientific - Olympus Plastics | 32-103 | Used to make M9 bath and Disseciton Buffer bath for washing worms prior to dissection. |
| Phenol:Chloroform:IAA | Ambion | AM9730 | Used in the isolation of total RNA |
| Potassium Chloride | Millipore Sigma | 529552 | needed for making Egg Salts |
| Potassium Phosphate Monobasic | Sigma-Aldrich | P0662-500G | needed for making M9 buffer |
| Qubit 3 Fluorometer | Invitrogen | Q33216 | Accompanies the Qubit RNA HS Assay Kit. Can be used to quantify RNA prior to running sample on the Agilent ScreenTape. |
| Qubit RNA HS Assay Kit | Invitrogen | Q32852 | Can be used to quantify RNA prior to running sample on the Agilent ScreenTape. |
| RNasin Ribonuclease Inhibitor | Promega | N2111 | Broad spectrum inhibition of common eukaryotic Rnases |
| RNase-Free DNase Set | Qiagen | 79254 | used for on-column DNA digestion during RNA isolation protocol. |
| RNeasy Micro Kit | Qiagen | 74004 | Used for isolation of total RNA from worm intestines/worms |
| Standard Glass Capillaries | World Precision Instruments | 1B100F-4 | 4 in OD 1.2 mm standard borosilicate glass capillaries used to make microcapillary pipettes for dissection |
| Sodium Chloride | Fisher | S271 | needed for making Egg Salts |
| Sodium phosphate dibasic heptahydrate | Fisher Scientific | S373-500 | needed for making M9 buffer |
| Syringe filter (0.2 micrometer SCFA) | Thermo Fisher | 72302520 | Optional for use with the mouth aspirator tube when mouth pipetting. |
| 4150 TapeStation System | Agilent | G2992AA | Accompanies the RNA ScreenTape reagents for assessing RNA quality and quantity |
| TaqPath BactoPure Microbial Detection Master Mix | Applied Biosystems | A52699 | master mix used for qPCR |
| TRIzol Reagent | Thermo Fisher Scientific | 15596026 | Nucleic acid isolation and preservation. QIAzol (Qiagen; 79306) can be substituted if preferred. |
| Worm Pick | NA | NA | Made in house from a pasteur pipette and a platinum wire. See wormbook for details. |
참고문헌
- Riddel, D. L., Blumenthal, T., Meyer, B. J., Priess, J. . C. elegans II. , (1997).
- Corsi, A. K., Wightman, B., Chalfie, M. A transparent window into biology: A primer on Caenorhabditis elegans. Genetics. 200 (2), 387-407 (2015).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 485 (2017).
- Roy, P. J., Stuart, J. M., Lund, J., Kim, S. K. Chromosomal clustering of muscle-expressed genes in Caenorhabditis elegans. Nature. 418 (6901), 975-979 (2002).
- Pauli, F., Liu, Y., Kim, Y. A., Chen, P. -. J., Kim, S. K. Chromosomal clustering and GATA transcriptional regulation of intestine-expressed genes in C. elegans. Development. 133 (2), 287-295 (2005).
- Spencer, W. C., et al. A spatial and temporal map of C. elegans gene expression. Genome Research. 21 (2), 325-341 (2011).
- Spencer, W. C., et al. Isolation of specific neurons from C. elegans larvae for gene expression profiling. PLoS One. 9 (11), 112102 (2014).
- Ma, X., et al. Analysis of C. elegans muscle transcriptome using trans-splicing-based RNA tagging (SRT). Nucleic Acids Research. 44 (21), 156 (2016).
- Blazie, S. M., et al. Comparative RNA-Seq analysis reveals pervasive tissue-specific alternative polyadenylation in Caenorhabditis elegans intestine and muscles. BMC Biology. 13 (1), 4 (2015).
- Blazie, S. M., et al. Alternative polyadenylation directs tissue-specific miRNA targeting in Caenorhabditis elegans somatic tissues. Genetics. 206 (2), 757-774 (2017).
- Gómez-Saldivar, G., et al. et al.Tissue-specific transcription footprinting using RNA PoI DamID (RAPID) in Caenorhabditis elegans. Genetics. 216 (4), 931-945 (2020).
- Katsanos, D., Barkoulas, M. Targeted DamID in C. elegans reveals a direct role for LIN-22 and NHR-25 in antagonizing the epidermal stem cell fate. Science Advances. 8 (5), 3141 (2022).
- Kaletsky, R., et al. The C. elegans adult neuronal IIS/FOXO transcriptome reveals adult phenotype regulators. Nature. 529 (7584), 92-96 (2015).
- Kaletsky, R., et al. Transcriptome analysis of adult Caenorhabditis elegans cells reveals tissue-specific gene and isoform expression. PLoS Genetics. 14 (8), 1007559 (2018).
- Mathies, L. D., et al. mRNA profiling reveals significant transcriptional differences between a multipotent progenitor and its differentiated sister. BMC Genomics. 20 (1), 427 (2019).
- Liang, X., Calovich-Benne, C., Norris, A. Sensory neuron transcriptomes reveal complex neuron-specific function and regulation of mec-2/ Stomatin splicing. Nucleic Acids Research. 50 (5), 2401-2416 (2021).
- Glenwinkel, L., et al. In silico analysis of the transcriptional regulatory logic of neuronal identity specification throughout the C. elegans nervous system. eLife. 10, 64906 (2021).
- Taylor, S. R., et al. Molecular topography of an entire nervous system. Cell. 184 (16), 4329-4347 (2021).
- Charest, J., et al. Combinatorial action of temporally segregated transcription factors. Developmental Cell. 55 (4), 483-499 (2020).
- Steiner, F. A., Talbert, P. B., Kasinathan, S., Deal, R. B., Henikoff, S. Cell-type-specific nuclei purification from whole animals for genome-wide expression and chromatin profiling. Genome Research. 22 (4), 766-777 (2012).
- Tintori, S. C., Nishimura, E. O., Golden, P., Lieb, J. D., Goldstein, B. A transcriptional lineage of the early C. embryo. Developmental Cell. 38 (4), 430-444 (2016).
- Hashimshony, T., Wagner, F., Sher, N., Yanai, I. CEL-Seq: Single-cell RNA-Seq by multiplexed linear amplification. Cell Reports. 2 (3), 666-673 (2012).
- Hashimshony, T., Feder, M., Levin, M., Hall, B. K., Yanai, I. Spatiotemporal transcriptomics reveals the evolutionary history of the endoderm germ layer. Nature. 519 (7542), 219-222 (2015).
- Packer, J. S., et al. A lineage-resolved molecular atlas of C. elegans embryogenesis at single-cell resolution. Science. 365 (6459), 1971 (2019).
- Warner, A. D., Gevirtzman, L., Hillier, L. W., Ewing, B., Waterston, R. H. The C. elegans embryonic transcriptome with tissue, time, and alternative splicing resolution. Genome Research. 29 (6), 1036-1045 (2019).
- Cao, J., et al. Comprehensive single-cell transcriptional profiling of a multicellular organism. Science. 357 (6352), 661-667 (2017).
- Durham, T. J., et al. Comprehensive characterization of tissue-specific chromatin accessibility in L2 Caenorhabditis elegans nematodes. Genome Research. 31 (10), 1952-1969 (2021).
- King, D. C., et al. The transcription factor ELT-2 positively and negatively impacts direct target genes to modulate the Caenorhabditis elegans intestinal transcriptome. bioRxiv. , (2021).
- Kocsisova, Z., Mohammad, A., Kornfeld, K., Schedl, T. Cell cycle analysis in the C. elegans germline with the thymidine analog EdU. Journal of Visualized Experiments. (140), e58339 (2018).
- Han, S., et al. Mono-unsaturated fatty acids link H3K4me3 modifiers to C. elegans lifespan. Nature. 544 (7649), 185-190 (2017).
- Edgar, L. G., Wolf, N., Wood, W. B. Early transcription in Caenorhabditis elegans embryos. Development. 120 (2), 443-451 (1994).
- Edgar, L. G., Goldstein, B. Culture and manipulation of embryonic cells. Methods in Cell Biology. 107, 151-175 (2012).
- Nishimura, E. O., Zhang, J. C., Werts, A. D., Goldstein, B., Lieb, J. D. Asymmetric transcript discovery by RNA-seq in C. elegans blastomeres identifies neg-1, a gene important for anterior morphogenesis. PLoS Genetics. 11 (4), 1005117 (2015).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Cerón, J. Basic Caenorhabditis elegans methods: Synchronization and observation. Journal of Visualized Experiments. (64), e4019 (2012).
- Fay, D. S., Fluet, A., Johnson, C. J., Link, C. D. In vivo aggregation of β-amyloid peptide variants. Journal of Neurochemistry. 71 (4), 1616-1625 (1998).
- Altun, Z. F., Hall, D. H. WormAtas Hermaphrodite Handbook – Introduction to C. elegans Anatomy. WormAtlas. , (2006).
- Tan, M. -. W., Mahajan-Miklos, S., Ausubel, F. M. Killing of Caenorhabditis elegans by Pseudomonas aeruginosa used to model mammalian bacterial pathogenesis. Proceedings of the National Academy of Sciences of the United States of America. 96 (2), 715-720 (1999).
- Oesterle, A. Pipette Cookbook 2018: P-97 & P-1000 Micropipette Pullers. Sutter Instrument Company. , (2018).
- Dineen, A., Nishimura, E. O., Goszczynski, B., Rothman, J. H., McGhee, J. D. Quantitating transcription factor redundancy: The relative roles of the ELT-2 and ELT-7 GATA factors in the C. elegans endoderm. Developmental Biology. 435 (2), 150-161 (2018).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유