Method Article
Handdissektion von Caenorhabditis elegans Darm
In diesem Artikel
Zusammenfassung
Das vorliegende Protokoll beschreibt ein Verfahren zur Isolierung von Darm aus adulten Caenorhabditis elegans-Nematodenwürmern von Hand zur Eingabe in Genomik, Proteomik, Mikrobiom oder anderen Assays.
Zusammenfassung
Der Caenorhabditis elegans-Darm besteht aus nur 20 Zellen und ist der Nexus vieler lebenserhaltender Funktionen, einschließlich Verdauung, Stoffwechsel, Alterung, Immunität und Umweltreaktion. Kritische Wechselwirkungen zwischen dem Wirt von C. elegans und seiner Umgebung konvergieren im Darm, wo sich die Darmmikrobiota konzentrieren. Daher ist die Fähigkeit, Darmgewebe vom Rest des Wurms zu isolieren, notwendig, um darmspezifische Prozesse zu beurteilen. Dieses Protokoll beschreibt eine Methode zur Handseparierung adulter C. elegans-Därme. Das Verfahren kann in fluoreszierend markierten Stämmen zur Vereinfachung oder zu Trainingszwecken durchgeführt werden. Sobald die Technik perfektioniert ist, kann der Darm von unmarkierten Würmern eines beliebigen Genotyps gesammelt werden. Dieser Mikrodissektionsansatz ermöglicht die gleichzeitige Erfassung von Wirtsdarmgewebe und Darmmikrobiota, ein Vorteil für viele Mikrobiomstudien. Daher können nachgeschaltete Anwendungen für die durch dieses Protokoll erzeugten Darmpräparate die RNA-Isolierung aus Darmzellen und die DNA-Isolierung aus eingefangenen Mikrobiota umfassen, sind aber nicht darauf beschränkt. Insgesamt bietet die Handdissektion von C. elegans-Därmen eine einfache und robuste Methode, um kritische Aspekte der Darmbiologie zu untersuchen.
Einleitung
Der Fadenwurm Caenorhabditis elegans ist mit nur 959 Zellen und einem 4-tägigen Ei-zu-Ei-Lebenszyklus ein ideales Modellsystem für viele Genetik-, Genomik- und Entwicklungsstudien 1,2. Die Leichtigkeit des Vorwärts- und Rückwärts-Genscreenings, die Prävalenz von technisch hergestellten Fluoreszenzmarkern, die Fähigkeit, nukleotidspezifische Genom-Editierung durchzuführen, und die zahlreichen gemeinschaftsweiten Ressourcen haben alle zu wichtigen Entdeckungen und Erkenntnissen im C. elegans-System beigetragen. Ein wesentlicher Nachteil ist jedoch die Schwierigkeit, reine Populationen von Zellen, Geweben oder Organen zu erhalten, die klein, zerbrechlich und miteinander verbunden sein können. Da reine Zellpopulationen für genomische Assays wie RNA-seq, ChIP-seq und ATAC-seq wichtig sind, haben sich mehrere Ansätze entwickelt, um reine Präparate von C. elegans-Zellen, -Geweben und -Organen zu erhalten. Hier wird eine Methode zur Handzerlegung von Därmen in großen Abschnitten aus erwachsenen C. elegans-Würmern beschrieben. Die resultierenden Präparate eignen sich für nachgeschaltete genomische Assays (Abbildung 1).
Die hier beschriebene feinskalige Gewebedissektionsmethode (Abbildung 2) ist nur ein Ansatz. Andere alternative Techniken - wie molekulare Markierung, Disaggregation von Würmern und Reinigung von Zelltypen von Interesse mit fluoreszenzaktivierter Zellsortierung (FACS) und Post-hoc-Analyse - wurden ebenfalls erfolgreich eingesetzt, um die gewebespezifischen Merkmale der Molekularbiologie von C. elegans zu untersuchen. Ein Vorteil der Handdissektion gegenüber diesen anderen Ansätzen ist jedoch, dass sie verwendet werden kann, um gleichzeitig die Eigenschaften des C. elegans-Darms und seines bakteriellen Inhaltszu erforschen 3,4,5. Dies ermöglicht die Sequenzierung von 16S rRNA-Genen und erleichtert Mikrobiomstudien innerhalb des C. elegans-Systems. Eine wichtige Einschränkung ist jedoch, dass Darmzellen nicht einzeln isoliert werden.
Die molekulare Markierung verleiht Molekülen nur innerhalb des spezifizierten Gewebes oder der Zellen von Interesse eine zelltypspezifische Markierung. Diese Tags können dann aus den gesamten Wurmpräparaten isoliert werden. Auf diese Weise haben gewebespezifische Promotoren, die ein markiertes PolyA-bindendes Protein oder einen gespleißten Leader antreiben, ein gewebespezifisches Transkriptom-Profiling 6,7,8,9,10 und 3'UTR-Mapping 11,12 ermöglicht. In ähnlicher Weise wurden gewebespezifische Transkriptionsfaktorprofile unter Verwendung von ChIP-seq und DamID durchgeführt, bei denen promotorspezifische Transkriptionsfaktorvarianten mit Tags oder Enzymfusionen 13,14 angehängt wurden.
FACS ermöglicht die Isolierung von Zelltypen von Interesse von dissoziierten Würmern basierend auf ihren intrinsischen zellulären Eigenschaften und fluoreszierenden Eigenschaften. Dieser Ansatz hat gewebespezifische Transkriptome aus verschiedenen Organen 8,15,16 und einzelnen neuronalen Zelltypen 8,9,15,16,17,18 erzeugt und wurde verwendet, um eine Expressionskarte des gesamten C. elegans-Nervensystems zu erstellen 19,20 . FACS und sein Cousin, die fluoreszenzaktivierte Kernsortierung (FANS), wurden ebenfalls verwendet, um zellspezifische Chromatinprofile21,22 zu erzeugen.
Schließlich kann eine Post-hoc-Analyse in Einzelzellauflösungs-Assays durchgeführt werden. Bei dieser Methode werden alle einzelnen Zellen untersucht, der Zelltyp jeder einzelnen wird in der Analysephase zugeordnet und die interessierenden Zelltypen werden selektiv für weitere Untersuchungen gefiltert. Die Post-hoc-Analyse wurde erfolgreich eingesetzt, um Transkriptome von sich entwickelnden Zellen mit hoher räumlicher und zeitlicher Auflösung in C. elegans-Embryonen im Stadium 23,24,25,26,27 und L1 28 zu erhalten. Die Zugänglichkeit von Chromatin wurde auch unter Verwendung von ATAC-seq anstelle von RNA-seq unter Verwendung einer ähnlichen Strategiecharakterisiert 29.
Jeder Ansatz hat seine Vor- und Nachteile. Für den Darm von C. elegans ist eine Wurmdisaggregation und FACS-Isolierung von Darmzellen im Embryo- und Larvenstadium30 möglich, bei Erwachsenen jedoch eine Herausforderung. Es wird angenommen, dass dies auf die großen, endo-reduplizierten und stark adhärenten Zellen des Darms zurückzuführen ist, die es schwierig machen, unbeschädigt zu dissoziieren. Die hier beschriebene Handdissektionsmethode umgeht diese Herausforderungen und ermöglicht die Isolierung großer Teile des Darms des erwachsenen Wurms. Die Praxis, Gonaden von Hand aus diesem Stadium zu sezieren, ist weit verbreitet und unkompliziert. Die Darmdissektion ähnelt der Gonadendissektion, wird jedoch seltener durchgeführt32. Das hier vorgestellte Protokoll basiert auf einem längeren, unveröffentlichten Protokoll, das von Dr. James McGhee und Barb Goszczynski entwickelt wurde. Dieses optimierte Protokoll entlehnt Techniken zur Isolierung von Blastomeren aus Embryonen im Frühstadium 23,33,34,35. Obwohl die Handdissektion für die Isolierung der meisten Zell- oder Gewebetypen in C. elegans nicht möglich ist, ist sie ideal für die Isolierung von Darm von erwachsenen Würmern. Daher ergänzt die Handdissektion andere Mittel zur Gewinnung darmspezifischer Zellpräparate.
Protokoll
CL2122-Würmer wurden für die vorliegende Studie verwendet. Die Würmer wurden durch das Caenorhabditis Genetics Center (CGC, siehe Table of Materials) gewonnen, das vom NIH Office of Research Infrastructure Programs (P40 OD010440) finanziert wird.
1. Züchtung von Würmern zur Dissektion
- Züchten Sie eine große Platte gemischtstufiger CL2122-Würmer zur Synchronisation nach Standardkulturverfahren (d. h. NGM-Platten, die mit E. coli OP50 ausgesät wurden)36,37.
HINWEIS: Es dauert im Allgemeinen ~ 96 Stunden, um die maximale Eiablagekapazität zu erreichen.- Der Embryo bereitet die Würmer innerhalb der 72-96 h Periode vor. Lassen Sie die Embryonen nach der Vorbereitung des Embryos 48 h lang in M9 schlüpfen (siehe Tabelle 1). Dies führt zu einer synchronen Population von Würmern im L1-Stadium.
HINWEIS: CL2122-Würmer beherbergen ein integriertes Transgen-GFP (grün fluoreszierendes Protein), das vom darmspezifischen mtl-2 (MeTaLlothionein 2) -Promotor38 vertrieben wird. Dieser Promotor ist spezifisch für das intestinale Zellzytoplasma und ermöglicht es, den Darm auf einem fluoreszierenden Dissektionsmikroskop sichtbar zu machen. Nach dem Training finden Benutzer möglicherweise keine Fluoreszenzanleitung erforderlich.
- Der Embryo bereitet die Würmer innerhalb der 72-96 h Periode vor. Lassen Sie die Embryonen nach der Vorbereitung des Embryos 48 h lang in M9 schlüpfen (siehe Tabelle 1). Dies führt zu einer synchronen Population von Würmern im L1-Stadium.
- Plattensynchronisierte L1-Schnecken auf mindestens zwei kleinen Platten. Züchten Sie Würmer auf NGM-Tellern mit ausreichend Nahrung, bis sie das Erwachsenenstadium erreichen (identifiziert durch das Vorhandensein von Embryonen, die in der Lage sind, Eier zu legen). Dies dauert zwischen 38-46 h bei 20 °C.
- Achten Sie darauf, dass das gleiche Entwicklungsstadium über Replikate und Vergleichsstämme hinweg geerntet wird.
HINWEIS: Die Dauer des Wachstums der Würmer hängt vom Stamm, der Temperatur, der Nahrungsquelle und dem angestrebten Entwicklungsstadium ab39. Andere Stadien in der Nähe des Erwachsenenstadiums können ebenfalls verwendet werden, wie das L4- und ältere Erwachsenenstadium. Für die Planung von Mikrobiomexperimenten können verschiedene bakterielle Nahrungsquellen über die traditionelle Nahrungsquelle (E. coli OP50) hinaus verwendet werden, um Würmer zu züchten, wie die CeMbio-Stämme4 oder einen Erreger von Interesse40.
- Achten Sie darauf, dass das gleiche Entwicklungsstadium über Replikate und Vergleichsstämme hinweg geerntet wird.
2. Herstellung von Stammlösungen und Mikrokapillarpipetten
ANMERKUNG: Tabelle 1 enthält die Einzelheiten aller für die vorliegende Studie verwendeten Puffer und Lösungen.
- Bereiten Sie 50 ml Eisalze (auch bekannt als Eipuffer) in einem nukleasefreien konischen Röhrchen vor (siehe Materialtabelle). Nach der Herstellung bei Raumtemperatur lagern.
- Bereiten Sie 10 ml 100 mM Levamisol-Stammlösung in einem nukleasefreien konischen Röhrchen vor. Nach der Zubereitung 500 μL Aliquots herstellen und bei −20 °C lagern. Aliquots können aufgetaut und 1 Woche lang bei 4 °C gelagert werden.
- Bereiten Sie 1 ml 20 mg/ml acetylierte Rinderserumalbuminlösung (BSA) in einem nukleasefreien Mikrozentrifugenröhrchen vor. Nach der Zubereitung 50 μL Einwegaliquots herstellen und bei −20 °C lagern.
HINWEIS: Es ist wichtig, die acetylierte Version von BSA zu verwenden, da dies die einzige Form von BSA ist, die nukleasefrei ist. Nicht-acetyliertes BSA kann eine signifikante Quelle für Nukleasen sein und somit zum Abbau von Proben führen. - Bereiten Sie mindestens fünf 50-μm- und 100-μm-Mikrokapillarpipetten vor, die jeweils die folgenden Schritte ausführen.
- Ziehen Sie zunächst Standardglaskapillaren (4 Zoll lang und 1,2 mm Außendurchmesser) mit einem Nadelzieher (siehe Materialtabelle) und Bedingungen für "Adherent Cell, C. elegans, & Drosophila" aus dem Sutter Instruments Pipette Cookbook41 (Bedingungen: Wärme = Rampe + 5; Zug = 100; = 75; Verzögerung = 90; Druck = 500; 2,5 mm x 2,5 mm Box).
- Als nächstes werden die Mikrokapillarpipetten entweder auf 50 μm (drei Häkchen, wie durch das Mikroforge-Augenlineal unter dem M5/0,1-Objektiv angezeigt) oder 100 μm (fünf Häkchen unter den gleichen Bedingungen) mit einer Mikroschmiede geschmiedet (siehe Materialtabelle). Diese Größen stellen den geschätzten Öffnungsdurchmesser der Mikrokapillarpipette dar (Abbildung 3).
- Befestigen Sie dann einen Mundsaugröhrchen an den Mikrokapillarpipetten.
HINWEIS: Aspiratorpipetten werden traditionell durch Mundpipettieren gesteuert, aber viele moderne Sicherheitsprotokolle verbieten diese Methode. So können Benutzer die Aspiration mit dem Mundsaugerschlauch steuern, indem sie den Schlauch zwischen Finger und Daumen einklemmen. Zusätzlich kann ein Spritzenfilter im Mundsaugerschlauchsystem installiert werden, um die Sicherheit zu erhöhen.
3. Versuchsvorbereitung
- Bereiten Sie 5 ml Dissektionspuffer in einem nukleasefreien konischen Röhrchen vor. Bei Raumtemperatur lagern.
- Bereiten Sie 1 ml acetylierte BSA-Lösung in einem nukleasefreien Mikrozentrifugenröhrchen vor. Auf Eis bleiben.
- Stellen Sie 350 μL Levamisollösung in einem nukleasefreien Mikrozentrifugenröhrchen her. In dreifacher Ausfertigung zubereiten (eine Tube für ca. 20 Würmer). Auf Eis bleiben.
- Chelatpuffer wird in einem nukleasefreien Mikrozentrifugenröhrchen hergestellt (Tabelle 1). In replizieren (eine Röhre für ca. 10 sezierte Würmer). Auf Eis bleiben. Die Verwendung von Chelat-Puffer während der Handdissektion verbessert die RNA-Qualität und -Quantität (Abbildung 4).
- Bereiten Sie ein Mikrozentrifugenröhrchen pro Versuchsgruppe vor, das 500 μL Nukleinsäureisolierungsreagenz oder Kit-spezifiziertes Isolierreagenz (siehe Materialtabelle) in einem nukleasefreien Röhrchen enthält. Auf Eis bleiben. Dieses Röhrchen wird verwendet, um den endgültigen, isolierten Darm für die Lagerung oder spätere Verwendung zu sammeln.
- Bereiten Sie ein M9-Bad und ein Dissektionspufferbad vor. Um dies zu tun, erhalten Sie zwei sterile Petrischalen mit einem Durchmesser von 35 mm. Fügen Sie dann 2 ml M9 zu einer Schale und 2 ml Dissektionspuffer zur anderen hinzu. Zum Schluss fügen Sie 100 μL BSA-Arbeitslösung zu jedem Bad hinzu. Zum Mischen schwenken. Das Hinzufügen von BSA zu den Bädern verhindert, dass die Würmer am Kunststoff haften bleiben.
- Bereiten Sie das Dissektionsarray vor. Man erhält einen 2-Well-Konkavitätsobjektträger (siehe Materialtabelle) und gibt 150 μL Levamisollösung in die erste Vertiefung. Dann fügen Sie 150 μL Dissektionspuffer in die zweite Vertiefung hinzu. Zum Schluss fügen Sie 20 μL BSA-Arbeitslösung zu jeder Vertiefung hinzu. Das Hinzufügen von BSA zu jeder Vertiefung verhindert, dass die Würmer an der Folie haften bleiben.
4. Handdissektion des Darms von C. elegans
- Bewegen Sie 20 erwachsene Würmer mit einem Wurmpickel von der NGM-Platte in das M9-Bad (Schritt 3.6). Dies wird externe Bakterien von den Würmern waschen.
- Bewegen Sie dann alle 20 Würmer aus dem M9-Bad in das Dissektionspufferbad (Schritt 3.6). Dadurch werden externe Bakterien weiter weggespült und die Würmer im Dissektionspuffer ausgeglichen.
- Als nächstes werden Chargen von Würmern (d. h. in Sätzen von 10) aus dem Dissektionspufferbad in die Vertiefung mit Levamisollösung (Schritt 3.7) gegeben.
HINWEIS: Levamisol lähmt die Würmer vorübergehend.- Sobald sich die Wurmbewegungen verlangsamt haben, bewegen Sie sie schnell von der Levamisolmulde in die Vertiefung, die den Dissektionspuffer enthält (Schritt 3.7). Achten Sie darauf, die Würmer nicht übermäßig zu lähmen.
HINWEIS: Die Anzahl der Würmer, die in Chargen in die Levamisollösung verschoben werden, kann je nach Komfort des Benutzers variieren. Dies gilt auch für die Anzahl der Würmer, die zunächst in das M9-Bad aufgenommen und dann in das Dissektionspufferbad verbracht wurden. Es ist eine gute Praxis, zusätzliche Würmer zum Sezieren zur Verfügung zu haben, insbesondere während des Trainings, da der Benutzer lernt und sich mit dem Protokoll vertraut macht.
- Sobald sich die Wurmbewegungen verlangsamt haben, bewegen Sie sie schnell von der Levamisolmulde in die Vertiefung, die den Dissektionspuffer enthält (Schritt 3.7). Achten Sie darauf, die Würmer nicht übermäßig zu lähmen.
- Lassen Sie dann die Würmer beginnen, sich ein wenig im Dissektionspuffer zu bewegen, bevor Sie mit den Dissektionen beginnen.
HINWEIS: Der Darm extrudiert nicht gut aus übermäßig gelähmten Würmern (dh Würmern, die sich überhaupt nicht bewegen). - Wenn Sie fertig sind, sezieren Sie die Würmer unter einem fluoreszierenden Sezierfernrohr mit einer Injektionsnadel (dh 27 G x 1/2 Zoll) (siehe Materialtabelle), indem Sie einen Schnitt entweder direkt hinter dem Pharynx (Abbildung 5Aa) oder direkt vor dem Rektum (Abbildung 5Ab) machen. Dadurch entstehen zwei große Abschnitte des Darms, eine vordere mittlere Hälfte und eine mittlere hintere HälfteI. Setzen Sie dieses Muster für den Rest der Würmer fort und halten Sie die Anzahl der Darmabschnitte, die aus den vorderen, mittleren und mittleren hinteren Abschnitten erhalten werden, gleich, bis die gewünschte Gesamtzahl des Darms erworben ist.
HINWEIS: Das Anbringen der Injektionsnadel an einem leeren 1-ml-Spritzenzylinder kann bei der Manipulation während der Handdissektion helfen. Je nach Komfort und Erfahrung des Benutzers ist es nicht ungewöhnlich, einen Sezierschnitt zu machen, der kein Darmfragment ergibt. Mit der Praxis wird dies jedoch weitaus seltener. - Warten Sie etwa 1 Minute, bis der Darm maximal aus dem Körper herausragt. Sie nehmen typischerweise eine Schleifenform an und können an einem Teil der Gonaden haften (Abbildung 5A). Während des Wartens 50 μL Chelatpuffer in die Vertiefung geben, um den RNA-Abbau zu reduzieren (Abbildung 4).
- Verwenden Sie während des Wartens und zur weiteren Erleichterung der Darmextrusion die 100-μm-Mikrokapillarpipette, die an einem Mundsauger befestigt ist, und ziehen Sie den Darm/Wurm in die Pipette hinein und aus ihr heraus (Abbildung 5B). Dies wird auch helfen, den Darm von der Gonade zu befreien. Achten Sie darauf, den Darm nicht zu verlieren, indem Sie ihn versehentlich vollständig in die Mikrokapillarpipette aufsaugen.
HINWEIS: Der Benutzer kann zwischen den 100 μm und 50 μm Mikrokapillarpipetten wechseln, um den Darm vom Rest des Körpers und der Gonade zu befreien. Die Öffnung mit kleinerem Durchmesser der 50 μm Mikrokapillarpipette kann einen Vorteil für die Entfernung klebriger Stücke aus dem Darm bieten. Aspiratorpipetten werden traditionell durch Mundpipettieren gesteuert, aber viele moderne Sicherheitsprotokolle verbieten diese Methode. So können Benutzer die Aspiration mit dem Mundsaugerschlauch steuern, indem sie den Schlauch zwischen Finger und Daumen einklemmen. Zusätzlich kann ein Spritzenfilter im Mundsaugerschlauchsystem installiert werden, um die Sicherheit zu erhöhen. - Sobald ein Darm ausreichend extrudiert ist, verwenden Sie die 27 G Injektionsnadel, um ihn vom Rest des Körpers und der verbleibenden Keimzelle abzuschneiden. Die Größe des isolierten Darmabschnitts kann je nach Erfahrung des Harvesters, der Qualität des Schnitts und dem Grad der Lähmung des Wurms stark variieren.
HINWEIS: Die Integrität des Darms und der Darmfragmente kann während des gesamten Protokolls über ihre GFP-Fluoreszenz leicht überwacht werden. - Verwenden Sie nun die Mikrokapillarpipette, um den Darmabschnitt abzusaugen und ihn aus der Vertiefung in das Mikrozentrifugenröhrchen zu bewegen, das ein Nukleinsäureisolierungsreagenz oder ein Kit-spezifiziertes Isolierreagenz enthält. Halten Sie den isolierten Darm im Reagenz auf Eis und wiederholen Sie ihn für den restlichen Darm.
HINWEIS: Dem Mikrozentrifugenröhrchen, das das Nukleinsäureisolierungsreagenz oder ein anderes Kit-spezifiziertes Isolierreagenz enthält, können im Laufe des Tages mehrere Därme hinzugefügt werden. Auf Eis bleiben. Wenn nicht der gesamte Darm in 1 Tag isoliert werden kann, können diejenigen, die bereits isoliert und in einem Nukleinsäureisolierungsreagenz (das die Nukleinsäuren konserviert) gelagert sind, bei -80 ° C gehalten werden, bis die Isolierungen wieder aufgenommen werden und / oder abgeschlossen werden können. Hier sind die Proben langfristig stabil (d.h. mehrere Monate bis 1 Jahr) und können bleiben, bis sie bereit sind, Nukleinsäuren zu isolieren. Der Darm kann zu Wasser oder einem kitspezifischen Lysepuffer geerntet werden. Es muss jedoch berücksichtigt werden, wie lange der Darm in einem bestimmten Puffer auf Eis (oder einer anderen gewünschten Temperatur) bleiben kann, bevor zu nachgeschalteten Anwendungen übergegangen wird.
5. RNA-Isolierung aus sezierten Eingeweiden
- Homogenisieren Sie die erworbenen Gewebe, die im Nukleinsäureisolierungsreagenz gespeichert sind, indem Sie drei Gefrier- / Tau- / Wirbelzyklen durchführen. Verwenden Sie dazu ein 37 °C warmes Perlenbad und flüssigen Stickstoff (oder ein anderes vergleichbares Mittel).
- Dann fügen Sie 0,2 Volumen Phenol:Chloroform:IAA-Reagenz (siehe Materialtabelle) zur Probe hinzu und wirbeln Sie kurz. Zum Beispiel würden für ein Startprobenvolumen von 500 μL 0,2 Volumina Chloroform 100 μL sein.
- Schütteln Sie das Röhrchen 20 s von Hand und inkubieren Sie es dann bei Raumtemperatur für 3 min.
- Die Probenphasen werden durch Zentrifugation (10.000 x g, 18 min, 4 °C) getrennt.
- Entfernen Sie die wässrige Phase und überführen Sie sie in ein neues nukleasefreies Mikrozentrifugenröhrchen.
HINWEIS: Achten Sie darauf, die Schnittstelle nicht abzusaugen oder zu stören. - Dann fügen Sie ein gleiches Volumen von 100% Ethanol in die wässrige Phase hinzu und schütteln Sie die Probe von Hand für 20 s.
- 700 μL der Probe in die Spinsäule überführen (siehe Materialtabelle). Dann wird RNA mit Zentrifugation an die Säule geklebt (≥8.000 x g, 30 s, RT [Raumtemperatur]). Verwerfen Sie den Durchfluss. Wiederholen Sie diesen Vorgang für alle weiteren verbleibenden Proben.
- Geben Sie 350 μL RW1-Puffer (siehe Materialtabelle) in die Säule, um die Probe zu waschen. Dann zentrifugieren (≥8.000 x g, 30 s, RT) und den Durchfluss verwerfen.
- Führen Sie einen DNA-Aufschluss an der Säule durch. Fügen Sie 80 μL DNase I im RDD-Puffer (siehe Materialtabelle) zur Probenspalte hinzu. Dann 15 Minuten bei RT inkubieren.
- Fügen Sie dann 350 μL RW1-Puffer in die Säule ein, um die Probe zu waschen. Zentrifugieren (≥8.000 x g, 30 s, RT) und Kolonne in ein neues Auffangröhrchen überführen. Entsorgen Sie das Durchflussrohr und das alte Sammelrohr. Diese Wäsche entfernt die DNase.
- Fügen Sie 500 μL RPE (siehe Materialtabelle) zur Probensäule hinzu. Dann zentrifugieren (≥8.000 x g, 30 s, RT) und den Durchfluss verwerfen. Wiederholen Sie diesen Vorgang für eine zweite Wäsche.
- Dann für weitere 1 min zentrifugieren, um die Säulenmembran weiter zu trocknen (≥8.000 x g, 1 min, RT). Als nächstes wird die Probensäule in ein frisches nukleasefreies Mikrozentrifugensammelröhrchen mit Deckel überführt. Entsorgen Sie das Durchflussrohr und das alte Sammelrohr.
- 14 μL nukleasefreies Wasser direkt in die Membran der Probensäule geben. Dann inkubieren Sie die Probe für 2 min bei RT. Als nächstes zentrifugieren (≥8.000 x g, 1 min, RT), um die RNA zu eluieren.
- Lagern Sie die Probe auf Eis. Als nächstes bewerten Sie die RNA-Qualität und -Quantität der Probe mit kommerziell erhältlichen Assay-Kits (siehe Materialtabelle). Danach lagern Sie die Probe in einem Gefrierschrank von −80 °C.
HINWEIS: RNA ist im Allgemeinen stabil bei -80 °C für bis zu 1 Jahr ohne Abbau.
Ergebnisse
Das vorliegende Protokoll wurde verwendet, um große Teile des Darms von adulten C. elegans von Hand zu isolieren (Abbildung 2). Die endgültige Darmprobe für jede gezeigte Versuchsgruppe besteht aus einer gleichen Sammlung von vorder-mittleren und mittleren hinteren Darmabschnitten. Abhängig von der experimentellen Fragestellung könnte es sich aber auch um eine Sammlung von nur vorderen, mittleren oder hinteren Darmabschnitten handeln. Insgesamt werden drei repräsentative Ergebnisse für dieses Protokoll präsentiert. Die erste zeigt die erfolgreiche Dissektion und Isolierung des Darms (Abbildung 6). Die zweite berichtet über die Ergebnisse der RNA-Isolierung aus isolierten Eingeweiden (Abbildung 7). Die dritte zeigt die Ergebnisse der mikrobiellen Überwachung aus isolierten Eingeweiden (Abbildung 8).
Für das erste Ergebnis zeigt Abbildung 6A, wie ein extrudierter Darm nach einem primären Schnitt an der Stelle "a" in Abbildung 5A bei erwachsenen CL2122-Würmern aussieht. Da CL2122-Würmer den darmspezifischen mtl-2-Promotor beherbergen, der mit GFP (mtl-2p::GFP) verschmolzen ist, leuchten isolierte Därme unter dem fluoreszierenden Sezierfernrohr grün. Die erfolgreiche Dissektion eines Darmabschnitts ist dann in Abbildung 6B dargestellt. Dieser Darmabschnitt ist frei von sichtbaren Verunreinigungen wie Ablagerungen von der Gonade oder dem Kadaver. Im Gegensatz dazu zeigt Abbildung 6C Därme, die nicht erfolgreich seziert werden, da Gonade und Kadaver noch sichtbar am Darmsegment befestigt sind.
Für das zweite Ergebnis wurde der Darm zu einem Nukleinsäureisolierungsreagenz geerntet und am nächsten Tag mit einem Low-Input-Gesamt-RNA-Extraktionsprotokoll verarbeitet (siehe Materialtabelle). Eine abschließende Darmprobe von insgesamt 60 Darmabschnitten aus den vorder-mittleren und mittleren hinteren Abschnitten ergibt etwa 15 ng hochwertige Gesamt-RNA (Abbildung 7A). Diese Menge an Gesamtdarm kann leicht an einem einzigen Tag gewonnen werden, kann aber bei Bedarf auch über mehrere Tage aufgebrochen werden. RNA-Ausbeuten aus Handdissektion sind effizienter als Wurmdisaggregation und FACS-Isolierung von Darmzellen, da Hunderttausende von Darmzellen benötigt werden, um entsprechende Mengen an Gesamt-RNA zu erhalten (Abbildung 7B). Wichtig ist, dass die durch die Handdissektion erzeugten RNA-Ausbeuten als Input für kommerzielle RNA-seq-Bibliothekskits (d.h. NEBNext Ultra II und NEBNext Single Cell/Low Input) mehr als ausreichend sind, die nur 2 pg RNA aufnehmen können.
Für das dritte Ergebnis wurde der Darm zu sterilem ddH20 geerntet und noch am selben Tag mit einem kommerziellen mikrobiellen DNA-Isolierkit verarbeitet (siehe Materialtabelle). Eine abschließende Darmprobe von 40 gesamten Darmabschnitten aus den vorder-mittleren und mittleren hinteren Abschnitten ergibt etwa 0,009 pg gesamtmikrobielle DNA unter Verwendung eines panbakteriellen Nachweistests (siehe Materialtabelle) (Abbildung 8). Diese Größen sind für traditionelle Quantifizierungsmethoden zu gering und müssen aus qPCR-Standardkurven extrapoliert werden. Idealerweise sollten Anwender eine endgültige Gesamtmenge an Darm >40 anstreben, da dies die Nachweisgrenze und die Grenze des Signal-Rausch-Verhältnisses in Bezug auf die Reagenzkontamination erhöht.
Bei der Versuchsplanung ist immer die Entnahme geeigneter Kontrollproben erforderlich. Für transkriptomische Experimente kann eine geeignete experimentelle Kontrolle Präparate des gesamten Wurms umfassen, die auf die gleiche Weise wie Darm gesammelt wurden. Während der Dissektion und Isolierung des Darms von C. elegans ist es jedoch üblich, dass sich Kadaver und Gonaden an den Darmabschnitten festsetzen. Während diese kontaminierenden Gewebe idealerweise vor der Lagerung für die nachgeschaltete Verwendung aus dem Darm entfernt werden, können zusätzliche experimentelle Kontrollen die Sammlung der übrig gebliebenen Wurmkadaver nach der Darmdissektion und / oder die Sammlung von sezierten Gonaden umfassen. Für Mikrobiomexperimente können die Kontrollen neben herkömmlichen positiven (Bakterienkultur) und negativen (Wasser) Kontrollen auch Präparate des gesamten Wurms umfassen, die auf die gleiche Weise wie Darm gesammelt wurden.
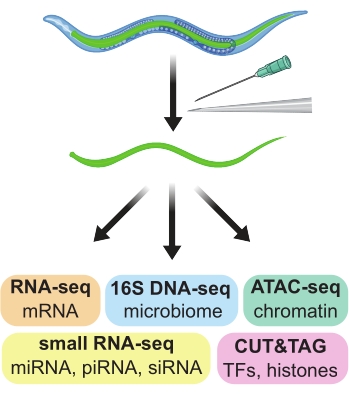
Abbildung 1: Handdissektion von Därmen aus adulten C. elegans, die zur Herstellung gewebespezifischer Präparate für den Einsatz in einer Vielzahl von nachgeschalteten Omics-Assays verwendet werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
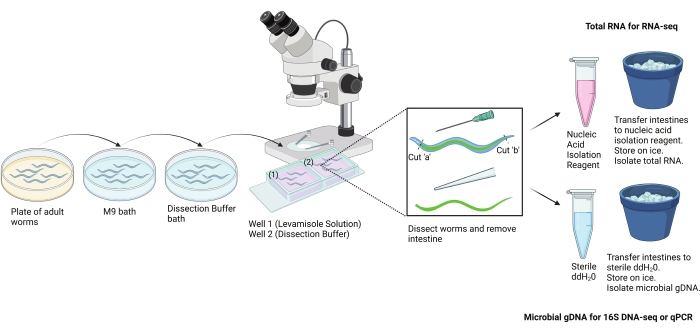
Abbildung 2: Schema für das Handsektionsprotokoll. Dieses Protokoll wurde verwendet, um große Teile des Darms von erwachsenen C. elegans von Hand zu isolieren. Der Darm kann für verschiedene nachgeschaltete Assays isoliert werden. Hier wird die Verwendung von Darm zur RNA-Isolierung und mikrobiellen DNA-Isolierung gezeigt. Bild erstellt mit BioRender.com. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
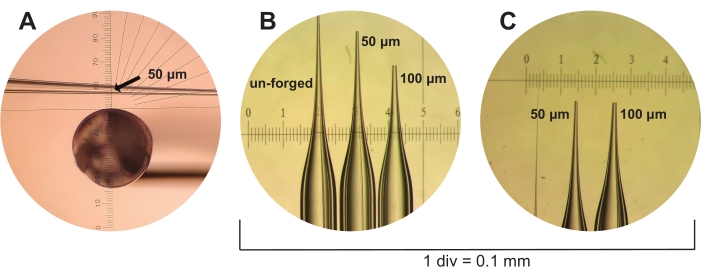
Abbildung 3: Herstellung von Mikrokapillarpipetten. (A) Eine frisch gezogene, aber ungeschmiedete Mikrokapillarpipette wird durch das Augenstück einer Mikroschmiede gezeigt. Ein Augenlineal wird verwendet, um 50 μm große Mikrokapillarpipetten bis zu einem geschätzten Innendurchmesser von drei Teilstrichen zu messen (dargestellt, durch Pfeil gekennzeichnet). (B) Ungeschmiedete, 50 μm und 100 μm Mikrokapillarpipetten werden unter dem Sezierfernrohr neben einem Kalibrierlineal mit 1 div = 0,1 mm gezeigt. (C) Die 50 μm und 100 μm Mikrokapillarpipetten von (B) werden noch einmal gezeigt, jedoch aus einem anderen Blickwinkel. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
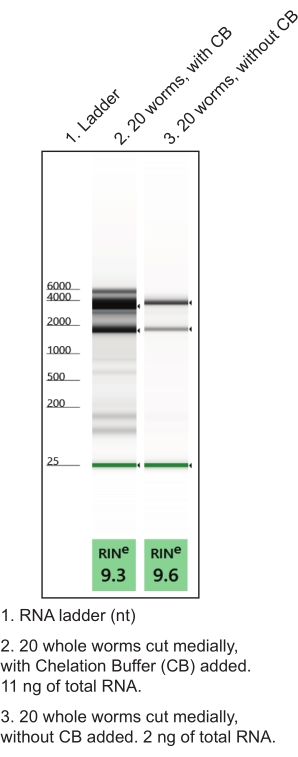
Abbildung 4: Die Verwendung von Chelat-Puffer (CB) während der Handdissektion zur Verbesserung der RNA-Ausbeute. Ein Low-Input-Gesamt-RNA-Extraktionsprotokoll erzeugte RNA-Präparate aus benannten Geweben. Repräsentative Gelelektrophoreseläufe zur Charakterisierung der isolierten Gesamt-RNA-Qualität (RIN, RNA-Integritätszahl) und -Quantität werden gezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
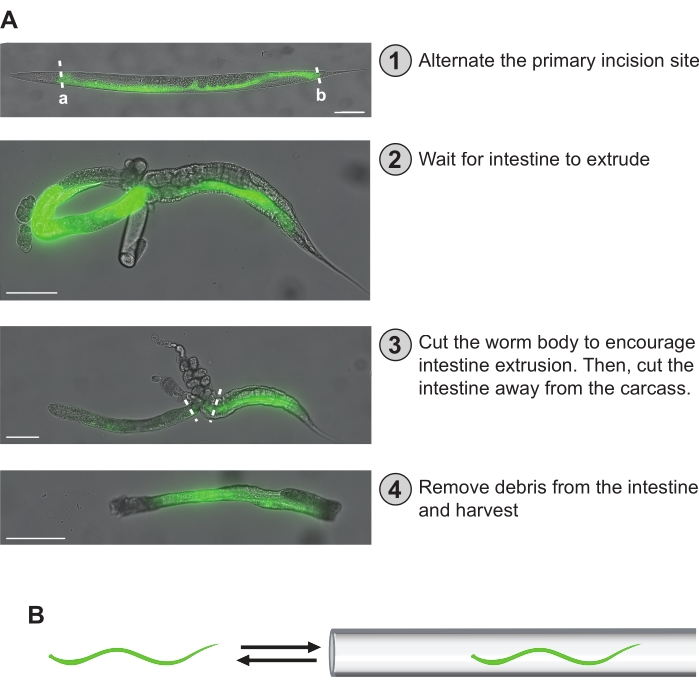
Abbildung 5: Schritte der Handdissektion . (A) Die im Protokoll beschriebenen Dissektionsschritte sind hier beschrieben. In (1) wird der Darm des Wurms durch GFP-Fluoreszenz sichtbar gemacht. Die primäre Inzisionsstelle kann gleichmäßig auf die Würmer verteilt werden, um eine gleichmäßige Abdeckung über die gesamte Darmlänge zu gewährleisten. Alternativ können entweder "a" oder "b" primäre Inzisionsstellen ausgewählt werden, um eine anterior-mittlere oder mittlere posteriore Darmfragment-spezifische Zubereitung zu erhalten. In (2) ist der Darm extrudiert und nimmt eine Schleifenform an. In (3) wird der Darm zuerst vom Wurmkörper abgeschnitten, während versucht wird, ihn vom Kadaver und der Gonade zu befreien. Der Darm wird dann von jedem verbleibenden Kadaver oder jeder Gonade entfernt, die nicht entfernt werden kann. In (4) wird ein gereinigter Abschnitt des Darms isoliert und zur Lagerung bereit. (B) Ein kritischer Schritt besteht darin, alle anhaftenden Trümmer (d. h. Gonaden, Kadaver) aus dem Darm zu entfernen, indem sie in die Mikrokapillarpipette am Ende des Mundsaugers ein- und ausgelagert werden. Maßstabsbalken = 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
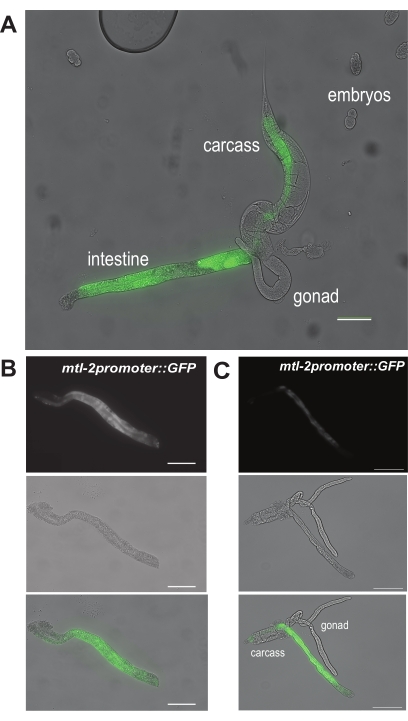
Abbildung 6: Repräsentative Ergebnisse der Handdissektionsschritte. Ein repräsentatives Bild, das (A) die Extrusion des Darms aus dem Wurm zeigt. Dies sind Würmer des CL2122-Genotyps, die GFP unter dem darmzellspezifischen mtl-2-Promotor exprimieren. Zwei repräsentative Bilder zeigen (B) einen gereinigten und vollständig isolierten Darmabschnitt und (C) einen isolierten Darm mit kontaminierendem Gonaden- und Kadavergewebe. Maßstabsbalken = 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
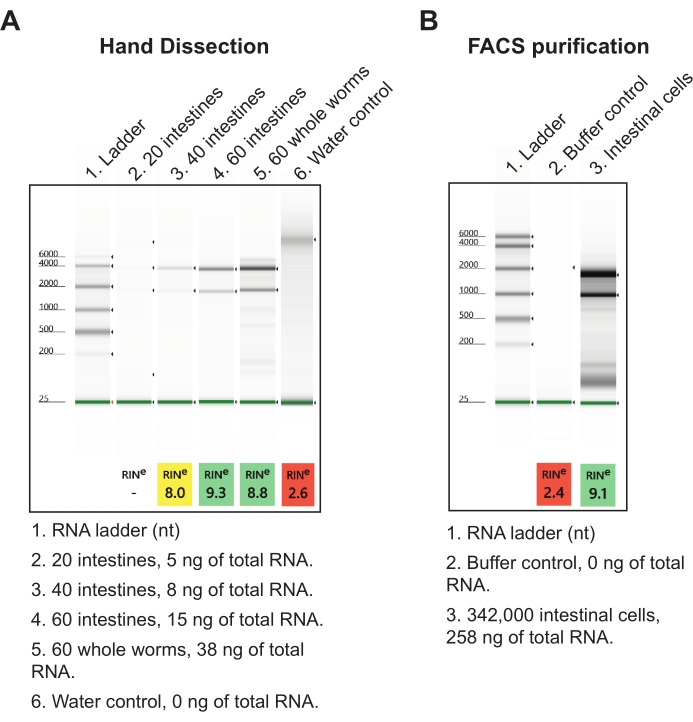
Abbildung 7: Repräsentative Ergebnisse der gesamten RNA-Extraktion aus isolierten Eingeweiden . (A) Es wird ein repräsentatives Bild von RNA-Präparaten gezeigt, die durch Agarose-Gelelektrophorese aufgelöst wurden, wobei die Qualität (RINs, RNA-Integritätszahl) und Quantität der isolierten Gesamt-RNAs charakterisiert werden. (B) Zum Vergleich wird ein repräsentatives Gel der Gesamt-RNA gezeigt, das aus Darmzellen isoliert wurde, die aus Würmern im L1-Stadium mittels fluoreszenzaktivierter Zellsortierung (FACS) gewonnen wurden. Bei 20 Zellen pro Darm kann gefolgert werden, dass die Handdissektionsmethode mehr Gesamt-RNA pro Darm liefert als FACS-gereinigte Darmzellen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
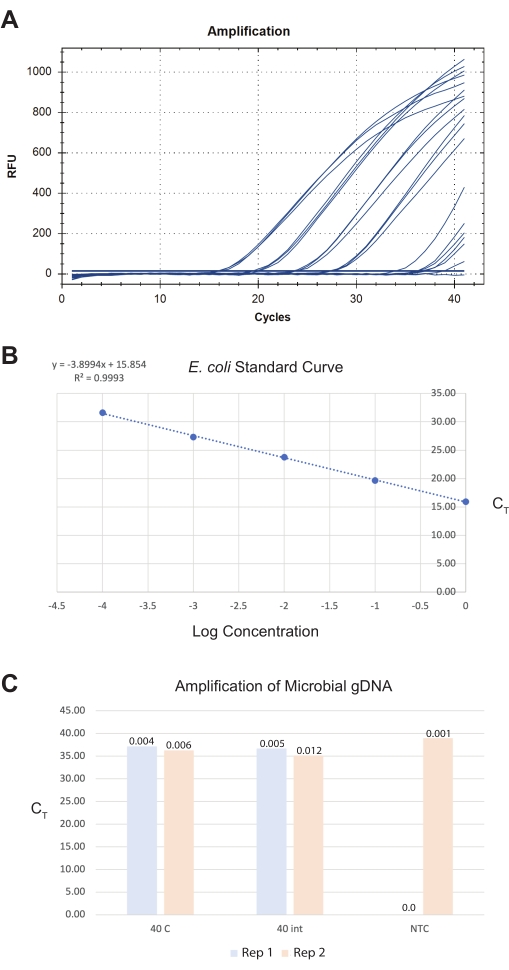
Abbildung 8: Repräsentative Ergebnisse für die gesamte mikrobielle DNA-Extraktion aus isolierten Därmen. Ein kommerzielles mikrobielles DNA-Isolierungskit erzeugte DNA-Präparate aus Darm, ganzen Würmern und Kontrollen. Ein panbakterieller Gentest wurde verwendet, um die Anzahl der Bakterien in den Proben zu quantifizieren. Ein repräsentatives Bild von (A) den erzeugten qPCR-Amplifikationskurven, (B) der E. coli OP50-Standardkurve, die zur Quantifizierung der Proben verwendet wurde, und (C) den Proben. In (B) werden die logarithmischen Ausgangskonzentrationen von E. coli-Standards mit ihrenCT-Werten verglichen. In (C) wurden zwei Replikate (Wiederholungen) von 40 ganzen Würmern medial geschnitten und 40 isolierte Darmabschnitte für die mikrobielle gDNA-Isolierung verarbeitet. Das No-Template-Control (NTC) aus dem qPCR-Lauf wird ebenfalls gezeigt. Die Y-Achse stellt dieC-T-Werte der Stichprobe dar. Die Menge an DNA, die aus PCRs quantifiziert wird, wird als Gesamtpikogramm (pg) -Werte über den Probenbalken angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Tabelle 1: Zusammensetzung der in dieser Studie verwendeten Puffer und Lösungen. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Dieser Artikel beschreibt das Schritt-für-Schritt-Protokoll für die Handzerlegung von Därmen aus adulten C. elegans und erzeugt reine Präparate für nachgeschaltete Assays. Zu den kritischen Schritten in diesem Protokoll gehören (1) die Sicherstellung, die Würmer nicht übermäßig zu lähmen, (2) genaue Sezierschnitte, (3) das Schmieden von Mikropipetten in angemessener Größe für die Dissektion und (4) die Sicherstellung der schnellen Wiederherstellung des gesunden Darms während der letzten Ernte. Aus diesen Gründen ist Vorsicht geboten, wenn Würmer der Levamisollösung ausgesetzt werden, und die Injektionsnadeln müssen häufig aufgefrischt werden, um eine maximale Schärfe zu gewährleisten. Die Handhabung des Darms mit der Mikrokapillarpipette und dem Mundsauger ist ein weiterer Schritt, der Übung erfordert. Richtig geschmiedete Mikropipetten der entsprechenden Größe machen auch einen wesentlichen Unterschied bei der Isolierung großer Darmabschnitte während der Dissektionen und verringern das Risiko, den Darm innerhalb der Mikropipette zu verlieren. Neue Protokollbenutzer verlieren häufig Därme am inneren Rand der Mikrokapillarpipette, bevor sie in das Isolierreagenz ausgestoßen werden können. Dieses Problem kann mit Übung und richtig geschmiedeten Mikrokapillarpipetten behoben werden.
Das hierin beschriebene Protokoll wurde für die Verwendung bei erwachsenen Würmern entwickelt. Vorläufige Studien belegen, dass dieses Protokoll auch für den Einsatz bei L4-Würmern und älteren erwachsenen Würmern wirksam ist. Die Wirksamkeit dieses Protokolls wurde jedoch bei Würmern im frühen Larvenstadium noch nicht untersucht. Eine Einschränkung dieses Ansatzes ist die geringe Menge an Material, die er liefert. Obwohl die Mengen für RNA-seq und PCR ausreichend sind, sind sie für andere Assays möglicherweise nicht ausreichend. Daher müssen Benutzer feststellen, ob der minimal erforderliche Input für einen Assay mit diesem Protokoll erfasst werden kann.
Unser Labor verwendet routinemäßig FACS, um Darmzellen nach der Isolierung zu reinigen 30, Post-hoc-Analysemethoden zur Identifizierung von Darmzellen und diese Handdissektionsmethode30,42. Die Handdissektion hat den Vorteil, dass sie für den Einsatz bei erwachsenen Würmern geeignet ist, wenn die Disaggregation von Würmern und die Zellisolierung weniger erfolgreich sind. Darüber hinaus sind die Effizienz und Qualität der Gesamt-RNA, die aus Handdissektionspräparaten extrahiert wird, hoch, wahrscheinlich weil die Gewebe schnell aus den Würmern gepflückt und dann schnell in einem Nukleinsäureisolierungsreagenz abgelagert werden, wodurch der RNA-Abbau reduziert wird. Ein weiterer Vorteil der Handdissektionsmethode ist, dass sie kostengünstig und leicht zu erlernen ist und keine spezielle Ausrüstung erfordert. Schließlich ermöglicht dieser Ansatz die Ernte und Isolierung von Darmbakterien aus dem Wurmdarm, was nachgeschaltete Mikrobiomstudien ermöglicht.
Das hier beschriebene Handdissektionsprotokoll zur Isolierung von Därmen aus adulten C. elegans stellt ein leistungsfähiges Werkzeug zur Untersuchung verschiedener Aspekte der Biologie von C. elegans dar. Mit einer reinen Darmvorbereitung können Forscher beispielsweise die Schnittstelle zwischen Immunität, Alterung, Stoffwechsel und Mikrobiom untersuchen.
Offenlegungen
Die Autoren erklären keine Interessenkonflikte.
Danksagungen
Wir sind der Pionierarbeit von James McGhee und Barb Goszczynski zu Dank verpflichtet, die ursprünglich die Darmdissektionsmethode entwickelt haben, von der dieses Protokoll adaptiert wird. Unsere Arbeit wird durch einen MIRA (R35) Award unterstützt, der vom National Institute of General Medical Sciences (National Institutes of Health, R35GM124877 an EON) und einem NSF-CAREER Award des NSF MCB Div Of Molecular and Cellular Bioscience (Award #2143849 an EON) betreut wird.
Materialien
| Name | Company | Catalog Number | Comments |
| Acetylated Bovine Serum Albumin (BSA) | VWR | 97061-420 | Nuclease free BSA |
| CL2122 worm strain | CGC (Caenorhabditis Genetics Center) | CL2122 | dvIs15 [(pPD30.38) unc-54(vector) + (pCL26) mtl-2::GFP]. Control strain for CL2120. Phenotype apparently WT. |
| Calcium Chloride Dihydrate | Fisher | C79 | needed for making Egg Salts |
| 50 mL Centrifuge Tubes, Bulk | Olympus Plastics | 28-108 | Nuclease free conical tube needed for solution making. |
| 15 mL Centrifuge Tubes, Bulk | Olympus Plastics | 28-103 | Nuclease free conical tube needed for solution making. |
| Concavity slide (2-well) | Electron Microscopy Sciences | 71878-08 | 12-pk of 2-well concavity slides |
| Ethylene glycol-bis(2-amino-ethylether)-N,N,N',N'-tetraacetic acid (EGTA) | Millipore Sigma | E3889 | needed for making chelation buffer |
| Fluorescent Dissection Microscope | Leica | M205 FCA | This is an optional piece of equipment that can be used with fluorescent C. elegans strains to help guid users during hand dissections |
| N-(2-Hydroxyethyl)piperazine-N′-(2-ethanesulfonic acid), 4-(2-Hydroxyethyl)piperazine-1-ethanesulfonic acid (HEPES) | Millipore Sigma | H4034 | needed for making dissection buffer |
| High Sensitivity RNA ScreenTape | Agilent | 5067-5579 | for assesment of total RNA quality and quantity |
| High Sensitivity RNA ScreenTape Ladder | Agilent | 5067-5581 | for assesment of total RNA quality and quantity |
| High Sensitivity RNA ScreenTape Sample Buffer | Agilent | 5067-5580 | for assesment of total RNA quality and quantity |
| HostZERO Microbial DNA Kit | Zymo Research | D4310 | Isolation of microbial DNA from worm intestines/worms |
| Hypodermic Needle (27G x 1/2") | BD Scientific | 305109 | needed for hand dissection of intestines |
| Levamisole (a.k.a. (-)-Tetramisole hydrochloride) | Millipore Sigma | L9756 | used to temporarily paralyze worms prior to hand dissection of intestines. |
| Luer-Lok General Use Disposable Syringe (1 mL) | BD Scientific | 309628 | Optional. Can be used to affix the hypodermic needle to, allowing easier manipulation of the needle during dissection. Remove the plunger. |
| Magnesium Chloride Hexahydrate | Fisher | M33 | needed for making Egg Salts |
| Magnesium Sulfate Hpetahydrate | Sigma-Aldrich | 230391-500G | needed for making M9 buffer |
| MF-900 Microforge | Narishige | MF-900 | Used to forge the microcapillary pipettes. Available through Tritech Research. |
| 1.7 mL Microtubes, Clear | Olympus Plastics | 22-282 | Nuclease free microfuge tube needed for solution making and sample storage. |
| Mouth Aspirator Tube | Millipore Sigma | A5177 | Mouth aspirator tube is needed in combination with the microcapillary pipette to allow aspiration of dissected intestines. |
| 16S Pan-Bacterial Control TaqMan Assay | Thermo Fisher | A50137 | Assay ID: Ba04930791_s1. Assay used for gut microbial detection via qPCR. |
| P-1000 Micropipette Puller | Sutter Instruments | Model P-1000 | Used to pull the microcapillary pippettes prior to forging. |
| Petri Dish (35 x 10 mm) | Genesee Scientific - Olympus Plastics | 32-103 | Used to make M9 bath and Disseciton Buffer bath for washing worms prior to dissection. |
| Phenol:Chloroform:IAA | Ambion | AM9730 | Used in the isolation of total RNA |
| Potassium Chloride | Millipore Sigma | 529552 | needed for making Egg Salts |
| Potassium Phosphate Monobasic | Sigma-Aldrich | P0662-500G | needed for making M9 buffer |
| Qubit 3 Fluorometer | Invitrogen | Q33216 | Accompanies the Qubit RNA HS Assay Kit. Can be used to quantify RNA prior to running sample on the Agilent ScreenTape. |
| Qubit RNA HS Assay Kit | Invitrogen | Q32852 | Can be used to quantify RNA prior to running sample on the Agilent ScreenTape. |
| RNasin Ribonuclease Inhibitor | Promega | N2111 | Broad spectrum inhibition of common eukaryotic Rnases |
| RNase-Free DNase Set | Qiagen | 79254 | used for on-column DNA digestion during RNA isolation protocol. |
| RNeasy Micro Kit | Qiagen | 74004 | Used for isolation of total RNA from worm intestines/worms |
| Standard Glass Capillaries | World Precision Instruments | 1B100F-4 | 4 in OD 1.2 mm standard borosilicate glass capillaries used to make microcapillary pipettes for dissection |
| Sodium Chloride | Fisher | S271 | needed for making Egg Salts |
| Sodium phosphate dibasic heptahydrate | Fisher Scientific | S373-500 | needed for making M9 buffer |
| Syringe filter (0.2 micrometer SCFA) | Thermo Fisher | 72302520 | Optional for use with the mouth aspirator tube when mouth pipetting. |
| 4150 TapeStation System | Agilent | G2992AA | Accompanies the RNA ScreenTape reagents for assessing RNA quality and quantity |
| TaqPath BactoPure Microbial Detection Master Mix | Applied Biosystems | A52699 | master mix used for qPCR |
| TRIzol Reagent | Thermo Fisher Scientific | 15596026 | Nucleic acid isolation and preservation. QIAzol (Qiagen; 79306) can be substituted if preferred. |
| Worm Pick | NA | NA | Made in house from a pasteur pipette and a platinum wire. See wormbook for details. |
Referenzen
- Riddel, D. L., Blumenthal, T., Meyer, B. J., Priess, J. . C. elegans II. , (1997).
- Corsi, A. K., Wightman, B., Chalfie, M. A transparent window into biology: A primer on Caenorhabditis elegans. Genetics. 200 (2), 387-407 (2015).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 485 (2017).
- Roy, P. J., Stuart, J. M., Lund, J., Kim, S. K. Chromosomal clustering of muscle-expressed genes in Caenorhabditis elegans. Nature. 418 (6901), 975-979 (2002).
- Pauli, F., Liu, Y., Kim, Y. A., Chen, P. -. J., Kim, S. K. Chromosomal clustering and GATA transcriptional regulation of intestine-expressed genes in C. elegans. Development. 133 (2), 287-295 (2005).
- Spencer, W. C., et al. A spatial and temporal map of C. elegans gene expression. Genome Research. 21 (2), 325-341 (2011).
- Spencer, W. C., et al. Isolation of specific neurons from C. elegans larvae for gene expression profiling. PLoS One. 9 (11), 112102 (2014).
- Ma, X., et al. Analysis of C. elegans muscle transcriptome using trans-splicing-based RNA tagging (SRT). Nucleic Acids Research. 44 (21), 156 (2016).
- Blazie, S. M., et al. Comparative RNA-Seq analysis reveals pervasive tissue-specific alternative polyadenylation in Caenorhabditis elegans intestine and muscles. BMC Biology. 13 (1), 4 (2015).
- Blazie, S. M., et al. Alternative polyadenylation directs tissue-specific miRNA targeting in Caenorhabditis elegans somatic tissues. Genetics. 206 (2), 757-774 (2017).
- Gómez-Saldivar, G., et al. et al.Tissue-specific transcription footprinting using RNA PoI DamID (RAPID) in Caenorhabditis elegans. Genetics. 216 (4), 931-945 (2020).
- Katsanos, D., Barkoulas, M. Targeted DamID in C. elegans reveals a direct role for LIN-22 and NHR-25 in antagonizing the epidermal stem cell fate. Science Advances. 8 (5), 3141 (2022).
- Kaletsky, R., et al. The C. elegans adult neuronal IIS/FOXO transcriptome reveals adult phenotype regulators. Nature. 529 (7584), 92-96 (2015).
- Kaletsky, R., et al. Transcriptome analysis of adult Caenorhabditis elegans cells reveals tissue-specific gene and isoform expression. PLoS Genetics. 14 (8), 1007559 (2018).
- Mathies, L. D., et al. mRNA profiling reveals significant transcriptional differences between a multipotent progenitor and its differentiated sister. BMC Genomics. 20 (1), 427 (2019).
- Liang, X., Calovich-Benne, C., Norris, A. Sensory neuron transcriptomes reveal complex neuron-specific function and regulation of mec-2/ Stomatin splicing. Nucleic Acids Research. 50 (5), 2401-2416 (2021).
- Glenwinkel, L., et al. In silico analysis of the transcriptional regulatory logic of neuronal identity specification throughout the C. elegans nervous system. eLife. 10, 64906 (2021).
- Taylor, S. R., et al. Molecular topography of an entire nervous system. Cell. 184 (16), 4329-4347 (2021).
- Charest, J., et al. Combinatorial action of temporally segregated transcription factors. Developmental Cell. 55 (4), 483-499 (2020).
- Steiner, F. A., Talbert, P. B., Kasinathan, S., Deal, R. B., Henikoff, S. Cell-type-specific nuclei purification from whole animals for genome-wide expression and chromatin profiling. Genome Research. 22 (4), 766-777 (2012).
- Tintori, S. C., Nishimura, E. O., Golden, P., Lieb, J. D., Goldstein, B. A transcriptional lineage of the early C. embryo. Developmental Cell. 38 (4), 430-444 (2016).
- Hashimshony, T., Wagner, F., Sher, N., Yanai, I. CEL-Seq: Single-cell RNA-Seq by multiplexed linear amplification. Cell Reports. 2 (3), 666-673 (2012).
- Hashimshony, T., Feder, M., Levin, M., Hall, B. K., Yanai, I. Spatiotemporal transcriptomics reveals the evolutionary history of the endoderm germ layer. Nature. 519 (7542), 219-222 (2015).
- Packer, J. S., et al. A lineage-resolved molecular atlas of C. elegans embryogenesis at single-cell resolution. Science. 365 (6459), 1971 (2019).
- Warner, A. D., Gevirtzman, L., Hillier, L. W., Ewing, B., Waterston, R. H. The C. elegans embryonic transcriptome with tissue, time, and alternative splicing resolution. Genome Research. 29 (6), 1036-1045 (2019).
- Cao, J., et al. Comprehensive single-cell transcriptional profiling of a multicellular organism. Science. 357 (6352), 661-667 (2017).
- Durham, T. J., et al. Comprehensive characterization of tissue-specific chromatin accessibility in L2 Caenorhabditis elegans nematodes. Genome Research. 31 (10), 1952-1969 (2021).
- King, D. C., et al. The transcription factor ELT-2 positively and negatively impacts direct target genes to modulate the Caenorhabditis elegans intestinal transcriptome. bioRxiv. , (2021).
- Kocsisova, Z., Mohammad, A., Kornfeld, K., Schedl, T. Cell cycle analysis in the C. elegans germline with the thymidine analog EdU. Journal of Visualized Experiments. (140), e58339 (2018).
- Han, S., et al. Mono-unsaturated fatty acids link H3K4me3 modifiers to C. elegans lifespan. Nature. 544 (7649), 185-190 (2017).
- Edgar, L. G., Wolf, N., Wood, W. B. Early transcription in Caenorhabditis elegans embryos. Development. 120 (2), 443-451 (1994).
- Edgar, L. G., Goldstein, B. Culture and manipulation of embryonic cells. Methods in Cell Biology. 107, 151-175 (2012).
- Nishimura, E. O., Zhang, J. C., Werts, A. D., Goldstein, B., Lieb, J. D. Asymmetric transcript discovery by RNA-seq in C. elegans blastomeres identifies neg-1, a gene important for anterior morphogenesis. PLoS Genetics. 11 (4), 1005117 (2015).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Cerón, J. Basic Caenorhabditis elegans methods: Synchronization and observation. Journal of Visualized Experiments. (64), e4019 (2012).
- Fay, D. S., Fluet, A., Johnson, C. J., Link, C. D. In vivo aggregation of β-amyloid peptide variants. Journal of Neurochemistry. 71 (4), 1616-1625 (1998).
- Altun, Z. F., Hall, D. H. WormAtas Hermaphrodite Handbook – Introduction to C. elegans Anatomy. WormAtlas. , (2006).
- Tan, M. -. W., Mahajan-Miklos, S., Ausubel, F. M. Killing of Caenorhabditis elegans by Pseudomonas aeruginosa used to model mammalian bacterial pathogenesis. Proceedings of the National Academy of Sciences of the United States of America. 96 (2), 715-720 (1999).
- Oesterle, A. Pipette Cookbook 2018: P-97 & P-1000 Micropipette Pullers. Sutter Instrument Company. , (2018).
- Dineen, A., Nishimura, E. O., Goszczynski, B., Rothman, J. H., McGhee, J. D. Quantitating transcription factor redundancy: The relative roles of the ELT-2 and ELT-7 GATA factors in the C. elegans endoderm. Developmental Biology. 435 (2), 150-161 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten