Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Evaluación de la salud mitocondrial en fibroblastos asociados al cáncer aislados de esferoides tumorales de pulmón multicelulares 3D
* Estos autores han contribuido por igual
En este artículo
Resumen
Se prepararon esferoides tumorales 3D multicelulares con células de adenocarcinoma de pulmón, fibroblastos y monocitos, seguidos del aislamiento de fibroblastos asociados al cáncer (CAF) de estos esferoides. Los CAF aislados se compararon con fibroblastos normales para evaluar la salud mitocondrial mediante el estudio del potencial transmembrana mitocondrial, las especies reactivas de oxígeno y las actividades enzimáticas.
Resumen
Los fibroblastos asociados al cáncer (CAF) se encuentran entre las células estromales más abundantes presentes en el microambiente tumoral, lo que facilita el crecimiento y la progresión del tumor. La complejidad dentro del microambiente tumoral, incluyendo el secretoma tumoral, la inflamación de bajo grado, la hipoxia y el desequilibrio redox, fomenta la interacción heterotípica y permite la transformación de fibroblastos residentes inactivos para convertirse en CAF activos. Los CAF se distinguen metabólicamente de los fibroblastos normales (NF) ya que son más activos glucolíticamente, producen niveles más altos de especies reactivas de oxígeno (ROS) y sobreexpresan el exportador de lactato MCT-4, lo que lleva a la apertura del poro de transición de permeabilidad mitocondrial (MPTP). Aquí se ha descrito un método para analizar la salud mitocondrial de CAF activados aislados de los esferoides tumorales 3D multicelulares que comprenden células de adenocarcinoma de pulmón humano (A549), monocitos humanos (THP-1) y células de fibroblastos de pulmón humano (MRC5). Los esferoides tumorales se desintegraron en diferentes intervalos de tiempo y, mediante la clasificación celular activada magnéticamente, se aislaron los CAF. El potencial de membrana mitocondrial de los CAF se evaluó mediante colorante JC-1, la producción de ROS mediante tinción de 2',7'-diclorodihidrofluoresceína diacetato (DCFDA) y la actividad enzimática en los CAF aislados. El análisis de la salud mitocondrial de CAF aisladas proporciona una mejor comprensión del efecto Warburg inverso y también se puede aplicar para estudiar las consecuencias de los cambios mitocondriales de CAF, como los flujos metabólicos y los mecanismos reguladores correspondientes sobre la heterogeneidad del cáncer de pulmón. Por lo tanto, el presente estudio aboga por una comprensión de las interacciones tumor-estroma en la salud mitocondrial. Proporcionaría una plataforma para verificar la eficacia de los candidatos a fármacos mitocondriales específicos contra los CAF como posibles terapias en el microambiente tumoral, evitando así la participación de CAF en la progresión del cáncer de pulmón.
Introducción
Los tumores sólidos están compuestos por poblaciones celulares heterogéneas que son guiadas por el microambiente tumoral (TME), sin embargo, el origen de la mayoría de las células aún no se ha descubierto. Principalmente las células estromales e inmunes (fibroblastos, células endoteliales, monocitos, macrófagos, células dendríticas, células B, células T y sus subconjuntos) reflejan la heterogeneidad tumoral en los cánceres de pulmón, mama, riñón y otros cánceres sólidos 1,2,3. Comprender el origen de cada subtipo y su potencial de transdiferenciación es de suma necesidad para desarrollar terapias avanzadas contra estos cánceres. El análisis de esta población celular diversa en biopsias humanas se presenta con varios desafíos debido al tipo de tumor, sitio, etapa, limitación de la cantidad de muestra y variabilidades específicas del paciente4. Por lo tanto, se necesita un modelo experimental, que no solo sea confiable sino que también pueda simular la condición tumoral in vivo, demostrando ser ideal para estudiar la diafonía tumor-estroma y su participación en la fisiopatología de la enfermedad.
Los cultivos tridimensionales (3D) multicelulares de esferoides tumorales (MCTS) son un sistema modelo de tumores in vitro ventajoso debido a su parecido con sus contrapartes naturales. MCTS puede replicar mejor aspectos de tumores sólidos que los modelos de cultivo celular 2D, incluida su arquitectura espacial, respuestas fisiológicas, liberación de mediadores solubles, patrones de expresión génica y mecanismos de resistencia a los medicamentos. Además, una ventaja principal de MCTS es que se puede utilizar para estudiar la heterogeneidad tumoral y el microambiente tumoral (TME). El método de caída colgante es la herramienta más comúnmente empleada para desarrollar y analizar MCTS5. En este método, las diferentes células con medios se suspenden en forma de gotas, lo que permite su crecimiento de forma coherente en 3D agregado y es fácil de acceder para su examen. La técnica es sencilla; No requiere muchas células y elimina la necesidad de un sustrato especializado como la agarosa para el desarrollo de esferoides6. Una ventaja adicional de este método radica en la reproducibilidad de su técnica. Además, este método también se ha utilizado para cocultivar poblaciones de células mixtas, como células endoteliales y células tumorales, para simular la angiogénesis tumoral temprana7.
En este estudio, se prepararon esferoides tumorales pulmonares 3D multicelulares con células de adenocarcinoma de pulmón, fibroblastos y monocitos utilizando el método de gota colgante que imita el microambiente del tumor pulmonar. Luego, la población de fibroblastos asociados al cáncer (CAF) se aisló para investigar la salud mitocondrial. La idea principal detrás del desarrollo de estos esferoides es aislar los CAF, ya que la diafonía entre las células en los esferoides podría transformar los fibroblastos en un estado de CAF activado similar al miofibroblasto. En segundo lugar, este estudio también puede representar cómo la producción aberrante de ROS y la disfunción mitocondrial impulsan a los fibroblastos normales hacia el fenotipo CAF más agresivo. Se encontró que los fibroblastos ensamblados dentro de los esferoides tumorales ganaron características miofibroblásticas, con el aumento de la actividad de ROS y la inducción de la expresión génica metabólica. Este protocolo destaca la importancia del microambiente tumoral en la activación de CAF y podría ser un excelente modelo para la generación in vitro y el estudio de las características fenotípicas de CAF.
Protocolo
1. Cultivo celular
- Cultivo de adenocarcinoma de pulmón humano línea celular A549, y línea celular monocítica humana THP-1 en medios RPMI1640 suplementados con 10% FBS y 1% de penicilina-estreptomicina a 37 °C en una cámara humidificada con 5% deCO2.
- Cultivo de células de fibroblastos pulmonares humanos MRC-5 en medio DMEM suplementado con FBS al 10% y solución de penicilina-estreptomicina al 1% a 37 °C en una cámara humidificada con 5% deCO2.
2. Preparación de esferoides tumorales multicelulares utilizando línea celular de adenocarcinoma de pulmón A549, fibroblastos MRC5 y monocitos THP-1
NOTA: Los esferoides 3D tumorigénicos multicelulares y no tumorigénicos se prepararon utilizando el método de gota colgante en una placa de cultivo celular de 90 mm. Una descripción detallada del desarrollo de estos esferoides se da a continuación. Todos los reactivos de cultivo celular, como el medio completo, PBS y la solución de tripsina-EDTA al 0,25%, deben calentarse previamente a 37 °C antes de su uso, a menos que se indique lo contrario.
- Preparación de la suspensión celular
- Cultivar células adherentes a A549 y MRC5 (5 x 106 células cada una) en medio de crecimiento completo DMEM (medio de águila modificada de Dulbecco [DMEM] + 10% de suero bovino fetal [FBS] + 1% de penicilina-estreptomicina) en matraces T25 a 37 °C en una cámara humidificada con 5% deCO2.
- Para las células THP-1, cultivar la suspensión celular (5 x 106 células) en medio de crecimiento completo (RPMI1640 + 10% FBS + 1% penicilina-estreptomicina) en matraces T25. Para un mejor crecimiento, colocar los matraces T25 a 37 °C en una cámara humidificada con un 5% deCO2 en posición de pie.
- Después de 3 días, al 80%-85% de confluencia, lavar las células A549 y MRC5 con PBS (solución salina de fosfato) libre de calcio y magnesio añadiendo 1 ml de PBS (25-30 °C) en cada matraz durante 1 min y aspirándolo.
- Extraer las células A549 y MRC5 del matraz incubándolas con 500 μL de solución de tripsina-EDTA al 0,25% durante 5 min a 37 °C en una incubadora humidificada con 5% deCO2. Inmediatamente después, agregue 4 ml de medios de crecimiento completos para inactivar la tripsina.
- Recoger la suspensión celular del matraz T25 en un tubo de 15 ml y granularla a 125 x g durante 5 min. Retire el sobrenadante y resuspenda las células en 5 ml de medio de crecimiento completo.
- Establecimiento de esferoides tumorales multicelulares
NOTA: Todos los pasos de la formación del esferoide tumoral deben realizarse dentro del gabinete de bioseguridad para mantener condiciones estériles. El volumen máximo de la suspensión celular se ha estandarizado como una gota de 25 μL para preparar una gota de tal manera que no se caiga mientras se invierten las tapas.- Cuente los números de células A549, MRC5 y THP-1 con un contador de celdas. Para cada gota de esferoide (25 μL) mantener los siguientes números celulares: 5.000 células A549, 4.000 células MRC5 y 1.000 células THP-1, siguiendo el protocolo descrito por Arora et al.7. Calcule los números de celda en consecuencia para un volumen de 1 ml.
NOTA: Los esferoides se prepararon inicialmente con tres recuentos celulares diferentes por esferoide (es decir, 5000, 8000 y 10,000). Además, se verificaron diferentes proporciones celulares de células tumorales/fibroblastos/monocitos (1:1:1, 2:2:1, 4:2:1, 5:2:1 y 5:4:1). Finalmente, se observó una formación exitosa de esferoides multicelulares 3D con una proporción de 5: 4: 1 y se utilizó para el estudio. La concentración de fibroblastos aumentó en función de su proporción en el microambiente tumoral, lo que mejoró aún más la rigidez de los esferoides tumorales. El procedimiento detallado fue reportado en una publicación reciente de Arora et al.7. - Prepare la suspensión celular en una proporción de 5 (A549, 2 x 105 células/ml) a 4 (MRC5, 1,6 x 10 5 células/ml) a 1 (THP-1,5 x 104 células/ml) y enrasar el volumen a 1 ml con DMEM completo.
- Colocar una gota de 25 μL de mezcla de suspensión celular sobre la cubierta de una placa de cultivo de 90 mm (aproximadamente 50 gotas/placa de 90 mm). Llene el fondo de la placa de cultivo de 90 mm con 10 ml de agua estéril.
- Invierta cuidadosamente la tapa sobre la cámara de hidratación llena de agua y coloque el plato en una incubadora de cultivo celular durante 3 días.
- Monitoree los esferoides bajo el microscopio con un aumento de 10x el día 4. Para adquirir imágenes, encienda el interruptor de encendido, coloque el plato de 60 mm con cuidado en el escenario y seleccione el aumento (10x). Ajuste las lentes y examine las células para analizar la agregación y proliferación celular. Pulse los botones Congelar y Guardar que se incluyen en el microscopio para capturar la imagen.
- Cambie el medio de crecimiento el día 4 aspirando cuidadosamente 20 μL de medio de cada gota y reemplazándolo con un medio de crecimiento completo fresco.
- Cuente los números de células A549, MRC5 y THP-1 con un contador de celdas. Para cada gota de esferoide (25 μL) mantener los siguientes números celulares: 5.000 células A549, 4.000 células MRC5 y 1.000 células THP-1, siguiendo el protocolo descrito por Arora et al.7. Calcule los números de celda en consecuencia para un volumen de 1 ml.
3. Análisis de muerte viva de esferoides tumorales
- Los días 7 y 10, invierta cuidadosamente el plato de 90 mm en el gabinete de bioseguridad y use una pipeta de 200 μL para recoger los esferoides de cada gota. Recoja cinco esferoides cada uno en un tubo de 1,5 ml.
- Añadir 500 μL de 1x PBS al tubo de 1,5 ml que contiene esferoides y centrifugar a 125 x g durante 5 min. Deseche el sobrenadante con cuidado y resuspenda los esferoides en 200 μL de 1x PBS. No pipetear rigurosamente para evitar la desintegración del esferoide.
- Pipetear los esferoides utilizando una pipeta de 200 μL en una placa de 60 mm para la tinción de calceína-AM y yoduro de propidio.
- Poner 5 μL de solución de calceína-AM de 1 μM y 5 μL de solución de yoduro de propidio de 2 mg/ml sobre los esferoides. Incubar durante 10 min. Después de completar el período de incubación, lave los esferoides suavemente con 1x PBS dos veces.
- Coloque la antena parabólica de 60 mm que contiene esferoides bajo el microscopio invertido fluorescente, observe y capture imágenes con un aumento de 10x seleccionando la opción de fluorescencia en el software del microscopio y seleccionando el FITC (canal de fluorescencia verde; excitación 490 nm, emisión 515 nm) para calceína y canal rojo de Texas (TXR; excitación 535 nm, emisión 617 nm).
- Para adquirir la imagen, encienda Ctr Adv, que es el interruptor 1, seguido de encender la CPU. Espere a que arranque el sistema de software. Cuando arranque, coloque el plato de 60 mm con cuidado en el escenario y seleccione los canales de aumento (10x) y fluorescentes (FITC, TXR). Ajuste las lentes y busque la imagen.
- Para ver la imagen en el sistema, seleccione la opción Live y ver la imagen. Ajuste la intensidad de la fluorescencia para una optimización adecuada. Guarde la imagen haciendo clic en el botón Guardar .
NOTA: En esta etapa, los esferoides serán visibles a simple vista. Bajo el microscopio óptico, los esferoides aparecerán como esferas redondas y rígidas con un aumento de 10x. Se pueden recolectar múltiples esferoides a la vez usando una pipeta de 200 μL.
4. Desintegración y suspensión celular de los esferoides tumorales
- Recolectar 200 esferoides tumorales cada uno los días 7 y 10, usando una pipeta de 1 ml en un tubo de 15 ml.
NOTA: Antes de la recolección, verifique cuidadosamente la forma de un solo esferoide después de transferirlo a un portaobjetos microscópico o en un plato de 30 mm y observe bajo el microscopio. - Granular los esferoides por centrifugación a 125 x g durante 5 min. Aspirar el sobrenadante cuidadosamente sin alterar los esferoides tumorales.
- Lave cuidadosamente los esferoides con 200 μL de PBS, centrifugar a 125 x g durante 5 min y desechar con precaución el sobrenadante.
- Para la desintegración del esferoide, añadir 400 μL de solución de tripsina-EDTA al 0,25% y mantener a 37 °C durante 10 min. Realizar un pipeteo vigoroso para la desintegración completa de los esferoides.
- Neutralizar la tripsina añadiendo 1 ml de medio de crecimiento DMEM completo. Centrifugar a 125 x g durante 5 min y desechar el sobrenadante con cuidado.
- Resuspender el pellet en 1 mL de medio DMEM completo y contar el número total de células.
5. Aislamiento de fibroblastos asociados al cáncer (CAF) a través de microperlas
- Para el aislamiento de CAF de los esferoides tumorales, resuspender 1 x 107 células en 80 μL de tampón de clasificación de células activadas magnéticas frías (MACS) (PBS a pH 7.2 que contiene 0.5% de albúmina sérica bovina [BSA] y 2 mM de ácido etilendiamina tetraacético [EDTA]).
- Incubar la suspensión celular (que contiene 1 x 107 células) con 20 μL de microperlas antifibroblastos. Mezclar bien golpeando suavemente el tubo e incubar durante 30 minutos a temperatura ambiente.
- Lave las celdas con 1 ml de tampón MACS frío, centrifugar a 125 x g durante 5 min y aspirar el sobrenadante. Resuspender las células en 500 μL de tampón MACS.
- Para la separación celular basada en perlas magnéticas, prepare la columna MACS enjuagándola con 3 ml de tampón MACS.
- Coloque la suspensión celular en la columna seguida de la colección de flujo que contiene la población celular no etiquetada.
- Lave la columna tres veces con 3 ml de tampón MACS. Retire la columna del separador y colóquela en el tubo de recolección.
- Recoja las células marcadas con microperlas antifibroblastos agregando 5 ml de tampón MACS y empujando firmemente el émbolo hacia la columna.
- Centrifugar las celdas marcadas a 125 x g durante 5 min. Proceda con los fibroblastos aislados para aplicaciones posteriores.
6. Análisis basado en citometría de flujo de la expresión de ACTA2 en CAFs aisladas
- Cuente el número de CAF aisladas y use aproximadamente 6 x 104 celdas. Lave las células una vez con PBS, centrifugar a 125 x g durante 5 min y aspirar el sobrenadante.
- Añadir 100 μL de tampón de permeabilización celular (PBS + 0,5% BSA + 0,3% v/v Triton X-100) e incubar las células a 4 °C durante 30 min.
- Vórtice las células intermitentemente para mantener una suspensión de una sola célula. Centrifugar y resuspender las células en 100 μL de tampón de permeabilización celular.
- Añadir 2 μL de anticuerpo antihumano α-SMA conjugado APC e incubar a 4 °C durante 45 min. Después de la incubación, añadir 1 ml de tampón de permeabilización y centrifugar a 125 x g durante 5 min para eliminar el exceso de anticuerpos.
- Resuspender el pellet en 400 μL de tampón de permeabilización para análisis citométrico de flujo. Adquirir un total de 10.000 eventos de cada muestra en un citómetro de flujo. Seleccione las células positivas para ACTA2 después de distinguir las poblaciones de células en función de su dispersión hacia adelante y hacia el lado, seleccionando la población de singletes seguida de la selección de las células positivas para ACTA2 que aparecen como un pico único en el histograma de parámetro único.
7. Tinción JC-1 para determinar el potencial de la membrana mitocondrial
- Contar el número de CAF aislados y utilizar aproximadamente 6 x 104 células para la tinción de 5,5,6,6'-tetracloro-1,1',3,3' tetraetilbenzimi-dazoylcarbocyanine yoduro (JC-1) mediante citometría de flujo.
- Lave bien las células con PBS, centrifugar a 125 x g durante 5 min, aspirar el sobrenadante y agregar 100 μL de tampón de tinción celular.
- Añadir el colorante JC-1 a una concentración de trabajo de 2 μM e incubar a temperatura ambiente durante 30 min. Al finalizar la incubación, lavar las células con PBS, centrifugar a 125 x g durante 5 min y volver a suspender en un volumen final de 400 μL.
- Adquirir un total de 20.000 eventos de cada muestra en un citómetro de flujo. Cuantificar el potencial de membrana mitocondrial midiendo los agregados JC-1 desplazados al rojo en el canal FL-2 y los monómeros desplazados al verde en el canal FL-1.
8. Tinción DCFDA para estimar los niveles de especies reactivas de oxígeno celular (ROS)
- Utilice los esferoides enteros (50 números), así como los CAF aislados (6 x 104 células) de los esferoides para la tinción de diacetato de 2',7'-diclorodihidrofluoresceína (DCFDA).
- Lave bien las células con PBS, centrifugar a 125 x g durante 5 min, aspirar el sobrenadante y agregar 100 μL de tampón de tinción celular.
- Añadir colorante DCFDA a una concentración de trabajo de 1 μM e incubar a temperatura ambiente durante 30 min. Lave las células dos veces con PBS, centrifugar a 125 x g durante 5 min y luego volver a suspender en un volumen final de 400 μL.
- Adquirir un total de 20.000 eventos de cada muestra en un citómetro de flujo. Evaluar los niveles de ROS midiendo la fluorescencia en el día 7 y el día 10 para los esferoides, así como los CAF aislados.
9. Análisis RT-qPCR de marcadores CAF y genes glucolíticos
- Extraiga el ARN de los CAF clasificados utilizando un kit de lisis unicelular siguiendo el protocolo del fabricante. Prepare ADNc a partir de 100 ng de ARN utilizando un kit de síntesis de ADNc siguiendo las pautas del fabricante.
- Realizar RT-qPCR para analizar los marcadores relativos CAF (ACTA2 8 y COL1A29) y la expresión del gen glucolítico (GLUT1 10 y MCT4 11). Realice un análisis de la curva de fusión después de la extensión final para garantizar la especificidad de los productos. Normalizar los datos utilizando la expresión de GAPDH como gen de referencia. Las secuencias de cebadores utilizadas para la RT-qPCR se enumeran en la Tabla Suplementaria 1.
10. Extracción y cuantificación de la proteína celular de CAFs
NOTA: Realice todos los pasos de extracción de proteínas en hielo para evitar la degradación de proteínas.
- Resuspender aproximadamente 4 x 106 células CAF en 100 μL de tampón de lisis RIPA helado (que contiene 30 mM de HEPES, 150 mM de NaCl, 1% de NP-40, 0,1% de SDS y 0,5% de desoxicolato de sodio con 5 mM de cóctel de inhibidores de la proteasa y fosfatasa Halt, y 5 mM de EDTA a pH 7,4).
- Vórtice rigurosamente para una lisis celular adecuada. Sonicar la suspensión celular a una frecuencia de 20 Hz, una amplitud del 20% durante 15 s y un pulso de 3x.
- Después de la sonicación, centrifugar el extracto de proteína a 13.000 x g durante 15 minutos a 4 °C. Transfiera el sobrenadante a un tubo preenfriado de 1,5 ml. Realice el ensayo de proteína BCA para cuantificar la proteína.
11. Análisis espectrofotométrico de actividades enzimáticas en CAFs
NOTA: Las siguientes actividades enzimáticas se analizan en CAF derivadas de esferoides tumorales.
- Medición de la actividad de la succinato deshidrogenasa
- Para evaluar la actividad de la succinato deshidrogenasa (SDH) en CAFs, prepare tampón SHE con sacarosa de 250 mM, ácido 4-(2-hidroxietil)-1-piperazinaetanosulfónico (HEPES) de 10 mM y 1 mM de etilenglicol-bis(β-aminoetil éter)-N,N,N′,N′-ácido tetraacético (EGTA) y ajuste el pH a 7.3. Añadir 0,4 mM de metosulfato de fenazina (PMS), 0,2 mM de 2,6-dicloroindofenol (DCPIP), 50 mM de MgCl2, Triton X-100 al 0,02% y cianuro de 1 mM en el tampón SHE y mantenerlo a 37 °C.
- Extraiga la proteína celular de los CAF como se describe en el paso 10 (2 x 106 células). En cada pocillo de una placa de 96 pocillos, incubar 100 μL de 0,1 mg de proteína celular con 100 μL de mezcla de reacción SHE a 37 °C durante 15 min.
- Para iniciar la actividad enzimática, añadir 10 mM de succinato12. Calcular la actividad enzimática en nM/min/mL midiendo los cambios en la absorbancia a 600 nm.
- El factor de conversión para DCIP es 0.0215 A/nM basado en su coeficiente de extinción molar12. Calcule la actividad relativa de SDH utilizando la fórmula mencionada:
Actividad relativa (nM/min/ml/enzima) =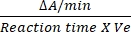 X X
X X  V
V
Donde ΔA/min = velocidad de reacción enzimática (A inicial- A final)/(tiempofinal- A/min inicial), Ve = volumen de la muestra, y V = volumen de la reacción.
- Estimación de la actividad de la citocromo c oxidasa
- Para evaluar la actividad de la citocromo c oxidasa (COX) en CAFs, añadir 0,2 mg/ml de proteína celular en una solución tampón de reacción KME que contenga tampón KME (125 mM KCl, 20 mM 3-(N-morfolino)propano sulfónico ácido [MOPS] y 1 mM EGTA, pH 7,4), 0,02 % Triton X-100 y 5 mM de ascorbato de sodio.
- En un tubo separado, mezclar 50 μM de citocromo c del corazón de caballo con 5 mM de ascorbato de sodio e incubar a 37 °C durante 5 min13. Después de la incubación, añadir 20 μL de citocromo c reducido a 500 μL de solución tampón de reacción KME que contenga proteína celular.
- Calcular la actividad enzimática en unidades/μL midiendo los cambios en la absorbancia a 550 nm. La absorción del citocromo c a 550 nm cambia con su estado de oxidación. La diferencia en los coeficientes de extinción (mM) entre el citocromo c reducido y oxidado es de 21,84 a 550 nm14. Calcule la cantidad de actividad enzimática en el lisado celular como:
Unidades/μL =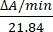
Donde ΔA/min = A/minmuestra-A/minen blanco y 21,84 = ΔґmM entre el citocromo c oxidado y el citocromo c reducido a 550 nm
- Evaluación de la actividad de la lactato deshidrogenasa
- Realice un ensayo de lactato deshidrogenasa (LDH) utilizando un kit de ensayo de células de lactato deshidrogenasa siguiendo las instrucciones del fabricante.
- Añadir reactivo de prueba de LDH (10 μL) a 0,5 mg/ml de proteínas celulares e incubar durante 5 min a 37 °C. Al finalizar la incubación, medir la absorbancia a 45 nm cada 1 min durante 8 min.
- Preparar los estándares NADH para la detección colorimétrica utilizando 0 (en blanco), 2.5, 5, 7.5, 10 y 12.5 nM/pocillo de NADH seguido de la adición de reactivo de prueba LDH a un volumen final de 50 μL.
- Trazar las diferencias en la absorbancia entre Tinicial y Tfinal a la curva estándar de NADH para determinar la cantidad de generación de NADH. Evalúe la actividad de la LDH utilizando la fórmula mencionada:
Actividad LDH =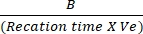 X factor de dilución de la muestra
X factor de dilución de la muestra
Donde B = cantidad (nmole) de NADH generada entre T inicial y T final, tiempo de reacción = Tfinal - Tinicial (min), y Ve = volumen de muestra (mL) agregado al pozo.
Resultados
La Figura 1 muestra el desarrollo de esferoides tumorales multicelulares utilizando tres poblaciones celulares diferentes: A549 (adenocarcinoma de pulmón), MRC-5 (fibroblastos) y THP-1 (monocitos), por el método de gota colgante como se observó en el día 7 y el día 10 bajo el microscopio. El día 7, los esferoides eran compactos y rígidos con un diámetro de 260 ± 5,3 μm, y el día 10, los esferoides tenían 480 ± 7,5 μm de diámetro (Figura 1A panel s...
Discusión
El presente estudio introduce el desarrollo de esferoides tumorales multicelulares que comprenden células tumorales, población de células estromales (es decir, fibroblastos) y población de células inmunes (es decir, monocitos) utilizando un método modificado de gota colgante. Los fibroblastos y los monocitos/macrófagos se encuentran entre las poblaciones más significativas que constituyen el microambiente tumoral (EMT), y su presencia a menudo está relacionada con un mal pronósticodel paciente...
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Este trabajo fue apoyado por el Proyecto del Premio a la Excelencia de las Mujeres SERBIAS, India (SB/WEA-02/2017) y el Proyecto del Premio de Investigación de Carrera Temprana SERB, India (ECR/2017/000892) al PD. Los autores, LA y SR reconocen a IIT Ropar y MHRD por sus becas de investigación. MK reconoce a ICMR por su beca de investigación.
Materiales
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| APC anti-human α-SMA | R&D systems | Cat# IC1420A | |
| Anti-fibroblast microbeads | Miltenyi Biotec | Cat# 130-050-601 | |
| Cell lines | |||
| A549 lung adenocarcinoma cells | NCCS Pune | - | |
| MRC-5 fetal lung fibroblasts | ATCC | CCL-171 | |
| THP-1 Human monocytes | NCCS Pune | - | |
| Chemicals | |||
| BSA | Himedia | Cat# 9048-46-8 | |
| 2,6-dichloroindophenol (DCPIP) | SRL | Cat# 55287 | |
| Calcein-AM | Thermo Fisher Scientific | Cat# C3099 | |
| DAPI | Thermo Fisher Scientific | Cat# D1306 | |
| DCFDA | Sigma | Cat# D6883 | |
| DMEM | Gibco | Cat# 11995073 | |
| DPBS | Gibco | Cat# 14190-144 | |
| EDTA | Thermo fisher scientific | Cat# 17892 | |
| EGTA | SRL | Cat# 62858 | |
| EZcoun Lactate Dehydrogenase Cell Assay Kit | HiMedia | Cat# CCK036 | |
| FBS | Gibco | Cat# 10082147 | |
| Halt Protease and Phosphatase Inhibitor Cocktail (100X) | Thermo Fisher Scientific | Cat# 87786 | |
| HEPES | Thermo Fisher Scientific | Cat# 15630080 | |
| Horse heart Cytochrome c | SRL | Cat# 81551 | |
| Image-iT Red hypoxia reagent | Thermo Fisher Scientific | Cat# H10498 | |
| JC-1 Dye | Thermo Fisher Scientific | Cat# T3168 | |
| KCl | Merck | Cat# P9541 | |
| MgCl2 | Merck | Cat# M8266 | |
| MOPS | Thermo Fisher Scientific | Cat# 69824 | |
| Nacl | Sigma-Aldrich | Cat# S9888 | |
| NADH MB Grade | SRL | Cat# 54941 | |
| NP-40 | Thermo Fisher Scientific | Cat# 85124 | |
| Penicillin/Streptomycin | Gibco | Cat# 15140122 | |
| Phenazine methosulfate (PMS) | SRL | Cat# 55782 | |
| Propidium iodide | Thermo fisher scientific | Cat# P1304MP | |
| RPMI 1640 | Gibco | Cat# 11875093 | |
| Single Cell Lysis Kit | Thermo Fisher Scientific | Cat# 4458235 | |
| Sodium ascorbate | Merck | Cat# A7631 | |
| Sodium cyanide | Sigma | Cat# 205222 | |
| Sodium Deoxycholate | Thermo Fisher Scientific | Cat# 89904 | |
| Sodium dodecyl sulphate | Sigma-Aldrich | Cat# L3771 | |
| Sodium succinate hexahydrate | SRL | Cat# 36313 | |
| Sucrose | Sigma | Cat# S0389 | |
| SuperScript VILO cDNA synthesis kit | Thermo Fisher Scientific | Cat# 11754-050 | |
| Triton X-100 | Sigma | Cat# T8787 | |
| Trypsin 0.25% EDTA | Gibco | Cat# 25200072 | |
| Universal SYBR Green Supermix | BIO-RAD | Cat# 172-5124 | |
| Plasticware | |||
| MACS LS Columns | Miltenyi Biotec | Cat# 130-042-401 | |
| Equipment | |||
| Countess II FL Automated Cell Counter | Thermo Fisher Scientific | Cat# AMQAF1000 | |
| EVOS XL core imaging system | Thermo Fisher Scientific | Serial Number F0518-1727-0191 | |
| LAS X software | Leica Microsystems | ||
| Leica fluorescent inverted microscope | s | DMi8 automated S/N 424150) | |
| Midi MACS separator | Miltenyi Biotec | Cat# 130-042-302 |
Referencias
- Kim, N., et al. Single-cell RNA sequencing demonstrates the molecular and cellular reprogramming of metastatic lung adenocarcinoma. Nature Communications. 11 (1), 1-5 (2020).
- Davidson, S., et al. Single-cell RNA sequencing reveals a dynamic stromal niche that supports tumor growth. Cell Reports. 31 (7), 107628 (2020).
- Zhang, Y., et al. Single-cell analyses of renal cell cancers reveal insights into tumor microenvironment, cell of origin, and therapy response. Proceedings of the National Academy of Sciences. 118 (24), (2021).
- Bray, L. J., Hutmacher, D. W., Bock, N. Addressing patient specificity in the engineering of tumor models. Frontiers in Bioengineering and Biotechnology. 7, 217 (2019).
- Timmins, N. E., Nielsen, L. K. Generation of multicellular tumor spheroids by the hanging-drop method. Tissue Engineering. , 141-151 (2007).
- Dituri, F., et al. Complex tumor spheroid formation and one-step cancer-associated fibroblasts purification from hepatocellular carcinoma tissue promoted by inorganic surface topography. Nanomaterials. 11 (12), 3233 (2021).
- Arora, L., et al. Development of a multicellular 3D tumor model to study cellular heterogeneity and plasticity in NSCLC tumor microenvironment. Frontiers in Oncology. 12, 881207 (2022).
- Nurmik, M., Ullmann, P., Rodriguez, F., Haan, S., Letellier, E. In search of definitions: Cancer-associated fibroblasts and their markers. International Journal of Cancer. 146 (4), 895-905 (2020).
- Zhang, Y., et al. HIF-1α is necessary for activation and tumour-promotion effect of cancer-associated fibroblasts in lung cancer. Journal of Cellular and Molecular Medicine. 25 (12), 5457-5469 (2021).
- Bu, L., et al. Biological heterogeneity and versatility of cancer-associated fibroblasts in the tumor microenvironment. Oncogene. 38 (25), 4887-4901 (2019).
- Whitaker-Menezes, D., et al. Evidence for a stromal-epithelial "lactate shuttle" in human tumors: MCT4 is a marker of oxidative stress in cancer-associated fibroblasts. Cell cycle. 10 (11), 1772-1783 (2011).
- Mandujano-Tinoco, E. A., Gallardo-Pérez, J. C., Marín-Hernández, A., Moreno-Sánchez, R., Rodríguez-Enríquez, S. Anti-mitochondrial therapy in human breast cancer multi-cellular spheroids. Biochimica et Biophysica Acta (BBA)-Molecular Cell Research. 1833 (3), 541-551 (2013).
- Bregman, A. A. . Laboratory Investigations in Cell and Molecular Biology. , (2002).
- Berry, E. A., Trumpower, B. L. Simultaneous determination of hemes a, b, and c from pyridine hemochrome spectra. Analytical Biochemistry. 161 (1), 1-15 (1987).
- Avagliano, A., et al. Metabolic reprogramming of cancer associated fibroblasts: the slavery of stromal fibroblasts. BioMed Research International. , (2018).
- Lorusso, G., Rüegg, C. The tumor microenvironment and its contribution to tumor evolution toward metastasis. Histochemistry and Cell Biology. 130 (6), 1091-1103 (2008).
- Liu, T., Zhou, L., Li, D., Andl, T., Zhang, Y. Cancer-associated fibroblasts build and secure the tumor microenvironment. Frontiers in Cell and Developmental Biology. 7, 60 (2019).
- Sebastian, A., et al. Single-cell transcriptomic analysis of tumor-derived fibroblasts and normal tissue-resident fibroblasts reveals fibroblast heterogeneity in breast cancer. Cancers. 12 (5), 1307 (2020).
- Elyada, E., et al. Cross-species single-cell analysis of pancreatic ductal adenocarcinoma reveals antigen-presenting cancer-associated fibroblasts. Cancer Discovery. 9 (8), 1102-1123 (2019).
- Ganguly, D., et al. Cancer-associated fibroblasts: Versatile players in the tumor microenvironment. Cancers. 12 (9), 2652 (2020).
- Harryvan, T. J., Verdegaal, E. M., Hardwick, J. C., Hawinkels, L. J., vander Burg, S. H. Targeting of the cancer-associated fibroblast-T-cell axis in solid malignancies. Journal of Clinical Medicine. 8 (11), 1989 (2019).
- Santi, A., Kugeratski, F. G., Zanivan, S. Cancer associated fibroblasts: the architects of stroma remodeling. Proteomics. 18 (5-6), 1700167 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados