È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Valutazione della salute mitocondriale nei fibroblasti associati al cancro isolati da sferoidi tumorali polmonari multicellulari 3D
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Gli sferoidi tumorali 3D multicellulari sono stati preparati con cellule di adenocarcinoma polmonare, fibroblasti e monociti, seguiti dall'isolamento dei fibroblasti associati al cancro (CAF) da questi sferoidi. I CAF isolati sono stati confrontati con fibroblasti normali per valutare la salute mitocondriale studiando il potenziale transmembrana mitocondriale, le specie reattive dell'ossigeno e le attività enzimatiche.
Abstract
I fibroblasti associati al cancro (CAF) sono tra le cellule stromali più abbondanti presenti nel microambiente tumorale, facilitando la crescita e la progressione del tumore. La complessità all'interno del microambiente tumorale, tra cui secretoma tumorale, infiammazione di basso grado, ipossia e squilibrio redox, favorisce l'interazione eterotipica e consente la trasformazione dei fibroblasti residenti inattivi per diventare CAF attivi. I CAF sono metabolicamente distinti dai fibroblasti normali (NF) in quanto sono più glicoliticamente attivi, producono livelli più elevati di specie reattive dell'ossigeno (ROS) e sovraesprimono l'esportatore di lattato MCT-4, portando all'apertura del poro di transizione della permeabilità mitocondriale (MPTP). Qui è stato descritto un metodo per analizzare la salute mitocondriale di CAF attivati isolati dagli sferoidi tumorali 3D multicellulari comprendenti cellule di adenocarcinoma polmonare umano (A549), monociti umani (THP-1) e cellule di fibroblasti polmonari umani (MRC5). Gli sferoidi tumorali sono stati disintegrati a diversi intervalli di tempo e attraverso la selezione cellulare attivata magneticamente, i CAF sono stati isolati. Il potenziale di membrana mitocondriale dei CAF è stato valutato utilizzando il colorante JC-1, la produzione di ROS mediante colorazione con 2',7'-diclorodiidrofluoresceina diacetato (DCFDA) e l'attività enzimatica nei CAF isolati. L'analisi della salute mitocondriale dei CAF isolati fornisce una migliore comprensione dell'effetto Warburg inverso e può anche essere applicata per studiare le conseguenze dei cambiamenti mitocondriali della CAF, come i flussi metabolici e i corrispondenti meccanismi regolatori sull'eterogeneità del cancro del polmone. Pertanto, il presente studio sostiene una comprensione delle interazioni tumore-stroma sulla salute mitocondriale. Fornirebbe una piattaforma per verificare i candidati farmaci mitocondriali specifici per la loro efficacia contro i CAF come potenziali terapie nel microambiente tumorale, prevenendo così il coinvolgimento del CAF nella progressione del cancro del polmone.
Introduzione
I tumori solidi sono composti da popolazioni cellulari eterogenee che sono guidate dal microambiente tumorale (TME), tuttavia, l'origine della maggior parte delle cellule deve ancora essere scoperta. Principalmente le cellule stromali e immunitarie (fibroblasti, cellule endoteliali, monociti, macrofagi, cellule dendritiche, cellule B, cellule T e loro sottogruppi) riflettono l'eterogeneità tumorale nei tumori polmonari, mammari, renali e altri tumori solidi 1,2,3. Comprendere l'origine di ciascun sottotipo e il loro potenziale di trans-differenziazione è della massima necessità per lo sviluppo di terapie avanzate contro questi tumori. L'analisi di questa variegata popolazione cellulare nelle biopsie umane presenta diverse sfide a causa del tipo di tumore, del sito, dello stadio, della limitazione della quantità del campione e delle variabilità specifiche del paziente4. Pertanto, è necessario un modello sperimentale, che non sia solo affidabile, ma possa anche simulare la condizione tumorale in vivo, dimostrandosi ideale per lo studio della diafonia tumore-stroma e il suo coinvolgimento nella fisiopatologia della malattia.
Le colture tridimensionali (3D) di sferoidi tumorali multicellulari (MCTS) sono un sistema modello in vitro vantaggioso di tumori a causa della loro somiglianza con le controparti naturali. MCTS può replicare meglio gli aspetti dei tumori solidi rispetto ai modelli di coltura cellulare 2D, compresa la loro architettura spaziale, le risposte fisiologiche, il rilascio di mediatori solubili, i modelli di espressione genica e i meccanismi di resistenza ai farmaci. Inoltre, uno dei principali vantaggi della MCTS è che può essere utilizzato per studiare l'eterogeneità tumorale e il microambiente tumorale (TME). Il metodo hanging-drop è lo strumento più comunemente impiegato per lo sviluppo e l'analisi di MCTS5. In questo metodo, le diverse cellule con i media sono sospese sotto forma di goccioline, il che consente la sua crescita in modo coerente aggregato 3D ed è semplice da accedere per l'esame. La tecnica è semplice; Non richiede molte cellule ed elimina la necessità di un substrato specializzato come l'agarosio per lo sviluppo di sferoidi6. Un ulteriore vantaggio di questo metodo risiede nella riproducibilità della sua tecnica. Inoltre, questo metodo è stato utilizzato anche per co-coltivare popolazioni cellulari miste, come cellule endoteliali e cellule tumorali, per simulare l'angiogenesi tumorale precoce7.
In questo studio, sferoidi tumorali polmonari 3D multicellulari sono stati preparati con cellule di adenocarcinoma polmonare, fibroblasti e monociti utilizzando il metodo della goccia sospesa che imita il microambiente del tumore polmonare. Quindi la popolazione di fibroblasti associati al cancro (CAF) è stata isolata per studiare la salute mitocondriale. L'idea principale alla base dello sviluppo di questi sferoidi è quella di isolare i CAF poiché la diafonia tra le cellule negli sferoidi potrebbe trasformare i fibroblasti in uno stato CAF attivato simile ai miofibroblasti. In secondo luogo, questo studio può anche descrivere come la produzione aberrante di ROS e la disfunzione mitocondriale spingano i fibroblasti normali verso il fenotipo CAF più aggressivo. È stato scoperto che i fibroblasti assemblati all'interno degli sferoidi tumorali hanno acquisito caratteristiche miofibroblastiche con una maggiore attività ROS e l'induzione dell'espressione genica metabolica. Questo protocollo evidenzia l'importanza del microambiente tumorale nell'attivazione della CAF e potrebbe essere un ottimo modello per la generazione in vitro e lo studio delle caratteristiche fenotipiche della CAF.
Protocollo
1. Coltura cellulare
- Linea cellulare di adenocarcinoma polmonare umano di coltura A549 e linea cellulare monocitica umana THP-1 in terreni RPMI1640 integrati con 10% FBS e 1% penicillina-streptomicina a 37 °C in camera umidificata con 5% di CO2.
- Coltura di cellule di fibroblasti polmonari umani MRC-5 in terreno DMEM integrato con soluzione di FBS al 10% e soluzione di penicillina-streptomicina all'1% a 37 °C in camera umidificata con CO2 al 5%.
2. Preparazione di sferoidi tumorali multicellulari utilizzando la linea cellulare di adenocarcinoma polmonare A549, fibroblasti MRC5 e monociti THP-1
NOTA: Gli sferoidi 3D tumorigenici e non tumorigenici multicellulari sono stati preparati utilizzando il metodo della goccia sospesa in un piatto di coltura cellulare da 90 mm. Di seguito viene fornita una descrizione dettagliata dello sviluppo di questi sferoidi. Tutti i reagenti di coltura cellulare come terreno completo, PBS e soluzione di tripsina-EDTA allo 0,25% devono essere preriscaldati a 37 °C prima dell'uso, salvo diversa indicazione.
- Preparazione della sospensione cellulare
- Coltivare cellule aderenti A549 e MRC5 (5 x 106 cellule ciascuna) in terreno di coltura completo DMEM (terreno di aquila modificato di Dulbecco [DMEM] + 10% siero bovino fetale [FBS] + 1% penicillina-streptomicina) in palloni T25 a 37 °C in camera umidificata con 5% di CO2.
- Per le cellule THP-1, far crescere la sospensione cellulare (5 x 106 cellule) in un mezzo di crescita completo (RPMI1640 + 10% FBS + 1% penicillina-streptomicina) in flaconi T25. Per una migliore crescita, posizionare i palloni T25 a 37 °C in una camera umidificata con il 5% di CO2 in posizione eretta.
- Dopo 3 giorni, all'80%-85% di confluenza, lavare le cellule A549 e MRC5 con PBS (soluzione salina tampone fosfato) privo di calcio e magnesio aggiungendo 1 mL di PBS (25-30 °C) in ciascun matraccio per 1 minuto e aspirandolo.
- Raccogliere le cellule A549 e MRC5 dal matraccio incubandole con 500 μL di soluzione di tripsina-EDTA allo 0,25% per 5 minuti a 37 °C in un incubatore umidificato con CO2 al 5%. Subito dopo, aggiungere 4 ml di terreno di coltura completo per inattivare la tripsina.
- Raccogliere la sospensione cellulare dal matraccio T25 in un tubo da 15 ml e pellettarla a 125 x g per 5 minuti. Rimuovere il surnatante e risospendere le cellule in 5 ml di terreno di crescita completo.
- Istituzione di sferoidi tumorali multicellulari
NOTA: Tutte le fasi della formazione dello sferoide tumorale devono essere eseguite all'interno dell'armadio di biosicurezza al fine di mantenere condizioni sterili. Il volume massimo della sospensione cellulare è stato standardizzato come una goccia di 25 μL per preparare una goccia in modo tale che non cada mentre si capovolgono i coperchi.- Contare i numeri di celle A549, MRC5 e THP-1 utilizzando un contatore di celle. Per ogni goccia sferoide (25 μL) mantenere i seguenti numeri di cellule: 5.000 cellule A549, 4.000 cellule MRC5 e 1.000 cellule THP-1, seguendo il protocollo descritto da Arora et al.7. Calcolare i numeri di cella di conseguenza per un volume di 1 ml.
NOTA: Gli sferoidi sono stati inizialmente preparati con tre diversi conteggi cellulari per sferoide (cioè 5000, 8000 e 10.000). Inoltre, sono stati controllati diversi rapporti cellulari di cellule tumorali / fibroblasti / monociti (1: 1: 1, 2: 2: 1, 4: 2: 1, 5: 2: 1 e 5: 4: 1). Infine, è stata osservata una formazione sferoide multicellulare 3D di successo con un rapporto di 5: 4: 1 ed è stata utilizzata per lo studio. La concentrazione di fibroblasti è stata aumentata in base alla sua proporzione nel microambiente tumorale, che ha ulteriormente migliorato la rigidità degli sferoidi tumorali. La procedura dettagliata è stata riportata in una recente pubblicazione di Arora et al.7. - Preparare la sospensione cellulare in un rapporto di 5 (A549, 2 x 10 5 cellule/ml) a 4 (MRC5, 1,6 x 10 5 cellule/ml) a 1 (THP-1,5 x 104 celle/ml) e portare il volume a 1 mL con DMEM completo.
- Introdurre una goccia di 25 μL di miscela di sospensione cellulare sul coperchio di un piatto di coltura da 90 mm (circa 50 gocce/piatto da 90 mm). Riempire il fondo del piatto di coltura da 90 mm con 10 ml di acqua sterile.
- Capovolgere con attenzione il coperchio sopra la camera di idratazione piena d'acqua e posizionare il piatto in un incubatore di coltura cellulare per 3 giorni.
- Monitorare gli sferoidi al microscopio con un ingrandimento 10x il giorno 4. Per acquisire immagini, accendere l'interruttore di alimentazione, posizionare con attenzione il piatto da 60 mm sul palco e selezionare l'ingrandimento (10x). Regolare le lenti ed esaminare le cellule per analizzare l'aggregazione e la proliferazione cellulare. Premere i pulsanti Freeze e Save forniti sul microscopio per acquisire l'immagine.
- Cambiare il terreno di coltura il giorno 4 aspirando con cura 20 μL di terreno da ciascuna goccia e sostituendolo con un nuovo substrato di crescita completo.
- Contare i numeri di celle A549, MRC5 e THP-1 utilizzando un contatore di celle. Per ogni goccia sferoide (25 μL) mantenere i seguenti numeri di cellule: 5.000 cellule A549, 4.000 cellule MRC5 e 1.000 cellule THP-1, seguendo il protocollo descritto da Arora et al.7. Calcolare i numeri di cella di conseguenza per un volume di 1 ml.
3. Analisi dei morti vivi degli sferoidi tumorali
- Nei giorni 7 e 10, capovolgere con attenzione il piatto da 90 mm nell'armadio di biosicurezza e utilizzare una pipetta da 200 μL per raccogliere gli sferoidi da ciascuna goccia. Raccogliere cinque sferoidi ciascuno in un tubo da 1,5 ml.
- Aggiungere 500 μL di 1x PBS al tubo da 1,5 mL contenente sferoidi e centrifugare a 125 x g per 5 minuti. Scartare attentamente il surnatante e risospendere gli sferoidi in 200 μL di 1x PBS. Non pipettare rigorosamente per evitare la disintegrazione sferoide.
- Pipetta gli sferoidi utilizzando una pipetta da 200 μL su un piatto da 60 mm per la colorazione con calcein-AM e ioduro di propidio.
- Mettere sugli sferoidi 5 μL di soluzione di calceina-AM da 1 μM e 5 μL di soluzione di ioduro di propidio 2 mg/ml. Incubare per 10 min. Dopo il completamento del periodo di incubazione, lavare delicatamente gli sferoidi con 1x PBS due volte.
- Posizionare il piatto da 60 mm contenente sferoidi sotto il microscopio invertito fluorescente, osservare e acquisire immagini con un ingrandimento 10x selezionando l'opzione di fluorescenza nel software del microscopio e selezionando il FITC (canale di fluorescenza verde; eccitazione 490 nm, emissione 515 nm) per calceina e canale rosso Texas (TXR; eccitazione 535 nm, emissione 617 nm).
- Per acquisire l'immagine, attivare Ctr Adv, che è l'interruttore 1, seguito dall'accensione della CPU. Attendere l'avvio del sistema software. Una volta avviato, posizionare con attenzione il piatto da 60 mm sul palco e selezionare l'ingrandimento (10x) e i canali fluorescenti (FITC, TXR). Regolare le lenti ed eseguire la scansione dell'immagine.
- Per visualizzare l'immagine nel sistema, selezionare l'opzione Live e visualizzare l'immagine. Regolare l'intensità della fluorescenza per un'ottimizzazione appropriata. Salvare l'immagine facendo clic sul pulsante Salva .
NOTA: In questa fase, gli sferoidi saranno visibili ad occhio nudo. Sotto il microscopio ottico, gli sferoidi appariranno come sfere rotonde e rigide con un ingrandimento 10x. È possibile raccogliere più sferoidi contemporaneamente utilizzando una pipetta da 200 μL.
4. Disintegrazione e sospensione cellulare di sferoidi tumorali
- Raccogliere 200 sferoidi tumorali ciascuno nei giorni 7 e 10, utilizzando una pipetta da 1 mL in una provetta da 15 ml.
NOTA: Prima della raccolta, controllare attentamente la forma e la forma di un singolo sferoide dopo averlo trasferito su un vetrino microscopico o su un piatto da 30 mm e osservare al microscopio. - Pellettare gli sferoidi mediante centrifugazione a 125 x g per 5 min. Aspirare il surnatante con attenzione senza disturbare gli sferoidi tumorali.
- Lavare accuratamente gli sferoidi con 200 μL di PBS, centrifugare a 125 x g per 5 minuti e scartare con cautela il surnatante.
- Per la disintegrazione dello sferoide, aggiungere 400 μL di soluzione di tripsina-EDTA allo 0,25% e mantenere a 37 °C per 10 minuti. Eseguire un pipettaggio vigoroso per la completa disintegrazione degli sferoidi.
- Neutralizzare la tripsina aggiungendo 1 mL di terreno di crescita DMEM completo. Centrifugare a 125 x g per 5 minuti ed eliminare accuratamente il surnatante .
- Risospendere il pellet in 1 mL di mezzo DMEM completo e contare il numero totale di cellule.
5. Isolamento dei fibroblasti associati al cancro (CAF) attraverso microsfere
- Per l'isolamento dei CAF dagli sferoidi tumorali, risospendere 1 x 10 7 cellule in 80 μL di tampone MACS (cold magnetic activated cell sorting) (PBS a pH7,2 contenente lo 0,5% di sieroalbumina bovina [BSA] e 2 mM di acido etilendiamminatraacetico [EDTA]).
- Incubare la sospensione cellulare (contenente 1 x 107 cellule) con 20 μL di microsfere anti-fibroblasti. Mescolare bene picchiettando delicatamente il tubo e incubare per 30 minuti a temperatura ambiente.
- Lavare le celle con 1 mL di tampone MACS freddo, centrifugare a 125 x g per 5 minuti e aspirare il surnatante. Risospendere le celle in 500 μL di buffer MACS.
- Per la separazione delle cellule basata su sfere magnetiche, preparare la colonna MACS risciacquandola con 3 ml di tampone MACS.
- Posizionare la sospensione cellulare nella colonna seguita dalla raccolta del flusso contenente la popolazione cellulare non etichettata.
- Lavare la colonna tre volte con 3 ml di tampone MACS. Rimuovere la colonna dal separatore e posizionarla sul tubo di raccolta.
- Raccogliere le cellule marcate con microsfere anti-fibroblasti aggiungendo 5 ml di tampone MACS e spingendo saldamente lo stantuffo nella colonna.
- Centrifugare le celle marcate a 125 x g per 5 minuti. Procedere con i fibroblasti isolati per applicazioni a valle.
6. Analisi basata sulla citometria a flusso dell'espressione di ACTA2 in CAF isolati
- Contare il numero di CAF isolati e utilizzare circa 6 x 104 celle. Lavare le cellule una volta con PBS, centrifugare a 125 x g per 5 minuti e aspirare il surnatante.
- Aggiungere 100 μL di tampone di permeabilizzazione cellulare (PBS + 0,5% BSA + 0,3% v/v Triton X-100) e incubare le cellule a 4 °C per 30 minuti.
- Vortice le cellule a intermittenza per mantenere una singola sospensione cellulare. Centrifugare e risospendere le cellule in 100 μL di tampone di permeabilizzazione cellulare.
- Aggiungere 2 μL di anticorpo anti-α SMA anti-umano coniugato APC e incubare a 4 °C per 45 minuti. Dopo l'incubazione, aggiungere 1 mL di tampone di permeabilizzazione e centrifugare a 125 x g per 5 minuti per rimuovere l'anticorpo in eccesso.
- Risospendere il pellet in 400 μL di tampone di permeabilizzazione per analisi citometrica a flusso. Acquisire un totale di 10.000 eventi di ciascun campione in un citometro a flusso. Selezionare le cellule ACTA2 positive dopo aver distinto le popolazioni di cellule in base alla loro dispersione diretta e laterale, selezionando la popolazione di singoletto seguita selezionando le cellule ACTA2 positive che appaiono come un singolo picco sull'istogramma a singolo parametro.
7. Colorazione JC-1 per determinare il potenziale della membrana mitocondriale
- Contare il numero di CAF isolati e utilizzare circa 6 x 104 cellule per la colorazione con 5,5,6,6'-tetracloro-1,1',3,3' tetraetilbenzimi-dazoilcarbocianina ioduro (JC-1) utilizzando la citometria a flusso.
- Lavare accuratamente le cellule con PBS, centrifugare a 125 x g per 5 minuti, aspirare il surnatante e aggiungere 100 μL di tampone colorante cellulare.
- Aggiungere il colorante JC-1 ad una concentrazione di lavoro di 2 μM e incubare a temperatura ambiente per 30 minuti. Al termine dell'incubazione, lavare le cellule con PBS, centrifugare a 125 x g per 5 minuti e risospendere in un volume finale di 400 μL.
- Acquisire un totale di 20.000 eventi di ciascun campione in un citometro a flusso. Quantificare il potenziale della membrana mitocondriale misurando gli aggregati JC-1 spostati verso il rosso sul canale FL-2 e i monomeri spostati verso il verde sul canale FL-1.
8. Colorazione DCFDA per stimare i livelli di specie reattive dell'ossigeno cellulare (ROS)
- Utilizzare gli sferoidi interi (50 numeri) e i CAF isolati (6 x 104 cellule) dagli sferoidi per la colorazione con diacetato di 2',7'-diclorodiidrofluoresceina (DCFDA).
- Lavare accuratamente le cellule con PBS, centrifugare a 125 x g per 5 minuti, aspirare il surnatante e aggiungere 100 μL di tampone colorante cellulare.
- Aggiungere il colorante DCFDA ad una concentrazione di lavoro di 1 μM e incubare a temperatura ambiente per 30 minuti. Lavare le celle due volte con PBS, centrifugare a 125 x g per 5 minuti, quindi risospendere in un volume finale di 400 μL.
- Acquisire un totale di 20.000 eventi di ciascun campione in un citometro a flusso. Valutare i livelli di ROS misurando la fluorescenza al giorno 7 e al giorno 10 per gli sferoidi e i CAF isolati.
9. Analisi RT-qPCR di marcatori CAF e geni glicolitici
- Estrarre l'RNA dai CAF selezionati utilizzando un kit di lisi a singola cellula seguendo il protocollo del produttore. Preparare cDNA da 100 ng di RNA utilizzando un kit di sintesi del cDNA seguendo le linee guida del produttore.
- Eseguire RT-qPCR per analizzare i relativi marcatori CAF (ACTA2 8 e COL1A29) e l'espressione del gene glicolitico (GLUT1 10 e MCT4 11). Eseguire l'analisi della curva di fusione dopo l'estensione finale per garantire la specificità dei prodotti. Normalizzare i dati usando l'espressione di GAPDH come gene di riferimento. Le sequenze di primer utilizzate per RT-qPCR sono elencate nella Tabella supplementare 1.
10. Estrazione e quantificazione della proteina cellulare dai CAF
NOTA: Eseguire tutte le fasi di estrazione delle proteine sul ghiaccio per evitare la degradazione delle proteine.
- Risospendere circa 4 x 106 cellule CAF in 100 μL di tampone di lisi RIPA ghiacciato (contenente 30 mM HEPES, 150 mM NaCl, 1% NP-40, 0,1% SDS e 0,5% desossicolato di sodio con 5 mM di cocktail di inibitori della proteasi e della fosfatasi Halt e 5 mM di EDTA a pH 7,4).
- Vortice rigorosamente per una corretta lisi cellulare. Sonicare la sospensione cellulare alla frequenza di 20 Hz, al 20% di ampiezza per 15 s e all'impulso 3x.
- Dopo la sonicazione, centrifugare l'estratto proteico a 13.000 x g per 15 minuti a 4 °C. Trasferire il surnatante in una provetta pre-raffreddata da 1,5 ml. Eseguire il test della proteina BCA per quantificare la proteina.
11. Analisi spettrofotometrica delle attività enzimatiche nei CAF
NOTA: Le seguenti attività enzimatiche sono analizzate nei CAF derivati da sferoidi tumorali.
- Misurazione dell'attività della succinato deidrogenasi
- Per valutare l'attività della succinato deidrogenasi (SDH) nei CAF, preparare tampone SHE con 250 mM di saccarosio, 10 mM di acido 4-(2-idrossietil)-1-piperazineetanosolfonico (HEPES) e 1 mM di glicole etilenico-bis(β-amminoetiletere)-N,N,N′,N′-tetraacetico (EGTA) e regolare il pH a 7,3. Aggiungere 0,4 mM di fenazina metosolfato (PMS), 0,2 mM di 2,6-dicloroindofenolo (DCPIP), 50 mM MgCl2, 0,02% Triton X-100 e 1 mM di cianuro nel tampone SHE e mantenere a 37 °C.
- Estrarre la proteina cellulare dai CAF come descritto al punto 10 (2 x 106 cellule). In ciascun pozzetto di una piastra a 96 pozzetti, incubare 100 μL di 0,1 mg di proteine cellulari con 100 μL di miscela di reazione SHE a 37 °C per 15 minuti.
- Per iniziare l'attività enzimatica, aggiungere 10 mM di succinato12. Calcolare l'attività enzimatica in nM/min/mL misurando le variazioni di assorbanza a 600 nm.
- Il fattore di conversione per DCIP è 0,0215 A / nM in base al suo coefficiente di estinzione molare12. Calcolare l'attività relativa di SDH utilizzando la formula menzionata:
Attività relativa (nM/min/mL/enzima) =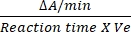 X X
X X  V
V
Dove ΔA/min = velocità di reazione enzimatica (iniziale A- A finale)/(tempofinale- A/mininiziale), Ve = volume del campione e V = volume della reazione.
- Stima dell'attività della citocromo c ossidasi
- Per valutare l'attività della citocromo c ossidasi (COX) nei CAF, aggiungere 0,2 mg/ml di proteina cellulare in una soluzione tampone di reazione KME contenente tampone KME (125 mM KCl, 20 mM di acido 3-(N-morfolino)propano solfonico [MOPS] e 1 mM EGTA, pH 7,4), 0,02% Triton X-100 e 5 mM di ascorbato di sodio.
- In una provetta separata, mescolare 50 μM di citocromo c del cuore di cavallo con 5 mM di ascorbato di sodio e incubare a 37 °C per 5 min13. Dopo l'incubazione, aggiungere 20 μL di citocromo c ridotto a 500 μL di soluzione tampone di reazione KME contenente proteine cellulari.
- Calcolare l'attività enzimatica in unità/μL misurando le variazioni di assorbanza a 550 nm. L'assorbimento del citocromo c a 550 nm cambia con il suo stato di ossidazione. La differenza nei coefficienti di estinzione (mM) tra citocromo c ridotto e ossidato è 21,84 a 550 nm14. Calcola la quantità di attività enzimatica nel lisato cellulare come:
Unità/μL =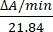
Dove ΔA/min = A/mincampione-A/minbianco e 21,84 = ΔӑmM tra citocromo c ossidato e citocromo c ridotto a 550 nm
- Valutazione dell'attività della lattato deidrogenasi
- Eseguire un test della lattato deidrogenasi (LDH) utilizzando un kit di analisi delle cellule della lattato deidrogenasi seguendo le istruzioni del produttore.
- Aggiungere il reagente per il test LDH (10 μL) a 0,5 mg/ml di proteine cellulari e incubare per 5 minuti a 37 °C. Al termine dell'incubazione, misurare l'assorbanza a 45 nm ogni 1 minuto per 8 minuti.
- Preparare gli standard NADH per il rilevamento colorimetrico utilizzando 0 (bianco), 2,5, 5, 7,5, 10 e 12,5 nM/pozzetto di NADH seguito dall'aggiunta del reagente di prova LDH a un volume finale di 50 μL.
- Tracciare le differenze di assorbanza tra Tiniziale e Tfinale alla curva standard NADH per determinare la quantità di generazione di NADH. Valutare l'attività di LDH utilizzando la formula menzionata:
Attività LDH =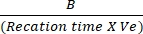 fattore di diluizione del campione X
fattore di diluizione del campione X
Dove B = quantità (nmole) di NADH generata tra Tiniziale e T finale, tempo di reazione = Tfinale - Tiniziale (min) e Ve = volume del campione (ml) aggiunto al pozzo.
Risultati
La Figura 1 mostra lo sviluppo di sferoidi tumorali multicellulari utilizzando tre diverse popolazioni cellulari - A549 (adenocarcinoma polmonare), MRC-5 (fibroblasti) e THP-1 (monociti) - con il metodo della goccia sospesa come osservato il giorno 7 e il giorno 10 al microscopio. Il giorno 7, gli sferoidi erano compatti e rigidi con un diametro di 260 ± 5,3 μm, e il giorno 10, gli sferoidi avevano un diametro di 480 ± 7,5 μm (Figura 1A pannello superiore,
Discussione
Il presente studio introduce lo sviluppo di sferoidi tumorali multicellulari comprendenti cellule tumorali, popolazione di cellule stromali (cioè fibroblasti) e popolazione di cellule immunitarie (cioè monociti) utilizzando un metodo di goccia sospesa modificata. I fibroblasti e i monociti/macrofagi sono tra le popolazioni più significative che costituiscono il microambiente tumorale (TME) e la loro presenza è spesso correlata a una prognosi infausta16. Quando presenti nella TME, i fibroblasti...
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto dal SERB-Women Excellence Award Project, India (SB/WEA-02/2017) e dal SERB-Early Career Research Award Project, India (ECR/2017/000892) a DP. Gli autori, LA e SR riconoscono IIT Ropar e MHRD per le loro borse di ricerca. MK riconosce l'ICMR per la sua borsa di ricerca.
Materiali
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| APC anti-human α-SMA | R&D systems | Cat# IC1420A | |
| Anti-fibroblast microbeads | Miltenyi Biotec | Cat# 130-050-601 | |
| Cell lines | |||
| A549 lung adenocarcinoma cells | NCCS Pune | - | |
| MRC-5 fetal lung fibroblasts | ATCC | CCL-171 | |
| THP-1 Human monocytes | NCCS Pune | - | |
| Chemicals | |||
| BSA | Himedia | Cat# 9048-46-8 | |
| 2,6-dichloroindophenol (DCPIP) | SRL | Cat# 55287 | |
| Calcein-AM | Thermo Fisher Scientific | Cat# C3099 | |
| DAPI | Thermo Fisher Scientific | Cat# D1306 | |
| DCFDA | Sigma | Cat# D6883 | |
| DMEM | Gibco | Cat# 11995073 | |
| DPBS | Gibco | Cat# 14190-144 | |
| EDTA | Thermo fisher scientific | Cat# 17892 | |
| EGTA | SRL | Cat# 62858 | |
| EZcoun Lactate Dehydrogenase Cell Assay Kit | HiMedia | Cat# CCK036 | |
| FBS | Gibco | Cat# 10082147 | |
| Halt Protease and Phosphatase Inhibitor Cocktail (100X) | Thermo Fisher Scientific | Cat# 87786 | |
| HEPES | Thermo Fisher Scientific | Cat# 15630080 | |
| Horse heart Cytochrome c | SRL | Cat# 81551 | |
| Image-iT Red hypoxia reagent | Thermo Fisher Scientific | Cat# H10498 | |
| JC-1 Dye | Thermo Fisher Scientific | Cat# T3168 | |
| KCl | Merck | Cat# P9541 | |
| MgCl2 | Merck | Cat# M8266 | |
| MOPS | Thermo Fisher Scientific | Cat# 69824 | |
| Nacl | Sigma-Aldrich | Cat# S9888 | |
| NADH MB Grade | SRL | Cat# 54941 | |
| NP-40 | Thermo Fisher Scientific | Cat# 85124 | |
| Penicillin/Streptomycin | Gibco | Cat# 15140122 | |
| Phenazine methosulfate (PMS) | SRL | Cat# 55782 | |
| Propidium iodide | Thermo fisher scientific | Cat# P1304MP | |
| RPMI 1640 | Gibco | Cat# 11875093 | |
| Single Cell Lysis Kit | Thermo Fisher Scientific | Cat# 4458235 | |
| Sodium ascorbate | Merck | Cat# A7631 | |
| Sodium cyanide | Sigma | Cat# 205222 | |
| Sodium Deoxycholate | Thermo Fisher Scientific | Cat# 89904 | |
| Sodium dodecyl sulphate | Sigma-Aldrich | Cat# L3771 | |
| Sodium succinate hexahydrate | SRL | Cat# 36313 | |
| Sucrose | Sigma | Cat# S0389 | |
| SuperScript VILO cDNA synthesis kit | Thermo Fisher Scientific | Cat# 11754-050 | |
| Triton X-100 | Sigma | Cat# T8787 | |
| Trypsin 0.25% EDTA | Gibco | Cat# 25200072 | |
| Universal SYBR Green Supermix | BIO-RAD | Cat# 172-5124 | |
| Plasticware | |||
| MACS LS Columns | Miltenyi Biotec | Cat# 130-042-401 | |
| Equipment | |||
| Countess II FL Automated Cell Counter | Thermo Fisher Scientific | Cat# AMQAF1000 | |
| EVOS XL core imaging system | Thermo Fisher Scientific | Serial Number F0518-1727-0191 | |
| LAS X software | Leica Microsystems | ||
| Leica fluorescent inverted microscope | s | DMi8 automated S/N 424150) | |
| Midi MACS separator | Miltenyi Biotec | Cat# 130-042-302 |
Riferimenti
- Kim, N., et al. Single-cell RNA sequencing demonstrates the molecular and cellular reprogramming of metastatic lung adenocarcinoma. Nature Communications. 11 (1), 1-5 (2020).
- Davidson, S., et al. Single-cell RNA sequencing reveals a dynamic stromal niche that supports tumor growth. Cell Reports. 31 (7), 107628 (2020).
- Zhang, Y., et al. Single-cell analyses of renal cell cancers reveal insights into tumor microenvironment, cell of origin, and therapy response. Proceedings of the National Academy of Sciences. 118 (24), (2021).
- Bray, L. J., Hutmacher, D. W., Bock, N. Addressing patient specificity in the engineering of tumor models. Frontiers in Bioengineering and Biotechnology. 7, 217 (2019).
- Timmins, N. E., Nielsen, L. K. Generation of multicellular tumor spheroids by the hanging-drop method. Tissue Engineering. , 141-151 (2007).
- Dituri, F., et al. Complex tumor spheroid formation and one-step cancer-associated fibroblasts purification from hepatocellular carcinoma tissue promoted by inorganic surface topography. Nanomaterials. 11 (12), 3233 (2021).
- Arora, L., et al. Development of a multicellular 3D tumor model to study cellular heterogeneity and plasticity in NSCLC tumor microenvironment. Frontiers in Oncology. 12, 881207 (2022).
- Nurmik, M., Ullmann, P., Rodriguez, F., Haan, S., Letellier, E. In search of definitions: Cancer-associated fibroblasts and their markers. International Journal of Cancer. 146 (4), 895-905 (2020).
- Zhang, Y., et al. HIF-1α is necessary for activation and tumour-promotion effect of cancer-associated fibroblasts in lung cancer. Journal of Cellular and Molecular Medicine. 25 (12), 5457-5469 (2021).
- Bu, L., et al. Biological heterogeneity and versatility of cancer-associated fibroblasts in the tumor microenvironment. Oncogene. 38 (25), 4887-4901 (2019).
- Whitaker-Menezes, D., et al. Evidence for a stromal-epithelial "lactate shuttle" in human tumors: MCT4 is a marker of oxidative stress in cancer-associated fibroblasts. Cell cycle. 10 (11), 1772-1783 (2011).
- Mandujano-Tinoco, E. A., Gallardo-Pérez, J. C., Marín-Hernández, A., Moreno-Sánchez, R., Rodríguez-Enríquez, S. Anti-mitochondrial therapy in human breast cancer multi-cellular spheroids. Biochimica et Biophysica Acta (BBA)-Molecular Cell Research. 1833 (3), 541-551 (2013).
- Bregman, A. A. . Laboratory Investigations in Cell and Molecular Biology. , (2002).
- Berry, E. A., Trumpower, B. L. Simultaneous determination of hemes a, b, and c from pyridine hemochrome spectra. Analytical Biochemistry. 161 (1), 1-15 (1987).
- Avagliano, A., et al. Metabolic reprogramming of cancer associated fibroblasts: the slavery of stromal fibroblasts. BioMed Research International. , (2018).
- Lorusso, G., Rüegg, C. The tumor microenvironment and its contribution to tumor evolution toward metastasis. Histochemistry and Cell Biology. 130 (6), 1091-1103 (2008).
- Liu, T., Zhou, L., Li, D., Andl, T., Zhang, Y. Cancer-associated fibroblasts build and secure the tumor microenvironment. Frontiers in Cell and Developmental Biology. 7, 60 (2019).
- Sebastian, A., et al. Single-cell transcriptomic analysis of tumor-derived fibroblasts and normal tissue-resident fibroblasts reveals fibroblast heterogeneity in breast cancer. Cancers. 12 (5), 1307 (2020).
- Elyada, E., et al. Cross-species single-cell analysis of pancreatic ductal adenocarcinoma reveals antigen-presenting cancer-associated fibroblasts. Cancer Discovery. 9 (8), 1102-1123 (2019).
- Ganguly, D., et al. Cancer-associated fibroblasts: Versatile players in the tumor microenvironment. Cancers. 12 (9), 2652 (2020).
- Harryvan, T. J., Verdegaal, E. M., Hardwick, J. C., Hawinkels, L. J., vander Burg, S. H. Targeting of the cancer-associated fibroblast-T-cell axis in solid malignancies. Journal of Clinical Medicine. 8 (11), 1989 (2019).
- Santi, A., Kugeratski, F. G., Zanivan, S. Cancer associated fibroblasts: the architects of stroma remodeling. Proteomics. 18 (5-6), 1700167 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon