Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Évaluation de la santé mitochondriale dans les fibroblastes associés au cancer isolés à partir de sphéroïdes de tumeurs pulmonaires multicellulaires 3D
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Des sphéroïdes tumoraux 3D multicellulaires ont été préparés avec des cellules d’adénocarcinome pulmonaire, des fibroblastes et des monocytes, suivis de l’isolement de fibroblastes associés au cancer (CAF) à partir de ces sphéroïdes. Les CAF isolés ont été comparés aux fibroblastes normaux pour évaluer la santé mitochondriale en étudiant le potentiel transmembranaire mitochondrial, les espèces réactives de l’oxygène et les activités enzymatiques.
Résumé
Les fibroblastes associés au cancer (CAF) sont parmi les cellules stromales les plus abondantes présentes dans le microenvironnement tumoral, facilitant la croissance et la progression tumorales. La complexité du microenvironnement tumoral, y compris le sécrétome tumoral, l’inflammation de bas grade, l’hypoxie et le déséquilibre redox, favorise l’interaction hétérotypique et permet la transformation des fibroblastes résidents inactifs pour devenir des CAF actifs. Les CAF se distinguent métaboliquement des fibroblastes normaux (NF) car ils sont plus glycolytiquement actifs, produisent des niveaux plus élevés d’espèces réactives de l’oxygène (ROS) et surexpriment l’exportateur de lactate MCT-4, conduisant à l’ouverture du pore de transition de perméabilité mitochondriale (MPTP). Ici, une méthode a été décrite pour analyser la santé mitochondriale des CAF activés isolés des sphéroïdes tumoraux 3D multicellulaires comprenant des cellules d’adénocarcinome pulmonaire humain (A549), des monocytes humains (THP-1) et des cellules de fibroblastes pulmonaires humains (MRC5). Les sphéroïdes tumoraux ont été désintégrés à différents intervalles de temps et grâce au tri cellulaire activé magnétiquement, les CAF ont été isolés. Le potentiel membranaire mitochondrial des CAF a été évalué à l’aide du colorant JC-1, de la production de ROS par coloration au diacétate de 2',7'-dichlorodihydrofluorescéine (DCFDA) et de l’activité enzymatique dans les CAF isolés. L’analyse de la santé mitochondriale des CAF isolés permet de mieux comprendre l’effet Warburg inverse et peut également être appliquée pour étudier les conséquences des changements mitochondriaux des CAF, telles que les flux métaboliques et les mécanismes de régulation correspondants sur l’hétérogénéité du cancer du poumon. Ainsi, la présente étude préconise une compréhension des interactions tumeur-stroma sur la santé mitochondriale. Il fournirait une plate-forme pour vérifier l’efficacité des candidats médicaments spécifiques aux mitochondries contre les CAF en tant que thérapeutiques potentielles dans le microenvironnement tumoral, empêchant ainsi l’implication des CAF dans la progression du cancer du poumon.
Introduction
Les tumeurs solides sont composées de populations cellulaires hétérogènes qui sont guidées par le microenvironnement tumoral (TME), cependant, l’origine de la plupart des cellules n’a pas encore été découverte. Principalement les cellules stromales et immunitaires (fibroblastes, cellules endothéliales, monocytes, macrophages, cellules dendritiques, cellules B, cellules T et leurs sous-ensembles) reflètent l’hétérogénéité tumorale dans les cancers du poumon, du sein, du rein et d’autres solides 1,2,3. Comprendre l’origine de chaque sous-type et leur potentiel de différenciation des transgenres est absolument nécessaire pour développer des thérapies innovantes contre ces cancers. L’analyse de cette population cellulaire diversifiée dans les biopsies humaines présente plusieurs défis en raison du type de tumeur, du site, du stade, de la limitation de la quantité d’échantillon et des variabilités spécifiques au patient4. Ainsi, un modèle expérimental est nécessaire, qui est non seulement fiable, mais peut également simuler l’état tumoral in vivo, se révélant idéal pour étudier la diaphonie tumeur-stroma et son implication dans la physiopathologie de la maladie.
Les cultures tridimensionnelles (3D) de tumeurs sphéroïdes multicellulaires (MCTS) sont un système modèle in vitro avantageux de tumeurs en raison de leur ressemblance avec leurs homologues naturels. Les SCTM peuvent mieux reproduire certains aspects des tumeurs solides que les modèles de culture cellulaire 2D, y compris leur architecture spatiale, leurs réponses physiologiques, la libération de médiateurs solubles, les modèles d’expression génique et les mécanismes de résistance aux médicaments. De plus, l’un des principaux avantages des SCTM est qu’ils peuvent être utilisés pour étudier l’hétérogénéité tumorale et le microenvironnement tumoral (TME). La méthode de la goutte suspendue est l’outil le plus couramment utilisé pour développer et analyser les SCTM5. Dans cette méthode, les différentes cellules avec des milieux sont suspendues sous forme de gouttelettes, ce qui permet sa croissance de manière agrégée 3D cohérente et est facile d’accès pour examen. La technique est simple; Il ne nécessite pas beaucoup de cellules et élimine le besoin d’un substrat spécialisé comme l’agarose pour le développement sphéroïde6. Un avantage supplémentaire de cette méthode réside dans la reproductibilité de sa technique. En outre, cette méthode a également été utilisée pour co-cultiver des populations de cellules mixtes, telles que des cellules endothéliales et des cellules tumorales, afin de simuler l’angiogenèse tumorale précoce7.
Dans cette étude, des sphéroïdes multicellulaires 3D de tumeurs pulmonaires ont été préparés avec des cellules d’adénocarcinome pulmonaire, des fibroblastes et des monocytes en utilisant la méthode de la goutte suspendue qui imite le microenvironnement de la tumeur pulmonaire. Ensuite, la population de fibroblastes associés au cancer (CAF) a été isolée pour étudier la santé mitochondriale. L’idée principale derrière le développement de ces sphéroïdes est d’isoler les CAF, car la diaphonie entre les cellules dans les sphéroïdes pourrait transformer les fibroblastes en un état CAF activé de type myo-fibroblaste. Deuxièmement, cette étude peut également décrire comment la production aberrante de ROS et le dysfonctionnement mitochondrial conduisent les fibroblastes normaux vers le phénotype CAF plus agressif. Il a été constaté que les fibroblastes assemblés dans les sphéroïdes tumoraux ont acquis des caractéristiques myofibroblastiques avec une activité ROS accrue et l’induction de l’expression génique métabolique. Ce protocole souligne l’importance du microenvironnement tumoral dans l’activation de la CAF et pourrait être un excellent modèle pour la génération in vitro et l’étude des caractéristiques phénotypiques de la CAF.
Protocole
1. Culture cellulaire
- Culture de la lignée cellulaire A549 de l’adénocarcinome pulmonaire humain et de la lignée cellulaire monocytaire humaine THP-1 dans un milieu RPMI1640 supplémentée avec 10% FBS et 1% pénicilline-streptomycine à 37 °C dans une chambre humidifiée avec 5% de CO2.
- Culture de cellules de fibroblastes pulmonaires humains MRC-5 en milieu DMEM supplémentée avec 10% de FBS et 1% de solution de pénicilline-streptomycine à 37 °C dans une chambre humidifiée avec 5% de CO2.
2. Préparation de sphéroïdes tumoraux multicellulaires à l’aide d’une lignée cellulaire d’adénocarcinome pulmonaire A549, de fibroblastes MRC5 et de monocytes THP-1
REMARQUE: Les sphéroïdes 3D tumorigènes multicellulaires et non tumorigènes ont été préparés en utilisant la méthode de la goutte suspendue dans une boîte de culture cellulaire de 90 mm. Une description détaillée du développement de ces sphéroïdes est donnée ci-dessous. Tous les réactifs de culture cellulaire tels que le milieu complet, le PBS et la solution trypsine-EDTA à 0,25 % doivent être préchauffés à 37 °C avant utilisation, sauf indication contraire.
- Préparation de la suspension cellulaire
- Cultiver des cellules adhérentes A549 et MRC5 (5 x 106 cellules chacune) dans un milieu de croissance complet DMEM (milieu aigle modifié de Dulbecco [DMEM] + 10 % de sérum fœtal bovin [FBS] + 1 % de pénicilline-streptomycine) dans des flacons T25 à 37 °C dans une chambre humidifiée contenant 5 % de CO2.
- Pour les cellules THP-1, cultiver la suspension cellulaire (5 x 106 cellules) dans un milieu de croissance complet (RPMI1640 + 10% FBS + 1% pénicilline-streptomycine) dans des flacons T25. Pour une meilleure croissance, placez les flacons T25 à 37 °C dans une chambre humidifiée contenant 5% de CO2 en position debout.
- Après 3 jours, à 80%-85% de confluence, laver les cellules A549 et MRC5 avec du PBS (phosphate tampon salin) sans calcium et magnésium en ajoutant 1 mL de PBS (25-30 °C) dans chaque fiole pendant 1 min et en l’aspirant.
- Récolter les cellules A549 et MRC5 du ballon en les incubant avec 500 μL de solution trypsine-EDTA à 0,25% pendant 5 min à 37 °C dans un incubateur humidifié à 5% de CO2. Immédiatement après, ajouter 4 mL de milieu de culture complet pour inactiver la trypsine.
- Recueillir la suspension cellulaire de la fiole T25 dans un tube de 15 ml et l’enduire en granulés à 125 x g pendant 5 min. Retirer le surnageant et remettre les cellules en suspension dans 5 mL de milieu de croissance complet.
- Etablissement de sphéroïdes tumoraux multicellulaires
REMARQUE: Toutes les étapes de la formation sphéroïde tumorale doivent être effectuées à l’intérieur de l’armoire de biosécurité afin de maintenir des conditions stériles. Le volume maximal de la suspension cellulaire a été normalisé comme une goutte de 25 μL pour préparer une gouttelette de sorte qu’elle ne tombe pas en retournant les couvercles.- Comptez les numéros de cellules A549, MRC5 et THP-1 à l’aide d’un compteur de cellules. Pour chaque gouttelette sphéroïde (25 μL), maintenez le nombre de cellules suivant : 5 000 cellules A549, 4 000 cellules MRC5 et 1 000 cellules THP-1, en suivant le protocole décrit par Arora et coll.7. Calculez les numéros de cellules en conséquence pour un volume de 1 mL.
REMARQUE: Les sphéroïdes ont été initialement préparés avec trois comptages de cellules différents par sphéroïde (c.-à-d. 5000, 8000 et 10 000). En outre, différents rapports cellulaires de cellules tumorales / fibroblastes / monocytes (1: 1: 1, 2: 2: 1, 4: 2: 1, 5: 2: 1 et 5: 4: 1) ont été vérifiés. Enfin, une formation de sphéroïdes multicellulaires 3D réussie a été observée avec un rapport de 5: 4: 1 et a été utilisée pour l’étude. La concentration de fibroblastes a été augmentée en fonction de sa proportion dans le microenvironnement tumoral, ce qui a encore renforcé la rigidité des sphéroïdes tumoraux. La procédure détaillée a été rapportée dans une publication récente d’Arora et al.7. - Préparer la suspension cellulaire dans un rapport de 5 (A549, 2 x 10 5 cellules/mL) à 4 (MRC5, 1,6 x 10 5 cellules/mL) à 1 (THP-1,5 x 104 cellules/mL) et compléter le volume à 1 mL avec DMEM complet.
- Déposer une goutte de 25 μL de mélange de suspension cellulaire sur le couvercle d’une boîte de culture de 90 mm (environ 50 gouttes/boîte de 90 mm). Remplir le fond de la capsule de culture de 90 mm avec 10 ml d’eau stérile.
- Retournez soigneusement le couvercle sur la chambre d’hydratation remplie d’eau et placez le plat dans un incubateur de culture cellulaire pendant 3 jours.
- Surveillez les sphéroïdes au microscope à un grossissement de 10x le jour 4. Pour acquérir des images, allumez l’interrupteur d’alimentation, placez soigneusement l’antenne parabolique de 60 mm sur la scène et sélectionnez le grossissement (10x). Ajustez les lentilles et examinez les cellules pour analyser l’agrégation et la prolifération cellulaires. Appuyez sur les boutons Figer et Enregistrer fournis sur le microscope pour capturer l’image.
- Changez le milieu de croissance au jour 4 en aspirant soigneusement 20 μL de milieu de chaque gouttelette et en le remplaçant par un milieu de croissance complet frais.
- Comptez les numéros de cellules A549, MRC5 et THP-1 à l’aide d’un compteur de cellules. Pour chaque gouttelette sphéroïde (25 μL), maintenez le nombre de cellules suivant : 5 000 cellules A549, 4 000 cellules MRC5 et 1 000 cellules THP-1, en suivant le protocole décrit par Arora et coll.7. Calculez les numéros de cellules en conséquence pour un volume de 1 mL.
3. Analyse des sphéroïdes tumoraux
- Les jours 7 et 10, retourner soigneusement la capsule de 90 mm dans l’enceinte de biosécurité et utiliser une pipette de 200 μL pour recueillir les sphéroïdes de chaque gouttelette. Prélever cinq sphéroïdes chacun dans un tube de 1,5 mL.
- Ajouter 500 μL de 1x PBS dans le tube de 1,5 mL contenant des sphéroïdes et centrifuger à 125 x g pendant 5 min. Jeter soigneusement le surnageant et remettre les sphéroïdes en suspension dans 200 μL de 1x PBS. Ne pas pipeter rigoureusement pour éviter la désintégration sphéroïde.
- Extraire les sphéroïdes à l’aide d’une pipette de 200 μL sur une capsule de 60 mm pour la coloration à la calcéine-AM et à l’iodure de propidium.
- Mettre 5 μL de solution de calcéine-AM 1 μM et 5 μL de solution d’iodure de propidium à 2 mg/mL sur les sphéroïdes. Incuber pendant 10 min. Une fois la période d’incubation terminée, lavez doucement les sphéroïdes avec 1x PBS deux fois.
- Placez la parabole de 60 mm contenant des sphéroïdes sous le microscope fluorescent inversé, observez et capturez des images à un grossissement de 10x en sélectionnant l’option de fluorescence dans le logiciel de microscope et en sélectionnant le FITC (canal de fluorescence vert; excitation 490 nm, émission 515 nm) pour la calcéine et le canal rouge du Texas (TXR; excitation 535 nm, émission 617 nm).
- Pour acquérir l’image, activez Ctr Adv, qui est le commutateur 1, puis allumez le processeur. Attendez que le système logiciel démarre. Une fois démarré, placez soigneusement l’antenne de 60 mm sur la scène et sélectionnez le grossissement (10x) et les canaux fluorescents (FITC, TXR). Ajustez les objectifs et numérisez l’image.
- Pour afficher l’image dans le système, sélectionnez l’option En direct et affichez l’image. Ajustez l’intensité de la fluorescence pour une optimisation appropriée. Enregistrez l’image en cliquant sur le bouton Enregistrer .
REMARQUE: À ce stade, les sphéroïdes seront visibles à l’œil nu. Au microscope optique, les sphéroïdes apparaîtront sous forme de sphères rondes et rigides à un grossissement de 10x. Plusieurs sphéroïdes peuvent être collectés à la fois à l’aide d’une pipette de 200 μL.
4. Désintégration et suspension cellulaire des sphéroïdes tumoraux
- Prélever 200 sphéroïdes tumoraux chacun les jours 7 et 10, à l’aide d’une pipette de 1 ml dans un tube de 15 ml.
REMARQUE: Avant le prélèvement, vérifiez soigneusement la forme d’un seul sphéroïde après l’avoir transféré sur une lame microscopique ou sur une antenne de 30 mm et observez-le au microscope. - Enduire les sphéroïdes par centrifugation à 125 x g pendant 5 min. Aspirer le surnageant avec précaution sans déranger les sphéroïdes tumoraux.
- Lavez soigneusement les sphéroïdes avec 200 μL de PBS, centrifugez à 125 x g pendant 5 minutes et jetez prudemment le surnageant.
- Pour la désintégration sphéroïde, ajouter 400 μL de solution trypsine-EDTA à 0,25% et maintenir à 37 °C pendant 10 min. Effectuer un pipetage vigoureux pour une désintégration complète des sphéroïdes.
- Neutraliser la trypsine en ajoutant 1 mL de milieu de croissance DMEM complet. Centrifuger à 125 x g pendant 5 min et jeter soigneusement le surnageant.
- Remettez la pastille en suspension dans 1 mL de milieu DMEM complet et comptez le nombre total de cellules.
5. Isolement des fibroblastes associés au cancer (CAF) par des microbilles
- Pour l’isolement des CAF des sphéroïdes tumoraux, remettre en suspension 1 x 10 7 cellules dans 80 μL de tampon de tri cellulaire activé magnétique à froid (MACS) (PBS à pH7,2 contenant 0,5% d’albumine sérique bovine [BSA] et 2 mM d’acide éthylènediamine tétraacétique [EDTA]).
- Incuber la suspension cellulaire (contenant 1 x 107 cellules) avec 20 μL de microbilles anti-fibroblastes. Bien mélanger en tapotant doucement le tube et incuber pendant 30 min à température ambiante.
- Laver les cellules avec 1 mL de tampon MACS froid, centrifuger à 125 x g pendant 5 min et aspirer le surnageant. Remettez les cellules en suspension dans 500 μL de tampon MACS.
- Pour la séparation cellulaire à base de billes magnétiques, préparez la colonne MACS en la rinçant avec 3 mL de tampon MACS.
- Placez la suspension cellulaire dans la colonne, suivie de la collection d’écoulement continu contenant la population cellulaire non marquée.
- Lavez la colonne trois fois avec 3 ml de tampon MACS. Retirez la colonne du séparateur et placez-la sur le tube de collecte.
- Recueillir les cellules marquées par des microbilles antifibroblastes en ajoutant 5 mL de tampon MACS et en poussant fermement le piston dans la colonne.
- Centrifuger les cellules marquées à 125 x g pendant 5 min. Procéder avec les fibroblastes isolés pour les applications en aval.
6. Analyse basée sur la cytométrie de flux de l’expression d’ACTA2 dans des CAF isolés
- Comptez le nombre de CAF isolés et utilisez environ 6 x 104 cellules. Lavez les cellules une fois avec du PBS, centrifugez à 125 x g pendant 5 minutes et aspirez le surnageant.
- Ajouter 100 μL de tampon de perméabilisation cellulaire (PBS + 0,5% BSA + 0,3% v/v Triton X-100) et incuber les cellules à 4 °C pendant 30 min.
- Vortex les cellules par intermittence pour maintenir une suspension unicellulaire. Centrifuger et remettre en suspension les cellules dans 100 μL de tampon de perméabilisation cellulaire.
- Ajouter 2 μL d’anticorps anti-α-SMA humains conjugués APC et incuber à 4 °C pendant 45 min. Après l’incubation, ajouter 1 mL de tampon de perméabilisation et centrifuger à 125 x g pendant 5 minutes pour éliminer l’excès d’anticorps.
- Resuspendre la pastille dans 400 μL de tampon de perméabilisation pour l’analyse cytométrique en flux. Acquérir un total de 10 000 événements de chaque échantillon dans un cytomètre en flux. Sélectionnez les cellules positives ACTA2 après avoir distingué les populations de cellules en fonction de leur diffusion directe et latérale, en sélectionnant la population singulet suivie de la sélection des cellules positives ACTA2 qui apparaissent comme un seul pic sur l’histogramme à paramètre unique.
7. Coloration JC-1 pour déterminer le potentiel de la membrane mitochondriale
- Compter le nombre de CAF isolés et utiliser environ 6 x 104 cellules pour la coloration à l’iodure de tétraéthylbenzimi-dazoylcarbocyanine (JC-1) au 5,5,6,6'-tétrachloro-1,1',3,3' tétraéthylbenzimi-dazoylcarbocyanine (JC-1) par cytométrie en flux.
- Lavez soigneusement les cellules avec du PBS, centrifugez à 125 x g pendant 5 min, aspirez le surnageant et ajoutez 100 μL de tampon de coloration cellulaire.
- Ajouter le colorant JC-1 à une concentration de travail de 2 μM et incuber à température ambiante pendant 30 min. À la fin de l’incubation, laver les cellules avec du PBS, centrifuger à 125 x g pendant 5 min et remettre en suspension dans un volume final de 400 μL.
- Acquérir un total de 20 000 événements de chaque échantillon dans un cytomètre en flux. Quantifier le potentiel de la membrane mitochondriale en mesurant les agrégats JC-1 décalés vers le rouge sur le canal FL-2 et les monomères décalés vers le vert sur le canal FL-1.
8. Coloration DCFDA pour estimer les niveaux d’espèces réactives de l’oxygène cellulaire (ROS)
- Utilisez les sphéroïdes entiers (50 chiffres) ainsi que les CAF isolés (6 x 104 cellules) des sphéroïdes pour la coloration au diacétate de 2',7'-dichlorodihydrofluorescéine (DCFDA).
- Lavez soigneusement les cellules avec du PBS, centrifugez à 125 x g pendant 5 min, aspirez le surnageant et ajoutez 100 μL de tampon de coloration cellulaire.
- Ajouter le colorant DCFDA à une concentration de travail de 1 μM et incuber à température ambiante pendant 30 min. Laver les cellules deux fois avec du PBS, centrifuger à 125 x g pendant 5 min, puis remettre en suspension dans un volume final de 400 μL.
- Acquérir un total de 20 000 événements de chaque échantillon dans un cytomètre en flux. Évaluer les niveaux de ROS en mesurant la fluorescence au jour 7 et au jour 10 pour les sphéroïdes ainsi que les CAF isolés.
9. Analyse RT-qPCR des marqueurs CAF et des gènes glycolytiques
- Extraire l’ARN des CAF triés à l’aide d’un kit de lyse unicellulaire suivant le protocole du fabricant. Préparer l’ADNc à partir de 100 ng d’ARN à l’aide d’un kit de synthèse d’ADNc en suivant les directives du fabricant.
- Effectuer la RT-qPCR pour analyser les marqueurs CAF relatifs (ACTA2 8 et COL1A29) et l’expression du gène glycolytique (GLUT1 10 et MCT4 11). Effectuer une analyse de la courbe de fusion après l’extension finale pour s’assurer de la spécificité des produits. Normaliser les données en utilisant l’expression de GAPDH comme gène de référence. Les séquences d’amorce utilisées pour la RT-qPCR sont énumérées dans le tableau supplémentaire 1.
10. Extraction et quantification de la protéine cellulaire des CAF
REMARQUE: Effectuez toutes les étapes de l’extraction des protéines sur la glace pour éviter la dégradation des protéines.
- Resuspendre environ 4 x 106 cellules CAF dans 100 μL de tampon de lyse RIPA glacé (contenant 30 mM HEPES, 150 mM NaCl, 1% NP-40, 0,1% SDS et 0,5% de désoxycholate de sodium avec 5 mM de cocktail de protéase et d’inhibiteur de phosphatase Halt, et 5 mM d’EDTA à pH 7,4).
- Vortex rigoureusement pour une lyse cellulaire appropriée. Sonicer la suspension cellulaire à une fréquence de 20 Hz, une amplitude de 20 % pendant 15 s et une impulsion de 3x.
- Après sonication, centrifuger l’extrait protéique à 13 000 x g pendant 15 min à 4 °C. Transférer le surnageant dans un tube prérefroidi de 1,5 mL. Effectuer le dosage des protéines BCA pour quantifier les protéines.
11. Analyse spectrophotométrique des activités enzymatiques dans les CAF
REMARQUE: Les activités enzymatiques suivantes sont analysées dans les CAF dérivés de sphéroïdes tumoraux.
- Mesure de l’activité de la succinate déshydrogénase
- Pour évaluer l’activité de la succinate déshydrogénase (SDH) dans les CAF, préparer le tampon SHE avec 250 mM de saccharose, 10 mM d’acide 4-(2-hydroxyéthyl)-1-pipérazinééthanésulfonique (HEPES) et 1 mM d’acide éthylèneglycol-bis(β-aminoéthyléther)-N,N,N′,N′-tétraacétique (EGTA) et ajuster le pH à 7,3. Ajouter 0,4 mM de méthosulfate de phénazine (PMS), 0,2 mM de 2,6-dichloroindophénol (DCPIP), 50 mM de MgCl2, 0,02 % de Triton X-100 et 1 mM de cyanure dans le tampon SHE et maintenir à 37 °C.
- Extraire la protéine cellulaire des CAF comme décrit à l’étape 10 (2 x 106 cellules). Dans chaque puits d’une plaque de 96 puits, incuber 100 μL de 0,1 mg de protéine cellulaire avec 100 μL de mélange réactionnel SHE à 37 °C pendant 15 min.
- Pour démarrer l’activité enzymatique, ajouter 10 mM de succinate12. Calculer l’activité enzymatique en nM/min/mL en mesurant les changements d’absorbance à 600 nm.
- Le facteur de conversion du DCIP est de 0,0215 A/nM sur la base de son coefficient d’extinction molaire12. Calculez l’activité relative de SDH en utilisant la formule mentionnée:
Activité relative (nM/min/mL/enzyme) =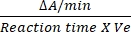 X X
X X  V
V
Où ΔA/min = vitesse de réaction enzymatique (A initial- A final)/(tempsfinal - A/mininitial), Ve = volume de l’échantillon, et V = volume de la réaction.
- Estimation de l’activité de la cytochrome c oxydase
- Pour évaluer l’activité de la cytochrome c oxydase (COX) dans les CAF, ajouter 0,2 mg/mL de protéine cellulaire dans une solution tampon réactionnelle KME contenant du tampon KME (125 mM KCl, 20 mM d’acide 3-(N-morpholino)propanesulfonique [MOPS] et 1 mM d’EGTA, pH 7,4), 0,02 % de Triton X-100 et 5 mM d’ascorbate de sodium.
- Dans un tube séparé, mélanger 50 μM de cytochrome c cardiaque de cheval avec 5 mM d’ascorbate de sodium et incuber à 37 °C pendant 5 min13. Après l’incubation, ajouter 20 μL de cytochrome c réduit à 500 μL de solution tampon réactionnelle KME contenant des protéines cellulaires.
- Calculer l’activité enzymatique en unités/μL en mesurant les changements d’absorbance à 550 nm. L’absorption du cytochrome c à 550 nm change avec son état d’oxydation. La différence de coefficients d’extinction (mM) entre le cytochrome c réduit et oxydé est de 21,84 à 550 nm14. Calculer la quantité d’activité enzymatique dans le lysat cellulaire comme:
Unités/μL =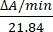
Où ΔA/min = A/minéchantillon A/minblanc et 21,84 = Δt͑ mM entre le cytochrome c oxydé et le cytochrome c réduit à 550 nm
- Évaluation de l’activité de la lactate déshydrogénase
- Effectuer un test de lactate déshydrogénase (LDH) à l’aide d’une trousse de dosage de cellules de lactate déshydrogénase en suivant les instructions du fabricant.
- Ajouter le réactif de test LDH (10 μL) à 0,5 mg/mL de protéines cellulaires et incuber pendant 5 min à 37 °C. À la fin de l’incubation, mesurer l’absorbance à 45 nm toutes les 1 min pendant 8 min.
- Préparer les étalons NADH pour la détection colorimétrique en utilisant 0 (blanc), 2,5, 5, 7,5, 10 et 12,5 nM/puits de NADH, puis ajouter du réactif de test LDH à un volume final de 50 μL.
- Tracer les différences d’absorbance entre Tinitial et Tfinal à la courbe standard NADH pour déterminer la quantité de NADH générée. Évaluer l’activité de la LDH en utilisant la formule mentionnée :
Activité de la LDH =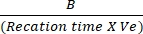 X facteur de dilution de l’échantillon
X facteur de dilution de l’échantillon
Où B = quantité (nmole) de NADH générée entre T initial et Tfinal, temps de réaction = Tfinal - Tinitial (min) et Ve = volume d’échantillon (mL) ajouté au puits.
Résultats
La figure 1 montre le développement de sphéroïdes tumoraux multicellulaires utilisant trois populations cellulaires différentes - A549 (adénocarcinome pulmonaire), MRC-5 (fibroblastes) et THP-1 (monocytes) - par la méthode de la goutte suspendue observée au jour 7 et au jour 10 au microscope. Au jour 7, les sphéroïdes étaient compacts et rigides avec un diamètre de 260 ± 5,3 μm, et au jour 10, les sphéroïdes avaient un diamètre de 480 ± 7,5 μm (figure 1...
Discussion
La présente étude introduit le développement de sphéroïdes tumoraux multicellulaires comprenant des cellules tumorales, une population de cellules stromales (fibroblastes) et une population de cellules immunitaires (monocytes) à l’aide d’une méthode de goutte suspendue modifiée. Les fibroblastes et les monocytes/macrophages font partie des populations les plus importantes qui constituent le microenvironnement tumoral (TME), et leur présence est souvent liée à un mauvais pronostic du patient
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Ce travail a été soutenu par le projet SERB-Women Excellence Award, Inde (SB/WEA-02/2017) et le SERB-Early Career Research Award Project, Inde (ECR/2017/000892) au Programme du diplôme. Les auteurs, LA et SR remercient IIT Ropar et MHRD pour leurs bourses de recherche. MK remercie l’ICMR pour sa bourse de recherche.
matériels
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| APC anti-human α-SMA | R&D systems | Cat# IC1420A | |
| Anti-fibroblast microbeads | Miltenyi Biotec | Cat# 130-050-601 | |
| Cell lines | |||
| A549 lung adenocarcinoma cells | NCCS Pune | - | |
| MRC-5 fetal lung fibroblasts | ATCC | CCL-171 | |
| THP-1 Human monocytes | NCCS Pune | - | |
| Chemicals | |||
| BSA | Himedia | Cat# 9048-46-8 | |
| 2,6-dichloroindophenol (DCPIP) | SRL | Cat# 55287 | |
| Calcein-AM | Thermo Fisher Scientific | Cat# C3099 | |
| DAPI | Thermo Fisher Scientific | Cat# D1306 | |
| DCFDA | Sigma | Cat# D6883 | |
| DMEM | Gibco | Cat# 11995073 | |
| DPBS | Gibco | Cat# 14190-144 | |
| EDTA | Thermo fisher scientific | Cat# 17892 | |
| EGTA | SRL | Cat# 62858 | |
| EZcoun Lactate Dehydrogenase Cell Assay Kit | HiMedia | Cat# CCK036 | |
| FBS | Gibco | Cat# 10082147 | |
| Halt Protease and Phosphatase Inhibitor Cocktail (100X) | Thermo Fisher Scientific | Cat# 87786 | |
| HEPES | Thermo Fisher Scientific | Cat# 15630080 | |
| Horse heart Cytochrome c | SRL | Cat# 81551 | |
| Image-iT Red hypoxia reagent | Thermo Fisher Scientific | Cat# H10498 | |
| JC-1 Dye | Thermo Fisher Scientific | Cat# T3168 | |
| KCl | Merck | Cat# P9541 | |
| MgCl2 | Merck | Cat# M8266 | |
| MOPS | Thermo Fisher Scientific | Cat# 69824 | |
| Nacl | Sigma-Aldrich | Cat# S9888 | |
| NADH MB Grade | SRL | Cat# 54941 | |
| NP-40 | Thermo Fisher Scientific | Cat# 85124 | |
| Penicillin/Streptomycin | Gibco | Cat# 15140122 | |
| Phenazine methosulfate (PMS) | SRL | Cat# 55782 | |
| Propidium iodide | Thermo fisher scientific | Cat# P1304MP | |
| RPMI 1640 | Gibco | Cat# 11875093 | |
| Single Cell Lysis Kit | Thermo Fisher Scientific | Cat# 4458235 | |
| Sodium ascorbate | Merck | Cat# A7631 | |
| Sodium cyanide | Sigma | Cat# 205222 | |
| Sodium Deoxycholate | Thermo Fisher Scientific | Cat# 89904 | |
| Sodium dodecyl sulphate | Sigma-Aldrich | Cat# L3771 | |
| Sodium succinate hexahydrate | SRL | Cat# 36313 | |
| Sucrose | Sigma | Cat# S0389 | |
| SuperScript VILO cDNA synthesis kit | Thermo Fisher Scientific | Cat# 11754-050 | |
| Triton X-100 | Sigma | Cat# T8787 | |
| Trypsin 0.25% EDTA | Gibco | Cat# 25200072 | |
| Universal SYBR Green Supermix | BIO-RAD | Cat# 172-5124 | |
| Plasticware | |||
| MACS LS Columns | Miltenyi Biotec | Cat# 130-042-401 | |
| Equipment | |||
| Countess II FL Automated Cell Counter | Thermo Fisher Scientific | Cat# AMQAF1000 | |
| EVOS XL core imaging system | Thermo Fisher Scientific | Serial Number F0518-1727-0191 | |
| LAS X software | Leica Microsystems | ||
| Leica fluorescent inverted microscope | s | DMi8 automated S/N 424150) | |
| Midi MACS separator | Miltenyi Biotec | Cat# 130-042-302 |
Références
- Kim, N., et al. Single-cell RNA sequencing demonstrates the molecular and cellular reprogramming of metastatic lung adenocarcinoma. Nature Communications. 11 (1), 1-5 (2020).
- Davidson, S., et al. Single-cell RNA sequencing reveals a dynamic stromal niche that supports tumor growth. Cell Reports. 31 (7), 107628 (2020).
- Zhang, Y., et al. Single-cell analyses of renal cell cancers reveal insights into tumor microenvironment, cell of origin, and therapy response. Proceedings of the National Academy of Sciences. 118 (24), (2021).
- Bray, L. J., Hutmacher, D. W., Bock, N. Addressing patient specificity in the engineering of tumor models. Frontiers in Bioengineering and Biotechnology. 7, 217 (2019).
- Timmins, N. E., Nielsen, L. K. Generation of multicellular tumor spheroids by the hanging-drop method. Tissue Engineering. , 141-151 (2007).
- Dituri, F., et al. Complex tumor spheroid formation and one-step cancer-associated fibroblasts purification from hepatocellular carcinoma tissue promoted by inorganic surface topography. Nanomaterials. 11 (12), 3233 (2021).
- Arora, L., et al. Development of a multicellular 3D tumor model to study cellular heterogeneity and plasticity in NSCLC tumor microenvironment. Frontiers in Oncology. 12, 881207 (2022).
- Nurmik, M., Ullmann, P., Rodriguez, F., Haan, S., Letellier, E. In search of definitions: Cancer-associated fibroblasts and their markers. International Journal of Cancer. 146 (4), 895-905 (2020).
- Zhang, Y., et al. HIF-1α is necessary for activation and tumour-promotion effect of cancer-associated fibroblasts in lung cancer. Journal of Cellular and Molecular Medicine. 25 (12), 5457-5469 (2021).
- Bu, L., et al. Biological heterogeneity and versatility of cancer-associated fibroblasts in the tumor microenvironment. Oncogene. 38 (25), 4887-4901 (2019).
- Whitaker-Menezes, D., et al. Evidence for a stromal-epithelial "lactate shuttle" in human tumors: MCT4 is a marker of oxidative stress in cancer-associated fibroblasts. Cell cycle. 10 (11), 1772-1783 (2011).
- Mandujano-Tinoco, E. A., Gallardo-Pérez, J. C., Marín-Hernández, A., Moreno-Sánchez, R., Rodríguez-Enríquez, S. Anti-mitochondrial therapy in human breast cancer multi-cellular spheroids. Biochimica et Biophysica Acta (BBA)-Molecular Cell Research. 1833 (3), 541-551 (2013).
- Bregman, A. A. . Laboratory Investigations in Cell and Molecular Biology. , (2002).
- Berry, E. A., Trumpower, B. L. Simultaneous determination of hemes a, b, and c from pyridine hemochrome spectra. Analytical Biochemistry. 161 (1), 1-15 (1987).
- Avagliano, A., et al. Metabolic reprogramming of cancer associated fibroblasts: the slavery of stromal fibroblasts. BioMed Research International. , (2018).
- Lorusso, G., Rüegg, C. The tumor microenvironment and its contribution to tumor evolution toward metastasis. Histochemistry and Cell Biology. 130 (6), 1091-1103 (2008).
- Liu, T., Zhou, L., Li, D., Andl, T., Zhang, Y. Cancer-associated fibroblasts build and secure the tumor microenvironment. Frontiers in Cell and Developmental Biology. 7, 60 (2019).
- Sebastian, A., et al. Single-cell transcriptomic analysis of tumor-derived fibroblasts and normal tissue-resident fibroblasts reveals fibroblast heterogeneity in breast cancer. Cancers. 12 (5), 1307 (2020).
- Elyada, E., et al. Cross-species single-cell analysis of pancreatic ductal adenocarcinoma reveals antigen-presenting cancer-associated fibroblasts. Cancer Discovery. 9 (8), 1102-1123 (2019).
- Ganguly, D., et al. Cancer-associated fibroblasts: Versatile players in the tumor microenvironment. Cancers. 12 (9), 2652 (2020).
- Harryvan, T. J., Verdegaal, E. M., Hardwick, J. C., Hawinkels, L. J., vander Burg, S. H. Targeting of the cancer-associated fibroblast-T-cell axis in solid malignancies. Journal of Clinical Medicine. 8 (11), 1989 (2019).
- Santi, A., Kugeratski, F. G., Zanivan, S. Cancer associated fibroblasts: the architects of stroma remodeling. Proteomics. 18 (5-6), 1700167 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon