Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Aplicación de AlDeSense para estratificar células de cáncer de ovario basadas en la actividad de aldehído deshidrogenasa 1A1
* Estos autores han contribuido por igual
En este artículo
Resumen
Los métodos para medir la actividad de ALDH1A1 en células vivas son críticos en la investigación del cáncer debido a su estado como biomarcador de tallo. En este estudio, empleamos una sonda fluorogénica selectiva de isoformas para determinar los niveles relativos de actividad de ALDH1A1 en un panel de cinco líneas celulares de cáncer de ovario.
Resumen
La recaída después del tratamiento del cáncer a menudo se atribuye a la persistencia de una subpoblación de células tumorales conocidas como células madre cancerosas (CSC), que se caracterizan por su notable capacidad de iniciación y autorenovación tumoral. Dependiendo del origen del tumor (por ejemplo, ovarios), el perfil de biomarcadores de superficie de CSC puede variar drásticamente, lo que hace que la identificación de tales células a través de la tinción inmunohistoquímica sea un esfuerzo difícil. Por el contrario, la aldehído deshidrogenasa 1A1 (ALDH1A1) se ha convertido en un excelente marcador para identificar las CSC, debido a su perfil de expresión conservado en casi todas las células progenitoras, incluidas las CSC. La isoforma ALDH1A1 pertenece a una superfamilia de 19 enzimas que son responsables de la oxidación de varios aldehídos endógenos y xenobióticos a los productos de ácido carboxílico correspondientes. Chan et al. desarrollaron recientemente AlDeSense, una sonda de "encendido" selectiva de isoformas para la detección de la actividad de ALDH1A1, así como un reactivo de control de coincidencia no reactivo (Ctrl-AlDeSense) para tener en cuenta la tinción fuera del objetivo. Esta herramienta selectiva de isoformas ya ha demostrado ser una herramienta química versátil a través de la detección de la actividad de ALDH1A1 en células de leucemia mielógena K562, mamosferas y xenoinjertos CSC derivados del melanoma. En este artículo, la utilidad de la sonda se mostró a través de fluorimetría adicional, microscopía confocal y experimentos de citometría de flujo donde se determinó la actividad relativa de ALDH1A1 en un panel de cinco líneas celulares de cáncer de ovario.
Introducción
Las células madre cancerosas (CSC) son una subpoblación de células tumorales que exhiben propiedades similares a las células madre1. Al igual que sus contrapartes no cancerosas, las CSC poseen la extraordinaria capacidad de autorrenovarse y proliferar. Junto con otros mecanismos incorporados, como la regulación positiva de los transportadores de casetes de unión a ATP, las CSC a menudo se salvan de los esfuerzos quirúrgicos iniciales de citorreducción, así como de la terapia adyuvante posterior2. Debido a su papel crítico en la resistencia al tratamiento3, la recaída4 y la metástasis5, las CSC se han convertido en una prioridad en la investigación del cáncer. A pesar de que hay una variedad de antígenos de superficie celular (por ejemplo, CD133) que pueden ser utilizados para identificar CSCs6, el aprovechamiento de la actividad enzimática de las aldehído deshidrogenasas (ALDHs) encontradas en el citoplasma ha surgido como una alternativa atractiva7. Los ALDH son una superfamilia de 19 enzimas responsables de catalizar la oxidación de aldehídos endógenos y xenobióticos reactivos a los productos de ácido carboxílico correspondientes8.
En general, la desintoxicación de aldehído es crucial para proteger las células de eventos de reticulación indeseables y estrés oxidativo que pueden dañar la integridad de las células madre9. Además, la isoforma 1A1 controla el metabolismo del ácido retinoico, que a su vez influye en el tallo a través de la señalización del retinaldehído10. Recientemente se desarrolló AlDeSense 11,12, una sonda de detección basada en la actividad de moléculas pequeñas (ABS) para detectar selectivamente la actividad de ALDH1A1. Los diseños de ABS logran la detección de analitos a través de un cambio químico en lugar de un evento vinculante, lo que permite una alta selectividad y una disminución de las respuestas fuera del objetivo13,14,15,16. El principio de diseño de la sonda fluorogénica selectiva de isoforma se basa en un mecanismo de enfriamiento de transferencia de electrones fotoinducida por donante (d-PeT)17, originado en el grupo funcional aldehído, que sirve para suprimir la firma fluorescente de la sonda18. Tras la conversión mediada por ALDH1A1 al ácido carboxílico, la relajación radiativa se desbloquea para producir un producto altamente fluorescente. Debido a que el enfriamiento de d-PeT nunca es 100% eficiente, la fluorescencia residual que puede conducir a posibles resultados falsos positivos se consideró al establecer este ensayo a través del desarrollo de Ctrl-AlDeSense, un reactivo que no responde con características fotofísicas coincidentes (por ejemplo, rendimiento cuántico) y un patrón de tinción citoplasmática idéntico en las células. Cuando se usa en conjunto, este emparejamiento único puede distinguir de manera confiable las células con alta actividad ALDH1A1 de aquellas que exhiben niveles bajos a través de fluorimetría, imágenes moleculares y citometría de flujo. Varias ventajas clave están asociadas con el uso de colorantes activables selectivos de isoformas sobre los métodos inmunohistoquímicos tradicionales. Por ejemplo, se presume que las CSC están enterradas profundamente dentro de un tumor y, por lo tanto, son más accesibles a una molécula pequeña en relación con los anticuerpos grandes19. Además, el producto fluorescente volteado no modifica covalentemente ningún componente celular, lo que significa que se puede eliminar fácilmente a través de ciclos de lavado para dejar un CSC en un estado sin modificar. Por último, la respuesta de encendido solo identifica células y funciones viables, al igual que el ensayo MTT, debido a su dependencia del cofactor NAD +.
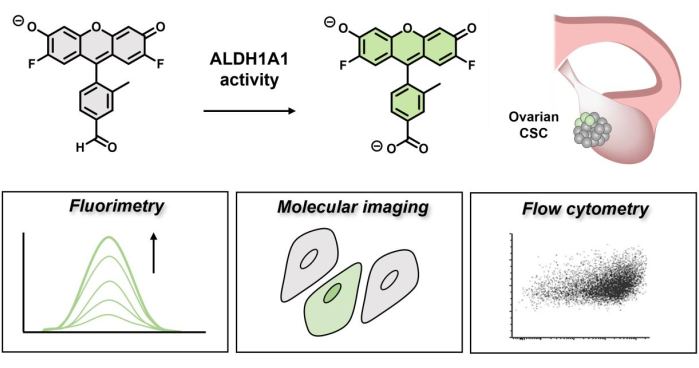
Figura 1: Esquema que demuestra el encendido fluorescente de AlDeSense. El colorante selectivo de isoforma es activado por ALDH1A1 y se puede utilizar para identificar la actividad elevada de ALDH1A1 en células de cáncer de ovario a través de fluorimetría, imágenes moleculares y citometría de flujo. Haga clic aquí para ver una versión más grande de esta figura.
En trabajos anteriores, el ensayo de sonda fluorogénica selectiva de isoformas estratificó con éxito células ALDH altas (ALDH +) de células ALDH bajas (ALDH-) en células de leucemia crónica humana K562, células de cáncer de mama humano MDA-MB-231 y células de melanoma murino B16F0. Esto es importante porque, para muchos tipos de cáncer, la expresión alta de la proteína ALDH1A1 significa un peor pronóstico clínico20. Esto supone que los niveles elevados de ALDH1A1 son indicativos de CSC que pueden evadir el tratamiento, desarrollar resistencia y diseminarse por todo el cuerpo. Sin embargo, en el caso del cáncer de ovario, hay estudios que reportan el hallazgo contrario (la alta expresión de ALDH1A1 está relacionada con una mejor supervivencia de la paciente)21,22,23,24. Si bien esto puede parecer contradictorio a primera vista, la expresión no necesariamente se correlaciona con la actividad enzimática, que puede estar influenciada por cambios en el microambiente tumoral (por ejemplo, flujo de pH, gradientes de oxígeno), disponibilidad del cofactor NAD + o sustratos aldehídos, niveles de ácidos carboxílicos (inhibición del producto) y modificaciones postraduccionales que pueden alterar la actividad enzimática25 . Además, el cáncer de ovario se divide en cinco tipos histológicos principales (seroso de alto grado, seroso de bajo grado, endometrioide, de células claras y mucinoso), que hipotetizamos que presentarán niveles variables de actividad ALDH1A126. Con el objetivo de investigar la actividad de ALDH1A1 en tumores de ovario, se empleó un ensayo de sonda fluorogénica selectiva de isoforma para identificar poblaciones de ALDH1A1+ en un panel de cinco líneas celulares de cáncer de ovario pertenecientes a los diferentes tipos histológicos mencionados anteriormente. Las líneas celulares probadas en este estudio incluyen células BG-1, Caov-3, IGROV-1, OVCAR-3 y PEO4, que cubren histotipos de células claras y serosas. Aquí, se destacó la versatilidad y generalización de la sonda para identificar CSC para los investigadores que buscan realizar estudios similares en otras líneas celulares de cáncer inmortalizadas, así como muestras de pacientes. El uso de AlDeSense arrojará luz sobre las vías bioquímicas involucradas en el mantenimiento de CSC en microambientes tisulares complejos y potencialmente servirá como una herramienta clínica para determinar el pronóstico y medir la agresividad del cáncer.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Medir la actividad total de ALDH1A1 en homogeneizados de células de cáncer de ovario a través de fluorimetría
- Descongelar 1 × 106 células en un matraz de cultivo celular T25 en 5 ml de los siguientes medios de cultivo celular:
- IGROV-1 y PEO4: Roswell Park Memorial Institute (RPMI) 1640 medio con 10% de suero fetal bovino (FBS) y 1% de penicilina/estreptomicina (P/S).
- BG-1 y Caov-3: Dulbecco's Modified Eagle's Medium (DMEM) con 10% FBS y 1% P/S.
- OVCAR-3: RPMI 1640 con 20% FBS, 1% P/S y 0,01 mg/ml de insulina.
- Mantener las células en una incubadora a 37 °C y 5% deCO2 durante dos o tres pasos. Asegúrese de que las células no excedan el 80% -90% de confluencia antes del paso.
- Tripsinizar las células en tripsina al 0,25% durante 10 min, contarlas con un contador de células automatizado y granular 1 × 107 células mediante centrifugación (180 × g) a 25 °C durante 5 min.
- Retire el sobrenadante cuidadosamente por aspiración, lave el pellet resuspendiendo las células en 1 ml de 1x PBS y vuelva a granular las células mediante centrifugación en las mismas condiciones que se describen en el paso 4.
- Resuspender las células en una solución de inhibidor de proteasa 1x en 1x PBS. Use 1 ml de esta solución por cada 2,5 × 106 células.
- Sonicar la suspensión celular en hielo durante 2 min (pulso de 1 s, 40% de amplitud) con una sonda homogeneizadora celular.
- Fracciones insolubles/membrana del pellet por centrifugación (3.200 × g) a 25 °C durante 15 min. Retire y mantenga el sobrenadante, ya que este es el homogeneizado que se utilizará en los pasos posteriores. Separar los homogeneizados en tres cubetas para realizar el experimento por triplicado.
- Añadir la sonda al homogeneizado para obtener una concentración final de la sonda de 4 μM. Pipetear hacia arriba y hacia abajo tres veces para mezclar bien la solución.
- Mida la señal fluorescente inmediatamente después de la adición y después de los tiempos de incubación deseados en un fluorímetro. El tiempo de incubación se puede optimizar para ver el encendido máximo del pliegue (1 h en este experimento). El tiempo de incubación debe ser constante en todas las líneas celulares comparadas.
- Establezca la longitud de onda de excitación en 496 nm.
- Ajuste la emisión a 510-600 nm.
- Ajuste el ancho de la hendidura a 0,5 mm.
- Pipeta la solución en una cubeta de cuarzo de 1 ml, colóquela en el fluorímetro y presione escanear.
- Divida la intensidad de fluorescencia final a 516 nm (longitud de onda fluorescente máxima) por la intensidad en la misma longitud de onda de la lectura inicial para determinar la activación del pliegue del colorante selectivo de isoforma.
2. Uso de microscopía de fluorescencia para obtener imágenes de células con alta actividad ALDH1A1
- Descongelar 1 × 106 células en un matraz de cultivo celular T25 en 5 ml del medio de cultivo celular apropiado.
- Mantener las células en una incubadora a 37 °C y 5% deCO2 durante dos o tres pasos. Asegúrese de que las células no excedan el 80% -90% de confluencia antes del paso.
- El día anterior a la toma de imágenes confocales, la placa 4 × 105 células en un portaobjetos de cámara de 8 pocillos.
- Cubra el fondo de cada pocillo con poli-L-lisina (0,1 mg/ml, 100 μL por pocillo) durante 10 min, aspirando posteriormente.
- Lave cada pocillo 3 veces agregando agua de grado de cultivo celular (100 μL por pocillo) y aspirando.
- Tripsinizar las células en tripsina al 0,25% durante 10 minutos, contarlas con un contador de células automatizado y colocar las células en 4 × 105 células por pocillo.
- Deje que las células se asienten y se adhieran durante la noche (12-16 h).
- Aspirar el medio de crecimiento y añadir medios libres de suero (500 μL por pocillo), complementados con 2 μM de la sonda o la sonda de control.
- Incubar con la sonda a temperatura ambiente durante 30 minutos y obtener imágenes de las células inmediatamente.
- Encienda el microscopio confocal y ajuste la muestra.
- Cargue la muestra cuidadosamente con el menor aumento; Encuentre las celdas para garantizar un posicionamiento adecuado.
- Asegúrese de que la lente del objetivo tenga un aumento de 10x y ubique las celdas.
- Para este experimento, se requieren el canal FITC (excitación: láser de 488 nm; emisión: 516-521 nm) y el canal T-PMT (luz transmitida). Localice y concéntrese en las células usando T-PMT para enfocarse en el mismo plano z durante todo el experimento para eliminar el sesgo.
- Ajuste la potencia del láser y la ganancia FITC a la configuración adecuada, donde la señal de las muestras Ctrl-AlDeSense AM es mínimamente detectable, mientras sigue viendo la señal en las muestras AlDeSense AM. Cada parámetro se puede ajustar deslizando la barra correspondiente. Es posible que la configuración deba ajustarse varias veces para identificar los parámetros correctos. Una vez optimizado, completa el resto del experimento dentro de esa línea celular usando parámetros idénticos.
- Tome tres imágenes por pocillo para un total de tres pozos por condición de tratamiento (nueve imágenes en total). Concéntrese en el plano adecuado usando T-PMT para evitar el sesgo en lugar de usar el canal de fluorescencia o la imagen combinada.
- Procese las imágenes para determinar el porcentaje de células ALDH1A1+.
- Usando el software de procesamiento de imágenes, divida el archivo czi en diferentes canales.
- Cuente el número total de células y el número total de células fluorescentes.
- Para determinar el porcentaje de células ALDH1A1+, divida el número de células fluorescentes por el número total de células en cada imagen. Es imperativo contar de la misma manera para cada imagen sin manipular las imágenes, ya que, por ejemplo, ajustar el brillo puede agregar una variable de confusión.
3. Aplicación de citometría de flujo para identificar células con alta actividad ALDH1A1
- Descongelar 1 × 106 células en un matraz de cultivo celular T25 en 5 ml del medio de cultivo celular apropiado.
- Mantener las células en una incubadora a 37 °C y 5% deCO2 durante dos o tres pasos. Asegúrese de que las células no excedan el 80% -90% de confluencia antes del paso.
- Tripsinizar, contar y granular las células en un tubo de centrífuga de 15 ml mediante centrifugación (180 × g) a 25 °C durante 5 min.
- Resuspender las células en 1 ml de solución de sonda de sonda/sonda de control de 2 μM en PBS. Mece las células a temperatura ambiente durante 60 minutos para garantizar que la exposición al tinte sea uniforme.
- Después del período de incubación, granular las células por centrifugación (180 × g) a 25 °C durante 5 min. Resuspender las células en 0,5 mL de PBS. Pase las células a través de un colador celular (malla de nylon de 35 μm) para eliminar los grupos de células que pueden obstruir el citómetro de flujo. Coloque inmediatamente las celdas en hielo.
- Encienda el instrumento y ejecute el protocolo de inicio.
- Compruebe si hay líquido de vaina y residuos vacíos.
- Ejecute las líneas con 10% de lejía y agua durante 5 minutos cada una.
- Ejecute perlas de control de calidad para garantizar el funcionamiento adecuado.
- En la pestaña de configuración, seleccione FSC (dispersión directa), SSC (dispersión lateral) y FITC (isotiocianato de fluoresceína) para el filtro de fluorescencia .
- Dibuje los siguientes gráficos en la puerta para la viabilidad, singletes y fluorescencia. Optimice la potencia del láser (específica para el instrumento del usuario) para que las poblaciones celulares estén dentro de los parámetros dados para su posterior análisis.
- Diagrama de dispersión FSC-A versus SSC-A: la población de células principales está cerca del centro del gráfico.
- Diagrama de dispersión FSC-A versus FSC-W: banda horizontal estrecha indicativa de singletes (en lugar de grupos celulares).
- Diagrama de dispersión FITC-A versus FSC-A: observe la distribución de las células clasificadas por la sonda de encendido fluorogénico selectivo de isoformas.
- Histograma FITC-A: observe el cambio en la población basado en FITC para determinar el porcentaje de células ALDH1A1+.
- Para optimizar la potencia del láser FIFC, ejecute una muestra con la sonda de modo que la cola derecha de la curva del histograma esté cerca de la señal FITC-A máxima. Posteriormente, ejecute una muestra con la sonda de control. Un cambio de población debe ser observable para revelar el rango dinámico máximo. El paso de optimización de la potencia del láser puede tener que repetirse varias veces, pero la potencia del láser no debe alterarse en las muestras una vez que se ha designado un ajuste para un experimento.
- Ejecute cada muestra durante 10,000 recuentos (hecho por triplicado).
- Repita el paso 3.12 para cada línea celular, ya que habrá variabilidad en la absorción y la actividad de ALDH1A1.
- Después de completar la recolección de muestras, ejecute las líneas con cloro al 10% y agua durante 5 minutos cada una, luego inicie el apagado del instrumento.
- Procese los datos utilizando el software de citometría de flujo y compruebe la población celular deseada. Establezca las puertas para que todos los eventos caigan en la puerta ALDH1A1- o ALDH1A1+. Con la selección de la puerta rectangular, configure la puerta ALDH1A1- de modo que el >99,5% de los eventos en las muestras de la sonda de control se produzcan dentro de esta puerta. Las células restantes se considerarán ALDH1A1+. Estas mismas puertas se pueden aplicar a la muestra de la sonda para cuantificar el número de eventos considerados ALDH1A1- y ALDH1A1+.
Access restricted. Please log in or start a trial to view this content.
Resultados
Actividad total de ALDH1A1 de los homogeneizados de células de cáncer de ovario
Los giros de pliegue promedio para cada línea celular obtenida de este ensayo son: BG-1 (1.12 ± 0.01); IGROV-1 (1,30 ± 0,03); Caov-3 (1,72 ± 0,06); PEO4 (2,51 ± 0,29); y OVCAR-3 (10,25 ± 1,46) (Figura 2).
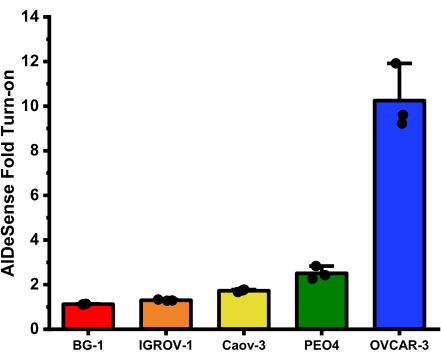
Figura...
Access restricted. Please log in or start a trial to view this content.
Discusión
La panselectividad es una limitación importante de muchas sondas ALDH; Sin embargo, recientemente se han reportado varios ejemplos selectivos de isoformas 32,33,34,35,36,37,38,39,40,41. ...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Divulgamos una patente pendiente (US20200199092A1) para la tecnología AlDeSense.
Agradecimientos
Este trabajo fue apoyado por los Institutos Nacionales de Salud (R35GM133581 a JC) y el Centro de Cáncer en Illinois Graduate Scholarship (otorgado a SG). JC agradece el apoyo de la Fundación Camille y Henry Dreyfus. Los autores agradecen al Dr. Thomas E. Bearrood por su contribución inicial a la preparación de las existencias de AlDeSense y AlDeSense AM. Agradecemos al Sr. Oliver D. Pichardo Peguero y al Sr. Joseph A. Forzano por su ayuda en la preparación de diversos precursores sintéticos. Agradecemos al Prof. Erik Nelson (Departamento de Fisiología Molecular e Integrativa, UIUC) por las células Caov-3, IGROV-1 y PEO4. Agradecemos al Prof. Paul Hergenrother (Departamento de Química, UIUC) por las células BG-1. Agradecemos a las instalaciones centrales del Instituto Carl R. Woese de Biología Genómica por el acceso al microscopio confocal Zeiss LSM 700 y al software correspondiente. Agradecemos a la Flow Cytometry Facility por el acceso al analizador BD LSR II CMtO. Sandra McMasters y al Cell Media Facility por su ayuda en la preparación de los medios de cultivo celular.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin, 0.1% EDTA in HBSS w/o Calcium, Magnesium and Sodium Bicarbonate | Corning | 25-050-CI | |
| 1x Phosphate Buffer Saline | Corning | 21-040-CMX12 | |
| AccuSpin Micro 17R | Fisher Scientific | 13-100-675 | |
| AlDeSense | Synthesized in-house | ||
| BG-1 | A gift provided by the Prof. Paul Hergenrother Lab, University of Illinois Urbana-Champaign | ||
| BioLite 25cm2 Flask | Thermo Fisher Scientific | 130189 | |
| Biosafety Cabinet 1300 series A2 | Thermo Fisher Scientific | ||
| Caov-3 | A gift provided by the Prof. Erik Nelson Lab, University of Illinois Urbana-Champaign | ||
| Cell homogenizer | Fisher Scientific | ||
| Centrifuge 5180R | Eppendorf | 22627040 | |
| Contrl-AlDeSense | Synthesized in-house | ||
| DMEM, 10% FBS, 1% P/S | Prepared by UIUC cell media facility | ||
| Falcon Round-Bottom Polystyrene Test Tubes with Cell Strainer Snap Cap, 5mL | Corning | 352003 | |
| FCS Express 6 | Provided by UIUC CMtO | ||
| FL microscope | EVOS | ||
| Fluorometer | Photon Technology International | ||
| Forma Series II Water-Jacketed CO2 Incubator | Fisher Scientific | 3110 | |
| IGROV-1 | A gift provided by the Prof. Erik Nelson Lab, University of Illinois Urbana-Champaign | ||
| ImageJ | NIH | ||
| Innova 42R Incubated Shaker | |||
| LSM 700 | Zeiss | ||
| LSR II | BD | ||
| Nunc Lab-Tek Chambered #1.0 Borosicilate Coverglass System | Thermo Fisher Scientific | 155383 | |
| OVCAR-3 | ATCC | HTB-161 | |
| PEO4 | A gift provided by the Prof. Erik Nelson Lab, University of Illinois Urbana-Champaign | ||
| Pierce Protease Inhibitor Tablets | Thermo Scientific | A32963 | |
| Poly-L-Lysine | Cultrex | 3438-100-01 | |
| Rocker | VWR | ||
| RPMI, 10% FBS, 1% P/S | Prepared by UIUC cell media facility | ||
| RPMI, 20% FBS, 1% P/S, 0.01 mg/mL Insulin | Prepared by UIUC cell media facility |
Referencias
- Bonnet, D., Dick, J. E. Human acute myeloid leukaemia is organised as a heirarchy that originates from a primitive haematopoetic cell. Nature Medicine. 3 (7), 730-737 (1997).
- Begicevic, R. R., Falasca, M. ABC transporters in cancer stem cells: Beyond chemoresistance. International Journal of Molecular Sciences. 18 (11), 2362(2017).
- Cojoc, M., Mäbert, K., Muders, M. H., Dubrovska, A. A role for cancer stem cells in therapy resistance: Cellular and molecular mechanisms. Seminars in Cancer Biology. 31, 16-27 (2015).
- Islam, F., Gopalan, V., Smith, R. A., Lam, A. K. Y. Translational potential of cancer stem cells: A review of the detection of cancer stem cells and their roles in cancer recurrence and cancer treatment. Experimental Cell Research. 335 (1), 135-147 (2015).
- Li, F., Tiede, B., Massagué, J., Kang, Y. Beyond tumorigenesis: Cancer stem cells in metastasis. Cell Research. 17 (1), 3-14 (2007).
- Kim, W. T., Ryu, C. J. Cancer stem cell surface markers on normal stem cells. BMB Reports. 50 (6), 285-298 (2017).
- Pors, K., Moreb, J. S. Aldehyde dehydrogenases in cancer: An opportunity for biomarker and drug development. Drug Discovery Today. 19 (12), 1953-1963 (2014).
- Jackson, B., et al. Update on the aldehyde dehydrogenase gene (ALDH) superfamily. Human Genomics. 5 (4), 283-303 (2011).
- Vasiliou, V., Pappa, A., Petersen, D. R. Role of aldehyde dehydrogenases in endogenous and xenobiotic metabolism. Chemico-Biological Interactions. 129 (1-2), 1-19 (2000).
- Tomita, H., Tanaka, K., Tanaka, T., Hara, A. Aldehyde dehydrogenase 1A1 in stem cells and cancer. Oncotarget. 7 (10), 11018-11032 (2016).
- Anorma, C., et al. Surveillance of cancer stem cell plasticity using an isoform-selective fluorescent probe for aldehyde dehydrogenase 1A1. ACS Central Science. 4 (8), 1045-1055 (2018).
- Bearrood, T. E., Aguirre-Figueroa, G., Chan, J. Rational design of a red fluorescent sensor for ALDH1A1 displaying enhanced cellular uptake and reactivity. Bioconjugate Chemistry. 31 (2), 224-228 (2020).
- Chan, J., Dodani, S. C., Chang, C. J. Reaction-based small-molecule fluorescent probes for chemoselective bioimaging. Nature Chemistry. 4 (12), 973-984 (2012).
- East, A. K., Lucero, M. Y., Chan, J. New directions of activity-based sensing for in vivo NIR imaging. Chemical Science. 12 (10), 3393-3405 (2021).
- Yadav, A. K., et al. Activity-based NIR bioluminescence probe enables discovery of diet-induced modulation of the tumor microenvironment via nitric oxide. ACS Central Science. 8 (4), 461-472 (2022).
- Yadav, A. K., et al. An activity-based sensing approach for the detection of cyclooxygenase-2 in Live Cells. Angewandte Chemie. 59 (8), 3307-3314 (2020).
- Ueno, T., et al. Rational principles for modulating fluorescence properties of fluorescein. Journal of the American Chemical Society. 126 (43), 14079-14085 (2004).
- Tanaka, F., Mase, N., Barbas 3rd, C. F. Design and use of fluorogenic aldehydes for monitoring the progress of aldehyde transformations. Journal of the American Chemical Society. 126 (12), 3692-3693 (2004).
- Thurber, G. M., Schmidt, M. M., Wittrup, K. D. Antibody tumor penetration: transport opposed by systemic and antigen-mediated clearance. Advanced Drug Delivery Reviews. 60 (12), 1421-1434 (2008).
- Marcato, P., Dean, C. A., Giacomantonio, C. A., Lee, P. W. K. Aldehyde dehydrogenase its role as a cancer stem cell marker comes down to the specific isoform. Cell Cycle. 10 (9), 1378-1384 (2011).
- Meng, E., et al. ALDH1A1 maintains ovarian cancer stem cell-like properties by altered regulation of cell cycle checkpoint and DNA repair network signaling. PLoS One. 9 (9), e107142(2014).
- Kaipio, K., et al. ALDH1A1-related stemness in high-grade serous ovarian cancer is a negative prognostic indicator but potentially targetable by EGFR/mTOR-PI3K/aurora kinase inhibitors. The Journal of Pathology. 250 (2), 159-169 (2020).
- Deng, S., et al. Distinct expression levels and patterns of stem cell marker, aldehyde dehydrogenase isoform 1 (ALDH1), in human epithelial cancers. PLoS One. 5 (4), e10277(2010).
- Chang, B., et al. ALDH1 expression correlates with favorable prognosis in ovarian cancers. Modern Pathology. 22 (6), 817-823 (2009).
- Gardner, S. H., Reinhardt, C. J., Chan, J. Advances in activity-based sensing probes for isoform-selective imaging of enzymatic activity. Angewandte Chemie. 60 (10), 5000-5009 (2021).
- Reid, B. M., Permuth, J. B., Sellers, T. A. Epidemiology of ovarian cancer: a review. Cancer Biology and Medicine. 14 (1), 9-32 (2017).
- Tulake, W., et al. Upregulation of stem cell markers ALDH1A1 and OCT4 as potential biomarkers for the early detection of cervical carcinoma. Oncology Letters. 16 (5), 5525-5534 (2018).
- Nwani, N. G., et al. A novel ALDH1A1 inhibitor targets cells with stem cell characteristics in ovarian cancer. Cancers. 11 (4), 502(2019).
- Roy, M., Connor, J., Al-Niaimi, A., Rose, S. L., Mahajan, A. Aldehyde dehydrogenase 1A1 (ALDH1A1) expression by immunohistochemistry is associated with chemo-refractoriness in patients with high-grade ovarian serous carcinoma. Human Pathology. 73, 1-6 (2018).
- Landen Jr, C. N., et al. Targeting aldehyde dehydrogenase cancer stem cells in ovarian cancer. Molecular Cancer Therapeutics. 9 (12), 3186-3199 (2010).
- Condello, S., et al. β-catenin-regulated ALDH1A1 is a target in ovarian cancer spheroids. Oncogene. 34 (18), 2297-2308 (2015).
- Storms, R. W., et al. Isolation of primitive human hematopoietic progenitors on the basis of aldehyde dehydrogenase activity. Proceedings of the National Academy of Sciences. 96 (16), 9118-9123 (1999).
- Duellman, S. J., et al. A bioluminescence assay for aldehyde dehydrogenase activity. Analytical Biochemistry. 434 (2), 226-232 (2013).
- Minn, I., et al. A red-shifted fluorescent substrate for aldehyde dehydrogenase. Nature Communications. 5, 3662(2014).
- Dollé, L., Boulter, L., Leclercq, I. A., van Grunsven, L. A. Next generation of ALDH substrates and their potential to study maturational lineage biology in stem and progenitor cells. American Journal of Physiology. Gastrointestinal and Liver Physiology. 308 (7), 573-578 (2015).
- Yagishita, A., et al. Development of highly selective fluorescent probe enabling flow-cytometric isolation of ALDH3A1-positive viable cells. Bioconjugate Chemistry. 28 (2), 302-306 (2017).
- Maity, S., et al. Thiophene bridged aldehydes (TBAs) image ALDH activity in cells: Via modulation of intramolecular charge transfer. Chemical Science. 8 (10), 7143-7151 (2017).
- Koenders, S. T. A., et al. Development of a retinal-based probe for the profiling of retinaldehyde dehydrogenases in cancer cells. ACS Central Science. 5 (12), 1965-1974 (2019).
- Oe, M., et al. Deep-red/near-infrared turn-on fluorescence probes for aldehyde dehydrogenase 1A1 in cancer stem cells. ACS Sensors. 6 (9), 3320-3329 (2021).
- Yagishita, A., Ueno, T., Tsuchihara, K., Urano, Y. Amino BODIPY-based blue fluorescent probes for aldehyde dehydrogenase 1-expressing cells. Bioconjugate Chemistry. 32 (2), 234-238 (2021).
- Okamoto, A., et al. Identification of breast cancer stem cells using a newly developed long-acting fluorescence probe, C5S-A, targeting ALDH1A1. Anticancer Research. 42 (3), 1199-1205 (2022).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados