Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Application d’AlDeSense pour stratifier les cellules cancéreuses de l’ovaire en fonction de l’activité de l’aldéhyde déshydrogénase 1A1
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Les méthodes de mesure de l’activité de l’ALDH1A1 dans les cellules vivantes sont essentielles dans la recherche sur le cancer en raison de son statut de biomarqueur de la souches. Dans cette étude, nous avons utilisé une sonde fluorogénique sélective isoforme pour déterminer les niveaux relatifs d’activité ALDH1A1 dans un panel de cinq lignées cellulaires de cancer de l’ovaire.
Résumé
La rechute après un traitement anticancéreux est souvent attribuée à la persistance d’une sous-population de cellules tumorales appelées cellules souches cancéreuses (CSC), qui se caractérisent par leur remarquable capacité d’initiation tumorale et d’auto-renouvellement. Selon l’origine de la tumeur (par exemple, les ovaires), le profil de biomarqueur de surface du CSC peut varier considérablement, ce qui rend l’identification de ces cellules par coloration immunohistochimique une entreprise difficile. Au contraire, l’aldéhyde déshydrogénase 1A1 (ALDH1A1) est apparue comme un excellent marqueur pour identifier les CSC, en raison de son profil d’expression conservé dans presque toutes les cellules progénitrices, y compris les CSC. L’isoforme ALDH1A1 appartient à une superfamille de 19 enzymes responsables de l’oxydation de divers aldéhydes endogènes et xénobiotiques en produits d’acide carboxylique correspondants. Chan et al. ont récemment développé AlDeSense, une sonde isoforme-sélective pour la détection de l’activité ALDH1A1, ainsi qu’un réactif de contrôle d’appariement non réactif (Ctrl-AlDeSense) pour tenir compte de la coloration hors cible. Cet outil sélectif des isoformes s’est déjà avéré être un outil chimique polyvalent grâce à la détection de l’activité de l’ALDH1A1 dans les cellules leucémiques myéloïdes K562, les mammosphères et les xénogreffes CSC dérivées du mélanome. Dans cet article, l’utilité de la sonde a été démontrée par des expériences supplémentaires de fluorimétrie, de microscopie confocale et de cytométrie en flux où l’activité relative de l’ALDH1A1 a été déterminée dans un panel de cinq lignées cellulaires de cancer de l’ovaire.
Introduction
Les cellules souches cancéreuses (CSC) sont une sous-population de cellules tumorales qui présentent des propriétés semblables à celles des cellules souches1. À l’instar de leurs homologues non cancéreux, les CSC possèdent une capacité extraordinaire de se renouveler et de proliférer. Avec d’autres mécanismes intégrés, tels que la régulation positive des transporteurs de cassettes liant l’ATP, les CSC sont souvent épargnés par les efforts initiaux de réduction tumorale chirurgicale, ainsi que par le traitement adjuvant ultérieur2. En raison de leur rôle essentiel dans la résistance au traitement3, la rechute4 et les métastases5, les CSC sont devenus une priorité dans la recherche sur le cancer. Bien qu’il existe une variété d’antigènes de surface cellulaire (p. ex. CD133) qui peuvent être utilisés pour identifier les CSC6, tirer parti de l’activité enzymatique des aldéhydes déshydrogénases (ALDH) trouvées dans le cytoplasme est apparu comme une alternative intéressante7. Les ALDH sont une superfamille de 19 enzymes responsables de catalyser l’oxydation des aldéhydes endogènes et xénobiotiques réactifs en produits d’acide carboxyliquecorrespondants 8.
En général, la détoxification des aldéhydes est cruciale pour protéger les cellules contre les événements de réticulation indésirables et le stress oxydatif qui peuvent endommager l’intégrité des cellules souches9. De plus, l’isoforme 1A1 contrôle le métabolisme de l’acide rétinoïque, qui à son tour influence la souche via la signalisation du rétinaldéhyde10. AlDeSense 11,12, une sonde de détection basée sur l’activité (ABS) à petites molécules pour détecter sélectivement l’activité ALDH1A1, a récemment été développée. Les conceptions ABS permettent la détection de l’analyte par un changement chimique plutôt que par un événement de liaison, ce qui permet une sélectivité élevée et une diminution des réponses hors cible13,14,15,16. Le principe de conception de la sonde fluorogénique isoforme-sélective repose sur un mécanisme de trempe par transfert d’électrons photoinduit par donneur (d-PeT)17, issu du groupe fonctionnel aldéhyde, qui sert à supprimer la signature fluorescente de la sonde18. Lors de la conversion médiée par ALDH1A1 en acide carboxylique, la relaxation radiative est débloquée pour produire un produit hautement fluorescent. Étant donné que la trempe d-PeT n’est jamais efficace à 100 %, la fluorescence résiduelle pouvant conduire à d’éventuels résultats faussement positifs a été prise en compte lors de l’établissement de ce test grâce au développement de Ctrl-AlDeSense, un réactif non réactif présentant des caractéristiques photophysiques correspondantes (p. ex., rendement quantique) et un schéma de coloration cytoplasmique identique dans les cellules. Lorsqu’il est utilisé en tandem, cet appariement unique peut distinguer de manière fiable les cellules à forte activité ALDH1A1 de celles qui présentent de faibles niveaux via la fluorimétrie, l’imagerie moléculaire et la cytométrie en flux. Plusieurs avantages clés sont associés à l’utilisation de colorants activables isoformels par rapport aux méthodes immunohistochimiques traditionnelles. Par exemple, on suppose que les CSC sont enfouis profondément dans une tumeur et sont donc plus accessibles à une petite molécule que les gros anticorps19. De plus, le produit fluorescent retourné ne modifie pas de manière covalente un composant cellulaire, ce qui signifie qu’il peut être facilement éliminé par des cycles de lavage pour laisser un CSC dans un état non modifié. Enfin, la réponse d’activation n’identifie que les cellules et les fonctions viables, tout comme le test MTT, en raison de sa dépendance au cofacteur NAD +.
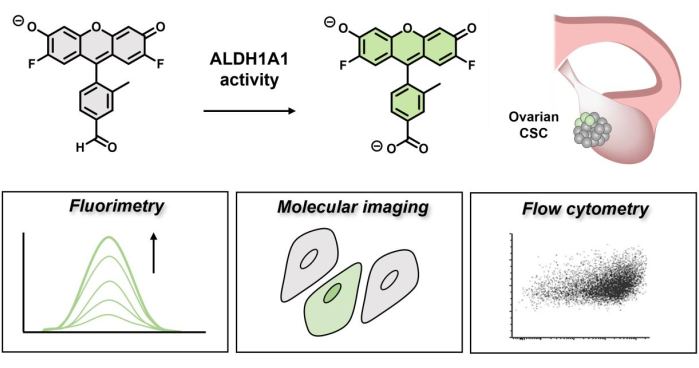
Figure 1 : Schéma montrant l’activation fluorescente d’AlDeSense. Le colorant isoforme-sélectif est activé par ALDH1A1 et peut être utilisé pour identifier une activité élevée d’ALDH1A1 dans les cellules cancéreuses de l’ovaire par fluorimétrie, imagerie moléculaire et cytométrie en flux. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Dans des travaux antérieurs, le test de sonde fluorogénique sélective isoforme a réussi à stratifier les cellules ALDH élevées (ALDH+) des cellules ALDH faibles (ALDH-) dans les cellules leucémiques chroniques humaines K562, les cellules cancéreuses du sein humain MDA-MB-231 et les cellules de mélanome murin B16F0. Ceci est important car, pour de nombreux types de cancer, une expression élevée de la protéine ALDH1A1 signifie un pronostic clinique moins sombre20. Cela suppose que des niveaux élevés d’ALDH1A1 sont indicatifs de CSC qui peuvent échapper au traitement, développer une résistance et se propager dans tout le corps. Cependant, dans le cas du cancer de l’ovaire, certaines études rapportent le résultat inverse (une expression élevée d’ALDH1A1 est liée à une amélioration de la survie des patientes)21,22,23,24. Bien que cela puisse sembler contradictoire à première vue, l’expression n’est pas nécessairement corrélée à l’activité enzymatique, qui peut être influencée par des changements dans le microenvironnement tumoral (par exemple, flux de pH, gradients d’oxygène), la disponibilité du cofacteur NAD+ ou des substrats d’aldéhyde, les niveaux d’acides carboxyliques (inhibition du produit) et les modifications post-traductionnelles qui peuvent modifier l’activité enzymatique25 . De plus, le cancer de l’ovaire est divisé en cinq types histologiques principaux (séreuse de haut grade, séreuse de bas grade, endométrioïde, à cellules claires et mucineuse), dont nous supposons qu’elles présenteront des niveaux variables d’activité ALDH1A126. Dans le but d’étudier l’activité de l’ALDH1A1 dans les tumeurs ovariennes, un test de sonde fluorogénique sélective isoforme a été utilisé pour identifier les populations ALDH1A1+ dans un panel de cinq lignées cellulaires de cancer de l’ovaire appartenant aux différents types histologiques mentionnés ci-dessus. Les lignées cellulaires testées dans cette étude comprennent des cellules BG-1, Caov-3, IGROV-1, OVCAR-3 et PEO4, couvrant les histotypes à cellules claires et séreuses. Ici, la polyvalence et la généralisabilité de la sonde ont été soulignées pour identifier les CSC pour les chercheurs qui cherchent à effectuer des études similaires sur d’autres lignées cellulaires cancéreuses immortalisées ainsi que sur des échantillons de patients. L’utilisation d’AlDeSense permettra de faire la lumière sur les voies biochimiques impliquées dans le maintien du SCC dans des microenvironnements tissulaires complexes et pourrait servir d’outil clinique pour déterminer le pronostic et mesurer l’agressivité du cancer.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Mesurer l’activité totale de l’ALDH1A1 dans les homogénats de cellules cancéreuses de l’ovaire par fluorimétrie
- Décongeler 1 × 106 cellules dans une fiole de culture cellulaire T25 dans 5 mL des milieux de culture cellulaire suivants :
- IGROV-1 et PEO4: Roswell Park Memorial Institute (RPMI) 1640 medium avec 10% de sérum bovin fœtal (FBS) et 1% de pénicilline / streptomycine (P / S).
- BG-1 et Caov-3 : Dulbecco’s Modified Eagle’s Medium (DMEM) avec 10% FBS et 1% P/S.
- OVCAR-3 : RPMI 1640 avec 20 % de FBS, 1 % P/S et 0,01 mg/mL d’insuline.
- Maintenir les cellules dans un incubateur à 37 °C et 5% de CO2 pendant deux à trois passages. Assurez-vous que les cellules ne dépassent pas 80% à 90% de confluence avant de passer.
- Trypsiniser les cellules dans 0,25% de trypsine pendant 10 min, les compter à l’aide d’un compteur de cellules automatisé et granuler 1 × 107 cellules par centrifugation (180 × g) à 25 °C pendant 5 min.
- Retirer le surnageant avec précaution par aspiration, laver la pastille en remettant les cellules en suspension dans 1 mL de 1x PBS et regranuler les cellules par centrifugation dans les mêmes conditions que celles décrites à l’étape 4.
- Resuspendre les cellules dans une solution de 1x inhibiteur de protéase dans 1x PBS. Utilisez 1 mL de cette solution par 2,5 × 106 cellules.
- Soniquez la suspension cellulaire sur glace pendant 2 min (impulsion de 1 s, amplitude de 40 %) avec une sonde homogénéisante de cellules.
- Fractions insolubles/membranaires des pastilles par centrifugation (3 200 × g) à 25 °C pendant 15 min. Retirez et conservez le surnageant, car c’est l’homogénat qui sera utilisé dans les étapes suivantes. Séparer les homogénats en trois cuvettes pour réaliser l’expérience en trois exemplaires.
- Ajouter la sonde à l’homogénat pour obtenir une concentration finale de sonde de 4 μM. Pipeter trois fois de haut en bas pour bien mélanger la solution.
- Mesurer le signal fluorescent immédiatement après l’ajout et après les temps d’incubation souhaités sur un fluorimètre. Le temps d’incubation peut être optimisé pour voir l’activation maximale du pli (1 h dans cette expérience). Le temps d’incubation doit être constant sur toutes les lignées cellulaires comparées.
- Réglez la longueur d’onde d’excitation sur 496 nm.
- Réglez l’émission sur 510-600 nm.
- Réglez la largeur de la fente sur 0,5 mm.
- Pipeter la solution dans une cuvette de quartz de 1 mL, placer dans le fluorimètre et frapper le balayage.
- Diviser l’intensité finale de fluorescence à 516 nm (longueur d’onde fluorescente maximale) par l’intensité à la même longueur d’onde à partir de la lecture initiale pour déterminer l’activation du pli du colorant isoforme-sélectif.
2. Utilisation de la microscopie à fluorescence pour imager des cellules à forte activité ALDH1A1
- Décongeler 1 × 106 cellules dans un ballon de culture cellulaire T25 dans 5 mL du milieu de culture cellulaire approprié.
- Maintenir les cellules dans un incubateur à 37 °C et 5% de CO2 pendant deux à trois passages. Assurez-vous que les cellules ne dépassent pas 80% à 90% de confluence avant de passer.
- La veille de l’imagerie confocale, plaquez 4 × 105 cellules dans une lame de chambre à 8 puits.
- Enduire le fond de chaque puits de poly-L-lysine (0,1 mg/mL, 100 μL par puits) pendant 10 minutes, puis en aspirant.
- Lavez chaque puits 3x en ajoutant de l’eau de culture cellulaire (100 μL par puits) et en aspirant.
- Trypsiniser les cellules dans 0,25% de trypsine pendant 10 min, les compter à l’aide d’un compteur de cellules automatisé et plaquer les cellules à 4 × 105 cellules par puits.
- Laissez les cellules se déposer et se fixer pendant la nuit (12-16 h).
- Aspirer le milieu de croissance et ajouter un milieu sans sérum (500 μL par puits), complété par 2 μM de la sonde ou de la sonde témoin.
- Incuber avec la sonde à température ambiante pendant 30 min et imager les cellules immédiatement.
- Allumez le microscope confocal et ajustez-le pour l’échantillon.
- Charger soigneusement l’échantillon au grossissement le plus bas; Trouvez les cellules pour assurer un bon positionnement.
- Assurez-vous que la lentille de l’objectif est à un grossissement de 10x et localisez les cellules.
- Pour cette expérience, le canal FITC (excitation: laser 488 nm; émission: 516-521 nm) et le canal T-PMT (lumière transmise) sont nécessaires. Localisez et concentrez-vous sur les cellules en utilisant T-PMT pour se concentrer sur le même plan z pendant toute l’expérience afin d’éliminer les biais.
- Ajustez la puissance laser et le gain FITC au réglage approprié, où le signal des échantillons Ctrl-AlDeSense AM est détectable au minimum, tout en voyant le signal dans les échantillons AlDeSense AM. Chaque paramètre peut être ajusté en faisant glisser la barre correspondante. Les paramètres devront peut-être être ajustés plusieurs fois pour identifier les paramètres corrects. Une fois optimisé, complétez le reste de l’expérience au sein de cette lignée cellulaire en utilisant des paramètres identiques.
- Prenez trois images par puits pour un total de trois puits par condition de traitement (neuf images au total). Concentrez-vous sur le plan approprié en utilisant T-PMT pour éviter les biais plutôt que d’utiliser le canal de fluorescence ou l’image fusionnée.
- Traitez les images pour déterminer le pourcentage de cellules ALDH1A1+.
- À l’aide d’un logiciel de traitement d’image, divisez le fichier czi en différents canaux.
- Comptez le nombre total de cellules et le nombre total de cellules fluorescentes.
- Pour déterminer le pourcentage de cellules ALDH1A1+, divisez le nombre de cellules fluorescentes par le nombre total de cellules dans chaque image. Il est impératif de compter de la même manière pour chaque image sans manipuler les images, car, par exemple, le réglage de la luminosité peut ajouter une variable confondante.
3. Application de la cytométrie de flux pour identifier les cellules ayant une activité ALDH1A1 élevée
- Décongeler 1 × 106 cellules dans un ballon de culture cellulaire T25 dans 5 mL du milieu de culture cellulaire approprié.
- Maintenir les cellules dans un incubateur à 37 °C et 5% de CO2 pendant deux à trois passages. Assurez-vous que les cellules ne dépassent pas 80% à 90% de confluence avant de passer.
- Trypsiniser, compter et granuler les cellules dans un tube à centrifuger de 15 mL par centrifugation (180 × g) à 25 °C pendant 5 min.
- Resuspendre les cellules dans 1 mL de solution de sonde/sonde témoin 2 μM dans du PBS. Balancez les cellules à température ambiante pendant 60 minutes pour vous assurer que l’exposition au colorant est uniforme.
- Après la période d’incubation, pelleter les cellules par centrifugation (180 × g) à 25 °C pendant 5 min. Remettez les cellules en suspension dans 0,5 mL de PBS. Faites passer les cellules à travers une passoire cellulaire (maille de nylon de 35 μm) pour éliminer les amas cellulaires qui peuvent obstruer le cytomètre en flux. Placez immédiatement les cellules sur de la glace.
- Allumez l’instrument et exécutez le protocole de démarrage.
- Vérifiez s’il y a du liquide de gaine et des déchets vides.
- Faites fonctionner les lignes avec 10% d’eau de Javel et d’eau pendant 5 minutes chacune.
- Exécutez des perles de contrôle de la qualité pour assurer le bon fonctionnement.
- Dans l’onglet Paramètres, sélectionnez FSC (diffusion directe), SSC (diffusion latérale) et FITC (isothiocyanate de fluorescéine) pour le filtre à fluorescence.
- Dessinez les graphiques suivants pour la viabilité, les singulettes et la fluorescence. Optimiser la puissance laser (spécifique à l’instrument de l’utilisateur) afin que les populations cellulaires soient dans les paramètres donnés pour une analyse plus approfondie.
- Nuage de points FSC-A versus SSC-A : la population cellulaire principale est proche du centre du graphique.
- Nuage de points FSC-A versus FSC-W : bande horizontale étroite indiquant des singulets (plutôt que des amas de cellules).
- Diagramme de dispersion FITC-A versus FSC-A : observez la distribution des cellules triées par la sonde d’activation fluorogénique sélective isoforme.
- Histogramme FITC-A : observez le déplacement de la population en fonction de la FITC pour déterminer le pourcentage de cellules ALDH1A1+.
- Pour optimiser la puissance laser FITC, exécutez un échantillon avec la sonde de sorte que la queue droite de la courbe de l’histogramme soit proche du signal FITC-A maximal. Ensuite, exécutez un échantillon avec la sonde de contrôle. Un déplacement de population doit être observable pour révéler la plage dynamique maximale. L’étape d’optimisation de la puissance laser peut devoir être répétée plusieurs fois, mais la puissance laser ne doit pas être modifiée entre les échantillons une fois qu’un réglage a été désigné pour une expérience.
- Exécutez chaque échantillon pour 10 000 comptages (en trois exemplaires).
- Répétez l’étape 3.12 pour chaque lignée cellulaire, car il y aura une variabilité dans l’absorption et l’activité ALDH1A1.
- Une fois le prélèvement de l’échantillon terminé, faites fonctionner les conduites avec 10 % d’eau de Javel et d’eau pendant 5 minutes chacune, puis lancez l’arrêt de l’instrument.
- Traitez les données à l’aide du logiciel de cytométrie en flux et portez la population cellulaire souhaitée. Réglez les portes de sorte que tous les événements tombent dans la porte ALDH1A1 ou ALDH1A1+. À l’aide de la sélection de porte rectangulaire, réglez la porte ALDH1A1- de sorte que >99,5 % des événements dans les échantillons de sonde de contrôle se produisent à l’intérieur de cette porte. Les cellules restantes seront considérées comme ALDH1A1+. Ces mêmes portes peuvent ensuite être appliquées à l’échantillon de sonde pour quantifier le nombre d’événements considérés ALDH1A1- et ALDH1A1+.
Access restricted. Please log in or start a trial to view this content.
Résultats
Activité totale de l’ALDH1A1 des homogénats de cellules cancéreuses de l’ovaire
Les activations moyennes de pli pour chaque lignée cellulaire obtenue à partir de ce test sont les suivantes: BG-1 (1,12 ± 0,01); IGROV-1 (1,30 ± 0,03); Caov-3 (1,72 ± 0,06); PEO4 (2,51 ± 0,29); et OVCAR-3 (10,25 ± 1,46) (figure 2).
Access restricted. Please log in or start a trial to view this content.
Discussion
La pansélectivité est une limitation majeure de nombreuses sondes ALDH ; Cependant, plusieurs exemples sélectifs d’isoformes ont récemment été rapportés 32,33,34,35,36,37,38,39,40,41.<...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Nous divulguons un brevet en instance (US20200199092A1) pour la technologie AlDeSense.
Remerciements
Ce travail a été soutenu par les National Institutes of Health (R35GM133581 à JC) et le Cancer Center at Illinois Graduate Scholarship (décerné à SG). JC remercie la Fondation Camille et Henry Dreyfus pour son soutien. Les auteurs remercient le Dr Thomas E. Bearrood pour sa contribution initiale à la préparation des stocks d’AlDeSense et d’AlDeSense AM. Nous remercions M. Oliver D. Pichardo Peguero et M. Joseph A. Forzano de leur aide dans la préparation de divers précurseurs synthétiques. Nous remercions le professeur Erik Nelson (Département de physiologie moléculaire et intégrative, UIUC) pour les cellules Caov-3, IGROV-1 et PEO4. Nous remercions le professeur Paul Hergenrother (Département de chimie, UIUC) pour les cellules BG-1. Nous remercions les installations principales de l’Institut Carl R. Woese de biologie génomique pour leur accès au microscope confocal Zeiss LSM 700 et au logiciel correspondant. Nous remercions l’installation de cytométrie en flux pour l’accès à l’analyseur BD LSR II CMtO. Nous remercions la Dre Sandra McMasters et l’installation des milieux cellulaires pour leur aide dans la préparation des milieux de culture cellulaire.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin, 0.1% EDTA in HBSS w/o Calcium, Magnesium and Sodium Bicarbonate | Corning | 25-050-CI | |
| 1x Phosphate Buffer Saline | Corning | 21-040-CMX12 | |
| AccuSpin Micro 17R | Fisher Scientific | 13-100-675 | |
| AlDeSense | Synthesized in-house | ||
| BG-1 | A gift provided by the Prof. Paul Hergenrother Lab, University of Illinois Urbana-Champaign | ||
| BioLite 25cm2 Flask | Thermo Fisher Scientific | 130189 | |
| Biosafety Cabinet 1300 series A2 | Thermo Fisher Scientific | ||
| Caov-3 | A gift provided by the Prof. Erik Nelson Lab, University of Illinois Urbana-Champaign | ||
| Cell homogenizer | Fisher Scientific | ||
| Centrifuge 5180R | Eppendorf | 22627040 | |
| Contrl-AlDeSense | Synthesized in-house | ||
| DMEM, 10% FBS, 1% P/S | Prepared by UIUC cell media facility | ||
| Falcon Round-Bottom Polystyrene Test Tubes with Cell Strainer Snap Cap, 5mL | Corning | 352003 | |
| FCS Express 6 | Provided by UIUC CMtO | ||
| FL microscope | EVOS | ||
| Fluorometer | Photon Technology International | ||
| Forma Series II Water-Jacketed CO2 Incubator | Fisher Scientific | 3110 | |
| IGROV-1 | A gift provided by the Prof. Erik Nelson Lab, University of Illinois Urbana-Champaign | ||
| ImageJ | NIH | ||
| Innova 42R Incubated Shaker | |||
| LSM 700 | Zeiss | ||
| LSR II | BD | ||
| Nunc Lab-Tek Chambered #1.0 Borosicilate Coverglass System | Thermo Fisher Scientific | 155383 | |
| OVCAR-3 | ATCC | HTB-161 | |
| PEO4 | A gift provided by the Prof. Erik Nelson Lab, University of Illinois Urbana-Champaign | ||
| Pierce Protease Inhibitor Tablets | Thermo Scientific | A32963 | |
| Poly-L-Lysine | Cultrex | 3438-100-01 | |
| Rocker | VWR | ||
| RPMI, 10% FBS, 1% P/S | Prepared by UIUC cell media facility | ||
| RPMI, 20% FBS, 1% P/S, 0.01 mg/mL Insulin | Prepared by UIUC cell media facility |
Références
- Bonnet, D., Dick, J. E. Human acute myeloid leukaemia is organised as a heirarchy that originates from a primitive haematopoetic cell. Nature Medicine. 3 (7), 730-737 (1997).
- Begicevic, R. R., Falasca, M. ABC transporters in cancer stem cells: Beyond chemoresistance. International Journal of Molecular Sciences. 18 (11), 2362(2017).
- Cojoc, M., Mäbert, K., Muders, M. H., Dubrovska, A. A role for cancer stem cells in therapy resistance: Cellular and molecular mechanisms. Seminars in Cancer Biology. 31, 16-27 (2015).
- Islam, F., Gopalan, V., Smith, R. A., Lam, A. K. Y. Translational potential of cancer stem cells: A review of the detection of cancer stem cells and their roles in cancer recurrence and cancer treatment. Experimental Cell Research. 335 (1), 135-147 (2015).
- Li, F., Tiede, B., Massagué, J., Kang, Y. Beyond tumorigenesis: Cancer stem cells in metastasis. Cell Research. 17 (1), 3-14 (2007).
- Kim, W. T., Ryu, C. J. Cancer stem cell surface markers on normal stem cells. BMB Reports. 50 (6), 285-298 (2017).
- Pors, K., Moreb, J. S. Aldehyde dehydrogenases in cancer: An opportunity for biomarker and drug development. Drug Discovery Today. 19 (12), 1953-1963 (2014).
- Jackson, B., et al. Update on the aldehyde dehydrogenase gene (ALDH) superfamily. Human Genomics. 5 (4), 283-303 (2011).
- Vasiliou, V., Pappa, A., Petersen, D. R. Role of aldehyde dehydrogenases in endogenous and xenobiotic metabolism. Chemico-Biological Interactions. 129 (1-2), 1-19 (2000).
- Tomita, H., Tanaka, K., Tanaka, T., Hara, A. Aldehyde dehydrogenase 1A1 in stem cells and cancer. Oncotarget. 7 (10), 11018-11032 (2016).
- Anorma, C., et al. Surveillance of cancer stem cell plasticity using an isoform-selective fluorescent probe for aldehyde dehydrogenase 1A1. ACS Central Science. 4 (8), 1045-1055 (2018).
- Bearrood, T. E., Aguirre-Figueroa, G., Chan, J. Rational design of a red fluorescent sensor for ALDH1A1 displaying enhanced cellular uptake and reactivity. Bioconjugate Chemistry. 31 (2), 224-228 (2020).
- Chan, J., Dodani, S. C., Chang, C. J. Reaction-based small-molecule fluorescent probes for chemoselective bioimaging. Nature Chemistry. 4 (12), 973-984 (2012).
- East, A. K., Lucero, M. Y., Chan, J. New directions of activity-based sensing for in vivo NIR imaging. Chemical Science. 12 (10), 3393-3405 (2021).
- Yadav, A. K., et al. Activity-based NIR bioluminescence probe enables discovery of diet-induced modulation of the tumor microenvironment via nitric oxide. ACS Central Science. 8 (4), 461-472 (2022).
- Yadav, A. K., et al. An activity-based sensing approach for the detection of cyclooxygenase-2 in Live Cells. Angewandte Chemie. 59 (8), 3307-3314 (2020).
- Ueno, T., et al. Rational principles for modulating fluorescence properties of fluorescein. Journal of the American Chemical Society. 126 (43), 14079-14085 (2004).
- Tanaka, F., Mase, N., Barbas 3rd, C. F. Design and use of fluorogenic aldehydes for monitoring the progress of aldehyde transformations. Journal of the American Chemical Society. 126 (12), 3692-3693 (2004).
- Thurber, G. M., Schmidt, M. M., Wittrup, K. D. Antibody tumor penetration: transport opposed by systemic and antigen-mediated clearance. Advanced Drug Delivery Reviews. 60 (12), 1421-1434 (2008).
- Marcato, P., Dean, C. A., Giacomantonio, C. A., Lee, P. W. K. Aldehyde dehydrogenase its role as a cancer stem cell marker comes down to the specific isoform. Cell Cycle. 10 (9), 1378-1384 (2011).
- Meng, E., et al. ALDH1A1 maintains ovarian cancer stem cell-like properties by altered regulation of cell cycle checkpoint and DNA repair network signaling. PLoS One. 9 (9), e107142(2014).
- Kaipio, K., et al. ALDH1A1-related stemness in high-grade serous ovarian cancer is a negative prognostic indicator but potentially targetable by EGFR/mTOR-PI3K/aurora kinase inhibitors. The Journal of Pathology. 250 (2), 159-169 (2020).
- Deng, S., et al. Distinct expression levels and patterns of stem cell marker, aldehyde dehydrogenase isoform 1 (ALDH1), in human epithelial cancers. PLoS One. 5 (4), e10277(2010).
- Chang, B., et al. ALDH1 expression correlates with favorable prognosis in ovarian cancers. Modern Pathology. 22 (6), 817-823 (2009).
- Gardner, S. H., Reinhardt, C. J., Chan, J. Advances in activity-based sensing probes for isoform-selective imaging of enzymatic activity. Angewandte Chemie. 60 (10), 5000-5009 (2021).
- Reid, B. M., Permuth, J. B., Sellers, T. A. Epidemiology of ovarian cancer: a review. Cancer Biology and Medicine. 14 (1), 9-32 (2017).
- Tulake, W., et al. Upregulation of stem cell markers ALDH1A1 and OCT4 as potential biomarkers for the early detection of cervical carcinoma. Oncology Letters. 16 (5), 5525-5534 (2018).
- Nwani, N. G., et al. A novel ALDH1A1 inhibitor targets cells with stem cell characteristics in ovarian cancer. Cancers. 11 (4), 502(2019).
- Roy, M., Connor, J., Al-Niaimi, A., Rose, S. L., Mahajan, A. Aldehyde dehydrogenase 1A1 (ALDH1A1) expression by immunohistochemistry is associated with chemo-refractoriness in patients with high-grade ovarian serous carcinoma. Human Pathology. 73, 1-6 (2018).
- Landen Jr, C. N., et al. Targeting aldehyde dehydrogenase cancer stem cells in ovarian cancer. Molecular Cancer Therapeutics. 9 (12), 3186-3199 (2010).
- Condello, S., et al. β-catenin-regulated ALDH1A1 is a target in ovarian cancer spheroids. Oncogene. 34 (18), 2297-2308 (2015).
- Storms, R. W., et al. Isolation of primitive human hematopoietic progenitors on the basis of aldehyde dehydrogenase activity. Proceedings of the National Academy of Sciences. 96 (16), 9118-9123 (1999).
- Duellman, S. J., et al. A bioluminescence assay for aldehyde dehydrogenase activity. Analytical Biochemistry. 434 (2), 226-232 (2013).
- Minn, I., et al. A red-shifted fluorescent substrate for aldehyde dehydrogenase. Nature Communications. 5, 3662(2014).
- Dollé, L., Boulter, L., Leclercq, I. A., van Grunsven, L. A. Next generation of ALDH substrates and their potential to study maturational lineage biology in stem and progenitor cells. American Journal of Physiology. Gastrointestinal and Liver Physiology. 308 (7), 573-578 (2015).
- Yagishita, A., et al. Development of highly selective fluorescent probe enabling flow-cytometric isolation of ALDH3A1-positive viable cells. Bioconjugate Chemistry. 28 (2), 302-306 (2017).
- Maity, S., et al. Thiophene bridged aldehydes (TBAs) image ALDH activity in cells: Via modulation of intramolecular charge transfer. Chemical Science. 8 (10), 7143-7151 (2017).
- Koenders, S. T. A., et al. Development of a retinal-based probe for the profiling of retinaldehyde dehydrogenases in cancer cells. ACS Central Science. 5 (12), 1965-1974 (2019).
- Oe, M., et al. Deep-red/near-infrared turn-on fluorescence probes for aldehyde dehydrogenase 1A1 in cancer stem cells. ACS Sensors. 6 (9), 3320-3329 (2021).
- Yagishita, A., Ueno, T., Tsuchihara, K., Urano, Y. Amino BODIPY-based blue fluorescent probes for aldehyde dehydrogenase 1-expressing cells. Bioconjugate Chemistry. 32 (2), 234-238 (2021).
- Okamoto, A., et al. Identification of breast cancer stem cells using a newly developed long-acting fluorescence probe, C5S-A, targeting ALDH1A1. Anticancer Research. 42 (3), 1199-1205 (2022).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon