Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Medición de los tiempos de relajación del tumor T2* después de la administración de nanopartículas de óxido de hierro
En este artículo
Resumen
Presentamos un protocolo estandarizado para la cuantificación de los tiempos de relajación T2* de los tumores utilizando software externo. Las imágenes de eco de gradiente de eco múltiple se adquieren y se introducen en el software para crear mapas T2* del tumor y medir los tiempos de relajación del tumor T2*.
Resumen
La relaxometría T2* es uno de los métodos establecidos para medir el efecto de las nanopartículas superparamagnéticas de óxido de hierro en los tejidos tumorales con imágenes por resonancia magnética (RM). Las nanopartículas de óxido de hierro acortan los tiempos de relajación T1, T2 y T2* de los tumores. Si bien el efecto T1 es variable en función del tamaño y la composición de las nanopartículas, los efectos T2 y T2* suelen ser predominantes, y las mediciones T2* son las más eficientes en el tiempo en un contexto clínico. Aquí, presentamos nuestro enfoque para medir los tiempos de relajación T2* del tumor, utilizando secuencias de eco de gradiente de eco múltiple, software externo y un protocolo estandarizado para crear un mapa T2* con software independiente del escáner. Esto facilita la comparación de datos de imágenes de diferentes escáneres clínicos, diferentes proveedores y trabajos de investigación coclínicos (es decir, datos de tumores T2* obtenidos en modelos de ratón y pacientes). Una vez instalado el software, el complemento T2 Fit Map debe instalarse desde el administrador de complementos. Este protocolo proporciona detalles de procedimiento paso a paso, desde la importación de las secuencias de eco de gradiente de eco múltiple en el software, hasta la creación de mapas T2* codificados por colores y la medición de los tiempos de relajación T2* del tumor. El protocolo se puede aplicar a tumores sólidos en cualquier parte del cuerpo y ha sido validado en base a datos de imágenes preclínicas y datos clínicos en pacientes. Esto podría facilitar las mediciones de T2* tumoral para ensayos clínicos multicéntricos y mejorar la estandarización y reproducibilidad de las mediciones de T2* tumoral en análisis de datos coclínicos y multicéntricos.
Introducción
La cuantificación no invasiva de los tiempos de relajación T2* tumoral en diversos tejidos del cuerpo con resonancia magnética (RM) está ampliamente establecida1. La justificación de este artículo es proporcionar un protocolo para la medición de los tiempos de relajación T2* del tumor que es independiente del software de escáner como Osirix2. Esto permitirá análisis uniformes de datos de imágenes de diferentes centros, diferentes escáneres y diferentes proveedores. De hecho, miles de usuarios podrían utilizar el mismo enfoque, aumentando así la estandarización de las mediciones de T2* tumoral. Las mediciones de T2* son utilizadas para diferentes propósitos por neurorradiólogos, expertos en imágenes cardíacas y expertos en imágenes abdominales, entre otros. Se han aplicado y optimizado secuencias de pulsos de resonancia magnética para medir los tiempos de relajación tisular T2* para la evaluación de hemorragias intracraneales3, contenido de hierro hepático 1,4 y contenido de hierro cardíaco 5,6, entre otros. Otros investigadores han utilizado mediciones T2* para generar estimaciones cuantitativas de acumulaciones de nanopartículas de óxido de hierro en tumores malignos 7,8. Sin embargo, muchos de estos enfoques anteriores utilizaban software institucional o software de escáner específico, que se limitaría a su uso en una institución específica o para procesar datos obtenidos en un escáner específico. Aquí, describimos un enfoque universalmente aplicable para generar mapas T2* tumorales y tiempos de relajación T2* tumorales basados en datos de resonancia magnética preclínicos o clínicos de cualquier escáner que pueda generar imágenes de eco de gradiente de eco múltiple. La secuencia de eco de gradiente requerida debe tener tiempos de primer eco muy cortos y un espaciado entre ecos cercanode 9,10. Las imágenes de eco de gradiente de eco múltiple se introducen en el software externo, se calculan los mapas T2* del tumor y se miden los tiempos de relajación del tumor T2*. El complemento T2 Fit Map en las curvas de decaimiento T2* de los modelos externos como un ajuste monoexponencial a S(t) = S o e-t/T2* 11 donde S(t) representa el valor de señaloproceso en un momento dado t; S 0 es el valor inicial de la señal o proceso en t =0; t denota tiempo; T2*, también conocido como el tiempo de relajación transversal aparente, caracteriza la tasa de decaimiento de la señal o proceso; y e es la base del logaritmo natural (aproximadamente igual a 2,71828). La ecuación describe una disminución exponencial, donde la señal o proceso disminuye con el tiempo en función de la tasa de decaimiento T2*. Cuanto mayor sea el valor de T2*, más lenta será la tasa de desintegración, y viceversa. El mismo software también se puede utilizar para introducir imágenes de eco de espín de eco múltiple y generar valores de T2 tumoral ajustando la curva de decaimiento T2 a S(t) = So e-t/T2. El ajuste de la curva se realizó utilizando software externo, sin incorporar un desplazamiento constante. Ambas curvas de decaimiento exhiben un comportamiento exponencial único, con T2* demostrando una duración más corta en comparación con T2.
En pacientes con hemosiderosis y hemocromatosis, la cuantificación del contenido de hierro hepático por biopsia tisular es el estándar de oro, mientras que la RM no invasiva es el punto de atención para establecer valores basales y monitorear los cambios a lo largo del tiempo de forma no invasiva12,13. Si bien la generación de mapas T2* para la cuantificación del hierro hepático está bien establecida4, no existe un protocolo estandarizado para medir los tiempos de relajación de T2* del tumor. Si bien los mapas T2* también pueden ser generados por el software del escáner, está limitado a un escáner y proveedor específicos. En el campo de la oncología, los estudios de imágenes en serie de un paciente determinado a menudo ocurren en diferentes escáneres, y los datos de resonancia magnética multicéntricos se adquieren en función de los estudios de imágenes de diferentes escáneres y diferentes proveedores. Además, la investigación de imágenes coclínicas se está implementando cada vez más y requiere la comparación de datos de resonancia magnética de pacientes y modelos de ratón que simulan su tumor. El propósito de este protocolo es proporcionar un protocolo para la medición de los tiempos de relajación T2* del tumor que sean independientes del software del escáner. Esto permitirá un análisis uniforme de los datos de imágenes de diferentes centros y diferentes escáneres. De hecho, miles de usuarios podrían utilizar el mismo enfoque, aumentando así la estandarización y la reproducibilidad de las mediciones de T2* tumoral. Nuestro protocolo utiliza software externo, que se puede descargar de Internet. Las imágenes de eco de gradiente de eco múltiple se introducen en el software y se ajustan a una fórmula de decaimiento monoexponencial para generar un mapa T2*, en el que se pueden medir los tiempos de relajación T2* del tumor utilizando regiones de interés (ROI) definidas por el operador5. Las nanopartículas de óxido de hierro pueden ser infundidas a diferentes dosis 14, En nuestro estudio, el paciente recibió una inyección de ferumoxitol (30 mg/ml) que contenía 510 mg de hierro elemental en un volumen de17 mL, a una dosis de 5 mg de hierro elemental por kg de peso corporal. Posteriormente se obtuvieron secuencias de eco de gradiente de eco múltiple15 utilizando parámetros de secuencia establecidos para la adquisición de datos.
Protocolo
Este protocolo se ha generado para un ensayo clínico prospectivo e investigación coclínica. El estudio cumplió con la Ley de Portabilidad y Responsabilidad del Seguro de Salud (HIPAA) y fue aprobado por la junta de revisión institucional (IRB) de la Universidad de Stanford. Todos los pacientes o su representante legalmente autorizado firmaron un consentimiento informado por escrito, y todos los niños entre 7 y 18 años de edad firmaron un formulario de consentimiento.
1. Instalación e inicio del plugin T2 Fit Map
- Inicie el software Osirix. Instale el complemento T2 Fit Map desde el administrador de complementos y reinicie el software.
- En la barra de menús, haga clic en el botón Plugins. Haga clic en el menú desplegable y seleccione Instalar un paquete de complementos (Figura 1).
- Una vez cargado el administrador de complementos, seleccione los complementos disponibles en el menú desplegable y luego T2 Fit Map (Figura 2).
- Haga clic en descargar e instalar. Cierre el administrador de complementos y reinicie el software.
- Cargue las imágenes de secuencia de eco de gradiente de eco múltiple como archivos DICOM en el software.
- Cambie la función del botón del ratón para dibujar una región de interés (ROI) (Figura 3).
- Con esta función de botón del ratón, defina una forma para el ROI requerido: seleccione Polígono ovalado o cerrado, o la forma deseada en el menú desplegable (Figura 4).
- Dibuje ROI en las imágenes requeridas con diferentes tiempos de eco (TE).
- Seleccione los ROI en todas las imágenes con diferentes TE para las que se requiere el mapa T2*.
- Haga clic en el botón Plugins , seleccione Filtros de imagen en el menú desplegable y, a continuación, seleccione T2 Fit Map.
- Haga clic en T2 Fit Map. Se abrirá un cuadro de diálogo; haga clic en Generar mapa (que se encuentra en la parte inferior del cuadro de diálogo) (Figura 5).
NOTA: Se genera una curva de ajuste con valores T2* mínimos, medios y máximos para los ROI seleccionados con varios TE (ms). El valor medio de T2* se calcula y se muestra debajo de la curva (Figura 6).
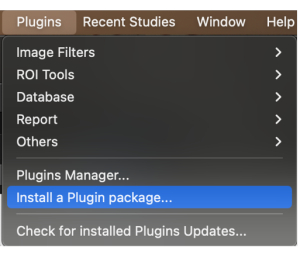
Figura 1: Seleccionando 'Instalar un paquete de complementos' en el menú desplegable. Haga clic aquí para ver una versión más grande de esta figura.
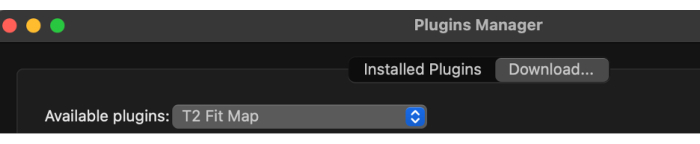
Figura 2: Seleccionando 'T2 Fit Map' de los plugins disponibles. Haga clic aquí para ver una versión más grande de esta figura.
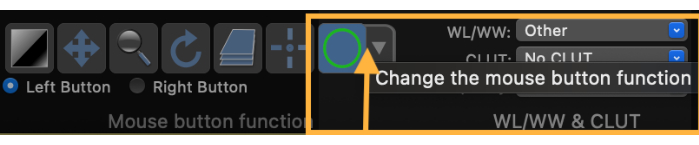
Figura 3: Captura de pantalla que muestra cómo cambiar la función del botón del ratón para dibujar una región de interés (ROI). Haga clic aquí para ver una versión más grande de esta figura.
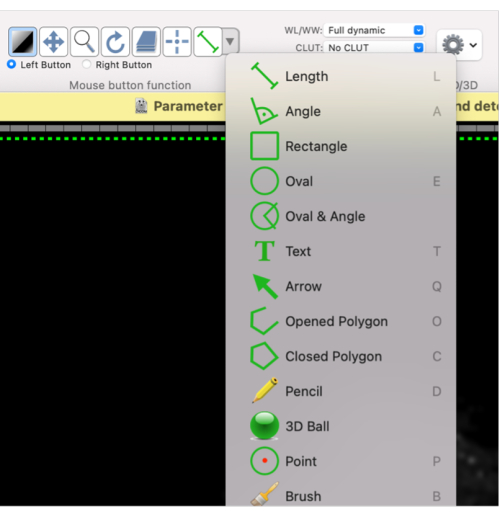
Figura 4: Captura de pantalla que muestra cómo seleccionar diferentes formas para el ROI. Haga clic aquí para ver una versión más grande de esta figura.
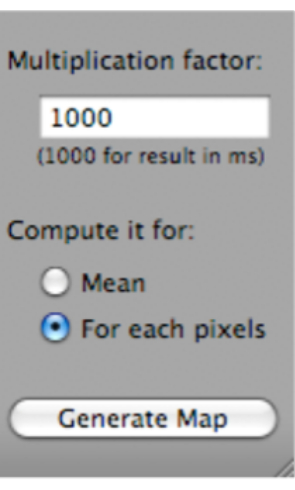
Figura 5: Captura de pantalla que muestra cómo seleccionar 'Generar mapa' después de seleccionar 'T2 Fit Map'. Haga clic aquí para ver una versión más grande de esta figura.
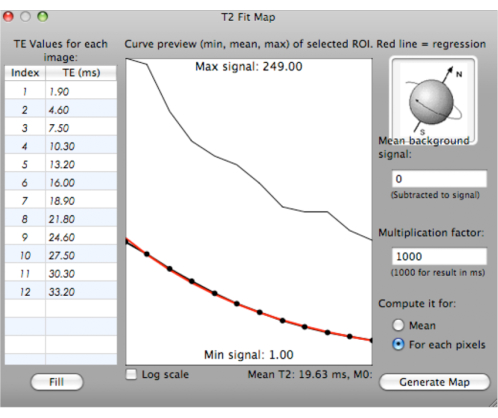
Figura 6: Captura de pantalla que muestra la generación de una curva de ajuste para valores T2*. Haga clic aquí para ver una versión más grande de esta figura.
2. Exclusión del ruido mediante la definición de mascarilla
NOTA: Para definir una máscara en el primer eco de los datos de T2map_MSME utilizados para calcular los mapas paramétricos, establezca un umbral de señal de primer eco inferior para el cálculo del valor T2* (el volumen del mapa T2* ahora excluirá los píxeles con señales demasiado bajas para un cálculo preciso en el primer eco multi-spin multi-echo (MSME). El umbral se puede aumentar para excluir más píxeles o disminuir para incluir más píxeles).
- Para definir el ROI de la máscara en una serie de imágenes fuera de los datos paramétricos, abra la serie deseada (por ejemplo, primer eco del T2map_MSME con TE = 15) y seleccione un sector.
- En la parte inferior del menú desplegable ROI, selecciona Grow Region....
- Seleccione el botón de opción Región de cultivo 3D... (Figura 7).
- En el menú desplegable Algoritmo, seleccione Umbral (límites inferior/superior).
- Establezca los umbrales inferior y superior en 0 y X% de la señal del músculo contralateral de la pantorrilla, respectivamente (p. ej., establezca el umbral en el valor que enmascarará la mayor cantidad de moteado en el ROI del tumor y dejará el tumor T2 (*) más evaluable).
- Establezca el nombre del ROI como desee.
- Haga clic en la imagen para colocar una semilla para el crecimiento del ROI.
- Haga clic en el botón Computar .
- En el menú ROI, seleccione Guardar todos los ROI de esta serie... (Figura 8).
NOTA: Ahora, el ROI que define el área a enmascarar de los mapas paramétricos se ha guardado y se puede aplicar a los datos paramétricos. - Abra el dataset Parametric en el visor 4D.
- En el menú ROI, seleccione Importar ROI(s)....
NOTA: El ROI de la máscara está ahora en la primera serie paramétrica. - Compruebe que los ROI están en el primer volumen 3D y no en 4D.
- Aplique máscara para asignar los datos. Para ello, en la parte inferior del menú desplegable ROI, seleccione Establecer valores de píxeles en. A continuación, seleccione aplicar a: ROI con el mismo nombre... (Figura 9).
- Marque la casilla Propagar a la serie 4D .
- Establezca píxeles que estén dentro de ROIs.
- Establecer en este nuevo valor: como 0.
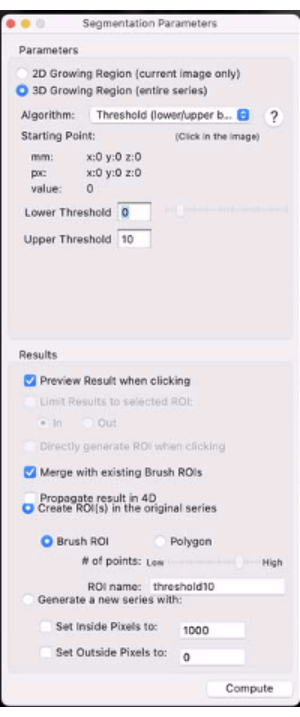
Figura 7: Captura de pantalla que muestra los parámetros de segmentación. Haga clic aquí para ver una versión más grande de esta figura.
Figura 8: Captura de pantalla que muestra cómo seleccionar "Guardar todos los ROI de esta serie...". Haga clic aquí para ver una versión más grande de esta figura.
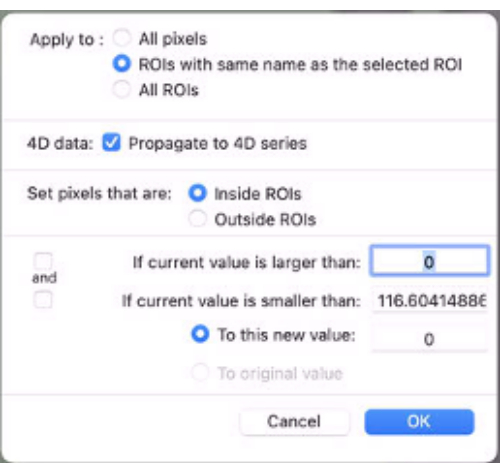
Figura 9: Captura de pantalla que muestra los valores que se ingresarán en "Establecer valores de píxeles en". Haga clic aquí para ver una versión más grande de esta figura.
Resultados

Figura 10: El mapa T2* con un ROI superpuesto en la lesión de osteosarcoma metastásico que muestra la media y el valor de desviación estándar T2*. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Nuestro protocolo nos permite medir los tiempos de relajación de T2* del tumor basados en secuencias de eco de gradiente de eco múltiple, un software externo y un complemento para crear mapas T2*. Los pasos críticos dentro del protocolo son la inclusión de la secuencia de gradiente de eco de eco múltiple con TE muy cortos en el protocolo de escaneo, y el ajuste monoexponencial de las imágenes de eco de gradiente de eco de eco múltiple utilizando software externo. Es importante organizar las imágenes de eco de gra...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado en parte por una subvención del Instituto Nacional del Cáncer, número de subvención U24CA264298. Agradecemos a Dawn Holley, Kim Halbert y Mehdi Khalighi del Centro de Servicios Metabólicos de PET / MRI por su ayuda con la adquisición de escaneos PET / MRI en el Centro de Investigación Lucas en Stanford. Agradecemos a los miembros del laboratorio de Daldrup-Link por sus valiosos aportes y discusiones sobre este proyecto.
Materiales
| Name | Company | Catalog Number | Comments |
| OsiriX | Pixmeo SARL | https://www.osirix-viewer.com/ | |
| 3T GE MR 750 | GE Healthcare, Chicago, IL | ||
| FERAHEME (ferumoxytol injection) | AMAG Pharmaceuticals, Inc. 1100 Winter Street Waltham, MA 02451 |
Referencias
- Garbowski, M. W., et al. Biopsy-based calibration of T2* magnetic resonance for estimation of liver iron concentration and comparison with R2 Ferriscan. Journal of Cardiovascular Magnetic Resonance. 16 (1), 40 (2014).
- . OsiriXDICOM Viewer Available from: https://www.osirix-viewer.com/ (2023)
- Linfante, I., Llinas, R. H., Caplan, L. R., Warach, S. MRI features of intracerebral hemorrhage within 2 hours from symptom onset. Stroke. 30 (11), 2263-2267 (1999).
- Labranche, R., et al. Liver iron quantification with MR imaging: a primer for radiologists. Radiographics. 38 (2), 392-412 (2018).
- Triadyaksa, P., Oudkerk, M., Sijens, P. E. Cardiac T2* mapping: Techniques and clinical applications. Journal of Magnetic Resonance Imaging. 52 (5), 1340-1351 (2020).
- Anderson, L. J., et al. Cardiovascular T2-star (T2*) magnetic resonance for the early diagnosis of myocardial iron overload. European Heart Journal. 22 (23), 2171-2179 (2001).
- Seo, M., et al. Estimation of T2* relaxation time of breast cancer: correlation with clinical, imaging and pathological features. Korean Journal of Radiology. 18 (1), 238-248 (2017).
- Serkova, N. J. Nanoparticle-based magnetic resonance imaging on tumor-associated macrophages and inflammation. Frontiers in Immunology. 8, 590 (2017).
- Chen, X., Qiu, B. A pilot study of short T2* measurements with ultrashort echo time imaging at 0.35 T. BioMedical Engineering OnLine. 17 (1), 70 (2018).
- Yi, J., Lee, Y. H., Song, H. -. T., Suh, J. -. S. Comparison of T2* between regular echo time and ultrashort echo time with 3D cones at 3 tesla for knee meniscus. Medicine. 97 (48), e13443 (2018).
- Weishaupt, D., et al. . How Does MRI Work?: An Introduction to the Physics and Function of Magnetic Resonance Imaging. , (2006).
- Wood, J. C. Guidelines for quantifying iron overload. Hematology. American Society of Hematology. 2014 (1), 210-215 (2014).
- Branisso, P. P. F., et al. Non-invasive methods for iron overload evaluation in dysmetabolic patients. Annals of Hepatology. 27 (4), 100707 (2022).
- Schaefer, B., Meindl, E., Wagner, S., Tilg, H., Zoller, H. Intravenous iron supplementation therapy. Molecular Aspects of Medicine. 75, 100862 (2020).
- Haacke, E. M., Mittal, S., Wu, Z., Neelavalli, J., Cheng, Y. -. C. N. Susceptibility-weighted imaging: technical aspects and clinical applications, part 1. AJNR: American Journal of Neuroradiology. 30 (1), 19-30 (2009).
- . T2 Mapping Slicer Extension Available from: https://github.com/gattia/Slicer-T2mapping (2021)
- 3D Slicer image computing platform. 3D Slicer Available from: https://slicer.org/ (2023)
- Messroghli, D. R., et al. An open-source software tool for the generation of relaxation time maps in magnetic resonance imaging. BMC Medical Imaging. 10, 16 (2010).
- . GNU Octave Available from: https://octave.org/ (2023)
- Chavhan, G. B., Babyn, P., Thomas, B., Shroff, M., Haacke, E. M. Principles, techniques, and applications of T2*-based MR imaging and its special applications. Radiographics. 29 (5), 1433-1449 (2009).
- Aghighi, M., et al. Magnetic resonance imaging of tumor associated macrophages: clinical translation. Clinical Cancer Research. 24 (17), 4110-4118 (2018).
- Trujillo-Alonso, V., et al. FDA-approved ferumoxytol displays anti-leukaemia efficacy against cells with low ferroportin levels. Nature Nanotechnology. 14 (6), 616-622 (2019).
- Ishiyama, K., et al. Tumor-liver contrast and subjective tumor conspicuity of respiratory-triggered T2-weighted fast spin-echo sequence compared with T2*-weighted gradient recalled-echo sequence for ferucarbotran-enhanced magnetic resonance imaging of hepatic malignant tumors. Journal of Magnetic Resonance Imaging. 27 (6), 1322-1326 (2008).
- Hirokawa, Y., et al. Hepatic lesions: improved image quality and detection with the periodically rotated overlapping parallel lines with enhanced reconstruction technique-evaluation of SPIO-enhanced T2-weighted MR images. Radiology. 251 (2), 388-397 (2009).
- Tonan, T., et al. Evaluation of small (≤2cm) dysplastic nodules and well-differentiated hepatocellular carcinomas with ferucarbotran-enhanced MRI in a 1.0-T MRI unit: Utility of T2*-weighted gradient echo sequences with an intermediate-echo time. European Journal of Radiology. 64 (1), 133-139 (2007).
- Rief, M., et al. Detection of focal liver lesions in unenhanced and ferucarbotran-enhanced magnetic resonance imaging: a comparison of T2-weighted breath-hold and respiratory-triggered sequences. Magnetic Resonance Imaging. 27 (9), 1223-1229 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados