Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Messung der Tumor-T2*-Relaxationszeiten nach Verabreichung von Eisenoxid-Nanopartikeln
In diesem Artikel
Zusammenfassung
Wir stellen ein standardisiertes Protokoll zur Quantifizierung der T2*-Relaxationszeiten von Tumoren mit Hilfe externer Software vor. Multi-Echo-Gradienten-Echobilder werden aufgenommen und in die Software eingespeist, um Tumor-T2*-Karten zu erstellen und Tumor-T2*-Relaxationszeiten zu messen.
Zusammenfassung
Die T2*-Relaxometrie ist eine der etablierten Methoden, um die Wirkung von superparamagnetischen Eisenoxid-Nanopartikeln auf Tumorgewebe mittels Magnetresonanztomographie (MRT) zu messen. Eisenoxid-Nanopartikel verkürzen die T1-, T2- und T2*-Relaxationszeiten von Tumoren. Während der T1-Effekt je nach Größe und Zusammensetzung der Nanopartikel variiert, sind die T2- und T2*-Effekte in der Regel vorherrschend, und T2*-Messungen sind im klinischen Kontext am zeiteffizientesten. Hier stellen wir unseren Ansatz zur Messung der Tumor-T2*-Relaxationszeiten vor, indem wir Multi-Echo-Gradientenecho-Sequenzen, externe Software und ein standardisiertes Protokoll zur Erstellung einer T2*-Karte mit scannerunabhängiger Software verwenden. Dies erleichtert den Vergleich von Bildgebungsdaten verschiedener klinischer Scanner, verschiedener Anbieter und koklinischer Forschungsarbeiten (d. h. Tumor-T2*-Daten, die in Mausmodellen und Patienten gewonnen wurden). Sobald die Software installiert ist, muss das T2 Fit Map-Plugin über den Plugin-Manager installiert werden. Dieses Protokoll bietet Schritt-für-Schritt-Verfahrensdetails, vom Import der Multi-Echo-Gradientenechosequenzen in die Software über die Erstellung farbcodierter T2*-Karten bis hin zur Messung der T2*-Relaxationszeiten des Tumors. Das Protokoll kann auf solide Tumore in jedem Körperteil angewendet werden und wurde auf der Grundlage präklinischer Bildgebungsdaten und klinischer Daten bei Patienten validiert. Dies könnte Tumor-T2*-Messungen für multizentrische klinische Studien erleichtern und die Standardisierung und Reproduzierbarkeit von Tumor-T2*-Messungen in koklinischen und multizentrischen Datenanalysen verbessern.
Einleitung
Die nichtinvasive Quantifizierung von Tumor-T2*-Relaxationszeiten in verschiedenen Geweben des Körpers mittels Magnetresonanztomographie (MRT) ist weithin etabliert1. Der Grundgedanke für diesen Artikel besteht darin, ein Protokoll für die Messung der Tumor-T2*-Relaxationszeiten bereitzustellen, das unabhängig von Scannersoftware wie Osirix2 ist. Dies ermöglicht eine einheitliche Analyse von Bilddaten aus verschiedenen Zentren, verschiedenen Scannern und verschiedenen Anbietern. In der Tat könnten potenziell Tausende von Anwendern den gleichen Ansatz verwenden und damit die Standardisierung von Tumor-T2*-Messungen erhöhen. T2*-Messungen werden unter anderem von Neuroradiologen, Experten für Herzbildgebung und Bildgebung des Abdomens für verschiedene Zwecke verwendet. MRT-Pulssequenzen zur Messung der Gewebe-T2*-Relaxationszeiten wurden u.a. für die Beurteilung von intrakraniellen Blutungen3, hepatischem Eisengehalt 1,4 und kardialem Eisengehalt 5,6 angewendet und optimiert. Andere Forscher haben T2*-Messungen verwendet, um quantitative Schätzungen der Anhäufung von Eisenoxid-Nanopartikeln in bösartigen Tumoren zu erstellen 7,8. Viele dieser früheren Ansätze verwendeten jedoch institutionelle Software oder spezifische Scanner-Software, die auf den Einsatz in einer bestimmten Institution oder auf die Verarbeitung von Daten beschränkt war, die mit einem bestimmten Scanner gewonnen wurden. In dieser Arbeit beschreiben wir einen universell anwendbaren Ansatz zur Erstellung von Tumor-T2*-Karten und Tumor-T2*-Relaxationszeiten auf der Grundlage präklinischer oder klinischer MRT-Daten von jedem Scanner, der Multi-Echo-Gradientenechobilder erzeugen kann. Die erforderliche Gradientenechosequenz sollte sehr kurze erste Echozeiten und einen engen Abstand zwischen den Echos 9,10 aufweisen. Die Multi-Echo-Gradientenechobilder werden dann in die externe Software eingespeist, Tumor-T2*-Karten werden berechnet und Tumor-T2*-Relaxationszeiten gemessen. Das T2 Fit Map-Plugin in den T2*-Abklingkurven der externen Modelle als monoexponentielle Anpassung an S(t) = So e-t/T2* 11, wobei S(t) den Signal- oder Prozesswert zu einem bestimmten Zeitpunkt t darstellt; S 0 ist der Anfangswert des Signals oder Prozesses bei t =0; t steht für Zeit; T2*, auch als scheinbare transversale Relaxationszeit bekannt, charakterisiert die Abklingrate des Signals oder Prozesses; und e ist die Basis des natürlichen Logarithmus (ungefähr gleich 2,71828). Die Gleichung beschreibt einen exponentiellen Zerfall, bei dem das Signal oder der Prozess im Laufe der Zeit in Abhängigkeit von der Abklingrate T2* abnimmt. Je größer der Wert von T2* ist, desto langsamer ist die Zerfallsrate und umgekehrt. Dieselbe Software kann auch verwendet werden, um Multi-Echo-Spin-Echo-Bilder einzugeben und Tumor-T2-Werte zu erzeugen, indem die T2-Zerfallskurve an S(t) = So e-t/T2 angepasst wird. Die Kurvenanpassung wurde mit einer externen Software durchgeführt, ohne einen konstanten Versatz zu berücksichtigen. Beide Zerfallskurven zeigen ein einzelnes exponentielles Verhalten, wobei T2* im Vergleich zu T2 eine kürzere Dauer aufweist.
Bei Patienten mit Hämosiderose und Hämochromatose ist die Quantifizierung des Eisengehalts in der Leber durch Gewebebiopsie der Goldstandard, während die nichtinvasive MRT-Bildgebung der Point-of-Care ist, um Ausgangswerte zu ermitteln und Veränderungen im Zeitverlauf nichtinvasiv zu überwachen12,13. Während die Erstellung von T2*-Karten für die Quantifizierung von Lebereisen gut etabliert ist4, gibt es kein standardisiertes Protokoll zur Messung der T2*-Relaxationszeiten von Tumoren. T2*-Karten können zwar auch von Scanner-Software generiert werden, sind jedoch auf einen bestimmten Scanner und Anbieter beschränkt. Im Bereich der Onkologie werden serielle Bildgebungsstudien eines bestimmten Patienten häufig auf verschiedenen Scannern durchgeführt, und multizentrische MRT-Daten werden auf der Grundlage von Bildgebungsstudien von verschiedenen Scannern und verschiedenen Anbietern erfasst. Darüber hinaus wird zunehmend koklinische Bildgebungsforschung betrieben, die den Vergleich von MRT-Daten von Patienten und Mausmodellen, die ihren Tumor simulieren, erfordert. Der Zweck dieses Protokolls ist es, ein Protokoll für die Messung der Tumor-T2*-Relaxationszeiten bereitzustellen, das unabhängig von der Scanner-Software ist. Dies ermöglicht eine einheitliche Analyse von Bilddaten aus verschiedenen Zentren und verschiedenen Scannern. In der Tat könnten potenziell Tausende von Anwendern den gleichen Ansatz verwenden und dadurch die Standardisierung und Reproduzierbarkeit von Tumor-T2*-Messungen erhöhen. Unser Protokoll verwendet externe Software, die aus dem Internet heruntergeladen werden kann. Multi-Echo-Gradientenechobilder werden in die Software eingespeist und an eine Formel für den monoexponentiellen Zerfall angepasst, um eine T2*-Karte zu erstellen, auf der die T2*-Relaxationszeiten des Tumors mit Hilfe von vom Bediener definierten Regions of Interest (ROIs) gemessen werden können5. Eisenoxid-Nanopartikel können in verschiedenen Dosen infundiert werden14, In unserer Studie erhielt der Patient eine Ferumoxytol-Injektion (30 mg/ml) mit 510 mg elementarem Eisen in einem Volumen von 17 ml in einer Dosierung von 5 mg elementarem Eisen pro kg Körpergewicht. Anschließend wurden Multi-Echo-Gradientenechosequenzen15 unter Verwendung festgelegter Sequenzparameter für die Datenerfassung erhalten.
Access restricted. Please log in or start a trial to view this content.
Protokoll
Dieses Protokoll wurde für eine prospektive klinische Studie und koklinische Forschung erstellt. Die Studie entsprach dem Health Insurance Portability and Accountability Act (HIPAA) und wurde vom Institutional Review Board (IRB) der Stanford University genehmigt. Alle Patienten oder ihr gesetzlicher Vertreter unterschrieben eine schriftliche Einverständniserklärung, und alle Kinder zwischen 7 und 18 Jahren unterschrieben eine Einverständniserklärung.
1. Installieren und Starten des T2 Fit Map Plugins
- Starten Sie die Osirix-Software. Installieren Sie das T2 Fit Map-Plugin über den Plugin-Manager und starten Sie die Software neu.
- Klicken Sie in der Menüleiste auf die Schaltfläche Plugins. Klicken Sie auf das Dropdown-Menü und wählen Sie Plug-in-Paket installieren (Abbildung 1).
- Sobald der Plugin-Manager geladen ist, wählen Sie die verfügbaren Plugins aus dem Dropdown-Menü und dann T2 Fit Map aus (Abbildung 2).
- Klicken Sie auf Herunterladen &; Installieren. Schließen Sie den Plugin-Manager und starten Sie die Software neu.
- Laden Sie die Multi-Echo-Gradienten-Echo-Sequenzbilder als DICOM-Dateien in die Software.
- Ändern Sie die Maustastenfunktion, um eine Region of Interest (ROI) zu zeichnen (Abbildung 3).
- Definieren Sie mit dieser Maustastenfunktion eine Form für den gewünschten ROI: Wählen Sie Oval oder Geschlossenes Polygon oder die gewünschte Form aus dem Dropdown-Menü (Abbildung 4).
- Zeichnen Sie ROIs in den gewünschten Bildern mit unterschiedlichen Echozeiten (TEs).
- Wählen Sie die ROIs in allen Bildern mit unterschiedlichen TEs aus, für die die T2*-Karte erforderlich ist.
- Klicken Sie auf die Schaltfläche Plugins , wählen Sie Bildfilter aus dem Dropdown-Menü aus, und wählen Sie dann T2 Fit Map aus.
- Klicke auf T2 Fit Map. Es öffnet sich ein Dialogfenster. Klicken Sie auf Karte generieren (am unteren Rand des Dialogfelds) (Abbildung 5).
HINWEIS: Es wird eine Anpassungskurve mit minimalen, mittleren und maximalen T2*-Werten für die ausgewählten ROIs mit verschiedenen TEs (ms) generiert. Der mittlere T2*-Wert wird berechnet und unterhalb der Kurve angezeigt (Abbildung 6).
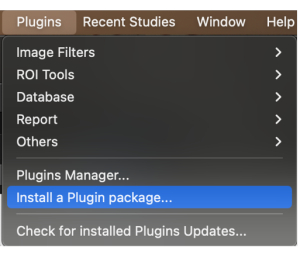
Abbildung 1: Auswahl von "Plugin-Paket installieren" aus dem Dropdown-Menü. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
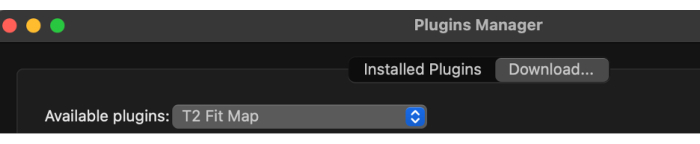
Abbildung 2: Auswahl von 'T2 Fit Map' aus den verfügbaren Plugins. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
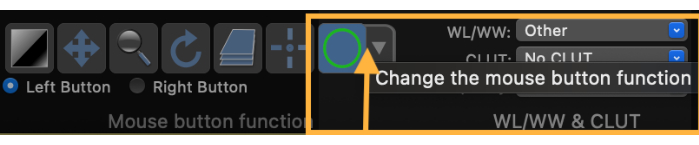
Abbildung 3: Screenshot, der zeigt, wie die Maustastenfunktion geändert wird, um eine Region of Interest (ROI) zu zeichnen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
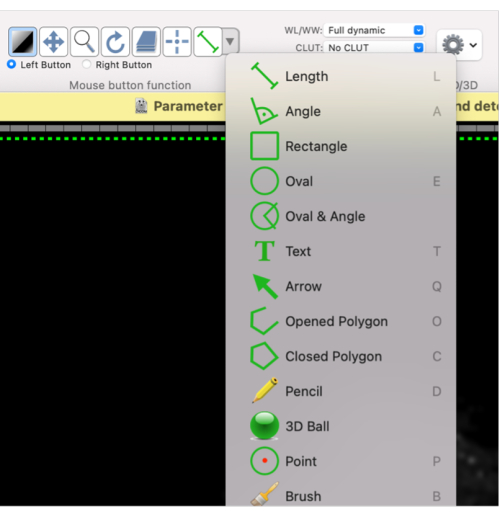
Abbildung 4: Screenshot, der zeigt, wie verschiedene Formen für den ROI ausgewählt werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
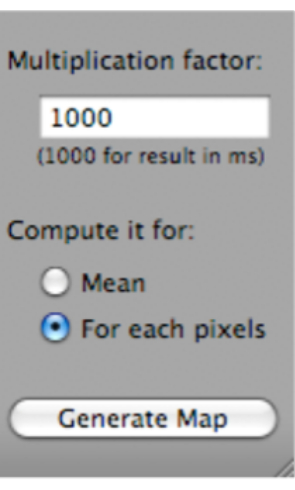
Abbildung 5: Screenshot, der zeigt, wie man "Karte generieren" auswählt, nachdem man "T2 Fit Map" ausgewählt hat. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
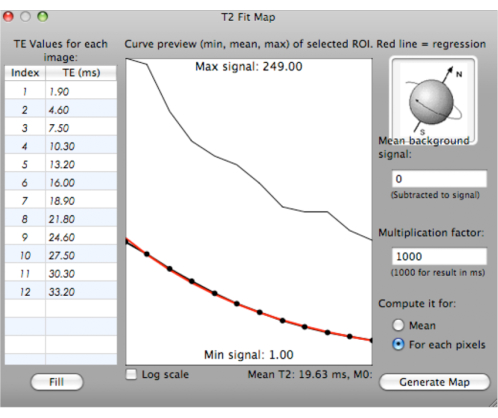
Abbildung 6: Screenshot, der die Erzeugung einer Anpassungskurve für T2*-Werte zeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
2. Ausschluss von Lärm durch die Definition der Maske
HINWEIS: Um eine Maske für das erste Echo der T2map_MSME Daten zu definieren, die zur Berechnung der parametrischen Karten verwendet werden, legen Sie einen niedrigeren Schwellenwert für das erste Echosignal für die Berechnung des T2*-Werts fest (das T2*-Map-Volumen schließt jetzt die Pixel mit Signalen aus, deren Signale für eine genaue Berechnung beim ersten Multi-Spin-Multi-Echo (MSME) zu niedrig sind. Der Schwellenwert kann erhöht werden, um mehr Pixel auszuschließen, oder verringert werden, um mehr Pixel einzuschließen).
- Um den Masken-ROI für eine Bildserie außerhalb der parametrischen Daten zu definieren, öffnen Sie die gewünschte Reihe (z. B. erstes Echo der T2map_MSME mit TE = 15) und wählen Sie eine Schicht aus.
- Wählen Sie unten im Dropdown-Menü "ROI" die Option " Region anbauen" aus.
- Wählen Sie das Optionsfeld 3D-Anbauregion... aus (Abbildung 7).
- Wählen Sie im Dropdown-Menü Algorithmus die Option Schwellenwert (untere/obere Grenze) aus.
- Legen Sie den unteren und oberen Schwellenwert auf 0 bzw. X % des kontralateralen Wadenmuskelsignals fest (z. B. setzen Sie den Schwellenwert auf den Wert, der die meisten Speckles im Tumor-ROI ausblendet und die auswertbarsten Tumor-T2(*)s verlässt).
- Legen Sie den ROI-Namen wie gewünscht fest.
- Klicken Sie auf das Bild, um einen Samen für den ROI-Anbau zu platzieren.
- Klicken Sie auf die Schaltfläche Berechnen .
- Wählen Sie im Menü "ROI" die Option " Alle ROIs dieser Serie speichern" aus (Abbildung 8).
ANMERKUNG: Jetzt wurde der ROI, der den Bereich definiert, der aus den parametrischen Karten ausgeblendet werden soll, gespeichert und kann auf die parametrischen Daten angewendet werden. - Öffnen Sie das parametrische Dataset im 4D viewer.
- Wählen Sie im Menü ROI die Option ROI(s) importieren....
ANMERKUNG: Der ROI der Maske befindet sich jetzt in der ersten parametrischen Reihe. - Überprüfen Sie, ob die ROIs im ersten 3D-Volume und nicht im 4D-Volume liegen.
- Wenden Sie eine Maske an, um die Daten zuzuordnen. Wählen Sie dazu unten im Dropdown-Menü "ROI" die Option " Pixelwerte festlegen auf" aus. Wählen Sie dann " Anwenden auf: ROIs mit demselben Namen" aus (Abbildung 9).
- Aktivieren Sie das Kontrollkästchen An 4D Serie weitergeben .
- Legen Sie Pixel fest, die sich innerhalb von ROIs befinden.
- Legen Sie Auf diesen neuen Wert: als 0 fest.
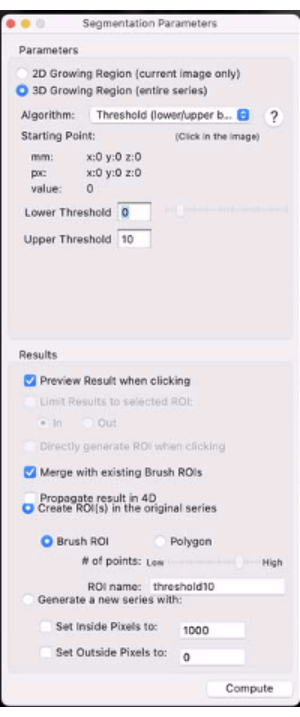
Abbildung 7: Screenshot mit den Segmentierungsparametern. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Abbildung 8: Screenshot, der zeigt, wie Sie "Alle ROIs dieser Serie speichern..." auswählen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
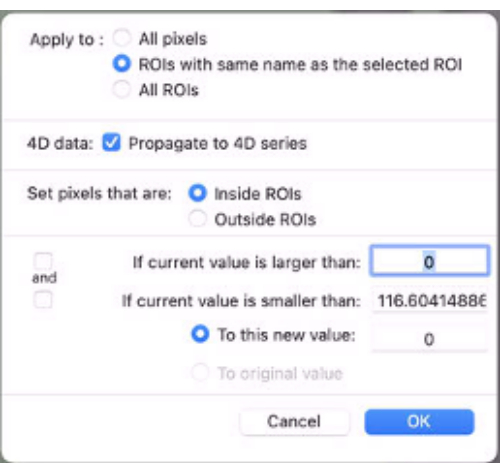
Abbildung 9: Screenshot mit den Werten, die in "Pixelwerte festlegen auf" eingegeben werden sollen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse

Abbildung 10: Die T2*-Karte mit einem ROI, der der metastasierten Osteosarkomläsion überlagert ist und den Mittelwert und die Standardabweichung T2* zeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Access restricted. Please log in or start a trial to view this content.
Diskussion
Unser Protokoll ermöglicht es uns, die T2*-Relaxationszeiten von Tumoren auf der Grundlage von Multi-Echo-Gradienten-Echo-Sequenzen, einer externen Software und einem Plugin zur Erstellung von T2*-Karten zu messen. Die entscheidenden Schritte innerhalb des Protokolls sind die Aufnahme der Multi-Echo-Gradient-Echo-Sequenz mit sehr kurzen TEs in das Scanning-Protokoll und die monoexponentielle Anpassung der Multi-Echo-Gradient-Echo-Bilder mit externer Software. Es ist wichtig, die eingegebenen Multi-Echo-Gradienten-Echo-B...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Arbeit wurde zum Teil durch ein Stipendium des National Cancer Institute, Grant Nummer U24CA264298, unterstützt. Wir danken Dawn Holley, Kim Halbert und Mehdi Khalighi vom PET/MRT Metabolic Service Center für ihre Unterstützung bei der Beschaffung von PET/MRT-Scans am Lucas Research Center in Stanford. Wir danken den Mitgliedern des Daldrup-Link-Labors für wertvolle Anregungen und Diskussionen zu diesem Projekt.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| OsiriX | Pixmeo SARL | https://www.osirix-viewer.com/ | |
| 3T GE MR 750 | GE Healthcare, Chicago, IL | ||
| FERAHEME (ferumoxytol injection) | AMAG Pharmaceuticals, Inc. 1100 Winter Street Waltham, MA 02451 |
Referenzen
- Garbowski, M. W., et al. Biopsy-based calibration of T2* magnetic resonance for estimation of liver iron concentration and comparison with R2 Ferriscan. Journal of Cardiovascular Magnetic Resonance. 16 (1), 40(2014).
- OsiriXDICOM Viewer. , Available from: https://www.osirix-viewer.com/ (2023).
- Linfante, I., Llinas, R. H., Caplan, L. R., Warach, S. MRI features of intracerebral hemorrhage within 2 hours from symptom onset. Stroke. 30 (11), 2263-2267 (1999).
- Labranche, R., et al. Liver iron quantification with MR imaging: a primer for radiologists. Radiographics. 38 (2), 392-412 (2018).
- Triadyaksa, P., Oudkerk, M., Sijens, P. E. Cardiac T2* mapping: Techniques and clinical applications. Journal of Magnetic Resonance Imaging. 52 (5), 1340-1351 (2020).
- Anderson, L. J., et al. Cardiovascular T2-star (T2*) magnetic resonance for the early diagnosis of myocardial iron overload. European Heart Journal. 22 (23), 2171-2179 (2001).
- Seo, M., et al. Estimation of T2* relaxation time of breast cancer: correlation with clinical, imaging and pathological features. Korean Journal of Radiology. 18 (1), 238-248 (2017).
- Serkova, N. J. Nanoparticle-based magnetic resonance imaging on tumor-associated macrophages and inflammation. Frontiers in Immunology. 8, 590(2017).
- Chen, X., Qiu, B. A pilot study of short T2* measurements with ultrashort echo time imaging at 0.35 T. BioMedical Engineering OnLine. 17 (1), 70(2018).
- Yi, J., Lee, Y. H., Song, H. -T., Suh, J. -S. Comparison of T2* between regular echo time and ultrashort echo time with 3D cones at 3 tesla for knee meniscus. Medicine. 97 (48), e13443(2018).
- Weishaupt, D., et al. How Does MRI Work?: An Introduction to the Physics and Function of Magnetic Resonance Imaging. , Springer. Berlin, Heidelberg. (2006).
- Wood, J. C. Guidelines for quantifying iron overload. Hematology. American Society of Hematology. 2014 (1), 210-215 (2014).
- Branisso, P. P. F., et al. Non-invasive methods for iron overload evaluation in dysmetabolic patients. Annals of Hepatology. 27 (4), 100707(2022).
- Schaefer, B., Meindl, E., Wagner, S., Tilg, H., Zoller, H. Intravenous iron supplementation therapy. Molecular Aspects of Medicine. 75, 100862(2020).
- Haacke, E. M., Mittal, S., Wu, Z., Neelavalli, J., Cheng, Y. -C. N. Susceptibility-weighted imaging: technical aspects and clinical applications, part 1. AJNR: American Journal of Neuroradiology. 30 (1), 19-30 (2009).
- T2 Mapping Slicer Extension. , Available from: https://github.com/gattia/Slicer-T2mapping (2021).
- 3D Slicer image computing platform. 3D Slicer. , Available from: https://slicer.org/ (2023).
- Messroghli, D. R., et al. An open-source software tool for the generation of relaxation time maps in magnetic resonance imaging. BMC Medical Imaging. 10, 16(2010).
- GNU Octave. , Available from: https://octave.org/ (2023).
- Chavhan, G. B., Babyn, P., Thomas, B., Shroff, M., Haacke, E. M. Principles, techniques, and applications of T2*-based MR imaging and its special applications. Radiographics. 29 (5), 1433-1449 (2009).
- Aghighi, M., et al. Magnetic resonance imaging of tumor associated macrophages: clinical translation. Clinical Cancer Research. 24 (17), 4110-4118 (2018).
- Trujillo-Alonso, V., et al. FDA-approved ferumoxytol displays anti-leukaemia efficacy against cells with low ferroportin levels. Nature Nanotechnology. 14 (6), 616-622 (2019).
- Ishiyama, K., et al. Tumor-liver contrast and subjective tumor conspicuity of respiratory-triggered T2-weighted fast spin-echo sequence compared with T2*-weighted gradient recalled-echo sequence for ferucarbotran-enhanced magnetic resonance imaging of hepatic malignant tumors. Journal of Magnetic Resonance Imaging. 27 (6), 1322-1326 (2008).
- Hirokawa, Y., et al. Hepatic lesions: improved image quality and detection with the periodically rotated overlapping parallel lines with enhanced reconstruction technique-evaluation of SPIO-enhanced T2-weighted MR images. Radiology. 251 (2), 388-397 (2009).
- Tonan, T., et al. Evaluation of small (≤2cm) dysplastic nodules and well-differentiated hepatocellular carcinomas with ferucarbotran-enhanced MRI in a 1.0-T MRI unit: Utility of T2*-weighted gradient echo sequences with an intermediate-echo time. European Journal of Radiology. 64 (1), 133-139 (2007).
- Rief, M., et al. Detection of focal liver lesions in unenhanced and ferucarbotran-enhanced magnetic resonance imaging: a comparison of T2-weighted breath-hold and respiratory-triggered sequences. Magnetic Resonance Imaging. 27 (9), 1223-1229 (2009).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten