Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
In vivo Imágenes de calcio de conjuntos neuronales en redes de neuronas sensoriales primarias en ganglios intactos de la raíz dorsal
En este artículo
Resumen
Este protocolo describe la exposición quirúrgica del ganglio de la raíz dorsal (GRD) seguida de GCaMP3 (indicador de Ca2+ codificado genéticamente; Proteína fluorescente verde-Calmodulina-M13 Proteína 3) Imagen de Ca2+ de los conjuntos neuronales utilizando ratones Pirt-GCaMP3 mientras se aplica una variedad de estímulos a la pata trasera ipsilateral.
Resumen
Las imágenes de Ca 2+ se pueden utilizar como un proxy de la actividad celular, incluidos los potenciales de acción y varios mecanismos de señalización que implican la entrada de Ca 2+ en el citoplasma o la liberación de reservas intracelulares de Ca2+. Las imágenes de Ca2+ basadas en Pirt-GCaMP3 de neuronas sensoriales primarias del ganglio de la raíz dorsal (DRG) en ratones ofrecen la ventaja de la medición simultánea de un gran número de células. Se pueden monitorizar hasta 1.800 neuronas, lo que permite estudiar redes neuronales y procesos somatosensoriales como un conjunto en su contexto fisiológico normal a nivel poblacional in vivo. El gran número de neuronas monitoreadas permite la detección de patrones de actividad que serían difíciles de detectar utilizando otros métodos. Los estímulos se pueden aplicar a la pata trasera del ratón, lo que permite estudiar los efectos directos de los estímulos en el conjunto de neuronas DRG. El número de neuronas que producen transitorios de Ca 2+, así como la amplitud de los transitorios de Ca2+ indica sensibilidad a modalidades sensoriales específicas. El diámetro de las neuronas proporciona evidencia de tipos de fibras activadas (mecanos no nocivos vs. fibras nocivas del dolor, fibras Aβ, Aδ y C). Las neuronas que expresan receptores específicos pueden ser marcadas genéticamente con td-Tomato y Cre específicas recombinasas junto con Pirt-GCaMP. Por lo tanto, las imágenes Pirt-GCaMP3 Ca2+ de DRG proporcionan una poderosa herramienta y modelo para el análisis de modalidades sensoriales específicas y subtipos de neuronas que actúan como un conjunto a nivel poblacional para estudiar el dolor, la picazón, el tacto y otras señales somatosensoriales.
Introducción
Las neuronas sensoriales primarias inervan directamente la piel y llevan la información somatosensorial de vuelta al sistema nervioso central 1,2. Los ganglios de la raíz dorsal (GRD) son grupos de células corporales de 10.000-15.000 neuronas sensoriales primarias 3,4. Las neuronas DRG presentan diversos tamaños, niveles de mielinización y patrones de expresión de genes y receptores. Las neuronas de diámetro más pequeño incluyen neuronas sensibles al dolor y las neuronas de mayor diámetro típicamente responden a estímulos mecánicos no dolorosos 5,6. Los trastornos en las neuronas sensoriales primarias, como lesiones, inflamación crónica y neuropatías periféricas, pueden sensibilizar estas neuronas a diversos estímulos y contribuir al dolor crónico, alodinia e hipersensibilidad al dolor 7,8. Por lo tanto, el estudio de las neuronas DRG es importante para comprender tanto la somatosensación en general como muchos trastornos de dolor y picazón.
Las neuronas que disparan in vivo son esenciales para la somatosensación, pero hasta hace poco, las herramientas para estudiar los ganglios intactos in vivo se han limitado a un número relativamente pequeño de células 9. Aquí, describimos un método poderoso para estudiar los potenciales de acción o actividades de las neuronas a nivel de población in vivo como un conjunto. El método emplea imágenes basadas en la dinámica citoplasmática de Ca2+. Los indicadores fluorescentes sensibles al Ca 2+ son buenos indicadores para medir la actividad celular debido a la concentración normalmente baja de Ca2+ citoplasmático. Estos indicadores han permitido el monitoreo simultáneo de cientos a varios miles de neuronas sensoriales primarias en ratones 9,10,11,12,13,14,15,16 y ratas 17. El método de imágenes de Ca2+ in vivo descrito en este estudio se puede utilizar para observar directamente las respuestas a nivel poblacional a estímulos mecánicos, fríos, térmicos y químicos.
La proteína de membrana de unión a fosfoinositida, Pirt se expresa en altos niveles en casi todas (>95%) neuronas sensoriales primarias18,19 y se puede utilizar para impulsar la expresión del sensor Ca 2+, GCaMP3, para monitorear la actividad neuronal in vivo20. En este protocolo, se describen técnicas para realizar cirugía DRG in vivo, imágenes de Ca2+ y análisis en el lado derecho lumbar 5 (L5) DRG de ratones Pirt-GCaMP314 utilizando microscopía de barrido láser confocal (LSM).
Protocolo
Todos los procedimientos descritos aquí se realizaron de acuerdo con un protocolo aprobado por el Comité Institucional de Cuidado y Uso de Animales del Centro de Ciencias de la Salud de la Universidad de Texas en San Antonio.
NOTA: Una vez iniciada, la cirugía con animales (paso 1) y las imágenes (paso 2) deben completarse de manera continua. El análisis de datos (paso 3) se puede realizar más adelante.
1. Cirugía y fijación del animal para la imagen L5 DRG del lado derecho
NOTA: En este estudio se utilizaron ratones Pirt-GCaMP3 C57BL / 6J machos y hembras de 8 semanas de edad o más. Si bien cualquiera de los sexos se puede visualizar igualmente bien, los ratones deben tener al menos 8 semanas de edad debido a la expresión débil o intermitente de Pirt en ratones más jóvenes. Los ratones Pirt-GCaMP3 C57BL / 6J se generaron en la Universidad Johns Hopkins14. Se pueden obtener imágenes de DRG de cualquier lado y se pueden obtener imágenes de otros DRG lumbares (por ejemplo, lumbar 4). Los tiempos dados son estimaciones para un cirujano experimentado. Los problemas técnicos ocasionales, como el aumento del sangrado, pueden aumentar el tiempo requerido.
- Prepare una solución salina estéril que contenga 40 mg/ml de ketamina y 6 mg/ml de xilazina. El volumen total debe ser de al menos 9 μL/g de masa corporal tanto para la cirugía como para la eutanasia después de la toma de imágenes.
PRECAUCIÓN: La ketamina es dañina si se inyecta, se ingiere o cuando entra en contacto con el ojo. Manejar con cuidado. - Asegúrese de que todas las herramientas quirúrgicas estén limpias y esterilizadas en autoclave u otro método aprobado por la Guía de los NIH para el cuidado y uso de animales de laboratorio.
- Entre 15 y 25 min antes de la cirugía, inyecte un ratón Pirt-GCaMP3 por vía intraperitoneal (i.p.) con ~2.25 μL de ketamina/xilazina por cada gramo de peso corporal (90 mg/kg de ketamina, 13,5 mg/kg de xilazina). No exceda los 120 mg/kg de ketamina.
- Dentro de 15 a 25 minutos después de la inyección de anestesia (paso 1.3), compruebe si el ratón ha alcanzado el plano quirúrgico de la anestesia pellizcando la pata trasera contralateral (no la pata trasera ipsilateral/derecha). La ausencia de reflejo de retirada de las extremidades posteriores asegura que se logre un plano quirúrgico de anestesia.
NOTA: El reflejo de retirada de las extremidades posteriores se utiliza durante todo el experimento para controlar la anestesia. Utilice siempre la pata trasera contralateral. - Coloque el ratón sobre una almohadilla térmica para mantener la temperatura corporal a 37 °C.
NOTA: Puede ser útil mantener la cabeza del ratón en su lugar con un marco estereotáxico (consulte la Tabla de materiales) u otro marco basado en la preferencia del investigador. - Localice el agrandamiento lumbar palpando el hueso pélvico del ratón. Afeite la parte posterior del ratón por encima del área de agrandamiento lumbar. Este paso debería tomar ~ 90 s.
NOTA: El ratón se puede quitar brevemente de la almohadilla térmica para afeitarse. - Haga una incisión rectangular de tres lados (8 mm x 20 mm) por encima del agrandamiento lumbar con tijeras y doble la piel con fórceps (Figura 1A). Este paso toma ~2 min.
NOTA: Los investigadores también pueden usar pinzas hemostáticas o un retractor para mantener abierta la incisión. Esta no es una cirugía de supervivencia, por lo que no es necesaria una limpieza adicional del área quirúrgica; sin embargo, se puede hacer usando povidona yodada. El tamaño de incisión más grande aceptable se da aquí. Una incisión más pequeña es preferible a una incisión más grande. - Use las tijeras de disección de resorte de 13 mm para hacer incisiones de 3-4 mm en el lado derecho de la columna vertebral. Use tijeras para cortar la piel y los músculos a los lados con el fin de exponer la columna vertebral (Figura 1B). Este paso toma ~3 min.
- Use tijeras de 8 mm para limpiar el proceso transversal del DRG L5 del lado derecho cortando el músculo y el tejido conectivo mientras trata de minimizar el sangrado. Use algodón y/o espuma de gel para absorber la sangre. La vértebra L5 es la primera vértebra rostral al hueso de la pelvicio.
NOTA: Este paso dura ~ 3 minutos y puede tomar más tiempo si el animal sangra más de lo normal. - Abra el proceso transversal L5 del lado derecho utilizando rongeurs de Friedman-Pearson o pinzas finas fuertes. Tenga cuidado de no tocar el DRG (Figura 1C).
NOTA: Este paso dura ~ 2 minutos, pero puede tomar más tiempo si el animal sangra más de lo normal. - No proceda hasta que el sangrado se detenga por completo. Evite el sangrado en la superficie de DRG usando espuma de gel o algodón. Este paso toma 1-4 min.
- Mueva el ratón y la almohadilla térmica al escenario personalizado (Figura 2A,B). Use cinta adhesiva para asegurar al animal y almohadilla térmica en su lugar. Coloque la nariz del animal en el cono de la nariz para que el animal pueda recibir anestesia continua con isoflurano. Asegure la pata trasera derecha que sobresale del escenario para que los estímulos se puedan aplicar fácilmente a la pata. Este paso dura 3 minutos.
- Asegure la columna vertebral en su lugar con las pinzas de la etapa sobre la piel en las vértebras y / o el hueso pélvico solo rostral y caudal hasta el L5 DRG. Ajuste las abrazaderas y la platina para que la superficie del DRG esté lo más nivelada posible (Figura 2B, C).
NOTA: Puede ser necesario recortar el tejido que se encuentra entre el DRG y el objetivo. - Coloque la platina debajo del microscopio de modo que el objetivo quede 8 mm directamente por encima del DRG cuando se baje (Figura 3A, B). Inserte el termómetro rectal.
NOTA: La distancia desde el GRD hasta el objetivo puede variar según el objetivo, el microscopio y el animal. - Conecte las líneas eléctricas a la almohadilla térmica y al termómetro rectal. Conecte el cono de la nariz a las líneas de gas isoflurano.
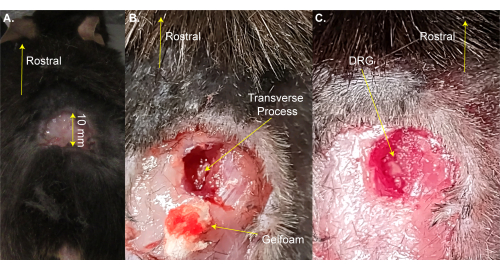
Figura 1: Ejemplo de cirugía de exposición a DRG . (A) Se afeitó un área pequeña y se cortó la piel y se dobló hacia atrás. La incisión es de ~10 mm en el eje rostral-caudal. (B) Se realizó una incisión en el lado derecho de la columna vertebral y se cortaron los músculos y el tejido conectivo, exponiendo el proceso transversal del lado derecho L5. La sangre se absorbió con espuma de gel. (C) Se limpió la apófisis transversal y se extrajo el hueso sobre el DRG. Haga clic aquí para ver una versión más grande de esta figura.
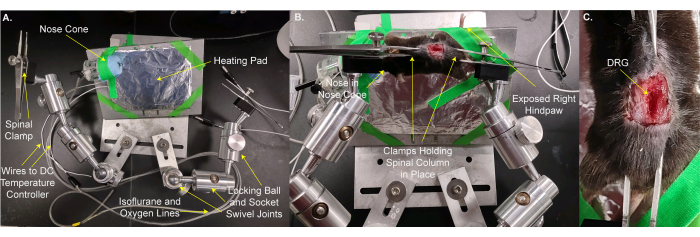
Figura 2: Montaje del ratón en una etapa personalizada para imágenes DRG . (A) Se muestra la etapa personalizada. Consiste en una placa base y una placa para el animal. La placa de montaje del animal está en una bola de bloqueo y una junta giratoria de zócalo. Un cono nasal con líneas para suministrar mezcla de oxígeno / isoflurano y una línea de gas residual junto con una almohadilla térmica envuelta en papel de aluminio están pegadas a la placa de montaje del animal. Dos brazos, cada uno hecho de tres juntas giratorias de bola de bloqueo y zócalo, están atornillados a la placa base. Cada brazo tiene una abrazadera hecha de pinzas con un tornillo para apretar y aflojar. (B) El animal está montado en la placa de montaje del animal. Su nariz se coloca en el cono de la nariz. Las pinzas se colocan sobre la piel sosteniendo la columna vertebral y el hueso pélvico. La pata trasera derecha (ipsilateral) está pegada con cinta adhesiva para sobresalir y facilitar el acceso para aplicar estímulos. (C) Una imagen en primer plano de la columna vertebral y el hueso pélvico sujetados. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: El animal en la etapa personalizada se coloca debajo del objetivo del microscopio . (A) Una vista de gran angular del escenario, el animal y el microscopio. Los cables al controlador de temperatura de CC y las líneas a la toma de oxígeno / isoflurano y la línea de gases residuales son visibles a la izquierda. (B) Una vista de primer plano del animal debajo del objetivo del microscopio. El DRG está ~8 mm por debajo del objetivo. Se inserta el termómetro rectal y la nariz está dentro del cono nasal. Haga clic aquí para ver una versión más grande de esta figura.
2. Imágenes DRG
- Utilice un microscopio confocal vertical 10x/0.4 DIC objetivo y el software asociado (consulte la Tabla de materiales) para obtener imágenes. Utilice la configuración del filtro verde (FITC): excitación 495 nm, emisión 519 nm, longitud de onda de detección 500-580 nm, dispositivo de imagen GaAsP-Pmt1, detector GaAsP-PMT.
NOTA: Para otros microscopios, utilice la configuración recomendada por el fabricante. - Encuentra la superficie del DRG con el microscopio. Ajuste las abrazaderas en el escenario de modo que la superficie DRG esté lo más nivelada posible y que el área de superficie máxima se visualice en el plano focal.
NOTA: La elección del objetivo puede variar dependiendo del microscopio utilizado y la preferencia del usuario. La elección del software depende del microscopio. Un DRG nivelado da como resultado imágenes más claras, permite que el software construya películas más claras, permite obtener imágenes de más neuronas y hace que el análisis sea más fácil y preciso. - Suministre 1% -1.5% de isoflurano en el flujo de oxígeno al cono de la nariz para garantizar que el ratón permanezca anestesiado. Controle al animal cuidadosamente durante todo el procedimiento para mantener la anestesia con isoflurano sin sobredosis.
NOTA: La cantidad de isoflurano necesaria para mantener la anestesia puede variar según el animal individual y el estímulo. Por lo general, el 1,5% es suficiente. Si el animal se mueve durante el estímulo, se debe aumentar el isoflurano. Si la respiración se vuelve superficial, se debe disminuir el isoflurano. Antes de cada estímulo, pruebe el reflejo de retirada de la extremidad posterior en la pata trasera contralateral.
PRECAUCIÓN: El isoflurano puede ser dañino o causar mareos o somnolencia si se inhala. Evite la inhalación y úselo solo en un área bien ventilada. - Cargue el protocolo de escaneo rápido del microscopio.
- Utilice los ajustes típicos para el escaneo rápido: tamaño de vóxel 2,496 μm x 2,496 μm x 16 μm, 512 x 512 píxeles, 10 pila Z de corte óptico, 1 unidad aireada (AU)/32 μm, potencia láser de 1% 488 nm 5 mW, tiempo de píxeles 1,52 μs, tiempo de línea 0,91 ms, tiempo de fotogramas 465 ms, velocidad de escaneo LSM 8, escaneo bidireccional, Ganancia del detector GaAsP-PMT 650 V, ganancia digital 1. Los ajustes óptimos pueden variar según el microscopio y el animal.
- Para configurar un protocolo de escaneo rápido, haga clic en la pestaña Adquisición . En Parámetros de adquisición, haga clic en la pestaña Marcos . Haga clic en Ajustes preestablecidos > 512 x 512 para configurar el microscopio para grabar una imagen de 512 píxeles x 512 píxeles. Esto a su vez establecerá los valores X e Y del tamaño del vóxel en función del tamaño de la imagen, que está determinado por el software del microscopio.
- En Parámetros de adquisición, haga clic en la pestaña Canales . Haga clic en el cuadro Track2 . Use el menú desplegable junto al cuadro Track2 para seleccionar Verde (FITC). Se abrirá una nueva pestaña debajo de Track2.
- Junto a Láseres, haga clic en el cuadro 488 . Esto establecerá las longitudes de onda de excitación y emisión.
- Junto al control deslizante de 488 nm, ajuste Potencia láser al 1%. Haga clic en el botón 1 AU para configurar la apertura de 1 unidad Airy. En FITC, establezca Ganancia maestra en 650 V, Desplazamiento digital en 0 y Ganancia digital en 1.
- En la pestaña Adquisición , marque la casilla Z-Stack . Haga clic en el botón Live para ver una imagen en vivo del ganglio. Gire la perilla del plano focal hacia arriba hasta que solo se vea un pequeño arco de neuronas.
- En Parámetros de adquisición, haga clic en la pestaña Z-Stack > botón Establecer último . Gire la perilla del plano focal hacia abajo hasta que solo se vea un pequeño arco de neuronas. En Parámetros de adquisición, haga clic en la pestaña Z-Stack > botón Establecer primero . Haga clic en el botón Live para desactivar la imagen en vivo.
- En Parámetros de adquisición, haga clic en la pestaña Z-Stack . Rellene el campo Sectores con 10. Esto establecerá 10 cortes ópticos y determinará automáticamente la profundidad del vóxel.
- En la pestaña Adquisición, haga clic en el cuadro Serie temporal . Aparecerá una nueva pestaña Serie temporal en Parámetros de adquisición. Haga clic en la pestaña Serie temporal > campo Ciclos con el número de ciclos que desea realizar a continuación. En este caso, es 8.
- Establezca la velocidad y la dirección del escaneo. En Parámetros de adquisición, ficha Modo de adquisición > > Dirección > Flecha de doble punta para el escaneo birdireccional. Seleccione la pestaña Modo de adquisición > el control deslizante Frame > Scan Speed Slider > 8.
NOTA: Normalmente se pueden cargar los ajustes de un experimento anterior y sólo hay que ajustar la potencia del láser si la imagen es demasiado brillante o tenue y ajustar el Z-Stack Set Last y Set First.
- Realice un breve escaneo de 8 ciclos del DRG haciendo clic en Iniciar experimento en la pestaña Adquisición . Cree una película haciendo una proyección ortogonal de escaneos (un escaneo por cuadro) a lo largo del tiempo y verifique manualmente la claridad de la imagen y los artefactos de imagen, como "ondas" de brillo que cruzan el DRG. Ajuste la posición de la abrazadera y el grosor de la sección óptica y repita este paso hasta que se logre una película clara y de alta calidad.
NOTA: Este paso debe repetirse si el animal se mueve o es movido por el investigador. Los problemas a buscar incluyen áreas del ganglio (no solo neuronas individuales) que parecen volverse más claras y oscuras en el transcurso del experimento, creando una apariencia ondulada o haciendo que las áreas desaparezcan o se vuelvan más brillantes. El movimiento superior a aproximadamente la mitad del diámetro de una neurona pequeña (<20 μm) es otro problema importante. La ondulación a menudo se puede arreglar acercando la primera y la última posición de la Z-Stack (consulte el paso 2.4.4 anterior) y reduciendo el grosor del corte óptico. La película 1 proporciona un ejemplo de un ganglio ondulado antes de nivelar y establecer el grosor de corte óptico correcto. La película 2 es después de corregir la nivelación y el grosor de la rebanada óptica. La diferencia es sutil, pero tiene un efecto tremendo en el análisis. - Cargue el protocolo de escaneo de alta resolución del microscopio.
- Utilice los ajustes típicos para el escaneo de alta resolución: tamaño de vóxel 1,248 μm x 1,248 μm x 14 μm, 1024 x 1024 píxeles, 6 pila Z de corte óptico, 1,2 unidad aireada (AU)/39 μm, 5% de potencia láser de 488 nm/25 mW, tiempo de píxeles 2,06 μs, tiempo de línea 4,95 ms, tiempo de fotogramas 5,06 s, velocidad de escaneo LSM 6, escaneo bidireccional, Ganancia del detector GaAsP-PMT 650 V, ganancia digital 1. Los ajustes óptimos pueden variar según el microscopio y el animal.
- Para configurar un protocolo de escaneo de alta resolución, haga clic en la pestaña Adquisición . En Parámetros de adquisición, haga clic en la pestaña Marcos . Haga clic en Ajustes preestablecidos > 1024 x 1024 para configurar el microscopio para grabar una imagen de 1024 píxeles x 1024 píxeles. Esto a su vez establecerá los valores X e Y del tamaño del vóxel en función del tamaño de la imagen, que está determinado por el software del microscopio.
- En Parámetros de adquisición, haga clic en la pestaña Canales . Haga clic en el cuadro Track2 . Use el menú desplegable junto al cuadro Track2 para seleccionar Verde (FITC). Se abrirá una nueva pestaña debajo de Track2.
- Junto a Láseres, haga clic en el cuadro 488 . Esto establecerá las longitudes de onda de excitación y emisión. Haga clic en el cuadro Rango láser de alta intensidad .
- Junto al control deslizante de 488 nm, ajuste Potencia láser al 5%. Haga clic en el botón 1 AU para configurar la apertura de 1 unidad Airy. En FITC establezca Ganancia maestra en 650 V, Desplazamiento digital en 0 y Ganancia digital en 1.
- En la pestaña Adquisición , marque la casilla Z-Stack . Haga clic en el botón Live para ver una imagen en vivo del ganglio. Gire la perilla del plano focal hacia arriba hasta que solo se vea un pequeño arco de neuronas.
- En Parámetros de adquisición, haga clic en la pestaña Z-Stack > botón Establecer último . Gire la perilla del plano focal hacia abajo hasta que solo se vea un pequeño arco de neuronas. En Parámetros de adquisición, haga clic en la pestaña Z-Stack > botón Establecer primero . Haga clic en el botón Live para desactivar la imagen en vivo.
- En Parámetros de adquisición, haga clic en la pestaña Z-Stack . Rellene el campo Sectores con 6. Esto establecerá 6 cortes ópticos y determinará automáticamente la profundidad del vóxel.
- En la pestaña Adquisición , asegúrese de que la casilla Serie temporal no esté marcada (sin serie temporal ).
- Establezca la velocidad y la dirección del escaneo. En Parámetros de adquisición, haga clic en la pestaña Modo de adquisición > Ajustes preestablecidos de fotogramas > > 1024 x 1024. Seleccione la ficha Modo de adquisición > Fotograma > dirección > flecha de doble punta para el escaneo bidireccional. Seleccione la pestaña Modo de adquisición > Control deslizante Frame > Scan Speed Slider > 6.
- Si las células están marcadas con td-Tomate, configure el microscopio para escanear el canal rojo de excitación de 592 nm / emisión de 614 nm, longitud de onda de detección de 600-700 nm además del canal verde. Establezca esto yendo a Parámetros de adquisición y haga clic en la pestaña Canales > cuadro Track1 . Use el menú desplegable junto al cuadro Track1 para seleccionar Rojo (Texas Red). Siga el mismo proceso que en el paso 2.6.3, excepto que haga clic en el cuadro 561 en lugar del cuadro 488 . Ajuste la potencia del láser al 1%. Td-Tomato es mucho más brillante que GCaMP3 y requiere menor potencia láser.
- Haga una imagen de alta resolución del DRG haciendo clic en el botón Iniciar experimento en la pestaña Adquisición .
- Cargue el protocolo de escaneo rápido del microscopio (ver paso 2.4). Registrar la actividad espontánea en el GRD durante 80 ciclos (aproximadamente 10 min). Genere una película de proyección ortogonal y verifique que la imagen sea de calidad suficiente para el análisis.
NOTA: Las consideraciones de calidad son las mismas que en el paso 2.5. - Para aplicar estímulos, configure el microscopio para realizar 15-20 escaneos. Espere a que se completen los escaneos 1-5 para producir la línea de base. Aplique el estímulo durante las exploraciones 6-10. Espere al menos 5 minutos después de cada estímulo antes de aplicar el siguiente estímulo para evitar la desensibilización.
NOTA: Los estímulos mecánicos deben aplicarse primero, y luego estímulos fríos, térmicos y químicos. Se deben aplicar estímulos más débiles (p. ej., baja fuerza mecánica, temperaturas más cercanas a la temperatura ambiente) antes de estímulos más fuertes (p. ej., mayor fuerza mecánica, temperaturas más alejadas de la temperatura ambiente). Al aplicar estímulos, asegúrese de no causar ningún movimiento del DRG. Para estímulos térmicos fuertes en particular, a menudo es necesario aplicar isoflurano al 2% durante 1-2 minutos antes de comenzar el estímulo. En el microscopio utilizado en este estudio, cada escaneo se puede escuchar claramente, de modo que el investigador puede identificar fácilmente el final del escaneo, lo que permite la aplicación del estímulo inmediatamente después del escaneo 5. Sin embargo, cualquier método que facilite la aplicación de estímulos en un punto de tiempo consistente funcionará. - Para una prensa mecánica, sostenga la pinza del algometro con la pata entre las paletas sin tocar la pata, y pellizque comenzando inmediatamente después del final del escaneo 5 y deteniéndose inmediatamente después del escaneo 10. Controle la fuerza de prensa con un algometro (consulte la Tabla de materiales). Mantenga la fuerza de prensa lo más cerca posible de la fuerza deseada (aquí usamos un estímulo de prensa de 100 g) y asegúrese de que no exceda los 10 g por encima de la fuerza deseada.
NOTA: Uno puede detectar estímulos con tan solo 0.07 g de filamentos von Frey y hasta 600 g de fuerza de presión. - Para estímulos fríos y térmicos, enfríe o caliente un vaso de precipitados de agua justo por debajo (para frío) o por encima (para térmico) de la temperatura deseada y comience a escanear. Aquí, utilice un estímulo de 45 °C. Cuando el agua tenga la temperatura correcta, aplique el estímulo inmediatamente después del escaneo 5 sumergiendo la pata en el agua. Tire del vaso de precipitados inmediatamente después de escanear 10.
NOTA: La temperatura debe estar dentro de 1 °C de la temperatura deseada en el escaneo 5. Si la temperatura del vaso de precipitados es incorrecta, no aplique el estímulo porque esto podría desensibilizar las neuronas. En su lugar, vuelva a enfriar o recalentar el agua e inténtelo de nuevo. Hemos medido temperaturas tan bajas como 0 ° C (agua helada) y tan altas como 95 ° C. Sin embargo, recuerde que las temperaturas superiores a 50 ° C pueden dañar los tejidos y confundir experimentos posteriores. Del mismo modo, algunos productos químicos (por ejemplo, tetrodotoxina, capsaicina) son irreversibles o no se pueden lavar y pueden impedir nuevos experimentos en el animal. - Después de aplicar y registrar todos los estímulos, sacrificar al animal por sobredosis con ketamina/xilazina (200 mg/kg de ketamina, 30 mg/kg de xilazina o 5 μL por gramo de masa corporal de solución preparada en el paso 1.1) seguida de decapitación.
3. Análisis de datos
- Abra los archivos de imagen arrastrando y soltando en ImageJ. Después de abrir el archivo, seleccione el tipo de imagen en Imagen > Tipo > color RGB.
NOTA: El plugin StackReg21 bajo Plugins > StackReg es útil para corregir y alinear artefactos de movimiento. ImageJ puede leer la mayoría de los formatos de archivo de software de microscopio. La elección del tipo de imagen depende de las preferencias del usuario. RGB simplifica la producción de imágenes en color para su publicación. Los paquetes de software del fabricante del microscopio o cytoNet22 también pueden ayudar en el análisis. No es necesario descargar, instalar y usar StackReg, pero se recomienda. - Utilice la herramienta de región de interés (ROI) para seleccionar neuronas activas en Analizar > herramienta > ROI Manager. Dibuje ROI usando las herramientas de elipse o rectángulo en la barra de herramientas y colóquelos en el archivo ROI presionando el botón Agregar en Agregar en la ventana del administrador de ROI, o presionando la tecla "t".
NOTA: Asegúrese de guardar con frecuencia el archivo ROI. Los usuarios siempre pueden restaurar los ROI arrastrando y soltando un archivo ROI en ImageJ y haciendo clic en el cuadro Mostrar todo en la parte inferior de la ventana ROI Manager. No se recomienda guardar como superposición. Por experiencia, guardar como un archivo ROI.zip simplifica el análisis. - En Analizar > establecer medidas, asegúrese de que la opción de valor gris medio esté marcada; desmarque todas las demás casillas en Establecer medidas. Utilice la herramienta multimedida en el menú del administrador de ROI para calcular la intensidad dentro de los ROI. Mida la intensidad utilizando la ventana ROI en Más > Multimedida.
NOTA: A veces, las neuronas adyacentes estarán demasiado juntas para dibujar ROI separados. Estas neuronas no se pueden usar para medir la intensidad transitoria, pero pueden incluirse en los recuentos de neuronas activas. - Guarde el archivo CSV generado por Multimeasure y abra el archivo CSV con cualquier software de hoja de cálculo.
NOTA: Consulte Archivo complementario 1 para obtener una plantilla de hoja de cálculo de ejemplo para facilitar el análisis. - Calcule la intensidad transitoria de Ca 2+ como ΔF / F 0 = (F t- F 0) / F 0, donde Ft es la intensidad de píxeles en un ROI en el punto de tiempo de interés, y F 0 es la intensidad de referencia determinada promediando las intensidades de los 2-4 cuadros antes del transitorio Ca 2+ para la actividad espontánea o los primeros 1-5 cuadros del ROI para Ca 2+ transitorios que ocurren durante la estimulación. Excluya cualquier neurona que produzca picos de Ca2+ antes del estímulo y no analice la actividad después de que termine el estímulo.
- Para el análisis de intensidad transitoria de Ca2+ , muestree aleatoriamente aproximadamente el mismo número de neuronas de cada ganglio para evitar sesgar los datos hacia los ganglios que produjeron el mayor número de neuronas que respondieron. Excluir picos donde ΔF / F 0 <0.15 .
NOTA: Existen métodos alternativos publicados de análisis e inclusión/exclusión de neuronas 11,12,23,24,25,26,27,28. - Mida los diámetros de las neuronas con la herramienta de línea de la barra de herramientas de ImageJ. Calcule el diámetro promedio a partir de las líneas dibujadas a lo largo de los diámetros más largo y más corto.
Resultados
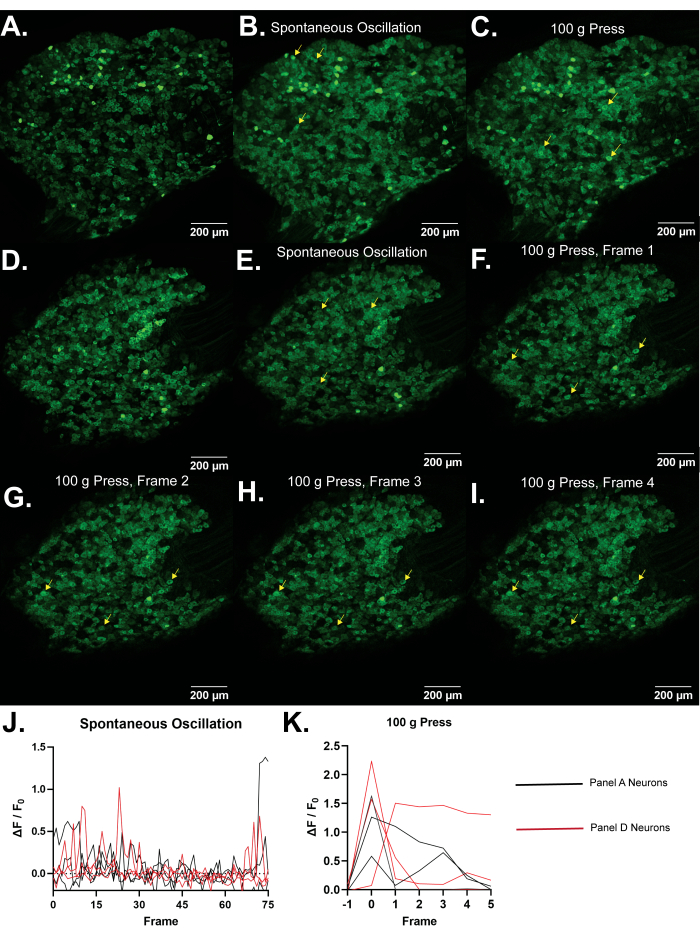
Figura 4: Imágenes representativas de los ganglios de la raíz dorsal L5 de ratones Pirt-GCaMP3. (A,D) Se muestran escaneos de alta resolución de fotograma único de los ganglios de la raíz dorsal L5 de ratones Pirt-GCaMP3. (B,E) . Proyecciones de intensidad media de 15 fotogramas de ganglios Pir...
Discusión
El dolor persistente está presente en una amplia gama de trastornos, debilitando y/o reduciendo la calidad de vida de aproximadamente 8% de las personas29. Las neuronas sensoriales primarias detectan estímulos nocivos en la piel, y su plasticidad contribuye al dolor persistente8. Si bien las neuronas se pueden estudiar en cultivo celular y explantes, al hacerlo las elimina de su contexto fisiológico normal. La exposición quirúrgica del DRG, seguida de imágenes Pirt-GC...
Divulgaciones
Los autores declaran que no hay intereses financieros contrapuestos.
Agradecimientos
Este trabajo fue apoyado por las subvenciones R01DE026677 y R01DE031477 de los Institutos Nacionales de Salud (a Y.S.K.), UTHSCSA startup fund (Y.S.K.) y un premio Rising STAR del sistema de la Universidad de Texas (YSK).
Materiales
| Name | Company | Catalog Number | Comments |
| Anased Injection (Xylazine) | Covetrus, Akorn | 33197 | |
| C Epiplan-Apochromat 10x/0.4 DIC | Cal Zeiss | 422642-9900-000 | |
| Cotton Tipped Applicators | McKesson | 24-106-1S | |
| Curved Hemostat | Fine Science Tools | 13007-12 | |
| DC Temperature Controller | FHC | 40-90-8D | |
| DC Temperature Controller Heating Pad | FHC | 40-90-2-05 | |
| Dumont Ceramic Coated Forceps | Fine Science Tools | 11252-50 | |
| FHC DC Temperature Controller | FHC | 40-90-8D | |
| Fluriso (Isoflurane) | MWI Animal Health, Piramal Group | 501017 | |
| Friedman-Pearson Rongeurs | Fine Science Tools | 16221-14 | |
| GelFoam | Pfizer | 09-0353-01 | |
| Ketaset (Ketamine) | Zoetis | KET-00002R2 | |
| Luminescent Green Stage Tape | JSITON/ Amazon | B803YW8ZWL | |
| Matrx VIP 3000 Isoflurane Vaporizer | Midmark | 91305430 | |
| Micro dissecting scissors | Roboz | RS-5882 | |
| Micro dissecting spring scissors | Fine Science Tools | 15023-10 | |
| Micro dissecting spring scissors | Roboz | RS-5677 | |
| Mini Rectal Thermistor Probe | FHC | 40-90-5D-02 | |
| Operating scissors | Roboz | RS-6812 | |
| Pirt-GCaMP3 C57BL/6J mice | Johns Hopkins University | N/A | Either sex can be imaged equally well. Mice should be at least 8 weeks old due to weak or intermittent Pirt promoter expression in younger mice. |
| SMALGO small animal algometer | Bioseb In vivo Research Instruments | BIO-SMALGO | |
| Stereotaxic frame | Kopf Model 923-B | 923-B | |
| td-Tomato C57BL/6J mice | Jackson Laboratory | 7909 | |
| Top Plate, 6 in x 10 in | Newport | 290-TP | |
| TrpV1-Cre C57BL/6J mice | Jackson Laboratory | 17769 | |
| Zeiss LSM 800 confocal microscope | Cal Zeiss | LSM800 | |
| Zeiss Zen 2.6 Blue Edition Software | Cal Zeiss | Zen (Blue Edition) 2.6 |
Referencias
- Rivero-Melián, C., Grant, G. Distribution of lumbar dorsal root fibers in the lower thoracic and lumbosacral spinal cord of the rat studied with choleragenoid horseradish peroxidase conjugate. The Journal of Comparative Neurology. 299 (4), 470-481 (1990).
- Wessels, W. J., Marani, E. A rostrocaudal somatotopic organization in the brachial dorsal root ganglia of neonatal rats. Clinical Neurology and Neurosurgery. 95, 3-11 (1993).
- Schmalbruch, H. The number of neurons in dorsal root ganglia L4-L6 of the rat. The Anatomical Record. 219 (3), 315-322 (1987).
- Sørensen, B., Tandrup, T., Koltzenburg, M., Jakobsen, J. No further loss of dorsal root ganglion cells after axotomy in p75 neurotrophin receptor knockout mice. The Journal of Comparative Neurology. 459 (3), 242-250 (2003).
- Basbaum, A. I., Woolf, C. J. Pain. Current Biology. 9 (12), 429-431 (1999).
- Liu, Y., Ma, Q. Generation of somatic sensory neuron diversity and implications on sensory coding. Current Opinion in Neurobiology. 21 (1), 52-60 (2011).
- Basbaum, A. I., Bautista, D. M., Scherrer, G., Julius, D. Cellular and molecular mechanisms of pain. Cell. 139 (2), 267-284 (2009).
- Stucky, C. L., Mikesell, A. R. Cutaneous pain in disorders affecting peripheral nerves. Neuroscience Letters. 765, 136233 (2021).
- Iseppon, F., Linley, J. E., Wood, J. N. Calcium imaging for analgesic drug discovery. Neurobiology of Pain. 11, 100083 (2022).
- Chen, Z., et al. Adjacent intact nociceptive neurons drive the acute outburst of pain following peripheral axotomy. Scientific Reports. 9 (1), 7651 (2019).
- Chisholm, K. I., Khovanov, N., Lopes, D. M., La Russa, F., McMahon, S. B. Large scale in vivo recording of sensory neuron activity with GCaMP6. eNeuro. 5 (1), (2018).
- Emery, E. C., et al. In vivo characterization of distinct modality-specific subsets of somatosensory neurons using GCaMP. Science Advances. 2 (11), 1600990 (2016).
- Ishida, H., et al. In vivo calcium imaging visualizes incision-induced primary afferent sensitization and its amelioration by capsaicin pretreatment. The Journal of Neuroscience. 41 (41), 8494-8507 (2021).
- Kim, Y. S., et al. Coupled activation of primary sensory neurons contributes to chronic pain. Neuron. 91 (5), 1085-1096 (2016).
- MacDonald, D. I., et al. Silent cold-sensing neurons contribute to cold allodynia in neuropathic pain. Brain. 144 (6), 1711-1726 (2021).
- Wang, F., et al. Sensory afferents use different coding strategies for heat and cold. Cell Reports. 23 (7), 2001-2013 (2018).
- Kucharczyk, M. W., et al. The impact of bone cancer on the peripheral encoding of mechanical pressure stimuli. Pain. 161 (8), 1894-1905 (2020).
- Kim, A. Y., et al. a phosphoinositide-binding protein, functions as a regulatory subunit of TRPV1. Cell. 133 (3), 475-485 (2008).
- Kim, Y. S., et al. Central terminal sensitization of TRPV1 by descending serotonergic facilitation modulates chronic pain. Neuron. 81 (4), 873-887 (2014).
- Tian, L., et al. Imaging neural activity in worms, flies and mice with improved GCaMP calcium indicators. Nature Methods. 6 (12), 875-881 (2009).
- Thévenaz, P., Ruttimann, U. E., Unser, M. A pyramid approach to subpixel registration based on intensity. IEEE Transactions on Image Processing. 7 (1), 27-41 (1998).
- Mahadevan, A. S., et al. cytoNet: Spatiotemporal network analysis of cell communities. PLoS Computational Biology. 18 (6), 1009846 (2022).
- Barretto, R. P., et al. The neural representation of taste quality at the periphery. Nature. 517 (7534), 373-376 (2015).
- Leijon, S. C. M., et al. Oral thermosensing by murine trigeminal neurons: modulation by capsaicin, menthol and mustard oil. The Journal of Physiology. 597 (7), 2045-2061 (2019).
- Sekiguchi, K. J., et al. Imaging large-scale cellular activity in spinal cord of freely behaving mice. Nature Communications. 7, 11450 (2016).
- Wu, A., Dvoryanchikov, G., Pereira, E., Chaudhari, N., Roper, S. D. Breadth of tuning in taste afferent neurons varies with stimulus strength. Nature Communications. 6, 8171 (2015).
- Ran, C., Hoon, M. A., Chen, X. The coding of cutaneous temperature in the spinal cord. Nature Neuroscience. 19 (9), 1201-1209 (2016).
- Yarmolinsky, D. A., et al. Coding and plasticity in the mammalian thermosensory system. Neuron. 92 (5), 1079-1092 (2016).
- Torrance, N., Smith, B. H., Bennett, M. I., Lee, A. J. The epidemiology of chronic pain of predominantly neuropathic origin. Results from a general population survey. The Journal of Pain. 7 (4), 281-289 (2006).
- Shannonhouse, J., et al. Meclizine and metabotropic glutamate receptor agonists attenuate severe pain and Ca(2+) activity of primary sensory neurons in chemotherapy-induced peripheral neuropathy. The Journal of Neuroscience. 42 (31), 6020-6037 (2022).
- Luiz, A. P., et al. Cold sensing by Na(V)1.8-positive and Na(V)1.8-negative sensory neurons. Proceedings of the National Academy of Sciences of the United States of America. 116 (9), 3811-3816 (2019).
- Hartung, J. E., Gold, M. S. GCaMP as an indirect measure of electrical activity in rat trigeminal ganglion neurons. Cell Calcium. 89, 102225 (2020).
- Chung, M. K., Wang, S., Oh, S. L., Kim, Y. S. Acute and chronic pain from facial skin and oral mucosa: Unique neurobiology and challenging treatment. International Journal of Molecular Sciences. 22 (11), 5810 (2021).
- Chan, S. L., Mayne, M., Holden, C. P., Geiger, J. D., Mattson, M. P. Presenilin-1 mutations increase levels of ryanodine receptors and calcium release in PC12 cells and cortical neurons. The Journal of Biological Chemistry. 275 (24), 18195-18200 (2000).
- Sierra, D. A., Popov, S., Wilkie, T. M. Regulators of G-protein signaling in receptor complexes. Trends in Cardiovascular Medicine. 10 (6), 263-268 (2000).
- Yoshihara, K., et al. Astrocytic Ca(2+) responses in the spinal dorsal horn by noxious stimuli to the skin. Journal of Pharmacological Sciences. 137 (1), 101-104 (2018).
- Tan, C. H., McNaughton, P. A. The TRPM2 ion channel is required for sensitivity to warmth. Nature. 536 (7617), 460-463 (2016).
- Akemann, W., Mutoh, H., Perron, A., Rossier, J., Knöpfel, T. Imaging brain electric signals with genetically targeted voltage-sensitive fluorescent proteins. Nature Methods. 7 (8), 643-649 (2010).
- Gong, Y., et al. High-speed recording of neural spikes in awake mice and flies with a fluorescent voltage sensor. Science. 350 (6266), 1361-1366 (2015).
- Grewe, B. F., Langer, D., Kasper, H., Kampa, B. M., Helmchen, F. High-speed in vivo calcium imaging reveals neuronal network activity with near-millisecond precision. Nature Methods. 7 (5), 399-405 (2010).
- Harada, K., et al. Red fluorescent protein-based cAMP indicator applicable to optogenetics and in vivo imaging. Scientific Reports. 7 (1), 7351 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados