Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Reacción en cadena de la polimerasa e hibridación dot-blot para la detección de leptospira en muestras de agua
En este artículo
Resumen
En este estudio, se diseñó una aplicación de mancha de puntos para detectar Leptospira de los tres clados principales en muestras de agua. Este método permite la identificación de cantidades mínimas de ADN específicamente dirigidas por una sonda marcada con digoxigenina, fácilmente detectables por un anticuerpo anti-digoxigenina. Este enfoque es una herramienta valiosa y satisfactoria para fines de detección.
Resumen
El dot-blot es una técnica sencilla, rápida, sensible y versátil que permite la identificación de cantidades mínimas de ADN específicamente objetivo de la hibridación de sonda en presencia de ADN portador. Se basa en la transferencia de una cantidad conocida de ADN a un soporte sólido inerte, como una membrana de nailon, utilizando el aparato de mancha y sin separación electroforética. Las membranas de nailon tienen la ventaja de una alta capacidad de unión a ácidos nucleicos (400 μg / cm2), alta resistencia y están cargadas positiva o neutramente. La sonda utilizada es un fragmento de ssDNA altamente específico de 18 a 20 bases de largo marcadas con digoxigenina (DIG). La sonda se conjugará con el ADN de Leptospira . Una vez que la sonda se ha hibridado con el ADN objetivo, es detectada por un anticuerpo anti-digoxigenina, lo que permite su fácil detección a través de sus emisiones reveladas en una película de rayos X. Los puntos con una emisión corresponderán a los fragmentos de ADN de interés. Este método emplea el marcaje no isotópico de la sonda, que puede tener una vida media muy larga. El inconveniente de este marcador inmunológico estándar es una menor sensibilidad que las sondas isotópicas. Sin embargo, se mitiga mediante el acoplamiento de la reacción en cadena de la polimerasa (PCR) y los ensayos de dot-blot. Este enfoque permite el enriquecimiento de la secuencia objetivo y su detección. Además, se puede utilizar como una aplicación cuantitativa cuando se compara con una dilución en serie de un patrón conocido. Aquí se presenta una aplicación de mancha de puntos para detectar Leptospira de los tres clados principales en muestras de agua. Esta metodología se puede aplicar a grandes cantidades de agua una vez que se han concentrado por centrifugación para proporcionar evidencia de la presencia de ADN de Leptospiral. Se trata de una herramienta valiosa y satisfactoria para fines generales de cribado, y puede utilizarse para otras bacterias no cultivables que puedan estar presentes en el agua, mejorando la comprensión del ecosistema.
Introducción
La leptospirosis en humanos se origina principalmente en fuentes ambientales 1,2. La presencia de Leptospira en lagos, ríos y arroyos es un indicador de transmisión de leptospirosis entre la fauna silvestre y los animales domésticos y de producción que eventualmente pueden entrar en contacto con estos cuerpos de agua 1,3,4. Además, se ha identificado Leptospira en fuentes no naturales, como aguas residuales, estancadas y del grifo 5,6.
Leptospira es una bacteria distribuida en todo el mundo 7,8, y el papel del medio ambiente en su preservación y transmisión ha sido bien reconocido. Leptospira puede sobrevivir en agua potable bajo pH variable y minerales9, y en cuerpos de agua naturales1. También puede sobrevivir por largos períodos en agua destilada10, y bajo pH constante (7.8), puede sobrevivir hasta 152 días11. Además, Leptospira puede interactuar en consorcios bacterianos para sobrevivir a condiciones adversas12,13. Puede formar parte de biofilms en agua dulce con Azospirillum y Sphingomonas e incluso es capaz de crecer y soportar temperaturas superiores a los 49 °C14,15. También puede multiplicarse en suelos anegados y permanecer viable hasta 379 días16, conservando su capacidad de causar la enfermedad hasta por un año17,18. Sin embargo, se sabe poco sobre la ecología dentro de los cuerpos de agua y cómo se distribuye dentro de ellos.
Desde su descubrimiento, el estudio del género Leptospira se basó en pruebas serológicas. No fue hasta el siglo actual cuando las técnicas moleculares se generalizaron en el estudio de esta espiroqueta. El dot-blot ha sido escasamente utilizado para su identificación utilizando (1) una sonda isotópica basada en el ARNr 16S y en una repetición de secuencia inter-simple (ISSR)19,20, (2) como un inmunoensayo basado en nanooro para la leptospirosis humana aplicada a la orina21, o (3) como un ensayo basado en anticuerpos para muestras de orina bovina22. La técnica cayó en desuso porque originalmente se basaba en sondas isotópicas. Sin embargo, es una técnica bien conocida que, junto con la PCR, produce mejores resultados, y se considera segura debido al uso de sondas no isotópicas. La PCR desempeña un papel crucial en el enriquecimiento del ADN de Leptospira al amplificar un fragmento de ADN específico que se puede encontrar en trazas en una muestra. Durante cada ciclo de PCR, la cantidad del fragmento de ADN objetivo se duplica en la reacción. Al final de la reacción, el amplicón se ha multiplicado por un factor de más de un millón23. El producto amplificado por PCR, a menudo no visible en la electroforesis de agarosa, se hace visible a través de la hibridación específica con una sonda marcada con DIG en el dot-blot 24,25,26.
La técnica de dot-blot es sencilla, robusta y adecuada para numerosas muestras, lo que la hace accesible a laboratorios con recursos limitados. Se ha empleado en una variedad de estudios de bacterias, incluyendo (1) bacterias orales27, (2) otros tipos de muestras como alimentos y heces28, y (3) la identificación de bacterias no cultivables29, a menudo de acuerdo con otras técnicas moleculares. Entre las ventajas que ofrece la técnica de dot-blot se encuentran: (1) La membrana tiene una alta capacidad de unión, capaz de unir más de 200 μg/cm2 de ácidos nucleicos y hasta 400 μg/cm2; (2) Los resultados de la mancha se pueden interpretar visualmente sin necesidad de equipo especial, y (3) se pueden almacenar convenientemente durante años a temperatura ambiente (RT).
El género Leptospira ha sido clasificado en clados patógenos, intermedios y saprófitos30,31. La distinción entre estos clados se puede lograr en función de genes específicos como lipL41, lipL32 y el ARNr 16S. LipL32 está presente en los clados patógenos y exhibe una alta sensibilidad en diversas herramientas serológicas y moleculares, mientras que está ausente en las especies de saprófitos21. El gen de mantenimiento lipL41 es conocido por su expresión estable y se utiliza en técnicas moleculares32, mientras que el gen 16S rRNA se utiliza para su clasificación.
Esta metodología se puede aplicar a grandes volúmenes de agua una vez que se han concentrado por centrifugación. Permite la evaluación de diversos puntos y profundidades dentro de un cuerpo de agua para detectar la presencia de ADN leptoespiral y el clado al que pertenece. Esta herramienta es valiosa tanto para fines ecológicos como para fines generales de detección y también se puede emplear para detectar otras bacterias no cultivables que pueden estar presentes en el agua.
Además, los ensayos de PCR y dot-blot son técnica y económicamente asequibles para una amplia gama de laboratorios, incluso para aquellos que carecen de equipos sofisticados o costosos. Este estudio tiene como objetivo aplicar el dot-blot basado en digoxigenina para la identificación de los tres clados de Leptospira en muestras de agua recolectadas de cuerpos de agua naturales.
Cepas bacterianas
Se incluyeron en este estudio doce serovares de Leptospira (Autumnalis, Bataviae, Bratislava, Canicola, Celledoni, Grippothyphosa, Hardjoprajitno, Icterohaemorrhagiae, Pomona, Pyrogenes, Tarassovi y Wolffi). Estos serovares forman parte de la colección del Departamento de Microbiología e Inmunología de la Facultad de Medicina Veterinaria y Zootecnia de la Universidad Nacional Autónoma de México, y actualmente se utilizan en el ensayo de microaglutinación (MAT).
Todos los serovares de Leptospira se cultivaron en EMJH y su ADN se extrajo utilizando un kit comercial de extracción de ADN (ver Tabla de Materiales). Se utilizó una mezcla de ADN genómico de los doce serovares como control positivo para el clado patógeno de Leptostpira . Como control positivo del clado intermedio de Leptospira , se incluyó ADN genómico de Leptospira fainei serovar cepa BUT6 de Hurstbridge, y como control positivo para el clado de saprófitos de Leptospira , también se incluyó ADN genómico de Leptospira biflexa serovar cepa Patoc Patoc I.
Los controles negativos consistieron en un plásmido vacío, ADN de bacterias no relacionadas (Ureaplasma urealyticum, Staphylococcus aureus, Brucella abortus, Salmonella typhimurium, Shigella boydii, Klebsiella pneumoniae, Acinetobacter baumannii y Escherichia coli) y agua de grado PCR, que sirvió como control no molde.
Muestras de agua
Se recolectaron doce muestras de prueba utilizando un método de muestreo estratificado-aleatorio del Centro de Investigación Biológica y Acuícola de Cuemanco (CIBAC) (19° 16' 54" N 99° 6' 11" W). Estas muestras se obtuvieron a tres profundidades: superficial, 10 y 30 cm (Figura 1A, B). Los procedimientos de recolección de agua no afectaron a ninguna especie en peligro de extinción o protegida. Cada muestra se recogió en un tubo de microcentrífuga estéril de 15 mL. Para recolectar la muestra, cada tubo se sumergió suavemente en el agua, se llenó a la profundidad seleccionada y luego se selló. Las muestras se mantuvieron a 22 °C y se transportaron rápidamente al laboratorio para su procesamiento.
Cada muestra se concentró por centrifugación en tubos de microcentrífuga estériles de 1,5 mL a 8000 x g durante 20 min a temperatura ambiente. Este paso se repitió hasta que todas las muestras se concentraron en un tubo, que luego se utilizó para la extracción de ADN (Figura 1C).
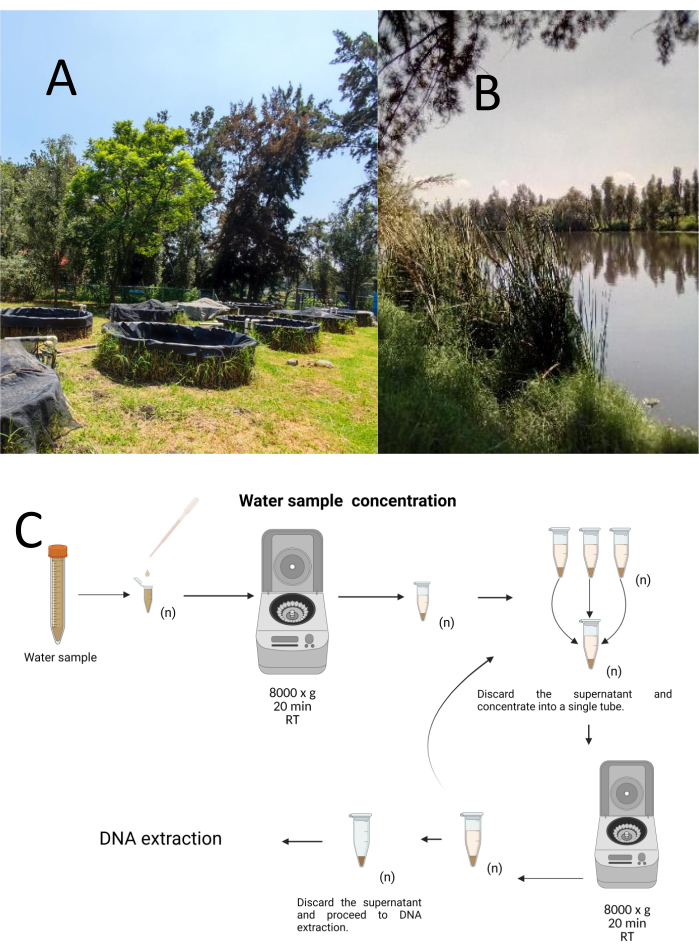
Figura 1: Concentración de muestras de agua por centrifugación. (A) Estanques de muestreo de agua, y (B) Arroyos naturales. (C) Procesamiento de muestras de agua basado en centrifugación en pasos repetidos tantas veces como sea necesario (n). Haga clic aquí para ver una versión más grande de esta figura.
Extracción de ADN
El ADN total se aisló utilizando un kit comercial de ADN genómico de acuerdo con las instrucciones del fabricante (ver Tabla de Materiales). Las extracciones de ADN se eluyeron en 20 μL de tampón de elución, y la concentración de ADN se determinó mediante un espectrofotómetro UV a 260-280 nm, y se almacenó a 4 °C hasta su uso.
Amplificación por PCR
Los objetivos de PCR fueron los genes 16S rRNA, lipL41 y lipL32, que identifican el ADN del género Leptospira y permiten distinguir entre los tres clados: patógeno, saprófito e intermedio. Tanto los cebadores como los diseños de las sondas se basaron en los trabajos previos de Ahmed et al., Azali et al., Bourhy et al., Weiss et al. y Branger et al.33,34,35,36,37. La secuencia de cada sonda, cebador y fragmento amplificado se describe en la Tabla 1, y su alineación con las secuencias de referencia se proporciona en el Archivo Suplementario 1, el Archivo Suplementario 2, el Archivo Suplementario 3, el Archivo Suplementario 4 y el Archivo Suplementario 5. Los reactivos de PCR y las condiciones de termociclado se describen en la sección de protocolo.
Los productos de amplificación se visualizaron por separación electroforética en un gel de agarosa al 1% en TAE (40 mM de base Tris, 20 mM de ácido acético y 1 mM de EDTA; pH 8,3), a 60 V durante 45 min con detección de bromuro de etidio, como se muestra en la Figura suplementaria 1. El ADN genómico obtenido de cada serovar se utilizó con concentraciones que oscilaron entre 6 x 106 y 1 x 104 copias genómicas equivalentes (GEq) en cada reacción de PCR, basándose en el tamaño del genoma de L. interrogans (4, 691, 184 pb)38 para Leptospira patógena, el tamaño del genoma de L. biflexa (3, 956, 088 pb)39 para Leptospira saprófita, y el tamaño del genoma de L. fainei serovar Hurstbridge cepa BUT6 (4, 267, 324 pb) con número de acceso AKWZ00000000.2.
La sensibilidad de las sondas se evaluó con ADN de cada serovar patógeno, L. biflexa serovar, cepa Patoc I, y L. fainei serovar, cepa BUT6 de Hurstbridge. Para evaluar la especificidad del ensayo de PCR e hibridación dot-blot, se incluyó ADN de bacterias no relacionadas.
Tabla 1: Cebadores y sondas de PCR para amplificar productos para la identificación de los clados patógenos, saprófitos e intermedios de Leptospira. Haga clic aquí para descargar esta tabla.
Ensayo de hibridación dot-blot
La técnica se denomina dot-blot porque los orificios en los que se coloca la muestra de ADN tienen forma de punto, y cuando se succionan para ser fijados en su lugar por succión al vacío, adquieren esta forma. Esta técnica fue desarrollada por Kafatos et al.40. La técnica permite la semicuantificación de Leptospira en cada muestra PCR positiva. El protocolo consiste en una desnaturalización con NaOH 0,4 M a temperatura ambiente, muestras con ADN de Leptospira de 30 ng a 0,05 ng, correspondientes a 6 x 106 a 1 x 104 leptospiras, se secan en una membrana de nailon con un aparato de puntos de 96 pocillos. Después de la inmovilización, el ADN se une a la membrana mediante la exposición a 120 mJ de luz ultravioleta. Cada sonda de ADN se conjuga con digoxigenina-11 dUTP mediante una etapa de catálisis de transferasa terminal en el extremo 3' (la digoxigenina es un esteroide vegetal obtenido de Digitalis purpurea, utilizado como reportero41). Tras la hibridación rigurosa de la sonda de ADN marcada (50 pmol) a la temperatura específica en el ADN objetivo, los híbridos de ADN se visualizan mediante la reacción de quimioluminiscencia con el anticuerpo anti-digoxigenina fosfatasa alcalina conjugado covalentemente con su sustrato CSPD. La luminiscencia se captura mediante la exposición a una película de rayos X (Figura 2).
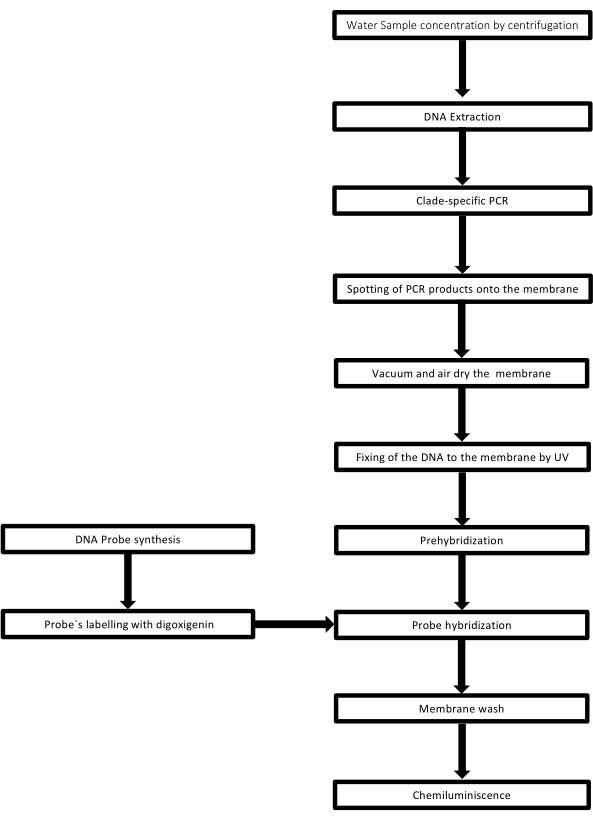
Figura 2: Pasos del procedimiento para el ensayo PCR-dot-blot. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
1. Preparación de la muestra
- Concentrar cada muestra de agua en tubos de microcentrífuga de 1,5 mL por centrifugación a 8.000 x g durante 10 min a 4 °C. Repita este paso, tantas veces como sea necesario, para concentrar la muestra en un volumen de 250 μL.
- Utilice el kit de extracción de ADN de acuerdo con las instrucciones del fabricante (consulte la tabla de materiales).
- Realizar la PCR específica según la sonda dot-blot que se vaya a utilizar (Tabla 1).
- Realizar las amplificaciones en tubos de PCR con un volumen final de 25 μL que contenga 1 X tampón, 2,5 unidades de Taq polimerasa, 1 μM de cada cebador, 0,2 mM de cada dNTP, 1,5 mM de MgCl2 y 100 ng de ADN objetivo de cada muestra de agua o ADN genómico de referencia.
- Programe la reacción de PCR en un termociclador de acuerdo con las condiciones de termociclado (Tabla 2).
- Conservar las reacciones a 4 °C hasta su uso.
- Coloque 10 μL de cada producto de PCR que se va a secar en un pocillo separado de una placa de 96 pocillos.
- Añadir 40 μL de TE a cada pocillo y mezclar mediante pipeteo.
NOTA: La placa de 96 pocillos se puede sellar y almacenar a 4 °C durante la noche. Siga las instrucciones proporcionadas en el Archivo Suplementario 6 para preparar búferes y soluciones para el protocolo. La figura 3 muestra los pasos de la preparación de la muestra.
Tabla 2: Condiciones de termociclado de la PCR para los genes 16S, lipL41 y lipL32. Haga clic aquí para descargar esta tabla.
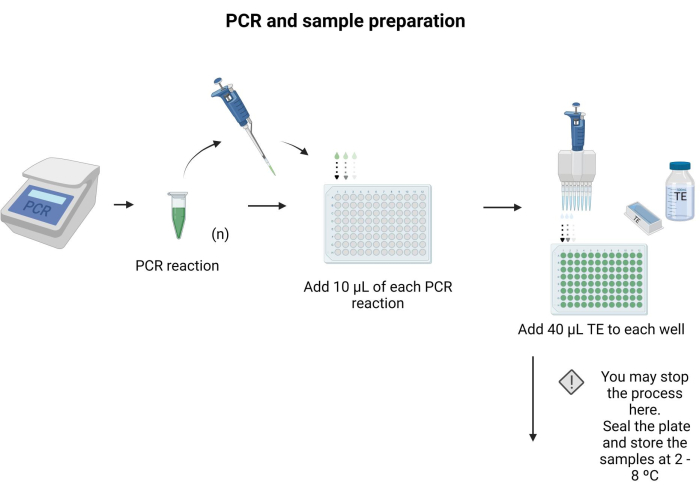
Figura 3: PCR y preparación de la muestra. Aplicando el protocolo específico de PCR, el producto de PCR se transfirió a una placa de microtitulación y se añadieron 40 μL de TE a cada pocillo. Haga clic aquí para ver una versión más grande de esta figura.
2. Montaje del aparato de mancha de puntos
NOTA: El montaje del aparato de punto y mancha se muestra en la Figura 4. Durante el procedimiento, use guantes para manipular las soluciones alcalinas y proteger la membrana de nailon de la contaminación.
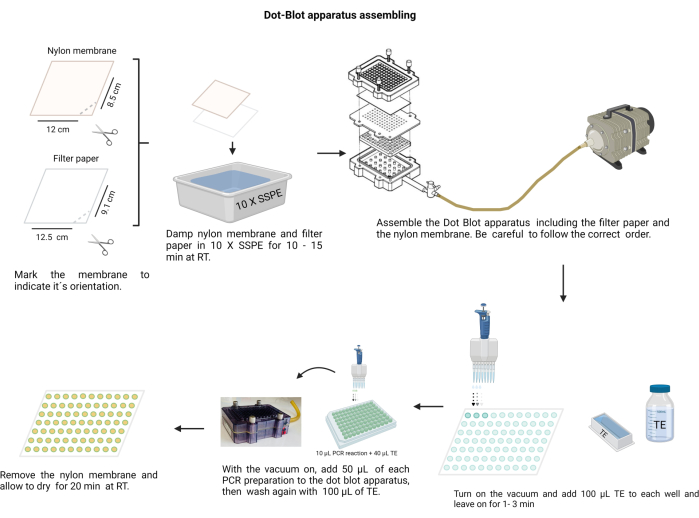
Figura 4: Montaje del aparato de mancha de puntos. El papel de filtro y la membrana de nylon (previamente humedecida en 10 X SSPE) deben estar dispuestos en el orden correcto. El conjunto debe asegurarse firmemente con los tornillos antes de aplicar la aspiradora. Cada pocillo debe lavarse con TE, y los productos de PCR se cargan en sus respectivos pocillos. Después de transferir el producto de PCR a través de la membrana, cada pocillo se lava nuevamente con TE y se deja secar. Haga clic aquí para ver una versión más grande de esta figura.
- Corta la membrana de nylon (ver Tabla de Materiales) y el papel de filtro en hojas de 12 x 8,5 cm.
- Marque la membrana con un marcador permanente.
- Haz una muesca con unas tijeras en un borde para indicar la orientación correcta. Utilice la marca para recordar el pedido de la muestra.
- Humedecer la membrana y el papel de filtro con tampón 10 X Solución salina-fosfato sódico-EDTA (SSPE; 3 M de NaCl, 0,2 M de NaH2PO4 y 0,02 M de EDTA, pH 7,4) (Archivo suplementario 6). Manipule la membrana con una pinza limpia y de punta roma.
- Ensamble la cámara de manchas de puntos; Primero, el papel de filtro, seguido de la membrana sobre el sello de plástico. Asegure la cubierta con los tornillos en forma transversal.
- Conecte la cámara a la aspiradora, coloque 100 μL de TE en cada pocillo, mantenga la aspiradora durante 1 minuto y luego deténgala.
- Encienda el vacío a baja velocidad y cargue 50 μL de cada muestra en el pocillo correspondiente de la membrana del aparato dot-blot (siguiendo la distribución de membrana preestablecida).
NOTA: Homogeneice cada muestra en la placa de hemaglutinación de 96 pocillos antes de colocarla en la cámara de transferencia de puntos. - Deje que la aspiradora seque la membrana. Si es necesario, golpee suavemente la cámara para liberar las burbujas de la muestra.
- Lave cada pocillo colocando 100 μL de TE en él con un vacío continuo y dejando que se seque.
NOTA: Después de completar la transferencia, es crucial apagar primero la bomba y desconectarla. De lo contrario, puede producirse la entrada de reflujo en el aparato. El ADN no necesita una etapa de desnaturalización si se utiliza una membrana de nailon cargada positivamente, pero si se utiliza otro soporte inerte, puede ser necesaria una etapa de predesnaturalización y una desnaturalización basada en álcalis41.
3. Desnaturalización y fijación del ADN
NOTA: La figura 5 ilustra el procedimiento de fijación de la membrana del ADN.
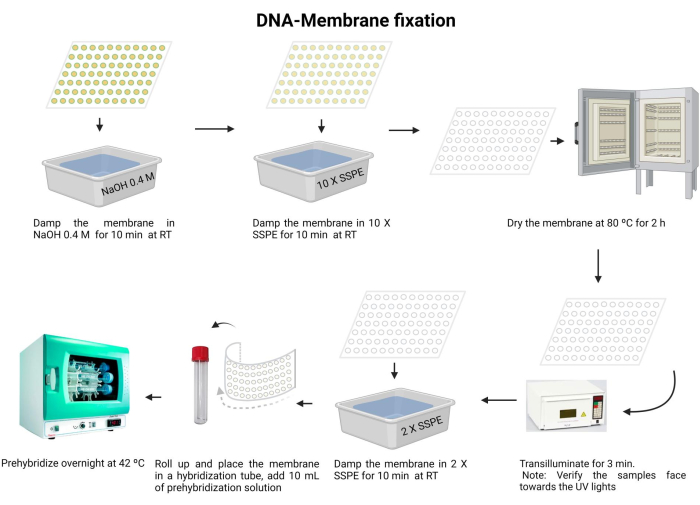
Figura 5: Procedimiento de fijación ADN-membrana. El ADN se desnaturaliza en una solución alcalina. A continuación, se neutraliza con 10 X SSPE y se seca la membrana. A continuación, se transilumina la membrana. La membrana se rehidrata con 2 X SSPE y se prehibrida durante la noche. Haga clic aquí para ver una versión más grande de esta figura.
- Incubar la membrana con NaOH 0,4 M durante 10 min a temperatura ambiente.
- Equilibre la membrana con 10 X SSPE durante 10 minutos a temperatura ambiente.
- Secar la membrana a 80 °C durante 2 h.
- Transilumina con luz UV de 120 mJ durante 3 min. Asegúrese de que las muestras estén orientadas boca abajo hacia la fuente de luz UV. Si utiliza un reticulante UV, confirme que las muestras estén orientadas hacia arriba y repita este paso dos veces.
NOTA: La membrana debe estar completamente seca antes de la reticulación UV. El ADN será inmovilizado por un enlace covalente a la membrana de nylon. Cada reticulación tarda aproximadamente 18'' a 1' con una dosis óptima de irradiación UV, para la mayoría de los experimentos de hibridación, de aproximadamente 0,6-0,8 kJ/m2.
La exposición a la radiación UV es perjudicial para los ojos y la piel. Use el equipo de protección adecuado y evite la exposición a la piel desnuda. - Lave la membrana con 2 X SSPE durante 10 min.
- Doble con cuidado la membrana e insértela en el tubo de hibridación (utilice un tubo de microcentrífuga de 15 ml).
- Añadir 10 mL de la solución de prehibridación (Ficha Suplementaria 6) al tubo e incubar a 42 °C durante la noche. Asegúrese de que el tubo esté correctamente sellado para evitar fugas.
4. Hibridación
- Retire 5 mL de la solución de prehibridación del tubo de hibridación. Para utilizar los 5 ml por segunda vez, guárdelo en otro tubo y guárdelo a -4 °C.
- Añadir 15 μL de la sonda marcada (Tabla 1) al tubo de hibridación. Enjuague la punta en la solución para asegurarse de que la sonda etiquetada se haya instilado completamente en la dilución.
NOTA: La unión de la sonda de ADN no es tan fuerte como la unión antígeno-anticuerpo. Por lo tanto, los cambios en la concentración de la sonda o el ADN en la muestra pueden influir en la intensidad de la fluorescencia emitida. Las sondas de ADN son estequiométricas, lo que significa que el número de moléculas de sonda unidas al ADN es equivalente al número de moléculas de ADN presentes en la solución. La figura 6 muestra la hibridación de la sonda. - Incubar a 42 °C durante la noche.
- Realice el etiquetado DIG de la sonda de ADN. Siga las instrucciones especificadas en la Tabla 3 para mezclar los reactivos, luego incube la mezcla a 37 °C durante 1 h. A continuación, añada 80 μL de agua destilada y guarde la sonda de cola a 4 °C.
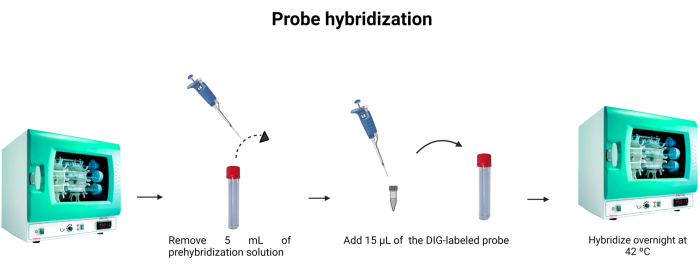
Figura 6: Hibridación de la sonda. Se ajusta el volumen del tampón de hibridación y se incorpora la sonda marcada con digoxigenina para permitir la hibridación de la sonda durante la noche. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 3: Reactivos para el marcaje de sondas con digoxigenina (DIG). Haga clic aquí para descargar esta tabla.
5. Quimioluminiscencia (etiquetado anti-DIG)
- Lave la membrana dos veces con 2 X SSPE/0.1% SDS a temperatura ambiente durante 10 min.
- Incubar la membrana con 5 X SSPE/0,1% SDS a la temperatura de recocido de la sonda durante 15 min.
NOTA: En este paso, use un recipiente con tapa para mantener la temperatura el mayor tiempo posible. Asegure una temperatura uniforme y constante en toda la membrana. El volumen aproximado en cada lavado es de 100 mL de la solución indicada. - Lave la membrana una vez con el tampón 1 (Archivo suplementario 6) a temperatura ambiente durante 5 min.
- Lave la membrana una vez con el tampón 2 (Archivo suplementario 6) a temperatura ambiente durante 30 min.
- Lavar la membrana con tampón 2 y añadir el anticuerpo anti-digoxigenina (15 μL) (ver Tabla de Materiales), luego incubar durante 45 min (el tiempo de incubación puede variar de 30-60 min). Este anticuerpo secundario marca la sonda para su detección.
NOTA: La membrana puede almacenarse en el tampón 2 con el anticuerpo anti-digoxigenina durante la noche a 4-8 °C. El marcaje anti-DIG del proceso de quimioluminiscencia se muestra en la Figura 7.
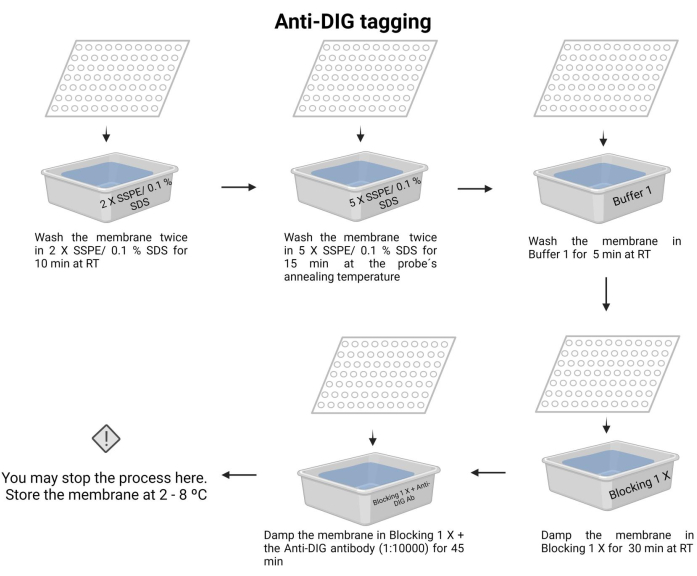
Figura 7: Marcaje anti-DIG del proceso de quimioluminiscencia. Los ácidos nucleicos no unidos se eliminan con soluciones tampón. La sonda se alinea con el ADN objetivo y se elimina el exceso. La membrana se bloquea con el tampón de bloqueo 1 X y se añade el anticuerpo anti-DIG (1:10000). Haga clic aquí para ver una versión más grande de esta figura.
6. Quimioluminiscencia (aplicación de sustrato)
- Lave la membrana dos veces en el tampón 1 a temperatura ambiente durante 15 min.
- Lave una vez en el tampón 3 (consulte la ficha complementaria 6) a temperatura ambiente durante 5 min, permitiendo que la membrana drene la mayor parte del tampón, dejándolo casi seco.
- Añadir CSPD listo para usar (3-(4-metoxispiro {1,2-dioxetano-3,2′-(5′-cloro) triciclo, [3.3.1.13,7] decan}-4-il) fenilo fosfato, ver Tabla de Materiales), y dejar reposar durante 5 min, (con los dedos humedecidos homogeneizar la CSPD de un lado a otro).
- Coloque la membrana en una bolsa de plástico transparente (12,5 x 9 cm) que se ajuste a la dimensión de la membrana. Retire con cuidado las burbujas de aire, distribuya uniformemente el CSPD y selle la bolsa con calor.
- Incubar la membrana en un baño de agua a 37 °C durante 15 min (puede ser hasta 30 min), y asegurarse de que la membrana con muestras esté colocada hacia abajo para que quede bien sumergida. Asegúrese de que la temperatura permanezca constante y bien distribuida sobre la membrana.
- Seque la bolsa de plástico y fíjela con cinta adhesiva sobre una película radiográfica ya usada. Esto permitirá un fácil manejo durante el procedimiento de exposición.
NOTA: La Figura 8 muestra la aplicación del sustrato del proceso de quimioluminiscencia.

Figura 8: Aplicación de sustrato del proceso de quimioluminiscencia. Se elimina el anticuerpo libre y se añade el sustrato CSPD (1:250) a la membrana. La reacción se activa por incubación a 37 °C y la membrana está dispuesta para registrar la quimioluminiscencia en una película de rayos X. Haga clic aquí para ver una versión más grande de esta figura.
7. Quimioluminiscencia (detección)
- Realice el procedimiento de exposición. En el cuarto oscuro, prepare las soluciones de revelador y fijador (consulte la Tabla de materiales) en las bandejas respectivas.
- En la oscuridad, coloque cuidadosamente la membrana fija frente a una nueva película radiográfica e insértela en un casete radiográfico (consulte la Tabla de Materiales).
- Registre el tiempo de exposición. El tiempo de exposición puede variar de 1 min a 30 min o más. Comience con 5 minutos y ajuste el tiempo en consecuencia.
- Humedecer la película de rayos X en solución reveladora durante 1-3 min y enjuagar suavemente con agua del grifo (15-45 s).
- Humedecer la película de rayos X en solución fijadora durante 1-3 min y enjuagar suavemente con agua del grifo (45 s).
- Deje que la película de rayos X se seque al aire y documente el ensayo en una caja de luz blanca visible.
NOTA: Evite agitar la membrana durante el tiempo de exposición a los rayos X. El proceso de detección se muestra en la Figura 9. - Proceda a la interpretación del resultado. En la película de rayos X expuesta, los puntos con emisión se pueden localizar visualmente y corresponden a los fragmentos de ADN con la hibridación de la sonda. La intensidad de la señal emitida depende de la luminiscencia y de la duración de la exposición.
NOTA: Las membranas se pueden almacenar secas entre papel de filtro durante varios meses a temperatura ambiente.
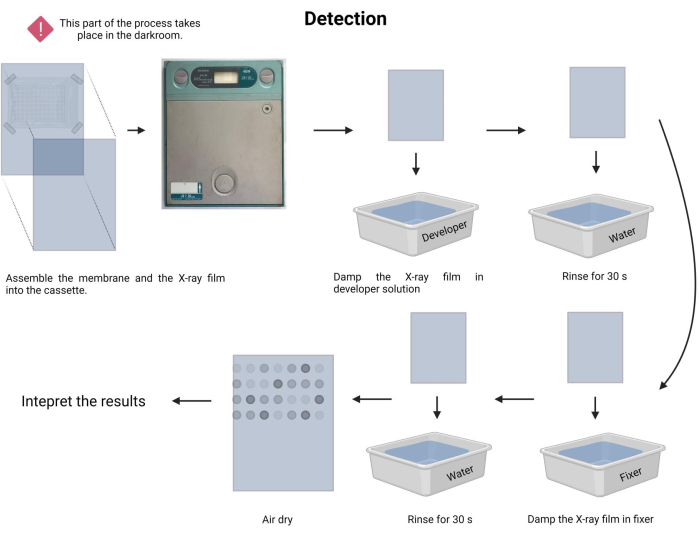
Figura 9: Detección del proceso de quimioluminiscencia. En condiciones de oscuridad, la membrana se expone a una película de rayos X dentro de un casete de rayos X. A continuación, se dejó reposar durante el tiempo de exposición, y luego se reveló y fijó la película de rayos X. Finalmente, se secó al aire y se interpretó. Haga clic aquí para ver una versión más grande de esta figura.
8. Procedimiento de deshibridación de membranas
- Lave la membrana dos veces durante 10 minutos con agua destilada.
- Lavar la membrana durante 20 min con NaOH 0,4 M a 53 °C (dos veces).
- Lave dos veces la membrana durante 10 minutos con 2 X SSPE.
- Deje que la membrana se seque a temperatura ambiente.
- Mantenga la membrana y vuelva al paso 3.5 del protocolo.
NOTA: La membrana puede almacenarse en el tampón de prehibridación durante la noche a 4-8 °C. Al día siguiente, reiniciar con una incubación a 42 °C durante 1 h, permitiendo que el tampón de prehibridación alcance la temperatura. A continuación, continúe con el paso 3.5 del protocolo.
Resultados
Para evaluar la efectividad de la técnica, se utilizó ADN genómico de cultivos puros de cada Leptospira serovar, junto con la sonda específica del clado. Las membranas se prepararon con 100 ng de ADN genómico por reacción de PCR para cada serovar, seguido de ocho ADN genómico de bacterias no relacionadas y concentraciones variables de ADN genómico de los serovares ad hoc de Leptospira. Cada ensayo incluyó control positivo, negativo y no especial. Este ADN genómico no relacionado no mostró afi...
Discusión
Los pasos críticos de la técnica de dot-blot incluyen (1) la inmovilización del ADN, (2) el bloqueo de los sitios de unión libres en la membrana con ADN no homólogo, (3) la complementariedad entre la sonda y el fragmento objetivo en condiciones de recocido, (4) la eliminación de la sonda no hibridada y (5) la detección de la molécula reportera41.
El PCR-Dot-blot tiene ciertas limitaciones, como que la técnica no proporciona información sobre el tamaño del fra...
Divulgaciones
Los autores declaran que no existe conflicto de intereses.
Agradecimientos
Estamos en deuda con la colección Leptospira del Departamento de Microbiología e Inmunología de la Facultad de Medicina Veterinaria y Zootecnia de la Universidad Nacional Autónoma de México. Agradecemos la generosa donación de las cepas de referencia de Leptuspira ; Leptospira fainei serovar Hurstbridge cepa BUT6 y Leptospira biflexa serovar Patoc cepa Patoc I al Dr. Alejandro de la Peña Moctezuma. Agradecemos al Dr. José Antonio Ocampo Cervantes, Coordinador del CIBAC, y al personal por su apoyo logístico. EDT se enmarcó en el programa Proyecto Terminal para estudiantes de pregrado de la Universidad Autónoma Metropolitana-Campus Cuajimalpa. Reconocemos el software Biorender.com para la creación de las figuras 1 y 3 a 9.
Materiales
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| Purelink DNA extraction kit | Invitrogen | K182002 | |
| Gotaq Flexi DNA Polimerase (End-Point PCR Taq polymerase kit) | Promega | M3001 | |
| Whatman filter paper, grade 1, | Merk | WHA1001325 | |
| Nylon Membranes, positively charged Roll 30cm x 3 m | Roche | 11417240001 | |
| Anti-Digoxigenin-AP, Fab fragments Sheep Polyclonal Primary-antibody | Roche | 11093274910 | |
| Medium Base EMJH | Difco | S1368JAA | |
| Leptospira Enrichment EMJH | Difco | BD 279510 | |
| Blocking Reagent | Roche | 11096176001 | |
| CSPD ready to use Disodium 3-(4-methoxyspiro {1,2-dioxetane-3,2′-(5′-chloro) tricyclo [3.3.1.13,7] decan}8-4-yl) phenyl phosphate | Merk | 11755633001 | |
| Deoxyribonucleic acid from herring sperm | Sigma Aldrich | D3159 | |
| Developer Carestream | Carestream Health Inc | GBX5158621 | |
| Digoxigenin-11-ddUTP | Roche | 11363905910 | |
| EDTA, Disodium Salt (Dihydrate) | Promega | H5032 | |
| Ficoll 400 | Sigma Aldrich | F8016 | |
| Fixer Carestream | Carestream Health Inc | GBX 5158605 | |
| Lauryl sulfate Sodium Salt (Sodium dodecyl sulfate; SDS) C12H2504SNa | Sigma Aldrich | L5750 | |
| N- Lauroylsarcosine sodium salt CH3(CH2)10CON(CH3) CH2COONa | Sigma Aldrich | L-9150 | It is an anionic surfactant |
| Polivinylpyrrolidone (PVP-40) | Sigma Aldrich | PVP40 | |
| Polyethylene glycol Sorbitan monolaurate (Tween 20) | Sigma Aldrich | 9005-64-5 | |
| Sodium Chloride (NaCl) | Sigma Aldrich | 7647-14-5 | |
| Sodium dodecyl sulfate (SDS) | Sigma Aldrich | 151-21-3 | |
| Sodium hydroxide (NaOH) | Sigma Aldrich | 1310-73-2 | |
| Sodium phosphate dibasic (NaH2PO4) | Sigma-Aldrich | 7558-79-4 | |
| Terminal transferase, recombinant | Roche | 3289869103 | |
| Tris hydrochloride (Tris HCl) | Sigma-Aldrich | 1185-53-1 | |
| SSPE 20X | Sigma-Aldrich | S2015-1L | It can be Home-made following Supplementary File 6 |
| Primers | Sigma-Aldrich | On demand | Follow table 1 |
| Probes | Sigma-Aldrich | On demand | Follow table 1 |
| Equipment | |||
| Nanodrop™ One Spectrophotometer | Thermo-Scientific | ND-ONE-W | |
| Refrigerated microcentrifuge Sigma 1-14K, suitable for centrifugation of 1.5 ml microcentrifuge tubes at 14,000 rpm | Sigma-Aldrich | 1-14K | |
| Disinfected adjustable pipettes, range 2-20 µl, 20-200 µl | Gilson | SKU:F167360 | |
| Disposable 1.5 ml microcentrifuge tubes (autoclaved) | Axygen | MCT-150-SP | |
| Disposable 600 µl microcentrifuge tubes (autoclaved) | Axygen | 3208 | |
| Disposable Pipette tips 1-10 µl | Axygen | T-300 | |
| Disposable Pipette tips 1-200 µl | Axygen | TR-222-Y | |
| Dot-Blot apparatus Bio-Dot | BIORAD | 1706545 | |
| Portable Hergom Suction | Hergom | 7E-A | |
| Scientific Light Box (Visible-light PH90-115V) | Hoefer | PH90-115V | |
| UV Crosslinker | Hoefer | UVC-500 | |
| Thermo Hybaid PCR Express Thermocycler | Hybaid | HBPX110 | |
| Radiographic cassette with IP Plate14 X 17 | Fuji |
Referencias
- Bierque, E., Thibeaux, R., Girault, D., Soupé-Gilbert, M. E., Goarant, C. A systematic review of Leptospira in water and soil environments. PLOS One. 15 (1), e0227055 (2020).
- Haake, D. A., Levett, P. N. Leptospirosis in humans. Current Topics in Microbiology and Immunology. 387, 65-97 (2015).
- Tripathy, D. N., Hanson, L. E. Leptospires from water sources at Dixon Springs Agricultural Center. Journal of Wildlife Diseases. 9 (3), 209-212 (1973).
- Smith, D. J., Self, H. R. Observations on the survival of Leptospira australis A in soil and water. The Journal of Hygiene. 53 (4), 436-444 (1955).
- Karpagam, K. B., Ganesh, B. Leptospirosis: a neglected tropical zoonotic infection of public health importance-an updated review. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 39 (5), 835-846 (2020).
- Casanovas-Massana, A., et al. Spatial and temporal dynamics of pathogenic Leptospira in surface waters from the urban slum environment. Water Research. 130, 176-184 (2018).
- Costa, F., et al. Global morbidity and mortality of Leptospirosis: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003898 (2015).
- Mwachui, M. A., Crump, L., Hartskeerl, R., Zinsstag, J., Hattendorf, J. Environmental and behavioural determinants of Leptospirosis transmission: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003843 (2015).
- Andre-Fontaine, G., Aviat, F., Thorin, C. Waterborne Leptospirosis: Survival and preservation of the virulence of pathogenic Leptospira spp. in fresh water. Current Microbiology. 71 (1), 136-142 (2015).
- Trueba, G., Zapata, S., Madrid, K., Cullen, P., Haake, D. Cell aggregation: A mechanism of pathogenic Leptospira to survive in freshwater. International Microbiology: the Official Journal of the Spanish Society for Microbiology. 7 (1), 35-40 (2004).
- Smith, C. E., Turner, L. H. The effect of pH on the survival of leptospires in water. Bulletin of the World Health Organization. 24 (1), 35-43 (1961).
- Barragan, V. A., et al. Interactions of Leptospira with environmental bacteria from surface water. Current Microbiology. 62 (6), 1802-1806 (2011).
- Abdoelrachman, R. Comparative investigations into the influence of the presence of bacteria on the life of pathogenic and apathogenic leptospirae. Antonie van Leeuwenhoek. 13 (1), 21-32 (1947).
- Singh, R., et al. Microbial diversity of biofilms in dental unit water systems. Applied and Environmental Microbiology. 69 (6), 3412-3420 (2003).
- Kumar, K. V., Lall, C., Raj, R. V., Vedhagiri, K., Vijayachari, P. Coexistence and survival of pathogenic leptospires by formation of biofilm with Azospirillum. FEMS Microbiology Ecology. 91 (6), 051 (2015).
- Yanagihara, Y., et al. Leptospira Is an environmental bacterium that grows in waterlogged soil. Microbiology Spectrum. 10 (2), 0215721 (2022).
- Gillespie, R. W., Ryno, J. Epidemiology of leptospirosis. American Journal of Public Health and Nation’s Health. 53 (6), 950-955 (1963).
- Bierque, E., et al. Leptospira interrogans retains direct virulence after long starvation in water. Current Microbiology. 77 (10), 3035-3043 (2020).
- Zhang, Y., Dai, B. Marking and detection of DNA of leptospires in the dot-blot and situ hybridization with digoxigenin-labeled probes. Journal of West China University of Medical Sciences. 23 (4), 353-435 (1992).
- Mérien, F., Amouriaux, P., Perolat, P., Baranton, G., Saint Girons, I. Polymerase chain reaction for detection of Leptospira spp. in clinical samples. Journal of Clinical Microbiology. 30 (9), 2219-2224 (1992).
- Veerapandian, R., et al. Silver enhanced nano-gold dot-blot immunoassay for leptospirosis. Journal of Microbiological Methods. 156, 20-22 (2019).
- Junpen, S., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Ishmael, F. T., Stellato, C. Principles and applications of polymerase chain reaction: basic science for the practicing physician. Annals of Allergy, Asthma & Immunology: Official Publication of the American College of Allergy, Asthma, & Immunology. 101 (4), 437-443 (2008).
- Boerner, B., Weigelt, W., Buhk, H. J., Castrucci, G., Ludwig, H. A sensitive and specific PCR/Southern blot assay for detection of bovine herpesvirus 4 in calves infected experimentally. Journal of Virological Methods. 83 (1-2), 169-180 (1999).
- Curry, E., Pratt, S. L., Kelley, D. E., Lapin, D. R., Gibbons, J. R. Use of a Combined duplex PCR/Dot-blot assay for more sensitive genetic characterization. Biochemistry Insights. 1, 35-39 (2008).
- Pilatti, M. M., Ferreira, S. d. e. A., de Melo, M. N., de Andrade, A. S. Comparison of PCR methods for diagnosis of canine visceral leishmaniasis in conjunctival swab samples. Research in Veterinary Science. 87 (2), 255-257 (2009).
- Conrads, G., et al. PCR reaction and dot-blot hybridization to monitor the distribution of oral pathogens within plaque samples of periodontally healthy individuals. Journal of Periodontology. 67 (10), 994-1003 (1996).
- Langa, S., et al. Differentiation of Enterococcus faecium from Lactobacillus delbrueckii subsp. bulgaricus and Streptococcus thermophilus strains by PCR and dot-blot hybridisation. International Journal of Food Microbiology. 88 (2-3), 197-200 (2003).
- Francesca, C., Lucilla, I., Marco, F., Giuseppe, C., Marisa, M. Identification of the unculturable bacteria Candidatus arthromitus in the intestinal content of trouts using dot-blot and Southern blot techniques. Veterinary Microbiology. 156 (3-4), 389-394 (2012).
- Arent, Z., Pardyak, L., Dubniewicz, K., Plachno, B. J., Kotula-Balak, M. Leptospira taxonomy: then and now. Medycyna Weterynaryjna. 78 (10), 489-496 (2022).
- Thibeaux, R., et al. Biodiversity of environmental Leptospira: Improving identification and revisiting the diagnosis. Frontiers in Microbiology. 9, 816 (2018).
- Carrillo-Casas, E. M., Hernández-Castro, R., Suárez-Güemes, F., de la Peña-Moctezuma, A. Selection of the internal control gene for real-time quantitative RT-PCR assays in temperature treated Leptospira. Current Microbiology. 56 (6), 539-546 (2008).
- Azali, M. A., Yean Yean, C., Harun, A., Aminuddin Baki A, N. N., Ismail, N. Molecular characterization of Leptospira spp. in environmental samples from North-Eastern Malaysia revealed a pathogenic strain, Leptospira alstonii. Journal of Tropical Medicine. 2016, 2060241 (2016).
- Ahmed, N., et al. Multilocus sequence typing method for identification and genotypic classification of pathogenic Leptospira species. Annals of Clinical Microbiology and Antimicrobials. 5, 28 (2006).
- Bourhy, P., Collet, L., Brisse, S., Picardeau, M. Leptospira mayottensis sp. nov., a pathogenic species of the genus Leptospira isolated from humans. International Journal of Systematic and Evolutionary Microbiology. 64, 4061-4067 (2014).
- Weiss, S., et al. An extended Multilocus Sequence Typing (MLST) scheme for rapid direct typing of Leptospira from clinical samples. PLOS Neglected Tropical Diseases. 10 (9), e0004996 (2016).
- Branger, C., et al. Polymerase chain reaction assay specific for pathogenic Leptospira based on the gene hap1 encoding the hemolysis-associated protein-1. FEMS Microbiology Letters. 243 (2), 437-445 (2005).
- Ren, S. X., et al. Unique physiological and pathogenic features of Leptospira interrogans revealed by whole-genome sequencing. Nature. 422 (6934), 888-893 (2003).
- Picardeau, M., et al. Genome sequence of the saprophyte Leptospira biflexa provides insights into the evolution of Leptospira and the pathogenesis of leptospirosis. PLOS One. 3 (2), e1607 (2008).
- Kafatos, F. C., Jones, C. W., Efstratiadis, A. Determination of nucleic acid sequence homologies and relative concentrations by a dot hybridization procedure. Nucleic Acids Research. 7 (6), 1541-1552 (1979).
- Bhat, A. I., Rao, G. P. Dot-blot hybridization technique. Characterization of Plant Viruses. , 303-321 (2020).
- Yadav, J. P., Batra, K., Singh, Y., Singh, M. Comparative evaluation of indirect-ELISA and Dot-blot assay for serodetection of Mycoplasma gallisepticum and Mycoplasma synoviae antibodies in poultry. Journal of Microbiological Methods. 189, 106317 (2021).
- Malinen, E., Kassinen, A., Rinttilä, T., Palva, A. Comparison of real-time PCR with SYBR Green I or 5'-nuclease assays and dot-blot hybridization with rDNA-targeted oligonucleotide probes in quantification of selected faecal bacteria. Microbiology. 149, 269-277 (2003).
- Wyss, C., et al. Treponema lecithinolyticum sp. nov., a small saccharolytic spirochaete with phospholipase A and C activities associated with periodontal diseases. International Journal of Systematic Bacteriology. 49, 1329-1339 (1999).
- Shah, J. S., I, D. C., Ward, S., Harris, N. S., Ramasamy, R. Development of a sensitive PCR-dot-blot assay to supplement serological tests for diagnosing Lyme disease. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 37 (4), 701-709 (2018).
- Niu, C., Wang, S., Lu, C. Development and evaluation of a dot-blot assay for rapid determination of invasion-associated gene ibeA directly in fresh bacteria cultures of E. coli. Folia microbiologica. 57 (6), 557-561 (2012).
- Wetherall, B. L., McDonald, P. J., Johnson, A. M. Detection of Campylobacter pylori DNA by hybridization with non-radioactive probes in comparison with a 32P-labeled probe. Journal of Medical Microbiology. 26 (4), 257-263 (1988).
- Kolk, A. H., et al. Detection of Mycobacterium tuberculosis in clinical samples by using polymerase chain reaction and a nonradioactive detection system. Journal of Clinical Microbiology. 30 (10), 2567-2575 (1992).
- Scherer, L. C., et al. PCR colorimetric dot-blot assay and clinical pretest probability for diagnosis of Pulmonary Tuberculosis in smear-negative patients. BMC Public Health. 7, 356 (2007).
- Armbruster, D. A., Pry, T. Limit of blank, limit of detection and limit of quantitation. The Clinical Biochemist Reviews. 29, S49-S52 (2008).
- Zhang, Y., Dai, B. Detection of Leptospira by dot-blot hybridization with photobiotin- and 32P-labeled DNA. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 130-132 (1992).
- Terpstra, W. J., Schoone, G. J., ter Schegget, J. Detection of leptospiral DNA by nucleic acid hybridization with 32P- and biotin-labeled probes. Journal of Medical Microbiology. 22 (1), 23-28 (1986).
- Shukla, J., Tuteja, U., Batra, H. V. DNA probes for identification of leptospires and disease diagnosis. The Southeast Asian Journal of Tropical Medicine and Public Health. 35 (2), 346-352 (2004).
- Jiang, N., Jin, B., Dai, B., Zhang, Y. Identification of pathogenic and nonpathogenic leptospires by recombinant probes. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 26 (1), 1-5 (1995).
- Fach, P., Trap, D., Guillou, J. P. Biotinylated probes to detect Leptospira interrogans on dot-blot hybridization or by in situ hybridization. Letters in Applied Microbiology. 12 (5), 171-176 (1991).
- Huang, N., Dai, B. Assay of genomic DNA homology among strains of different virulent leptospira by DNA hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 122-125 (1992).
- Dong, X., Dai, B., Chai, J. Homology study of leptospires by molecular hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (1), 1-4 (1992).
- Komminoth, P. Digoxigenin as an alternative probe labeling for in situ hybridization. Diagnostic Molecular Pathology: The American Journal of Surgical Pathology, part B. 1 (2), 142-150 (1992).
- Saengjaruk, P., et al. Diagnosis of human leptospirosis by monoclonal antibody-based antigen detection in urine. Journal of Clinical Microbiology. 40 (2), 480-489 (2002).
- Okuda, M., et al. Enzyme-linked immunosorbent assay for the detection of canine Leptospira antibodies using recombinant OmpL1 protein. The Journal of Veterinary Medical Science. 67 (3), 249-254 (2005).
- Suwimonteerabutr, J., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Kanagavel, M., et al. Peptide-specific monoclonal antibodies of Leptospiral LigA for acute diagnosis of leptospirosis. Scientific reports. 7 (1), 3250 (2017).
- Levett, P. N. Leptospirosis. Clinical Microbiology Reviews. 14 (2), 296-326 (2001).
- Monahan, A. M., Callanan, J. J., Nally, J. E. Proteomic analysis of Leptospira interrogans shed in urine of chronically infected hosts. Infection and Immunity. 76 (11), 4952-4958 (2008).
- Rojas, P., et al. Detection and quantification of leptospires in urine of dogs: a maintenance host for the zoonotic disease leptospirosis. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 29 (10), 1305-1309 (2010).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados