È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Reazione a catena della polimerasi e ibridazione dot-blot per la rilevazione di leptospira in campioni d'acqua
In questo articolo
Riepilogo
In questo studio, è stata progettata un'applicazione di dot-blot per rilevare Leptospira dai tre cladi principali nei campioni d'acqua. Questo metodo consente l'identificazione di quantità minime di DNA specificamente bersaglio di una sonda marcata con digossigenina, facilmente rilevabili da un anticorpo anti-digossigenina. Questo approccio è uno strumento prezioso e soddisfacente ai fini dello screening.
Abstract
Il dot-blot è una tecnica semplice, veloce, sensibile e versatile che consente l'identificazione di quantità minime di DNA specificamente bersaglio dell'ibridazione della sonda in presenza di DNA vettore. Si basa sul trasferimento di una quantità nota di DNA su un supporto solido inerte, come una membrana di nylon, utilizzando l'apparato dot-blot e senza separazione elettroforetica. Le membrane in nylon hanno il vantaggio di un'elevata capacità di legame dell'acido nucleico (400 μg/cm2), un'elevata resistenza e sono caricate positivamente o neutramente. La sonda utilizzata è un frammento di ssDNA altamente specifico di 18-20 basi lunghe marcate con digossigenina (DIG). La sonda si coniugherà con il DNA di Leptospira . Una volta che la sonda si è ibridata con il DNA bersaglio, viene rilevata da un anticorpo anti-digossigenina, consentendone una facile rilevazione attraverso le sue emissioni rivelate in una pellicola radiografica. I punti con un'emissione corrisponderanno ai frammenti di DNA di interesse. Questo metodo impiega la marcatura non isotopica della sonda, che può avere un'emivita molto lunga. Lo svantaggio di questa immuno-marcatura standard è una sensibilità inferiore rispetto alle sonde isotopiche. Tuttavia, è mitigato dall'accoppiamento della reazione a catena della polimerasi (PCR) e dai saggi dot-blot. Questo approccio consente l'arricchimento della sequenza di destinazione e il relativo rilevamento. Inoltre, può essere utilizzato come applicazione quantitativa se confrontato con una diluizione seriale di uno standard noto. Qui viene presentata un'applicazione dot-blot per rilevare Leptospira dai tre cladi principali nei campioni d'acqua. Questa metodologia può essere applicata a grandi quantità di acqua una volta che sono state concentrate mediante centrifugazione per fornire prove della presenza di DNA leptospirale. Questo è uno strumento prezioso e soddisfacente per scopi di screening generali e può essere utilizzato per altri batteri non coltivabili che possono essere presenti nell'acqua, migliorando la comprensione dell'ecosistema.
Introduzione
La leptospirosi nell'uomo ha origine principalmente da fonti ambientali 1,2. La presenza di Leptospira in laghi, fiumi e torrenti è un indicatore della trasmissione della leptospirosi tra la fauna selvatica e gli animali domestici e da produzione che possono eventualmente entrare in contatto con questi corpi idrici 1,3,4. Inoltre, Leptospira è stata identificata in fonti non naturali, tra cui acque reflue, stagnanti e di rubinetto 5,6.
Leptospira è un batterio distribuito in tutto il mondo 7,8 e il ruolo dell'ambiente nella sua conservazione e trasmissione è stato ben riconosciuto. La Leptospira può sopravvivere nell'acqua potabile a pH e minerali variabili9 e nei corpi idrici naturali1. Può anche sopravvivere per lunghi periodi in acqua distillata10 e, a pH costante (7,8), può sopravvivere fino a 152 giorni11. Inoltre, Leptospira può interagire in consorzi batterici per sopravvivere a condizioni difficili12,13. Può far parte di biofilm in acqua dolce con Azospirillum e Sphingomonas ed è anche in grado di crescere e sopportare temperature superiori a 49 °C14,15. Può anche moltiplicarsi in terreni impregnati d'acqua e rimanere vitale fino a 379 giorni16, preservando la sua capacità di causare la malattia fino a un anno17,18. Tuttavia, si sa poco sull'ecologia all'interno dei corpi idrici e su come è distribuita al loro interno.
Fin dalla sua scoperta, lo studio del genere Leptospira si è basato su test sierologici. Non è stato fino al secolo in corso che le tecniche molecolari sono diventate più diffuse nello studio di questa spirocheta. Il dot-blot è stato scarsamente utilizzato per la sua identificazione utilizzando (1) una sonda isotopica basata sull'rRNA 16S e su una ripetizione di sequenza intersemplice (ISSR)19,20, (2) come test immunologico basato su nanogold per la leptospirosi umana applicata all'urina21, o (3) come test basato su anticorpi per campioni di urina bovina22. La tecnica cadde in disuso perché originariamente si basava su sonde isotopiche. Tuttavia, è una tecnica ben nota che, abbinata alla PCR, produce risultati migliori ed è considerata sicura grazie all'uso di sonde non isotopiche. La PCR svolge un ruolo cruciale nell'arricchimento del DNA di Leptospira amplificando uno specifico frammento di DNA che può essere trovato in tracce in un campione. Durante ogni ciclo di PCR, la quantità del frammento di DNA bersaglio viene raddoppiata nella reazione. Alla fine della reazione, l'amplicone è stato moltiplicato per un fattore di oltre un milione23. Il prodotto amplificato mediante PCR, spesso non visibile nell'elettroforesi dell'agarosio, diventa visibile attraverso un'ibridazione specifica con una sonda marcata con DIG nel dot-blot 24,25,26.
La tecnica del dot-blot è semplice, robusta e adatta a numerosi campioni, rendendola accessibile ai laboratori con risorse limitate. È stato impiegato in una varietà di studi sui batteri, tra cui (1) batteri orali27, (2) altri tipi di campioni come cibo e feci28 e (3) l'identificazione di batteri non coltivabili29, spesso in accordo con altre tecniche molecolari. Tra i vantaggi offerti dalla tecnica del dot-blot ci sono: (1) La membrana ha un'elevata capacità legante, in grado di legare oltre 200 μg/cm2 di acidi nucleici e fino a 400 μg/cm2; (2) I risultati del dot-blot possono essere interpretati visivamente senza richiedere attrezzature speciali e (3) possono essere comodamente conservati per anni a temperatura ambiente (RT).
Il genere Leptospira è stato classificato in cladi patogeni, intermedi e saprofiti30,31. La distinzione tra questi cladi può essere ottenuta in base a geni specifici come lipL41, lipL32 e l'rRNA 16S. LipL32 è presente nei cladi patogeni e mostra un'elevata sensibilità in vari strumenti sierologici e molecolari, mentre è assente nelle specie di saprofiti21. Il gene housekeeping lipL41 è noto per la sua espressione stabile e utilizzato nelle tecniche molecolari32, mentre il gene 16S rRNA è utilizzato per la loro classificazione.
Questa metodologia può essere applicata a grandi volumi d'acqua una volta che sono stati concentrati per centrifugazione. Consente la valutazione di vari punti e profondità all'interno di un corpo idrico per rilevare la presenza di DNA leptospirale e del clade a cui appartiene. Questo strumento è prezioso sia per scopi ecologici che di screening generale e può essere utilizzato anche per rilevare altri batteri non coltivabili che possono essere presenti nell'acqua.
Inoltre, i test PCR e dot-blot sono tecnicamente ed economicamente accessibili a un'ampia gamma di laboratori, anche a quelli privi di attrezzature sofisticate o costose. Questo studio mira ad applicare il dot-blot a base di digoxigenina per l'identificazione dei tre cladi di Leptospira in campioni d'acqua raccolti da corpi idrici naturali.
Ceppi batterici
In questo studio sono stati inclusi dodici sierotipi di Leptospira (Autumnalis, Bataviae, Bratislava, Canicola, Celledoni, Grippothyphosa, Hardjoprajitno, Icterohaemorrhagiae, Pomona, Pyrogenes, Tarassovi e Wolffi). Questi sierotipi fanno parte della collezione del Dipartimento di Microbiologia e Immunologia, Facoltà di Medicina Veterinaria e Zootecnica, Università Nazionale Autonoma del Messico, e sono attualmente utilizzati nel test di microagglutinazione (MAT).
Tutti i sierotipi di Leptospira sono stati coltivati in EMJH e il loro DNA è stato estratto utilizzando un kit commerciale per l'estrazione del DNA (vedi Tabella dei materiali). Una miscela di DNA genomico dei dodici sierotipi è stata utilizzata come controllo positivo per il clade patogeno Leptospira . Come controllo positivo del clade intermedio di Leptospira , è stato incluso il DNA genomico del ceppo BUT6 del sierotipo Hurstbridge di Leptospira fainei e, come controllo positivo per il clade saprofita di Leptospira , è stato incluso anche il DNA genomico del ceppo Patoc Patoc I.
I controlli negativi consistevano in un plasmide vuoto, DNA di batteri non imparentati (Ureaplasma urealyticum, Staphylococcus aureus, Brucella abortus, Salmonella typhimurium, Shigella boydii, Klebsiella pneumoniae, Acinetobacter baumannii ed Escherichia coli) e acqua di grado PCR, che fungeva da controllo non stampo.
Campioni d'acqua
Dodici campioni di prova sono stati raccolti utilizzando un metodo di campionamento stratificato e casuale dal Cuemanco Biological and Aquaculture Research Center (CIBAC) (19° 16' 54" N 99° 6' 11" W). Questi campioni sono stati ottenuti a tre profondità: superficiale, 10 e 30 cm (Figura 1A, B). Le procedure di raccolta dell'acqua non hanno avuto alcun impatto su alcuna specie in via di estinzione o protetta. Ogni campione è stato raccolto in una provetta sterile da microcentrifuga da 15 ml. Per raccogliere il campione, ogni tubo è stato delicatamente immerso nell'acqua, riempito alla profondità selezionata e quindi sigillato. I campioni sono stati mantenuti a 22 °C e prontamente trasportati in laboratorio per l'elaborazione.
Ogni campione è stato concentrato mediante centrifugazione in provette sterili da microcentrifuga da 1,5 mL a 8000 x g per 20 minuti a temperatura ambiente. Questo passaggio è stato ripetuto fino a quando tutti i campioni sono stati concentrati in una provetta, che è stata poi utilizzata per l'estrazione del DNA (Figura 1C).
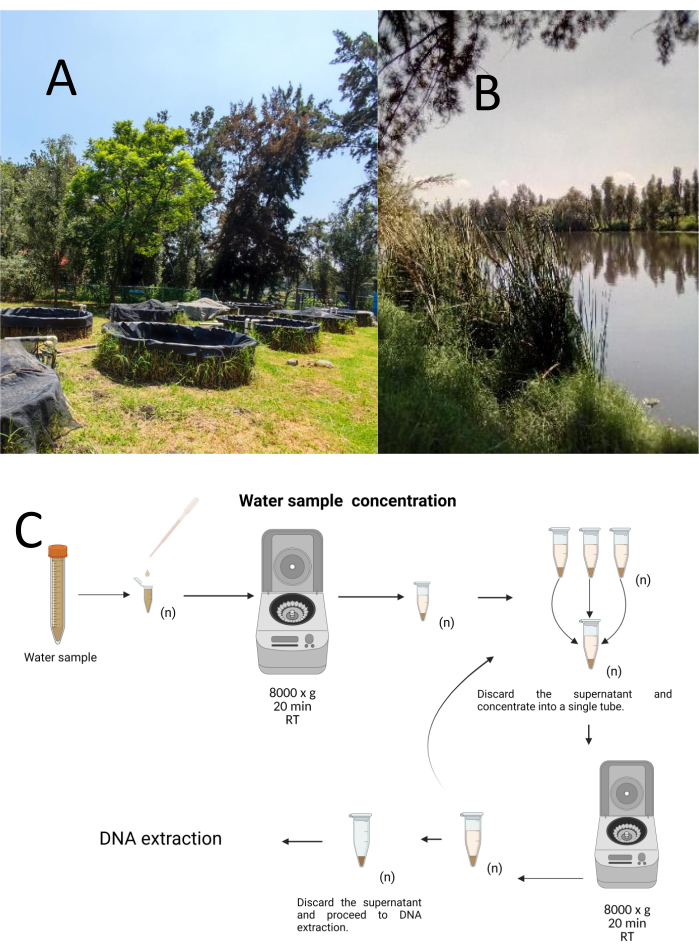
Figura 1: Concentrazione di campioni d'acqua per centrifugazione. (A) Stagni di campionamento dell'acqua e (B) Corsi d'acqua naturali. (C) Trattamento del campione d'acqua basato sulla centrifugazione in fasi ripetute tutte le volte necessarie (n). Fare clic qui per visualizzare una versione più grande di questa figura.
Estrazione del DNA
Il DNA totale è stato isolato utilizzando un kit di DNA genomico commerciale secondo le istruzioni del produttore (vedi Tabella dei materiali). Le estrazioni di DNA sono state eluite in 20 μL di tampone di eluizione e la concentrazione di DNA è stata determinata da uno spettrofotometro UV a 260-280 nm e conservata a 4 °C fino all'uso.
Amplificazione PCR
I bersagli della PCR erano i geni 16S rRNA, lipL41 e lipL32, che identificano il DNA del genere Leptospira e consentono la distinzione tra i tre cladi: patogeno, saprofita e intermedio. Sia i primer che i progetti delle sonde si basavano sui precedenti lavori di Ahmed et al., Azali et al., Bourhy et al., Weiss et al. e Branger et al.33,34,35,36,37. La sequenza di ciascuna sonda, primer e frammento amplificato è descritta nella Tabella 1 e il loro allineamento con le sequenze di riferimento è fornito nel File supplementare 1, nel File supplementare 2, nel File supplementare 3, nel File supplementare 4 e nel File supplementare 5. I reagenti PCR e le condizioni di termociclo sono descritti nella sezione del protocollo.
I prodotti di amplificazione sono stati visualizzati mediante separazione elettroforetica su un gel di agarosio all'1% in TAE (40 mM di base Tris, 20 mM di acido acetico e 1 mM di EDTA; pH 8,3), a 60 V per 45 minuti con rilevamento di etidio-bromuro, come mostrato nella Figura 1 supplementare. Il DNA genomico ottenuto da ciascun sierotipo è stato utilizzato con concentrazioni comprese tra 6 x 106 e 1 x 104 copie genomiche equivalenti (GEq) in ciascuna reazione di PCR, in base alla dimensione del genoma di L. interrogans (4, 691, 184 bp)38 per la Leptospira patogena, la dimensione del genoma di L. biflexa (3, 956, 088 bp)39 per la Leptospira saprofita, e la dimensione del genoma del sierotipo di L. fainei ceppo di Hurstbridge BUT6 (4, 267, 324 bp) con numero di adesione AKWZ00000000.2.
La sensibilità delle sonde è stata valutata con il DNA di ciascun sierotipo patogeno, L. biflexa sierotipo Patoc ceppo Patoc I e L. fainei sierotipo Hurstbridge ceppo BUT6 in ogni esperimento. Per valutare la specificità del test di ibridazione PCR e dot-blot, è stato incluso il DNA di batteri non correlati.
Tabella 1: Primer e sonde PCR per amplificare i prodotti per l'identificazione dei cladi patogeni, saprofiti e intermedi di Leptospira. Fare clic qui per scaricare questa tabella.
Saggio di ibridazione dot-blot
La tecnica è chiamata dot-blot perché i fori in cui viene posizionato il campione di DNA hanno una forma a punti, e quando vengono aspirati per essere fissati in posizione mediante aspirazione sottovuoto, acquisiscono questa forma. Questa tecnica è stata sviluppata da Kafatos et al.40. La tecnica consente la semi-quantificazione di Leptospira in ogni campione positivo alla PCR. Il protocollo consiste in una denaturazione con NaOH 0,4 M a temperatura ambiente, campioni con DNA di Leptospira da 30 ng a 0,05 ng, corrispondenti a 6 x 106 a 1 x 104 leptospiri, vengono tamponati su una membrana di nylon con un apparato dot-blot a 96 pozzetti. Dopo l'immobilizzazione, il DNA viene legato alla membrana mediante esposizione a 120 mJ di luce UV. Ogni sonda di DNA è coniugata con digoxigenina-11 dUTP mediante una fase di catalisi della transferasi terminale all'estremità 3' (la digoxigenina è uno steroide vegetale ottenuto dalla Digitalis purpurea, usato come reporter41). A seguito della rigorosa ibridazione della sonda di DNA marcata (50 pmol) alla temperatura specifica sul DNA bersaglio, gli ibridi di DNA vengono visualizzati mediante la reazione di chemiluminescenza con l'anticorpo anti-digoxigenina fosfatasi alcalina coniugato in modo covalente con il suo substrato CSPD. La luminescenza viene catturata dall'esposizione a una pellicola radiografica (Figura 2).
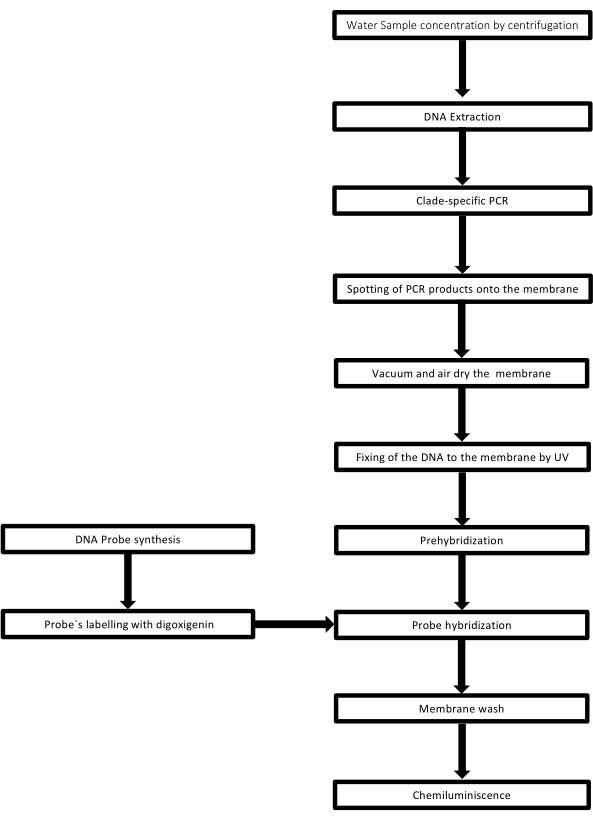
Figura 2: Fasi della procedura per il test PCR-dot-blot. Fare clic qui per visualizzare una versione più grande di questa figura.
Protocollo
1. Preparazione del campione
- Concentrare ogni campione d'acqua in provette da microcentrifuga da 1,5 mL mediante centrifugazione a 8.000 x g per 10 minuti a 4 °C. Ripetere questo passaggio, tutte le volte che è necessario, per concentrare il campione in un volume di 250 μl.
- Utilizzare il kit di estrazione del DNA secondo le istruzioni del produttore (vedere la tabella dei materiali).
- Eseguire la PCR specifica in base alla sonda dot-blot che verrà utilizzata (Tabella 1).
- Eseguire le amplificazioni in provette PCR con il volume finale di 25 μL contenenti 1 tampone X, 2,5 unità di Taq polimerasi, 1 μM di ciascun primer, 0,2 mM di ciascun dNTP, 1,5 mM di MgCl2 e 100 ng di DNA target da ciascun campione d'acqua o DNA genomico di riferimento.
- Programmare la reazione PCR in un termociclatore in base alle condizioni di termociclo (Tabella 2).
- Conservare le reazioni a 4 °C fino al momento dell'uso.
- Porre 10 μl di ciascun prodotto della PCR da asciugare in un pozzetto separato di una piastra a 96 pozzetti.
- Aggiungere 40 μL di TE in ciascun pozzetto e miscelare mediante pipettaggio.
NOTA: La piastra a 96 pozzetti può essere sigillata e conservata a 4 °C durante la notte. Seguire le istruzioni fornite nel file supplementare 6 per preparare buffer e soluzioni per il protocollo. La Figura 3 illustra le fasi di preparazione del campione.
Tabella 2: Condizioni di termociclo PCR per i geni 16S, lipL41 e lipL32. Fare clic qui per scaricare questa tabella.
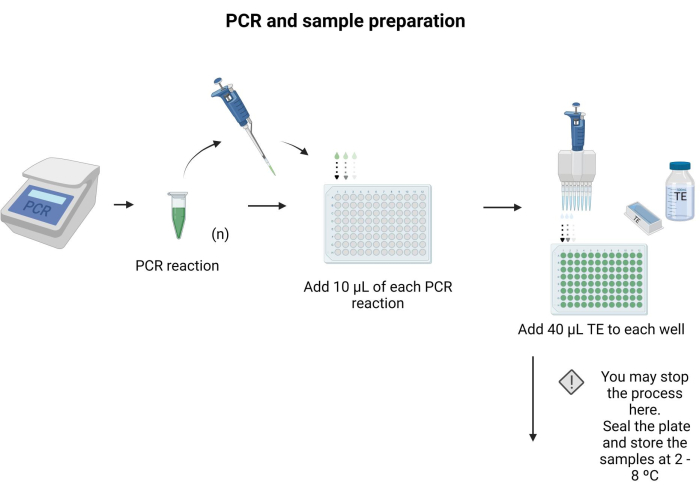
Figura 3: PCR e preparazione del campione. Applicando il protocollo PCR specifico, il prodotto PCR è stato trasferito in una piastra per microtitolazione e sono stati aggiunti 40 μL di TE a ciascun pozzetto. Fare clic qui per visualizzare una versione più grande di questa figura.
2. Assemblaggio dell'apparecchio a macchie di punti
NOTA: L'assemblaggio dell'apparato a macchie di punti è mostrato nella Figura 4. Durante la procedura, indossare guanti per maneggiare le soluzioni alcaline e proteggere la membrana di nylon dalla contaminazione.
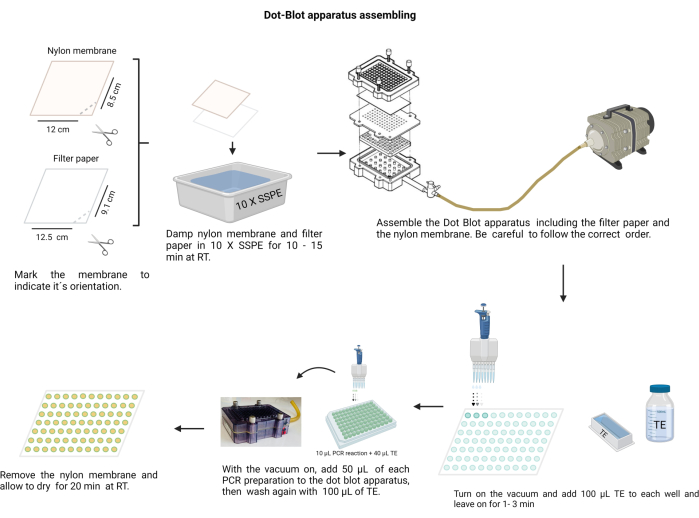
Figura 4: Assemblaggio dell'apparato dot-blot. La carta da filtro e la membrana di nylon (precedentemente inumidita in 10 X SSPE) devono essere disposte nell'ordine corretto. Il gruppo deve essere fissato saldamente con le viti prima di applicare il vuoto. Ogni pozzetto deve essere lavato con TE e i prodotti PCR vengono caricati nei rispettivi pozzetti. Dopo aver trasferito il prodotto della PCR attraverso la membrana, ogni pozzetto viene nuovamente lavato con TE e lasciato asciugare. Fare clic qui per visualizzare una versione più grande di questa figura.
- Tagliare la membrana di nylon (vedi Tabella dei materiali) e la carta da filtro in fogli di dimensioni 12 x 8,5 cm.
- Segna la membrana con un pennarello indelebile.
- Fai una tacca con le forbici su un bordo per indicare il giusto orientamento. Usa il segno per ricordare l'ordine del campione.
- Inumidire la membrana e la carta da filtro con 10 tamponi SALINE-sodio fosfato-EDTA (SSPE; 3 M NaCl, 0,2 M NaH2PO4 e 0,02 M EDTA, pH 7,4) (File supplementare 6). Maneggiare la membrana con una pinzetta pulita e smussata.
- Assemblare la camera dot-blot; In primo luogo, la carta da filtro, seguita dalla membrana sopra la guarnizione di plastica. Fissare il coperchio con le viti in modo trasversale.
- Collegare la camera al vuoto, posizionare 100 μL di TE in ciascun pozzetto, mantenere il vuoto per 1 minuto, quindi fermarlo.
- Accendere il vuoto a bassa velocità e caricare 50 μL di ciascun campione nel pozzetto corrispondente sulla membrana dell'apparecchio dot-blot (seguendo la distribuzione della membrana prestabilita).
NOTA: Omogeneizzare ogni campione nella piastra a 96 pozzetti per emoagglutinazione prima di posizionarlo nella camera dot-blot. - Lasciare asciugare la membrana dal vuoto. Se necessario, colpire delicatamente la camera per rilasciare le bolle nel campione.
- Lavare ogni pozzetto inserendo 100 μL di TE con un vuoto continuo e lasciandolo asciugare.
NOTA: Dopo aver completato il trasferimento, è fondamentale spegnere prima la pompa e staccarla. In caso contrario, potrebbe verificarsi l'ingresso di reflusso nell'apparecchio. Il DNA non necessita di una fase di denaturazione se viene utilizzata una membrana di nylon caricata positivamente, ma se si utilizza un altro supporto inerte, potrebbe essere necessaria una fase di pre-denaturazione e una denaturazione a base alcalina41.
3. Denaturazione e fissazione del DNA
NOTA: La Figura 5 illustra la procedura di fissazione della membrana del DNA.
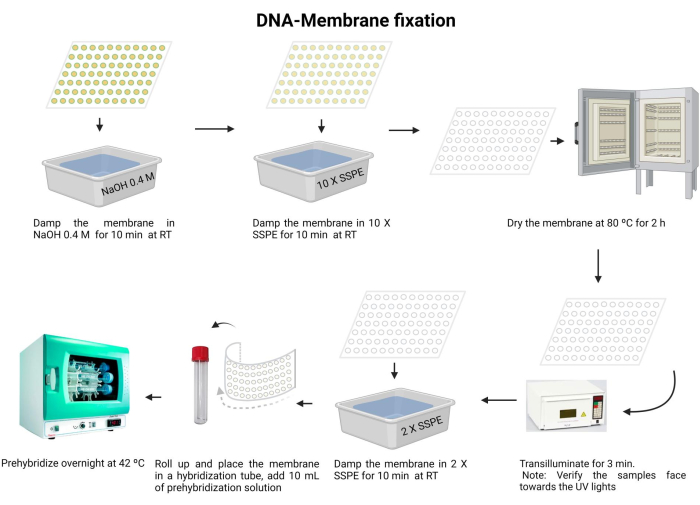
Figura 5: Procedura di fissazione della membrana del DNA. Il DNA viene denaturato in una soluzione alcalina. Successivamente, viene neutralizzato con 10 X SSPE e la membrana viene asciugata. Successivamente, la membrana viene transilluminata. La membrana viene reidratata con 2 X SSPE e pre-ibridata durante la notte. Fare clic qui per visualizzare una versione più grande di questa figura.
- Incubare la membrana con NaOH 0,4 M per 10 minuti a temperatura ambiente.
- Equilibrare la membrana con 10 X SSPE per 10 minuti a temperatura ambiente.
- Asciugare la membrana a 80 °C per 2 ore.
- Transilluminare con luce UV da 120 mJ per 3 min. Assicurarsi che i campioni siano orientati a faccia in giù verso la sorgente di luce UV. Se si utilizza un reticolante UV, verificare che i campioni siano rivolti verso l'alto e ripetere questo passaggio due volte.
NOTA: La membrana deve essere completamente asciutta prima della reticolazione UV. Il DNA sarà immobilizzato da un legame covalente alla membrana di nylon. Ogni reticolazione richiede da 18'' a 1' con una dose di irradiazione UV ottimale, per la maggior parte degli esperimenti di ibridazione, di circa 0,6-0,8 kJ/m2.
L'esposizione all'irradiazione UV è dannosa per gli occhi e la pelle. Indossare dispositivi di protezione adeguati ed evitare l'esposizione alla pelle nuda. - Lavare la membrana con 2 X SSPE per 10 min.
- Piegare con cautela la membrana e inserirla nella provetta di ibridazione (utilizzare una provetta da microcentrifuga da 15 mL).
- Aggiungere 10 mL della soluzione di pre-ibridazione (File supplementare 6) alla provetta e incubare a 42 °C per una notte. Assicurarsi che il tubo sia stato adeguatamente sigillato per evitare perdite.
4. Ibridazione
- Rimuovere 5 mL della soluzione di pre-ibridazione dalla provetta di ibridazione. Per utilizzare i 5 mL per una seconda volta, tenerli in un'altra provetta e conservarli a -4 °C.
- Aggiungere 15 μl della sonda marcata (Tabella 1) alla provetta di ibridazione. Sciacquare la punta nella soluzione per assicurarsi che la sonda etichettata sia stata accuratamente instillata nella diluizione.
NOTA: Il legame della sonda DNA non è forte come il legame antigene-anticorpo. Pertanto, i cambiamenti nella concentrazione della sonda o del DNA nel campione possono influenzare l'intensità della fluorescenza emessa. Le sonde di DNA sono stechiometriche, il che significa che il numero di molecole di sonda legate al DNA è equivalente al numero di molecole di DNA presenti nella soluzione. La Figura 6 illustra l'ibridazione della sonda. - Incubare a 42 °C per una notte.
- Eseguire l'etichettatura DIG della sonda DNA. Seguire le istruzioni specificate nella tabella 3 per miscelare i reattivi, quindi incubare la miscela a 37 °C per 1 ora. Aggiungere quindi 80 μl di acqua distillata e conservare la sonda a coda a 4 °C.
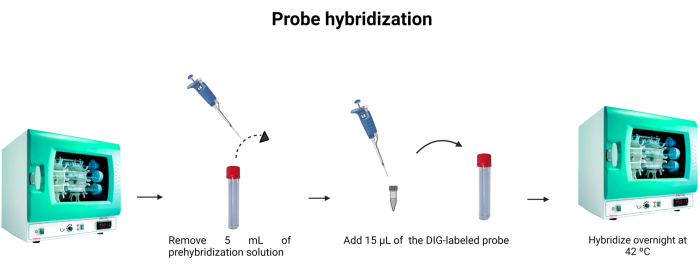
Figura 6: Ibridazione della sonda. Il volume del tampone di ibridazione viene regolato e la sonda marcata con digoxigenina viene incorporata per consentire l'ibridazione della sonda durante la notte. Fare clic qui per visualizzare una versione più grande di questa figura.
Tabella 3: Reagenti per la marcatura delle sonde con digossigenina (DIG). Fare clic qui per scaricare questa tabella.
5. Chemiluminescenza (etichettatura anti-DIG)
- Lavare la membrana due volte con 2 X SSPE/0,1% SDS a temperatura ambiente per 10 min.
- Incubare la membrana con 5 X SSPE/0,1% SDS alla temperatura di ricottura della sonda per 15 minuti.
NOTA: In questo passaggio, utilizzare un contenitore con coperchio per mantenere la temperatura il più a lungo possibile. Garantire una temperatura uniforme e costante su tutta la membrana. Il volume approssimativo in ogni lavaggio è di 100 ml della soluzione indicata. - Lavare la membrana una volta con il tampone 1 (file supplementare 6) a temperatura ambiente per 5 min.
- Lavare una volta la membrana con il tampone 2 (file supplementare 6) a temperatura ambiente per 30 min.
- Lavare la membrana con il tampone 2 e aggiungere l'anticorpo anti-digossigenina (15 μL) (vedere la tabella dei materiali), quindi incubare per 45 minuti (il tempo di incubazione può variare da 30 a 60 minuti). Questo anticorpo secondario contrassegna la sonda per il rilevamento.
NOTA: La membrana può essere conservata nel tampone 2 con l'anticorpo anti-digossigenina per una notte a 4-8 °C. L'etichettatura anti-DIG del processo di chemiluminescenza è illustrata nella Figura 7.
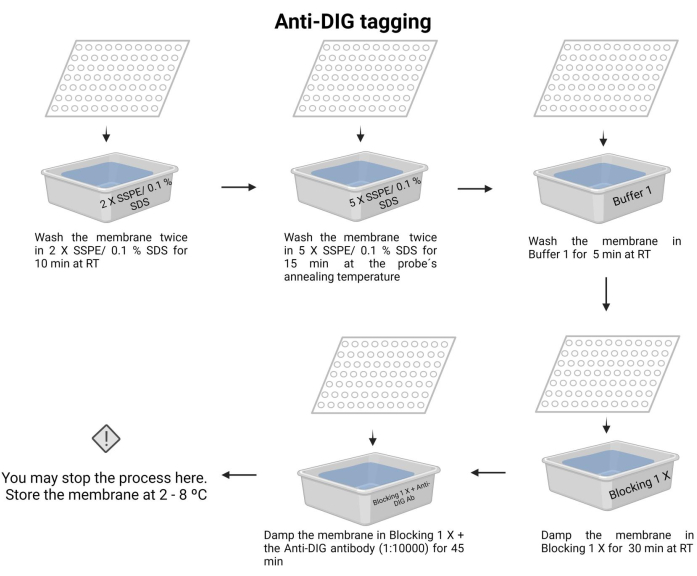
Figura 7: Etichettatura anti-DIG del processo di chemiluminescenza. Gli acidi nucleici non legati vengono rimossi con soluzioni tampone. La sonda è allineata con il DNA bersaglio e l'eccesso viene rimosso. La membrana viene bloccata con il tampone bloccante 1 X e viene aggiunto l'anticorpo anti-DIG (1:10000). Fare clic qui per visualizzare una versione più grande di questa figura.
6. Chemiluminescenza (applicazione del substrato)
- Lavare la membrana due volte nel tampone 1 a temperatura ambiente per 15 min.
- Lavare una volta nel tampone 3 (vedi file supplementare 6) a temperatura ambiente per 5 minuti, lasciando che la membrana dreni la maggior parte del tampone, lasciandolo quasi asciutto.
- Aggiungere CSPD pronto all'uso (disodio 3-(4-metossispiro {1,2-diossano-3,2′-(5′-cloro) triciclo-[3.3.1.13,7] decan}-4-il) fenil fosfato, vedi Tabella dei materiali), e lasciare agire per 5 minuti, (con le dita inumidite omogeneizzare il CSPD da un lato all'altro).
- Posizionare la membrana in un sacchetto di plastica trasparente (12,5 x 9 cm) che si adatti alle dimensioni della membrana. Rimuovere con cura le bolle d'aria, distribuire uniformemente il CSPD e sigillare a caldo il sacchetto.
- Incubare la membrana in un bagno d'acqua a 37 °C per 15 minuti (può arrivare fino a 30 minuti) e assicurarsi che la membrana con i campioni sia posizionata verso il basso in modo che sia ben sommersa. Assicurarsi che la temperatura rimanga costante e ben distribuita sulla membrana.
- Asciugare il sacchetto di plastica e fissarlo con lo scotch su una pellicola radiografica già utilizzata. Ciò consentirà una facile manipolazione durante la procedura di esposizione.
NOTA: La Figura 8 mostra l'applicazione del processo di chemiluminescenza al substrato.

Figura 8: Applicazione del substrato del processo di chemiluminescenza. L'anticorpo libero viene rimosso e il substrato CSPD (1:250) viene aggiunto alla membrana. La reazione viene attivata mediante incubazione a 37 °C e la membrana viene disposta per registrare la chemiluminescenza in una pellicola radiografica. Fare clic qui per visualizzare una versione più grande di questa figura.
7. Chemiluminescenza (rilevamento)
- Eseguire la procedura di esposizione. Nella camera oscura, preparare le soluzioni di sviluppo e fissaggio (vedere Tabella dei materiali) nei rispettivi vassoi.
- Al buio, posizionare con cura la membrana fissa di fronte a una nuova pellicola radiografica e inserirla in una cassetta radiografica (vedi Tabella dei materiali).
- Registrare il tempo di esposizione. Il tempo di esposizione può variare da 1 minuto a 30 minuti o più. Inizia con 5 minuti e regola il tempo di conseguenza.
- Inumidire la pellicola radiografica in soluzione di sviluppo per 1-3 minuti e risciacquare delicatamente con acqua di rubinetto (15-45 s).
- Inumidire la pellicola radiografica in soluzione fissante per 1-3 minuti e risciacquare delicatamente con acqua di rubinetto (45 s).
- Lasciare asciugare la pellicola radiografica all'aria e documentare il test in una lightbox bianca visibile.
NOTA: Evitare di scuotere la membrana durante il tempo di esposizione ai raggi X. Il processo di rilevamento è illustrato nella Figura 9. - Procedere all'interpretazione del risultato. Nella pellicola radiografica esposta, i punti con emissione possono essere localizzati visivamente e corrispondono ai frammenti di DNA con l'ibridazione della sonda. L'intensità del segnale emesso dipende dalla luminescenza e dalla durata dell'esposizione.
NOTA: Le membrane possono essere conservate essiccate tra la carta da filtro per diversi mesi a temperatura ambiente.
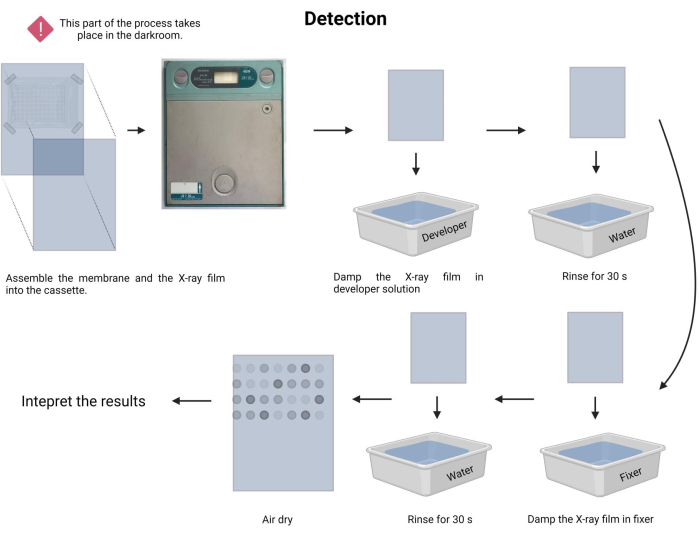
Figura 9: Rilevamento del processo di chemiluminescenza. In condizioni di oscurità, la membrana è esposta a una pellicola radiografica all'interno di una cassetta radiografica. Successivamente, è stato lasciato riposare durante il periodo di esposizione, quindi la pellicola a raggi X è stata sviluppata e fissata. Infine, è stato asciugato all'aria e interpretato. Fare clic qui per visualizzare una versione più grande di questa figura.
8. Procedura di deibridazione della membrana
- Lavare la membrana due volte per 10 minuti con acqua distillata.
- Lavare la membrana per 20 minuti con NaOH 0,4 M a 53 °C (due volte).
- Lavare due volte la membrana per 10 minuti con 2 X SSPE.
- Lasciare asciugare la membrana a temperatura ambiente.
- Mantieni la membrana e torna al passaggio 3.5 del protocollo.
NOTA: La membrana può essere conservata nel tampone di pre-ibridazione per una notte a 4-8 °C. Il giorno successivo, ricominciare con un'incubazione a 42 °C per 1 ora, permettendo al tampone di pre-ibridazione di raggiungere la temperatura. Quindi, procedere al passaggio 3.5 del protocollo.
Risultati
Per valutare l'efficacia della tecnica, è stato utilizzato DNA genomico da colture pure di ciascun sierotipo di Leptospira , insieme alla sonda specifica per clade. Le membrane sono state preparate con 100 ng di DNA genomico per reazione PCR per ciascun sierotipo, seguito da otto DNA genomico di batteri non correlati e concentrazioni variabili di DNA genomico dei sierotipi Leptospira ad hoc . Ogni test includeva un controllo positivo, negativo e non modello. Questi DNA genomici non correlati non hanno ...
Discussione
Le fasi critiche della tecnica dot-blot includono (1) immobilizzazione del DNA, (2) blocco dei siti di legame liberi sulla membrana con DNA non omologo, (3) la complementarità tra la sonda e il frammento bersaglio in condizioni di ricottura, (4) rimozione della sonda non ibridata e (5) il rilevamento della molecola reporter41.
La PCR-Dot-blot presenta alcune limitazioni, come la tecnica che non fornisce informazioni sulle dimensioni del frammento37
Divulgazioni
Gli autori dichiarano che non vi è alcun conflitto di interessi.
Riconoscimenti
Siamo in debito con la collezione Leptospira del Dipartimento di Microbiologia e Immunologia, Facoltà di Medicina Veterinaria e Zootecnica, Università Nazionale Autonoma del Messico. Siamo grati per la generosa donazione dei ceppi di riferimento Leptospira ; Leptospira fainei sierotipo Hurstbridge ceppo BUT6 e Leptospira biflexa sierovariante Patoc ceppo Patoc I al Dr. Alejandro de la Peña Moctezuma. Ringraziamo il Dott. José Antonio Ocampo Cervantes, Coordinatore del CIBAC, e il personale per il loro supporto logistico. EDT faceva parte del programma Terminal Project per studenti universitari della Metropolitan Autonomous University-Campus Cuajimalpa. Ringraziamo il software Biorender.com per la creazione delle figure 1 e da 3 a 9.
Materiali
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| Purelink DNA extraction kit | Invitrogen | K182002 | |
| Gotaq Flexi DNA Polimerase (End-Point PCR Taq polymerase kit) | Promega | M3001 | |
| Whatman filter paper, grade 1, | Merk | WHA1001325 | |
| Nylon Membranes, positively charged Roll 30cm x 3 m | Roche | 11417240001 | |
| Anti-Digoxigenin-AP, Fab fragments Sheep Polyclonal Primary-antibody | Roche | 11093274910 | |
| Medium Base EMJH | Difco | S1368JAA | |
| Leptospira Enrichment EMJH | Difco | BD 279510 | |
| Blocking Reagent | Roche | 11096176001 | |
| CSPD ready to use Disodium 3-(4-methoxyspiro {1,2-dioxetane-3,2′-(5′-chloro) tricyclo [3.3.1.13,7] decan}8-4-yl) phenyl phosphate | Merk | 11755633001 | |
| Deoxyribonucleic acid from herring sperm | Sigma Aldrich | D3159 | |
| Developer Carestream | Carestream Health Inc | GBX5158621 | |
| Digoxigenin-11-ddUTP | Roche | 11363905910 | |
| EDTA, Disodium Salt (Dihydrate) | Promega | H5032 | |
| Ficoll 400 | Sigma Aldrich | F8016 | |
| Fixer Carestream | Carestream Health Inc | GBX 5158605 | |
| Lauryl sulfate Sodium Salt (Sodium dodecyl sulfate; SDS) C12H2504SNa | Sigma Aldrich | L5750 | |
| N- Lauroylsarcosine sodium salt CH3(CH2)10CON(CH3) CH2COONa | Sigma Aldrich | L-9150 | It is an anionic surfactant |
| Polivinylpyrrolidone (PVP-40) | Sigma Aldrich | PVP40 | |
| Polyethylene glycol Sorbitan monolaurate (Tween 20) | Sigma Aldrich | 9005-64-5 | |
| Sodium Chloride (NaCl) | Sigma Aldrich | 7647-14-5 | |
| Sodium dodecyl sulfate (SDS) | Sigma Aldrich | 151-21-3 | |
| Sodium hydroxide (NaOH) | Sigma Aldrich | 1310-73-2 | |
| Sodium phosphate dibasic (NaH2PO4) | Sigma-Aldrich | 7558-79-4 | |
| Terminal transferase, recombinant | Roche | 3289869103 | |
| Tris hydrochloride (Tris HCl) | Sigma-Aldrich | 1185-53-1 | |
| SSPE 20X | Sigma-Aldrich | S2015-1L | It can be Home-made following Supplementary File 6 |
| Primers | Sigma-Aldrich | On demand | Follow table 1 |
| Probes | Sigma-Aldrich | On demand | Follow table 1 |
| Equipment | |||
| Nanodrop™ One Spectrophotometer | Thermo-Scientific | ND-ONE-W | |
| Refrigerated microcentrifuge Sigma 1-14K, suitable for centrifugation of 1.5 ml microcentrifuge tubes at 14,000 rpm | Sigma-Aldrich | 1-14K | |
| Disinfected adjustable pipettes, range 2-20 µl, 20-200 µl | Gilson | SKU:F167360 | |
| Disposable 1.5 ml microcentrifuge tubes (autoclaved) | Axygen | MCT-150-SP | |
| Disposable 600 µl microcentrifuge tubes (autoclaved) | Axygen | 3208 | |
| Disposable Pipette tips 1-10 µl | Axygen | T-300 | |
| Disposable Pipette tips 1-200 µl | Axygen | TR-222-Y | |
| Dot-Blot apparatus Bio-Dot | BIORAD | 1706545 | |
| Portable Hergom Suction | Hergom | 7E-A | |
| Scientific Light Box (Visible-light PH90-115V) | Hoefer | PH90-115V | |
| UV Crosslinker | Hoefer | UVC-500 | |
| Thermo Hybaid PCR Express Thermocycler | Hybaid | HBPX110 | |
| Radiographic cassette with IP Plate14 X 17 | Fuji |
Riferimenti
- Bierque, E., Thibeaux, R., Girault, D., Soupé-Gilbert, M. E., Goarant, C. A systematic review of Leptospira in water and soil environments. PLOS One. 15 (1), e0227055 (2020).
- Haake, D. A., Levett, P. N. Leptospirosis in humans. Current Topics in Microbiology and Immunology. 387, 65-97 (2015).
- Tripathy, D. N., Hanson, L. E. Leptospires from water sources at Dixon Springs Agricultural Center. Journal of Wildlife Diseases. 9 (3), 209-212 (1973).
- Smith, D. J., Self, H. R. Observations on the survival of Leptospira australis A in soil and water. The Journal of Hygiene. 53 (4), 436-444 (1955).
- Karpagam, K. B., Ganesh, B. Leptospirosis: a neglected tropical zoonotic infection of public health importance-an updated review. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 39 (5), 835-846 (2020).
- Casanovas-Massana, A., et al. Spatial and temporal dynamics of pathogenic Leptospira in surface waters from the urban slum environment. Water Research. 130, 176-184 (2018).
- Costa, F., et al. Global morbidity and mortality of Leptospirosis: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003898 (2015).
- Mwachui, M. A., Crump, L., Hartskeerl, R., Zinsstag, J., Hattendorf, J. Environmental and behavioural determinants of Leptospirosis transmission: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003843 (2015).
- Andre-Fontaine, G., Aviat, F., Thorin, C. Waterborne Leptospirosis: Survival and preservation of the virulence of pathogenic Leptospira spp. in fresh water. Current Microbiology. 71 (1), 136-142 (2015).
- Trueba, G., Zapata, S., Madrid, K., Cullen, P., Haake, D. Cell aggregation: A mechanism of pathogenic Leptospira to survive in freshwater. International Microbiology: the Official Journal of the Spanish Society for Microbiology. 7 (1), 35-40 (2004).
- Smith, C. E., Turner, L. H. The effect of pH on the survival of leptospires in water. Bulletin of the World Health Organization. 24 (1), 35-43 (1961).
- Barragan, V. A., et al. Interactions of Leptospira with environmental bacteria from surface water. Current Microbiology. 62 (6), 1802-1806 (2011).
- Abdoelrachman, R. Comparative investigations into the influence of the presence of bacteria on the life of pathogenic and apathogenic leptospirae. Antonie van Leeuwenhoek. 13 (1), 21-32 (1947).
- Singh, R., et al. Microbial diversity of biofilms in dental unit water systems. Applied and Environmental Microbiology. 69 (6), 3412-3420 (2003).
- Kumar, K. V., Lall, C., Raj, R. V., Vedhagiri, K., Vijayachari, P. Coexistence and survival of pathogenic leptospires by formation of biofilm with Azospirillum. FEMS Microbiology Ecology. 91 (6), 051 (2015).
- Yanagihara, Y., et al. Leptospira Is an environmental bacterium that grows in waterlogged soil. Microbiology Spectrum. 10 (2), 0215721 (2022).
- Gillespie, R. W., Ryno, J. Epidemiology of leptospirosis. American Journal of Public Health and Nation’s Health. 53 (6), 950-955 (1963).
- Bierque, E., et al. Leptospira interrogans retains direct virulence after long starvation in water. Current Microbiology. 77 (10), 3035-3043 (2020).
- Zhang, Y., Dai, B. Marking and detection of DNA of leptospires in the dot-blot and situ hybridization with digoxigenin-labeled probes. Journal of West China University of Medical Sciences. 23 (4), 353-435 (1992).
- Mérien, F., Amouriaux, P., Perolat, P., Baranton, G., Saint Girons, I. Polymerase chain reaction for detection of Leptospira spp. in clinical samples. Journal of Clinical Microbiology. 30 (9), 2219-2224 (1992).
- Veerapandian, R., et al. Silver enhanced nano-gold dot-blot immunoassay for leptospirosis. Journal of Microbiological Methods. 156, 20-22 (2019).
- Junpen, S., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Ishmael, F. T., Stellato, C. Principles and applications of polymerase chain reaction: basic science for the practicing physician. Annals of Allergy, Asthma & Immunology: Official Publication of the American College of Allergy, Asthma, & Immunology. 101 (4), 437-443 (2008).
- Boerner, B., Weigelt, W., Buhk, H. J., Castrucci, G., Ludwig, H. A sensitive and specific PCR/Southern blot assay for detection of bovine herpesvirus 4 in calves infected experimentally. Journal of Virological Methods. 83 (1-2), 169-180 (1999).
- Curry, E., Pratt, S. L., Kelley, D. E., Lapin, D. R., Gibbons, J. R. Use of a Combined duplex PCR/Dot-blot assay for more sensitive genetic characterization. Biochemistry Insights. 1, 35-39 (2008).
- Pilatti, M. M., Ferreira, S. d. e. A., de Melo, M. N., de Andrade, A. S. Comparison of PCR methods for diagnosis of canine visceral leishmaniasis in conjunctival swab samples. Research in Veterinary Science. 87 (2), 255-257 (2009).
- Conrads, G., et al. PCR reaction and dot-blot hybridization to monitor the distribution of oral pathogens within plaque samples of periodontally healthy individuals. Journal of Periodontology. 67 (10), 994-1003 (1996).
- Langa, S., et al. Differentiation of Enterococcus faecium from Lactobacillus delbrueckii subsp. bulgaricus and Streptococcus thermophilus strains by PCR and dot-blot hybridisation. International Journal of Food Microbiology. 88 (2-3), 197-200 (2003).
- Francesca, C., Lucilla, I., Marco, F., Giuseppe, C., Marisa, M. Identification of the unculturable bacteria Candidatus arthromitus in the intestinal content of trouts using dot-blot and Southern blot techniques. Veterinary Microbiology. 156 (3-4), 389-394 (2012).
- Arent, Z., Pardyak, L., Dubniewicz, K., Plachno, B. J., Kotula-Balak, M. Leptospira taxonomy: then and now. Medycyna Weterynaryjna. 78 (10), 489-496 (2022).
- Thibeaux, R., et al. Biodiversity of environmental Leptospira: Improving identification and revisiting the diagnosis. Frontiers in Microbiology. 9, 816 (2018).
- Carrillo-Casas, E. M., Hernández-Castro, R., Suárez-Güemes, F., de la Peña-Moctezuma, A. Selection of the internal control gene for real-time quantitative RT-PCR assays in temperature treated Leptospira. Current Microbiology. 56 (6), 539-546 (2008).
- Azali, M. A., Yean Yean, C., Harun, A., Aminuddin Baki A, N. N., Ismail, N. Molecular characterization of Leptospira spp. in environmental samples from North-Eastern Malaysia revealed a pathogenic strain, Leptospira alstonii. Journal of Tropical Medicine. 2016, 2060241 (2016).
- Ahmed, N., et al. Multilocus sequence typing method for identification and genotypic classification of pathogenic Leptospira species. Annals of Clinical Microbiology and Antimicrobials. 5, 28 (2006).
- Bourhy, P., Collet, L., Brisse, S., Picardeau, M. Leptospira mayottensis sp. nov., a pathogenic species of the genus Leptospira isolated from humans. International Journal of Systematic and Evolutionary Microbiology. 64, 4061-4067 (2014).
- Weiss, S., et al. An extended Multilocus Sequence Typing (MLST) scheme for rapid direct typing of Leptospira from clinical samples. PLOS Neglected Tropical Diseases. 10 (9), e0004996 (2016).
- Branger, C., et al. Polymerase chain reaction assay specific for pathogenic Leptospira based on the gene hap1 encoding the hemolysis-associated protein-1. FEMS Microbiology Letters. 243 (2), 437-445 (2005).
- Ren, S. X., et al. Unique physiological and pathogenic features of Leptospira interrogans revealed by whole-genome sequencing. Nature. 422 (6934), 888-893 (2003).
- Picardeau, M., et al. Genome sequence of the saprophyte Leptospira biflexa provides insights into the evolution of Leptospira and the pathogenesis of leptospirosis. PLOS One. 3 (2), e1607 (2008).
- Kafatos, F. C., Jones, C. W., Efstratiadis, A. Determination of nucleic acid sequence homologies and relative concentrations by a dot hybridization procedure. Nucleic Acids Research. 7 (6), 1541-1552 (1979).
- Bhat, A. I., Rao, G. P. Dot-blot hybridization technique. Characterization of Plant Viruses. , 303-321 (2020).
- Yadav, J. P., Batra, K., Singh, Y., Singh, M. Comparative evaluation of indirect-ELISA and Dot-blot assay for serodetection of Mycoplasma gallisepticum and Mycoplasma synoviae antibodies in poultry. Journal of Microbiological Methods. 189, 106317 (2021).
- Malinen, E., Kassinen, A., Rinttilä, T., Palva, A. Comparison of real-time PCR with SYBR Green I or 5'-nuclease assays and dot-blot hybridization with rDNA-targeted oligonucleotide probes in quantification of selected faecal bacteria. Microbiology. 149, 269-277 (2003).
- Wyss, C., et al. Treponema lecithinolyticum sp. nov., a small saccharolytic spirochaete with phospholipase A and C activities associated with periodontal diseases. International Journal of Systematic Bacteriology. 49, 1329-1339 (1999).
- Shah, J. S., I, D. C., Ward, S., Harris, N. S., Ramasamy, R. Development of a sensitive PCR-dot-blot assay to supplement serological tests for diagnosing Lyme disease. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 37 (4), 701-709 (2018).
- Niu, C., Wang, S., Lu, C. Development and evaluation of a dot-blot assay for rapid determination of invasion-associated gene ibeA directly in fresh bacteria cultures of E. coli. Folia microbiologica. 57 (6), 557-561 (2012).
- Wetherall, B. L., McDonald, P. J., Johnson, A. M. Detection of Campylobacter pylori DNA by hybridization with non-radioactive probes in comparison with a 32P-labeled probe. Journal of Medical Microbiology. 26 (4), 257-263 (1988).
- Kolk, A. H., et al. Detection of Mycobacterium tuberculosis in clinical samples by using polymerase chain reaction and a nonradioactive detection system. Journal of Clinical Microbiology. 30 (10), 2567-2575 (1992).
- Scherer, L. C., et al. PCR colorimetric dot-blot assay and clinical pretest probability for diagnosis of Pulmonary Tuberculosis in smear-negative patients. BMC Public Health. 7, 356 (2007).
- Armbruster, D. A., Pry, T. Limit of blank, limit of detection and limit of quantitation. The Clinical Biochemist Reviews. 29, S49-S52 (2008).
- Zhang, Y., Dai, B. Detection of Leptospira by dot-blot hybridization with photobiotin- and 32P-labeled DNA. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 130-132 (1992).
- Terpstra, W. J., Schoone, G. J., ter Schegget, J. Detection of leptospiral DNA by nucleic acid hybridization with 32P- and biotin-labeled probes. Journal of Medical Microbiology. 22 (1), 23-28 (1986).
- Shukla, J., Tuteja, U., Batra, H. V. DNA probes for identification of leptospires and disease diagnosis. The Southeast Asian Journal of Tropical Medicine and Public Health. 35 (2), 346-352 (2004).
- Jiang, N., Jin, B., Dai, B., Zhang, Y. Identification of pathogenic and nonpathogenic leptospires by recombinant probes. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 26 (1), 1-5 (1995).
- Fach, P., Trap, D., Guillou, J. P. Biotinylated probes to detect Leptospira interrogans on dot-blot hybridization or by in situ hybridization. Letters in Applied Microbiology. 12 (5), 171-176 (1991).
- Huang, N., Dai, B. Assay of genomic DNA homology among strains of different virulent leptospira by DNA hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 122-125 (1992).
- Dong, X., Dai, B., Chai, J. Homology study of leptospires by molecular hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (1), 1-4 (1992).
- Komminoth, P. Digoxigenin as an alternative probe labeling for in situ hybridization. Diagnostic Molecular Pathology: The American Journal of Surgical Pathology, part B. 1 (2), 142-150 (1992).
- Saengjaruk, P., et al. Diagnosis of human leptospirosis by monoclonal antibody-based antigen detection in urine. Journal of Clinical Microbiology. 40 (2), 480-489 (2002).
- Okuda, M., et al. Enzyme-linked immunosorbent assay for the detection of canine Leptospira antibodies using recombinant OmpL1 protein. The Journal of Veterinary Medical Science. 67 (3), 249-254 (2005).
- Suwimonteerabutr, J., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Kanagavel, M., et al. Peptide-specific monoclonal antibodies of Leptospiral LigA for acute diagnosis of leptospirosis. Scientific reports. 7 (1), 3250 (2017).
- Levett, P. N. Leptospirosis. Clinical Microbiology Reviews. 14 (2), 296-326 (2001).
- Monahan, A. M., Callanan, J. J., Nally, J. E. Proteomic analysis of Leptospira interrogans shed in urine of chronically infected hosts. Infection and Immunity. 76 (11), 4952-4958 (2008).
- Rojas, P., et al. Detection and quantification of leptospires in urine of dogs: a maintenance host for the zoonotic disease leptospirosis. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 29 (10), 1305-1309 (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon