É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Reação em cadeia da polimerase e hibridização de ponto-blot para detecção de Leptospira em amostras de água
Neste Artigo
Resumo
Neste estudo, uma aplicação de dot-blot foi projetada para detectar Leptospira dos três clados principais em amostras de água. Este método permite a identificação de quantidades mínimas de DNA especificamente direcionadas por uma sonda marcada com digoxigenina, facilmente detectada por um anticorpo anti-digoxigenina. Essa abordagem é uma ferramenta valiosa e satisfatória para fins de triagem.
Resumo
O dot-blot é uma técnica simples, rápida, sensível e versátil que permite a identificação de quantidades mínimas de DNA especificamente direcionadas pela hibridização da sonda na presença de DNA transportador. Baseia-se na transferência de uma quantidade conhecida de DNA para um suporte sólido inerte, como uma membrana de náilon, utilizando o aparelho de dot-blot e sem separação eletroforética. As membranas de nylon têm a vantagem de alta capacidade de ligação de ácidos nucleicos (400 μg/cm2), alta resistência e são carregadas positiva ou neutramente. A sonda usada é um fragmento de ssDNA altamente específico de 18 a 20 bases marcadas com digoxigenina (DIG). A sonda se conjugará com o DNA da Leptospira . Uma vez que a sonda tenha hibridizado com o DNA alvo, ela é detectada por um anticorpo anti-digoxigenina, permitindo sua fácil detecção por meio de suas emissões reveladas em um filme de raios-X. Os pontos com uma emissão corresponderão aos fragmentos de DNA de interesse. Este método emprega a marcação não isotópica da sonda, que pode ter uma meia-vida muito longa. A desvantagem deste imunomarcador padrão é uma sensibilidade mais baixa do que as sondas isotópicas. No entanto, é mitigado pelo acoplamento da reação em cadeia da polimerase (PCR) e ensaios de dot-blot. Essa abordagem permite o enriquecimento da sequência alvo e sua detecção. Além disso, pode ser usado como uma aplicação quantitativa quando comparado com uma diluição em série de um padrão bem conhecido. Uma aplicação de dot-blot para detectar Leptospira dos três clados principais em amostras de água é apresentada aqui. Esta metodologia pode ser aplicada a grandes quantidades de água, uma vez que tenham sido concentradas por centrifugação para fornecer evidências da presença de DNA de Leptospira. Esta é uma ferramenta valiosa e satisfatória para fins gerais de triagem, podendo ser utilizada para outras bactérias não cultiváveis que possam estar presentes na água, melhorando a compreensão do ecossistema.
Introdução
A leptospirose em humanos tem origem principalmente em fontes ambientais 1,2. A presença de Leptospira em lagos, rios e córregos é um indicador de transmissão de leptospirose entre animais silvestres e animais domésticos e de produção que podem eventualmente entrar em contato com esses corpos d'água 1,3,4. Além disso, Leptospira foi identificada em fontes não naturais, incluindo esgoto, água estagnada e água da torneira 5,6.
Leptospira é uma bactéria distribuída mundialmente 7,8, e o papel do ambiente em sua preservação e transmissão tem sido bem reconhecido. Leptospira pode sobreviver em água potável sob pH e minerais variáveis9 e em corpos d'água naturais1. Também pode sobreviver por longos períodos em água destilada10 e, sob pH constante (7,8), pode sobreviver até 152 dias11. Além disso, Leptospira pode interagir em consórcios bacterianos para sobreviver a condições adversas12,13. Pode fazer parte de biofilmes em água doce com Azospirillum e Sphingomonas e é capaz de crescer e suportar temperaturas superiores a 49 °C14,15. Também pode se multiplicar em solo encharcado e permanecer viável por até 379 dias16, preservando sua capacidade de causar a doença por até um ano17,18. No entanto, pouco se sabe sobre a ecologia dentro dos corpos d'água e como ela se distribui dentro deles.
Desde sua descoberta, o estudo do gênero Leptospira foi baseado em testes sorológicos. Não foi até o século atual que as técnicas moleculares se tornaram mais prevalentes no estudo deste espiroqueta. O dot-blot tem sido pouco utilizado para sua identificação usando (1) uma sonda isotópica baseada no 16S rRNA e em uma repetição de sequência inter-simples (ISSR) 19 , 20 , (2) como um imunoensaio baseado em nanoouro para leptospirose humana aplicada à urina21, ou (3) como um ensaio baseado em anticorpos para amostras de urina bovina22. A técnica caiu em desuso porque foi originalmente baseada em sondas isotópicas. No entanto, é uma técnica bem conhecida que, juntamente com a PCR, produz resultados aprimorados, sendo considerada segura devido ao uso de sondas não isotópicas. A PCR desempenha um papel crucial no enriquecimento do DNA de Leptospira, amplificando um fragmento de DNA específico que pode ser encontrado em pequenas quantidades em uma amostra. Durante cada ciclo de PCR, a quantidade do fragmento de DNA alvo é dobrada na reação. No final da reação, o amplicon foi multiplicado por um fator de mais de um milhão23. O produto amplificado por PCR, muitas vezes não visível na eletroforese de agarose, torna-se visível por hibridização específica com uma sonda marcada com DIG no ponto-blot 24,25,26.
A técnica dot-blot é simples, robusta e adequada para inúmeras amostras, tornando-a acessível a laboratórios com recursos limitados. Tem sido empregado em uma variedade de estudos de bactérias, incluindo (1) bactérias orais27, (2) outros tipos de amostras, como alimentos e fezes28, e (3) a identificação de bactérias não cultiváveis29, muitas vezes de acordo com outras técnicas moleculares. Entre as vantagens oferecidas pela técnica dot-blot estão: (1) A membrana possui alta capacidade de ligação, capaz de ligar mais de 200 μg/cm2 de ácidos nucléicos e até 400 μg/cm2; (2) Os resultados do dot-blot podem ser interpretados visualmente sem a necessidade de equipamento especial e (3) podem ser convenientemente armazenados por anos em temperatura ambiente (RT).
O gênero Leptospira foi classificado em clados patogênicos, intermediários e saprófitos30,31. A distinção entre esses clados pode ser alcançada com base em genes específicos, como lipL41, lipL32 e 16S rRNA. LipL32 está presente nos clados patogênicos e exibe alta sensibilidade em várias ferramentas sorológicas e moleculares, enquanto está ausente em espécies saprófitas21. O gene housekeeping lipL41 é conhecido por sua expressão estável e utilizado em técnicas moleculares32, enquanto o gene16 S rRNA é utilizado para sua classificação.
Esta metodologia pode ser aplicada a grandes volumes de água, uma vez que tenham sido concentrados por centrifugação. Ele permite a avaliação de vários pontos e profundidades dentro de um corpo d'água para detectar a presença de DNA leptospira e o clado ao qual ele pertence. Esta ferramenta é valiosa para fins de triagem ecológica e geral e também pode ser empregada para detectar outras bactérias não cultiváveis que possam estar presentes na água.
Além disso, os ensaios de PCR e dot-blot são técnica e economicamente acessíveis a uma ampla gama de laboratórios, mesmo aqueles que não possuem equipamentos sofisticados ou caros. Este estudo tem como objetivo aplicar o dot-blot à base de digoxigenina para a identificação dos três clados de Leptospira em amostras de água coletadas de corpos d'água naturais.
Cepas bacterianas
Doze sorovares de Leptospira (Autumnalis, Bataviae, Bratislava, Canicola, Celledoni, Grippothyphosa, Hardjoprajitno, Icterohaemorrhagiae, Pomona, Pyrogenes, Tarassovi e Wolffi) foram incluídos neste estudo. Esses sorovares fazem parte da coleção do Departamento de Microbiologia e Imunologia da Faculdade de Medicina Veterinária e Zootecnia da Universidade Nacional Autônoma do México, e atualmente são utilizados no teste de microaglutinação (SAM).
Todos os sorovares de Leptospira foram cultivados em MCEM e seu DNA foi extraído usando um kit comercial de extração de DNA (ver Tabela de Materiais). Uma mistura de DNA genômico dos doze sorovares foi usada como controle positivo para o clado patogênico Leptospira . Como controle positivo do clado intermediário Leptospira , foi incluído o DNA genômico do sorovar Leptospira fainei Hurstbridge cepa BUT6, e como controle positivo para o clado saprófito Leptospira , o DNA genômico do sorovar Leptospira biflexa Patoc cepa Patoc I.
Os controles negativos consistiram em um plasmídeo vazio, DNA de bactérias não relacionadas (Ureaplasma urealyticum, Staphylococcus aureus, Brucella abortus, Salmonella typhimurium, Shigella boydii, Klebsiella pneumoniae, Acinetobacter baumannii e Escherichia coli) e água de grau PCR, que serviu como controle não modelo.
Amostras de água
Doze amostras experimentais foram coletadas usando um método de amostragem estratificada aleatória do Centro de Pesquisa Biológica e Aquacultural de Cuemanco (CIBAC) (19° 16' 54" N 99° 6' 11" W). Essas amostras foram obtidas em três profundidades: superficial, 10 e 30 cm (Figura 1A, B). Os procedimentos de coleta de água não afetaram nenhuma espécie ameaçada ou protegida. Cada amostra foi coletada em um tubo de microcentrífuga estéril de 15 mL. Para coletar a amostra, cada tubo foi submerso suavemente na água, preenchido na profundidade selecionada e depois selado. As amostras foram mantidas a 22 °C e prontamente transportadas para o laboratório para processamento.
Cada amostra foi concentrada por centrifugação em tubos de microcentrífuga estéreis de 1,5 mL a 8000 x g por 20 min à temperatura ambiente. Essa etapa foi repetida até que todas as amostras estivessem concentradas em um tubo, que foi então usado para extração de DNA (Figura 1C).
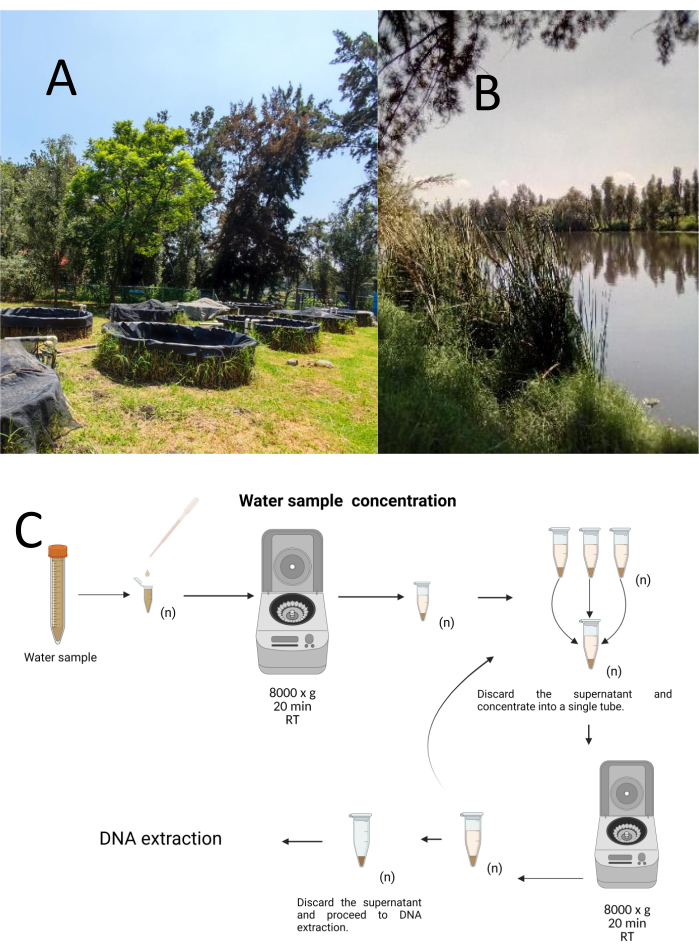
Figura 1: Concentração de amostras de água por centrifugação. (A) Lagoas de amostragem de água e (B) Córregos naturais. (C) Processamento de amostras de água baseado em centrifugação em etapas repetidas quantas vezes forem necessárias (n). Clique aqui para ver uma versão maior desta figura.
Extração de DNA
O DNA total foi isolado usando um kit comercial de DNA genômico de acordo com as instruções do fabricante (ver Tabela de Materiais). As extrações de DNA foram eluídas em 20 μL de tampão de eluição, e a concentração de DNA foi determinada por um espectrofotômetro UV a 260-280 nm e armazenada a 4 °C até o uso.
amplificação por PCR
Os alvos da PCR foram os 16 genesS rRNA, lipL41 e lipL32, que identificam o DNA do gênero Leptospira e permitem a distinção entre os três clados: patogênico, saprófito e intermediário. Tanto os primers quanto os designs de sondas foram baseados nos trabalhos anteriores de Ahmed et al., Azali et al., Bourhy et al., Weiss et al. e Branger et al.33,34,35,36,37. A sequência de cada sonda, primer e fragmento amplificado é descrita na Tabela 1, e seu alinhamento com as sequências de referência é fornecido no Arquivo Suplementar 1, Arquivo Suplementar 2, Arquivo Suplementar 3, Arquivo Suplementar 4 e Arquivo Suplementar 5. Os reagentes de PCR e as condições de termociclagem são descritos na seção de protocolo.
Os produtos de amplificação foram visualizados por separação eletroforética em um gel de agarose a 1% em TAE (40 mM de base Tris, 20 mM de ácido acético e 1 mM de EDTA; pH 8,3), a 60 V por 45 min com detecção de brometo de etídio, conforme mostrado na Figura Suplementar 1. O DNA genômico obtido de cada sorovar foi usado com concentrações variando de 6 x 106 a 1 x 104 cópias equivalentes genômicas (GEq) em cada reação de PCR, com base no tamanho do genoma de L. interrogans (4, 691, 184 pb)38 para Leptospira patogênica, o tamanho do genoma de L. biflexa (3, 956, 088 pb)39 para Leptospira saprófita, e o tamanho do genoma de L. fainei sorovar Hurstbridge cepa BUT6 (4, 267, 324 pb) com número de acesso AKWZ00000000.2.
A sensibilidade das sondas foi avaliada com DNA de cada sorovar patogênico, sorovar L. biflexa Patoc cepa Patoc I e L. fainei sorovar Hurstbridge cepa BUT6 em cada experimento. Para avaliar a especificidade do ensaio de PCR e hibridização dot-blot, foi incluído DNA de bactérias não relacionadas.
Tabela 1: Primers e sondas de PCR para amplificar produtos para identificar os clados patogênicos, saprófitos e intermediários de Leptospira. Clique aqui para baixar esta tabela.
Ensaio de hibridização de ponto-borrão
A técnica é chamada de dot-blot porque os orifícios nos quais a amostra de DNA é colocada têm a forma de um ponto e, quando são sugados para serem fixados no lugar por sucção a vácuo, adquirem essa forma. Essa técnica foi desenvolvida por Kafatos et al.40. A técnica permite a semiquantificação de Leptospira em cada amostra positiva para PCR. O protocolo consiste em uma desnaturação com NaOH 0,4 M à temperatura ambiente, amostras com DNA de Leptospira de 30 ng a 0,05 ng, correspondendo a 6 x 106 a 1 x 104 leptospiras, são borradas em uma membrana de náilon com um aparelho de dot-blot de 96 poços. Após a imobilização, o DNA é ligado à membrana por exposição à luz UV de 120 mJ. Cada sonda de DNA é conjugada com digoxigenina-11 dUTP por uma etapa de catálise de transferase terminal na extremidade 3 '(Digoxigenina é um esteróide vegetal obtido de Digitalis purpurea, usado como repórter41). Após a hibridização rigorosa da sonda de DNA marcada (50 pmol) na temperatura específica no DNA alvo, os híbridos de DNA são visualizados pela reação de quimioluminescência com o anticorpo anti-fosfatase alcalina antidigoxigenina conjugado covalentemente com seu substrato CSPD. A luminescência é capturada pela exposição a um filme de raios-X (Figura 2).
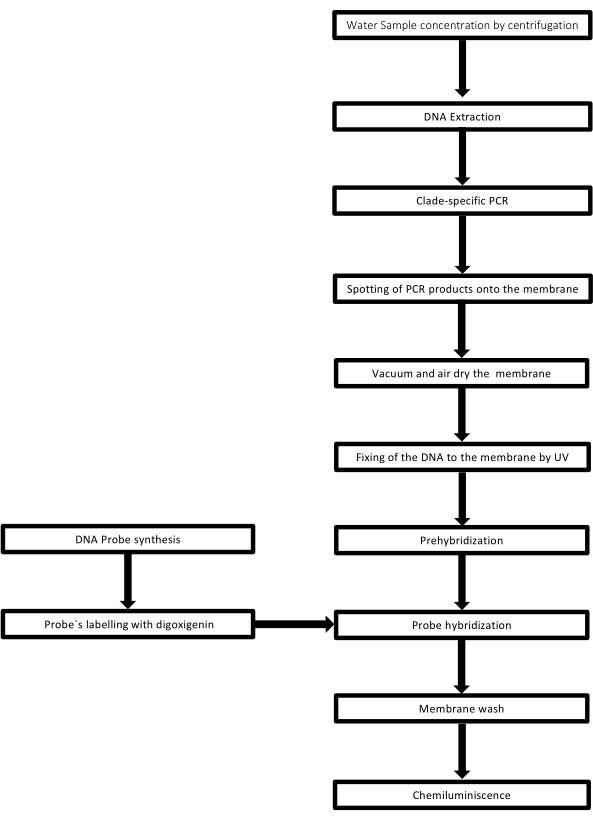
Figura 2: Etapas do procedimento para o ensaio PCR-dot-blot. Clique aqui para ver uma versão maior desta figura.
Protocolo
1. Preparação da amostra
- Concentre cada amostra de água em tubos de microcentrífuga de 1,5 mL por centrifugação a 8.000 x g por 10 min a 4 ° C. Repita esta etapa, quantas vezes forem necessárias, para concentrar a amostra em um volume de 250 μL.
- Use o kit de extração de DNA de acordo com as instruções do fabricante (consulte a Tabela de Materiais).
- Realize a PCR específica de acordo com a sonda dot-blot que será utilizada (Tabela 1).
- Realizar as amplificações em tubos de PCR com o volume final de 25 μL contendo 1 tampão X, 2,5 unidades de Taq polimerase, 1 μM de cada primer, 0,2 mM de cada dNTP, 1,5 mM MgCl2 e 100 ng de DNA alvo de cada amostra de água ou DNA genômico de referência.
- Programe a reação de PCR em um termociclador de acordo com as condições de termociclagem (Tabela 2).
- Conservar as reacções a 4 °C até à utilização.
- Coloque 10 μL de cada produto de PCR a ser blotted em um poço separado de uma placa de 96 poços.
- Adicione 40 μL de TE a cada poço e misture por pipetagem.
NOTA: A placa de 96 poços pode ser selada e armazenada a 4 °C durante a noite. Siga as instruções fornecidas no Arquivo Suplementar 6 para preparar buffers e soluções para o protocolo. A Figura 3 mostra as etapas de preparo da amostra.
Tabela 2: Condições de termociclagem de PCR para os genes 16S, lipL41 e lipL32. Clique aqui para baixar esta tabela.
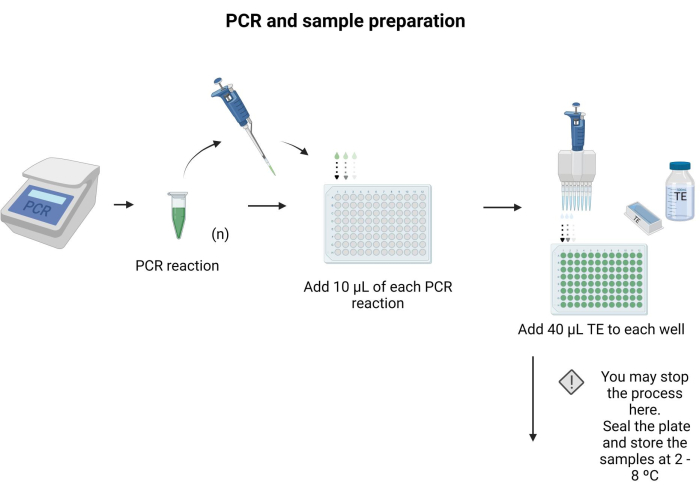
Figura 3: PCR e preparo de amostras. Aplicando o protocolo de PCR específico, o produto da PCR foi transferido para uma placa de microtitulação e 40 μL de TE foram adicionados a cada poço. Clique aqui para ver uma versão maior desta figura.
2. Montagem do aparelho de dot-blot
NOTA: A montagem do aparelho de dot-blot é mostrada na Figura 4. Durante o procedimento, use luvas para manusear as soluções alcalinas e proteja a membrana de náilon contra contaminação.
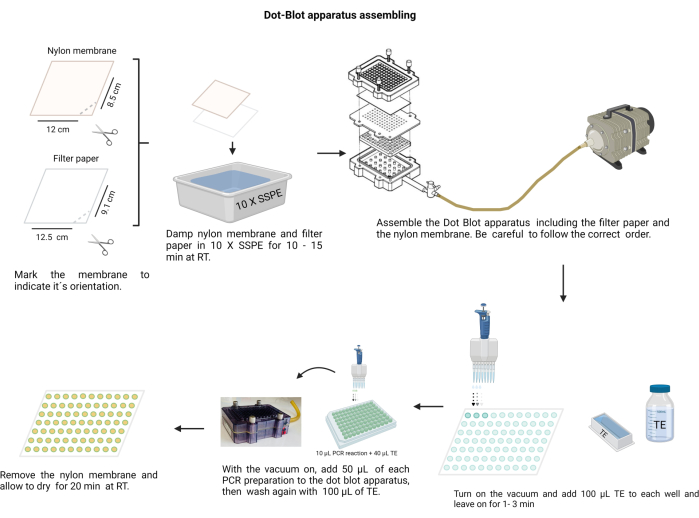
Figura 4: Montagem do aparelho de dot-blot. O papel de filtro e a membrana de nylon (previamente umedecida em 10 X SSPE) devem ser dispostos na ordem correta. O conjunto deve ser fixado com os parafusos firmemente antes de aplicar o vácuo. Cada poço precisa ser lavado com TE e os produtos de PCR são carregados em seus respectivos poços. Após a transferência do produto de PCR através da membrana, cada poço é lavado novamente com TE e deixado secar. Clique aqui para ver uma versão maior desta figura.
- Corte a membrana de náilon (consulte a Tabela de Materiais) e o papel de filtro em folhas de tamanho 12 x 8,5 cm.
- Marque a membrana com um marcador permanente.
- Faça um entalhe com uma tesoura em uma das bordas para indicar a orientação correta. Use a marca para lembrar o pedido da amostra.
- Umedeça a membrana e o papel de filtro com 10 X tampão de fosfato de sódio salino-EDTA (SSPE; 3 M NaCl, 0.2 M NaH2PO4 e 0.02 M EDTA, pH 7.4) (Arquivo Suplementar 6). Manuseie a membrana com uma pinça limpa e sem corte.
- Monte a câmara de mancha de pontos; Primeiro, o papel de filtro, seguido pela membrana sobre o selo de plástico. Prenda a tampa com os parafusos transversalmente.
- Conecte a câmara ao vácuo, coloque 100 μL de TE em cada poço, mantenha o vácuo por 1 min e depois pare-o.
- Ligue o vácuo em baixa velocidade e carregue 50 μL de cada amostra no poço correspondente na membrana do aparelho de dot-blot (seguindo a distribuição de membrana pré-decidida).
NOTA: Homogeneizar cada amostra na placa de hemaglutinação de 96 poços antes de colocá-la na câmara de mancha de pontos. - Deixe o vácuo secar a membrana. Se necessário, bata suavemente na câmara para liberar as bolhas na amostra.
- Lave cada poço colocando 100 μL de TE nele com um vácuo contínuo e deixando-o secar.
NOTA: Depois de concluir a transferência, é crucial primeiro desligar a bomba e desconectá-la. Não fazer isso pode resultar em refluxo entrando no aparelho. O DNA não precisa de uma etapa de desnaturação se uma membrana de náilon carregada positivamente for usada, mas se estiver usando outro suporte inerte, uma etapa de pré-desnaturação e desnaturação à base de álcalis pode ser necessária41.
3. Desnaturação e fixação do ADN
NOTA: A Figura 5 ilustra o procedimento de fixação da membrana de DNA.
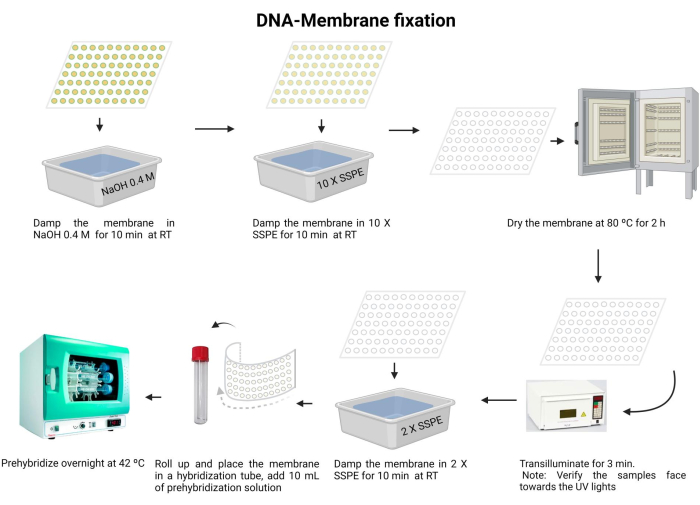
Figura 5: Procedimento de fixação da membrana de DNA. O DNA é desnaturado em uma solução alcalina. Em seguida, é neutralizado com 10 X SSPE e a membrana é seca. Em seguida, a membrana é transiluminada. A membrana é reidratada com 2 X SSPE e pré-hibridizada durante a noite. Clique aqui para ver uma versão maior desta figura.
- Incube a membrana com NaOH 0,4 M por 10 min em temperatura ambiente.
- Equilibre a membrana com 10 X SSPE por 10 min em temperatura ambiente.
- Secar a membrana a 80 °C durante 2 h.
- Transilumine com luz UV de 120 mJ por 3 min. Certifique-se de que as amostras estejam orientadas para baixo em direção à fonte de luz UV. Se estiver usando um reticulador UV, confirme se as amostras estão voltadas para cima e repita esta etapa duas vezes.
NOTA: A membrana deve estar completamente seca antes da reticulação UV. O DNA será imobilizado por uma ligação covalente com a membrana de náilon. Cada reticulação leva cerca de 18 '' a 1 '' com uma dose ideal de irradiação UV, para a maioria dos experimentos de hibridização, de aproximadamente 0,6-0,8 kJ / m2.
A exposição à irradiação UV é prejudicial aos olhos e à pele. Use equipamento de proteção adequado e evite a exposição à pele nua. - Lave a membrana com 2 X SSPE por 10 min.
- Dobre cuidadosamente a membrana e insira-a no tubo de hibridização (use um tubo de microcentrífuga de 15 mL).
- Adicionar 10 ml da solução de pré-hibridização (lima suplementar 6) ao tubo e incubar a 42 °C durante a noite. Certifique-se de que o tubo foi devidamente vedado para evitar qualquer vazamento.
4. Hibridização
- Remova 5 mL da solução de pré-hibridização do tubo de hibridização. Para utilizar os 5 ml pela segunda vez, mantenha-os noutro tubo e guarde-os a -4 °C.
- Adicione 15 μL da sonda marcada (Tabela 1) ao tubo de hibridização. Enxágue a ponta na solução para garantir que a sonda rotulada foi completamente instilada na diluição.
NOTA: A ligação da sonda de DNA não é tão forte quanto a ligação antígeno-anticorpo. Portanto, mudanças na concentração da sonda ou DNA na amostra podem influenciar a intensidade da fluorescência emitida. As sondas de DNA são estequiométricas, o que significa que o número de moléculas de sonda ligadas ao DNA é equivalente ao número de moléculas de DNA presentes na solução. A Figura 6 mostra a hibridização da sonda. - Incubar a 42 °C durante a noite.
- Execute a marcação DIG da sonda de DNA. Siga as instruções especificadas na Tabela 3 para misturar os reagentes e, em seguida, incubar a mistura a 37 °C por 1 h. Em seguida, adicione 80 μL de água destilada e armazene a sonda de cauda a 4 °C.
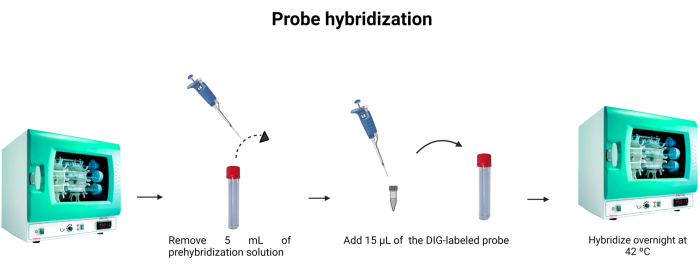
Figura 6: Hibridização da sonda. O volume do tampão de hibridização é ajustado e a sonda marcada com digoxigenina é incorporada para permitir a hibridização da sonda durante a noite. Clique aqui para ver uma versão maior desta figura.
Tabela 3: Reagentes para a marcação das sondas com digoxigenina (DIG). Clique aqui para baixar esta tabela.
5. Quimioluminescência (marcação anti-DIG)
- Lave a membrana duas vezes com 2 X SSPE/0.1% SDS em temperatura ambiente por 10 min.
- Incube a membrana com 5 X SSPE/0,1% SDS na temperatura de recozimento da sonda por 15 min.
NOTA: Nesta etapa, use um recipiente com tampa para manter a temperatura o maior tempo possível. Garanta uma temperatura uniforme e constante em toda a membrana. O volume aproximado em cada lavagem é de 100 mL da solução indicada. - Lave a membrana uma vez com o Tampão 1 (Arquivo Suplementar 6) em temperatura ambiente por 5 min.
- Lave a membrana uma vez com o Tampão 2 (Arquivo Suplementar 6) em temperatura ambiente por 30 min.
- Lave a membrana com o tampão 2 e adicione o anticorpo anti-digoxigenina (15 μL) (consulte a Tabela de Materiais) e incube por 45 min (o tempo de incubação pode variar de 30 a 60 min). Este anticorpo secundário marca a sonda para detecção.
NOTA: A membrana pode ser armazenada no tampão 2 com o anticorpo anti-digoxigenina durante a noite a 4-8 °C. A marcação anti-DIG do processo de quimioluminescência é representada na Figura 7.
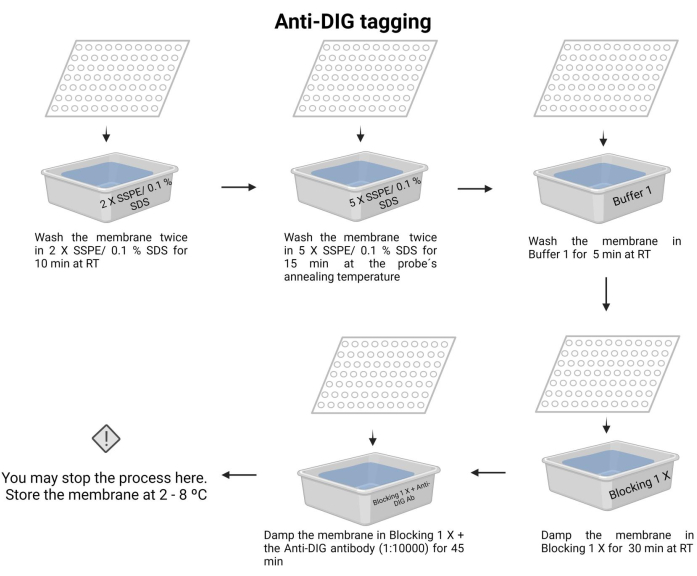
Figura 7: Marcação anti-DIG do processo de quimioluminescência. Os ácidos nucléicos não ligados são removidos com soluções tampão. A sonda é alinhada com o DNA alvo e o excesso é removido. A membrana é bloqueada com o tampão 1 X de bloqueio e o anticorpo anti-DIG é adicionado (1:10000). Clique aqui para ver uma versão maior desta figura.
6. Quimioluminescência (aplicação de substrato)
- Lave a membrana duas vezes no tampão 1 em temperatura ambiente por 15 min.
- Lave uma vez no tampão 3 (consulte o arquivo suplementar 6) em temperatura ambiente por 5 min, permitindo que a membrana drene a maior parte do tampão, deixando-o quase seco.
- Adicione CSPD pronto para uso (3-(4-metoxispiro dissódico {1,2-dioxetano-3,2′-(5′-cloro) triciclo-[3.3.1.13,7] decanto}-4-il) fenilfosfato, consulte a Tabela de Materiais) e deixe descansar por 5 min (com os dedos umedecidos homogeneizar o CSPD de um lado para o outro).
- Coloque a membrana em um saco plástico transparente (12,5 x 9 cm) que se ajuste à dimensão da membrana. Remova cuidadosamente as bolhas de ar, distribua uniformemente o CSPD e sele o saco a quente.
- Incubar a membrana em banho-maria a 37 °C durante 15 min (pode ser até 30 min) e assegurar que a membrana com as amostras é colocada para baixo de modo a ficar bem submersa. Certifique-se de que a temperatura permaneça constante e bem distribuída sobre a membrana.
- Seque o saco plástico e fixe-o com fita adesiva em um filme radiográfico já usado. Isso permitirá um manuseio fácil durante o procedimento de exposição.
NOTA: A Figura 8 mostra a aplicação do substrato do processo de quimioluminescência.

Figura 8: Aplicação do substrato do processo de quimioluminescência. O anticorpo livre é removido e o substrato CSPD (1:250) é adicionado à membrana. A reação é ativada por incubação a 37 °C e a membrana é disposta para registrar a quimioluminescência em um filme de raios-X. Clique aqui para ver uma versão maior desta figura.
7. Quimioluminescência (detecção)
- Execute o procedimento de exposição. Na câmara escura, prepare as soluções de revelador e fixador (ver Tabela de Materiais) nas respectivas bandejas.
- No escuro, coloque cuidadosamente a membrana fixa voltada para um novo filme radiográfico e insira-a em um radiográfico (consulte a Tabela de Materiais).
- Registre o tempo de exposição. O tempo de exposição pode variar de 1 min a 30 min ou mais. Comece com 5 min e ajuste o tempo de acordo.
- Umedeça o filme de raios-X na solução reveladora por 1-3 min e enxágue suavemente com água da torneira (15-45 s).
- Umedeça o filme de raios-X em solução fixadora por 1-3 min e enxágue suavemente com água da torneira (45 s).
- Deixe o filme de raios-X secar ao ar e documente o ensaio em uma caixa de luz branca visível.
NOTA: Evite agitar a membrana durante o tempo de exposição aos raios X. O processo de detecção é representado na Figura 9. - Prossiga para a interpretação do resultado. No filme de raios-X exposto, os pontos com emissão podem ser localizados visualmente e correspondem aos fragmentos de DNA com a hibridização da sonda. A intensidade do sinal emitido depende da luminescência e da duração da exposição.
NOTA: As membranas podem ser armazenadas secas entre o papel de filtro por vários meses em temperatura ambiente.
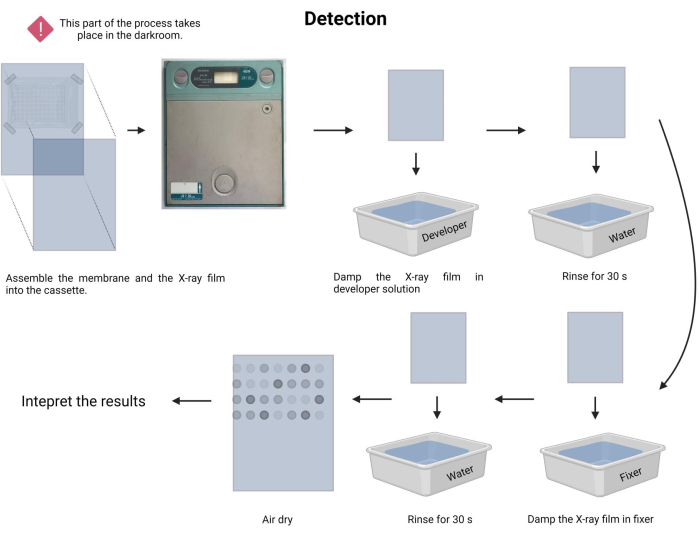
Figura 9: Detecção do processo de quimioluminescência. Em condições escuras, a membrana é exposta a um filme de raios-X dentro de um de raios-X. Em seguida, foi permitido permanecer durante o tempo de exposição e, em seguida, o filme de raios-X foi revelado e fixado. Finalmente, foi seco ao ar e interpretado. Clique aqui para ver uma versão maior desta figura.
8. Procedimento de deshibridização por membrana
- Lave a membrana duas vezes por 10 min com água destilada.
- Lave a membrana por 20 min com NaOH 0,4 M a 53 °C (duas vezes).
- Lave duas vezes a membrana por 10 min com 2 X SSPE.
- Deixe a membrana secar à temperatura ambiente.
- Guarde a membrana e retorne à etapa 3.5 do protocolo.
NOTA: A membrana pode ser armazenada no tampão de pré-hibridização durante a noite a 4-8 °C. No dia seguinte, reiniciar com uma incubação a 42 °C durante 1 h, permitindo que o tampão de pré-hibridização atinja a temperatura. Em seguida, prossiga para a etapa 3.5 do protocolo.
Resultados
Para avaliar a eficácia da técnica, foi utilizado DNA genômico de culturas puras de cada sorovar de Leptospira , juntamente com a sonda específica do clado. As membranas foram preparadas com 100 ng de DNA genômico por reação de PCR para cada sorovar, seguido por oito DNA genômico de bactérias não relacionadas e concentrações variáveis de DNA genômico dos sorovares ad hoc de Leptospira . Cada ensaio incluiu controle positivo, negativo e sem modelo. Esses DNAs genômicos não relacionados n?...
Discussão
As etapas críticas da técnica de dot-blot incluem (1) imobilização de DNA, (2) bloqueio dos locais de ligação livres na membrana com DNA não homólogo, (3) a complementaridade entre a sonda e o fragmento alvo sob condições de recozimento, (4) remoção da sonda não hibridizada e (5) a detecção da molécula repórter41.
O PCR-Dot-blot tem certas limitações, como a técnica não fornece informações sobre o tamanho do fragmento hibridizado...
Divulgações
Os autores declaram não haver conflito de interesses.
Agradecimentos
Estamos em dívida com a coleção Leptospira do Departamento de Microbiologia e Imunologia da Faculdade de Medicina Veterinária e Zootecnia da Universidade Nacional Autônoma do México. Agradecemos a generosa doação das cepas de referência de Leptospira ; Leptospira fainei sorovar Hurstbridge cepa BUT6 e Leptospira biflexa sorovar Patoc cepa Patoc I para o Dr. Alejandro de la Peña Moctezuma. Agradecemos ao Dr. José Antonio Ocampo Cervantes, Coordenador do CIBAC, e ao pessoal pelo apoio logístico. A EDT estava no âmbito do programa Terminal Project para alunos de graduação da Universidade Autônoma Metropolitana-Campus Cuajimalpa. Reconhecemos o Biorender.com software para a criação das figuras 1 e 3 a 9.
Materiais
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| Purelink DNA extraction kit | Invitrogen | K182002 | |
| Gotaq Flexi DNA Polimerase (End-Point PCR Taq polymerase kit) | Promega | M3001 | |
| Whatman filter paper, grade 1, | Merk | WHA1001325 | |
| Nylon Membranes, positively charged Roll 30cm x 3 m | Roche | 11417240001 | |
| Anti-Digoxigenin-AP, Fab fragments Sheep Polyclonal Primary-antibody | Roche | 11093274910 | |
| Medium Base EMJH | Difco | S1368JAA | |
| Leptospira Enrichment EMJH | Difco | BD 279510 | |
| Blocking Reagent | Roche | 11096176001 | |
| CSPD ready to use Disodium 3-(4-methoxyspiro {1,2-dioxetane-3,2′-(5′-chloro) tricyclo [3.3.1.13,7] decan}8-4-yl) phenyl phosphate | Merk | 11755633001 | |
| Deoxyribonucleic acid from herring sperm | Sigma Aldrich | D3159 | |
| Developer Carestream | Carestream Health Inc | GBX5158621 | |
| Digoxigenin-11-ddUTP | Roche | 11363905910 | |
| EDTA, Disodium Salt (Dihydrate) | Promega | H5032 | |
| Ficoll 400 | Sigma Aldrich | F8016 | |
| Fixer Carestream | Carestream Health Inc | GBX 5158605 | |
| Lauryl sulfate Sodium Salt (Sodium dodecyl sulfate; SDS) C12H2504SNa | Sigma Aldrich | L5750 | |
| N- Lauroylsarcosine sodium salt CH3(CH2)10CON(CH3) CH2COONa | Sigma Aldrich | L-9150 | It is an anionic surfactant |
| Polivinylpyrrolidone (PVP-40) | Sigma Aldrich | PVP40 | |
| Polyethylene glycol Sorbitan monolaurate (Tween 20) | Sigma Aldrich | 9005-64-5 | |
| Sodium Chloride (NaCl) | Sigma Aldrich | 7647-14-5 | |
| Sodium dodecyl sulfate (SDS) | Sigma Aldrich | 151-21-3 | |
| Sodium hydroxide (NaOH) | Sigma Aldrich | 1310-73-2 | |
| Sodium phosphate dibasic (NaH2PO4) | Sigma-Aldrich | 7558-79-4 | |
| Terminal transferase, recombinant | Roche | 3289869103 | |
| Tris hydrochloride (Tris HCl) | Sigma-Aldrich | 1185-53-1 | |
| SSPE 20X | Sigma-Aldrich | S2015-1L | It can be Home-made following Supplementary File 6 |
| Primers | Sigma-Aldrich | On demand | Follow table 1 |
| Probes | Sigma-Aldrich | On demand | Follow table 1 |
| Equipment | |||
| Nanodrop™ One Spectrophotometer | Thermo-Scientific | ND-ONE-W | |
| Refrigerated microcentrifuge Sigma 1-14K, suitable for centrifugation of 1.5 ml microcentrifuge tubes at 14,000 rpm | Sigma-Aldrich | 1-14K | |
| Disinfected adjustable pipettes, range 2-20 µl, 20-200 µl | Gilson | SKU:F167360 | |
| Disposable 1.5 ml microcentrifuge tubes (autoclaved) | Axygen | MCT-150-SP | |
| Disposable 600 µl microcentrifuge tubes (autoclaved) | Axygen | 3208 | |
| Disposable Pipette tips 1-10 µl | Axygen | T-300 | |
| Disposable Pipette tips 1-200 µl | Axygen | TR-222-Y | |
| Dot-Blot apparatus Bio-Dot | BIORAD | 1706545 | |
| Portable Hergom Suction | Hergom | 7E-A | |
| Scientific Light Box (Visible-light PH90-115V) | Hoefer | PH90-115V | |
| UV Crosslinker | Hoefer | UVC-500 | |
| Thermo Hybaid PCR Express Thermocycler | Hybaid | HBPX110 | |
| Radiographic cassette with IP Plate14 X 17 | Fuji |
Referências
- Bierque, E., Thibeaux, R., Girault, D., Soupé-Gilbert, M. E., Goarant, C. A systematic review of Leptospira in water and soil environments. PLOS One. 15 (1), e0227055 (2020).
- Haake, D. A., Levett, P. N. Leptospirosis in humans. Current Topics in Microbiology and Immunology. 387, 65-97 (2015).
- Tripathy, D. N., Hanson, L. E. Leptospires from water sources at Dixon Springs Agricultural Center. Journal of Wildlife Diseases. 9 (3), 209-212 (1973).
- Smith, D. J., Self, H. R. Observations on the survival of Leptospira australis A in soil and water. The Journal of Hygiene. 53 (4), 436-444 (1955).
- Karpagam, K. B., Ganesh, B. Leptospirosis: a neglected tropical zoonotic infection of public health importance-an updated review. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 39 (5), 835-846 (2020).
- Casanovas-Massana, A., et al. Spatial and temporal dynamics of pathogenic Leptospira in surface waters from the urban slum environment. Water Research. 130, 176-184 (2018).
- Costa, F., et al. Global morbidity and mortality of Leptospirosis: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003898 (2015).
- Mwachui, M. A., Crump, L., Hartskeerl, R., Zinsstag, J., Hattendorf, J. Environmental and behavioural determinants of Leptospirosis transmission: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003843 (2015).
- Andre-Fontaine, G., Aviat, F., Thorin, C. Waterborne Leptospirosis: Survival and preservation of the virulence of pathogenic Leptospira spp. in fresh water. Current Microbiology. 71 (1), 136-142 (2015).
- Trueba, G., Zapata, S., Madrid, K., Cullen, P., Haake, D. Cell aggregation: A mechanism of pathogenic Leptospira to survive in freshwater. International Microbiology: the Official Journal of the Spanish Society for Microbiology. 7 (1), 35-40 (2004).
- Smith, C. E., Turner, L. H. The effect of pH on the survival of leptospires in water. Bulletin of the World Health Organization. 24 (1), 35-43 (1961).
- Barragan, V. A., et al. Interactions of Leptospira with environmental bacteria from surface water. Current Microbiology. 62 (6), 1802-1806 (2011).
- Abdoelrachman, R. Comparative investigations into the influence of the presence of bacteria on the life of pathogenic and apathogenic leptospirae. Antonie van Leeuwenhoek. 13 (1), 21-32 (1947).
- Singh, R., et al. Microbial diversity of biofilms in dental unit water systems. Applied and Environmental Microbiology. 69 (6), 3412-3420 (2003).
- Kumar, K. V., Lall, C., Raj, R. V., Vedhagiri, K., Vijayachari, P. Coexistence and survival of pathogenic leptospires by formation of biofilm with Azospirillum. FEMS Microbiology Ecology. 91 (6), 051 (2015).
- Yanagihara, Y., et al. Leptospira Is an environmental bacterium that grows in waterlogged soil. Microbiology Spectrum. 10 (2), 0215721 (2022).
- Gillespie, R. W., Ryno, J. Epidemiology of leptospirosis. American Journal of Public Health and Nation’s Health. 53 (6), 950-955 (1963).
- Bierque, E., et al. Leptospira interrogans retains direct virulence after long starvation in water. Current Microbiology. 77 (10), 3035-3043 (2020).
- Zhang, Y., Dai, B. Marking and detection of DNA of leptospires in the dot-blot and situ hybridization with digoxigenin-labeled probes. Journal of West China University of Medical Sciences. 23 (4), 353-435 (1992).
- Mérien, F., Amouriaux, P., Perolat, P., Baranton, G., Saint Girons, I. Polymerase chain reaction for detection of Leptospira spp. in clinical samples. Journal of Clinical Microbiology. 30 (9), 2219-2224 (1992).
- Veerapandian, R., et al. Silver enhanced nano-gold dot-blot immunoassay for leptospirosis. Journal of Microbiological Methods. 156, 20-22 (2019).
- Junpen, S., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Ishmael, F. T., Stellato, C. Principles and applications of polymerase chain reaction: basic science for the practicing physician. Annals of Allergy, Asthma & Immunology: Official Publication of the American College of Allergy, Asthma, & Immunology. 101 (4), 437-443 (2008).
- Boerner, B., Weigelt, W., Buhk, H. J., Castrucci, G., Ludwig, H. A sensitive and specific PCR/Southern blot assay for detection of bovine herpesvirus 4 in calves infected experimentally. Journal of Virological Methods. 83 (1-2), 169-180 (1999).
- Curry, E., Pratt, S. L., Kelley, D. E., Lapin, D. R., Gibbons, J. R. Use of a Combined duplex PCR/Dot-blot assay for more sensitive genetic characterization. Biochemistry Insights. 1, 35-39 (2008).
- Pilatti, M. M., Ferreira, S. d. e. A., de Melo, M. N., de Andrade, A. S. Comparison of PCR methods for diagnosis of canine visceral leishmaniasis in conjunctival swab samples. Research in Veterinary Science. 87 (2), 255-257 (2009).
- Conrads, G., et al. PCR reaction and dot-blot hybridization to monitor the distribution of oral pathogens within plaque samples of periodontally healthy individuals. Journal of Periodontology. 67 (10), 994-1003 (1996).
- Langa, S., et al. Differentiation of Enterococcus faecium from Lactobacillus delbrueckii subsp. bulgaricus and Streptococcus thermophilus strains by PCR and dot-blot hybridisation. International Journal of Food Microbiology. 88 (2-3), 197-200 (2003).
- Francesca, C., Lucilla, I., Marco, F., Giuseppe, C., Marisa, M. Identification of the unculturable bacteria Candidatus arthromitus in the intestinal content of trouts using dot-blot and Southern blot techniques. Veterinary Microbiology. 156 (3-4), 389-394 (2012).
- Arent, Z., Pardyak, L., Dubniewicz, K., Plachno, B. J., Kotula-Balak, M. Leptospira taxonomy: then and now. Medycyna Weterynaryjna. 78 (10), 489-496 (2022).
- Thibeaux, R., et al. Biodiversity of environmental Leptospira: Improving identification and revisiting the diagnosis. Frontiers in Microbiology. 9, 816 (2018).
- Carrillo-Casas, E. M., Hernández-Castro, R., Suárez-Güemes, F., de la Peña-Moctezuma, A. Selection of the internal control gene for real-time quantitative RT-PCR assays in temperature treated Leptospira. Current Microbiology. 56 (6), 539-546 (2008).
- Azali, M. A., Yean Yean, C., Harun, A., Aminuddin Baki A, N. N., Ismail, N. Molecular characterization of Leptospira spp. in environmental samples from North-Eastern Malaysia revealed a pathogenic strain, Leptospira alstonii. Journal of Tropical Medicine. 2016, 2060241 (2016).
- Ahmed, N., et al. Multilocus sequence typing method for identification and genotypic classification of pathogenic Leptospira species. Annals of Clinical Microbiology and Antimicrobials. 5, 28 (2006).
- Bourhy, P., Collet, L., Brisse, S., Picardeau, M. Leptospira mayottensis sp. nov., a pathogenic species of the genus Leptospira isolated from humans. International Journal of Systematic and Evolutionary Microbiology. 64, 4061-4067 (2014).
- Weiss, S., et al. An extended Multilocus Sequence Typing (MLST) scheme for rapid direct typing of Leptospira from clinical samples. PLOS Neglected Tropical Diseases. 10 (9), e0004996 (2016).
- Branger, C., et al. Polymerase chain reaction assay specific for pathogenic Leptospira based on the gene hap1 encoding the hemolysis-associated protein-1. FEMS Microbiology Letters. 243 (2), 437-445 (2005).
- Ren, S. X., et al. Unique physiological and pathogenic features of Leptospira interrogans revealed by whole-genome sequencing. Nature. 422 (6934), 888-893 (2003).
- Picardeau, M., et al. Genome sequence of the saprophyte Leptospira biflexa provides insights into the evolution of Leptospira and the pathogenesis of leptospirosis. PLOS One. 3 (2), e1607 (2008).
- Kafatos, F. C., Jones, C. W., Efstratiadis, A. Determination of nucleic acid sequence homologies and relative concentrations by a dot hybridization procedure. Nucleic Acids Research. 7 (6), 1541-1552 (1979).
- Bhat, A. I., Rao, G. P. Dot-blot hybridization technique. Characterization of Plant Viruses. , 303-321 (2020).
- Yadav, J. P., Batra, K., Singh, Y., Singh, M. Comparative evaluation of indirect-ELISA and Dot-blot assay for serodetection of Mycoplasma gallisepticum and Mycoplasma synoviae antibodies in poultry. Journal of Microbiological Methods. 189, 106317 (2021).
- Malinen, E., Kassinen, A., Rinttilä, T., Palva, A. Comparison of real-time PCR with SYBR Green I or 5'-nuclease assays and dot-blot hybridization with rDNA-targeted oligonucleotide probes in quantification of selected faecal bacteria. Microbiology. 149, 269-277 (2003).
- Wyss, C., et al. Treponema lecithinolyticum sp. nov., a small saccharolytic spirochaete with phospholipase A and C activities associated with periodontal diseases. International Journal of Systematic Bacteriology. 49, 1329-1339 (1999).
- Shah, J. S., I, D. C., Ward, S., Harris, N. S., Ramasamy, R. Development of a sensitive PCR-dot-blot assay to supplement serological tests for diagnosing Lyme disease. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 37 (4), 701-709 (2018).
- Niu, C., Wang, S., Lu, C. Development and evaluation of a dot-blot assay for rapid determination of invasion-associated gene ibeA directly in fresh bacteria cultures of E. coli. Folia microbiologica. 57 (6), 557-561 (2012).
- Wetherall, B. L., McDonald, P. J., Johnson, A. M. Detection of Campylobacter pylori DNA by hybridization with non-radioactive probes in comparison with a 32P-labeled probe. Journal of Medical Microbiology. 26 (4), 257-263 (1988).
- Kolk, A. H., et al. Detection of Mycobacterium tuberculosis in clinical samples by using polymerase chain reaction and a nonradioactive detection system. Journal of Clinical Microbiology. 30 (10), 2567-2575 (1992).
- Scherer, L. C., et al. PCR colorimetric dot-blot assay and clinical pretest probability for diagnosis of Pulmonary Tuberculosis in smear-negative patients. BMC Public Health. 7, 356 (2007).
- Armbruster, D. A., Pry, T. Limit of blank, limit of detection and limit of quantitation. The Clinical Biochemist Reviews. 29, S49-S52 (2008).
- Zhang, Y., Dai, B. Detection of Leptospira by dot-blot hybridization with photobiotin- and 32P-labeled DNA. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 130-132 (1992).
- Terpstra, W. J., Schoone, G. J., ter Schegget, J. Detection of leptospiral DNA by nucleic acid hybridization with 32P- and biotin-labeled probes. Journal of Medical Microbiology. 22 (1), 23-28 (1986).
- Shukla, J., Tuteja, U., Batra, H. V. DNA probes for identification of leptospires and disease diagnosis. The Southeast Asian Journal of Tropical Medicine and Public Health. 35 (2), 346-352 (2004).
- Jiang, N., Jin, B., Dai, B., Zhang, Y. Identification of pathogenic and nonpathogenic leptospires by recombinant probes. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 26 (1), 1-5 (1995).
- Fach, P., Trap, D., Guillou, J. P. Biotinylated probes to detect Leptospira interrogans on dot-blot hybridization or by in situ hybridization. Letters in Applied Microbiology. 12 (5), 171-176 (1991).
- Huang, N., Dai, B. Assay of genomic DNA homology among strains of different virulent leptospira by DNA hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 122-125 (1992).
- Dong, X., Dai, B., Chai, J. Homology study of leptospires by molecular hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (1), 1-4 (1992).
- Komminoth, P. Digoxigenin as an alternative probe labeling for in situ hybridization. Diagnostic Molecular Pathology: The American Journal of Surgical Pathology, part B. 1 (2), 142-150 (1992).
- Saengjaruk, P., et al. Diagnosis of human leptospirosis by monoclonal antibody-based antigen detection in urine. Journal of Clinical Microbiology. 40 (2), 480-489 (2002).
- Okuda, M., et al. Enzyme-linked immunosorbent assay for the detection of canine Leptospira antibodies using recombinant OmpL1 protein. The Journal of Veterinary Medical Science. 67 (3), 249-254 (2005).
- Suwimonteerabutr, J., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Kanagavel, M., et al. Peptide-specific monoclonal antibodies of Leptospiral LigA for acute diagnosis of leptospirosis. Scientific reports. 7 (1), 3250 (2017).
- Levett, P. N. Leptospirosis. Clinical Microbiology Reviews. 14 (2), 296-326 (2001).
- Monahan, A. M., Callanan, J. J., Nally, J. E. Proteomic analysis of Leptospira interrogans shed in urine of chronically infected hosts. Infection and Immunity. 76 (11), 4952-4958 (2008).
- Rojas, P., et al. Detection and quantification of leptospires in urine of dogs: a maintenance host for the zoonotic disease leptospirosis. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 29 (10), 1305-1309 (2010).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados