Method Article
Preparación de núcleos cerebrales y de médula ósea de alta calidad para ensayos multioma de núcleos individuales
En este artículo
Resumen
El éxito de la transcriptómica y multiómica de una sola célula/núcleo único depende en gran medida de la calidad de las células/núcleos. Por lo tanto, el aislamiento de células/núcleos de los tejidos y su purificación debe estar altamente estandarizado. Este protocolo describe la preparación de núcleos del cerebro y la médula ósea para el ensayo multioma de un solo núcleo aguas abajo.
Resumen
El análisis de una sola célula se ha convertido en el enfoque de elección para desentrañar la complejidad de los procesos biológicos que requieren evaluar la variabilidad de las respuestas celulares individuales al tratamiento o la infección con resolución de una sola célula.
En los últimos 10 años se han desarrollado muchas técnicas para el perfil molecular de una sola célula, y se han comercializado varias tecnologías específicas. El perfil de una sola célula basado en gotas de 10X Genomics es una tecnología generalizada que ofrece reactivos listos para usar para el perfil transcriptómico y multiómico de una sola célula. La tecnología incluye flujos de trabajo para la secuenciación de ARN de una sola célula y de un solo núcleo (scRNA-Seq y snRNA-Seq, respectivamente), scATAC-Seq, perfil inmunitario de una sola célula (secuenciación BCR/TCR) y multioma. Este último combina información transcripcional (scRNA-Seq) y epigenética (scATAC-Seq) procedente de la misma célula.
La calidad (viabilidad, integridad, pureza) de las suspensiones unicelulares o de un solo núcleo aisladas de los tejidos y analizadas por cualquiera de estos enfoques es fundamental para generar datos de alta calidad. Por lo tanto, los protocolos de preparación de muestras deben adaptarse a las particularidades de cada tejido biológico y asegurar la generación de suspensiones celulares y núcleos de alta calidad.
En este artículo se describen dos protocolos para preparar muestras de cerebro y médula ósea para la canalización de genómica multiome 10X aguas abajo. Los protocolos se realizan paso a paso y cubren la disociación de tejidos, la clasificación celular, el aislamiento de núcleos y el control de calidad de la suspensión de núcleos preparados que se utiliza como material de partida para la partición celular y el código de barras, la preparación de bibliotecas y la secuenciación. Estos protocolos estandarizados producen bibliotecas de núcleos de alta calidad y datos robustos y confiables.
Introducción
Durante muchos años, las técnicas unicelulares han sido el estándar de oro para el análisis de procesos biológicos. Inicialmente se restringieron al fenotipado unicelular a través de microscopía, citometría de flujo y ensayos similares. Un gran avance en el análisis de una sola célula se produjo con el desarrollo de enfoques para el perfil molecular de una sola célula, en particular la secuenciación de ARN de una sola célula (scRNA-Seq) que permitió caracterizar todo el transcriptoma de células individuales. Muy potente, scRNA-Seq genera información sobre el estado transcripcional de una célula en una condición y un punto de tiempo específicos. Sin embargo, no proporciona visibilidad sobre la regulación génica que impulsa la transcripción, ni sobre las modificaciones moleculares que se producen a lo largo del tiempo. Para superar esta limitación, se han invertido muchos esfuerzos en el desarrollo de ensayos multiómicos unicelulares que permitan el análisis de múltiples factores y procesos a partir de una misma célula 1,2,3,4. La primera medición exitosa de dos modalidades dentro de células individuales se produjo mediante la vinculación de patrones de expresión de proteínas de superficie múltiple con el transcriptoma completo de células individuales en el enfoque CITE-Seq5. Las evoluciones más recientes combinan la expresión génica con la accesibilidad a la cromatina (Ensayo de cromatina accesible a la transposasa mediante secuenciación, ATAC-Seq), capturando así simultáneamente las modalidades transcriptómicas y epigenómicas en las mismas células (por ejemplo, sci-CAR)6. Las primeras soluciones comerciales que permitieron asociar la transcriptómica con el fenotipo celular o con cambios epigenéticos de la misma célula vinieron de la mano de 10X Genomics.
Los experimentos para el perfil molecular de una sola célula contienen los siguientes pasos: (1) disociación de tejidos o preparación de suspensiones de una sola célula; (2) purificación celular y/o aislamiento de núcleos; (3) partición y código de barras; (4) construcción de bibliotecas y control de calidad; (5) secuenciación de nueva generación; (6) Análisis de datos. Si bien los pasos (3)-(6) pueden variar significativamente según la tecnología empleada, los pasos iniciales son generalmente comunes a todos ellos. La calidad de la suspensión de células/núcleos preparada determinará el resultado general del experimento. Dependiendo del tipo de tejido, la obtención de suspensiones unicelulares/nucleales de alta calidad puede ser un desafío. Las particularidades de algunos tejidos, como el corazón, el músculo, el cerebro, el pulmón, el intestino y otros, requieren métodos de disrupción tisular y aislamiento de núcleos adaptados a cada tipo de muestra, con el fin de garantizar la producción de núcleos de alta calidad para el análisis molecular 7,8,9,10 . Los métodos de disrupción tisular y los protocolos de disociación pueden ser mecánicos, enzimáticos (p. ej., una mezcla de colagenasas y DNasa) o una combinación de ambos, y se pueden realizar manualmente o mediante instrumentos (p. ej., Qiagen DSC-400, gentleMACS).
Las técnicas unicelulares se han convertido en una herramienta de elección para la investigación biomédica. En neurobiología, la diversidad celular en el cerebro y la complejidad de sus funciones requieren análisis de alta resolución y alto rendimiento para la visualización de poblaciones celulares raras y para evaluar su heterogeneidad 11,12,13,14. La vinculación de la identidad celular y los mecanismos reguladores de genes de las células individuales proporciona información sobre el desarrollo y la fisiología del cerebro. Otro ejemplo son los estudios de la respuesta inmunitaria en el contexto de enfermedades infecciosas, autoinmunes o cancerosas, que se basan en gran medida en análisis de células individuales. La heterogeneidad de los subconjuntos de células inmunitarias y la complejidad de su actividad e interacciones con otros tipos de células requieren una resolución de una sola célula para descifrar los mecanismos subyacentes a la respuesta inmunitaria. Las células inmunitarias se originan en la médula ósea, donde los progenitores hematopoyéticos están compuestos por células que se diferencian gradualmente y adquieren y pierden marcadores de superficie celular a lo largo de un proceso escalonado antes de salir de la médula ósea a su hogar en la periferia. El análisis de una sola célula permite una caracterización minuciosa de las etapas de desarrollo celular. Se puede lograr a través del fenotipado de una sola célula, realizado convencionalmente mediante citometría de flujo multiparamétrica. Sin embargo, se ha demostrado que las firmas transcriptómicas de una sola célula revelan una identificación más precisa de los subtipos de células progenitoras, ya que estas células se distribuyen en grupos que caen entre sí y, por lo tanto, pueden identificarse erróneamente cuando se utiliza un enfoque de marcador de superficie celular gruesa15. Un número creciente de estudios descubren las modificaciones epigenéticas que las células madre hematopoyéticas y progenitoras (HSPC) pueden adquirir a partir de la exposición a diversos agentes, lo que lleva a un impacto significativo en la capacidad de respuesta a largo plazo del sistema inmune 16,17,18,19. Las novedosas tecnologías multiómicas permiten estudiar estos procesos con resolución de una sola célula.
Se han descrito muchos protocolos para el aislamiento de células y núcleos para muestras de cerebro 11,20,21,22 y médula ósea 23,24. Para minimizar el sesgo debido a la variabilidad experimental, es necesario validar protocolos optimizados de preparación de un solo núcleo para la secuenciación transcriptómica y epigenómica conjunta de una sola célula, asegurando así la reproducibilidad de los ensayos multiómicos de una sola célula.
Aquí, se describen dos protocolos robustos para la preparación de núcleos a partir de (1) tejido cerebral fresco congelado y (2) HSPC de médula ósea fresca para el análisis multioma de una sola célula posterior (Figura 1).
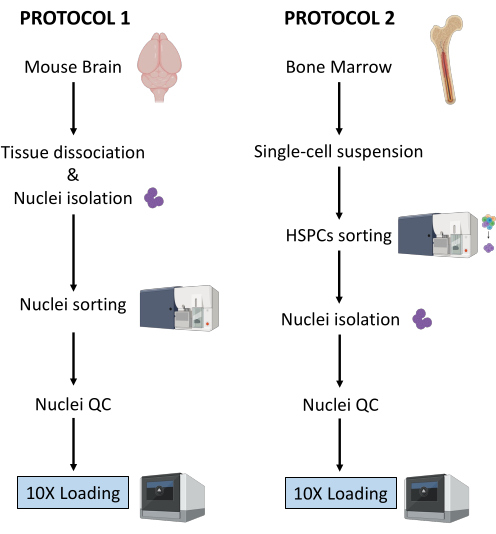
Figura 1: Representación esquemática de los protocolos para el aislamiento de núcleos de tejidos cerebrales y de médula ósea recién congelados. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
Los procedimientos experimentales se llevaron a cabo bajo el estricto cumplimiento normativo de los protocolos aprobados por el Comité de Ética en Experimentación Animal (CETEA). Para el aislamiento de los núcleos cerebrales se utilizaron ratones C57BL/6 de 3 meses de edad. Para el aislamiento de la médula ósea se utilizaron ratones hembra C57BL/6J de 8 semanas de edad con un peso de 18 g.
1. Purificación de núcleos del cerebro de ratón
NOTA: Use guantes de látex o nitrilo en todo momento durante el procedimiento. Se recomienda encarecidamente que dos personas realicen el experimento, que los pasos 1 a 3 (es decir, la preparación de la suspensión de un solo núcleo) sean realizados por una sola persona y que el paso 4 (es decir, la preparación del clasificador) sea realizado en paralelo por otra persona. Dado que el protocolo es muy sensible al tiempo, es fundamental minimizar el tiempo de procesamiento de muestras teniendo el clasificador listo tan pronto como se prepare la suspensión de núcleos individuales.
- Preparación de reactivos y materiales
- Esterilice cuidadosamente las herramientas de disección en autoclave (a 121 °C durante 20 min) y lávelas con etanol al 70% justo antes de su uso. Prepare una placa de Petri por muestra, llena con 2-3 ml de solución salina tamponada con fosfato (DPBS) helada de Dulbecco.
- Enfríe la microcentrífuga a 4 °C, llene un cubo con hielo y coloque el homogeneizador de vidrio sobre hielo.
- Prepare el tampón de lisis de núcleos agregando digitonina para una concentración final de 0.01%, 10 mL por muestra.
- Prepare el tampón de tinción añadiendo inhibidores de la ARNasa al tampón de tinción celular para obtener una concentración final de 0,2 U/μL, 20 ml por muestra.
- Prepare DPBS 0,04% BSA añadiendo inhibidores de la RNasa para una concentración final de 0,2 U/μL, 2 mL por muestra.
- Preparar 1 mL de tampón de núcleos diluidos según el protocolo Multiome25.
- Mantenga todos los reactivos y muestras en hielo.
- Disección de tejidos
- Sacrificar ratones utilizando protocolos aprobados por la institución. En este protocolo, los ratones fueron decapitados después de una sobredosis de ketamina/xilacina.
- Cortar la cabeza del ratón con unas tijeras y extraer el cerebro del cráneo como se describe en Meyerhoff et al.26. Transfiera inmediatamente el cerebro a una placa de Petri preparada con el DPBS 1x helado bajo un microscopio estereoscópico iluminado por diodos emisores de luz (LED).
- Corte el tejido cerebral con un bisturí para separar las áreas cerebrales de interés (p. ej., corteza entorrinal, hipocampo, corteza prefrontal) y transfiera cada región a una placa de Petri separada que contenga 1x DPBS helada. Mantener en hielo.
- Con un bisturí, pica el tejido en trozos de <0,5 cm para facilitar la homogeneización en el siguiente paso.
- Con una micropipeta P1000, transfiera el tejido picado y el DPBS 1x de la placa de Petri a un tubo de 1,5 ml. Asegúrese de usar tubos hechos de plástico de baja unión a proteínas. Deje que las piezas de tejido se separen por gravedad. Retire con cuidado el exceso de 1x DPBS con una micropipeta P1000.
NOTA: Después de este paso, es posible congelar el tejido picado transfiriendo los tubos de baja unión de proteínas a hielo seco y luego almacenándolos a -80 °C hasta proceder con el aislamiento de los núcleos.
- Aislamiento de núcleos
- Llene el vaso con 2 ml de tampón de lisis de núcleos helados con digitonina al 0,01%. Agrega los trozos de pañuelo en la doza.
NOTA: si trabaja con tejido fresco congelado, agregue el tejido congelado picado directamente al tampón de lisis de núcleos 0.01% digitonina; No deje que el tejido se descongele antes. - Homogeneizar usando un homogeneizador de tejido de vidrio 25 veces con el mortero A y luego 25 veces con el mortero B. Transfiera el homogeneizado a un tubo de 15 mL.
- Añadir 2 ml adicionales de tampón de lisis de núcleos helados con digitonina al 0,01% e incubar en hielo durante 5 min. Centrifugar los núcleos a 500 x g durante 5 min a 4 °C.
- Retirar el sobrenadante con una micropipeta y añadir 4 ml de tampón de lisis de núcleos helado con digitonina al 0,01%. Incubar en hielo durante 5 minutos y filtrar a través de un colador de células de 40 μm.
- Centrifugar los núcleos a 500 x g durante 5 min a 4 °C y retirar el sobrenadante con una micropipeta.
- Añadir 4 ml de tampón de tinción para lavar los núcleos y centrifugar a 500 x g durante 5 min a 4 °C. Retirar el sobrenadante con una micropipeta y volver a suspender el gránulo en 4 ml de tampón de tinción.
- Filtrar a través de un colador de células de 40 μm y centrifugar a 500 x g durante 5 min a 4 °C. Vuelva a suspender en 1 mL de PBS con 0.04% de BSA.
- Cuente los núcleos para garantizar la consistencia de la preparación de tejidos/núcleos en diferentes muestras. Se espera obtener recuentos de núcleos similares de las mismas regiones cerebrales:
- Añadir 10 μL de azul de tripán al 0,4% a un tubo vacío de 0,5 mL. Añadir 10 μL de los núcleos y mezclar 5 veces mediante pipeteo.
- Cuente los núcleos utilizando un contador celular automatizado siguiendo las recomendaciones del proveedor. Mantén los núcleos en hielo.
- Prepare los núcleos para la clasificación.
NOTA: Los núcleos extraídos incorporan 7-AAD, y esta tinción se utiliza para su purificación mediante clasificador celular activado por fluorescencia (FACS).- Transfiera 100 μL de núcleos a un tubo FACS para el control sin teñir. Añadir 10 μL de 7-AAD a los núcleos restantes y mantener 5 min a 4 °C.
- Clasifique un mínimo de 0,5 x 106 núcleos por FACS para eliminar dobletes y escombros.
- Llene el vaso con 2 ml de tampón de lisis de núcleos helados con digitonina al 0,01%. Agrega los trozos de pañuelo en la doza.
- Clasificación de núcleos mediante un FACS
NOTA: Si bien la clasificación de núcleos se puede realizar en una amplia variedad de clasificadores celulares, aquí se describe el procedimiento para usar los instrumentos BD FACSAria Fusion o BD FACSAria III. Se recomienda encarecidamente que la calibración y la configuración del clasificador de células se realicen bajo la supervisión o por un usuario experimentado del instrumento. Para reducir el tiempo de procesamiento de la muestra, es fundamental tener el clasificador listo tan pronto como se prepare la suspensión de un solo núcleo.- Calibración del instrumento FACS
- Encienda el clasificador de celdas y la computadora. Una vez que el software esté conectado al instrumento, inicie el procedimiento de puesta en marcha fluídica. Seleccione Citómetro > inicio de Fluidic en el menú principal y siga los cuatro pasos. Haga clic en Listo después de completar cada uno.
- Inserte la boquilla de 70 μm, encienda el chorro y deje que el chorro se estabilice durante 15 minutos. Ajusta la amplitud para obtener la formación de gotas y haz clic en Sweet Spot.
- Coloque el filtro de densidad neutra (N.D) 1.0 y abra la interfaz de configuración y seguimiento del citómetro (CST).
- Control de calidad diario: Diluir las perlas de CST en medio FACS (ver recomendaciones del proveedor) y realizar el control de CST. Una vez completado, reemplace el N.D 1.0 por el N.D 2.0.
- Diluya Accudrops en medio FACS (consulte las recomendaciones del proveedor) y realice el retardo de caída como se describe en los pasos 6 a 10.
- En la plantilla de experimento, seleccione el experimento Accudrop Drop Delay y abra el diseño de clasificación del tubo.
- Dentro de la ventana inferior de la cámara, haga clic en Voltaje y luego en Filtro óptico para habilitar la aplicación de carga en las placas de deflexión y el uso de un filtro óptico específico frente a la cámara. Asegúrese de que el cuadrante del lado derecho indique 100. Si es necesario, ajuste el tornillo láser rojo para optimizar el impacto del láser.
- Ajuste el caudal para alcanzar la velocidad de 1.000 a 3.000 eventos por segundo.
- Haga clic en Ordenar y cancelar. Asegúrese de que el cuadrante izquierdo sea igual a 100 y que el cuadrante derecho sea 0. Si el cuadrante izquierdo está por debajo de 95, realice el retardo automático.
- Haga clic en Voltaje, luego en Ordenar prueba. Controlar la calidad de los flujos laterales depositados en los tubos de recogida. Si es necesario, ajuste la posición de las secuencias laterales moviendo los controles deslizantes.
- Puesta en marcha de un instrumento FACS para la clasificación de núcleos.
- Iniciar la adquisición de los núcleos no teñidos. Estos se utilizan para definir las dispersiones directas y laterales, y el voltaje del detector para el parámetro 7-ADD. Establezca los parámetros de modo que la señal 7-AAD de la muestra no teñida se encuentre dentro de la primera década de la escala logarítmica en el diagrama de puntos.
- Comience a adquirir el tubo de núcleos teñidos con 7-AAD y defina las poblaciones de núcleos mediante el uso de una estrategia de compuerta basada en (1) FSC-A/SCC-A y luego FSC-H/SSC-H para el tamaño y la granularidad, (2) FSC-H/FSC-A para la discriminación de dobletes y (3) SSC-A/7-AAD para núcleos positivos de 7-AAD (ver Figura 2A).
- Asegúrese de que la corriente y la deflexión sean estables.
- En la cámara de flujo lateral, encienda la clasificación de prueba, encienda el voltaje y confirme la clasificación precisa de gotas en un tubo de 1,5 ml instalado en el lado izquierdo.
- En la ventana Diseño de ordenación , seleccione la población de interés, tal como se define en el paso 2 (anterior). En Eventos de destino, seleccione el umbral en Continuo para obtener un mínimo de 0,5 x 106 núcleos por muestra. En Precisión, seleccione Pureza de 4 vías.
- Una vez que esté listo, haga clic en Ordenar y Aceptar para comenzar con la clasificación de núcleos.
- Calibración del instrumento FACS
- Control de calidad y recuento de núcleos purificados
NOTA: Este paso debe realizarse solo durante el experimento piloto para la optimización de los pasos de preparación de muestras, con el objetivo de probar la pureza de los núcleos clasificados que se cargarán en el chip de cromo 10X. Una vez que el protocolo está completamente optimizado, no se recomienda realizar este paso de control de calidad en los experimentos de seguimiento para evitar el desperdicio innecesario de núcleos recolectados que pueden estar disponibles en cantidades bajas.- Control de pureza por citometría de flujo
- Transfiera 10 μL de los núcleos clasificados a un nuevo tubo FACS que contenga 90 μL de DPBS con suero fetal bovino inactivado por calor al 2% (HI-FBS).
- Adquiera y registre datos posteriores a la clasificación para verificar la pureza y viabilidad de la clasificación. Asegúrese de que al menos el 98% de los núcleos aparezcan en la puerta de interés, como se define en 4.2 (véase la figura 2B).
- Recuento de los núcleos purificados
- Centrifugar los núcleos clasificados durante 5 min a 500 x g y a 4 °C y retirar cuidadosamente el sobrenadante con una micropipeta. Vuelva a suspender en 100 μL de tampón de núcleos diluidos.
- Añadir 10 μL de azul de tripán al 0,4% a un tubo vacío de 0,5 mL. Añadir 10 μL de los núcleos clasificados y mezclar 5 veces mediante pipeteo.
- Cuente los núcleos utilizando un contador celular automatizado siguiendo las recomendaciones del proveedor. Ajuste la concentración de núcleos a 3,5 x 106/mL, es decir, 16.000 núcleos por 5 μL.
- Control de calidad de núcleos purificados por microscopía
NOTA: Este paso debe realizarse solo durante el experimento piloto para la optimización de los pasos de preparación de muestras para probar la calidad de los núcleos que se van a cargar en el chip de cromo 10X. Una vez que el protocolo está completamente optimizado, no se recomienda realizar este paso de control de calidad en los experimentos de seguimiento para evitar el desperdicio innecesario de núcleos recolectados que pueden estar disponibles en cantidades bajas.- Asegúrese de que los portaobjetos del microscopio y las cubiertas estén limpios y libres de polvo. Si es necesario, lave y enjuague los cubreobjetos con etanol absoluto y séquelos con toallitas sin pelusa.
- Distribuir 25 μL de poli-l-lisina en los pocillos portaobjetos que se van a utilizar e incubar durante 10 min a temperatura ambiente (RT), protegidos del polvo.
- Retirar el exceso de poli-l-lisina y añadir 10 μL de la suspensión de núcleos purificados. Incubar durante 5 min a RT, protegido del polvo.
- Agregue una gota de medio de montaje a cada pocillo, evitando burbujas.
- Coloque un cubreobjetos encima de los pocillos sembrados. Cubra con toallitas de papel y presione el cubreobjetos firmemente para eliminar el exceso de medio de montaje. Tenga cuidado de no mover el cubreobjetos y no limpie el exceso de medio de montaje.
- Tome varias imágenes con un microscopio invertido con luz de campo claro y un aumento mínimo de 40x.
- Control de pureza por citometría de flujo
- Realice un ensayo multioma.
- Proceda inmediatamente a la Guía del usuario de Chromium Next GEM Single Cell Multiome ATAC + Gene Expression (CG000338 - Rev F)25.
2. Purificación de núcleos de células madre y progenitoras hematopoyéticas de médula ósea de ratón (HSPC)
NOTA Este protocolo describe la purificación de núcleos de tres subconjuntos de HSPC de médula ósea: células madre hematopoyéticas (HSC) linaje-c-Kit+Sca-1+, linaje-c-Kit+Sca-1-CD34+FcγR- progenitores mieloides comunes (CMP) y linaje- c-Kit+Sca-1-CD34+FcγR+ progenitores de granulocitos-monocitos (GMP). Use guantes de látex o nitrilo en todo momento durante el procedimiento. Este protocolo es una adaptación del 10X Genomics Demonstrated Protocol - Nuclei Isolation for Single Cell Multiome ATAC + GEX sequencing (CG000365 - Rev C)27. Se han introducido modificaciones en el protocolo original para maximizar la recuperación de núcleos. Se recomienda encarecidamente que dos personas realicen el experimento, para tener pasos 1. a 3. (es decir, la preparación de la solución unicelular) realizada por una persona, y el paso 4 (es decir, la preparación del clasificador) realizado en paralelo por otra persona. Dado que el protocolo es muy sensible al tiempo, es fundamental minimizar el tiempo de procesamiento de muestras teniendo el clasificador listo tan pronto como se prepare la suspensión de una sola célula.
- Preparación de reactivos y materiales
- Llena un balde con hielo.
- Preparar el tampón FACS: DPBS con una solución de HI-FBS al 2% (aproximadamente 500 ml para 6 muestras) y filtrar a través de un filtro de 0,2 μm.
- Preparar el medio de recolección: DPBS con solución de HI-FBS al 10% (500 μL por muestra) y filtrar a través de un filtro de 0,2 μm.
- Aislamiento de células de la médula ósea
- Sacrificar ratones utilizando protocolos aprobados por la institución. En este experimento, los ratones fueron sacrificados por dislocación cervical después de una sobredosis de ketamina / xilacina.
- Rocíe el abdomen y las patas traseras de los ratones con etanol al 70%.
- Use pinzas y tijeras estériles para hacer una pequeña incisión en el centro de la parte inferior del abdomen y abrir el peritoneo desde la base de las patas traseras hasta el diafragma (Figura complementaria 1).
- Haga un corte adicional para cada pata trasera perpendicular al peritoneo abierto, luego agarre cada lado de uno de estos cortes adicionales y sepárelo para despegar la piel de ambas patas traseras más allá de la articulación del tobillo para exponer los músculos de ambas patas traseras (Figura suplementaria 1A).
- Alinee las tijeras a lo largo de la columna vertebral en la articulación de la cadera de una pata trasera para cortar la pierna sin cortar el fémur (Figura suplementaria 1B, C). Repite lo mismo para la otra pierna.
- Para aislar el fémur, corte la mayor parte del tejido muscular, luego sostenga el fémur y la tibia en cada mano con las yemas de los dedos en la articulación (Figura suplementaria 1D, E). Dobla suavemente la pierna contra la curva natural para dislocar la tibia del fémur (Figura suplementaria 1E) y luego corta con cuidado el tejido conectivo con unas tijeras para separar el fémur y la tibia.
- Use las tijeras con ligeros movimientos de torsión para dislocar el trozo de columna vertebral del extremo superior del fémur (Figura suplementaria 1E).
- Limpie el fémur aislado con papel de seda para eliminar el músculo y el tejido conectivo restantes.
- Manténgalo frío en hielo en una placa de 12 pocillos bien llena con 2 ml de DMEM (1x) + GlutaMAX-I.
- Una vez que se hayan recogido todos los fémures, asegúrese de que los tejidos musculares y fibrosos se eliminen por completo del hueso. No corte el hueso para (a) mantener la médula dentro estéril y (b) evitar la pérdida de células en el pocillo. Siga los siguientes pasos para limpiar las células de dos fémures de un ratón, adaptados de Haag y Murthy28.
- Prepare un tubo de 1,5 ml y otro de 0,5 ml. Agregue 150 μL del tampón FACS al tubo de 1,5 mL, luego haga un orificio en la parte inferior del tubo de 0,5 mL con una aguja de 18 G y coloque el tubo de 0,5 mL en el tubo de 1,5 mL.
- Abra la parte distal de cada fémur con unas tijeras quirúrgicas de ratón (Figura suplementaria 1F): Bloquee la epífisis distal entre las cuchillas y aplique una presión suave mientras gira las tijeras para separar suavemente la epífisis distal sin cortar el hueso con dureza. Si tiene éxito, 4 protuberancias deben ser visibles en el extremo de la fisis ahora expuesto (Figura suplementaria 1G).
- Coloque los dos fémures con el extremo abierto hacia abajo en el tubo preparado de 0,5 ml colocado dentro de un tubo de 1,5 ml que contenga tampón FACS (Figura suplementaria 1H).
- Coloque un colador de células de 70 μm en un tubo de 50 ml y humedezca previamente el colador con 2 ml del tampón FACS.
- Para lavar la médula ósea, centrifugue los tubos (con las tapas abiertas) a 12.000 x g hasta que la centrífuga alcance el valor de 12.000 x g , luego detenga inmediatamente la centrífuga.
- Verificar que las células de la médula ósea estén peletizadas en el tubo de 1,5 mL y que los fémures estén blancos (antes del lavado celular, son rojos) (Figura complementaria 1I). Deseche los tubos de 0,5 ml con los 2 fémures.
- Deseche el sobrenadante de 150 μL con una pipeta.
- Vuelva a suspender el gránulo con una micropipeta en 1 ml de tampón de lisado de amonio-cloruro-potasio (ACK) durante 1-2 minutos a RT para lisar los glóbulos rojos. Evite los tiempos de incubación más largos, ya que pueden resultar en una menor viabilidad de las células nucleadas.
- Transfiera al tubo de 50 ml a través del filtro de células de 70 μm previamente humedecido.
- Añadir 10 ml de tampón FACS para diluir el tampón de lisado ACK y detener así la lisis.
- Centrifugar a 400 x g durante 5 min a 4 °C. Vuelva a suspender en tampón FACS de 10 ml resuspendiendo primero en 1 ml y luego rellenando con 9 ml.
- Prepare las celdas para el conteo como se describe en 1.3.8.
- Cuente las celdas utilizando un contador de celdas automatizado siguiendo las recomendaciones del proveedor. Se espera que recoja aproximadamente 40 millones de células de 2 fémures.
- Tinción de HSPC de médula ósea
- Centrifugar las células a 400 x g durante 5 min a 4 °C y resuspender el pellet con una micropipeta en tampón FACS hasta una concentración final de 1 x 107 células/mL.
- Con una micropipeta P1000, transfiera la suspensión a un tubo FACS, filtrando a través de una tapa de filtro de células de 35 μm.
- Prepare muestras de tubo de ensayo de tinción única para cada anticuerpo enumerado en la Tabla 1 para establecer compensaciones de fluorocromos en el clasificador de células:
- Prepare un tubo de FACS por anticuerpo y llénelo con 200 μL de PBS.
- Agregue 15 μL de perlas de compensación de fluorocromo en cada tubo FACS de anticuerpo conjugado con fluorocromo. En los tubos FACS para células no teñidas y para células teñidas individuales vivas/muertas, agregue 500.000 células en lugar de perlas.
- Añadir 1 μL de cada anticuerpo conjugado con fluorocromo (ver Tabla 1) en su tubo FACS correspondiente. Agregue 0,5 μL de tinción viva/muerta en el tubo FACS de tinción única viva/muerta.
- Mantener en hielo durante 15 minutos protegido de la luz.
- Prepare las mezclas 1 y 2 como se indica en la Tabla 2.
NOTA: Los volúmenes de anticuerpos indicados en la Tabla 2 son válidos para los anticuerpos a los que se hace referencia en la Tabla de Materiales. Deben optimizarse para cualquier nueva referencia de anticuerpos o para un lote diferente de la misma referencia de anticuerpos. - Añadir 300 μL de Mix 1 en el tubo de muestra, volver a suspender y mantener durante 15 minutos sobre hielo protegido de la luz.
- Añadir 300 μL de Mix 2 en el tubo de muestra, volver a suspender y mantener durante 20 minutos sobre hielo protegido de la luz.
- Agregue 3 ml del tampón FACS a los tubos de tinción simple y a los tubos de muestra teñidos con mezcla. Girar a 400 x g durante 5 min a 4°C.
- Deseche cuidadosamente el sobrenadante con una micropipeta y vuelva a suspender el gránulo en 500 μL del tampón FACS.
- Prepare un tubo de 1,5 ml precargado con 500 μl de medio de recolección.
NOTA: La mezcla 1 se prepara en DPBS ya que contiene la tinción Vivo/Muerto significativamente afectada por HI-FBS. Una vez que las células son teñidas por Vivo/Muerto, se agrega Mix 2, que contiene los anticuerpos conjugados con fluorocromo resuspendidos en tampón FACS que contiene HI-FBS. La única excepción es el anticuerpo anti-CD16/32 que se incluye en la mezcla de anticuerpos 1 para servir como bloqueador del receptor Fc que evita la unión no específica de los otros anticuerpos agregados en el siguiente paso.
- Clasificación de celdas mediante un FACS
NOTA: Si bien la clasificación de células se puede realizar en una amplia variedad de clasificadores de células, aquí se describe el procedimiento para usar los instrumentos BD FACSAria Fusion o BD FACSAria III. Se recomienda encarecidamente que la calibración y la configuración del clasificador de células se realicen bajo supervisión o por un usuario experimentado del instrumento.- Calibración del instrumento FACS: Consulte el paso 4.1 del Protocolo 1.
- Configuración del instrumento FACS para la clasificación de células:
- Iniciar la adquisición de las células no teñidas. Estos se utilizan para definir las dispersiones directas y laterales y el voltaje del detector para cada fluoróforo. Establezca los parámetros de modo que la señal fluorescente de cada fluoróforo se encuentre dentro de la primera década de la escala logarítmica en el diagrama de puntos.
- Adquiera controles de un solo color para configurar las compensaciones manualmente (la mediana de las poblaciones positivas y negativas debe estar alineada) o utilice el software de cálculo automático (mediciones de pendiente). Asegúrese de que los controles de compensación coincidan con los fluorocromos experimentales y la configuración del detector. Registre 10.000 eventos para células y 5.000 eventos para cuentas.
- Utilice el tubo de muestra (es decir, células con tinción múltiple) para definir las poblaciones celulares de interés mediante la estrategia de compuerta que se muestra en la Figura 3A. Siga los pasos 4 a 6 (a continuación).
- Para identificar las tres HSPC de médula ósea de interés (HSC, CMP y GMP), comience la compuerta utilizando el tamaño (FSC-A) y la granularidad (SSC-A) para controlar los leucocitos, luego FSC-H/FSC-A para discriminar dobletes.
- Basado en el marcador SSC-A/células muertas, las células vivas de la puerta. Utilice Lineage/c-Kit para seleccionar células que son negativas para el linaje y que expresan niveles intermedios a altos de c-Kit. A través de c-Kit/Sca-1, compuerta en las HSC de linaje-c-Kit+ Sca-1+ (LKS+), una de las tres poblaciones de interés.
- Entre los progenitores mieloides (linaje-c-Kit+Sca-1-), se utiliza FcγR/CD34 para excluir los progenitores de megacariocitos y eritroides (MEP) CD34-FcγR-, al tiempo que se incluyen CD34+ FcγR-CMP, así como CD34+FcγR+ GMP en las poblaciones celulares que se van a clasificar.
- Asegúrese de que la corriente y la deflexión sean estables.
- En la cámara de flujo lateral, encienda la clasificación de prueba, encienda el voltaje y confirme la clasificación precisa de gotas en un tubo de 1,5 ml instalado en el lado izquierdo.
- En la ventana Diseño de clasificación , seleccione la(s) población(es) de interés (es decir, "LKS+" y "Progenitores mieloides CD34+ " que se muestran en este ejemplo). En Dispositivo, seleccione 2 tubos. En Precisión, seleccione Pureza. En Eventos de destino, seleccione Continuo para ordenar entre 160 000 y 200 000 progenitores mieloides LKS+ y CD34+ .
- Agregue 500 μL de tampón FACS a la suspensión celular y transfiera 1 mL de la muestra filtrándola a un nuevo tubo FACS de 35 μm tapado por filtro de células para garantizar que todas las células estén en una sola suspensión justo antes de la adquisición. Esto elimina los cúmulos celulares que podrían obstruir el instrumento.
- Una vez listo, haga clic en Ordenar y Aceptar para comenzar a ordenar. Ajuste el caudal para mantener la velocidad por debajo de los 10.000 eventos por segundo.
NOTA: La proporción esperada de progenitores mieloides LKS+ a CD34+ es de 1:3 para un ratón hembra C57BL/6J adulto (8-12 semanas de edad) en estado estacionario. Por lo general, los números de celda ordenados de destino se alcanzan dentro de los 30 minutos posteriores a la clasificación.
- Control de calidad y recuento de celdas clasificadas
NOTA: Este paso debe realizarse solo durante el experimento piloto para la optimización de los pasos de preparación de la muestra, con el objetivo de probar la pureza de las células clasificadas que se utilizarán para el aislamiento de núcleos. Una vez que el protocolo está completamente optimizado, no se recomienda realizar este paso de control de calidad en los experimentos de seguimiento para evitar el desperdicio innecesario de material de partida que puede estar disponible en cantidades bajas para el aislamiento de núcleos.- Control de pureza por citometría de flujo
- Transfiera 10 μL de las células clasificadas a un nuevo tubo FACS que contenga 90 μL de tampón FACS.
- Adquiera y registre datos posteriores a la clasificación para verificar la pureza y viabilidad de la clasificación. Asegúrese de que al menos el 95% de las celdas aparezcan en la puerta de interés, como se define en 3 a 6 y se ilustra en la Figura 3B.
- Control de pureza por citometría de flujo
- Aislamiento de núcleos de HSPC de médula ósea clasificadas
- Utilice el protocolo "Low Cell Input Nuclei Isolation" del Apéndice del Protocolo 10X Genomics Demonstrated - Nuclei Isolation for Single Cell Multiome ATAC + GEX sequencing (CG000365 - Rev C)27, con las siguientes modificaciones realizadas para optimizar la recuperación de núcleos:
- Tiempo de lisis: ejecute un experimento piloto para este protocolo con el fin de identificar el mejor tiempo de lisis para el aislamiento de núcleos. Asegúrese de lograr una lisis celular completa mientras mantiene los núcleos intactos.
NOTA: El paso f del protocolo27 de 10X Genomics mencionado anteriormente indica "incubar [en tampón de lisis] durante 3-5 minutos en hielo". Durante el experimento piloto, pruebe al menos durante 3 minutos, 4 minutos y 5 minutos y evalúe la cantidad de núcleos recuperados mediante recuento y calidad mediante citometría de flujo e imágenes de microscopía para elegir la duración óptima de la lisis (consulte la descripción de estas comprobaciones de control de calidad a continuación). Para ahorrar reactivos, reemplace el tampón de núcleos diluidos con PBS 0.04% BSA en el experimento piloto. Para las HSPC de médula ósea, se identificó 3 min como la duración óptima de la lisis. - Centrifugaciones celulares: Para todas las centrifugaciones en suspensión celular, centrifugar a 300 x g durante 7 min (en lugar de los 5 min en CG000365 - Rev C)27 a 4 °C.
- Centrifugaciones de núcleos: Realice todas las centrifugaciones en suspensión de núcleos a 500 x g durante 5 min según CG000365 - Rev C27.
- Recolección de núcleos: En el paso b, después de resuspender 50 μL de PBS 0,04% BSA y transferirlo a un tubo de 0,2 mL, agregar 50 μL de PBS 0.04% BSA al tubo original y pipetear la mezcla para recolectar las células sobrantes. Transfiera al tubo de 0,2 ml para alcanzar un volumen total de 100 μl.
- A partir de ahora, el volumen total será de 100 μL en lugar de los 50 μL del protocolo. Ajuste los pasos posteriores en consecuencia (por ejemplo, para el paso d, elimine 90 μL en lugar de 45 μL; para el paso e, agregue 90 μL de tampón de lisis en lugar de 45 μL).
- Para el paso m, vuelva a suspender el gránulo de núcleos en 12 μL de tampón de núcleos diluido en lugar de 7 μL.
- Cuenta los núcleos aislados. En un tubo vacío de 0,5 ml, agregue 10 μl de azul de tripán al 0,4 % y 8 μl de PBS al 0,04 % de BSA.
- Añadir 2 μl de núcleos al tubo y contar los núcleos como se describe en el punto 1.3.8. Utilice un contador de celdas automatizado siguiendo las recomendaciones del proveedor.
- Tiempo de lisis: ejecute un experimento piloto para este protocolo con el fin de identificar el mejor tiempo de lisis para el aislamiento de núcleos. Asegúrese de lograr una lisis celular completa mientras mantiene los núcleos intactos.
- Utilice el protocolo "Low Cell Input Nuclei Isolation" del Apéndice del Protocolo 10X Genomics Demonstrated - Nuclei Isolation for Single Cell Multiome ATAC + GEX sequencing (CG000365 - Rev C)27, con las siguientes modificaciones realizadas para optimizar la recuperación de núcleos:
- Control de pureza por citometría de flujo
NOTA: Este paso debe realizarse solo durante el experimento piloto para la optimización de los pasos de preparación de muestras para probar la pureza de los núcleos que se van a cargar en el chip de cromo 10X. Una vez que el protocolo está completamente optimizado, no se recomienda realizar este paso de control de calidad en los experimentos de seguimiento para evitar el desperdicio innecesario de núcleos recolectados que pueden estar disponibles en cantidades bajas.- Después de completar el aislamiento de los núcleos, transfiera 6 μL de resuspensión de núcleos en un nuevo tubo FACS precargado con 150 μL de tampón FACS. Añadir 3 μL de 7-AAD e incubar durante 5 min en hielo.
- Adquiera y registre datos posteriores a la clasificación para verificar la pureza y viabilidad de la clasificación. Asegúrese de que al menos el 95% de los núcleos aparezcan en la puerta de interés, tal como se define en el paso 4.2 del Protocolo 1 (véase la Figura 4).
- Control de calidad de núcleos purificados por microscopía:
NOTA: Este paso debe realizarse solo durante el experimento piloto para la optimización de los pasos de preparación de muestras para probar la calidad de los núcleos que se van a cargar en el chip 10X Chromium. Una vez que el protocolo está completamente optimizado, no se recomienda realizar este paso de control de calidad en los experimentos de seguimiento para evitar el desperdicio innecesario de núcleos recolectados que pueden estar disponibles en cantidades bajas.- Proceda como se describe en el paso 1.5.3.
- Realizar un ensayo multioma
- Proceda inmediatamente a la Guía del usuario de Chromium Next GEM Single Cell Multiome ATAC + Gene Expression (CG000338 - Rev F)25.
Resultados
Los dos protocolos descritos anteriormente detallan el aislamiento de núcleos a partir de dos tipos diferentes de tejido. Las diferencias y similitudes entre los dos protocolos se representan esquemáticamente en la Figura 1.
Purificación de núcleos del cerebro de ratón
En el protocolo aquí descrito, proponemos un método suave para la preparación de núcleos a partir de muestras de cerebro. Comienza con una disociación mecánica del tejido cerebral en un tampón de lisis, seguido de pasos de lavado y filtrado por colador que eliminan el tejido restante de la suspensión. La posterior eliminación de escombros, células no lisadas y partículas pequeñas se logra mediante FACS para garantizar que solo se carguen núcleos purificados para el protocolo Multiome aguas abajo. La Figura 2 muestra el perfil de los núcleos antes y después de la clasificación. Después del filtrado y antes de la clasificación de los núcleos, la muestra contiene una gran cantidad de residuos, con más del 99% de los "singletes" positivos para la tinción nuclear (7-AAD), lo que indica una lisis celular óptima (Figura 2A). Los núcleos se ordenan en función de la puerta positiva 7-AAD. Se adquiere una fracción del material clasificado para verificar la pureza de los núcleos preparados. La figura 2B muestra el perfil de los núcleos cerebrales después de la clasificación. La clasificación de los núcleos permitió un aumento en la pureza de los núcleos del 36% inicial (Figura 2A) a casi el 100% (Figura 2B).
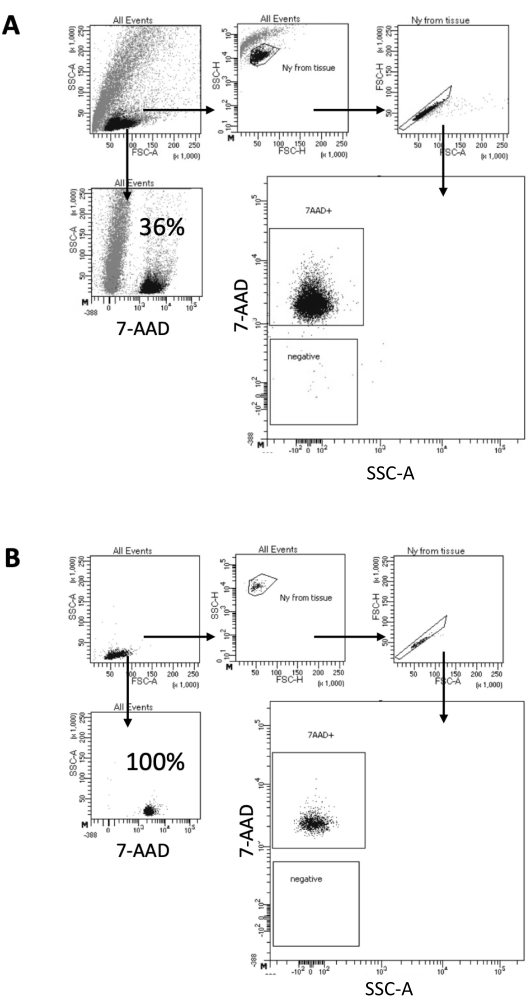
Figura 2: Estrategia de compuerta para la clasificación de núcleos y prueba de pureza después de la clasificación. Los núcleos se tiñeron con 7-AAD y se adquirieron mediante el clasificador celular. (A) Los núcleos se comprimen primero en función de su tamaño y granularidad (FSC-A y SSC-A, respectivamente). A continuación, se seleccionan las partículas individuales en función de sus propiedades FSC-A/FSC-H y la tinción con 7-AAD. (B) Después de la clasificación celular, se prueba la pureza de una fracción de los núcleos del tubo de recolección utilizando la misma estrategia de compuerta que en A. Haga clic aquí para ver una versión más grande de esta figura.
Purificación de progenitores de células madre hematopoyéticas (HSPC) de médula ósea de ratón
Tras el aislamiento de la médula ósea, se clasifican hasta 2 x 105 HSPC por FACS, de acuerdo con la estrategia de compuerta que se muestra en la Figura 3A. Se evalúa la eficacia de clasificación y la pureza de la muestra (Figura 3B).
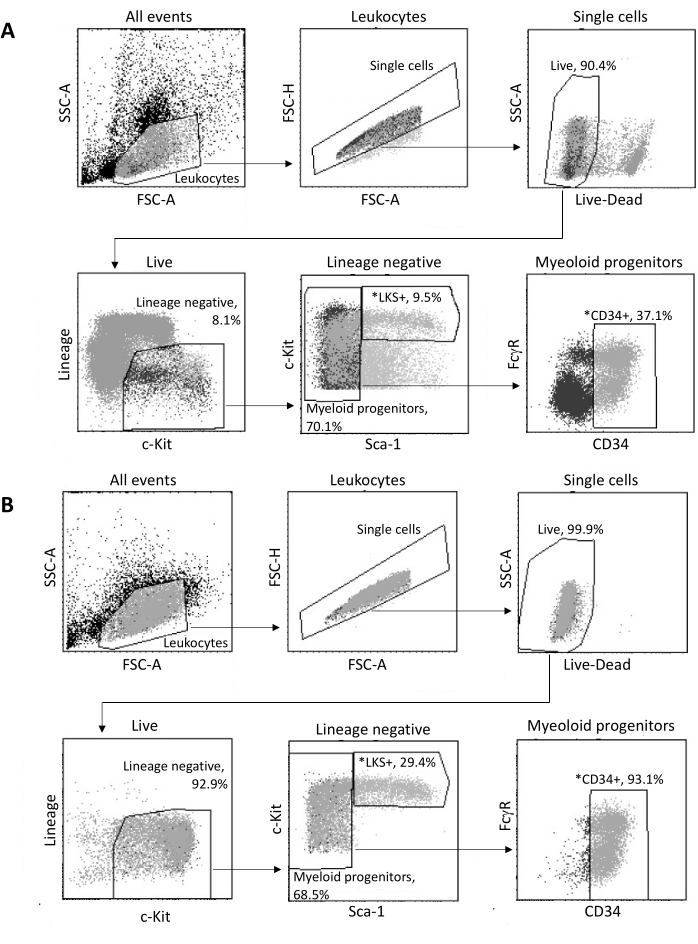
Figura 3: Estrategia de activación para la clasificación de HSPC de médula ósea. (A) Una estrategia representativa de activación de FACS para clasificar células madre hematopoyéticas LKS+ viables y progenitores mieloides CD34+ para el aislamiento de núcleos. (B) Gráficos representativos de FACS utilizados para la verificación de la pureza de la población celular clasificada. Se muestran las proporciones de diferentes subconjuntos celulares con respecto a la población parental. *Las dos poblaciones clasificadas. Haga clic aquí para ver una versión más grande de esta figura.
El protocolo "Low Cell Input Nuclei Isolation" permite el aislamiento de núcleos de muestras con un máximo de 105 células. Incluye un bajo número de pasos de centrifugación, minimizando así la pérdida de células/núcleos. Hemos ajustado el volumen de tampones de lisis y lavado proporcionalmente a la entrada de la célula y hemos aumentado el tiempo de centrifugación para una recuperación máxima de los núcleos. Hemos realizado un experimento piloto para evaluar la cantidad de núcleos recuperados mediante recuento y su calidad mediante citometría de flujo e imágenes de microscopía. La Figura 4 muestra la muestra de HSPCs después de la lisis celular. Este protocolo generó núcleos de alta calidad, como se observa en la Figura 5A, sin ningún residuo que pudiera afectar al protocolo multioma aguas abajo.
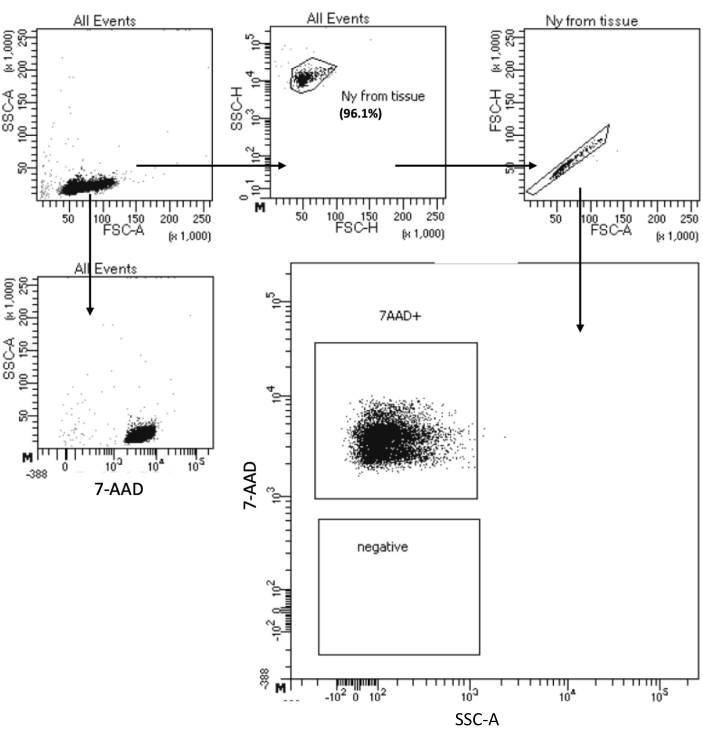
Figura 4: Clasificación de la prueba de pureza de núcleos HSPC aislados de médula ósea. Los núcleos se tiñeron con 7-AAD y se adquirieron mediante el clasificador celular. Los núcleos se comprimieron primero en función de su tamaño y granularidad (FSC-A y SSC-A, respectivamente) para evaluar la pureza de la muestra. La proporción de núcleos se indica con respecto a la población parental. Haga clic aquí para ver una versión más grande de esta figura.
| Número de tubo | Nombre del tubo | Entidad manchada | Cantidad de entidad teñida | Anticuerpo/colorante (μL) | Búfer de recolección (μL) |
| 1 | Sin manchar | Células | 5,00,000 | N/A | 200 |
| 2 | VIVO/MUERTO Tinción de células muertas Aqua reparable | Células | 5,00,000 | 0.5 | |
| 3 | APC/Cianina 7 anti-ratón CD16/32 (FcγR) | OneComp eBeads | 15 μL | 1 | |
| 4 | Cóctel de linaje anti-ratón Pacific Blue | OneComp eBeads | 15 μL | 1 | |
| 5 | PE anti-ratón Ly-6A/E (Sca-1) | OneComp eBeads | 15 μL | 1 | |
| 6 | APC anti-ratón CD117 (c-Kit) | OneComp eBeads | 15 μL | 1 | |
| 7 | FITC anti-ratón CD34 | OneComp eBeads | 15 μL | 1 |
Tabla 1: Controles de tinción única para los ajustes de compensación en el citómetro de flujo. Se indican los controles de tinción única requeridos, el número de células o perlas que se van a teñir y las cantidades de anticuerpos.
| Mezcla maestra | Reactivo | Dilución final | Anticuerpo/colorante (μL) | Tipo de búfer | Tampón (μL) |
| Mezcla 1 | APC/Cianina 7 anti-ratón CD16/32 (FcγR) | 1/500 | 1.2 | DPBS | 300 |
| VIVO/MUERTO Tinción de células muertas Aqua reparable | 1/250 | 2.4 | |||
| Mezcla 2 | Cóctel de linaje anti-ratón Pacific Blue | 1/20 | 30 | Búfer FACS | 300 |
| PE anti-ratón Ly-6A/E (Sca-1) | 1/200 | 3 | |||
| APC anti-ratón CD117 (c-Kit) | 1/200 | 3 | |||
| FITC anti-ratón CD34 | 1/50 | 12 | |||
| Volumen total de tinción | 600 |
Tabla 2: Composición de la mezcla de tinción para HSPC de médula ósea. Se muestran los volúmenes de reactivos necesarios para la tinción de una muestra que contiene 40 millones de células. Para teñir un mayor número de muestras, multiplique el volumen indicado por el número requerido de muestras y agregue la mitad de un volumen de muestra adicional para garantizar un volumen suficiente de la mezcla maestra.
Figura complementaria 1: Protocolo de aislamiento de células de médula ósea. (A) Abrir el peritoneo. Las líneas punteadas blancas indican la línea a lo largo de la cual se va a cortar. (B) Después de pelar la piel de la pata trasera, alinee las tijeras a lo largo de la columna vertebral en la articulación de la cadera para cortar la pierna sin cortar el fémur. (C) La apariencia de la pierna separada del cuerpo antes de la extirpación del músculo. (D) La apariencia de la pierna después de la extirpación del músculo. (E) El procedimiento para separar el fémur en la articulación de la rodilla, luego en la articulación de la cadera, teniendo cuidado de no abrir el fémur. Las flechas curvas blancas muestran el movimiento requerido. Las flechas punteadas blancas indican el área que se debe separar suavemente con unas tijeras para pellizcar. (F) Procedimiento para abrir la parte distal del fémur (es decir, la parte previamente unida a la tibia en la articulación de la rodilla) agarrando firmemente el cartílago y la epífisis distal con unas tijeras y volteándola hacia atrás para exponer la médula ósea. (G) Cuatro protuberancias, indicadas con flechas negras, deben ser visibles en el extremo de la fisis expuesta. (H) La aparición de un fémur con el extremo abierto hacia abajo en el tubo preparado de 0,5 ml colocado dentro de un tubo de 1,5 ml que contiene 150 μl de tampón FACS. (I) La apariencia de las células de médula ósea granuladas y el fémur ahora blanco después de una centrifugación rápida a 12.000 x g. Haga clic aquí para descargar este archivo.
Discusión
La preparación de suspensiones celulares o de núcleos de alta calidad es de crucial importancia para el éxito de los análisis RNA-Seq y multiómicos unicelulares o unicelulares 29,30,31. Aquí, hemos descrito protocolos para la preparación de muestras y el aislamiento de núcleos para ensayos multioma de dos tipos de tejido: cerebro y médula ósea.
El protocolo cerebral descrito en este trabajo permite la recuperación de núcleos de alta calidad a partir de tejido cerebral recién congelado. Incluye los siguientes pasos: disrupción del tejido congelado, aislamiento de núcleos, purificación de núcleos y control de calidad del material preparado. El tejido cerebral está compuesto por muchos tipos de células diferentes, y el procedimiento de disociación de tejidos y aislamiento de núcleos debe preservar las proporciones de poblaciones celulares presentes en el tejido inicial. Aquí, la composición del tampón de lisis y el tiempo de incubación se optimizaron para permitir una lisis completa y suave de todas las poblaciones celulares que componen el tejido.
El protocolo de HSPCs de médula ósea es algo diferente, ya que requiere un paso adicional al comienzo del experimento para aislar la población celular de interés de una suspensión celular heterogénea. Después de la recolección de tejido fresco, se lisan los glóbulos rojos y la muestra se enriquece para el subconjunto de células de interés. Se lisan las células objetivo, se aíslan los núcleos y se controla la calidad del material preparado.
10X Genomics proporciona varios protocolos validados para el aislamiento de núcleos en numerosos tejidos diferentes32,33. La empresa también comercializa un kit de aislamiento de núcleos con una línea sencilla para aislar núcleos de tejidos validados34. Sin embargo, estos protocolos necesitan una optimización adicional para adaptar las particularidades de ciertas muestras. Un ejemplo son las muestras que requieren trabajar con una entrada de celda baja. Para estas muestras, los pasos más desafiantes son las centrifugaciones, que deben ser lo suficientemente estrictas para limpiar la muestra y lo suficientemente suaves para evitar la pérdida de células/núcleos. Con el protocolo aquí descrito, hemos adaptado el Protocolo Demostrado de Genómica 10X - Aislamiento de Núcleos para Secuenciación de Célula Única Multioma ATAC + GEX (CG000365 - Rev C)27 para encontrar un buen equilibrio entre estos dos requisitos. Como se demuestra en el ejemplo de la preparación de núcleos a partir de HSPC clasificados, hemos mejorado la recuperación de núcleos sin afectar a la calidad de la muestra.
Un desafío adicional es el paso de lisis de las células purificadas para el aislamiento de núcleos. Las condiciones de lisis más duras y los tiempos de incubación más largos pueden dañar los núcleos y, por lo tanto, afectar la calidad de los datos de secuenciación. La Figura 5 muestra imágenes representativas de núcleos de muestras de médula ósea en diferentes tiempos de incubación con tampón de lisis e ilustra cuán diferente podría ser el estado de los núcleos dependiendo de la lisis celular. En el ejemplo de las HSPC, hemos identificado la lisis de 3 minutos como la condición que da lugar a la mayor proporción de núcleos intactos de aspecto saludable y la menor proporción de núcleos dañados. Los tiempos de incubación de lisis deben optimizarse para cada nuevo tipo de muestra.
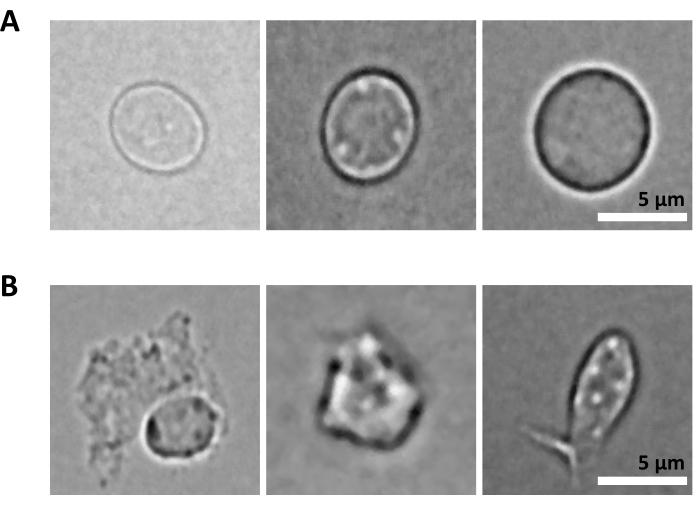
Figura 5: Control de calidad de los núcleos por microscopía. Se muestran imágenes representativas de campo claro de núcleos aislados de la médula ósea de ratón con (A) núcleos intactos y (B) dañados. Barra de escala 5 μm. Las imágenes se tomaron con un microscopio invertido utilizando un objetivo ELWD NA 0.60 de 40x y un zoom digital de 1.5x. Haga clic aquí para ver una versión más grande de esta figura.
Ambos protocolos detallados en este trabajo se basan en la purificación de células o núcleos específicos mediante instrumentos FACS de alto rendimiento. Este paso es de crucial importancia para los protocolos de preparación de una sola célula/núcleos en los que se van a aislar subconjuntos raros de células a partir de suspensiones heterogéneas. En estos, como en el ejemplo que se muestra aquí para la clasificación de HSPC, es posible que se requiera un panel de citometría de flujo de alta dimensión para habilitar la "compuerta" en la población celular de interés. La clasificación es extremadamente rápida y precisa, lo que lleva a una pureza superior al 95% de los subconjuntos de celdas clasificados. Este enfoque expone la suspensión celular a una presión de hasta 70 psi y, por lo tanto, puede ser limitante para la clasificación de células frágiles (por ejemplo, células dendríticas, neutrófilos), ya que puede causar la ruptura de su membrana celular. En estos casos, se deben seleccionar soluciones alternativas para la purificación celular, incluida la clasificación magnética, la aplicación de instrumentos de nueva generación (por ejemplo, CellenOne, Cellenion; MACSQuant Tyto, Miltenyi)35,36, o sistemas basados en gotas (p. ej., ODIN, Sensific)37. Sin embargo, la lenta velocidad de clasificación de estas tecnologías, con una clasificación celular que dura horas en lugar de minutos, es un fuerte factor limitante para la aplicación de estos enfoques en la preparación de células viables para Multiome y otras aplicaciones de una sola célula basadas en el análisis de grandes números de células.
Para la purificación de núcleos aislados del tejido, FACS es el método de elección debido a su rendimiento y a la pureza del material aislado. Los núcleos no son sensibles a la presión, y los aislados de tejido filtrados se pueden purificar fácilmente a través del clasificador de células. Si el laboratorio no está equipado con un instrumento FACS, existen otras alternativas, algo menos eficientes pero suficientemente buenas. Algunos ejemplos son la ultracentrifugación o el uso de pequeños equipos como MARS (Applied Cell) que separa las partículas en función de su diferencia de tamaño, mediante ondas acústicas; arandela laminar CURIOX que utiliza las propiedades hidrofóbicas de las suspensiones celulares/nucleos; o LEVITAS bio que se basa en las propiedades físicas de las células (levitación) para separarlas de los desechos.
Aquí, describimos protocolos para obtener un alto número de núcleos y la mejor pureza para el protocolo Multiome aguas abajo. La clasificación FACS y los pasos repetidos de centrifugación dan como resultado una pérdida sustancial del material inicial. Por esta razón, en el protocolo para la preparación de núcleos del cerebro que describimos aquí se requiere material de partida lo suficientemente abundante como para dar lugar a la recolección de al menos 500.000 núcleos después de la clasificación FACS. Se deben aplicar protocolos alternativos si no se puede cumplir este criterio. Cuando se trabaja con poblaciones celulares raras o pequeñas secciones de tejido, la cantidad disponible de material inicial puede ser un factor limitante. Para abordar este problema, es posible mejorar la recuperación de núcleos (a) reduciendo el volumen de lisis, (b) reduciendo el volumen de lavado, (c) utilizando un solo lavado con tiempo de centrifugación extendido para tratar de mejorar la recuperación como se indica en los protocolos de 10X Genomics para el aislamiento de núcleos de baja entrada celular. Para el análisis multiómico de material de bajo contenido, vale la pena considerar aplicaciones basadas en placas como scNMT, SNARE-seq y Paired-seq38 que requieren muchas menos muestras de entrada.
En resumen, hemos descrito dos protocolos robustos para la preparación de núcleos del cerebro y de la médula ósea para el análisis posterior de Multiome. Estos protocolos son aplicables en cualquier proyecto científico que requiera suspensiones de núcleos individuales de alta calidad a partir de estos dos tipos de tejidos, independientemente de la cuestión científica planteada. Nuestro grupo ha estado aplicando el protocolo de aislamiento de núcleos cerebrales en estudios de desarrollo cerebral tras la inactivación de varios genes diana y en estudios de respuesta inmune en el contexto de enfermedades neurológicas. Estamos utilizando el protocolo de aislamiento de núcleos de médula ósea para descifrar la participación de varias subpoblaciones hematopoyéticas en el establecimiento del sistema inmune.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Ana Jeemin Choi recibió un estipendio del Programa Internacional de Doctorado Pasteur - Universidad de París (PPU).
Materiales
| Name | Company | Catalog Number | Comments |
| 18 G x 1 ½ (1.2 mm x 38 mm) Agani needles | Terumo | AN*1838S1 | |
| 15 mL tubes | Falcon | 352097 | |
| 5 mL round bottom FACS tube with cell strainer cap 35 µm | falcon | 352235 | |
| 50 mL tubes | Falcon | 352070 | |
| 7-AAD | BD pharmagen | 559925 | |
| ACK Lysing Buffer | Gibco | A10492-01 | |
| APC anti-mouse CD117 (c-Kit) | BioLegend | 105812 | Clone: 2B8 |
| APC/Cyanine 7 anti-mouse CD16/32 (FcγR) | BioLegend | 101328 | Clone: 93 |
| BD FACSAria III | BD Biosciences | non-applicable | |
| BD FACSDiva Software v8.0.1 | BD Biosciences | non-applicable | |
| Bovine Serum Albumin stock solution 10% | Miltenyi Biotec | 130-091-376 | |
| Cell staining buffer | Biolegend | 420201 | |
| CFI Suprplan Fluor ELWD 40XC ON 0.6 | Nikon | non-applicable | |
| CMOS camera Prime 95B 25 mm | Photometrix | non-applicable | |
| Countess II FL Automated Cell Counter | Invitrogen | AMQAF1000 | |
| Countess cell counting chamber slide | Invitrogen | C10283 | |
| Coverglass 24 mm x 24 mm 0.13-0.17 mm | Brand | BR470819 | |
| Digitonine 5% | Invitrogen | BN2006 | |
| Disposable Scalpels | Swann-Morton | 0508 | |
| DMEM (1x) + GlutaMAX-I | Gibco | 31966-021 | |
| DPBS (10x) | Gibco | 14200-067 | |
| DTT | Sigma aldrich | 646563 | |
| Epifluorescence inverted microscope Nikon Ti2 -E | Nikon | non-applicable | |
| Eppendorf Safe-Lock Tubes 0.5 mL | Eppendorf | 30123603 | |
| Ethanol 70% | VWR | 83801.290 | |
| FITC anti-mouse CD34 | Invitrogen | 11-0341-85 | Clone: RAM34 |
| Forceps for dissection | FST | 11152-10 | |
| Heat-inactivated Fetal Bovine Serum (FBS) | Gibco | 11533387 | |
| Dounce Homogeniser 2 mL | Bellco glass | 1984-10002 | Pestle “A” Large Clearance: .0030-.0050″ and Pestle “B” Small Clearance: .0005-.0025″ |
| LIVE/DEAD fixable aqua dead cell stain kit | Invitrogen | L34957 | |
| Magnesium chloride solution 1 M | Sigma aldrich | M1028 | |
| Microcentrifuge | Eppendorf | 5424R | |
| Mounting medium Fluoromount-G | invitrogen | 00-4958-02 | |
| Nonidet P40 substitute | Sigma aldrich | 74385 | |
| Nuclease free water | ThermoFischer | AM9932 | |
| Nuclei buffer 20x | 10X Genomics | 2000153/2000207 | |
| Nuclei isolation kit EZ prep | Sigma Aldrich | NUC-101 | |
| OneComp eBeads compensation beads | Invitrogen | 01-1111-41 | |
| Pacific Blue anti-mouse lineage cocktail (including anti-mouse CD3, Ly-6G/Ly-6C, CD11b, CD45R/B220, TER-119) | BioLegend | 133310 | Clones (in the same order as the antibodies listed): 17A2, RB6-8C5, M1/70, RA3-6B2, Ter-119 |
| PCR Tube Strips 0.2 mL | Eppendorf | 951010022 | |
| PE anti-mouse Ly-6A/E (Sca-1) | BioLegend | 122507 | Clone: E13-161.7 |
| Petri dish 100 mm x 20 mm OPTILUX | Falcon | 353003 | |
| Ply-L-lysine 0.01% sterile-filtered suitable for cell culture | Sigma | P4707 | |
| Printed microscope slides 8 well 6 mm numbered | Epredia | ER-301B-CE24 | |
| Protein LoBind Tubes 1.5 mL | Eppendorf | 30108116 | |
| Recombinant Rnase inhibitor 5000 U | Takara | 2313A | |
| Scissors for dissection | FST | 14090-09 | |
| Sodium chloride solution 5 M | Sigma aldrich | 59222C | |
| Syringe filters, PES, 0.2 µm | Fisher Scientific | 15206869 | |
| Transparent nail polish | any | non-applicable | |
| Trizma Hydrochloride solution pH 7.4 | Sigma aldrich | T2194 | |
| Trypan Blue 0.4% | gibco | 15250061 | |
| Tween 20 | Biorad | 1662404 | |
| UltraPure Distilated Water Dnase/Rnase Free | Invitrogen | 10977-035 |
Referencias
- Clark, S. J., et al. scNMT-seq enables joint profiling of chromatin accessibility DNA methylation and transcription in single cells. Nature Communications. 9 (1), 781(2018).
- Lee, J., Hyeon, D. Y., Hwang, D. Single-cell multiomics: technologies and data analysis methods. Experimental & Molecular Medicine. 52 (9), 1428-1442 (2020).
- Cerrizuela, S., et al. High-throughput scNMT protocol for multiomics profiling of single cells from mouse brain and pancreatic organoids. STAR Protocols. 3 (3), 101555(2022).
- Dimitriu, M. A., Lazar-Contes, I., Roszkowski, M., Mansuy, I. M. Single-cell multiomics techniques: From conception to applications. Frontiers in Cell and Developmental Biology. 10, 854317(2022).
- Stoeckius, M., et al. Simultaneous epitope and transcriptome measurement in single cells. Nature Methods. 14 (9), 865-868 (2017).
- Cao, J., et al. Joint profiling of chromatin accessibility and gene expression in thousands of single cells. Science (New York, N.Y.). 361 (6409), 1380-1385 (2018).
- Narayanan, A., et al. Nuclei Isolation from Fresh Frozen Brain Tumors for Single-Nucleus RNA-seq and ATAC-seq. Journal of Visualized Experiments: JoVE. (162), e61542(2020).
- Kim, M., et al. Single-nucleus transcriptomics reveals functional compartmentalization in syncytial skeletal muscle cells. Nature Communications. 11 (1), 6375(2020).
- Santos, M. D., et al. Extraction and sequencing of single nuclei from murine skeletal muscles. STAR Protocols. 2 (3), 100694(2021).
- Safabakhsh, S., et al. Isolating nuclei from frozen human heart tissue for single-nucleus RNA sequencing. Current Protocols. 2 (7), e480(2022).
- Lau, S. -F., Cao, H., Fu, A. K. Y., Ip, N. Y. Single-nucleus transcriptome analysis reveals dysregulation of angiogenic endothelial cells and neuroprotective glia in Alzheimer's disease. Proceedings of the National Academy of Sciences of the United States of America. 117 (41), 25800-25809 (2020).
- Armand, E. J., Li, J., Xie, F., Luo, C., Mukamel, E. A. Single-cell sequencing of brain cell transcriptomes and epigenomes. Neuron. 109 (1), 11-26 (2021).
- Morabito, S., et al. Single-nucleus chromatin accessibility and transcriptomic characterization of Alzheimer's disease. Nature Genetics. 53 (8), 1143-1155 (2021).
- Chen, S., et al. Spatially resolved transcriptomics reveals genes associated with the vulnerability of middle temporal gyrus in Alzheimer's disease. Acta Neuropathologica Communications. 10 (1), 188(2022).
- Paul, F., et al. Transcriptional heterogeneity and lineage commitment in myeloid progenitors. Cell. 163 (7), 1663-1677 (2015).
- Kaufmann, E., et al. BCG educates hematopoietic stem cells to generate protective innate immunity against tuberculosis. Cell. 172 (1-2), 176-190.e19 (2018).
- Christ, A., et al. Western diet triggers NLRP3-dependent innate immune reprogramming. Cell. 172 (1-2), 162-175.e14 (2018).
- Moorlag, S. J. C. F. M., et al. β-Glucan Induces protective trained immunity against mycobacterium tuberculosis infection: A key role for IL-1. Cell Reports. 31 (7), 107634(2020).
- de Laval, B., et al. C/EBPβ-dependent epigenetic memory induces trained immunity in hematopoietic stem cells. Cell Stem Cell. 26 (5), 657-674.e8 (2020).
- Renthal, W., et al. Characterization of human mosaic Rett syndrome brain tissue by single-nucleus RNA sequencing. Nature Neuroscience. 21 (12), 1670-1679 (2018).
- Yang, A. C., et al. A human brain vascular atlas reveals diverse mediators of Alzheimer's risk. Nature. 603 (7903), 885-892 (2022).
- Lee, D. R., Zhang, Y., Rhodes, C. T., Petros, T. J. Generation of single-cell and single-nuclei suspensions from embryonic and adult mouse brains. STAR Protocols. 4 (1), 101944(2022).
- Corces, M. R., et al. Lineage-specific and single-cell chromatin accessibility charts human hematopoiesis and leukemia evolution. Nature Genetics. 48 (10), 1193-1203 (2016).
- Ranzoni, A. M., et al. Integrative single-cell RNA-seq and ATAC-seq analysis of human developmental hematopoiesis. Cell Stem Cell. 28 (3), 472-487.e7 (2021).
- 10X Genomics Chromium Next GEM Single Cell Multiome ATAC + Gene Expression User Guide, Document Number CG000338 Rev F. , At https://www.10xgenomics.com/support/single-cell-multiome-atac-plus-gene-expression/documentation/steps/library-prep/chromium-next-gem-single-cell-multiome-atac-plus-gene-expression-reagent-kits-user-guide (2022).
- Meyerhoff,, et al. Microdissection of mouse brain into functionally and anatomically different regions. Journal of Visualized Experiments: JoVE. (168), e61941(2021).
- 10X Genomics 10X Genomics Demonstrated Protocol - Nuclei Isolation for Single Cell Multiome ATAC + GEX sequencing (CG000365 - Rev C). , At https://www.10xgenomics.com/support/single-cell-multiome-atac-plus-gene-expression/documentation/steps/sample-prep/nuclei-isolation-for-single-cell-multiome-atac-plus-gene-expression-sequencing (2022).
- Haag, S., Murthy, A. Murine monocyte and macrophage culture. Bio-Protocol. 11 (6), e3928(2021).
- Haque, A., Engel, J., Teichmann, S. A., Lönnberg, T. A practical guide to single-cell RNA-sequencing for biomedical research and clinical applications. Genome Medicine. 9 (1), 75(2017).
- Jiang, P. Quality control of single-cell RNA-seq. Methods in Molecular Biology (Clifton, N.J). 1935, 1-9 (2019).
- Regan, C., Preall, J. Practical considerations for single-cell genomics. Current Protocols. 2 (8), e498(2022).
- 10X Genomics 10X Genomics Demonstrated Protocol - Nuclei Isolation for Single Cell ATAC Sequencing (CG000169 - Rev E). , At https://www.10xgenomics.com/support/single-cell-atac/documentation/steps/sample-prep/nuclei-isolation-for-single-cell-atac-sequencing (2022).
- 10X Genomics 10X Genomics Demonstrated Protocol - Nuclei Isolation from Complex Tissues for Single Cell Multiome ATAC + Gene Expression Sequencing. (CG000375 - Rev C). , At https://www.10xgenomics.com/support/single-cell-multiome-atac-plus-gene-expression/documentation/steps/sample-prep/nuclei-isolation-from-complex-tissues-for-single-cell-multiome-atac-plus-gene-expression-sequencing (2022).
- 10X Genomics 10X Genomics - Chromium Nuclei Isolation Kit (CG000505 - Rev A). , At https://www.10xgenomics.com/support/single-cell-gene-expression/documentation/steps/sample-prep/chromium-nuclei-isolation-kit-sample-prep-user-guide (2022).
- Shomroni, O., et al. A novel single-cell RNA-sequencing approach and its applicability connecting genotype to phenotype in ageing disease. Scientific Reports. 12 (1), 4091(2022).
- Ocañas, S. R., Pham, K. D., Blankenship, H. E., Machalinski, A. H., Chucair-Elliott, A. J., Freeman, W. M. Minimizing the ex vivo confounds of cell-isolation techniques on transcriptomic and translatomic profiles of purified microglia. eNeuro. 9 (2), (2022).
- Gérard, A., et al. High-throughput single-cell activity-based screening and sequencing of antibodies using droplet microfluidics. Nature Biotechnology. 38 (6), 715-721 (2020).
- Vandereyken, K., Sifrim, A., Thienpont, B., Voet, T. Methods and applications for single-cell and spatial multi-omics. Nature Reviews. Genetics. 24, 494-515 (2023).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados