Method Article
Ingeniería de proteínas por visualización de superficie de levadura
En este artículo
Resumen
Este protocolo describe los pasos esenciales para llevar a cabo campañas de selección de exhibición de superficie de levadura para enriquecer las variantes de proteínas que se unen a un antígeno de interés.
Resumen
La ingeniería de proteínas permite la mejora de las funciones existentes de una proteína determinada o la generación de nuevas funciones. Una de las herramientas más utilizadas y versátiles en el campo de la ingeniería de proteínas es la visualización de la superficie de la levadura, donde un grupo de proteínas aleatorias se expresa en la superficie de la levadura. La unión del fenotipo (por ejemplo, la unión de la proteína mostrada por la levadura al antígeno de interés) y el genotipo (el plásmido que codifica para la variante de proteína) permite la selección de esta biblioteca para las propiedades deseadas y la posterior secuenciación de variantes enriquecidas. Al combinar la selección magnética de perlas con la clasificación por citometría de flujo, se pueden seleccionar y enriquecer variantes de proteínas con una unión mejorada a un antígeno objetivo. En particular, además de la maduración de la afinidad, también se puede lograr la unión a un objetivo sin ninguna afinidad de enlace inicial. Aquí, proporcionamos un protocolo paso a paso que cubre todas las partes esenciales de una campaña de selección de visualización de superficies de levadura y brinda ejemplos de resultados típicos de visualización de superficies de levadura. Demostramos que la visualización de la superficie de la levadura es un método robusto y ampliamente aplicable que puede establecerse en cualquier laboratorio de biología molecular con acceso a la citometría de flujo.
Introducción
La visualización de la superficie de la levadura es una de las tecnologías clave en el campo de la ingeniería de proteínas. Permite la selección de variantes de proteínas con propiedades deseadas, como una mejor afinidad o estabilidad. Introducida por primera vez en 19971, es una de las tecnologías de visualización más utilizadas junto con la visualización de fagos 2,3, la visualización de ribosomas4 y la visualización de células de mamíferos 5,6,7. La proteína de interés (POI) se muestra en la superficie de las células de levadura fusionándola para anclar proteínas. Dispone de una gama de diferentes proteínas de anclaje y, lo más común, el POI se fusiona con el extremo C-terminal de la proteína de apareamiento aglutinina de levadura Aga2p 1,8. Además, el POI suele estar flanqueado por dos etiquetas, como una etiqueta de hemaglutinina (HA-tag) y una etiqueta c-myc, que permite la detección del nivel de visualización mediante el uso de anticuerpos marcados con fluorescencia y citometría de flujo (Figura 1A). Las campañas típicas de selección de levadura implican una combinación de selecciones de perlas magnéticas y clasificación por citometría de flujo. Las selecciones de perlas permiten el manejo de grandes cantidades de células y el enriquecimiento de variantes proteicas que se unen al antígeno diana también con bajas afinidades, ya que las interacciones polivalentes con las perlas cargadas de antígeno conducen a efectos de avidez y, por lo tanto, evitan la pérdida de variantes de baja afinidad (Figura 1B). El análisis y la selección de citometría de flujo ofrecen la ventaja de visualizar la unión de las variantes de POI mostradas al antígeno marcado. En consecuencia, las poblaciones de unión se pueden clasificar y cultivar, lo que conduce al enriquecimiento de variantes de proteínas con las características deseadas a lo largo de varias rondas de clasificación. Además, se pueden realizar rondas adicionales de mutagénesis aleatoria para aumentar aún más la diversidad y, por lo tanto, la probabilidad de encontrar mutaciones adicionales que contribuyan a la afinidad y/o estabilidad de la proteína.
La visualización de la superficie de la levadura presenta ciertas ventajas, como (a) la maquinaria de expresión eucariota, que permite el plegamiento oxidativo de proteínas, así como modificaciones eucariotas postraduccionales (como la N-glicosilación), (b) la normalización de la expresión debido a la detección de las dos etiquetas peptídicas que flanquean la proteína, (c) la inspección visual del progreso de la selección mediante citometría de flujo (por ejemplo, porcentaje de células de unión e intensidad de unión) y (d) la posibilidad de analizar mutantes de proteínas individuales en levadura (por ejemplo, analizando la termoestabilidad y la afinidad), presentando una alternativa que ahorra tiempo a la laboriosa expresión y purificación de proteínas9. De hecho, tanto las afinidades (valores de KD) como las estabilidades (valores de T50) de las proteínas mostradas en la superficie de la levadura han mostrado buenas correlaciones con los datos obtenidos mediante métodos biofísicos y proteínas solubles 9,10,11,12. La visualización de la superficie de la levadura se ha empleado para la ingeniería de una variedad de proteínas, por ejemplo, fragmentos de anticuerpos 13,14,15,16, el 10º dominio de fibronectina tipo III17,18, rcSso7d19,20 o nudos21. Del mismo modo, se han llevado a cabo numerosas investigaciones para optimizar los diseños de las bibliotecas de levaduras mediante la alteración de las posiciones aleatorias, así como el uso de codones de aminoácidos 17,22,23. Se ha demostrado que la visualización de la superficie de la levadura es exitosa para la ingeniería de la estabilidad 14,15,24,25, la afinidad 18,26,27, la actividad enzimática 28,29,30,31 y la expresión de proteínas 32. Además, se lograron aplicaciones más sofisticadas, como la unión condicional en presencia o ausencia de una molécula pequeña, utilizando la visualización de la superficie de levadura20.
En este protocolo, describimos todos los pasos esenciales para una campaña de selección con visualización de la superficie de la levadura con el ejemplo de la biblioteca G4 (basada en el10º dominio de fibronectina tipo III, Fn3) seleccionada contra el antígeno proteína 4 de unión al retinol humano (hRBP4) en presencia de la pequeña molécula A112020. Esta selección se llevó a cabo para producir una interacción proteína-proteína que depende de una pequeña molécula que se puede utilizar como interruptor molecular. Cabe destacar que, si bien son posibles enfoques alternativos con la visualización de la superficie de la levadura, las selecciones típicas de levadura generalmente apuntan a unirse a un antígeno objetivo sin ninguna afinidad de unión previa. Cubrimos todos los pasos de una campaña de selección de levaduras, que implica el cultivo de una biblioteca de levaduras, la selección de perlas, la clasificación por citometría de flujo y la maduración por afinidad mediante PCR propensa a errores (epPCR). Por lo tanto, este protocolo complementa los protocolos anteriores de visualización de la superficie de la levadura33,34 y puede utilizarse como base para las selecciones de visualización de la superficie de la levadura (Figura 1) con cualquier biblioteca de levadura dada y antígeno objetivo de elección.
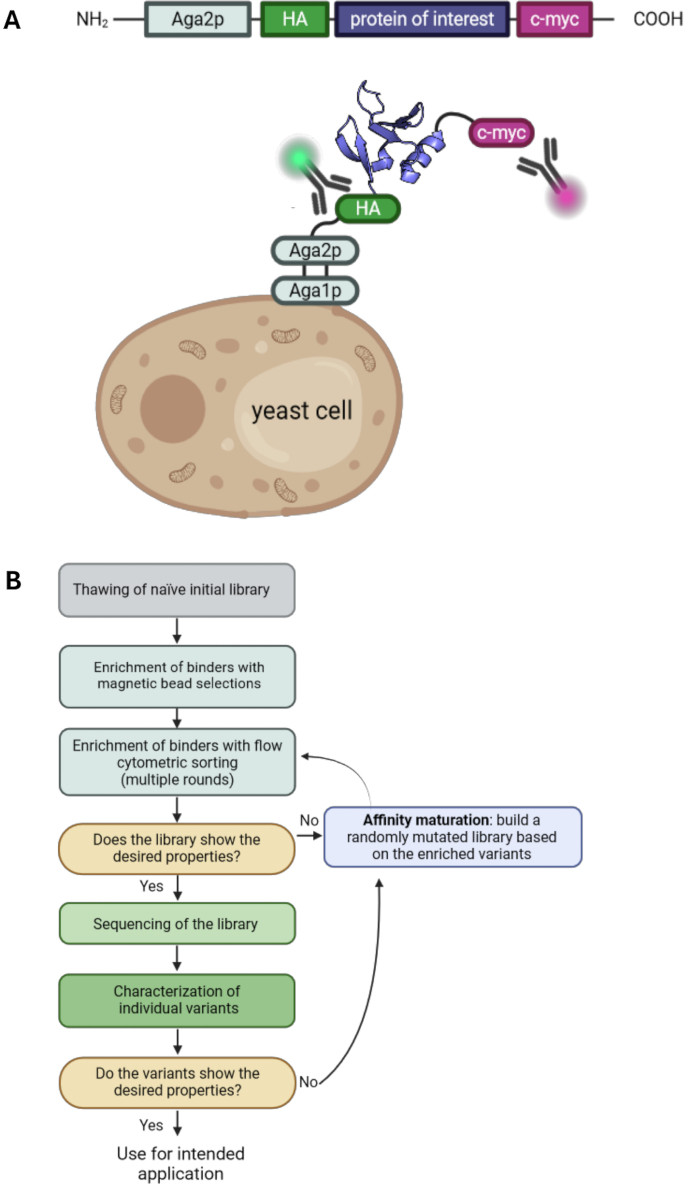
Figura 1: Principio de visualización de la superficie de la levadura y un flujo de trabajo típico para las selecciones de visualización de la superficie de la levadura. (A) El POI se clona en un vector de visualización de la superficie de levadura y, por lo general, está flanqueado por una etiqueta HA- N-terminal y una etiqueta c-myc C-terminal. La construcción se fusiona con la proteína de apareamiento de la levadura Aga2p para su visualización en la superficie. La proteína representada es el aglutinante diseñado "RS3" de PDB ID: 6QBA20. (B) Diagrama de flujo que ilustra un flujo de trabajo típico para campañas de selección de exhibición de superficie de levadura, que combinan el enriquecimiento de variantes de proteínas con las propiedades deseadas mediante selecciones de perlas y clasificación por citometría de flujo, así como epPCR para la maduración por afinidad. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
1. Descongelación y cultivo de bibliotecas de levadura
- Descongele suficientes alícuotas de la biblioteca de levadura congelada en RT y diluya inmediatamente en SD-CAA (ver Tabla 1) hasta un OD600 de aproximadamente 1 (esto corresponde a 1 x 107 células/mL).
NOTA: Asegúrese de cubrir la diversidad de la biblioteca al menos 10 veces. Por lo tanto, si el tamaño de la biblioteca es de 2,5 x 108 clones (esta es la diversidad de la biblioteca G4 utilizada en este ejemplo), inocule un mínimo de 2,5 x 109 células en 250 mL de SD-CAA para alcanzar un OD600 de 1. Este protocolo también se puede iniciar a partir de cultivos líquidos (células de levadura recién transformadas con una biblioteca). - Evaluar el número de células viables.
- Tome una alícuota de 100 μL de esta suspensión de levadura (a DO600 de 1) y realice una serie de dilución 1:10 con 900 μL de medio SD-CAA en tubos de microcentrífuga.
- Prepare diluciones de hasta 10-5 y coloque 100 μL de diluciones 10-3, 10-4 y 10-5 en placas SD-CAA (Tabla 1). Incubar las placas a 30 °C durante 2-3 días. Asegúrese de que el número de células viables después de la descongelación supere la diversidad de la biblioteca en al menos 10 veces.
- Mientras tanto, continúe con la suspensión celular restante y cultive el cultivo de levadura que contiene la biblioteca G4 en matraces de vidrio durante la noche a 30 °C mientras agita (180 rpm). Después de la incubación nocturna, continuar con la sección 2 (Inducción de la expresión de proteínas en la superficie de la levadura).
- Después de 2-3 días de incubación, cuente las colonias en las placas SD-CAA mediante inspección visual. Al calcular el número final de células viables después de la descongelación, considere el factor de dilución y la proporción de la placa (es decir, 100 μL) frente al volumen de cultivo original (es decir, 250 mL).
Células viables/mL = número de colonias en placa × factor de dilución
Células viables (total) = (células viables/mL) × volumen de cultivo
| Medio/tampón | Componente | Concentración [g/L] | Comentarios/Descripción | |||
| SD-CAA | D-glucosa | 20 | Disuelva todos los componentes del medio en 1000 mL ddH2O y filtrado estéril con filtros estériles desechables de 0,22 μm. | |||
| Base de nitrógeno de levadura | 6.7 | |||||
| Aminoácidos Casmino | 5 | |||||
| Ácido cítrico monohidratado | 7.4 | |||||
| Citrato trisódico dihidratado | 10.83 | |||||
| SG-CAA | D-galactosa | 20 | Disuelva todos los componentes del medio en 1000 mL ddH2O y filtrado estéril con filtros estériles desechables de 0,22 μm. | |||
| D-glucosa | 2 | |||||
| Base de nitrógeno de levadura | 6.7 | |||||
| Ácidos casaminos | 5 | |||||
| hidrogenofosfato disódico heptahidratado | 10.2 | |||||
| Dihidrógeno fosfato de sodio monohidratado | 8.56 | |||||
| Placas SD-CAA | Sorbitol | 182 | Disolver sorbitol, hidrogenofosfato disódico heptahidratado, dihidrógeno fosfato sódico monohidratado y agar-agar en 900 mL ddH2O y autoclave. Disolver y filtrar estérilmente los componentes restantes en 100 mL ddH2O y añadir cuando el medio esterilizado en autoclave esté tibio. | |||
| hidrogenofosfato disódico heptahidratado | 10.2 | |||||
| Dihidrógeno fosfato de sodio monohidratado | 7.44 | |||||
| Agar-agar | 15 | |||||
| D-glucosa | 20 | |||||
| Base de nitrógeno de levadura | 6.7 | |||||
| Ácidos casaminos | 5 | |||||
| YPD | Peptona | 20 | Prepare un caldo de D-glucosa 10x (200 g/L) y un filtrado estéril con filtros estériles desechables de 0,22 μm. Disolver peptona y extracto de levadura en 900 mL ddH2O y autoclave. Cuando esté tibio, agregue 100 mL 10x D-glucosa. | |||
| Extracto de levadura | 10 | |||||
| D-glucosa | 20 | |||||
| Placas YPD | Peptona | 20 | Prepare un caldo de D-glucosa 10x (200 g/L) y un filtrado estéril con filtros estériles desechables de 0,22 μm. Disolver peptona, extracto de levadura y agar-agar en 900 mL ddH2O y autoclave. Cuando esté tibio, agregue 100 mL 10x D-glucosa. | |||
| Extracto de levadura | 10 | |||||
| D-glucosa | 20 | |||||
| Agar-agar | 15 | |||||
| PBSA | BSA | 1 | Disuelva BSA en PBS y filtrado estéril con filtros estériles desechables de 0,22 μm. | |||
Tabla 1: Composición de medios y búferes.
2. Inducción de la expresión de proteínas en la superficie de la levadura
- Después de la incubación durante la noche, los cultivos de levadura suelen alcanzar un OD600 entre 2 y 20. Mida el OD600 con un fotómetro para determinar la densidad del cultivo de levadura.
NOTA: Las mediciones de OD solo son lineales en el rango de 0.1-1. Por lo tanto, realice diluciones (normalmente 1:20 y 1:50) en SD-CAA para determinar el OD600.- Diluya las células en SD-CAA hasta un OD600 de 1 y asegúrese de que la diversidad esté cubierta 10x (es decir, se utilizan2,5 x 10 9 células para la dilución de la biblioteca G4). Continúe la incubación a 30 °C mientras agita para permitir que las células alcancen una fase de crecimiento exponencial.
- Prepare las existencias de glicerol (que cubren la diversidad 50 veces) de las bibliotecas de levadura (en SD-CAA que contiene un 15% de glicerol) a partir del cultivo nocturno. Para ello, centrifugar el número necesario de células de levadura (2000 x g, 3 min, 4 °C), desechar el sobrenadante y volver a suspender el pellet de la célula en 1 ml de medio SD-CAA suplementado con glicerol al 15%. Congele inmediatamente las existencias de glicerol a -80 °C.
NOTA: La viabilidad de las células de levadura aumenta si los cultivos de levadura se cultivan hasta una fase estacionaria antes de la congelación. En rondas de selección posteriores, cuando la diversidad de las bibliotecas es menor, normalmente es suficiente utilizar 700 μL del cultivo de levadura y mezclarlo con 300 μL de SD-CAA suplementado con glicerol al 50%. En este caso, los cultivos de levadura suelen mostrar valores de OD600 de 15 en la fase estacionaria, pero el valor real puede depender del fotómetro que se utilice. Por lo tanto, la fase estacionaria se puede determinar mediante mediciones repetidas de OD600 . Si el valor de OD600 no aumenta durante un período de incubación de 2 h, se alcanza una fase estacionaria.
- Centrifugar un volumen adecuado de células para cubrir la diversidad 10x a 2000 x g durante 5 min, 4-6 h después de la dilución del cultivo de levadura (los valores típicos de OD600 están entre 4-6), y desechar el sobrenadante.
- Vuelva a suspender el pellet de celda en SG-CAA (ver Tabla 1) para producir un OD600 de 1 y transfiéralo a un matraz de vidrio nuevo.
NOTA: La resuspensión en un medio que contiene galactosa induce la expresión de la proteína en la superficie por el promotor inducible por galactosa. - Incubar los cultivos de levadura durante la noche a 20 °C mientras se agitan a 180 rpm para inducir la expresión superficial del POI. Después de la inducción durante la noche, los cultivos de levadura generalmente alcanzarán valores de OD600 entre 2 y 5; si la temperatura de inducción se eleva a 37 °C, espere valores de OD600 de 4-10.
NOTA: Reducir la temperatura a 20 °C ralentiza el crecimiento de las células y, por lo general, produce mejores niveles de visualización. Sin embargo, la inducción a 37 °C podría ser útil para aumentar la presión de selección hacia mutantes estabilizados35.
3. Primera ronda de selección de perlas de las bibliotecas de levadura (selección positiva)
NOTA: Un procedimiento estándar de selección de cuentas implica 6 pasos (Tabla 2).
| Día | Paso | |
| 0 | Cultura de la noche a la mañana | |
| 1 | Inducción de la expresión de proteínas en la superficie de las células de levadura | |
| 2 | Primera selección de cuentas con 1 selección positiva | |
| 3 | Eliminación de las perlas, paseo, inducción de la expresión de proteínas en la superficie de las células de levadura y congelación de la biblioteca | |
| 4 | Segunda selección de cuentas con 3 selecciones negativas y 1 positiva | |
| 5 | Retirada de las cuentas y congelación de la biblioteca | |
Tabla 2: Cronología típica para la realización de selecciones de perlas de una biblioteca de levaduras.
- Preparación de las cuentas
- Prepare las perlas (consulte la Tabla de materiales) para la primera selección de perlas: Para cada tubo, vuelva a suspender 10 μL de perlas magnéticas de aglutinante de biotina (4 × 105 perlas/μL) en 990 μL de PBSA (Tabla 1) para lavar, coloque el tubo en una rejilla magnética durante 2 min con la tapa abierta. Retire con cuidado el sobrenadante. Repita el paso de lavado con 1 mL de PBSA.
NOTA: Si se necesitan varios tubos de perlas con el mismo antígeno biotinilado, se pueden preparar en un tubo en este paso para producir una solución uniforme de perlas de antígeno. En este caso, la cantidad de antígeno debe aumentarse en consecuencia en el siguiente paso. - Vuelva a suspender las perlas en un volumen total de 1 mL de PBSA con 6,7-33 pmoles de antígeno biotinilado (aquí: 6,86 μL de hRBP4 con una concentración de 0,125 mg/mL) en un tubo de microcentrífuga de 1,5 mL.
NOTA: Si bien 6,7 pmoles de antígeno son suficientes, se deben usar 33 pmoles si el antígeno no es limitante. Si la selección se realiza para un antígeno unido a un fármaco o ligando de molécula pequeña, como en este caso, este fármaco/ligando debe estar presente durante la selección y debe añadirse en este paso (aquí: 5 μM A1120). - Incubar a 4 °C durante 2 h con agitación o rotación suaves.
NOTA: Las celdas se pueden preparar durante este paso. - Después de la incubación y antes de la adición a las células, coloque el tubo en una rejilla magnética durante 2 minutos con la tapa abierta. Retire el sobrenadante y lave las perlas cargadas de antígeno con 1 mL de PBSA como se describe anteriormente. Repita el paso de lavado.
NOTA: Si se selecciona un antígeno unido a un fármaco o ligando, el tampón de lavado debe contener este fármaco o ligando en exceso (aquí: 5 μM A1120). - Vuelva a suspender las perlas cargadas de antígeno en 50 μL de PBSA (en presencia del fármaco o ligando, si procede).
NOTA: Si se prepararon las mismas perlas cargadas de antígeno para varios tubos, ajuste el volumen en consecuencia (para la biblioteca G4, el volumen final aquí es de 100 μL, lo que produce perlas para dos tubos).
- Prepare las perlas (consulte la Tabla de materiales) para la primera selección de perlas: Para cada tubo, vuelva a suspender 10 μL de perlas magnéticas de aglutinante de biotina (4 × 105 perlas/μL) en 990 μL de PBSA (Tabla 1) para lavar, coloque el tubo en una rejilla magnética durante 2 min con la tapa abierta. Retire con cuidado el sobrenadante. Repita el paso de lavado con 1 mL de PBSA.
- Preparación de las células de levadura
- Mida el OD600 del cultivo de levadura inducido como se describe anteriormente.
- Pellet suficientes celdas para cubrir 10 veces la diversidad de la biblioteca (2000 x g, 10 min, 4 °C). Para la biblioteca G4, utilice celdas de 2,5 x 109 para la centrifugación. Retirar el sobrenadante y lavar las células con 10 mL de PBSA.
- Centrifugar de nuevo (2000 x g, 10 min, 4 °C) y resuspender las células hasta una concentración de 1,25 x 109 células/mL en el volumen adecuado de PBSA (en este caso: 2 mL) y transferir a un tubo de microcentrífuga.
NOTA: El volumen de PBSA depende del número de células. - Centrifugar (2000 x g, 3 min, 4 °C) y resuspender en 950 μL de PBSA.
- Incubación de células de levadura con perlas cargadas de antígeno
- Añadir 50 μL de perlas cargadas de antígeno a 950 μL de suspensión celular e incubar durante 2 h a 4 °C mientras se agita o se rota.
- Después de la incubación, coloque la solución de perlas de antígeno celular en una rejilla magnética con la tapa abierta. Pipetear el líquido desde la tapa hasta el tubo para someter todo el cultivo de levadura a una selección magnética. Espere 2 minutos antes de desechar con cuidado las celdas no unidas. Lavar inmediatamente con 1 mL de PBSA.
NOTA: Si las selecciones se realizan en presencia de un fármaco/ligando, en este ejemplo A1120, este debe estar presente en PBSA. - Después de la etapa de lavado con PBSA, vuelva a suspender las células rápidamente en 1 mL de SD-CAA para evitar que se sequen.
- Agregue 200 mL de SD-CAA y realice diluciones seriadas como se describe anteriormente. Colocar 100 μL en placas SD-CAA e incubar durante 2-3 días a 30 °C para determinar la nueva diversidad máxima. Aquí, la diversidad de la biblioteca G4 después de la primera selección de cuentas es de 1,25 x106 celdas.
NOTA: Dado que la biblioteca original se sobremuestreó 10 veces en esta ronda de selección, es razonable suponer que la mayoría de los clones estarán presentes varias veces en el grupo enriquecido después de la selección. Como consecuencia, la diversidad real será menor, y este número debe considerarse como un valor máximo. Por lo general, la diversidad se reduce al menos 100 veces dentro de esta primera ronda de selección. Incubar el cultivo restante (200 mL) a 30 °C con agitación (180 rpm) durante al menos 16 h. El protocolo se puede pausar después de la incubación nocturna colocando las células a 4 °C.
4. Retirada de las cuentas y cultivo antes de la siguiente ronda de selección de cuentas
- Mida OD600 como se describe anteriormente. Pellet suficientes celdas para cubrir la diversidad en al menos 10 veces (2000 x g, 3 min, RT). Deseche el sobrenadante y vuelva a suspender las células en 1 mL de SD-CAA.
- Transfiera a un tubo de microcentrífuga y colóquelo en una rejilla magnética durante 2 minutos con la tapa abierta. Transfiera las células no unidas a un nuevo tubo de microcentrífuga y repita el proceso de separación.
- Diluir las células no unidas en 100 mL de SD-CAA e incubar a 30 °C agitando a 180 rpm hasta alcanzar un OD600 de 3-4.
NOTA: Si el diámetro exterior de600 es inferior a 4 antes de la extracción de las perlas, las células se pueden inducir inmediatamente después sin incubación. - Cuando se alcance un diámetro exterior de600 grados de 3-4, se centrifugará un número suficiente de células para cubrir la diversidad al menos 10 veces (2000 x g, 3 min, RT) y se resuspenda en SG-CAA a un diámetro exterior de600 de 1 para la inducción de la expresión de proteínas e incubar a 20 °C mientras se agita a 180 rpm durante la noche.
- Cuando el cultivo de levadura SD-CAA restante ha alcanzado la fase de crecimiento estacionaria (OD600 por encima de 15), granule suficientes células para cubrir la diversidad 50x (2000 x g, 3 min, RT). Congelación de las células por centrifugación (2000 x g, 3 min), seguida de la resuspensión del pellet de la célula en SD-CAA que contiene un 15% de glicerol y almacenamiento a -80 °C.
5. Segunda ronda de selección de cuentas con 3 selecciones negativas y 1 positiva
- Prepare las células y las perlas de antígeno como se describió anteriormente. Prepare una solución de perlas desnudas (sin agregar el antígeno) para las selecciones negativas.
NOTA: Dado que se realizan 3 selecciones negativas, las cuentas se pueden preparar juntas para las tres rondas. - Después del lavado, vuelva a suspender las perlas de antígeno en 50 μL de PBSA y vuelva a suspender las perlas desnudas en 150 μL de PBSA (si estaban preparadas para las tres selecciones negativas).
- Para la primera selección negativa, añadir 50 μL de perlas desnudas lavadas a 950 μL de células lavadas en PBSA e incubar durante 1,5 h a 4 °C.
- Después de la incubación, coloque los tubos que contienen las suspensiones de perlas desnudas en una rejilla magnética con la tapa abierta. Pipetea cualquier líquido de la tapa en el tubo y espera 2 min. Transfiera las células no unidas a un tubo de microcentrífuga nuevo y agregue 50 μL de perlas desnudas lavadas.
- Repita el procedimiento para dos rondas más de selección negativa. Mientras tanto, prepare las perlas cargadas de antígeno para la selección positiva.
- Después de 3 rondas de selección negativas, añadir 50 μL de solución de perlas cargada de antígeno a las células e incubar durante 2 h a 4 °C.
- Coloque las células que contienen las perlas cargadas de antígeno en una rejilla magnética con la tapa abierta y pipetee cualquier líquido que esté en la tapa en el tubo. Espere 2 minutos antes de descartar las celdas no unidas.
- Realice todos los pasos restantes como se describe para la primera selección de antígeno-perla. La única diferencia con la primera selección positiva (paso 3.3.2) es el número de pasos de lavado: en lugar de lavar una vez con PBSA, lavar la suspensión de celda de perla dos veces con PBSA para aumentar la rigurosidad de la selección.
NOTA: Por lo general, la diversidad se reduce nuevamente entre 10 y 100 veces. La diversidad máxima de la biblioteca G4 después de esta segunda selección de cuentas fue de 3,45 x 105 celdas. Como se señaló anteriormente, se espera que la diversidad real sea menor debido al sobremuestreo de la biblioteca.
6. Selección de bibliotecas mediante clasificación por citometría de flujo
- Después de la inducción nocturna de la expresión superficial en SG-CAA, mida OD600 como se describe anteriormente. Pellet suficientes celdas para cubrir 10 veces la diversidad (2000 x g, 5 min, RT). Deseche el sobrenadante.
- Vuelva a suspender el pellet en PBSA y transfiéralo a tubos de microcentrífuga. Use 3 x 107 celdas para tintar cada tubo. Prepare tantos tubos como sea necesario, dependiendo de la diversidad. Prepare un tubo de control para la tinción sin el antígeno.
NOTA: Por ejemplo, si se deben clasificar 30 x 106 celdas, granule 300 x 106 celdas para lograr una celda visible y evitar la pérdida de celdas en los pasos posteriores. Vuelva a suspender en 5 mL de PBSA y transfiera de 3 a 4 alícuotas de 500 μL cada una a tubos de microcentrífuga. Prepare aproximadamente 3 veces más celdas que el número que se debe clasificar porque se perderá una fracción de las celdas durante el proceso de tinción. Si la selección se realiza en presencia de un fármaco/ligando, prepare un control adicional solo con el antígeno pero sin el fármaco/ligando. - Centrifugar de nuevo (2000 x g, 5 min, RT), resuspender el pellet en 200 μL de PBSA que contiene el antígeno (hRBP4 con 5 μM A1120) e incubar durante 1 h a 4 °C.
NOTA: Todos los pasos a partir de este punto deben realizarse con hielo y con reactivos fríos. Para evitar la depleción de anticuerpos o antígenos, todos los reactivos deben añadirse en exceso estequiométrico en comparación con el número total de proteínas de levadura que se muestran en el tubo. De lo contrario, podría existir el riesgo de limitar la unión de las variantes mostradas por la levadura debido a la disponibilidad limitada de antígenos y/o anticuerpos. Por lo general, se utilizan 5 x 104 moléculas por célula de levadura para el cálculo, y se recomienda agregar al menos un exceso estequiométrico de antígeno y anticuerpos. Si el antígeno es muy limitado, el exceso se puede reducir a 5 veces. Una explicación más detallada, incluyendo ejemplos de cálculos para evitar el agotamiento del ligando, se proporciona en un protocolo anterior9. - Después de la incubación, centrifugar las células (2000 x g, 5 min, 4 °C) y lavar con 1 mL de PBSA (si hay un fármaco/ligando durante las selecciones, como A1120 en este caso, debe estar presente en el tampón de lavado).
- Vuelva a suspender las células en 100 μL de PBSA frío que contenga los anticuerpos para la visualización de la tinción y la detección del antígeno unido e incube durante 30 min a 4 °C.
- Utilice el anticuerpo anti-c-myc (dilución final 1:100) para detectar la POI completa. Utilice el anticuerpo anti-HA (dilución final 1:50) para detectar la cantidad total de proteínas mostradas. Utilice el anticuerpo Penta-His (dilución final 1:20) o la estreptavidina (dilución final 1:200) para seleccionar células de levadura que se unan al antígeno soluble que contengan una etiqueta HIS 6x o biotina, respectivamente.
- Alterne entre los reactivos de detección entre las rondas de clasificación para evitar el enriquecimiento de las variantes de proteínas que se unen al reactivo secundario.
NOTA: Se pueden utilizar diferentes combinaciones de fluoróforos, pero Alexa Fluor 488 y Alexa Fluor 647 son buenas opciones, ya que no requieren compensación en la citometría de flujo. Para prevenir la depleción de anticuerpos, debe agregarse en exceso estequiométrico en comparación con el número total de proteínas de levadura que se muestran en el tubo. Por lo general, los cálculos se basan en 5 x 104 moléculas por célula de levadura. Una explicación más detallada se proporciona en los protocolos anteriores9.
- Después de la incubación, centrifugar las células (2000 x g, 5 min, 4 °C) y añadir 1 mL de PBSA (que contiene el fármaco/ligando, para la biblioteca G4 seleccionada por hRBP4 5 μM de A1120) al pellet y volver a centrifugar. Retire la mayor parte del sobrenadante y conserve solo 20-30 μL para evitar que el pellet se seque.
- Vuelva a suspender el pellet en PBSA frío solo justo antes de la clasificación y clasifique las células de levadura que muestren moléculas de Fn3 con las propiedades deseadas (es decir, que se unan a hRBP4 en presencia de A1120).
NOTA: La estrategia de compuerta se muestra en la Figura 2A. - Clasifique las células directamente en el medio SD-CAA, agregue más medio SD-CAA después de la clasificación e incube a 30 °C agitando a 180 rpm.
NOTA: En caso de que el volumen aumente significativamente después de la clasificación, se debe realizar un paso de centrifugación (2000 x g, 5 min, RT) para eliminar el exceso de líquido de la vaina, y las células se resuspenden en un medio SD-CAA fresco. Anote el número de celdas clasificadas en el nuevo tubo, ya que esto se convierte en la nueva diversidad máxima para la siguiente ronda de clasificación. Como se señaló anteriormente, la diversidad real de la biblioteca será menor debido al sobremuestreo de la biblioteca. - Repita las clasificaciones de citometría de flujo durante varias rondas para ver un enriquecimiento de las variantes de proteínas de unión en la biblioteca de levaduras (Figura 2B).
NOTA: Puede ser útil comparar las bibliotecas una al lado de la otra en un experimento analítico de citometría de flujo. En rondas de selección posteriores, la disminución de la concentración del antígeno puede ser beneficiosa para discriminar entre los aglutinantes de menor y mayor afinidad y clasificar específicamente las variantes de proteínas que muestran una señal de unión más alta al objetivo a bajas concentraciones (Figura 2C). En rondas de selección posteriores, también es posible clasificar para aumentar la termoestabilidad. Para ello, se realizó una mutagénesis aleatoria de la biblioteca mediante epPCR, tal y como se describe en la sección 7. Prepare las celdas de manera similar a otras rondas de clasificación de citometría de flujo por inducción, determinación de OD600 y lavado de celdas. Coloque las células de levadura en PBSA en hielo durante 10 minutos, seguido de una incubación de 10 minutos en un termociclador para el choque térmico a una temperatura elevada. Después, colóquelos de nuevo en hielo durante 10 minutos y proceda con el protocolo de tinción. Las variantes estabilizadas resistirán la desnaturalización térmica y, por lo tanto, mostrarán una unión mantenida después de la incubación por calor. Se pueden probar diferentes temperaturas para el choque térmico, dependiendo de la termoestabilidad inicial de la proteína de tipo salvaje. Por lo general, son adecuadas temperaturas que oscilan entre 40 y 80 °C. Sin embargo, por encima de los 48 °C, la viabilidad de las células de levadura disminuirá drásticamente. Por lo tanto, es necesario extraer los plásmidos después de clasificar la biblioteca de levadura con choque térmico, como se discutió en el paso 7.1. En este caso, las células EBY100 deben transformarse nuevamente con variantes enriquecidas antes de pasar a la siguiente ronda de clasificación36. Un protocolo más detallado para aumentar la termoestabilidad se puede encontrar en otro lugar37. Si la desnaturalización de la proteína mostrada es reversible, este método no funcionará. Alternativamente, induzca la expresión de proteínas a 37 °C en lugar de 20 °C el día anterior a la clasificación y clasifique las variantes de proteínas que aún muestren una visualización eficiente de cuerpo entero.
7. Maduración de la afinidad con epPCR para introducir mutaciones aleatorias
NOTA: La maduración por afinidad mediante epPCR se puede realizar antes de la primera ronda de clasificación por citometría de flujo o entre rondas de clasificación por citometría de flujo. Para la selección de la biblioteca G4 con hRBP4 en presencia de A1120, se llevó a cabo la maduración por afinidad antes de la primera ronda de clasificación por citometría de flujo. Esto también depende del tamaño de la biblioteca después de la selección del cordón y de la señal de unión que se puede detectar con la citometría de flujo. En particular, en los casos en los que las afinidades después de la selección de cordones no son suficientes para obtener una señal en los experimentos de citometría de flujo (porque el antígeno se disocia rápidamente durante los pasos de lavado), la epPCR puede generar variantes mejoradas que posteriormente pueden detectarse y seleccionarse mediante citometría de flujo.
- Realice una extracción de ADN de miniprep de levadura de la biblioteca de levadura enriquecida utilizando el kit comercial de miniprep II de plásmidos (consulte la tabla de materiales) con las siguientes modificaciones:
- Mida la densidad celular del cultivo durante la noche, diluya a un DO600 de 0,2 por la mañana e incube a 30 °C con agitación.
- Cuando OD600 alcance 0,6, coseche 3 mL de cultivo centrifugando las células a 600 x g durante 2 min.
- Añadir 200 μL de solución 1 al pellet. Añadir 6 μL de Zymoliasa. Vuelva a suspender el pellet mediante un movimiento de sacudida o vórtice leve e incube a 37 °C durante 3 h.
- Añadir 200 μL de solución 2 y mezclar suavemente. Añadir 400 μL de solución 3 y mezclar suavemente.
- Centrifugar a máxima velocidad durante 3 min.
- Transfiera el sobrenadante a la columna de espín I, centrifugue a la velocidad máxima durante 30 s y deseche el flujo.
- Lavar con 550 μL de tampón de lavado que contiene etanol y centrifugar a velocidad máxima durante 2 min.
- Transfiera la columna a un tubo de microcentrífuga limpio y centrifuga a máxima velocidad durante 2 minutos para eliminar el tampón de lavado residual.
- Transfiera la columna a un nuevo tubo de microcentrífuga, agregue 10 μL de agua libre de nucleasas y centrifugue durante 1 minuto a velocidad máxima para eluir el ADN.
- Realice una PCR inicial para amplificar la plantilla para epPCR.
- Amplifique el ADN por PCR utilizando cebadores recocidos en el exterior del inserto en el vector de visualización de la superficie de la levadura.
NOTA: Dado que se eluye una gran cantidad de ADN genómico de levadura junto con el plásmido de interés durante la minipreparación de levadura, se recomienda este paso para obtener un fragmento de ADN limpio que se utilizará como plantilla para la epPCR. - Para pCTCON2, use cebadores fwd 5'- GGCTCTGGTGGAGGCGGTAGCGGAGGCGGAG
GGTCGGCTAGC y rev 5'- CTATTACAAGTCC
TCTTCAGAAATAAGCTTTTGTGTTCGGATCC con los componentes de reacción descritos en la Tabla 3 y las condiciones de ciclo representadas en la Tabla 4. - Purifique la PCR con un kit de limpieza de PCR y ADN. Eluto en 20 μL de H2O libre de nucleasas.
- Amplifique el ADN por PCR utilizando cebadores recocidos en el exterior del inserto en el vector de visualización de la superficie de la levadura.
- Realizar epPCR para introducir mutaciones aleatorias.
- Realizar una epPCR con análogos de nucleótidos con los ingredientes indicados (Tabla 5) y condiciones (Tabla 6).
- Ejecute todo el producto epPCR en un gel de agarosa preparatorio al 1% a 120 V durante 45 min. Incluya un marcador y deje un carril entre el producto epPCR y la escalera. Extraer la banda deseada correspondiente al tamaño esperado del inserto, purificarla con un kit de extracción en gel y medir la concentración de ADN.
- Amplificar el ADN aleatorizado por PCR.
- Amplifica 50 ng del ADN con una polimerasa Q5. Configure 2 x 100 μL de reacción PCR para cada electroporación (Tabla 7) con las condiciones indicadas (Tabla 8). Utilice los mismos cebadores que se utilizaron para la primera PCR.
NOTA: Se requieren grandes cantidades de ADN para la electroporación en levadura. - Analice 5 μL del producto PCR en un gel de agarosa al 1%, ejecute con 120 V durante 45 min.
- Amplifica 50 ng del ADN con una polimerasa Q5. Configure 2 x 100 μL de reacción PCR para cada electroporación (Tabla 7) con las condiciones indicadas (Tabla 8). Utilice los mismos cebadores que se utilizaron para la primera PCR.
- Purificar el ADN amplificado con precipitación de etanol.
- Piscina de 2 x 100 μL juntos. Añadir 20 μL (10% del volumen) de acetato de sodio 3 M filtrado estéril, pH 5,2.
NOTA: Este tampón se puede preparar con antelación y almacenar durante unos meses en RT. - Agregue 440 μL de etanol al 100% (o al menos 2 veces el volumen) e incube a RT durante 2 min.
- Centrifugar a 20.000 x g durante 5 min a RT. Retirar el sobrenadante, añadir 500 μL de etanol al 70% y mezclar brevemente.
- Centrifugar a 20.000 x g durante 5 min a RT. Retirar el sobrenadante, añadir 500 μL de etanol al 100% y mezclar brevemente.
- Centrifugar a 20.000 x g durante 5 min a RT. Retirar el sobrenadante y dejar secar el pellet de ADN bajo una campana de flujo laminar hasta que se evapore todo el etanol.
NOTA: Utilice un bloque de calor a 48 °C para acelerar este proceso. Es posible que la bolita de ADN no sea visible en todo momento. - Disuelva el pellet de ADN seco en 10 μL de H2O libre de nucleasa.
NOTA: El ADN debe disolverse en el menor volumen posible. No mida la concentración de ADN.
- Piscina de 2 x 100 μL juntos. Añadir 20 μL (10% del volumen) de acetato de sodio 3 M filtrado estéril, pH 5,2.
| Volumen [μL] | Concentración final | |
| 5x Potenciador Q5 | 10 | 1 vez |
| 5x Búfer Q5 | 10 | 1 vez |
| Imprimación fwd 10 μM | 2.5 | 0,5 μM |
| Imprimación rev 10 μM | 2.5 | 0,5 μM |
| dNTPs 10 mM | 1 | 200 μM |
| Q5 polimerasa | 0.5 | 20 U/mL |
| ADN de la levadura miniprep | 10 | |
| Libre de nucleasas H2O | 13.5 |
Tabla 3: Condiciones para la PCR de1er paso para la amplificación de los genes POI a partir de la minipreparación de levadura aislada.
| Paso | Temperatura | Hora |
| Desnaturalización inicial | 98 °C | 30 s |
| 25 ciclos | 98 °C | 10 s |
| 72 °C | 30 s | |
| 72 °C | 30 s | |
| Prórroga definitiva | 72 °C | 2 minutos |
| Sostener | 4 °C |
Tabla 4: Condiciones de ciclo para la PCR de1er paso para la amplificación de los genes POI a partir de la minipreparación de levadura aislada.
| Volumen [μL] | Concentración final | |
| Libre de nucleasas H2O | hasta 50 | |
| 10x Tampón Thermopol | 5 | 1 vez |
| Primer_fwd (10 μM) | 2.5 | 0,5 μM |
| Primer_rev (10 μM) | 2.5 | 0,5 μM |
| dNTPs (10 mM) | 1 | 200 μM |
| 8-oxo-dGTP (100 μM) | 1 | 2 μM |
| dPTP (100 μM) | 1 | 2 μM |
| Producto PCR de 1ª PCR | XX | 50 ng |
| ADN polimerasa Taq | 0.5 | 0,05 U/μL |
Tabla 5: Condiciones para la epPCR que se realiza después de la amplificación del ADN POI con la PCR de1er paso.
| Paso | Temperatura | Hora |
| Desnaturalización inicial | 94 °C | 30 s |
| 15 ciclos | 94 °C | 45 s |
| 60 °C | 30 s | |
| 72 °C | 1 minuto | |
| Prórroga definitiva | 72 °C | 10 minutos |
| Sostener | 4 °C |
Tabla 6: Condiciones de ciclo para la epPCR.
| Volumen [μL] | Concentración final | |
| 5x Potenciador Q5 | 20 | 1 vez |
| 5x Búfer Q5 | 20 | 1 vez |
| Imprimación fwd 10 μM | 5 | 0,5 μM |
| Imprimación rev 10 μM | 5 | 0,5 μM |
| dNTPs 10 mM | 1 | 200 μM |
| Q5 polimerasa | 1 | 20 U/mL |
| 50 ng de ADN | XX | |
| ddH20 | hasta 100 |
Tabla 7: Condiciones para la PCR de2º paso para la amplificación del producto epPCR antes de la electroporación de las células EBY100.
| Paso | Temperatura | Hora |
| Desnaturalización inicial | 98 °C | 30 s |
| 25 ciclos | 98 °C | 10 s |
| 72 °C | 30 s | |
| 72 °C | 30 s | |
| Prórroga definitiva | 72 °C | 2 minutos |
| Sostener | 4 °C |
Tabla 8: Condiciones de ciclo para la PCR de2º paso para la amplificación del producto epPCR.
8. Linealización del vector de visualización de levadura para electroporación
- Linealice el vector de visualización de la superficie de la levadura pCTCON2 (plásmido Addgene #41843).
- Realice una digestión a gran escala y utilice este vector linealizado digerido para todas las electroporaciones posteriores.
- Digiera 2 x 200 μg de ADN plasmídico pCTCON2 con Sal I durante 24 h a 37 °C para una linealización inicial (Tabla 9).
- Digirir con NheI y BamHI durante 24 h a 37 °C para extirpar el inserto (Tabla 10).
- Por último, añadir 5 μL de cada enzima al tubo (Sal I, NheI, BamHI) y volver a incubar durante 24 h a 37 °C.
- Ejecute el vector digerido en un gel de agarosa preparativo. Eliminar las bandas y purificar con un kit de extracción en gel. Eluir el vector en 20 μL, medir la concentración de ADN y ajustarla a aproximadamente 2 μg/μL.
NOTA: La digestión del plásmido durante un tiempo prolongado asegura una escisión suficiente del inserto y dará como resultado menos fondo del vector después de la electroporación.
| ADN | 200 μg |
| 10x CutSmartBuffer | 50 μL |
| Sal I-HF (NEB) | 30 μL (60 U) |
| H2O | Hasta 500 μL |
Tabla 9: Condiciones para el primer paso del digestión a gran escala del vector de visualización de superficie de levadura pCTCON2.
| pCTCON2 (Sal que digierí) | 500 μL |
| 10x CutSmartBuffer | 37,5 μL |
| NheI-HF (NEB) | 15 μL (30 U) |
| BamHI-HF (NEB) | 15 μL (30 U) |
| H2O | hasta 875 μL |
Tabla 10: Condiciones para el segundo paso de la digestión a gran escala del vector de visualización de superficie de levadura pCTCON2.
9. Electroporación de EBY100 con ADN aleatorizado y vector linealizado
- Prepare las celdas de levadura EBY100 (ATCC, consulte la Tabla de materiales) extrajiéndolas de un caldo de glicerol 3 días antes de la electroporación. Dado que las células EBY100 no transformadas no contienen el vector de visualización de la superficie de levadura, utilice placas YPD de medios completos (Tabla 1). Incubar a 30 °C.
- Un día antes de la electroporación, inocular una sola colonia de EBY100 en 30 mL de medio YPD recién preparado (Tabla 1). Incubar durante la noche a 30 °C mientras se agita a 180 rpm.
NOTA: El medio YPD no debe tener más de 1 mes. - A la mañana siguiente, diluya el cultivo nocturno EBY100 a un diámetro exterior de600 de 0,2 en 100 ml de medio YPD e incube a 30 °C mientras agita a 180 rpm.
NOTA: Un volumen de cultivo de 50 mL es suficiente para 2 electroporaciones. Por lo general, 100 mL se preparan para tener suficientes celdas para los controles (solo celdas y celdas más vector). - Cuando se alcance un OD600 de 1,3-1,5, granule las células en tubos de 50 ml a 2000 x g durante 3 min y retire el sobrenadante.
- Vuelva a suspender el pellet en 25 mL de acetato de litio filtrado estéril de 100 mM (ver Tabla de Materiales). Añadir DTT filtrado estéril recién preparado (1 M) (ver Tabla de Materiales) hasta una concentración final de 10 mM.
NOTA: El volumen de acetato de litio debe ser la mitad del volumen original de YPD. Reduzca o aumente la escala si es necesario. El acetato de litio (100 mM) puede prepararse con antelación y almacenarse durante unos meses en RT, pero la culata de 1 M de TDT debe prepararse justo antes de su uso y mantenerse en hielo. - Incubar las células a 30 °C agitando durante 10 min.
- Centrifugar a 2000 x g durante 3 min, desechar el sobrenadante y colocar las células en hielo.
NOTA: Todos los pasos siguientes deben realizarse con hielo y con reactivos enfriados, cubetas y tubos de microcentrífuga. - Vuelva a suspender el pellet en 25 mL de centrífuga H2O. estéril fría a 2000 x g durante 3 min.
NOTA: El volumen H2O utilizado debe ser la mitad del volumen YPD original. - Volver a suspender en 350 μL de H2O estéril.
NOTA: El volumen total después de la resuspensión debe ser de 500 μL. Agregue más H2O si es necesario. - Prepare la biblioteca de ADN en paralelo: Coloque los tubos de microcentrífuga que contienen los insertos purificados con etanol en hielo y agregue 4 μg de vector pCTCON2 linealizado.
- Agregue 250 μL de las células al ADN y mezcle suavemente cortando. Transfiera a una cubeta de electroporación preenfriada de 2 mm y manténgala en hielo hasta la electroporación.
NOTA: Agregue controles (celdas solo sin ADN y celdas con vector linealizado solo sin inserto) para determinar el éxito de la electroporación. - Electroporar utilizando cubetas de electroporación de 2 mm (ver Tabla de Materiales) con las siguientes condiciones: protocolo de onda cuadrada, pulso único, 500 V, duración de pulso de 15 ms.
NOTA: Las caídas típicas son del 3% al 9%. Se pueden utilizar otros electroporadores, pero las condiciones indicadas están optimizadas para el instrumento utilizado en este estudio. - Rescate las células con 1 mL de YPD precalentado, transfiéralas a un tubo de microcentrífuga precalentado e incube sin agitar durante 1 h a 30 °C.
NOTA: Coloque el bloque calefactor junto al electroporador para asegurarse de que los tubos de recuperación permanezcan a la temperatura adecuada. - Placa de 100 μL de diluciones 10-3, 10-4, 10-5 y 10-6 de células en placas SD-CAA (ver Tabla 1) y permitir el crecimiento de transformantes a 30 °C.
NOTA: La diversidad teórica de la biblioteca se obtiene calculando hacia atrás el número de transformantes al volumen de cultivo original, aquí 1 mL. Normalmente, este protocolo de transformación produce aproximadamente 107-10 8 células transformadas. - Granular las células restantes a 2000 x g durante 3 min, resuspender en 200 mL SD-CAA e incubar a 30 °C con agitación a 180 rpm durante la noche. Las células transformadas que contienen la biblioteca aleatorizada de epPCR pueden pasarse en SD-CAA e inducirse para su expresión superficial en SG-CAA al día siguiente o almacenarse a 4 °C hasta su uso posterior. Congelar las existencias de glicerol de la biblioteca después de la epPCR después de determinar la diversidad. Dado que al día siguiente una fracción de las células seguirán siendo células no transformadas que se arrastraron en el procedimiento de transformación sin obtener un plásmido, se recomienda pasar al menos 20 veces la diversidad de la biblioteca en el primer paso de paso para compensar este efecto.
10. Secuenciación de las bibliotecas de levaduras después de varias rondas de selección
- Extraiga el ADN de la levadura realizando una minipreparación de levadura como se describió anteriormente.
- Electroporar células electrocompetentes comerciales de E. coli 10-beta (ver Tabla de Materiales) con el ADN aislado.
- Coloque las cubetas de electroporación (1 mm, ver Tabla de Materiales) y los tubos de microcentrífuga sobre hielo. Precalentar tubos de microcentrífuga con 950 μL de medio de crecimiento a 37 °C para recuperar las células después de la electroporación.
- Descongele las células en hielo y mezcle moviendo el tubo suavemente. Transfiera 50 μL de las células a un tubo de microcentrífuga refrigerado. Agregue 1 μL del ADN de la minipreparación de levadura.
- Transfiera con cuidado la mezcla de célula y ADN a una cubeta de electroporación refrigerada.
NOTA: Evite las burbujas y asegúrese de que las celdas se depositen en el fondo de la cubeta. - Electroporar con un dispositivo de electroporación utilizando las siguientes condiciones: 2,0 kV; 200 Ω; 25 μF.
NOTA: Las constantes de tiempo típicas son de 4,8 a 5,1 ms. Se pueden utilizar otros electroporadores, pero las condiciones indicadas están optimizadas para el instrumento utilizado en este estudio. - Añada inmediatamente 950 μL de medio de crecimiento precalentado a 37 °C (véase la tabla de materiales) a las cubetas, mezcle suavemente y vuelva a pasar a los tubos de microcentrífuga calientes.
NOTA: Coloque el bloque calefactor junto al electroporador para asegurarse de que los tubos de recuperación permanezcan a la temperatura adecuada. - Incubar durante 40 min a 37 °C y 300 rpm. Coloque 10 μL, 100 μL y el resto de las células (después de la centrifugación) en una placa LB selectiva precalentada (ver Tabla 1). Incubar las placas durante la noche a 37 °C.
NOTA: Para el vector pCTCON2, la ampicilina se utiliza para la selección de antibióticos. - Prepare una placa copiadora con agar LB y el antibiótico requerido (aquí se usa ampicilina): pipetee 200 μL de agar LB con 100 μg/mL de ampicilina en cada pocillo de una placa de 96 pocillos y deje que se solidifique.
- Prepare una placa de secuenciación de 96 pocillos con 50 μL de LB con 100 μg/mL de ampicilina por pocillo.
- Elija una sola colonia de las placas y primero inocule la placa de copia y el mismo pocillo de la placa de secuenciación.
NOTA: La placa de copia garantiza que los plásmidos deseados se puedan obtener fácilmente inoculando un cultivo líquido y realizando una minipreparación. - Incubar la placa de copia durante la noche a 37 °C y luego almacenarla a 4 °C.
- Incubar la placa de secuenciación durante 3-4 h a 37 °C mientras se agita (180 rpm) cubierta con un plástico o papel de aluminio.
- Selle la placa de secuenciación y envíela para su secuenciación junto con 500 μL de cebador de secuenciación pCTCON2 de 10 μM (5'-CGTTTGTCAGTAATTGCGGTTCTC).
- Para el análisis de las secuencias, traduzca las secuencias de ADN obtenidas a secuencias de aminoácidos utilizando un programa adecuado de su elección, por ejemplo, EMBOSS Transeq38. A continuación, utilice la Alineación de Secuencia Múltiple (MUSCLE)39 o una herramienta de alineación similar para alinear las secuencias variantes entre sí y con la proteína parental de tipo salvaje.
NOTA: Al realizar la secuenciación de bibliotecas de diferentes rondas de selección (por ejemplo, después de una ronda anterior de selección, así como después de rondas adicionales de selección con concentraciones de antígeno más bajas para enriquecer las variantes de alta afinidad), es posible identificar diferentes variantes. Esto depende en gran medida de la rigurosidad de las puertas aplicadas durante las rondas de clasificación y, por lo tanto, de la diversidad de las bibliotecas.
Resultados
La biblioteca G4 se seleccionó frente al antígeno hRBP4 unido al fármaco de molécula pequeña A1120. La tinción de las bibliotecas para la clasificación por citometría de flujo se realizó como se describe en el Método 6, y la estrategia de compuerta aplicada se muestra en la Figura 2A. Una puerta inicial incluyó todas las células en función de la morfología de la célula, y la segunda puerta (histograma de FSC-Width) mostró una estrategia de compuerta estricta que se aplicó para seleccionar células individuales y eliminar los agregados de células. La tercera y última puerta mostró la visualización de variantes proteicas (eje x) frente a la unión de antígenos (eje y). Se clasificaron las células de levadura que mostraban señales de visualización y unión. Es importante destacar que la puerta de clasificación se configuró de manera estricta para enriquecer los dominios de enlace con alta señal de enlace y, por lo tanto, alta afinidad. Esta selección rigurosa produjo un enriquecimiento de las células de levadura que se unen específicamente al antígeno objetivo a lo largo de la campaña de selección (Figura 2B). En rondas de clasificación por citometría de flujo posteriores, la concentración de antígeno disminuyó 10 veces (de 100 nM a 10 nM). Por lo tanto, la señal de unión general se redujo, y solo los aglutinantes con una alta afinidad seguían siendo detectables y clasificados (Figura 2C).
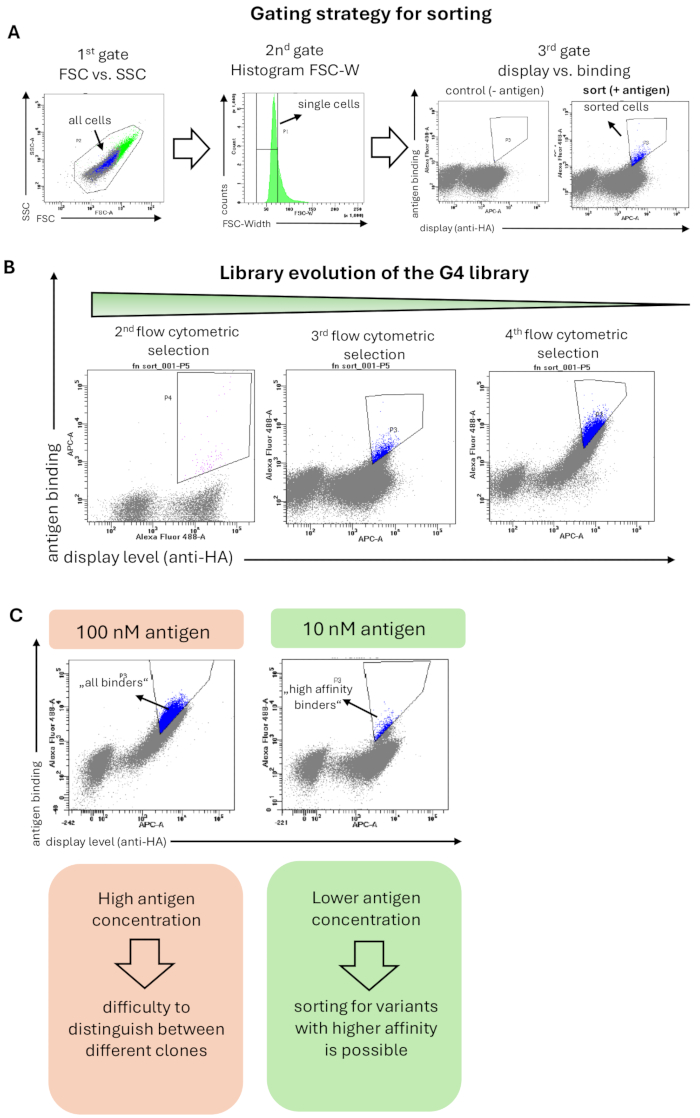
Figura 2: Resultados representativos de una selección de visualización de la superficie de levadura de la biblioteca G4 basada en Fn3 para unirse al antígeno (hRBP4 en presencia de A1120). (A) La estrategia general de compuerta para la clasificación de bibliotecas de levadura. La primera puerta (FSC vs. SSC) es seleccionar todas las células de levadura y excluir los eventos de dispersión; la segunda puerta (histograma de FSC-W) tiene como objetivo eliminar los agregados celulares y seleccionar solo células de levadura individuales. La tercera puerta traza el nivel de visualización de la superficie (detección de la etiqueta HA- o c-myc) frente a la unión al antígeno (aquí hRBP4 en presencia de 5 μM A1120, detectado por el anticuerpo anti-His). Además, la biblioteca se tiñó solo con anticuerpos secundarios (sin antígeno), donde no se espera unión al antígeno. Las celdas ordenadas se resaltan en azul. (B) Evolución de la biblioteca G4 a lo largo de 3 rondas de clasificación por citometría de flujo. Se puede observar un enriquecimiento de la población vinculante con cada ronda de selección. (C) El uso de concentraciones de antígeno más bajas permite la selección de variantes de proteínas con una mayor afinidad hacia el antígeno objetivo. Al reducir la concentración de antígeno (en este caso hRBP4) 10 veces, aparecen diferentes diagonales, lo que indica la presencia de clones con mayor afinidad (células ordenadas, azul) o menor. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
La visualización de la superficie de la levadura se ha convertido en uno de los métodos clave utilizados en la ingeniería de proteínas. Aunque se emplea comúnmente para la ingeniería de la afinidad 1,18,40,41, la expresión/estabilidad 24,27,42,43 y la actividad28,44, otros usos como el mapeo de epítopos45,46 o la caracterización de los mutantes individuales en la superficie de las células de levadura9 también son posibles. En este protocolo, proporcionamos los pasos básicos para iniciar una campaña de selección de visualización de superficie de levadura, incluida la selección con perlas magnéticas y la clasificación por citometría de flujo, así como la diversificación de la biblioteca de levaduras por epPCR para la maduración por afinidad.
Un requisito esencial para las selecciones convencionales de la superficie de la levadura es la disponibilidad de proteínas solubles de calidad suficiente. Comenzar con una proteína diana bien plegada con alta pureza y un estado de oligomerización definido (es decir, la proteína monomérica solo debe estar presente como monómero) proporciona la tasa de éxito más alta para seleccionar una variante de proteína que se una al antígeno diana con alta afinidad. Una alternativa para las proteínas diana difíciles de expresar son las selecciones basadas en células, que presentan una estrategia razonable para eludir esta limitación47. Sin embargo, la visualización de la superficie de la levadura ofrece muchas ventajas, como la posibilidad de caracterizar las variantes de proteínas resultantes directamente en la superficie de la levadura sin la necesidad de realizar una clonación laboriosa y laboriosa que requiere mucho tiempo, la expresión en un formato soluble y la purificación de proteínas. Tanto la afinidad como la estabilidad de las variantes se pueden analizar directamente en la superficie de la levadura9.
En este protocolo, mostramos cómo se seleccionó la biblioteca G4 de variantes proteicas, más concretamente del10º dominio tipo III de la fibronectina humana, para unirse al antígeno hRBP4 en presencia de la pequeña molécula A1120. La combinación de la selección de perlas y la clasificación por citometría de flujo produjo un enriquecimiento de variantes, que mostraron una mayor unión al antígeno objetivo a lo largo de las rondas de selección (Figura 2B). Demostramos que el uso de concentraciones más bajas de antígeno permite la selección de variantes de proteínas de alta afinidad (Figura 2C). Por lo general, las afinidades que se pueden lograr con las selecciones de visualización de levadura están en el rango de nanomolares o incluso picomolares18. Las afinidades finales dependen del antígeno diana, el número de rondas de selección y maduración de la afinidad, el andamiaje de unión utilizado y la estrategia de compuerta aplicada. La caracterización de las variantes individuales de proteínas no se aborda en este protocolo, pero se explica en detalle en nuestro trabajo anterior9. Aunque la visualización de levadura se empleó originalmente para la ingeniería de fragmentos de anticuerpos como scFvs 1,40, el método también se ha utilizado ampliamente para proteínas no basadas en anticuerpos10.
En resumen, la visualización de la superficie de la levadura es una poderosa herramienta de ingeniería de proteínas que permite la generación de variantes de proteínas con propiedades novedosas o mejoradas, como la unión a casi cualquier proteína objetivo y/o una mayor estabilidad.
Divulgaciones
M.W.T. recibe financiación de Miltenyi Biotec. Todos los autores son inventores de solicitudes de patentes para tecnologías y proteínas modificadas que se desarrollaron mediante el uso de la visualización de la superficie de la levadura.
Agradecimientos
Este trabajo fue apoyado por el Fondo Austriaco para la Ciencia (Proyecto FWF W1224 - Programa de Doctorado en Tecnología Biomolecular de Proteínas - BioToP y Proyecto FWF ESP 465-B), el Ministerio Federal de Asuntos Digitales y Económicos de Austria, la Fundación Nacional para la Investigación, Tecnología y Desarrollo de Austria a la Asociación de Investigación Christian Doppler (Laboratorio Christian Doppler para Células CAR-T de Próxima Generación), y por donaciones privadas al St. Anna Children's Cancer Research Institute (Viena, Austria). E.S. es beneficiaria de una beca DOC de la Academia Austriaca de Ciencias en el Instituto de Investigación del Cáncer Infantil de St. Anna. Las figuras fueron creadas con BioRender.com.
Materiales
| Name | Company | Catalog Number | Comments |
| 10-beta electrocompetent E. coli | NEB | C3020K | |
| Agar-Agar, Kobe I | Carl Roth | 5210.5 | |
| Ampicillin sodium salt | Carl Roth | K029.2 | |
| Anti-c-myc antibody, clone 9E10, AF488 | Invitrogen | MA1-980-A488 (Thermo Fisher) | |
| Anti-c-myc antibody, clone 9E10, AF647 | Invitrogen | MA1-980-A647 (Thermo Fisher) | |
| Anti-HA antibody, clone 16B12, AF488 | BioLegend | 901509 (Biozym) | |
| Anti-HA antibody, clone 16B12, AF647 | BioLegend | 682404 (Biozym) | |
| BamHI-HF | NEB | R3136S | |
| Bovine serum albumin, cold ethanol fraction | Sigma-Aldrich | A4503 | |
| Citric acid monohydrate | Sigma-Aldrich | C1909 | |
| D-Galactose | Carl Roth | 4987.2 | |
| D-Glucose | Sigma-Aldrich | G8270 | |
| Difco yeast nitrogen base | Becton Dickinson (BD) | 291940 | |
| Di-Sodium hydrogen phosphate heptahydrate | Carl Roth | X987.3 | |
| DL-Dithiothreitol | Sigma-Aldrich | D0632 | |
| D-Sorbitol | Carl Roth | 6213.1 | |
| Dulbecco’s phosphate buffered saline (10x) | Thermo Scientific | 14190169 | |
| Dynabeads Biotin Binder | Invitrogen | 11047 (Fisher Scientific) | |
| DynaMag-2 Magnet | Thermo Fisher | 12321D | |
| EBY100 | ATCC | MYA-4941 | |
| Electroporation cuvette 1 mm (for E.coli) | VWR | 732-1135 | |
| Electroporation cuvette 2 mm (for yeast) | VWR | 732-1136 | |
| Ethanol absolute | MERCK | 1070172511 | |
| GeneMorph II Random Mutagenesis Kit | Agilent Technologies | 200550 | |
| Gibco Bacto Casamino Acids | Becton Dickinson (BD) | 223120 | |
| Glycerol | AppliChem | 131339.1211 | |
| LE agarose | Biozym | 840004 | |
| Lithium acetate dihydrate | Sigma-Aldrich | L4158 | |
| Monarch DNA Gel Extraction Kit | NEB | T1020S | |
| Monarch PCR & DNA Cleanup Kit | NEB | T1030S | |
| Multifuge 1S-R | Heraeus | ||
| NheI-HF | NEB | R3131S | |
| Outgrowth medium | NEB | B9035S | |
| pCTCON2 | Addgene | #41843 | |
| Penicillin G sodium salt | Sigma-Aldrich | P3032 | |
| Penta-His antibody, AF488 | Qiagen | 35310 | |
| Penta-His antibody, AF647 | Qiagen | 35370 | |
| Peptone ex casein tryptically digested | Carl Roth | 8986.3 | |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491S | |
| SaII-HF | NEB | R3138S | |
| Sodium acetate | Sigma-Aldrich | S8750-1KG | |
| Sodium chloride | Carl Roth | 3957.2 | |
| Sodium dihydrogen phosphate monohydrate | Carl Roth | K300.2 | |
| Steritop threaded bottle top filter | MERCK | S2GPT01RE | |
| Streptavidin, AF488 | Invitrogen | S32354 (Thermo Fisher) | |
| Streptavidin, AF647 | Invitrogen | S32357 (Thermo Fisher) | |
| Streptomycin sulfate | Sigma-Aldrich | S6501 | |
| Tri-Sodium citrate dihydrate | Carl Roth | 4088.1 | |
| UV-Vis spectrophotometer | Agilent | 8453 | |
| Yeast extract, micro-granulated | Carl Roth | 2904.4 | |
| Zymoprep Yeast Plasmid Miniprep II | Zymo Research | D2004 |
Referencias
- Boder, E. T., Wittrup, K. D. Yeast surface display for screening combinatorial polypeptide libraries. Nat Biotechnol. 15 (6), 553-557 (1997).
- Scott, J. K., Smith, G. P. Searching for peptide ligands with an epitope library. Science. 249 (4967), 386-390 (1990).
- Smith, G. P. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 228 (4705), 1315-1317 (1985).
- Schaffitzel, C., Hanes, J., Jermutus, L., Pluckthun, A. Ribosome display: an in vitro method for selection and evolution of antibodies from libraries. J Immunol Methods. 231 (1-2), 119-135 (1999).
- Ho, M., Nagata, S., Pastan, I. Isolation of anti-CD22 Fv with high affinity by Fv display on human cells. Proc Natl Acad Sci U S A. 103 (25), 9637-9642 (2006).
- Wagner, A., et al. Identification of activating mutations in the transmembrane and extracellular domains of EGFR. Biochemistry. 61 (19), 2049-2062 (2022).
- Wagner, A., et al. PhosphoFlowSeq - A high-throughput kinase activity assay for screening drug resistance mutations in EGFR. J Mol Biol. 433 (22), 167210(2021).
- Cherf, G. M., Cochran, J. R. Applications of yeast surface display for protein engineering. Methods Mol Biol. 1319, 155-175 (2015).
- Zajc, C. U., Teufl, M., Traxlmayr, M. W. Affinity and stability analysis of yeast displayed proteins. Methods Mol Biol. 2491, 155-173 (2022).
- Gai, S. A., Wittrup, K. D. Yeast surface display for protein engineering and characterization. Curr Opin Struct Biol. 17 (4), 467-473 (2007).
- Orr, B. A., Carr, L. M., Wittrup, K. D., Roy, E. J., Kranz, D. M. Rapid method for measuring ScFv thermal stability by yeast surface display. Biotechnol Prog. 19 (2), 631-638 (2003).
- Teufl, M., Zajc, C. U., Traxlmayr, M. W. Engineering strategies to overcome the stability-function trade-off in proteins. ACS Synth Biol. 11 (3), 1030-1039 (2022).
- Wozniak-Knopp, G., et al. Introducing antigen-binding sites in structural loops of immunoglobulin constant domains: Fc fragments with engineered HER2/neu-binding sites and antibody properties. Protein Eng Des Sel. 23 (4), 289-297 (2010).
- Traxlmayr, M. W., et al. Directed evolution of stabilized IgG1-Fc scaffolds by application of strong heat shock to libraries displayed on yeast. Biochim Biophys Acta. 1824 (4), 542-549 (2012).
- Traxlmayr, M. W., et al. Directed evolution of Her2/neu-binding IgG1-Fc for improved stability and resistance to aggregation by using yeast surface display. Protein Eng Des Sel. 26 (4), 255-265 (2013).
- Hasenhindl, C., et al. Stability assessment on a library scale: a rapid method for the evaluation of the commutability and insertion of residues in C-terminal loops of the CH3 domains of IgG1-Fc. Protein Eng Des Sel. 26 (10), 675-682 (2013).
- Koide, A., Wojcik, J., Gilbreth, R. N., Hoey, R. J., Koide, S. Teaching an old scaffold new tricks: monobodies constructed using alternative surfaces of the FN3 scaffold. J Mol Biol. 415 (2), 393-405 (2012).
- Hackel, B. J., Kapila, A., Wittrup, K. D. Picomolar affinity fibronectin domains engineered utilizing loop length diversity, recursive mutagenesis, and loop shuffling. J Mol Biol. 381 (5), 1238-1252 (2008).
- Traxlmayr, M. W., et al. Strong enrichment of aromatic residues in binding sites from a charge-neutralized hyperthermostable Sso7d scaffold library. J Biol Chem. 291 (43), 22496-22508 (2016).
- Zajc, C. U., et al. A conformation-specific ON-switch for controlling CAR T cells with an orally available drug. Proc Natl Acad Sci U S A. 117 (26), 14926-14935 (2020).
- Moore, S. J., Cochran, J. R. Engineering knottins as novel binding agents. Methods Enzymol. 503, 223-251 (2012).
- Hackel, B. J., Ackerman, M. E., Howland, S. W., Wittrup, K. D. Stability and CDR composition biases enrich binder functionality landscapes. J Mol Biol. 401 (1), 84-96 (2010).
- Hackel, B. J., Wittrup, K. D. The full amino acid repertoire is superior to serine/tyrosine for selection of high affinity immunoglobulin G binders from the fibronectin scaffold. Protein Eng Des Sel. 23 (4), 211-219 (2010).
- Laurent, E., et al. Directed evolution of stabilized monomeric CD19 for monovalent CAR interaction studies and monitoring of CAR-T cell patients. ACS Synth Biol. 10 (5), 1184-1198 (2021).
- Julian, M. C., et al. Co-evolution of affinity and stability of grafted amyloid-motif domain antibodies. Protein Eng Des Sel. 28 (10), 339-350 (2015).
- Cochran, J. R., Kim, Y. S., Lippow, S. M., Rao, B., Wittrup, K. D. Improved mutants from directed evolution are biased to orthologous substitutions. Protein Eng Des Sel. 19 (6), 245-253 (2006).
- Kieke, M. C., et al. High affinity T cell receptors from yeast display libraries block T cell activation by superantigens. J Mol Biol. 307 (5), 1305-1315 (2001).
- Lipovsek, D., et al. Selection of horseradish peroxidase variants with enhanced enantioselectivity by yeast surface display. Chem Biol. 14 (10), 1176-1185 (2007).
- Fushimi, T., et al. Mutant firefly luciferases with improved specific activity and dATP discrimination constructed by yeast cell surface engineering. Appl Microbiol Biotechnol. 97 (9), 4003-4011 (2013).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2015).
- Park, S. W., Lee, D. S., Kim, Y. S. Engineering a U-box of E3 ligase E4B through yeast surface display-based functional screening generates a variant with enhanced ubiquitin ligase activity. Biochem Biophys Res Commun. 612, 147-153 (2022).
- Schutz, M., et al. Directed evolution of G protein-coupled receptors in yeast for higher functional production in eukaryotic expression hosts. Sci Rep. 6, 21508(2016).
- Angelini, A., et al. Protein engineering and selection using yeast surface display. Methods Mol Biol. 1319, 3-36 (2015).
- Chen, T. F., de Picciotto, S., Hackel, B. J., Wittrup, K. D. Engineering fibronectin-based binding proteins by yeast surface display. Methods Enzymol. 523, 303-326 (2013).
- Shusta, E. V., Holler, P. D., Kieke, M. C., Kranz, D. M., Wittrup, K. D. Directed evolution of a stable scaffold for T-cell receptor engineering. Nat Biotechnol. 18 (7), 754-759 (2000).
- Traxlmayr, M. W., Obinger, C. Directed evolution of proteins for increased stability and expression using yeast display. Arch Biochem Biophys. 526 (2), 174-180 (2012).
- Traxlmayr, M. W., Shusta, E. V. Directed evolution of protein thermal stability using yeast surface display. Methods Mol Biol. 1575, 45-65 (2017).
- Madeira, F., et al. The EMBL-EBI Job Dispatcher sequence analysis tools framework in 2024. Nucleic Acids Res. 52 (W1), W521-W525 (2024).
- Edgar, R. C. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Res. 32 (5), 1792-1797 (2004).
- Chao, G., et al. Isolating and engineering human antibodies using yeast surface display. Nat Protoc. 1 (2), 755-768 (2006).
- VanAntwerp, J. J., Wittrup, K. D. Fine affinity discrimination by yeast surface display and flow cytometry. Biotechnol Prog. 16 (1), 31-37 (2000).
- McConnell, A. D., et al. A general approach to antibody thermostabilization. MAbs. 6 (5), 1274-1282 (2014).
- Pavoor, T. V., Wheasler, J. A., Kamat, V., Shusta, E. V. An enhanced approach for engineering thermally stable proteins using yeast display. Protein Eng Des Sel. 25 (10), 625-630 (2012).
- Chen, I., Dorr, B. M., Liu, D. R. A general strategy for the evolution of bond-forming enzymes using yeast display. Proc Natl Acad Sci U S A. 108 (28), 11399-11404 (2011).
- Traxlmayr, M. W., et al. Construction of a stability landscape of the CH3 domain of human IgG1 by combining directed evolution with high throughput sequencing. J Mol Biol. 423 (3), 397-412 (2012).
- Chao, G., Cochran, J. R., Wittrup, K. D. Fine epitope mapping of anti-epidermal growth factor receptor antibodies through random mutagenesis and yeast surface display. J Mol Biol. 342 (2), 539-550 (2004).
- Stern, L. A., et al. Cellular-based selections aid yeast-display discovery of genuine cell-binding ligands: Targeting oncology vascular biomarker CD276. ACS Comb Sci. 21 (3), 207-222 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados